JoVE 비디오를 활용하시려면 도서관을 통한 기관 구독이 필요합니다. 전체 비디오를 보시려면 로그인하거나 무료 트라이얼을 시작하세요.
Method Article
마이크로위성 DNA 게노티핑 및 유동 세포분석분석분석포르민고정파라핀내장화증모금조직
요약
수다디형 두더지(Hydatidiform moles)는 어금니 게놈에 대한 부모의 기여와 형태학적 특징에 따라 분류될 수 있는 이기종 빈치학을 가진 비정상적인 인간 임신입니다. 여기서, 멀티플렉스 마이크로위성 DNA 의전성 및 포르말린 파라핀 내장 어금니 조직의 유동 세포분석프로토콜은 결과의 해석 및 통합과 함께 상세히 기술된다.
초록
수다디폼 두더지(HM)는 과도한 트로포블라아제 증식과 비정상적인 배아 발달을 특징으로 하는 비정상적인 인간 임신입니다. 현미경 형태 학적 평가, 완전한 HM (CHM) 및 부분 HM (PHM)에 따라 HM의 두 가지 유형이 있습니다. 이들은 어금니 게놈에 대한 부모의 기여에 기초하여 더 세분화될 수 있습니다. 형태학 및 유전자형 분석에 의한 HM의 이러한 특성화는 환자 관리및 이 흥미로운 병리학의 근본적인 이해에 매우 중요합니다. HM의 형태학적 분석은 광범위한 관찰자 간 가변성의 영향을 받으며 HM을 CHM 및 PHM으로 정확하게 분류하고 수경 비 어금니 낙태와 구별하기에 충분하지 않다는 것이 잘 문서화되어 있습니다. Genotyping 분석은 주로 포르말린 고정 파라핀 임베디드 (FFPE) 개념작용의 제품에서 DNA 와 조직에 수행되며, 이는 최적의 품질보다 적고 결과적으로 잘못된 결론으로 이어질 수 있습니다. 이 문서에서는, FFPE 어금니 조직의 다중 지질 분석 및 유동 세포 분석 분석을 위한 상세한 프로토콜은, 이 방법의 결과의 해석과 더불어, 그들의 문제 해결 및 형태학적 평가와의 통합과 더불어 제공됩니다 , p57KIP2 면역성 화학 및 현장 혼성화 (FISH)에서형광은 정확하고 견고한 진단에 도달합니다. 여기에서 저자는 약 400개의 개념 작용 제품을 분석하여 지난 10년 동안 배운 방법과 교훈을 공유합니다.
서문
수다디폼 두더지(HM)는 비정상적인 배아 발달, 트로포블라스트의 과증식, 융모융물(CV)의 수경 변성을 특징으로 하는 비정상적인 인간 임신입니다. 역사적으로, HM은 형태학적 평가1에기초하여 완전한 HM(CHM) 및 부분 HM(PHM)의 두 가지 유형으로 나뉘었다. 그러나, 형태학적 평가만으로는 HM을 두 개의 아류형(CHM 및 PHM)으로 분류하고 이를 비어치 유산2,3,4와구별하기에 충분하지 않은 것으로 나타났다.
CHM과 PHM은 악성 종양에 다른 성향을 가지고 있기 때문에, 환자에게 적절한 후속 및 관리를 제공하기 위해 HM의 유전자 형 유형을 정확하게 결정하는 것이 중요합니다. 따라서 지난 수십 년 동안 어금니 조직에 대한 부모의 기여를 확인하고 HM의 올바른 분류에 도달하기 위해 여러 가지 방법론이 개발되고 발전했습니다. 여기에는 핵형 분석, 염색체 밴딩 다형성, 인간 백혈구 항원 (HLA) 혈청학적 입력, 제한 단편 길이 다형성, 가변 수의 탠덤 반복, 마이크로 위성 유전자 변형, 유세포 분석 및 p57이 포함됩니다. KIP2 면역 화학. 이것은 그들의 게놈에 부모 기여에 근거를 둔 HM 개념의 정확한 세분화를 허용했습니다, 다음과 같이: CHM, 이는 이월 및 유전적 단정자 또는 디플로이드 안드로이노믹 분산증, 및 PHM, 이는 삼엽충, 99%에서 분산성 및 케이스5,6,7,8의1 %에서 단정자 . 게다가, 지난 2 년간 에서 나타난 HM의 또 다른 genotypic 모형이 있습니다, 이는 이형 양부모입니다. 후자는 주로 재발하고 단일 가족 구성원 (심플렉스 케이스) 또는 적어도 2 개의 가족 구성원 (가족 케이스)에 영향을 미칠 수 있습니다. 이들 디플로이드 두더지들은 주로 환자9,10,11,12에서 NLRP7 또는 KHDC3L의 열성 돌연변이에 의해 유발된다. NLRP7에서 열성 돌연변이를 가진 환자에서 디플로이드 양부모 HM은 형태학적 분석에 의해 CHM 또는 PHM으로 진단될 수 있으며 이는 환자13,14에서돌연변이의 중증도와 연관된 것으로 보인다. 그들의 유전자형에 따른 HM의 분류 이외에, 몇몇 유전형질 방법의 소개 그리고 사용은 이누엽 이완 양부모 개념과 같은 비 어금니 유산에서 각종 대구물 엔티티의 구별을 허용하고 개념의 다른 유형5,15. 이러한 개념은 HM의 일부 형태학적 특징을 어느 정도 모방하는 trophoblast 증식 및 비정상적인 융모 형태를 가질 수 있습니다.
이 문서의 목적은 포르말린 고정 파라핀 임베디드 (FFPE) 조직의 다중 지질 분석 및 유세포 분석에 대한 자세한 프로토콜을 제공하고, 이러한 방법의 결과와 다른 방법과의 통합에 대한 포괄적 인 분석을 제공하는 것입니다. 어금니 조직의 정확하고 결정적인 진단.
프로토콜
이 연구 는 McGill 기관 검토 위원회에 의해 승인 되었다. 모든 환자는 연구 결과에 참여하고 다양한 병리학 부에서 검색 된 개념작용 (POC)의 FFPE 제품을 가지고 서면 동의를 제공했습니다.
참고: 유세포 분석에 의한 지질형 분석 및 계략 측정을 위한 몇 가지 방법이 있지만, 여기에 제공된 프로토콜은 각각에 대해 하나의 플랫폼을 사용하여 하나의 분석 방법을 설명합니다.
1. 게놈
-
최고의 FFPE 블록 선택
- 개념작용의 각 FFPE 제품 (POC)를 위해, 현미경 검사법에 의하여 형태학적 평가를 위해 섹션 1.2 및 1.3에 기술된 대로 4 μm 두께의 헤마톡실린 및 에오신 (H&E) 염색 된 단면을 준비하십시오.
- H&E 슬라이드와 가벼운 현미경을 사용하여 가장 많은 양의 융모 융모(CV)를 가진 FFPE 블록을 선택하고, 가능하다면 모계 조직과 섞이지 않고 CV가 분리된 블록을 선택합니다.
-
단면화
- 선택한 블록을 얼음 위에 15분 동안 놓아 서 단면을 용이하게 합니다.
- 미세한 형태학적 평가를 위해 두께가 4 μm이고 DNA 추출을 위해 두께가 10 μm인 절편을 자르도록 마이크로토메를 조정합니다.
- 콜드 블록을 마이크로톤에 놓고 H&E 염색을 위해 각 블록에서 한 부분을 자르고 DNA 추출을 위해 블록의 CV 양에 따라 선택한 블록에서 10-30 개의 섹션을 잘라냅니다.
참고: CV로 가득 찬 블록의 경우 10개의 섹션이 DNA 추출에 충분합니다. 블록의 약 10 %만이 CV를 포함하고 나머지는 모성 조직인 경우 충분한 양의 DNA를 보장하기 위해 20-30 섹션이 필요합니다. - 집게를 사용하여 각 섹션을 45 °C 수조로 옮김하십시오. 수조에서 연필을 사용하여 샘플 식별 번호로 이전에 표시된 양전하슬라이드(재료 표)로섹션을 선택합니다.
- 단면이 들어 있는 슬라이드를 65°C의 오븐에 놓아 섹션이 슬라이드에 부착되도록 합니다. H&E슬라이드를 오븐에 25분 동안 보관하십시오.
참고: 배양 시간이 짧을수록 조직이 슬라이드에 약간 덜 부착되어 결과적으로 모계 조직의 제거를 용이하게합니다.
-
H&E 염색
- 슬라이드가 실온(10분)까지 식힙니다.
- 시약 준비
- 어신 Y 작업 솔루션 준비 (0.25%) 표 1에따라 . 잘 섞어서 실온에서 보관하세요.
- 물에 헤마톡슬린 5x의 재고 용액을 희석하여 작업 헤마톡실린 용액을 준비하십시오 (즉, 80 mL의 물과 헤마톡슬린 20 mL을 혼합하십시오).
참고: 저장을 위한 호일에 헤마톡실린의 스톡 용액을 감싸.
- 표 2에따라 연기 후드 아래에 올바른 시약으로 염색 항아리를 준비하십시오.
- 표 2에따라 슬라이드를 적절한 염색 용기에 적어 침지하여 H&E 염색을 수행합니다.
- 형태 학적 분석을 위해 4 μm 섹션을 장착 매체와 유리 커버 슬립(재료 표)와커버 슬립을 장착합니다.
참고: 지질형 지질 질에 대한 10 μm 섹션은 커버 미끄러져서는 안됩니다. - 독성 자일렌 냄새가 사라지도록 10 μm 섹션을 연기 후드 아래에 최소 3시간 동안 둡니다.
주의 사항: 모든 염색 단계는 연기 후드에서 수행해야합니다. 자일렌 냄새는 독성이 있기 때문에 자일렌 제품은 항상 후드 아래에 보관해야합니다. 또한 자일렌과 헤마톡실린은 특수 용기에 버려야합니다. 이러한 용기가 가득 차면 실험실의 안전 조직에서 권장하는 대로 폐기해야 합니다.
| 시약 | 수량 |
| 이신 Y 재고 솔루션 (1%) | 250 mL |
| 80% 에탄올 | 750 mL |
| 빙하 아세트산 (농축) | 5 mL |
표 1: 에신 Y 작업 용액 (0.25%) 준비.
| 시약 사용(쓰레기통당 100mL) | 기간 |
| 1) 자일렌 | 약 5분 |
| 2) 자일렌 | 약 5분 |
| 3) 100% 에탄올 | 2분 |
| 4) 95% 에탄올 | 2분 |
| 5) 70% 에탄올 | 2분 |
| 6) 50% 에탄올 | 2분 |
| 7) 증류수 | 약 5분 |
| 8) 헤마톡실린 | 4분 |
| 9) 증류수 | 약 5분 |
| 10) 에오신 | 1분 |
| 11) 95% 에탄올 | 약 5분 |
| 12) 100% 에탄올 | 약 5분 |
| 13) 자일렌 | 약 5분 |
| 14) 자일렌 | 약 5분 |
표 2: H&E 염색 프로토콜의 시약 및 지속 시간.
-
이력서의 격리
- 가벼운 입체 현미경으로, 집게와 물축 종이 물티슈(재료표)의작은 조각을 사용하여 H&E 염색 된 10 μm 두께의 섹션에서 원치 않는 모성 조직을 긁어 냅니다.
참고: 최종 목표는 슬라이드에 CV 또는 태아 막 (존재 할 때)만 유지하여 다른 모든 조직을 제거하는 것입니다. 이 단계는 세부 사항에 세심한주의가 필요하기 때문에 블록에 따라 많은 시간과 인내심이 필요할 수 있습니다. - 두 번째 사람이 청소 후 슬라이드를 다시 확인하여 모성 조직이 없는지 확인하십시오.
- 청소 슬라이드의 사진을 찍거나 데이터 해석을 돕기 위해 다음을 문서화하십시오 : 1) 조직이 청소하기 어려웠는지, 출혈성 인지 또는 매우 깨끗한지 여부, 2) 사용되는 섹션의 수 및 3) 청소 된 조직의 대략적인 양.
참고: 그림 1은 청소하기 쉬운 슬라이드의 예를 제공합니다. 대략 이 양의 CV를 포함하는 블록의 경우, 10개의 섹션은 DNA 추출에 충분합니다. 그림 2의 슬라이드에는 모계 조직과 혼합된 CV가 거의 없기 때문에 청소하기가 매우 어렵고 시간이 많이 걸립니다. 대략 이 양의 CV를 포함하는 블록의 경우 DNA 추출을 위해 30개의 섹션이 필요합니다. - 종이 물티슈의 작은 축축한 조각을 사용하여 이력서를 수집합니다. 집게를 사용하여 축축한 종이 물티슈에서 작은 조각을 찢어 이력서를 수집하는 데 사용합니다.
- 부착된 이력서가 부착된 종이 물티슈 조각을 라벨이 붙은 1.5mL 튜브에 넣습니다.
- 이 단계에서 사용되는 종이 물티슈의 양을 너무 많이 최소화하면 DNA 추출 컬럼이 막히게 되어 결과적으로 수집된 DNA의 최종 양을 줄일 수 있습니다. 평균적으로 샘플당 7개 미만의 작은 종이 물티슈를 사용하는 것을 목표로 합니다. 대량의 CV가 존재하기 때문에 이것이 불가능한 경우, 추출을 용이하게하기 위해 샘플을 두 개의 튜브로 분할하십시오.
- 가벼운 입체 현미경으로, 집게와 물축 종이 물티슈(재료표)의작은 조각을 사용하여 H&E 염색 된 10 μm 두께의 섹션에서 원치 않는 모성 조직을 긁어 냅니다.

그림 1: 유질이 없는 것을 위한 대표 슬라이드. 상단: 모성 조직이 없어지기 위해 "청소"해야 하는 슬라이드입니다. 아래쪽: 청소한 후 표시된 동일한 슬라이드에 DNA 추출을 위한 CV 외에는 아무것도 포함되어 없습니다. 이 그림의 더 큰 버전을 보려면 여기를 클릭하십시오.
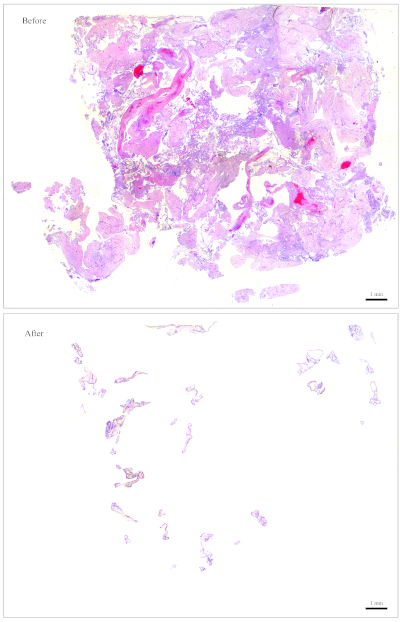
그림 2: 헤노티핑을 위한 대표 슬라이드. 상단: 모성 조직이 없어지기 위해 "청소"해야 하는 슬라이드입니다. 아래쪽: 청소한 후 표시된 동일한 슬라이드에 DNA 추출을 위한 CV 외에는 아무것도 포함되어 없습니다. 이 그림의 더 큰 버전을 보려면 여기를 클릭하십시오.
- FFPE키트(물자 표)로부터DNA 추출 프로토콜을 따라 DNA 추출을 수행한다.
참고: 일부 키트는 최종 용출을 위해 15−20 μL의 용출 버퍼를 사용하는 것이 좋습니다. 경험에서 15 μL의 용출 버퍼가있는 용출은 대부분의 샘플에 적합합니다. 희석은 필요에 따라 스톡 DNA로부터 제조될 수 있다.
-
DNA 정량화
- 실험실 분광광도계 장치를 사용하여 DNA 1 μL을 로드하고 정량화를 위해 260 nm에서 흡광도를 측정합니다.
- 2% 아가로즈 겔에 DNA 1 μL을 적재하고 질적 평가를 위해 80-100V의 전압으로 겔 전기 동동을 실행합니다.
- 단계 1.6.1 및 1.6.2의 결과에 기초하여, 멀티플렉스 숏탠덤 반복(STR) 중합효소 연쇄 반응(PCR) 증폭에 사용되는 DNA의 부피를 선택한다. 다음에 오는 PCR 증폭에 DNA의 최소 1000 ng를 사용하는 것을 목표로한다.
참고: 그림 3은 DNA의 농도(분광광도계 결과에 기초)와 함께 겔의 대표적인 예와 다음에 오는 멀티플렉스 STR PCR에 권장되는 DNA 용액의 부피를 보여줍니다.
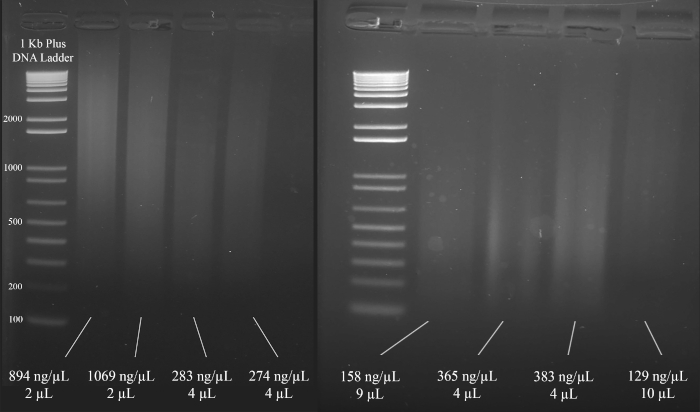
도 3: DNA 정량화를 위한 대표적인 겔. 분광광도계를 사용하여 측정된 각 DNA의 농도 및 멀티플렉스 PCR에 사용되는 양이 포함됩니다. 이 그림의 더 큰 버전을 보려면 여기를 클릭하십시오.
-
PCR 증폭
- 멀티플렉스 STR시스템(재료 표)을사용하여 형광 마이크로위성 지질검사를 수행합니다.
- 멀티플렉스 STR시스템(재료 표)을사용하여 PCR 증폭을 위해 도 4에 표시된 PCR 조건을 사용하십시오.
참고: D18S51, D21S11, TH01, D3S1358, 펜타 E, FGA, TPOX, D8S1179, vWA, 아멜로제닌, CSF1PO, D16S539, D7S820, D13S318, D13S318: 이 멀티플렉스 STR 시스템에서 사용된다.

도 4: 멀티플렉스 STR 시스템에 대한 PCR 사이클 조건. 이 그림의 더 큰 버전을 보려면 여기를 클릭하십시오.
-
모세관 전기 동공에 의해 PCR 제품을 해결합니다.
- 멀티플렉스 시스템의 내부 표준 레인의 0.5 μL에서 각 증폭 된 샘플의 1 μL과 고이온화된 포름아미드(재료 표)의9.5 μL을 일시 중단합니다.
- 모세관 전기동동계(표)를통해 시료를 실행하여 계측기 및 멀티플렉스 시스템의 염료 세트에 적합한 분리 매트릭스(재료표)를사용합니다.
- 데이터 분석
- DNA 단편 분석 소프트웨어로 데이터를 분석하고 POC 대열기를 부모 대열에 비교하여 기원을 결정합니다.
- 크기 표준을 설정합니다.
참고: 이를 통해 소프트웨어는 멀티플렉스 STR 시스템에 사용되는 사다리를 인식하고 사다리를 기반으로 앰플리톤에 베이스페어링을 할당할 수 있습니다. 다음 단계는 특정소프트웨어(재료 표)에대한 것이지만 다른 유형의 소프트웨어 설정에도 도움이 될 수 있습니다.- 소프트웨어를 엽니다. 새 프로젝트 시작을 클릭한 다음 새 크기 표준을클릭합니다.
- 크기 표준에 이름을 지정합니다(예: ABI_600).
- 새 크기 표준 정의를 입력함상자에 입력합니다: 60, 80, 100, 120, 140, 160, 180, 200, 225, 250, 275, 300, 325, 350, 375, 400, 450, 475, 500, 60, 60을 입력합니다. 그런 다음 크기 추가를 클릭합니다.
참고: 입력한 숫자는 오른쪽 상자 아래에 표시되며 현재 크기 표준 정의라는 이름입니다(그림 5참조). - 저장을 클릭합니다.
- 파일을 가져오고 분석하려면 파일 추가를클릭하고 분석할 fsa 파일을 선택합니다. 선택한 파일 추가를 클릭한 다음 확인을클릭합니다. 그런 다음 다음 단계를 수행합니다.
- 크기 표준 열을 찾아 ABI_600(또는 크기 표준에 지정된 이름 중 하나를 선택)을 선택합니다.
- 분석 방법에서 기본 크기 조정 - NPP를 클릭한 다음 녹색 분석 버튼을 클릭합니다.
- 이제 파일을 볼 준비가 되었습니다. 보기 옵션을 조정하여 원하는 대로 데이터를 봅니다.
- 문제 해결 - 분석 방법
참고: 소프트웨어가 피크를 식별하고 올바르게 정렬하지 못할 수 있습니다. 이것은 피크가 너무 낮거나 너무 높을 때 발생합니다. 다음 두 가지 분석 방법은 이를 수정할 수 있으며 샘플을 다시 테스트하기 전에 시도해야 합니다.- 높은 피크에 대한 분석 방법 1:
- 새 분석 방법을 클릭하고 높은 봉우리 (또는 개인 기본 설정에 따라 다른 이름)의 이름을 지정합니다.
- 분석 및 크기 조정을 위해 범위를 클릭한 다음 부분 범위를 클릭합니다. 그런 다음 시작 점 및 시작 크기에대해 100을 입력합니다.
- 중지 지점의경우 10,000을 입력합니다. 중지 크기를보려면 1000을 입력합니다.
- 그런 다음 최소 피크 높이를 클릭하고 색상의 피크 임계값이 다음과 같이되도록 숫자를 변경합니다: 파란색: 50; 녹색: 50; 노란색: 20; 빨간색: 100; 오렌지: 5000.
- 새 분석 방법을 저장합니다.
- 낮은 피크에 대한 분석 방법 2:
- 새 분석 방법을 클릭하고 낮은 봉우리 (또는 개인 기본 설정에 따라 다른 이름)의 이름을 지정합니다.
- 분석 및 크기 조정을 위해 범위를 클릭한 다음 부분 범위를 클릭합니다. 그런 다음 시작 점 및 시작 크기에대해 100을 입력합니다.
- 중지 지점의경우 10,000을 입력합니다. 중지 크기를보려면 1000을 입력합니다.
- 그런 다음 품질 플래그를 클릭하고 0.5에서 1로읽도록 패스 범위를 변경합니다. 0.0에서 0.0으로읽도록 낮은 품질 범위를 변경합니다. 선형성을 다음과 같은 것으로 가정변경: (bp) 100.0에서 (bp) 800.0.
- 새 분석 방법을 저장합니다.
참고: 이제 분석 방법에서 낮은 봉우리 또는 높은 피크를 선택한 다음 녹색 분석 버튼을 클릭하여 파일을 다시 분석할 수 있습니다.
- 높은 피크에 대한 분석 방법 1:

그림 5: 크기 표준 편집기표시 스크린샷입니다. 이 그림의 더 큰 버전을 보려면 여기를 클릭하십시오.
2. 유세포세포분석
-
이상적인 FFPE 블록 선택
- H&E 슬라이드와 가벼운 현미경을 사용하여 CV로 구성된 조직의 약 50-70%를 포함하는 FFPE 블록을 선택합니다.
참고: 도 6은 약 50% CV(섹션의 오른쪽 절반)와 50% 모계 조직(왼쪽 절반)으로 구성되기 때문에 유세포 분석에 적합한 블록의 대표적인 예입니다. 모계 조직의 존재는 디플로이드 피크에 대한 내부 통제 역할을하기 때문에 중요합니다. - 이상적인 양의 CV가 없는 블록의 경우 단면이 수행될 때 CV를 보강합니다. 이렇게 하려면 새로 절단된 섹션의 어느 면에 해당 H&E 슬라이드에 따라 더 많은 CV가 포함되어 있는지 확인합니다. 이를 바탕으로, 이력서를 보강하기 위해 폐기해야 하는 나머지 절반을 잘라내기 위해 블레이드를 사용합니다.
참고: 그림 7은 유세포 분석에 충분한 CV가 없는 블록을 보여 주었습니다. 이와 같은 블록의 경우, 그림과 같이 모계 조직에 대한 CV의 양을 늘리기 위해 CV를 적게 포함하는 절반이 버려지도록 섹션을 절단해야합니다. 버려진 부분을 보정하기 위해 더 많은 섹션을 잘라야 합니다.
- H&E 슬라이드와 가벼운 현미경을 사용하여 CV로 구성된 조직의 약 50-70%를 포함하는 FFPE 블록을 선택합니다.
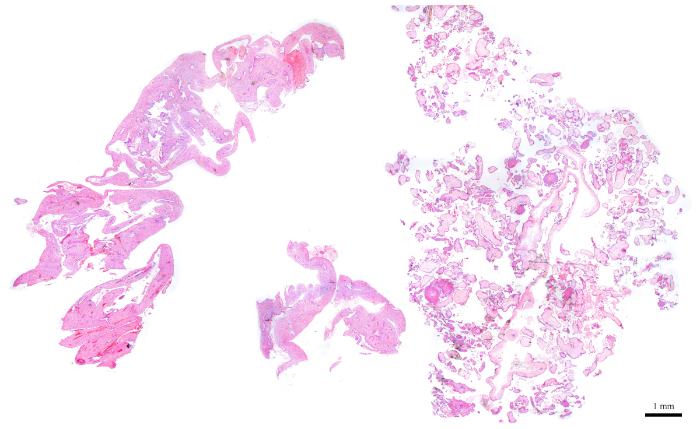
그림 6: 유세포측정에 이상적인 POC 블록을 나타내는 H&E 섹션. 이 그림의 더 큰 버전을 보려면 여기를 클릭하십시오.
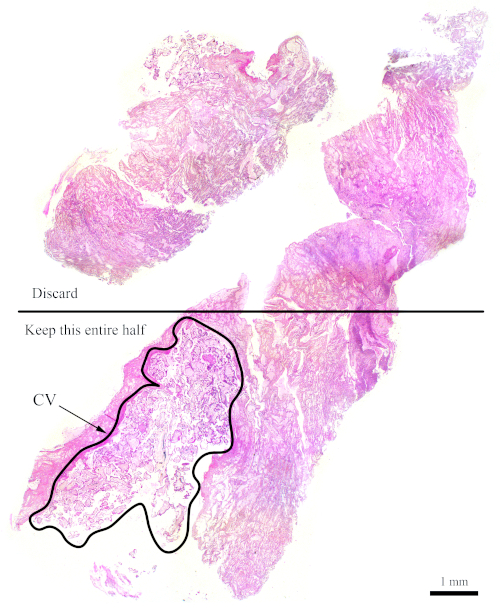
그림 7: 유세포측정에 대한 더 어려운 블록을 나타내는 H&E 섹션. 이 대표적인 H&E 섹션은 CV에 대한 보강을 목표로 이 섹션의 아래쪽 절반만 유세포 분석에 사용해야 함을 보여줍니다. "CV"라고 표시된 윤곽영역은 대부분 CV로 구성됩니다.
- 단면화
- 단면을 용이하게하기 위해 15 분 동안 얼음에 블록을 둡니다.
- 최상의 FFPE 블록을 사용하여 마이크로토메를 사용하여 두께 50 μm(또는 100μm 두께의 두 섹션 2개)인 4개의 단면을 잘라냅니다.
참고: 유세포분석의 경우 더 두꺼운 단면을 가지는 것이 바람직하다. - 이상적인 FFPE 블록을 사용할 수없는 경우, 그럼에도 불구하고 모계 조직에 대한 CV의 비율을 유지하는 것을 목표로합니다. 예를 들어, 블록의 30%만 이력서로 구성되어 있고 나머지는 모성 조직을 가지고 있는 경우, 모성 조직을 포함하는 섹션의 절반 이상을 제거하고 보상하기 위해 더 많은 섹션을 사용합니다(그림 7참조).
- 라벨이 부착된 15mL 튜브에 섹션을 놓습니다.
참고: 다음 단계에서 사용되는 유기 시약이 잉크를 용해하고 제거할 수 있으므로 라벨 위에 테이프를 붙이십시오.
- FFPE 조직에서 유세포 분석 프로토콜
- 탈파화 및 수화
- 연기 후드 아래 다음 세탁(표 3)을수행 합니다.
- 15 mL 튜브를 적절한 시약의 6 mL로 채우고, 표 3에 제시된 순서에 따라, 각각의 지속 기간 동안 시약에 섹션을 두고, 진공 흡입 및 유리 파스퇴르 피펫을 사용하여 시약을 제거한다.
- 각 단계 사이에 파스퇴르 파이펫을 먼저 70 % 에탄올에 담근 다음 증류수에 담근 다음 다음 다음 단계로 진행하십시오.
- 시약과 함께 조직의 조각을 제거하지 않도록 매우주의하십시오. 15 mL 튜브를 60도 각도로 기울여 조직을 그리지 않고 액체 시약의 흡입을 용이하게합니다.
주의 사항: 버려진 액체는 자일렌을 포함하고 자일렌 폐기물 용기에 폐기해야합니다.
- 솔루션 준비
- 이중 증류수 1L에 구연산 2g을 용해시켜 구연산 용액을 준비합니다. pH를 6으로 가져옵니다. 4 °C에서 보관하십시오.
- 펩신 용액을 0.01 g의 펩신을 천 NaCl 당 9 개의 2 mL, pH 1.64에 용해시킴으로써 준비한다. 이것은 하나의 샘플입니다.
주의 사항: 펩신은 독성이 있으며 쉽게 분산되어 공중에 떠 있게 될 수 있습니다. 펩신을 분말 형태로 다룰 때 마스크를 착용하고 사용 후 작업 영역을 모두 닦으십시오. - 프로피듐 요오드화물 (PI)-리보누올아제 한 샘플에 대한 용액 제제.
- PI 50 μL을 450 μL의 PBS와 혼합합니다(10x 희석).
- 혼합물에 리보클리스 A(1 mg/mL)의 50 μL을 첨가합니다. 항상 호일에 싸여 보관하십시오.
- 소화 와 염색
- 15 mL 튜브에 4 °C C 액을 추가 한 다음 2 시간 동안 80 °C 수조에 놓습니다.
- 용액을 실온(15분)까지 식힙니다. 시트레이트 용액을 제거합니다.
- 1x PBS, 소용돌이 6mL를 추가하고 1-2 분 동안 기다렸다가 조직이 바닥에 정착할 수 있도록 합니다. 진공 흡입 및 유리 파스퇴르 파이펫을 사용하여 1x PBS를 제거합니다.
- 펩신 용액 1 mL(예열된 37°C)을 37°C 건조 조에 넣고 30분마다 소용돌이에 놓고 10분마다 PI-ribonuclease A 용액을 이 배양 후 마지막 10분 동안 준비합니다.
- 1x PBS, 소용돌이 6mL를 추가하고 1-2 분 동안 기다렸다가 조직이 바닥에 정착할 수 있도록 합니다. 진공 흡입 및 유리 파스퇴르 파이펫을 사용하여 1x PBS를 제거합니다.
- PI-ribonuclease A 용액의 550 μL을 추가하고 샘플을 37 °C 건조 욕조에 30 분 동안 놓습니다.
참고: 이 시점에서, 시료는 호일에 싸서 다음날 아침까지 4°C에서 하룻밤 방치될 수 있다. - 48μm 여과 메쉬를 통해 용액을 필터링합니다. 흐름 세포계와 함께 사용할 수있는 폴리스티렌 라운드 바닥 튜브에서 여과액을 수집합니다. 포셉을 사용하여 튜브의 상단 부분에 5cm x 5cm 크기의 여과 메쉬를 배치하여 액체가 메쉬를 통해 튜브로 피펫화 될 수 있도록하십시오.
참고: 이제 샘플이 유량 세포계로 실행될 준비가 되었습니다. 그들이 실행 될 준비가 될 때까지 호일에 싸여 유지합니다.
- 조직의 유세포 분석 플랫폼 기술자의 도움을 받아 유량 세포계로 샘플을 실행합니다.
참고: PE 채널은 PI 염색 DNA를 검출하는 데 사용되며 유속은 수집 중에 느리게 설정되어야 합니다. 분석 및 해석을 용이하게 하기 위해 PE-A x축을 따라 디플로이드 피크가 대략 200에 도달하도록 전압이 선택되었는지 확인합니다. 샘플당 최소 20,000개의 이벤트를 기록하는 것을 목표로 합니다.
- 탈파화 및 수화
| 시약 사용(각 6mL) | 기간 |
| 1) 자일렌 | 2 x 10 분 |
| 2) 100% 에탄올 | 2 x 10 분 |
| 3) 95% 에탄올 | 약 10분 |
| 4) 70% 에탄올 | 약 10분 |
| 5) 50% 에탄올 | 약 10분 |
| 6) 증류수 | 2 x 10 분 |
표 3: 탈파화 및 재수화를 위한 시약 및 지속 시간.
-
유세포분석 데이터 분석
- 유세포분석소프트웨어(재료 표)로데이터를 분석합니다.
참고: 다음 단계는 특정소프트웨어(재료 표)에대한 것이지만 다른 유형의 소프트웨어 설정에도 도움이 될 수 있습니다.- 유량 세포계에서 샘플을 실행한 후 분석을 위해 FCS 2.0 파일을 다운로드합니다.
- 유세포분석 소프트웨어를 열고 File | 새 문서.
- 히스토그램 아이콘()을
 클릭한 다음 포인터를 드래그하여 사각형을 만듭니다.
클릭한 다음 포인터를 드래그하여 사각형을 만듭니다. - FCS 파일을 찾아보고 열기를클릭합니다. x축을 따라 FCS-A를 클릭한 다음 PE-A를선택합니다.
- 점 도표 아이콘
 ()을 클릭 한 다음 포인터를 드래그하여 히스토그램 플롯 아래에 다른 사각형을 만듭니다. 그런 다음 히스토그램에 대해 선택된 동일한 FCS 파일을 찾아봅슬합니다.
()을 클릭 한 다음 포인터를 드래그하여 히스토그램 플롯 아래에 다른 사각형을 만듭니다. 그런 다음 히스토그램에 대해 선택된 동일한 FCS 파일을 찾아봅슬합니다. - 점 플롯의 x축을 PE-A로 변경하고 y축을 PE-W로변경합니다.
참고: 그림 8A는 이 시점에서 플롯의 모양을 보여 줍니다. - 영역 아이콘 ()을
 클릭하고 그림 8의 점 도표와 같이 diploid 피크 (그림 8B의x 축에서 약 100)에서 시작하여 x 축에서 약 700 으로 끝나는 점 도표에 상자를 그립니다. B.
클릭하고 그림 8의 점 도표와 같이 diploid 피크 (그림 8B의x 축에서 약 100)에서 시작하여 x 축에서 약 700 으로 끝나는 점 도표에 상자를 그립니다. B.
참고: 그림 8의 디플로이드 피크는 x축에서 대략 200입니다. 이것은 샘플이 유동 세포계를 통해 기록될 때 임의로 선택되며, 단순히 결과의 분석및 해석을 용이하게 하기 위해서입니다. - 플롯을 클릭 | 지역/게이트 편집, 다음 전략에서 G0 셀 옆에 있는 셀에 R0을 입력합니다. 그런 다음 닫기를 클릭합니다.
- 히스토그램의 아무 곳이나 클릭한 다음 플롯에서 | 플롯/오버레이 형식 지정. 게이트에서 G0 = R0을 선택한 다음 확인을클릭합니다.
참고: 이것은 더 나은 ploidy 피크를 시각화 할 수 있습니다 게이팅 단계입니다. 히스토그램은 이제 그림 8B의히스토그램처럼 보입니다. 점 플롯의 특정 영역에 초점을 맞추기 위해 (단계 2.4.1.7에 그려진 상자를 이동하여) 생성 된 게이트를 가지고 놀 수 있습니다. - 플롯에 레이블을 지정하려면 텍스트 영역
 아이콘()을 클릭한 다음 포인터를 드래그하여 문서 맨 위에 상자를 만든 다음 환자 ID, POC ID 및 사용된 블록(하나의 POC에 대해 여러 블록이 있을 수 있음)을 입력합니다. 블록에 존재하는 백분율 CV, 샘플을 실행하는 데 사용되는 전압 및 날짜입니다.
아이콘()을 클릭한 다음 포인터를 드래그하여 문서 맨 위에 상자를 만든 다음 환자 ID, POC ID 및 사용된 블록(하나의 POC에 대해 여러 블록이 있을 수 있음)을 입력합니다. 블록에 존재하는 백분율 CV, 샘플을 실행하는 데 사용되는 전압 및 날짜입니다.
- 유세포분석소프트웨어(재료 표)로데이터를 분석합니다.
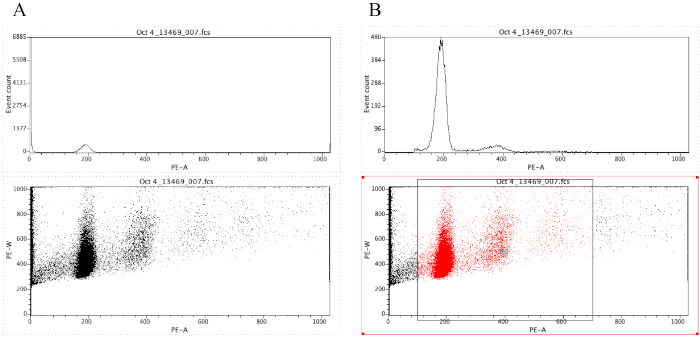
그림 8: 히스토그램과 대표 샘플의 도트 플롯을 표시하는 스크린샷(A) 및 게이트(B). 이 그림의 더 큰 버전을 보려면 여기를 클릭하십시오.
결과
어금니 조직의 복잡성과 다양한 유전자형이 있을 수 있다는 사실은 형태학적 평가, p57 면역 조직 화학, 마이크로 위성 유전형 분석, 유세포 분석 및 FISH와 같은 여러 방법의 엄격한 분석 및 사용을 필요로합니다. 예를 들어, 한 환자(1790)는 POCs의 마이크로어레이 분석에 의해서만 삼엽수인 것으로 밝혀진 2개의 PHM을 참조하였다. 환자는 그러므로 재발하는 PHM으로 진단되었습...
토론
HM은 이질적인 병인을 가진 이상한 인간 적인 임신이고 그들의 정확한 분류 및 진단을 도전하게 하는 다른 조직학 및 genotypic 모형이 있습니다. 조직 병리학 적 형태 학적 평가는 종종 부정확입증되었고 따라서 HM을 CHM 및 PHM으로 분류하고 비 어금니 유산과 구별하기 위해 자체적으로 신뢰할 수 없습니다. 따라서 HM의 정확한 진단은 멀티플렉스 마이크로위성 DNA 지질분석, 유세포분석에 의한 계략 분...
공개
저자는 공개 할 것이 없다.
감사의 말
저자는 원래 의 흐름 세포 분석 프로토콜을 공유 소피 패트리어와 마리안 파레시 감사, 공급 및 시약을 제공하는 프로메가와 Qiagen. 이 작품은 레소 Québécois en 생식및 건강 연구를 위한 캐나다 연구소 (MOP-130364)에 의해 R.S에 지원되었습니다.
자료
| Name | Company | Catalog Number | Comments |
| BD FACS Canto II | BD BioSciences | 338960 | |
| Capillary electrophoresis instrument: Genomes Applied Biosystems 3730xl DNA Analyzer | Applied biosystems | 313001R | Service offered by the Centre for Applied Genomics (http://www.tcag.ca) |
| Citric acid | Sigma | 251275 | |
| Cytoseal 60, histopathology mounting medium | Fisher | 23244257 | |
| Eosin Y stock solution (1%) | Fisher | SE23-500D | |
| FCSalyzer - flow cytometry analysis software | SourceForge | - | https://sourceforge.net/projects/fcsalyzer/ |
| FFPE Qiagen kit | Qiagen | 80234 | |
| Forceps | Fine Science Tools | 11295-51 | For sectioning and for the cleaning process |
| Glacial Acetic Acid (Concentrated) | Sigma | A6283-500mL | |
| Glass coverslips: Cover Glass | Fisher | 12-541a | |
| Hematoxylin | Fisher | CS401-1D | |
| Highly deionized formamide: Hi-Di Formamide | Thermofisher | 4311320 | |
| IHC platform: Benchmark Ultra | Roche | - | |
| Kimwipes | Ultident | 30-34120 | |
| Microtome | Leica | RM2135 | |
| Microtome blades | Fisher | 12-634-1C | |
| Nitex filtering mesh, 48 μm | Filmar | 74011 | http://www.filmar.qc.ca/index.php?filet=produits&id=51&lang=en ; any other filter is suitable, but this is an inexpensive and effective option from a non-research company |
| p57 antibody | Cell Marque | 457M | |
| Pasteur pipette | VWR | 53499-632 | |
| PCR machine | Perkin Elmer, Applied Biosystems | GeneAmp PCR System 9700 | |
| PeakScanner 1.0 | Applied Biosystems | 4381867 | Software for genotyping analysis. |
| Pepsin from porcine gastric mucosa | Sigma | P7012 | |
| Polystyrene round-bottom tubes | BD Falcon | 352058 | |
| Positively charged slides: Superfrost Plus 25x75mm | Fisher | 1255015 | |
| PowerPlex 16 HS System | Promega Corporation | DC2102 | |
| Propidium Iodide | Sigma | P4864 | |
| Ribonuclease A from bovine pancreas | Sigma | R4875 | |
| Separation matrix: POP-7 Polymer | Thermofisher | 4352759 | |
| UltraPure Agarose | Fisher | 16500-500 | |
| Xylene | Fisher | X3P1GAL |
참고문헌
- Szulman, A. E., Surti, U. The syndromes of hydatidiform mole. II. Morphologic evolution of the complete and partial mole. American Journal of Obstetrics & Gynecology. 132 (1), 20-27 (1978).
- Fukunaga, M., et al. Interobserver and intraobserver variability in the diagnosis of hydatidiform mole. The American Journal of Surgical Pathology. 29 (7), 942-947 (2005).
- Gupta, M., et al. Diagnostic reproducibility of hydatidiform moles: ancillary techniques (p57 immunohistochemistry and molecular genotyping) improve morphologic diagnosis for both recently trained and experienced gynecologic pathologists. The American Journal of Surgical Pathology. 36 (12), 1747-1760 (2012).
- Howat, A. J., et al. Can histopathologists reliably diagnose molar pregnancy?. Journal of Clinical Pathology. 46 (7), 599-602 (1993).
- Banet, N., et al. Characteristics of hydatidiform moles: analysis of a prospective series with p57 immunohistochemistry and molecular genotyping. Modern Pathology. 27 (2), 238-254 (2014).
- Lipata, F., et al. Precise DNA genotyping diagnosis of hydatidiform mole. Obstetrics & Gynecology. 115 (4), 784-794 (2010).
- Buza, N., Hui, P. Partial hydatidiform mole: histologic parameters in correlation with DNA genotyping. International Journal of Gynecologic Pathology. 32 (3), 307-315 (2013).
- Fisher, R. A., et al. Frequency of heterozygous complete hydatidiform moles, estimated by locus-specific minisatellite and Y chromosome-specific probes. Human Genetics. 82 (3), 259-263 (1989).
- Murdoch, S., et al. Mutations in NALP7 cause recurrent hydatidiform moles and reproductive wastage in humans. Nature Genetics. 38 (3), 300-302 (2006).
- Parry, D. A., et al. Mutations causing familial biparental hydatidiform mole implicate c6orf221 as a possible regulator of genomic imprinting in the human oocyte. American Journal of Human Genetics. 89 (3), 451-458 (2011).
- Nguyen, N. M., Slim, R. Genetics and Epigenetics of Recurrent Hydatidiform Moles: Basic Science and Genetic Counselling. Current Obstetrics and Gynecology Reports. 3, 55-64 (2014).
- Sebire, N. J., Savage, P. M., Seckl, M. J., Fisher, R. A. Histopathological features of biparental complete hydatidiform moles in women with NLRP7 mutations. Placenta. 34 (1), 50-56 (2013).
- Nguyen, N. M., et al. Comprehensive genotype-phenotype correlations between NLRP7 mutations and the balance between embryonic tissue differentiation and trophoblastic proliferation. Journal of Medical Genetics. 51 (9), 623-634 (2014).
- Brown, L., et al. Recurrent pregnancy loss in a woman with NLRP7 mutation: not all molar pregnancies can be easily classified as either "partial" or "complete" hydatidiform moles. International Journal of Gynecologic Pathology. 32 (4), 399-405 (2013).
- Colgan, T. J., Chang, M. C., Nanji, S., Kolomietz, E. A Reappraisal of the Incidence of Placental Hydatidiform Mole Using Selective Molecular Genotyping. The International Journal of Gynecological Cancer. 26 (7), 1345-1350 (2016).
- Murphy, K. M., McConnell, T. G., Hafez, M. J., Vang, R., Ronnett, B. M. Molecular genotyping of hydatidiform moles: analytic validation of a multiplex short tandem repeat assay. The Journal of Molecular Diagnostics. 11 (6), 598-605 (2009).
재인쇄 및 허가
JoVE'article의 텍스트 или 그림을 다시 사용하시려면 허가 살펴보기
허가 살펴보기This article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. 판권 소유