このコンテンツを視聴するには、JoVE 購読が必要です。 サインイン又は無料トライアルを申し込む。
Method Article
ホルマリン固定パラフィン埋め込みヒダチジフォームモル組織のマイクロサテライトDNAジェノタイピングとフローサイトメトリープロイディ解析
要約
ヒダチジフォームほくろは、形態学的特徴およびモルゲノムへの親の寄与に応じて分類することができる異種性腸科を有する異常なヒト妊娠である。ここでは、ホルマリン固定パラフィン埋め込みモル組織の多重マイクロサテライトDNAジェノタイピングおよびフローサイトメトリーのプロトコルを、結果の解釈および統合とともに詳細に説明する。
要約
ヒダチジフォームほくろ(HM)は、過剰な栄養芽細胞増殖および異常な胚発生を特徴とする異常なヒト妊娠である。顕微鏡形態学的評価に基づくHMには、完全HM(CHM)と部分HM(PHM)の2種類があります。これらは、モルゲノムに対する親の寄与に基づいてさらに細分化することができる。HMのこのような特性化は、形態学および遺伝子型分析によって、患者管理およびこの興味深い病理の基本的な理解のために重要である。HMの形態学的解析は広い観察者間変動の影響を受け、HMをCHMおよびPHMに正確に分類し、水中非モル中絶と区別するには十分ではないことが十分に文書化されている。ジェノタイピング分析は、主に、最適な品質を持たないホルムリン固定パラフィン埋め込み(FFPE)製品のDNAおよび組織に対して行われ、結果的に間違った結論につながる可能性があります。本稿では、FFPEモル組織の多重ゲノタイピングおよびフローサイトメトリー分析のための詳細なプロトコルを提供し、これらの方法の結果の解釈、トラブルシューティング、および形態学的評価との統合を提供する。、p57KIP2免疫組織化学、およびその中の蛍光ハイブリダイゼーション(FISH)は、正しく、堅牢な診断に達する。ここでは、約400の概念製品の分析から過去10年間に学んだ方法と教訓を共有する。
概要
ヒダチジフォームほくろ(HM)は、異常な胚発生、栄養芽細胞の過剰増殖、絨毛絨毛性絨毛(CV)の水文変性を特徴とする異常なヒト妊娠である。従来、HMは形態学的評価1のみに基づいて完全なHM(CHM)と部分HM(PHM)の2種類に分けられており、従来は2種類に分けられております。しかし、形態学的評価だけではHMを2つのサブタイプ(CHMおよびPHM)に分類し、非モル流産2、3、4と区別するには不十分であることが示されている。
CHMとPHMは悪性腫瘍に対する傾向が異なるため、患者に適切なフォローアップと管理を提供するために、HMのゲノムタイプを正確に決定することが重要です。その結果、過去数十年にわたり、モル組織に対する親の寄与を特定し、HMの正しい分類に達することを目的として、いくつかの方法論が開発され、進化してきました。これらには、核型分析、染色体バンディング多型、ヒト白血病抗原(HLA)血清、制限断片長多型、可変数のカンデム反復、マイクロサテライトジェノタイピング、フローサイトメトリー、およびp57が含まれます。KIP2免疫組織化学。これにより、ゲノムに対する親の貢献に基づくHM概念の正確な細分化が可能になりました:CHMは、ジプロイド・アンドロティック・モノスマティックまたはディプロイド・アンドロゲンティック・ディスパーミックであり、PHMは99%で、99%のディスパーミックであり、症例5、6、7、8の1%で単精子。さらに、過去20年間に出現した別のゲノムタイプのHMがあり、これは二重親である。後者は主に再発し、単一の家族(シンプレックス症例)または少なくとも2つの家族(家族の場合)に影響を与える可能性があります。これらの二重親モルは、主に患者9、10、11、12におけるNLRP7またはKHDC3Lの劣性変異によって引き起こされる。NLRP7における劣性変異を有する患者における二葉性双親HMは、形態学的分析によってCHMまたはPHMと診断され、これは患者13、14における変異の重症度と関連していると思われる。彼らの遺伝子型に従ったHMの分類に加えて、いくつかの遺伝子タイピング法の導入と使用は、異球体二親の概念のような非モル流産からの様々なモルエンティティの区別を可能にしました。概念の他のタイプ5,15.このような概念は、ある程度、HMのいくつかの形態学的特徴を模倣するいくつかの栄養芽細胞増殖および異常な絨毛形態を持ち込む可能性がある。
この記事の目的は、ホルマリン固定パラフィン埋め込み(FFPE)組織の多重ゲノタイピングとフローサイトメトリーの詳細なプロトコルを提供し、これらの方法の結果と他の方法との統合の包括的な分析を提供することです。モル組織の正しく、決定的な診断。
プロトコル
この研究は、マギル機関レビュー委員会によって承認されました.すべての患者は、研究に参加し、様々な病理学部門から受胎(POC)のFFPE製品を取得することに書面による同意を提供しました。
注:フローサイトメトリーによるジェノタイピングとプロディ決定にはいくつかの方法がありますが、ここで提供されるプロトコルは、それぞれに1つのプラットフォームを使用した1つの分析方法を説明します。
1. ジェノタイピング
-
最高のFFPEブロックの選択
- 概セプション(POC)の各FFPE産物について、セクション1.2および1.3に記載されている4μm厚のヘマトキシリンおよびエオシン(H&E)染色されたセクションを調製し、顕微鏡による形態学的評価を行う。
- H&Eスライドと小顕微鏡を使用して、最大量の絨毛膜絨毛(CV)を有するFFPEブロックを選択し、可能であれば、CVを持つブロックと母体組織とは分離せず、混ざり合わないブロックを選択します。
-
断面
- 選択したブロックを氷の上に15分間置き、断面を容易にします。
- 顕微鏡の形態学的評価のために4 μmの厚さ、DNA抽出のための10 μmの厚さのセクションを切断するためにマイクロトームを調整します。
- マイクロトームにコールドブロックを配置し、H&E染色のための各ブロックから1つのセクションをカットし、選択したブロックから10-30セクションを、ブロック内のCVの量に応じて、DNA抽出用に切断します。
注:CVがいっぱいのブロックの場合、DNA抽出には10のセクションで十分です。ブロックの約10%のみがCVを含み、残りが母体組織である場合、十分な量のDNAを確保するために20−30のセクションが必要です。 - 鉗子を使用して、45 °Cの水浴に各セクションを移します。水浴の断面を、鉛筆を使ってサンプル識別番号でラベル付けした正の電荷スライド(材料の表)で取り出します。
- セクションを含むスライドをオーブンに入れ、セクションがスライドに付着できるように65 °Cに置きます。H&Eのスライドをオーブンに25分間保管し、DNA抽出用のスライドをオーブンで20分間保管します。
注:インキュベーション時間が短いほど、組織はスライドにわずかに付着しなくなり、その結果、母体組織の除去が容易になります。
-
H&E染色
- スライドが室温(10分)まで冷却できるようにします。
- 試薬の調製
- エオシン Y ワーキング ソリューションの準備 (0.25%)表 1 に次のように。よく混ぜ、室温で保存します。
- ヘマトキシリン5xのストック溶液を水中で希釈して作業ヘマトキシリン溶液を調作する(すなわち、80mLの水をヘマトキシリンの20mLと混合する)。
注:貯蔵用の箔にヘマトキシリンのストック溶液をラップします。
- 表2に従って、ヒュームフードの下に正しい試薬で染色瓶を準備します。
- 表 2に従って、適切な期間にスライドを適切な染色瓶に浸して H&E 染色を実行します。
- 取り付け媒体とガラスカバースリップで形態解析のための形態解析のための4 μmセクションを取り付けます(材料の表)。
注:ジェノタイピングのための10 μmセクションはカバースリップしないでください。 - 有毒なキシレン臭が消散するために、ヒュームフードの下に10 μmのセクションを最低3時間放置してください。
注意:すべての染色ステップは、ヒュームフードの下で行う必要があります。キシレン製品は、キシレン臭が有毒であるため、常にフードの下に保管する必要があります。さらに、キシレンおよびヘマトキシリンは、特別な容器に廃棄する必要があります。これらの容器が満杯になったら、実験室の安全組織によって推薦される通り廃棄する必要がある。
| 試薬 | 量 |
| エオシンYストックソリューション (1%) | 250 mL |
| 80% エタノール | 750 mL |
| 氷河酢酸(濃縮) | 5 mL |
表 1: エオシン Y 作動ソリューション (0.25%)準備。
| 使用試薬(ビンあたり100mL) | 期間 |
| 1) キシレン | 5 分 |
| 2) キシレン | 5 分 |
| 3) 100% エタノール | 2 分 |
| 4) 95% エタノール | 2 分 |
| 5) 70% エタノール | 2 分 |
| 6) 50% エタノール | 2 分 |
| 7) 蒸留水 | 5 分 |
| 8) ヘマトキシリン | 4 分 |
| 9) 蒸留水 | 5 分 |
| 10) エオシン | 1 分 |
| 11) 95% エタノール | 5 分 |
| 12) 100% エタノール | 5 分 |
| 13) キシレン | 5 分 |
| 14) キシレン | 5 分 |
表 2: H&E 染色プロトコルの試薬および持続時間。
-
CVの分離
- 小光ステレオ顕微鏡の下では、鉗子と水湿紙ワイプ(材料の表)の小片を使用して、H&E染色された10μmの厚いセクションから不要な母体組織を掻き取ります。
注:最終的な目標は、スライド上のCVまたは胎児膜(存在する場合)を維持し、したがって、他のすべての組織を除去することです。このステップは、細部に細心の注意を払う必要があるため、ブロックによっては多くの時間と忍耐が必要な場合があります。 - 2人目の人に、クリーニング後にスライドを再確認し、母体組織がないことを確認します。
- きれいなスライドの写真を撮るか、データの解釈に役立つ次の文書:1)組織が洗浄が困難であったかどうか、出血性、または非常にきれいであったかどうか、2)使用されるセクションの数、および3)洗浄された組織のおおよその量。
注: 図 1は、簡単にクリーニングできるスライドの例を示しています。この量のCVを含むブロックの場合、DNA抽出には10のセクションで十分です。図2のスライドは、母体組織と混ざり合った非常に少数のCVを有し、洗浄が非常に困難で時間がかかります。この量のCVを含むブロックの場合、DNA抽出には30のセクションが必要です。 - 小さな湿った紙の拭き取りを使用してCVを収集します。鉗子を使用して、湿った紙の拭き取りから小さな部分を引き裂き、CVを収集するためにそれを使用します。
- 付属のCVで紙拭き取りをラベル付きの1.5 mLチューブに入れます。
- このステップで使用される紙拭き取りの量を最小限に抑えるには、DNA抽出カラムが詰まり、収集されたDNAの最終量が減少する可能性があります。平均して、サンプルあたり 7 個未満の小さな用紙拭き取りを使用することを目指します。大量のCVが存在することが不可能な場合は、抽出を容易にするために2つのチューブ間でサンプルを分割します。
- 小光ステレオ顕微鏡の下では、鉗子と水湿紙ワイプ(材料の表)の小片を使用して、H&E染色された10μmの厚いセクションから不要な母体組織を掻き取ります。

図1:ジェノタイピングの代表的なスライド。上:母体組織を解放するために「洗浄」する必要があるスライド。下: 洗浄後に表示される同じスライドで、DNA抽出用の CV しか含まれていません。この図のより大きなバージョンを表示するには、ここをクリックしてください。
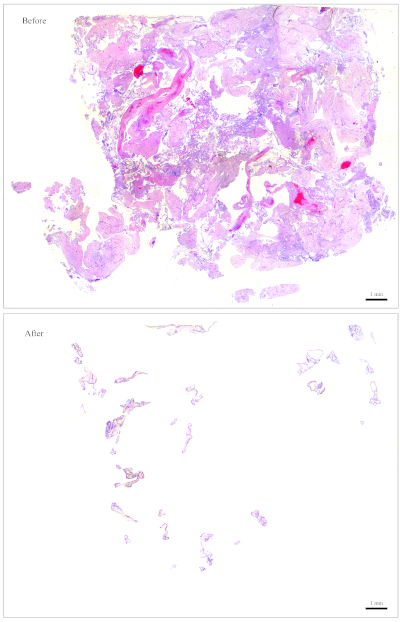
図2:ジェノタイピングの代表的なスライド。上:母体組織を解放するために「洗浄」する必要があるスライド。下: 洗浄後に表示される同じスライドで、DNA抽出用の CV しか含まれていません。この図のより大きなバージョンを表示するには、ここをクリックしてください。
- FFPEキット(材料の表)からのDNA抽出のプロトコルに従ってDNA抽出を実行します。
注:一部のキットでは、最終的な溶出に 15-20 μL の溶出バッファーを使用することをお勧めします。経験から、溶出バッファーの15 μLの溶出はほとんどのサンプルのためによく働く。希釈は、必要に応じてストックDNAから調製してもよい。
-
DNA定量
- ラボ分光光度計装置を使用して、DNAの1 μLをロードし、定量のために260 nmで吸光度を測定します。
- 2%のアガロースゲルにDNAの1 μLをロードし、定性的評価のために80−100Vの電圧でゲル電気泳動を実行します。
- ステップ1.6.1および1.6.2の結果に基づいて、多重短タンデムリピート(STR)ポリメラーゼ連鎖反応(PCR)増幅に使用するDNAの体積を選択します。その後のPCR増幅に最低1000ngのDNAを使用することを目指します。
注:図3は、DNAの濃度(分光光度計の結果に基づく)と、以下の多重STR PCRに推奨されるDNA溶液の体積と共に、ゲルの代表的な例を示す。
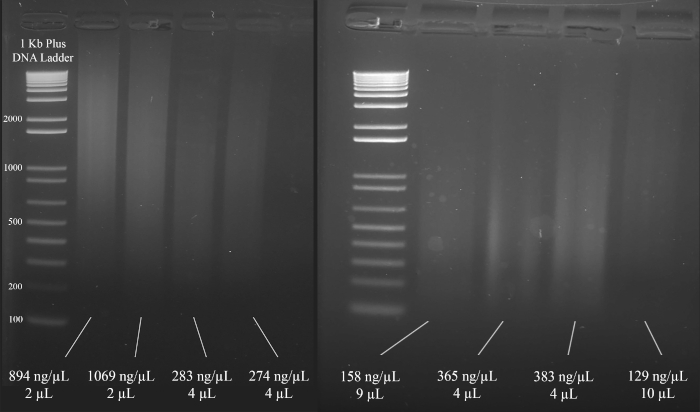
図3:DNA定量のための代表的なゲル。分光光度計を用いて測定した各DNAの濃度と、多重PCRに使用される量が含まれます。この図のより大きなバージョンを表示するには、ここをクリックしてください。
-
PCR増幅
- 多重STRシステム(材料の表)を使用して蛍光マイクロサテライトゲノタイピングを実行します。
- 多重STRシステム(材料の表)を使用してPCR増幅のために図4に示すPCR条件を使用してください。
注:このマルチプレックス STR システムでは、D18S51、D21S11、TH01、D3S1358、ペンタ E、FGA、TPOX、D8S1179、vWA、アメロゲニン、CSF1PO、D16S539、D7S820、D13S318、および D13S3181818、および D13S1817 で使用されています。

図4:多重STRシステムのPCRサイクル条件。この図のより大きなバージョンを表示するには、ここをクリックしてください。
-
毛細血管電気泳動によってPCR製品を解決します。
- 多重システムの内部標準レーンの0.5 μLおよび高度脱イオン化されたホルムアミドの9.5 μL(材料の表)の各増幅されたサンプルの1 μLを中断する。
- 器械および多重システムの色素セットのための適切な分離マトリックス(材料のテーブル)を使用して毛細血管電気泳動器械(材料の表)を通してサンプルを実行する。
- データ分析
- DNAフラグメント解析ソフトウェアを使用してデータを分析し、POC対向性を親の対向性に比較して、その起源を特定します。
- サイズ標準を設定します。
注:これにより、ソフトウェアはマルチプレックスSTRシステムで使用されるラダーを認識し、ラダーに基づいてアンプリコンにベースペアを割り当てることができます。次の手順は、1 つの特定のソフトウェア (マテリアルの表)を対象としていますが、他の種類のソフトウェアをセットアップする場合にも役立つ場合があります。- ソフトウェアを開きます。[新しいプロジェクトを開始]をクリックし、[新しいサイズの標準]をクリックします。
- サイズ標準に名前を付けます (ABI_600 など)。
- [新しいサイズ標準定義を入力]という名前のボックスに、次のように入力します: 60、 80、100、120、140、140、160、180、200、225、250、275、300、325、425、450、475、475、500、500、500、500。次に、[サイズを追加] をクリックします。
注: 入力した数値は右側のボックスの下に表示され、現在のサイズ標準定義という名前が付けられています(図 5 を参照)。 - [保存]をクリックします。
- ファイルをインポートして分析するには、[ファイルの追加]をクリックし、分析する fsa ファイルを選択します。[選択したファイルを追加]をクリックし、[OK] をクリックします。 次に、次の手順に従います。
- [サイズ標準]列を見つけて、ABI_600 (またはサイズ標準に指定された名前) を選択します。
- [解析方法]で、[既定のサイズ変更 - NPP]をクリックし、緑色の[分析]ボタンをクリックします。
- これで、ファイルを表示する準備ができました。表示オプションを調整して、必要に応じてデータを表示します。
- トラブルシューティング - 分析方法
注:ソフトウェアは、ピークを識別し、正しく位置合わせに失敗することがあります。これは、ピークが低すぎるか高すぎる場合に発生します。次の 2 つの分析方法は、これを修正し、サンプルを再テストする前に試す必要があります。- 高ピークの解析方法 1:
- 新しい分析方法をクリックし、[高ピーク](または個人の好みに基づいて別の名前)と付けます。
- [範囲]をクリックし、[部分範囲]をクリックして解析とサイズ変更を行います。次に、開始点と開始サイズに100と入力します。
- [ストップ ポイントの場合は、10,000 と入力してください。[ストップ サイズ]に「1000」と入力します。
- 次に、最小ピークの高さをクリックし、色のピークしきい値が次のようになるように数値を変更します: 青: 50;緑: 50;イエロー:20;赤: 100;オレンジ:5000。
- 新しい解析方法を保存します。
- 低ピークの解析方法 2:
- 新しい分析方法をクリックし、低ピーク(または個人の好みに基づいて別の名前) という名前を付けます。
- [範囲]をクリックし、[部分範囲]をクリックして解析とサイズ変更を行います。次に、開始点と開始サイズに100と入力します。
- [ストップ ポイントの場合は、10,000 と入力してください。[ストップ サイズ]に「1000」と入力します。
- 次に、品質フラグをクリックし、パス範囲を変更して、0.5から 1 に読み取るようにします。低品質範囲を0.0 から 0.0 に読み取るように変更します。(bp) 100.0 から (bp) 800.0 に対して、線形性を次のように変更します。
- 新しい解析方法を保存します。
注:[解析方法]で[低ピーク]または [高ピーク]を選択し、緑色の[分析]ボタンをクリックしてファイルを再分析できるようになりました。
- 高ピークの解析方法 1:

図 5: サイズ標準エディタを示すスクリーンショット。この図のより大きなバージョンを表示するには、ここをクリックしてください。
2. フローサイトメトリー
-
理想的な FFPE ブロックの選択
- H&Eスライドと小顕微鏡を使用して、CVで構成される組織の約50−70%を有するFFPEブロックを選択します。
注:図6は、約50%のCV(セクションの右半分)と50%の母体組織(左半分)で構成されているため、フローサイトメトリー分析に適したブロックの代表的な例です。彼らはジプロイドピークの内部制御として機能するので、母体組織の存在は重要です。 - CV の理想的な量を持たないブロックの場合は、セクション化が実行されるにつれて CV に富む。この方法を行うには、対応する H&E スライドに従って、新しく切断されたセクションのどちら側にさらに CV が含まれているかを特定します。それに基づいて、CVのために豊かにするために廃棄する必要がある残りの半分を切断するためにブレードを使用します。
注:図7は、フローサイトメトリー分析に十分なCVを持たないブロックを示しています。このようなブロックの場合、図に示すように、母体組織に対するCVの量を増やすために、CVを含む半分が廃棄されるようにセクションをカットする必要があります。破棄される部分を補正するために、より多くのセクションをカットしてください。
- H&Eスライドと小顕微鏡を使用して、CVで構成される組織の約50−70%を有するFFPEブロックを選択します。
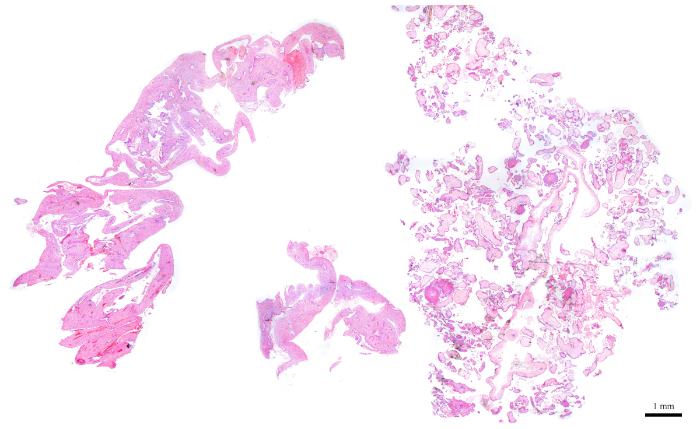
図 6: フロー サイトメトリーに最適な POC ブロックを表す H&E セクション。この図のより大きなバージョンを表示するには、ここをクリックしてください。
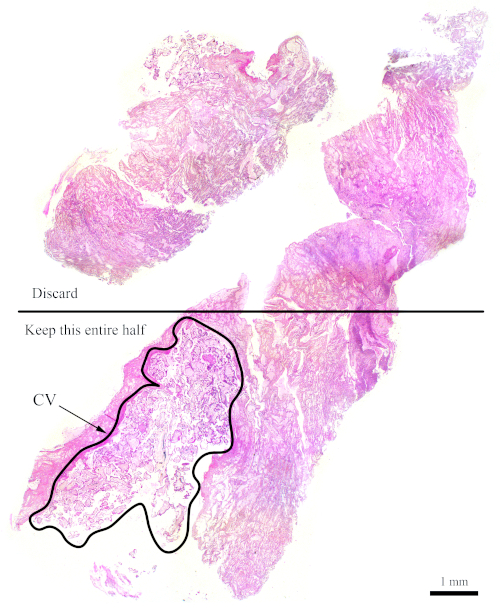
図 7: フローサイトメトリーのより困難なブロックを表す H&E セクション。この代表的な H&E セクションでは、このセクションの下半分だけをフロー サイトメトリー分析に使用し、CV を豊かにすることを目的としています。「CV」というラベルの付いた輪郭を描いた領域は、主にCVで構成されています。
- 断面
- 断面を容易にするために、15分間氷の上にブロックを残します。
- 可能な限り最高のFFPEブロックを使用して、マイクロトームを使用して50 μm厚い(または2つの100 μmの厚いセクション)の4つのセクションを切断します。
注:フローサイトメトリーの場合は、より厚い断面を有することが好ましい。 - 理想的なFFPEブロックが利用できない場合には、それにもかかわらず、母体組織に対するCVの比率を維持することを目指す。たとえば、ブロックの 30% のみが CV で構成され、残りの部分に母体組織がある場合は、母体組織を含むセクションの少なくとも半分を削除し、さらに多くのセクションを使用して補正します (図 7参照)。
- ラベル付きの 15 mL チューブにセクションを配置します。
注:次のステップで使用する有機試薬はインクを溶解および除去することができるので、ラベルの上にテープを貼るようにしてください。
- FFPE組織からのフローサイトメトリープロトコル
- 脱パラフィン化と水分補給
- ヒュームフードの下で以下の洗い物(表3)を行います。
- 15 mLチューブを適切な試薬の6 mLで充填し、表3に示す順序に従って、試薬内のセクションをそれぞれの持続時間に残し、真空吸引とガラスパスツールピペットを使用して試薬を除去する。
- 各ステップの間に、最初に70%のエタノールでパスツールピペットを浸し、次に蒸留水に浸してから、次のステップに進みます。
- 試薬と一緒に組織の断片を除去しないように非常に注意してください。15 mLチューブを60度の角度に傾け、ティッシュを引かずに液体試薬の吸引を容易にします。
注意:廃棄された液体にはキシレンが含まれており、キシレン廃棄物容器に入れるべきです。
- ソリューションの準備
- クエン酸2gを二重蒸留水の1Lに溶解してクエン酸溶液を調出す。pHを6にします。4 °Cで保存します。
- ペプシン溶液を0.01gのペプシンを2mLで9個のNaCl、pH 1.64に溶解して調出する。これは 1 つのサンプルです。
注意:ペプシンは有毒であり、容易に分散し、空気中になることができます。ペプシンを粉末状に取り扱う場合はマスクを着用し、使用後に作業領域をすべて拭き取ります。 - ヨウ化プロピジウム(PI)-リボヌクレアーゼ1サンプルに対する溶液製剤。
- 50 μL の PI を 450 μL の PBS と混合します(希釈 10 倍)。
- 混合物に50 μLのリボヌクレアーゼA(1mg/mL)を加えます。常にホイルに包んでおいてください。
- 消化と染色
- 15 mLチューブに4°Cのクエン酸溶液を加え、80°Cの水浴に2時間入れます。
- 溶液を室温(15分)まで冷やします。クレート溶液を取り除きます。
- 1x PBS、渦の6 mLを追加し、組織が底部に落ち着くように1-2分待ちます。真空吸引とガラスパスツールピペットを使用して1x PBSを取り外します。
- 1 mLのペプシン溶液(37°Cに予熱)を加え、37°Cの乾式浴に30分毎に30分間置き、このインキュベーションの最後の10分間でPI-リボヌクレアーゼA溶液を準備します。
- 1x PBS、渦の6 mLを追加し、組織が底部に落ち着くように1-2分待ちます。真空吸引とガラスパスツールピペットを使用して1x PBSを取り外します。
- PI-リボヌクレアーゼA溶液の550 μLを追加し、30分間37°Cの乾燥浴にサンプルを入れます。
注:この時点で、サンプルはホイルで包まれ、翌朝まで4°Cで一晩残すことができます。 - 48 μm の濾過メッシュを通して溶液をろ過します。フローサイトメーターで使用できるポリスチレン丸底チューブで濾液を収集します。鉗子を使用して、チューブの上部に 5 cm x 5 cm の濾過メッシュを配置し、液体をメッシュを通してチューブ内にピペテットできるようにします。
注:これで、サンプルをフローサイトメーターで実行する準備が整いました。彼らが実行する準備ができるまで、ホイルで包んでおいてください。
- 組織のフローサイトメトリープラットフォーム技術者の助けを借りて、フローサイトメーターでサンプルを実行します。
注:PEチャネルは、PI染色されたDNAを検出するために使用され、流量は取得時に遅く設定する必要があります。分析と解釈を容易にするために、ディップロイドピークがPE-A X軸に沿っておよそ200であるような電圧が選択されていることを確認してください。サンプルごとに最低 20,000 イベントを記録することを目指します。
- 脱パラフィン化と水分補給
| 使用試薬(各6mL) | 期間 |
| 1) キシレン | 2 x 10 分 |
| 2) 100% エタノール | 2 x 10 分 |
| 3) 95% エタノール | 10分 |
| 4) 70% エタノール | 10分 |
| 5) 50% エタノール | 10分 |
| 6) 蒸留水 | 2 x 10 分 |
表3:脱パラフィン化および水分補給のための試薬および持続時間。
-
フローサイトメトリーデータ分析
- フローサイトメトリー解析ソフトウェア(材料の表)を使用してデータを分析します。
注:次の手順は、1 つの特定のソフトウェア (マテリアルの表)を対象としていますが、他の種類のソフトウェアをセットアップする場合にも役立つ場合があります。- フロー サイトメーターでサンプルを実行した後、分析用に FCS 2.0 ファイルをダウンロードします。
- フローサイトメトリー解析ソフトウェアを開き、[ファイル] をクリックします。新しいドキュメント:
- ヒストグラムアイコン ()
 をクリックし、ポインタをドラッグして四角形を作成します。
をクリックし、ポインタをドラッグして四角形を作成します。 - FCS ファイルを参照し、[開く]をクリックします。X 軸に沿って、FCS-Aをクリックし、PE-A を選択します。
- ドットプロットアイコン()
 をクリックし、ポインタをドラッグしてヒストグラムプロットの下に別の長方形を作成します。次に、ヒストグラムに対して選択されたのと同じ FCS ファイルを参照します。
をクリックし、ポインタをドラッグしてヒストグラムプロットの下に別の長方形を作成します。次に、ヒストグラムに対して選択されたのと同じ FCS ファイルを参照します。 - ドットプロットのX軸をPE-Aに、Y 軸をPE-Wに変更します。
注: 図 8Aは、この時点でのプロットの外観を示しています。 - [領域] アイコン
 () をクリックし、ディプロイドピークの前 (図 8Bの x 軸で約 100) の前に始まり、X 軸で約 700 で終わるドット プロット上のボックスを描画します(図8のドット プロットに示すように)B.
() をクリックし、ディプロイドピークの前 (図 8Bの x 軸で約 100) の前に始まり、X 軸で約 700 で終わるドット プロット上のボックスを描画します(図8のドット プロットに示すように)B.
注:図 8のディプロイド ピークは、X 軸上の約 200 です。これは、サンプルがフローサイトメーターを介して記録され、単に結果の分析と解釈を容易にするために任意に選択されます。 - プロットをクリック|[領域/ゲートを編集]を行い、[ストラテジー] の下のG0セルの横にあるセルにR0と入力します。次に、をクリックします。
- ヒストグラム上の任意の場所をクリックし、プロットで|プロット/オーバーレイの書式設定 :[ゲート]で[G0 = R0]を選択し、[OK]をクリックします。
注:これは、プロイディピークをより良く視覚化することを可能にするゲーティングステップです。ヒストグラムは、図 8Bのヒストグラムのように見えるはずです。ドットプロットの特定の領域に焦点を当てるために(ステップ2.4.1.7で描かれたボックスを動かすことによって)作成されたゲートで遊ぶことができます。 - プロットにラベルを付けるには、[テキスト領域]
 アイコン () をクリックし、ポインターをドラッグしてドキュメントの上部にボックスを作成し、次の情報を入力します: 患者 ID、POC ID、および使用されるブロック (1 つの POC に対して複数のブロックが存在する可能性があるため)。ブロック上に存在するパーセント CV、サンプルの実行に使用される電圧、および日付。
アイコン () をクリックし、ポインターをドラッグしてドキュメントの上部にボックスを作成し、次の情報を入力します: 患者 ID、POC ID、および使用されるブロック (1 つの POC に対して複数のブロックが存在する可能性があるため)。ブロック上に存在するパーセント CV、サンプルの実行に使用される電圧、および日付。
- フローサイトメトリー解析ソフトウェア(材料の表)を使用してデータを分析します。
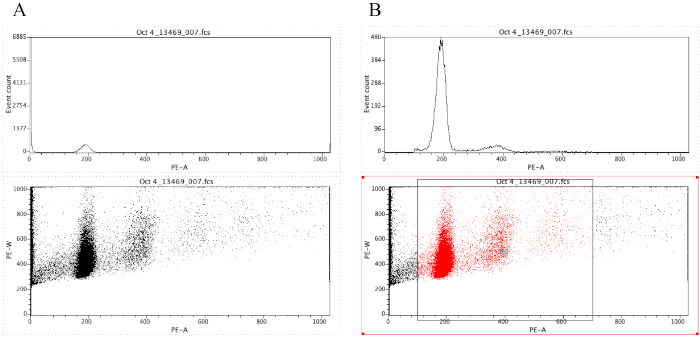
図8:ヒストグラムと、アンゲート(A)とゲート(B)の代表的なサンプルのドットプロットを表示するスクリーンショット。この図のより大きなバージョンを表示するには、ここをクリックしてください。
結果
モル組織の複雑さと様々な形のゲノムを持つ可能性があるという事実は、形態学的評価、p57免疫組織化学、マイクロサテライトゲノタイピング、フローサイトメトリー、FISHなどのいくつかの方法の厳格な分析と使用を必要とします。例えば、1人の患者(1790)は、POCのマイクロアレイ分析によってのみ三脚であることが判明した2つのPHMと呼ばれた。したがって、患者は?...
ディスカッション
HMは異種病因を有する異常なヒト妊娠であり、異なる組織学的および好奇数型を有し、正確な分類および診断を困難にする。ヒト病理学的形態学的評価はしばしば不正確であることが証明され、したがって、HMをCHMおよびPHMに分類し、非臼頭流産と区別するために単独で信頼性が低い。したがって、HMの正確な診断には、多重マイクロサテライトDNAジェノタイピング、フローサイトメトリーに?...
開示事項
著者は何も開示していない。
謝辞
著者らは、元のフローサイトメトリープロトコルを共有してくれたソフィー・パトリアーとマリアンヌ・パレシー、および供給と試薬を提供するためのプロメガとキアゲンに感謝しています。この研究は、レソー・ケベコイス・アン・リオー殖とカナダ保健研究所(MOP-130364)によってR.S.に支援されました。
資料
| Name | Company | Catalog Number | Comments |
| BD FACS Canto II | BD BioSciences | 338960 | |
| Capillary electrophoresis instrument: Genomes Applied Biosystems 3730xl DNA Analyzer | Applied biosystems | 313001R | Service offered by the Centre for Applied Genomics (http://www.tcag.ca) |
| Citric acid | Sigma | 251275 | |
| Cytoseal 60, histopathology mounting medium | Fisher | 23244257 | |
| Eosin Y stock solution (1%) | Fisher | SE23-500D | |
| FCSalyzer - flow cytometry analysis software | SourceForge | - | https://sourceforge.net/projects/fcsalyzer/ |
| FFPE Qiagen kit | Qiagen | 80234 | |
| Forceps | Fine Science Tools | 11295-51 | For sectioning and for the cleaning process |
| Glacial Acetic Acid (Concentrated) | Sigma | A6283-500mL | |
| Glass coverslips: Cover Glass | Fisher | 12-541a | |
| Hematoxylin | Fisher | CS401-1D | |
| Highly deionized formamide: Hi-Di Formamide | Thermofisher | 4311320 | |
| IHC platform: Benchmark Ultra | Roche | - | |
| Kimwipes | Ultident | 30-34120 | |
| Microtome | Leica | RM2135 | |
| Microtome blades | Fisher | 12-634-1C | |
| Nitex filtering mesh, 48 μm | Filmar | 74011 | http://www.filmar.qc.ca/index.php?filet=produits&id=51&lang=en ; any other filter is suitable, but this is an inexpensive and effective option from a non-research company |
| p57 antibody | Cell Marque | 457M | |
| Pasteur pipette | VWR | 53499-632 | |
| PCR machine | Perkin Elmer, Applied Biosystems | GeneAmp PCR System 9700 | |
| PeakScanner 1.0 | Applied Biosystems | 4381867 | Software for genotyping analysis. |
| Pepsin from porcine gastric mucosa | Sigma | P7012 | |
| Polystyrene round-bottom tubes | BD Falcon | 352058 | |
| Positively charged slides: Superfrost Plus 25x75mm | Fisher | 1255015 | |
| PowerPlex 16 HS System | Promega Corporation | DC2102 | |
| Propidium Iodide | Sigma | P4864 | |
| Ribonuclease A from bovine pancreas | Sigma | R4875 | |
| Separation matrix: POP-7 Polymer | Thermofisher | 4352759 | |
| UltraPure Agarose | Fisher | 16500-500 | |
| Xylene | Fisher | X3P1GAL |
参考文献
- Szulman, A. E., Surti, U. The syndromes of hydatidiform mole. II. Morphologic evolution of the complete and partial mole. American Journal of Obstetrics & Gynecology. 132 (1), 20-27 (1978).
- Fukunaga, M., et al. Interobserver and intraobserver variability in the diagnosis of hydatidiform mole. The American Journal of Surgical Pathology. 29 (7), 942-947 (2005).
- Gupta, M., et al. Diagnostic reproducibility of hydatidiform moles: ancillary techniques (p57 immunohistochemistry and molecular genotyping) improve morphologic diagnosis for both recently trained and experienced gynecologic pathologists. The American Journal of Surgical Pathology. 36 (12), 1747-1760 (2012).
- Howat, A. J., et al. Can histopathologists reliably diagnose molar pregnancy?. Journal of Clinical Pathology. 46 (7), 599-602 (1993).
- Banet, N., et al. Characteristics of hydatidiform moles: analysis of a prospective series with p57 immunohistochemistry and molecular genotyping. Modern Pathology. 27 (2), 238-254 (2014).
- Lipata, F., et al. Precise DNA genotyping diagnosis of hydatidiform mole. Obstetrics & Gynecology. 115 (4), 784-794 (2010).
- Buza, N., Hui, P. Partial hydatidiform mole: histologic parameters in correlation with DNA genotyping. International Journal of Gynecologic Pathology. 32 (3), 307-315 (2013).
- Fisher, R. A., et al. Frequency of heterozygous complete hydatidiform moles, estimated by locus-specific minisatellite and Y chromosome-specific probes. Human Genetics. 82 (3), 259-263 (1989).
- Murdoch, S., et al. Mutations in NALP7 cause recurrent hydatidiform moles and reproductive wastage in humans. Nature Genetics. 38 (3), 300-302 (2006).
- Parry, D. A., et al. Mutations causing familial biparental hydatidiform mole implicate c6orf221 as a possible regulator of genomic imprinting in the human oocyte. American Journal of Human Genetics. 89 (3), 451-458 (2011).
- Nguyen, N. M., Slim, R. Genetics and Epigenetics of Recurrent Hydatidiform Moles: Basic Science and Genetic Counselling. Current Obstetrics and Gynecology Reports. 3, 55-64 (2014).
- Sebire, N. J., Savage, P. M., Seckl, M. J., Fisher, R. A. Histopathological features of biparental complete hydatidiform moles in women with NLRP7 mutations. Placenta. 34 (1), 50-56 (2013).
- Nguyen, N. M., et al. Comprehensive genotype-phenotype correlations between NLRP7 mutations and the balance between embryonic tissue differentiation and trophoblastic proliferation. Journal of Medical Genetics. 51 (9), 623-634 (2014).
- Brown, L., et al. Recurrent pregnancy loss in a woman with NLRP7 mutation: not all molar pregnancies can be easily classified as either "partial" or "complete" hydatidiform moles. International Journal of Gynecologic Pathology. 32 (4), 399-405 (2013).
- Colgan, T. J., Chang, M. C., Nanji, S., Kolomietz, E. A Reappraisal of the Incidence of Placental Hydatidiform Mole Using Selective Molecular Genotyping. The International Journal of Gynecological Cancer. 26 (7), 1345-1350 (2016).
- Murphy, K. M., McConnell, T. G., Hafez, M. J., Vang, R., Ronnett, B. M. Molecular genotyping of hydatidiform moles: analytic validation of a multiplex short tandem repeat assay. The Journal of Molecular Diagnostics. 11 (6), 598-605 (2009).
転載および許可
このJoVE論文のテキスト又は図を再利用するための許可を申請します
許可を申請This article has been published
Video Coming Soon
Copyright © 2023 MyJoVE Corporation. All rights reserved