A subscription to JoVE is required to view this content. Sign in or start your free trial.
Method Article
מדידות FLIM-FRET של אינטראקציות חלבון-חלבון בחיידקים חיים.
In This Article
Summary
אנו מתארים כאן פרוטוקול לאפיון אינטראקציות חלבון-חלבון בין שני חלבונים מבוטאים בצורה שונה מאוד ב- Pseudomonas aeruginosa חי באמצעות מדידות FLIM-FRET. הפרוטוקול כולל מבני זני חיידקים, קיבוע חיידקים, הדמיה ושגרת ניתוח נתונים לאחר הדמיה.
Abstract
אינטראקציות חלבון-חלבון (PPIs) שולטות בתהליכי מפתח שונים בתאים. מיקרוסקופיית דימות אורך חיים פלואורסצנטי (FLIM) בשילוב עם העברת אנרגיה תהודה Förster (FRET) לספק מידע מדויק על PPIs בתאים חיים. FLIM-FRET מסתמך על מדידת הריקבון הנתולי של תורם FRET בכל פיקסל של תמונת FLIM, ומספק מידע כמותי ומדויק על PPIs וארגוני הסלולר המרחביים שלהם. אנו מציעים כאן פרוטוקול מפורט למדידות FLIM-FRET שהחלנו כדי לפקח על PPIs ב- Pseudomonas aeruginosa חי במקרה הספציפי של שני חלבונים אינטראקציה לידי ביטוי עם מספרי עותק שונים מאוד כדי להדגים את האיכות ואת החוסן של הטכניקה בחשיפת תכונות קריטיות של PPIs. פרוטוקול זה מתאר בפירוט את כל השלבים הדרושים לאפיון PPI - החל מבניית מוטנטים חיידקיים עד לניתוח הסופי באמצעות כלים שפותחו לאחרונה ומספקים אפשרויות ויזואליזציה מתקדמות לפרשנות פשוטה של נתוני FLIM-FRET מורכבים.
Introduction
אינטראקציות חלבון-חלבון (PPIs) לשלוט בתהליכי מפתח שוניםבתאים 1. התפקידים של PPIs שונים בהתבסס על הרכב חלבון, פונקציות זיקה ומיקומים בתאים2. PPIs ניתן לחקור באמצעות טכניקותשונות 3. לדוגמה, אימונופרציפציה הדדית היא טכניקה פשוטה יחסית, חזקה וזולה הנפוצה בזיהוי או אישור של ממשקי אבטחה. עם זאת, לימוד PPIs יכול להיות מאתגר כאשר החלבונים אינטראקציה יש רמות ביטוי נמוכות או כאשר האינטראקציות הן ארעיות או רלוונטי רק בסביבות ספציפיות. לימוד PPIs המתרחשים בין האנזימים השונים של מסלול הפיוברדין ב P. aeruginosa דורש כי הדיכוי של הדיכוי הכללי ברזל-co-factored Fur הוא הקלה כדי לאפשר את הביטוי של כל החלבונים של מסלול פיוברדין לבוא לידי ביטוי בתא 4,5,6. תקנה נפוצה זו עבור כל החלבונים של המסלול תוצאות ביטויים בזמן התא צפוי לקדם את האינטראקציות שלהם. המגוון במונח הגודל, הטבע, רמות הביטוי ומספר החלבונים במסלול מטבולי זה מקשה על המחקר במערכות משוחזרות6. חקר PPIs בסביבת התאים שלהם ולכן הוא קריטי כדי להבין עוד יותר את הפונקציות הביולוגיות של חלבונים בהקשר המקומי שלהם.
רק שיטות מעטות כולל פלואורסצנטיות מאפשרות לחקור PPIs בתאיםחיים 7. בין הפרמטרים פלואורסצנטיים שונים שניתן למדוד, החיים פלואורסצנטיים (כלומר, הזמן הממוצע פלואורופור נשאר במצב נרגש שלה לפני פליטת פוטון) הוא כנראה אחד הפרמטרים המעניינים ביותר לחקור בתאים חיים. אורך החיים הפלואורסצנטי של פלואורופור רגיש מאוד לסביבתו ולכן FLIM יכול לספק מידע כימי או פיזי לגבי סביבת הפלואורופור8. זה כולל את הנוכחות של העברת אנרגיה תהודה של פורסטר (FRET) שיכולה להתרחש בנוכחות "מקבל" של פלואורסצנטיות הממוקמת במרחק קצר של "תורם" פלואורסצנטי. העברת אנרגיה גורמת לקיצור משמעותי של משך החיים של פלואורסצנטיות התורם (איור 1A), מה שהופך את מיקרוסקופיית הדמיה לכל החיים של פלואורסצנטיות (FLIM) לגישה רבת עוצמה לחקור אינטראקציות חלבון-חלבון ישירות בתאים חיים. FLIM יכול גם לספק מידע מרחבי על איפה האינטראקציות מתרחשות בתאים7,8. גישה זו היא חזקה מאוד עבור חקירת PPIs במצבים שבהם תיוג עם פלואורופורים של שני השותפים אינטראקציה אפשרי.
כדי ש- FRET יתרחש - נדרשים תנאים קריטיים במרחק שבין שני פלואורופורים8,9. שני הפלואורופורים לא צריכים להיות רחוקים אחד מהשני ביותר מ-10 ננונום. לכן, יש לנקוט אזהרות בעת תכנון ניסויי FLIM-FRET כדי להבטיח כי התורם ומקבל הפלואורסצנטיות יש סיכוי להיות ממוקם קרוב זה לזה במתחם אינטראקציה. למרות שזה אולי נראה מגביל, זה למעשה יתרון אמיתי כמו התלות מרחוק של FRET מבטיח כי שני חלבונים מסומנים שעברו FRET צריך לקיים אינטראקציה פיזית (איור 1A). הקשיים לקבל תשובות ברורות על PPI בניסויי קולוקליזציה (שני חלבונים קולוקליזציה לא בהכרח אינטראקציה) ולכן אינם בעיה באמצעות FLIM-FRET.
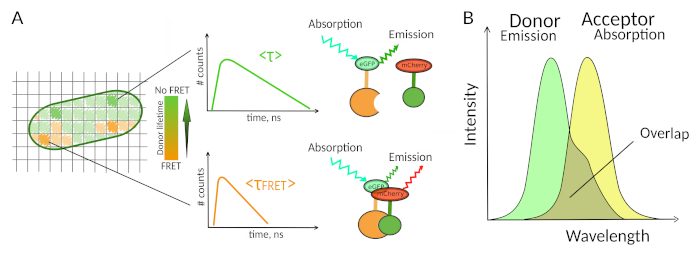
איור 1: עיקרון ניתוח FLIM-FRET. כל פיקסל בתמונה הרב-מימדית FLIM-FRET מכיל מידע על ריקבון הפלואורסצנטיות שנרשם במיקום מסוים זה (#counts = מספר הפוטונים שזוהו בערוץ t). (A)הייצוג הקלאסי של התמונה FLIM הוא בדרך כלל תמונת דו-מימד מקודדת בצבע כוזב (משמאל). ירידה בחיי הפלואורסצנטיות הממוצעים של התורם - כפי שניתן לראות על ידי שינוי בסולם הצבעים - ניתן לראות בנוכחות FRET והוא אינפורמטיבי על נוכחותם של PPIs באזור מרחבי זה. (B) חפיפה בין ספקטרום פליטת התורמים לבין ספקטרום ספיגת המקבלים נחוצה כדי ש- FRET יתרחש. אנא לחץ כאן כדי להציג גירסה גדולה יותר של איור זה.
דרישה שנייה עבור FRET היא כי ספקטרום הפליטה של התורם ואת ספקטרום הספיגה של המקבל צריךלחפוף 8 (איור 1B). העירור הפלואורסצנטי של התורם צריך להיות באורך גל שתורם מעט מאוד לרגשות הפלואורסצנטיות הישירה של המקבל. לא כל השילובים של פלואורופורים אפשריים ואנחנו ממליצים בנוסף להשתמש בתורמים עם ריקבון פלואורסצנטי מונו-ספונטני כדי להקל על פרשנויות FLIM-FRET10. מספר זוגות של חלבונים פלואורסצנטיים עומדים בדרישות אלה, כולל זוג eGFP-mCherryהפופולרי 11 (לסקירה על הצבעים של זוגות FRET חלבון פלואורסצנטי זמיןלראות 12,13).
FLIM-FRET מאפשר למדוד את הריקבון הנתולי של תורם FRET בכל פיקסל של תמונת FLIM (איור 1A). קיימות שתי טכניקות עיקריות לקביעת אורך החיים של הפלואורסצנטיות השונות ברכישה וניתוח: תחום תדירות (FD)14 ותחום זמן (TD). TD FLIM נפוץ יותר ומבוצע באמצעות תאורה פעמו בשילוב עם תצורות זיהוי אפשריות שונות כולל שיטות gating15, פסמצלמה 16 או זמן בקורלציה אחת ספירת פוטון (TCSPC)טכניקות 8. עבור טכניקות FD ו- TD, אורך החיים של הפלואורסצנטיות אינו נמדד ישירות אלא דורש ניתוח של הנתונים הנמדדים כדי להעריך את אורך החיים או את נוכחותם של אינטראקציות. עבור טכניקות TCSPC, הניתוח הנפוץ ביותר מסתמך על התאמת הריקבון עם פונקציות מעריכיות בודדות או מרובות באמצעות פחות פיתול ריבועי מחדש הממזער את הסכום המשוקלל של השאריות.
לבסוף, FLIM-FRET יכול להתבצע הן באמצעות עירור פוטון יחיד או multiphoton. האחרונה יש כמה יתרונות כמו הפחתת שפעת אוטומטית ו photodamage מתוך מישור המוקד. גירויים מרובי פוטונים מאפשרים גם עומק בעירור ארוך יותר אם עובדים בדגימות תלת-מימדעבות 8. להיפך, עירור פוטון יחיד הוא בדרך כלל יעיל יותר כמו שני פוטון ספיגת חתך של חלבונים פלואורסצנטימוגבלים 17.
כאן, אנו מציעים פרוטוקול עבור מדידות FLIM-FRET של PPIs בשידור חי P. aeruginosa במקרה מסוים של שני חלבונים אינטראקציה (PvdA ו PvdL) לידי ביטוי עם מספר שונה מאוד של עותקים כדי להדגים את האיכות ואת החוסן של הטכניקה בחשיפת תכונות קריטיות של PPIs. חלבוני PvdA ו- PvdL מעורבים בביוסינתזה של פיוברדין. PvdA הוא L-ornithine N5-oxygenase ומנסתז את L-N5-formyl-N5-hydroxyornithine מ L-ornithine על ידי הידרוקסילציה (PvdA) ו formylation (PvdF)18. PvdL הוא אנזים פפטיד לא ריבוזומלי (NRPS) המורכב מארבעה מודולים. המודול הראשון מקטלז את האצילציה של חומצה מירית. המודול השני ממזרז את ההפעלה של L-Glu ואת עיבויו ל- myristic-coA. לאחר מכן, המודול השלישי מתמצת חומצת אמינו L-Tyr כי הוא לאחר מכן isomerized ב- D-Tyr. לבסוף, המודול הרביעי קושר חומצת אמינו L-Dab (חומצה Diaminobutyric) כדי ליצור את טריפטיד acylated L-Glu / D-Tyr / L-Dab6. PvdL ולכן אחראי על הסינתזה של שלוש חומצות האמינו הראשונות של מבשר פיוברדין. האינטראקציה של חלבון PvdA עם PvdL מפתיעה מכיוון ש- PvdL, בניגוד ל- PvdI ו- PvdJ, אינו נושא מודול ספציפי עבור L-N5-formyl-N5-hydroxyornithine. אינטראקציה זו מציעה כי כל האנזימים האחראים על biosynthesis מבשר פיוברדין מסודרים במתחמים גדולים ארעיים ודינמיים רבאנזימטיים 19,20.
בדו"ח זה אנו מסבירים בפירוט כיצד לבנות את הזנים החיידקיים המבטאים באופן מקורי את שני החלבונים המסומנים eGFP ו- mCherry. אנו מתארים גם הכנה לדוגמה ותנאים להדמיית תאים יעילה של FLIM-FRET. לבסוף, אנו מציעים הדרכה שלב אחר שלב לניתוח תמונה, כולל כלי שפותח לאחרונה ומספק אפשרויות ויזואליזציה מתקדמות לפרשנות פשוטה של נתוני FLIM-FRET מורכבים. עם דו"ח זה, ברצוננו לשכנע לא רק הרפתקנים אלא רוב הביולוגים כי FRET-FLIM היא טכניקה נגישה ורבת עוצמה מסוגלת לענות על השאלות שלהם על PPIs ישירות בסביבה הסלולר המקומי.
Protocol
1. בניית פלסמיד
- הגבר על ידי שני PCR (PCR1 ו -3) את רצפי ה- DNA (השתמש ב- DNA הגנומי של P. aeruginosa PAO1) של 700 זוגות הבסיס במעלה ובמורד הזרם של האזורים המתאימים לאתר הכניסה בגנום P. aeruginosa עם פולימראז DNA נאמנות גבוהה. הוסף אתרי הגבלה לפרימרים בכחול וירוק והוסף רצף חופף עם mCherry לפרימרים באדום (איור 2).
- עבור PvdA שכותרתו ב C-terminus עם eGFP, להגביר את אזור 700 bp במעלה הזרם יחסית codon להפסיק על ידי פריימרים בכחול, להגביר את 700 bp במורד הזרם האזור המכיל את קודון להפסיק עם פריימרים בירוק.
- עבור PvdL המסומן ב- N-terminus עם mCherry, הגבר את אזור 700 bp במעלה הזרם לגן PvdL, כולל קודון ההתחלה, על-ידי פריימרים בכחול, והגדל את אזור 700 bp במורד הזרם עם פריימרים בירוק.
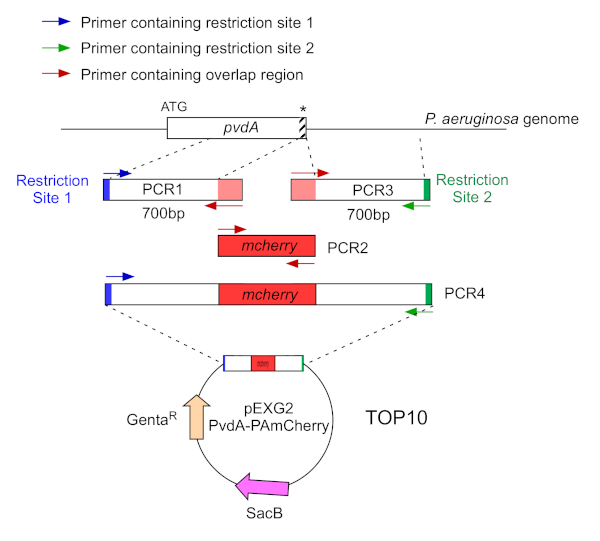
איור 2: סקירה כללית של אסטרטגיית PCR ובניית פלסמידים המשמשים לבניית PvdA-mCherry. ראה טקסט לפרטים - pvdA מקודד אנזים המעורב ביוסינתזה של פיוברדין siderophore, מטבוליט משני מעורב ברכישת ברזל. אנא לחץ כאן כדי להציג גירסה גדולה יותר של איור זה.
- הגבר את הדנ"א המקודד eGFP (ללא קודונים להתחיל ולעצור) עם פריימרים באדום עם פולימראז DNA פידליטי גבוה.
- לטהר את מוצרי PCR בעמודת ניקוי PCR (רשימת חומרים).
- ערבבו מוצרי PCR חופפים ביחס שיווי משקל ובצעו PCR שני באמצעות פריימרים עם אתר הגבלה המשמש ל-PCR 1 ו-3 (ירוק וכחול באיור 2).
- להעביר את המוצר PCR בג'ל אגרוז-1x TAE (טריס-בסיס אצטט EDTA pH 8.0), לחתוך את הלהקה המתאימה ולחלץ את האמפייקון עם ערכת ניקוי PCR(רשימת חומרים).
הערה: ניתן להשהות את הפרוטוקול כאן. - תקציר אמפייקון PCR ו pEXG2 פלסמיד באמצעות אנזימי ההגבלההמתאימים 21.
- Ligate plasmid ולהכניס עם T4 DNA ligase באמצעות 90 ננוגרם של פלסמיד ויחס מולקולרי 1:1 (plasmid:insert).
- להפוך בניית פלסמיד בתאים מוסמכים כימית E. coli TOP10 תאים על ידי ערבוב מוצר קשירה ו 100 μL של TOP10. הדגירה את תערובת חיידקים / פלסמיד מוסמך על קרח במשך 30 דקות לפני שתמשיך עם הלם חום 42 °C (60 °F) עבור 60 s. לאחר מכן, לשים את הצינור על קרח במשך 10 דקות.
- הוסף 1 מ"ל של מרק lysogeny (LB) לחיידקים דגירה ב 37 °C (60 °F) במשך 1 שעות.
- צלחת 100 μL חיידקים על אגר LB המכיל 15 מיקרוגרם / מ"ל ג'נטמיצין.
- דגירה לילה ב 37 מעלות צלזיוס.
- מסך הנוכחות של הכנס על ידי המושבה PCR: מתוך מושבה אחת שנאי מבודד, להרים כמות דקה של חיידקים להתווסף לתערובת PCR המכיל פריימרים הכלאה על פלסמיד באופן כזה נוכחות של אמפולייקון יכול להיות מזוהה על ידי הפעלת המוצר על ג'ל אגרוז (DNA פולימראז). מאותה מושבה המשמשת PCR, להעביר כמות קטנה של חיידקים על צלחת טרייה המכילה 15 מיקרוגרם / מ"ל gentamicin להיות מבודד המשמש מיצוי פלסמיד. לבסוף, לבודד ולטהר את פלסמיד (רשימת חומרים) ולאמת את ההוספה על ידי רצף.
- חנות TOP10 חיידקים המכילים את פלסמיד ב LB עם 20 % גליצרול ב 1.5 mL microtube ב -80 °C (60 °F) ואת פלסמיד מטוהר ב -20 °C (60 °F) בצינור 1.5 מ"ל.
הערה: ניתן להשהות את הפרוטוקול כאן
2. החדרת תג פלואורסצנטי לתוך הגנום הכרומוזומלי של P. aeruginosa (איור 3)
- לגדול P. aeruginosa, TOP10 ו E. coli עוזר חיידקים, כל אחד ב 5 מ"ל של LB ללא אנטיביוטיקה ב 30 °C (60 °F) תחת רעידותמסלולית לילה 22. צור החדרת תג פלואורסצנטי בגנום של P. aeruginosa על ידי העברת פלסמיד מ E. coli TOP10 לתוך זן PAO1 ושילוב הפלסמיד לתוך הגנום על ידי שילוב הומולוגי. אירוע חצייה-מעל שני החותך את הווקטור יוצר את המוטנט המתאים.
- למדוד את הצפיפות האופטית ב 600 ננומטר (OD600 ננומטר) של תרבות החיידקים ולערבב כמות שווה של P. aeruginosa (500 μL, OD600 ננומטר = 1.0) עם E. coli TOP10 pEXG2 (500 μL, OD600 ננומטר = 1.0) ו E. coli HB101 pRK600 עוזר (500 μL, OD600 ננומטר = 1.0) ב 1.5 mL microtube.
- צנטריפוגה 5 דקות ב 9,300 x גרם כדי גלולה את החיידקים.
הערה: ניתן להשתמש בכלים מקוונים כדי להמיר כוח g צנטריפוגלי לסיבוב לדקה (סל"ד) כדי להתאים את מהירות הצנטריפוגה. - שמור על גלולת החיידקים והשליך את העל-טבעי.
- תן מחדש את גלולה המכילה חיידקים ב 50 μL של LB.
- צלחת נקודה (~ 50 μL) של התערובת באמצע אגר LB (מחממים מראש ב 37 °C (69 °F) ו דגירה 5 שעות ב 37 °C (69 °F).
- לגרד את המקום עם לולאת חיסון סטרילית ולתלות מחדש ב 1 מ"ל של LB.
- צלחת 100 μL של השעיה חיידקית זו על אגר LB המכיל 10 מיקרוגרם / מ"ל כלוראמפניקול לחסל E. קולי (E. coli TOP10 pEXG2 ו E. coli HB101 pRK600 עוזר רגישים chloramphenicol אבל P. aeruginosa הוא עמיד באופן טבעי) ו 30 מיקרוגרם / מ"ל ג'נטמיצין ודגירה 2 ימים ב 37 °C (69 °F).
- Resuspend מושבה אחת ב 1 mL LB ודגירה ב 37 °C (69 °F) תחת אורביטאל רועד 4h.
- צנטריפוגה 3 דקות ב 9,300 x g ולהשליך 950 μL של על טבעי. תן את גלולה ב 50 μL של LB ולבודד את התערובת על LB אגר המכיל סוכרוז וללא NaCl.
- דגירה לילה ב 30 מעלות צלזיוס.
- ספוט מושבות מבודדות על אגר LB ו LB אגר המכיל 15 מ"ג / מ"ל גנטמיצין על מנת לבדוק את הרגישות ג'נטמיצין.
- ודא את החדרת eGFP או mCherry על ידי מושבות PCR (פולימראז DNA) וריווח באמצעות פריימרים ספציפיים.
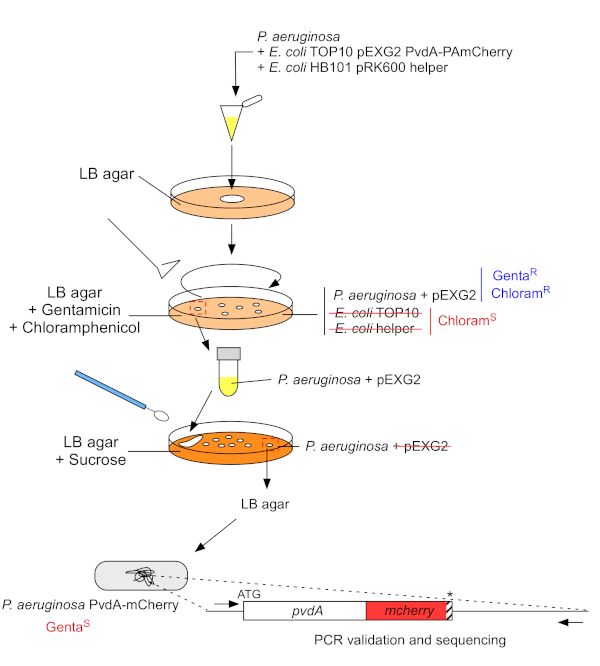
איור 3: פרוטוקול הבנייה של זני P. aeruginosa על ידי החדרת תג פלואורסצנטי. עיין בטקסט לקבלת פרטים. אנא לחץ כאן כדי להציג גירסה גדולה יותר של איור זה.
3. מדידת פיוברדין
- לגדל חיידקים ב 5 מ"ל של LB ב 30 מעלות צלזיוס תחת רעידות מסלולית לילה.
- חיידקי גלולה על ידי צנטריפוגה, לשטוף ולגדל אותם ב 5 מ"ל של SM (Succinate בינוני, הרכב: 6 g∙ L-1 K2HPO4, 3 g∙ L-1 KH2PO4, 1 g∙ L-1 (NH4)2 SO4, 0.2 גרם ∙ L-1 MgSO4, 7 H2O ו 4 g∙ L-1 נתרן succinate עם pH מותאם 7.0 על ידי הוספת NaOH) ב 30 °C (60 °F) תחת רעידות מסלולית לילה. SM הוא מדיום משוללי ברזל - היעדר ברזל יפעיל את הביטוי של החלבונים של מסלול הפיוברדין מודחק בדרך כלל בנוכחות ברזל.
- למדוד OD600 ננונום ולדלל חיידקים שוב במדיום SM טרי ב OD600 ננום = 0.1 ולגדל אותם ב 30 מעלות צלזיוס תחת רעידות מסלולית לילה.
- למדוד OD600 ננום כדי לקבוע את כמות החיידקים בכל מדגם.
- הכן קובט קוורץ המכיל 100 μL של תרבות P. aeruginosa ולהשלים עד 1 מ"ל של SM (900 μL). הכן קובט קוורץ המכיל 1 מ"ל של אמצעי SM (ריק).
- באמצעות ספקטרופוטומטר UV גלוי, למדוד את הספיגה במקסימום של שיא הספיגה. ב pH 7.0, המקסימום של ספיגה של פיוברדין יתרחש ב ~ 400 ננונום. לקבוע את ריכוז פיוברדין (טופס apo) מדגם באמצעות חוק Beer-Lambert באמצעות מקדם הכחדה טוחנת ב 400 ננוגרם של e = 19 000 M-1∙cm-1.
הערה: פיוברדין ניתן לכמת בטווח הספיגה מ ~ 0.1 עד ~ 1 (בהתאם ספקטרופוטומטר UV גלוי) שבו הספיגה ליניארית עולה עם ריכוז.
4. תרבות חיידקים ותנאים לתאים לבטא PvdA, PvdL ו- PvdJ
- ביום 1, לחסן צינור עם 5 מ"ל של LB מן המלאי גליצרול המתאים של חיידקים לגדל חיידקים במהלך הלילה ב 30 °C (60 °F) ב 200 סל"ד ב אינקובטור שייקר מסלולית.
- ביום 2, כדורי תאים על ידי צנטריפוגה ב 3,000 x גרם במשך 3 דקות ולהשליך את supernatant.
- תן שימוש חוזר בתאים ב- 10 מ"ל של SM.
- חזור על שלבים 4.2-4.3 פעם אחת ולגדל חיידקים SM לילה ב 30 °C (60 °F) 200 סל"ד.
- ביום 3, לדלל 1/10 את תרבות החיידקים ב- SM טרי.
- לגדל חיידקים מדוללים שוב בן לילה באותם תנאים.
הערה: נוכחות של פיוברדין ניתן לזהות חזותית כפי שהוא צבעים צהוב-ירוק המדיה ההולכת וגדלה. זה מראה כי הביטוי של החלבונים של מסלול פיוברדין הופעלו, כי אנזימים של עניין באים לידי ביטוי בתאים.
5. הכנת כרית אגרוז (איור 4)
- מניחים שקופית זכוכית מיקרוסקופ על משטח אופקי שטוח. סדר שתי מגלשות זכוכית ומעליהן שתי שכבות של סרט הדבקה בכל צד של השקופית הראשונית.
הערה: שמור על רווח של 1-2 מ"מ בין שלוש השקופיות המיושרות כדי למנוע מההתגלות המולטת להתפשט בסופו של דבר על השקופיות עם סרט הדבקה. - פיפטה ויוצקים טיפה של 70 μL של 1% נמס אגרוז על מגלשת הזכוכית. הוסף שקופית רביעית בחלק העליון כדי לשטח את טיפת agarose ולחץ למטה בעדינות. חכה בערך דקה.
- להוריד את השקופית העליונה ולשחרר עם פיפטה על 3 μL של חיידקים לתוך 3 עד 4 כתמים במקומות שונים על כרית אגרוז.
- מכסים בכיסוי זכוכית מיקרוסקופי (לדוגמה עובי 22x22 מ"מ #1.5).
הערה: שטוחות ועובי של כיסויים חשובים לעבודה עם עירור שני פוטון. כיסויי דיוק עם שטוחות אחידה מבוקרת ושפעת אוטומטית נמוכה הם בדרך כלל בחירה טובה. - תקן את המכסה בפרפין מומס כדי לאטום את הכיסויים על מגלשת הזכוכית. התחל על ידי תיקון ארבע הפינות של כיסוי.
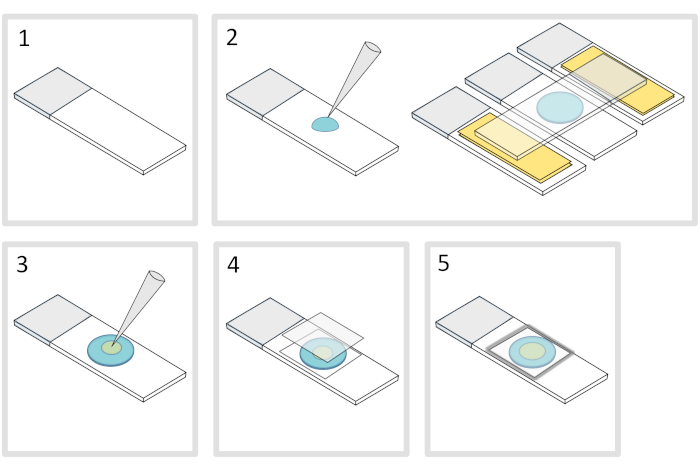
איור 4: הכנת כרית אגרוז. אנא לחץ כאן כדי להציג גירסה גדולה יותר של איור זה.
6. הדמיה עם הגדרת מיקרוסקופיה של שני פוטונים
הערה: אנו משתמשים במיקרוסקופ הפוך של סריקת עירור דו-פוטון תוצרת בית עם יעד טבילת מים 60x 1.2NA הפועל במצב איסוף פלואורסצנטיות ללא סריקה. אורך הגל של עירור שני פוטונים נקבע על 930 ננוגרם. הוא מסופק על ידי לייזר Ti:Sapphire (קצב חזרה של 80 מגה-הרץ, ≈ רוחב דופק של 70 fs) שעובד ב-10-20 מגה-ואט. פוטונים פלואורסצנטיים נאספו באמצעות מסנן מעבר קצר של 680 ננוגרם ומסנן מעבר פס של 525/50 ננוגרם לפני שהופנו לדיודה צילום מפולת סיבים המחוברת למודול ספירת פוטון יחיד (TCSPC) מתואם בזמן. המיקרוסקופ מצויד גם במנורת פלואורסצנטיות שידור. מספר מיקרוסקופים FLIM-FRET זמינים כעת מסחרית ומתקני הדמיה רבים מצוידים במערך מסוגל לבצע מדידות FLIM-FRET.
- השתמש במנורת הפלואורסצנטיות כדי למקד את המטרה במונוליאר של חיידקים בדגימה ולבחור אזורים מעניינים.
- בדוק כי תריס לייזר העירור סגור כי אור אינפרא אדום מגיע הלייזר חסום ולא להיכנס למיקרוסקופ.
זהירות: יש להקדיש תשומת לב זהירה ודריכות מתמדת לעבודה עם לייזרים פעימות IR מכיוון שאור הלייזר אינו ניתן לראות על ידי העיניים, אך כל תערוכה ישירה ואף השתקפות ישירה בלתי הפיכה או השתקפות לייזר יכולה להזיק מאוד וליצור נזקים בלתי הפיכים לעיניים. אנא עיינו בהליכי הבטיחות וההכשרה של הלייזר המקומי לפני השימוש בהגדרת מיקרוסקופיה. - מקם את החלקת המיקרוסקופיה על הבמה עם כיסויים הפונים למטרה.
- בדוק כי מנורת הפלואורסצנטיות היא על.
- סובב את צריח קוביית המסנן כדי לבחור את קוביית eGFP ולפתוח את תריס מנורת הפלואורסצנטיות.
- שלח את אור הפלואורסצנטי לכיוון העינית של המיקרוסקופ.
זהירות: ודאו שהמסנן המתאים מושלך בנתיב האור כדי להשליך אור בעירור ישיר המגיע ממנורה פלואורסצנטית שיכולה לגרום נזק לעיניים. - מקד את המטרה על חיידקים באמצעות ידית מיקרוסקופ.
- בחר אזור מעניין בדגימה על-ידי תרגום שלו באמצעות מוט ההיגוי השולט בשלב ממונע
הערה: התמקדות קלה יותר עם מדגם פלואורסצנטי מאוד המאפשר את הפלואורסצנטיות להיראות ישירות עם העיניים. - החלף את העירור עבור לייזר 2PE למדידות FLIM-FRET.
- שלח את נתיב פליטת הפלואורסצנטיות בחזרה לכיוון הגלאי.
- להחזיר את צריח קוביית המסנן כדי לבחור עבור קוביית dichroic עבור לייזר 930 ננומטריה.
- הגדר את כוח הלייזר ל-20 מגה-ואט.
- הגדר את גודל אזור העניין ל- 30 מיקרומטר. פעולה זו מתאימה את המתח המפעיל את מראות הגלבו ומגדירה את טווח תנועותיהם (איור 5).
- הפעל את הגלאי והתחל לסרוק את הדגימה - לחצני ההתחלה והעצירה השולטים בסריקה שולטים גם בפתיחה וסגירה של תריס הלייזר הן מטעמי בטיחות והן כדי להגביל את צילום הדגימה (איור 5).
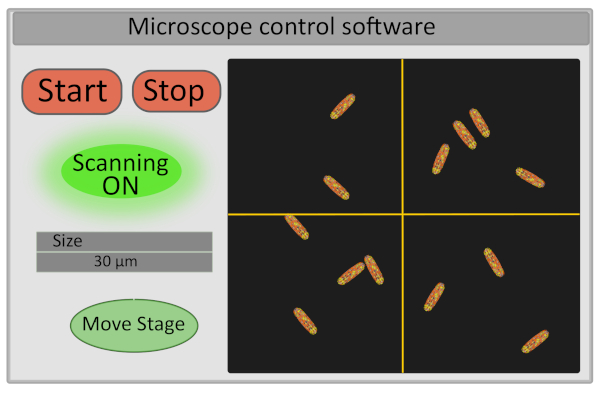
איור 5: ייצוג סכמטי של הממשק של תוכנת בקרת מיקרוסקופ. אנא לחץ כאן כדי להציג גירסה גדולה יותר של איור זה.
- במידת הצורך, התאימו את המיקוד על ידי הזזה קלה של ידית המיקוד העדינה של המיקרוסקופ.
- בחר את שדה התצוגה להדמיה על-ידי הזזה עדין של השלב מממשק המחשב. ניתן לעשות זאת בהגדרה על ידי הזזת הצלב על התמונה בתוכנת הבקרה של המיקרוסקופ (איור 5) שתגדיר את המרכז החדש של התמונה ותלחץ על הזז שלב. שדה מבט טוב לרכישה מתאים לתמונה עם 10-30 חיידקים ללא תנועה, כולם ממוקדים כראוי (כל החיידקים נמצאים באותו מישור). אם מעוניינים לחלץ תאים בודדים FLIM-FRET נתונים, להבטיח כי חיידקים הם אינדיבידואליים היטב (פילוח התמונה יהיה הרבה יותר קל).
- פתח את תוכנת SPCM (תוכנה מסחרית לרכישת נתונים) ובדוק שקצב ספירת הפוטונים אינו גבוה מדי כדי למנוע אפקט ערימה שיכול להשפיע על מדידות לכל החיים. במידת הצורך, הורידו את עוצמת הלייזר כדי לשמור על שיעור ספירת הפוטון נמוך (כ-1% מקצב החזרה על הלייזר).
הערה: אפקט הערימה מתאר את ההשפעות של פוטונים שאבדו בשיעורי ספירת פוטון גבוהים עקב הזמן המת של התקני ספירת פוטון יחידה בקורלציה בזמן (TCSPC). אם ערימה מתרחשת, אורך החיים הממוצע נמדד הופך קצר באופן מלאכותי עם מרכיב קצר יותר שיכול להופיע הריקבון בשל דגימת יתר של פוטונים פולטים במהירות. - התאם פרמטרי רכישה כולל זמן איסוף הרכש (בדרך כלל 60 s עד 180 s נדרשים כדי לאסוף מספיק פוטונים).
- לחץ על לחצן התחל והמתן להשלמת הרכישה.
- שמור את הנתונים.
- תפסיק לסרוק את הדגימה ותכבה את הגלאי.
- בחר שדה תצוגה אחר בשלבי דגימה וחזור על שלבים 6.14-6.22 או תמונה של שקופית מיקרוסקופיה חדשה על-ידי חזרה על שלבים 6.1-6.22.
הערה: P. aeruginosa יכול לחיות ולחלק עד 6-8 שעות בטמפרטורת החדר על כרית אגרוז (מתאים סוף סוף ~ 4 זמן הכפלה ב 20 מעלות צלזיוס). באופן אידיאלי, לא לחכות זמן רב מדי כדי לבצע מדידת FLIM-FRET, כדי למנוע התבוננות כרית מכוסה לחלוטין עם חיידקים.
7. ניתוח נתונים
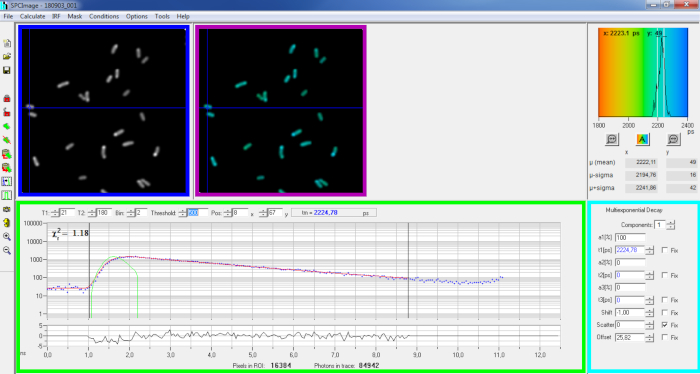
איור 6: הפאנל הראשי של חלון ניתוח הנתונים של תוכנת SPCImage. תמונת עוצמה (תיבה כחולה), תמונה לכל החיים (תיבה סגולה), היסטוגרמה לכל החיים (בפינה הימנית העליונה), עקומת ריקבון במיקום נבחר (תיבה ירוקה) ופרמטרי ריקבון במיקום נבחר (קופסת ציאן) של ריקבון PvdA-eGFP מייצג שנרשם בשידור חי P. aeruginosa באמצעות כרטיס רכישה bh SPC830 בהגדרת עירור דו-פוטון תוצרת בית-FLIM-FRET. את עקומת הריקבון הניסיונית של הפיקסל הצביעה בתמונה לעיל, ניתן לראות את התאמה מונו-מעריכית (עקומה אדומה) המפתלת את הריקבון מתפקוד התגובה האינסטרומנטלית המחושבת שלו (עקומה ירוקה) בלוח הירוק. אנא לחץ כאן כדי להציג גירסה גדולה יותר של איור זה.
- ניתוח בסיסי
- הפעל את תוכנת SPCImage.
- יבא את קובץ SPCM שנשמר. תמונת העוצמה מוצגת בלוח השמאלי העליון של התוכנה ( איור6 תיבה כחולה).
- בחן את חלון עקומת הריקבון (איור 6 תיבה ירוקה) המציג את נתוני הריקבון המתאימים לפיקסל שנבחר בתדמית העוצמתית (איור 6 תיבה כחולה). מספרי הפוטון של כל ערוץ זמן מוצגים כנקודות כחולות וההתקף של הריקבון מצויר כקו אדום. שים לב שלאחר טעינת הנתונים התוכנה מציגה את הפיקסל הבהיר ביותר של התמונה. הזיזו את הצלב הכחול לאורך התמונה כדי לבחון פיקסלים בעוצמה נמוכה יותר. חלון הריקבון יתרענן באופן אוטומטי בכל מיקום פיקסל חדש.
הערה: מדידת פונקציית התגובה האינסטרומנטלית (IRF) של מערכת סריקת לייזר היא קשה מאוד. IRF המחושב מהקצה העולה של עקומות ריקבון הפלואורסצנטיות בנתונים FLIM יכול לשמש לפירוק ריקבון. זוהי האפשרות שנעשית כברירת מחדל ב- SPCimage ( איור6 עקומה ירוקה). - כוונן את טווח ההתאמה על-ידי הזזת ערוצי ההתחלה והסיום של תיבת ההתאמה (T1 ו- T2 בתיבה הירוקה). T1 צריך להתחיל בערוצים הראשונים של הריקבון העולה T2 להגדיר את הערוץ האחרון בסוף הריקבון והוא יכול להיבחר כאחד הערוצים האחרונים של הריקבון עם מספר פוטונים מעל ספירת הפוטונים לקזז (כלומר, רמות הפוטונים נספרים לפני הריקבון עולה).
- בחר את הערך binning על-ידי שינוי הערך Bin. ריקבון העקומה משלב את ספירת הפוטונים של הפיקסל הנבחר יחד עם אזור של i פיקסלים סביב מיקום הסמן המוגדר על-ידי הפרמטר bin (הגדלת binning יגדיל את מספר הפוטונים בריקבון עשוי להיות מועיל להגיע ספירת פוטון הנדרש עבור מודלים מעריכיים מרובים).
- התאם את ערך הסף. פיקסלים ללא ערוץ אחד לפחות עם מספר פוטונים גבוהים פיקסלים סף לא ייכללו בהליך ההתאמה. כמובן ככל שמספר הפיקסלים שיתאים גבוה יותר, כך הניתוח ארוך יותר.
הערה: נתוני FLIM יכולים להכיל מספר עצום של פיקסלים וערוצי זמן. הגרסאות האחרונות של התוכנה מאפשרות שימוש במעבד הגרפי (יחידת מעבד גרפי) לעיבוד מספר רב של פיקסלים במקביל, מה שמפחית באופן מאסיבי את זמני העיבוד. זה יכול להיות מעניין להתאים את הפרמטרים binning ואת הסף באמצעות תמונות המתאימות מבני חיידקים המציגים את עוצמת הפלואורסצנטיות הנמוכה ביותר (למשל, עם זנים חיידקיים עם רמות הביטוי הנמוכות ביותר). זה יבטיח כי הריקבון הרלוונטי שנצפה בדגימות אלה יעמוד בקריטריוני הסינון וייכללו בניתוח. לאחר מכן ניתן להשתמש בפרמטרים אלה עבור כל התמונות. - התאימו, במידת הצורך, את פרמטרי הריקבון (קופסת ציאן). תן למשמרת להשתנות, רוב הזמן להתפזר ולקזז ניתן לתקן לאפס אם מבט על פונקציות ריקבון להראות כי תרומתם היא זניחה. ניתן להערך את הקיזוז בהתבוננות בערוצים הראשונים של הריקבון - שים לב כי הדמיה במשך זמן רב עקב פלואורסצנטיות נמוכה המדגם בדרך כלל גורמת לקיזוז שאינו אפס. פיזור מתרחש בעיקר בדגימות עבות והוא יכול להיחשב זניח אחרת.
- לפני הפעלת ההתאמה, בחר את אלגוריתם ההתאמה. פתח את חלון הגדרות האלגוריתם באפשרויות הצג/הסתר מודל. בחר אלגוריתם הערכת סבירות מרבית (MLE) (איור 7A).
- הפעל את התאמת התמונה על-ידי לחיצה על | מטריצת ריקבון. לאחר השלמתה, תמונת FLIM המקודדת לכל החיים מופיעה בחלונית 'תמונה לכל החיים'(איור 6 סגול).
הערה: בחלון עקומת הריקבון(איור 6 ירוק) ניתן לראות את ערך אורך החיים המתאים לכל פיקסל בתמונה על-ידי הזזת הצלב הכחול.
הערה: כדי לעבד מספר גדול של קבצי נתונים דומים של FLIM באופן אוטומטי, ניתן להשתמש במצב עיבוד אצווה. - בדוק את איכות מתאים מסתכל על שאריות (מחולק באופן אידיאלי באופן אקראי סביב 0) וערך ריבוע צ'י קרוב 1.
- ניתן לייצא נתונים מותאמים בתבניות שונות. כדי לייצא קבצים בקבצי txt, עבור אל | יצא . בחלון אפשרויות ייצוא ( איור7B), בחרו 'בחר הכל' ולחצו על 'ייצוא' .
- לבסוף שמור את קובץ הניתוח. קבצי ניתוח נשמרים כקבצי *.img ו ניתן לפתוח אותם מחדש ישירות ב- SPCImage.
הערה: במקרים מסוימים של כמויות לא מאוזנות של תורמים/מקבלים, FLIM-FRET יכול לחשוף תת-אוכלוסיות בתערובת של קומפלקסים של חלבונים אינטראקציה - במיוחד כאשר הריכוזים של שני בני הזוג שונים מאוד, וכתוצאה מכך תערובות של מינים מורכבים וחופשיים. מינים שאינם אינטראקציה (המאופיינים בדעיכה הדומה מאוד לריקבון רק התורם) יכולים להיות מופלים מאלה המקיימים אינטראקציה בהנחה של קבועות מרחבית של רכיבי החיים של התורם על פני ערכת הנתונים. באופן דומה, תסביך אינטראקציה לא סטואיכומטרי עם תורמים נוספים או מקבל יותר של פלואורסצנטיות עשוי להיווצר. את הריקבון הפלואורסצנטי של מתחמים כאלה קשה בדרך כלל לפרש. ניתן להשתמש בעלילה של דיאגרמת FLIM כדי לספק מידע קריטי אודות מצב סטואיכימטריה ואיגוד של PPIs20,23. התוויית דיאגרמת FLIM היא ייצוג גרפי של הרכיב הקצר ביותר לכל החיים כפונקציה של משרעתו. ניתן להשתמש בו כדי להמחיש פיקסלים עם חתימות ריקבון דומות. כדי לצייר ייצוגים כאלה, ריקבון פלואורסצנטי ניסיוני צריך להיות מצויד עם מודל מעריכי שני. השלבים הבאים יכולים להיות מדריך לאורך תהליך זה. - התחל על ידי ניתוח הנתונים של הבנייה רק התורם. זה יאפשר לקבוע את הערך לכל החיים של התורם. באופן אידיאלי, למדוד ערך זה על פני מספר תמונות שנרשמו באותם תנאים כמו מבנים התורם / מקבל כדי לאחזר ערך חיים חזק עבור התורם.
- לאחר שנקבע, להתאים את ריקבון פלואורסצנטי של מבנים תורם / מקבל עם מודל שני מעריכי. בתיבת הפרמטר דעיכה של ציאן( איור 6), הגדר את מספר הרכיבים ל- 2. תקן פרמטר t2(ps) לערך החיים החזק של התורם שנקבע בשלב 1 וסמן את התיבה כדי לתקן פרמטר זה.
הערה: חשוב לתקן את החיים ארוכים τ2 על מנת להגביל את התאמת יתר, כדי לשפר את התכנסות ההתאמה, ולקבל פרמטרים אמינים יותר התאמה דומעריכית 24,25,26. - שמור את *img קובץ וייצא נתונים כמו *.asc קבצים כמו בשלב 7.1.11.
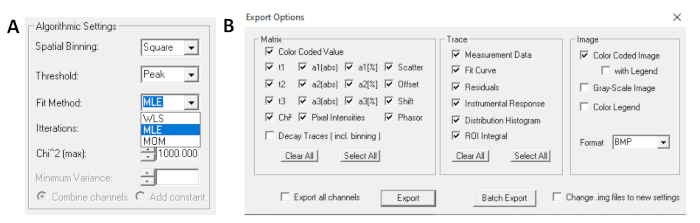
איור 7: (A) הגדרות אלגוריתם להתאמת הריקבון במודלים מעריכיים. בחירת MLE (אלגוריתם סבירות מרבית או הערכת סבירות מרבית, MLE) כמודל התאמה ו- (B) חלון אפשרויות ייצוא. אנא לחץ כאן כדי להציג גירסה גדולה יותר של איור זה.
- ניתוח מתקדם של תמונות FLIM ב- R
- התקן R (https://cran.r-project.org) ו- RStudio (https://rstudio.com) במידת הצורך.
- פתח את RStudio וצור פרוייקט חדש.
- העבר את כל הקובץ analysis *.asc בתיקיה הנקראת "נתונים" בתיקיה הראשית של הפרוייקט.
- פתח קובץ Script חדש (או פתח את קובץ ה- Script FLIM_analysis. ר').
- התקן את חבילת flimDiagRam הייעודית לניתוח נתונים https://github.com/jgodet/flimDiagRam. התקשר לחבילה בסביבת העבודה. (ראה את ההודעה HowTo_FlimDiagRam)
הערה: התקנת חבילות צריכה לעשות פעם אחת בלבד. לאחר ההתקנה, ניתן להתקשר לחבילות מכל הפעלת R חדשה. הורדת חבילות R מ-github דורשת התקנת חבילת 'devtools'. ההתקנה של 'devtools' יכול לקחת כמה דקות. חבילת flimDiagRam יכולה לשמש לייצוג הפרמטרים וההפצות של נתוני FLIM, לחילוץ נתוני FLIM ברמה של תאים בודדים מותאמים אישית, להשוואת תוצאות FLIM בין תנאים או זנים ולחקור נתוני FLIM באמצעות כלי הדמיה מתקדמים כמו התוויית דיאגרמת FLIM. - השתמש בקוד עם הערות שלב אחר שלב והנתונים זמינים כדי לשחזר באופן עצמאי את כל נתוני המשנה המוצגים בסעיף תוצאות מייצגות להלן. ערכת לימוד זו ניתן למצוא בהודעה HowTo_FlimDiagRam https://github.com/jgodet/flimDiagRam/blob/master/HowTo.pdf. ניתן לבצע חילוץ קל של הקוד כדי לנתח את הנתונים.
תוצאות
פונקציות התפלגות מצטברות אמפיריות (ecdf) של אורך החיים הפלואורסצנטי הנמדד עבור זנים חיידקיים שונים מוצגות איור 8. אם FRET מתרחשת, ecdfs מועברים לכיוון אורך החיים קצר יותר (איור 8A,8B). שים לב שכאשר האינטראקציה של שני החלבונים גורמת למרחקים ארוכים בין שני הפל...
Discussion
FLIM-FRET מציע כמה יתרונות מרכזיים על פני הדמיית FRET מבוססת עוצמה. חיים פלואורסצנטיים הם פרמטר מהותי של הפלואורופור. כתוצאה מכך, זה לא תלוי בריכוזים מקומיים של פלואורופורים ולא בעוצמת העירור האור. אורך החיים של הפלואורסצנטיות מושפע גם הוא בצורה גרועה מהלבנת תמונות. מעניין במיוחד לראות PPIs בתאים...
Disclosures
למחברים אין מה לחשוף.
Acknowledgements
אנו מודים לד"ר לודוביק ריצ'רט על עזרתו החשובה ברכישת נתוני FLIM ועל התחזוקה הטכנית והפיתוח של מערך FLIM. עבודה זו מומנה על ידי מענקים מפונדציה לשפוך la Recherche en Chimie (https://icfrc.fr/). VN ממומן על ידי פוביציה לשפוך la Recherche Médicale (FRM‐SPF201809006906). YM אסיר תודה למכון האוניברסיטאי של צרפת (IUF) על התמיכה ומתן זמן נוסף להקדשה למחקר. IJS ו- JG מודים למכון לאספקת סמים של שטרסבורג על תמיכתו הכספית.
Materials
| Name | Company | Catalog Number | Comments |
| 525/50 nm band-pass filter | F37-516, AHF, Germany | ||
| 680 nm short pass filter | F75-680, AHF, Germany | ||
| Agarose | Sigma-Aldrich | A9539 | |
| Ammonium Sulfate (NH4)2SO4 | Sigma-Aldrich | A4418 | |
| DreamTaq DNA polymerase 5U/μL | ThermoFisher Scientific | EP0714 | |
| E. coli TOP10 | Invitrogen | C404010 | |
| Fiber-coupled avalanche photo-diode | SPCM-AQR-14- FC, Perkin Elmer | ||
| Glass coverslips (Thickness No. 1.5, 20×20mm | Knitel glass | MS0011 | |
| High-Fidelity DNA polymerase Phusion 2U/μL | ThermoFisher Scientific | F530S | |
| Lysogeny broth (LB) | Millipore | 1.10285 | |
| Magnesium Sulfate Heptahydrate (MgSO4 . 7H2O) | Sigma-Aldrich | 10034-99-8 | |
| Microscope slides (25×75mm) | Knitel glass | MS0057 | |
| NucleoSpin Gel and PCR Clean-up | Macherey-Nagel | 740609.50 | |
| NucleoSpin Plasmid | Macherey-Nagel | 740588.10 | |
| Potassium Phosphate Dibasic (K2HPO4) | Sigma-Aldrich | RES20765 | |
| Potassium Phosphate Monobasic (KH2PO4) | Sigma-Aldrich | P5655 | |
| Sodium Succinate (Disodium) | Sigma-Aldrich | 14160 | |
| SPCImage, SPCM software | Becker & Hickl | ||
| Sterile inoculating loop | Nunc | 7648-1PAK | |
| T4 DNA ligase 1U/μL | ThermoFisher Scientific | 15224017 | |
| TCSPC module | SPC830, Becker & Hickl, Germany | ||
| Ti:Sapphire laser | Insight DeepSee, Spectra Physics | ||
| Tubes 50mL | Falcon | 352070 |
References
- Braun, P., Gingras, A. C. History of protein-protein interactions: From egg-white to complex networks. Proteomics. 12, 1478-1498 (2012).
- Nooren, I. M. A., Thornton, J. M. Structural characterisation and functional significance of transient protein-protein interactions. Journal of Molecular Biology. 325, 991-1018 (2003).
- Hayes, S., Malacrida, B., Kiely, M., Kiely, P. A. Studying protein-protein interactions: Progress, pitfalls and solutions. Biochemical Society Transactions. 44, 994-1004 (2016).
- Guillon, L., Altenburger, S., Graumann, P. L., Schalk, I. J. Deciphering protein dynamics of the siderophore pyoverdine pathway in Pseudomonas aeruginosa. PLoS ONE. 8, 1-9 (2013).
- Ringel, M. T., Brüser, T. The biosynthesis of pyoverdines. Microbial Cell. 5, 424-437 (2018).
- Schalk, I. J., Rigouin, C., Godet, J. An overview of siderophore biosynthesis among fluorescent Pseudomonads and new insights into their complex cellular organization. Environmental Microbiology. 22, 1447-1466 (2020).
- Cui, Y., et al. Techniques for detecting protein-protein interactions in living cells: principles, limitations, and recent progress. Science China Life Sciences. , (2019).
- Day, R. N., Mazumder, N., Sun, Y., Christopher, K. G. FRET microscopy: Basics, issues and advantages of FLIM-FRET imaging. Springer Series in Chemical Physics. 111, 249-276 (2015).
- Bastiaens, P. I. H., Squire, A. Fluorescence lifetime imaging microscopy: Spatial resolution of biochemical processes in the cell. Trends in Cell Biology. 9, 48-52 (1999).
- Yasuda, R. Imaging spatiotemporal dynamics of neuronal signaling using fluorescence resonance energy transfer and fluorescence lifetime imaging microscopy. Current Opinion in Neurobiology. 16, 551-561 (2006).
- Tramier, M., Zahid, M., Mevel, J. -. C., Masse, M. -. J., Coppey-Moisan, M. Sensitivity of CFP/YFP and GFP/mCherry Pairs to Donor Photobleaching on FRET Determination by Fluorescence Lifetime Imaging Microscopy in Living Cells. Microscopy Research and Technique. 71, 146-157 (2008).
- Bajar, B. T., Wang, E. S., Zhang, S., Lin, M. Z., Chu, J. A guide to fluorescent protein FRET pairs. Sensors (Switzerland). 16, 1-24 (2016).
- Piston, D. W., Kremers, G. J. Fluorescent protein FRET: the good, the bad and the ugly. Trends in Biochemical Sciences. 32, 407-414 (2007).
- Leray, A., et al. Optimized protocol of a frequency domain fluorescence lifetime imaging microscope for fret measurements. Microscopy Research and Technique. 72, 371-379 (2009).
- Elson, D. S., et al. Real-time time-domain fluorescence lifetime imaging including single-shot acquisition with a segmented optical image intensifier. New Journal of Physics. 6, 1-13 (2004).
- Rajoria, S., Zhao, L., Intes, X., Barroso, M. FLIM-FRET for Cancer Applications. Current Molecular Imaging. 3, 144-161 (2014).
- Drobizhev, M., Makarov, N. S., Tillo, S. E., Hughes, T. E., Rebane, A. Two-photon absorption properties of fluorescent proteins. Nature Methods. 8, 393-399 (2011).
- Visca, P., Ciervo, A., Orsi, N. Cloning and nucleotide sequence of the pvdA gene encoding the pyoverdin biosynthetic enzyme L-ornithine N5-oxygenase in Pseudomonas aeruginosa. Journal of Bacteriology. 176, 1128-1140 (1994).
- Imperi, F., Visca, P. Subcellular localization of the pyoverdine biogenesis machinery of Pseudomonas aeruginosa: A membrane-associated 'siderosome'. FEBS Letters. 587, 3387-3391 (2013).
- Gasser, V., et al. In cellulo FRET-FLIM and single molecule tracking reveal the supra-molecular organization of the pyoverdine bio-synthetic enzymes in Pseudomonas aeruginosa. Quarterly Reviews of Biophysics. , 1-11 (2019).
- Rietsch, A., Mekalanos, J. J. Metabolic regulation of type III secretion gene expression in Pseudomonas aeruginosa. Molecular Microbiology. 59, 807-820 (2006).
- Herrero, M., De Lorenzo, V., Timmis, K. N. Transposon vectors containing non-antibiotic resistance selection markers for cloning and stable chromosomal insertion of foreign genes in gram-negative bacteria. Journal of Bacteriology. 172, 6557-6567 (1990).
- Godet, J., Mély, Y. Exploring protein-protein interactions with large differences in protein expression levels using FLIM-FRET. Methods and Applications in Fluorescence. 8, 014007 (2019).
- El Meshri, S. E., et al. Role of the nucleocapsid domain in HIV-1 gag oligomerization and trafficking to the plasma membrane: A fluorescence lifetime imaging microscopy investigation. Journal of Molecular Biology. 427, 1480-1494 (2015).
- Becker, W. The bh TCSPC Handbook. Scanning. , 1 (2010).
- Richert, L., Didier, P., de Rocquigny, H., Mély, Y. Monitoring HIV-1 protein oligomerization by FLIM FRET microscopy. Springer Series in Chemical Physics. , 111 (2015).
- Fereidouni, F., Blab, G. A., Gerritsen, H. C. Phasor based analysis of FRET images recorded using spectrally resolved lifetime imaging. Methods and Applications in Fluorescence. 2, (2014).
- Fereidouni, F., Gorpas, D., Ma, D., Fatakdawala, H., Marcu, L. Rapid fluorescence lifetime estimation with modified phasor approach and Laguerre deconvolution: a comparative study. Methods and Applications in Fluorescence. 5, 035003 (2017).
- Margineanu, A., et al. Screening for protein-protein interactions using Förster resonance energy transfer (FRET) and fluorescence lifetime imaging microscopy (FLIM). Scientific Reports. 6, (2016).
- Guzmán, C., Oetken-Lindholm, C., Abankwa, D. Automated High-Throughput Fluorescence Lifetime Imaging Microscopy to Detect Protein-Protein Interactions. Journal of Laboratory Automation. 21, 238-245 (2016).
- Liu, W., Cui, Y., Ren, W., Irudayaraj, J. Epigenetic biomarker screening by FLIM-FRET for combination therapy in ER+ breast cancer. Clinical Epigenetics. 11, 1-9 (2019).
- Liu, X., et al. Fast fluorescence lifetime imaging techniques: A review on challenge and development. Journal of Innovative Optical Health Sciences. 12, 1-27 (2019).
- Padilla-Parra, S., Auduge, N., Coppey-Moisan, M., Tramier, M. Non fitting based FRET-FLIM analysis approaches applied to quantify protein-protein interactions in live cells. Biophysical Reviews. 3, 63-70 (2011).
- Padilla-Parra, S., Audugé, N., Coppey-Moisan, M., Tramier, M. Quantitative FRET analysis by fast acquisition time domain FLIM at high spatial resolution in living cells. Biophysical Journal. 95, 2976-2988 (2008).
- Stringari, C., et al. Phasor approach to fluorescence lifetime microscopy distinguishes different metabolic states of germ cells in a live tissue. Proceedings of the National Academy of Sciences of the United States of America. 108, 13582-13587 (2011).
- Digman, M. A., Caiolfa, V. R., Zamai, M., Gratton, E. The phasor approach to fluorescence lifetime imaging analysis. Biophysical Journal. 94, 14-16 (2008).
- Liang, Z., Lou, J., Scipioni, L., Gratton, E., Hinde, E. Quantifying nuclear wide chromatin compaction by phasor analysis of histone Förster resonance energy transfer (FRET) in frequency domain fluorescence lifetime imaging microscopy (FLIM) data. Data in Brief. 30, 105401 (2020).
- Grimm, J. B., Heckman, L. M., Lavis, L. D. The chemistry of small-molecule fluorogenic probes. Progress in Molecular Biology and Translational Science. 113, (2013).
- Li, L., Sun, H. Next Generation of Small-Molecule Fluorogenic Probes for Bioimaging. Biochemistry. 59, 216-217 (2020).
- Yao, R., Ochoa, M., Yan, P., Intes, X. Net-FLICS: fast quantitative wide-field fluorescence lifetime imaging with compressed sensing - deep learning approach. Light: Science and Applications. 8, 1-7 (2019).
- Smith, J. T., et al. Fast fit-free analysis of fluorescence lifetime imaging via deep learning. Proceedings of the National Academy of Sciences of the United States of America. 116, 24019-24030 (2019).
- Yao, R., Ochoa, M., Intes, X., Yan, P. Deep compressive macroscopic fluorescence lifetime imaging. Proceedings - International Symposium on Biomedical Imaging. 2018, 908-911 (2018).
Reprints and Permissions
Request permission to reuse the text or figures of this JoVE article
Request PermissionThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. All rights reserved