È necessario avere un abbonamento a JoVE per visualizzare questo. Accedi o inizia la tua prova gratuita.
Method Article
Misurazioni FLIM-FRET delle interazioni proteina-proteina nei batteri vivi.
In questo articolo
Riepilogo
Descriviamo qui un protocollo per caratterizzare le interazioni proteina-proteina tra due proteine espresse in modo molto diverso nelle Pseudomonas aeruginosa vive usando misurazioni FLIM-FRET. Il protocollo include costruzioni di ceppo batterico, immobilizzazione dei batteri, routine di analisi dei dati di imaging e post-imaging.
Abstract
Le interazioni proteina-proteina (IPP) controllano vari processi chiave nelle cellule. La microscopia a vita a fluorescenza (FLIM) combinata con il trasferimento di energia di risonanza di Förster (FRET) fornisce informazioni accurate sugli IPP nelle cellule vive. FLIM-FRET si basa sulla misurazione del decadimento a vita della fluorescenza di un donatore FRET ad ogni pixel dell'immagine FLIM, fornendo informazioni quantitative e accurate sugli IPP e sulle loro organizzazioni cellulari spaziali. Proponiamo qui un protocollo dettagliato per le misurazioni FLIM-FRET che abbiamo applicato per monitorare gli IPP nelle Pseudomonas aeruginosa vive nel caso particolare di due proteine interagenti espresse con numeri di copia molto diversi per dimostrare la qualità e la robustezza della tecnica nel rivelare le caratteristiche critiche degli IPP. Questo protocollo descrive in dettaglio tutti i passaggi necessari per la caratterizzazione PPI, a partire dalle costruzioni mutanti batteriche fino all'analisi finale utilizzando strumenti sviluppati di recente che forniscono possibilità di visualizzazione avanzate per un'interpretazione diretta di dati FLIM-FRET complessi.
Introduzione
Le interazioni proteina-proteina (IPP) controllano vari processi chiave nellecellule 1. I ruoli degli IPP differiscono in base alla composizione proteica, alle funzioni di affinità e alle posizioni nellecellule 2. Gli IPP possono essere studiati attraverso diverse tecniche3. Ad esempio, la co-immunoprecipitazione è una tecnica relativamente semplice, robusta ed economica comunemente utilizzata per identificare o confermare gli IPP. Tuttavia, studiare gli IPP può essere impegnativo quando le proteine interagenti hanno bassi livelli di espressione o quando le interazioni sono transitori o rilevanti solo in ambienti specifici. Lo studio degli IPP che si verificano tra i diversi enzimi della via della pioverdina in P. aeruginosa richiede che la repressione del repressore generale del ferro-co-fattorizzato Fur sia alleviata per consentire l'espressione di tutte le proteine della via della pioverdina da esprimere nellacella 4,5,6. Questa regolazione comune per tutte le proteine del percorso si traduce in espressioni tempestive nella cellula che dovrebbero promuovere le loro interazioni. La diversità in termini di dimensioni, natura, livelli di espressione e il numero di proteine di questa via metabolica rendono difficile lo studio nei sistemi ricostituiti6. Esplorare gli IPP nel loro ambiente cellulare è quindi fondamentale per comprendere ulteriormente le funzioni biologiche delle proteine nel loro contesto nativo.
Solo pochi metodi, tra cui la fluorescenza, consentono di esplorare gli IPP nelle celluleviventi 7. Tra i diversi parametri di fluorescenza che possono essere misurati, la durata della fluorescenza (cioè il tempo medio in cui un fluorofore rimane nel suo stato eccitato prima di emettere un fotone) è probabilmente uno dei parametri più interessanti da esplorare nelle cellule viventi. La durata della fluorescenza di un fluorofo è altamente sensibile al suo ambiente e FLIM può quindi fornire informazioni chimiche o fisiche sull'ambiente del fluoroforo8. Ciò include la presenza di trasferimento di energia di risonanza di Förster (FRET) che può verificarsi in presenza di un "accettore" di fluorescenza situato a breve distanza da un "donatore" di fluorescenza. Il trasferimento di energia si traduce in un significativo accorciamento della durata della fluorescenza del donatore (Figura 1A), rendendo la microscopia a vita a fluorescenza (FLIM) un potente approccio per esplorare le interazioni proteina-proteina direttamente nelle cellule vive. FLIM può inoltre fornire informazioni spaziali su dove si svolgono le interazioni nelle celle7,8. Questo approccio è estremamente potente per indagare gli IPP in situazioni in cui è possibile l'etichettatura con fluorofori dei due partner interagenti.
Affinché fret si verifichi - sono necessarie condizioni critiche sulla distanza tra due fluorofori8,9. I due fluorofori non dovrebbero essere distanti l'uno dall'altro di oltre 10 nm. Pertanto, è necessario prestare attenzione quando si progettano esperimenti FLIM-FRET per garantire che il donatore e l'accettore di fluorescenza abbiano la possibilità di trovarsi uno vicino all'altro nel complesso interagente. Anche se questo può sembrare vincolante, è in realtà un vero vantaggio in quanto la dipendenza dalla distanza del FRET garantisce che due proteine marcate sottoposte a FRET mentono interagiscano fisicamente (Figura 1A). Le difficoltà di ottenere risposte chiare sul PPI negli esperimenti di colocalizzazione (due proteine colocalizzate potrebbero non interagire necessariamente) non sono quindi un problema usando FLIM-FRET.
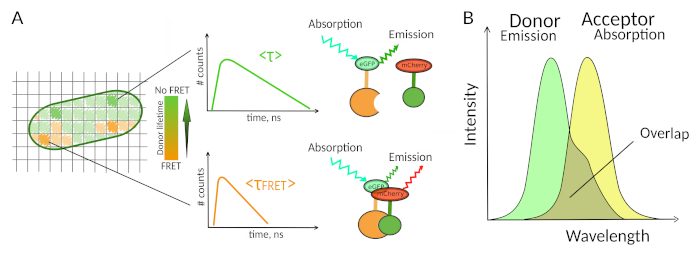
Figura 1: Principio di analisi FLIM-FRET. Ogni pixel dell'immagine multidimensionale FLIM-FRET contiene informazioni sul decadimento della fluorescenza registrato in questa particolare posizione (#counts = numero di fotoni rilevati nel canale t). (A) La rappresentazione classica dell'immagine FLIM è in genere un'immagine 2D codificata a vita di falsi colori (a sinistra). Una diminuzione della durata media della fluorescenza del donatore - come visto da un cambiamento nella scala dei colori - può essere osservata in presenza di FRET ed è informativa sulla presenza di IPP in quest'area spaziale. (B) La sovrapposizione tra lo spettro di emissione del donatore e lo spettro di assorbimento dell'accettore è necessaria affinché il FRET si verifichi. Clicca qui per visualizzare una versione più grande di questa figura.
Un secondo requisito per il FRET è che lo spettro di emissione del donatore e gli spettri di assorbimento dell'accettore si sovrapponganoa 8 (figura 1B). L'eccitazione a fluorescenza del donatore dovrebbe essere a lunghezze d'onda che contribuiscono molto poco all'eccitazione diretta della fluorescenza dell'accettore. Non tutte le combinazioni di fluorofori sono possibili e si consiglia inoltre di utilizzare preferenzialmente donatori con decadimenti di fluorescenza monoesonziali per facilitare le interpretazioni FLIM-FRET10. Diverse coppie di proteine a fluorescenza soddisfano questi requisiti, tra cui la popolare coppia eGFP-mCherry11 (per una recensione sulla tavolozza delle coppie fret proteiche fluorescenti disponibilivedi 12,13).
FLIM-FRET consente di misurare il decadimento a vita della fluorescenza di un donatore FRET ad ogni pixel di un'immagine FLIM (Figura 1A). Esistono due tecniche principali per determinare la durata della fluorescenza che differiscono nell'acquisizione e nell'analisi: il dominio della frequenza (FD)14 e il dominio del tempo (TD). TD FLIM è più diffuso e viene eseguito utilizzando un'illuminazione pulsata combinata con diverse possibili configurazioni di rilevamento tra cui i metodi di gating15,la fotocamera astrisce 16 o le tecniche di conteggio dei fotoni singoli correlate al tempo (TCSPC)8. Sia per le tecniche FD che TD, la durata della fluorescenza non viene misurata direttamente, ma richiede un'analisi dei dati misurati per stimare la durata o la presenza di interazioni. Per le tecniche TCSPC, l'analisi più utilizzata si basa sull'adattamento dei decadimenti con funzioni singole o multie esponenziali utilizzando ri-convoluzioni iterativi meno quadrate che riducono al minimo la somma ponderata dei residui.
Infine, FLIM-FRET può essere eseguito sia utilizzando singole eccitazioni fotone o multifotoni. Gli ultimi hanno diversi vantaggi come ridurre l'autofluorescenza e il danno fotografico dal piano focale. Le eccitazioni multifotono consentono anche una maggiore profondità di eccitazione se si lavora in campioni 3D spessi8. Al contrario, l'eccitazione di fotoni singoli è solitamente più efficiente in quanto le sezioni trasversali di assorbimento dei due fotoni delle proteine fluorescentisono limitate 17.
Qui proponiamo un protocollo per le misurazioni FLIM-FRET degli IPP in P. aeruginosa vivo nel caso particolare di due proteine interagenti (PvdA e PvdL) espresse con un numero molto diverso di copie per dimostrare la qualità e la robustezza della tecnica nel rivelare le caratteristiche critiche degli IPP. Le proteine PvdA e PvdL sono coinvolte nella biosintesi della pioverdina. La PvdA è una L-ornitina N5-ossigenasi e sintetizza l'L-N5-formil-N5-idrossiornitina da L-ornitina per idrossilazione (PvdA) e formilazione (PvdF)18. Il PvdL è un enzima di sintesi peptidica non ribosomiale (NRPS) composto da quattro moduli. Il primo modulo catalizza l'acilazione dell'acido mirmistico. Il secondo modulo catalizza l'attivazione di L-Glu e la sua condensa alla myristic-coA. Quindi, il terzo modulo condensa un amminoacido L-Tyr che viene poi isomerizzato in D-Tyr. Infine, il quarto modulo lega un amminoacido L-Dab (acido diaminobutirrico) per formare il tripeptide acilato L-Glu/D-Tyr/L-Dab6. PvdL è quindi responsabile della sintesi dei tre primi amminoacidi del precursore della pioverdina. L'interazione della proteina PvdA con PvdL è sorprendente in quanto PvdL, al contrario di PvdI e PvdJ, non porta un modulo specifico per l'L-N5-formil-N5-idrossiornitina. Questa interazione suggerisce che tutti gli enzimi responsabili della biosintesi precursore della pioverdina sono disposti in grandi complessi multienzimatici transitori e dinamici19,20.
In questa relazione spieghiamo in dettaglio come costruire i ceppi batterici esprimendo nativamente le due proteine interagenti etichettate eGFP e mCherry. Descriviamo anche la preparazione del campione e le condizioni per un'efficiente imaging cellulare FLIM-FRET. Infine, proponiamo un tutorial passo-passo per l'analisi delle immagini, incluso uno strumento sviluppato di recente che offre possibilità di visualizzazione avanzate per un'interpretazione diretta di dati FLIM-FRET complessi. Con questa relazione, vorremmo convincere non solo i più avventurosi, ma anche la maggior parte dei biologi che FRET-FLIM è una tecnica accessibile e potente in grado di affrontare le loro domande sugli IPP direttamente nell'ambiente cellulare nativo.
Protocollo
1. Costruzione plasmide
- Amplificare da due PCR (PCR1 e 3) le sequenze di DNA (utilizzare il DNA genomico di P. aeruginosa PAO1) delle 700 coppie di basi a monte e a valle delle regioni corrispondenti al sito di inserimento nel genoma di P. aeruginosa con DNA polimerasi ad alta fedeltà. Aggiungere siti di restrizione ai primer in blu e verde e aggiungere una sequenza sovrapposta con mCherry ai primer in rosso (Figura 2).
- Per il PvdA etichettato al C-terminale con eGFP, amplificare la regione di 700 bp a monte rispetto al codone di arresto dai primer in blu, e amplificare la regione a valle di 700 bp contenente il codone di arresto con i primer in verde.
- Per il PvdL etichettato all'N-terminale con mCherry, amplificare la regione di 700 bp a monte del gene PvdL, incluso il codone iniziale, dai primer in blu, e amplificare la regione a valle di 700 bp con i primer in verde.
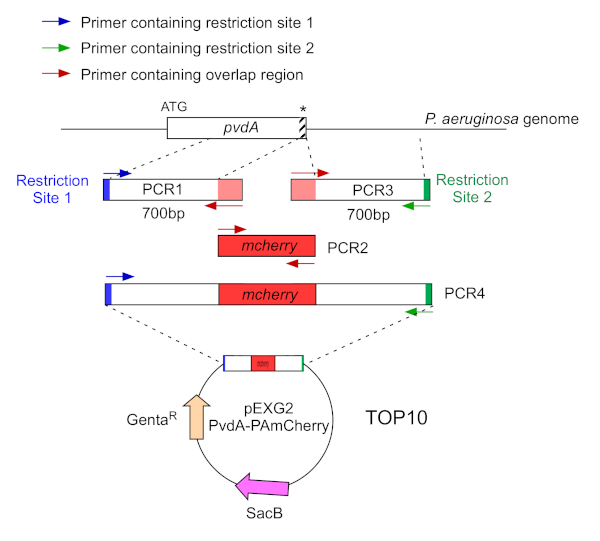
Figura 2: Panoramica della strategia PCR e della costruzione di plasmidi utilizzati per la costruzione di PvdA-mCherry. Vedi il testo per i dettagli - pvdA codifica un enzima coinvolto nella biosintesi della trasoverdina sideroforo, un metabolita secondario coinvolto nell'acquisizione del ferro. Clicca qui per visualizzare una versione più grande di questa figura.
- Amplificare il DNA codificante eGFP (senza i codoni start and stop) con primer in rosso con DNA polimerasi ad alta fedeltà.
- Purificare i prodotti PCR in una colonna di pulizia PCR (Table of Materials).
- Mescolare i prodotti PCR sovrapposti in rapporto equimolare ed eseguire una seconda PCR utilizzando primer con sito di restrizione utilizzato per PCR 1 e 3 (verde e blu nella figura 2).
- Migrare il prodotto PCR in gel Agarose-1x TAE (Tris-Base Acetato EDTA pH 8.0), tagliare la banda corrispondente ed estrarre l'amplicon con un kit di pulizia PCR(Table of Materials).
NOTA: Il protocollo può essere messo in pausa qui. - Digerire l'amplicon PCR e il plasmide pEXG2 utilizzando i corrispondenti enzimi direstrizione 21.
- Ligate plasmide e inserire con T4 DNA ligasi utilizzando 90 ng di plasmide e rapporto molecolare 1:1 (plasmide:inserto).
- Trasforma la costruzione dei plasmidi nelle cellule chimicamente competenti E. coli TOP10 miscelando il prodotto di legatura e 100 μL di TOP10. Incubare la miscela batteria/plasmide competente sul ghiaccio per 30 minuti prima di procedere con uno shock termico di 42 °C per 60 s. Quindi, metti il tubo sul ghiaccio per 10 minuti.
- Aggiungere 1 mL di brodo di lisogenia (LB) ai batteri e incubare a 37 °C per 1 h.
- Piastra 100 μL batteri su agar LB contenente 15 μg/mL gentamicina.
- Incubare durante la notte a 37 °C.
- Schermare la presenza dell'inserto per colonia PCR: da una colonia di trasformatori isolata, raccogliere una quantità minima di batteri da aggiungere a un mix PCR contenente primer che si ibridano sul plasmide in modo tale che la presenza dell'amplicon possa essere rilevata eseguendo il prodotto su un gel di agarosio (DNA polimerasi). Dalla stessa colonia utilizzata per la PCR, trasferire una piccola quantità di batteri su una piastra fresca contenente 15 μg/mL di gentamicina da isolare e utilizzare per l'estrazione del plasmide. Infine, isolare e purificare il plasmide (Table of Materials) e verificare l'inserto sequenziando.
- Conservare i batteri TOP10 contenenti il plasmide in LB con il 20 % di glicerolo in microtubo da 1,5 ml a -80 °C e il plasmide purificato a -20 °C in tubo da 1,5 ml.
NOTA: Il protocollo può essere messo in pausa qui
2. Inserimento di tag fluorescenti nel genoma cromosomico di P. aeruginosa (Figura 3)
- Coltivare batteri aiutanti P. aeruginosa,TOP10 ed E. coli, ognuno su 5 mL di LB senza antibiotico a 30 °C sotto scuotimento orbitale durante lanotte 22. Generare l'inserimento di tag fluorescenti nel genoma di P. aeruginosa trasferendo il plasmide da E. coli TOP10 nel ceppo PAO1 e integrando il plasmide nel genoma mediante ricombinazione omologa. Un secondo evento di crossing-over che eccita il vettore genera il mutante corrispondente.
- Misurare la densità ottica a 600 nm (OD600 nm)della coltura batterica e mescolare una quantità uguale di P. aeruginosa (500 μL, OD600 nm = 1,0) con E. coli TOP10 pEXG2 (500 μL, OD600 nm = 1,0) ed E. coli HB101 pRK600 helper (500 μL, OD600 nm = 1,0) in microtubo da 1,5 mL.
- Centrifugare 5 min a 9.300 x g per pellettizzare i batteri.
NOTA: Gli strumenti online possono essere utilizzati per convertire la forza g centrifuga in rotazione al minuto (rpm) per regolare la velocità della centrifuga. - Conservare il pellet batterico e scartare il supernatante.
- Resuspend il pellet contenente batteri in 50 μL di LB.
- Placcare un punto (~50 μL) della miscela al centro dell'agar LB (preriscaldare a 37 °C) e incubare 5 ore a 37 °C.
- Rottamare il punto con un circuito di inoculazione sterile e rimospendare in 1 mL di LB.
- Piastra 100 μL di questa sospensione batterica su agar LB contenente 10 μg/mL di cloramfenicolo per eliminare E. coli (E. coli TOP10 pEXG2 ed E. coli HB101 pRK600 aiutante sono sensibili al cloramfenicolo ma P. aeruginosa è naturalmente resistente) e 30 μg/mL gentamicina e incubare 2 giorni a 37 °C.
- Resuspend una colonia in 1 mL LB e incubare a 37 °C sotto scuotimento orbitale 4h.
- Centrifugare 3 min a 9.300 x g e scartare 950 μL di supernatante. Rimescolare il pellet in 50 μL di LB e isolare la miscela su agar LB contenente saccarosio e senza NaCl.
- Incubare durante la notte a 30 °C.
- Individuare colonie isolate su agar LB e agar LB contenenti gentamicina 15 mg/mL al fine di controllare la sensibilità alla gentamicina.
- Verificare l'inserimento eGFP o mCherry da parte delle colonie PCR (DNA polimerasi) e sequenziare utilizzando primer specifici.
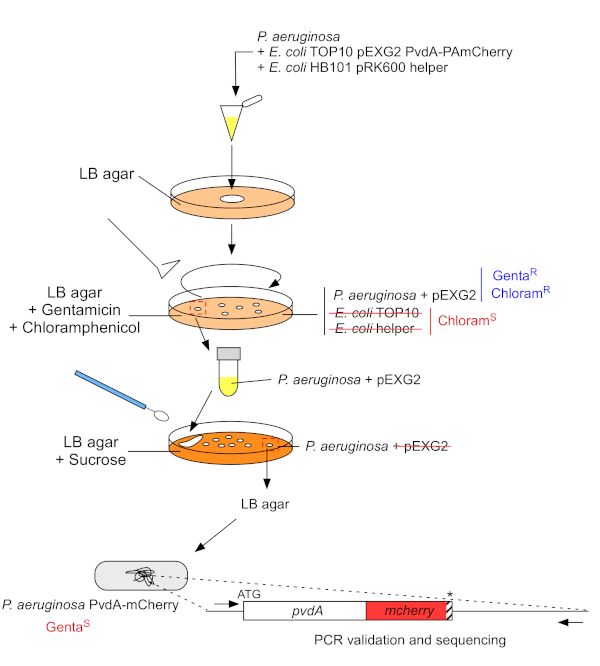
Figura 3: Protocollo di costruzione dei ceppi di P. aeruginosa mediante inserimento di tag fluorescenti. Vedi il testo per i dettagli. Clicca qui per visualizzare una versione più grande di questa figura.
3. Misurazione della pilodina
- Far crescere i batteri in 5 mL di LB a 30 °C sotto scuotimento orbitale durante la notte.
- I batteri pellet per centrifugazione, li lavano e li coltivano in 5 mL di SM (Medium Succinato, composizione: 6 g-L−1 K2HPO4, 3 g-L−1 KH2PO4, 1 g-L−1 (NH4)2 SO4, 0,2 g -L−1 MgSO4, 7 H2O e 4 g-L−1 succinato di sodio con il pH regolato a 7,0 aggiungendo NaOH) a 30 °C sotto scuotimento orbitale durante la notte. SM è un mezzo privato di ferro - l'assenza di ferro attiverà l'espressione delle proteine della via della pioverdina normalmente repressa in presenza di ferro.
- Misurare OD600 nm e diluire nuovamente i batteri in mezzo SM fresco a OD600 nm = 0,1 e coltivarli a 30 °C sotto scuotimento orbitale durante la notte.
- Misurare OD600 nm per determinare la quantità di batteri in ogni campione.
- Preparare una cuvetta al quarzo contenente 100 μL di coltura di P. aeruginosa e completa fino a 1 mL di SM (900 μL). Preparare una cuvetta al quarzo contenente 1 mL di mezzo SM (vuoto).
- Utilizzando uno spettrofotometro visibile ai raggi UV, misurare l'assorbanza al massimo del picco di assorbimento. A pH 7.0, il massimo assorbimento di pyoverdine avverrà a ~ 400 nm. Determinare la concentrazione di pyoverdine (forma apo) nel campione usando la legge di Beer-Lambert usando un coefficiente di estinzione molare a 400 nm di e = 19 000 M-1-cm-1.
NOTA: La pioverdina può essere quantificata nell'intervallo di assorbanza da ~0,1 a ~1 (a seconda dello spettrofotometro visibile ai raggi UV) in cui l'assorbanza aumenta linearmente con la concentrazione.
4. Coltura batterica e condizioni per le cellule per esprimere PvdA, PvdL e PvdJ
- Il primo giorno, inoculare un tubo con 5 ml di LB dall'appropriato stock di glicerolo di batteri e far crescere i batteri durante la notte a 30 °C a 200 giri/min in un incubatore di agitatori orbitali.
- Il secondo giorno, le celle a pellet per centrifugazione a 3.000 x g per 3 minuti e scartano il supernatante.
- Rimosi le cellule in 10 mL di SM.
- Ripetere i passaggi 4.2-4.3 una volta e far crescere i batteri in SM durante la notte a 30 °C 200 giri/min.
- Il giorno 3, diluire 1/10 la coltura batterica in SM fresco.
- Far crescere di nuovo i batteri diluiti durante la notte nelle stesse condizioni.
NOTA: La presenza di pyoverdine può essere rilevata visivamente mentre colora in giallo-verde il supporto in crescita. Mostra che l'espressione delle proteine della via della pioverdina è stata attivata e che gli enzimi di interesse si esprimono nelle cellule.
5. Preparazione del tampone di agarosio(figura 4)
- Posizionare uno scivolo di vetro al microscopio su una superficie orizzontale piatta. Disporre due vetrini sormontati da due strati di nastro adesivo su ciascun lato dello scivolo iniziale.
NOTA: Mantenere uno spazio di 1-2 mm tra le tre diapositive allineate per evitare che l'agarosio corpo a corpo si diffonda infine sugli scivoli con nastro adesivo. - Pipettare e versare una goccia di 70 μL di agarosio fuso all'1% sullo scivolo di vetro. Aggiungere una quarta diapositiva sulla parte superiore per appiattire la goccia di agarosio e premere delicatamente verso il basso. Aspetta circa un minuto.
- Togliere lo scivolo superiore e cadere con una pipetta di circa 3 μL di batteri in 3-4 punti in diverse posizioni sul tampone di agarosio.
- Coprire con una coverlip in vetro al microscopio (ad esempio uno spessore di 22x22 mm #1,5 mm).
NOTA: La planarità e lo spessore delle copertine sono importanti per lavorare con eccitazioni a due fotoni. Le coverlips di precisione con planarità uniforme controllata e bassa autofluorescenza sono di solito una buona scelta. - Fissare il coverslip con paraffina fusa per sigillare il coverslip sullo scivolo di vetro. Inizia fissando i quattro angoli del coverslip.
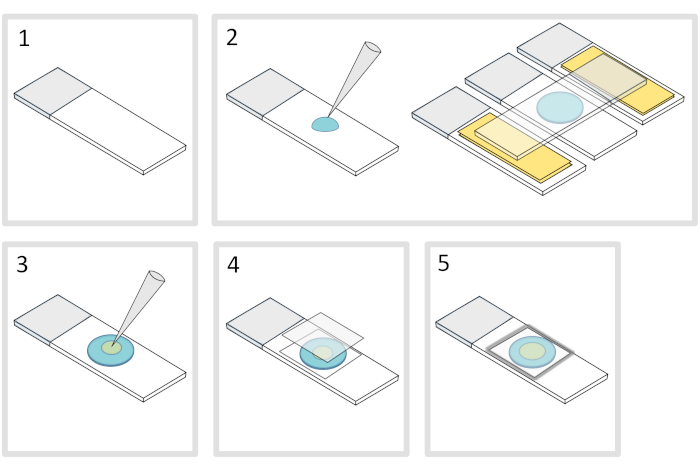
Figura 4: Preparazione del tampone di agarosio. Clicca qui per visualizzare una versione più grande di questa figura.
6. Imaging con configurazione di microscopia a due fotoni
NOTA: Stiamo utilizzando un microscopio invertito a scansione di eccitazione a due fotoni fatto in casa con un obiettivo di immersione in acqua 60x 1.2NA che opera in modalità di raccolta della fluorescenza de-scansionata. La lunghezza d'onda dell'eccitazione a due fotoni è impostata a 930 nm. È fornito da un laser Ti:Sapphire (frequenza di ripetizione di 80 MHz, ≈ 70 fs di larghezza dell'impulso) che funziona a 10-20 mW. I fotoni a fluorescenza sono stati raccolti attraverso un filtro passa corto da 680 nm e un filtro passa banda da 525/50 nm prima di essere indirizzati a un fotodiodo a valanga accoppiato in fibra collegato a un modulo TCSPC (Single Photon Counting) correlato al tempo. Il microscopio è inoltre dotato di una lampada a fluorescenza a trasmissione. Diversi microscopi FLIM-FRET sono ora disponibili in commercio e molte strutture di imaging sono dotate di configurazioni in grado di eseguire misurazioni FLIM-FRET.
- Utilizzare la lampada a fluorescenza per concentrare l'obiettivo sul monostrato di batteri nel campione e selezionare le regioni di interesse.
- Verificare che l'otturatore laser ad eccitazione sia chiuso e che la luce infrarossa proveniente dal laser sia bloccata e non entri nel microscopio.
Attenzione: Si dovrebbe prestare attenzione e vigilanza costante lavorando con i laser pulsati IR in quanto la luce laser non può essere vista dagli occhi, ma qualsiasi esposizione diretta transitoria o riflessione laser può essere estremamente dannosa e creare danni irreversibili agli occhi. Si prega di fare riferimento alle procedure di sicurezza laser locali e all'allenamento prima di utilizzare le configurazioni di microscopia. - Posizionare la diapositiva di microscopia sul palco con le coverlips rivolte verso l'obiettivo.
- Verificare che la lampada a fluorescenza sia ON.
- Ruotare la torretta del cubo del filtro per selezionare il cubo eGFP e aprire l'otturatore della lampada a fluorescenza.
- Inviare la luce di fluorescenza verso l'oculare del microscopio.
Attenzione:assicurarsi che nel percorso della luce vengano smaltiti filtri appropriati per scartare la luce di eccitazione diretta proveniente dalla lampada a fluorescenza che può danneggiare gli occhi. - Focalizzare l'obiettivo sui batteri utilizzando la manopola del microscopio.
- Selezionare una regione di interesse nel campione traducendola utilizzando il joystick che controlla lo stadio motorizzato
NOTA: La messa a fuoco è più facile con un campione altamente fluorescente che consente di vedere la fluorescenza direttamente con gli occhi. - Cambiare l'eccitazione per il laser 2PE per le misurazioni FLIM-FRET.
- Rimandare il percorso di emissione di fluorescenza verso il rivelatore.
- Tornare indietro la torretta del cubo del filtro per selezionare il cubo dicroico per il laser a 930 nm.
- Impostare la potenza del laser su 20 mW.
- Impostare la dimensione della regione di interesse su 30 μm. Questa operazione regola la tensione che opera i galvo-specchi e definisce l'intervallo dei loro movimenti (Figura 5).
- Accendere il rilevatore e iniziare a scansionare il campione: i pulsanti start e stop che controllano la scansione controllano anche l'apertura e la chiusura dell'otturatore laser sia per motivi di sicurezza che per limitare la fotobleaching del campione (Figura 5).
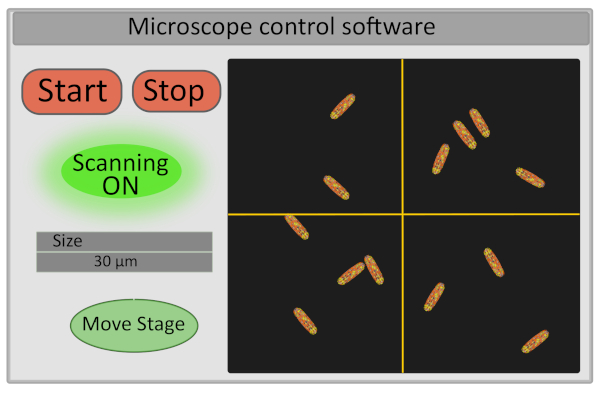
Figura 5: Rappresentazione schematica dell'interfaccia del software di controllo del microscopio. Clicca qui per visualizzare una versione più grande di questa figura.
- Se necessario, regolare la messa a fuoco spostando leggermente la manopola di messa a fuoco fine del microscopio.
- Scegliere il campo visivo per l'imaging spostando finemente lo stage dall'interfaccia del computer. Questa impostazione può essere eseguita spostando la croce sull'immagine nel software di controllo del microscopio (Figura 5) che definirà il nuovo centro dell'immagine e premendo Move Stage. Un buon campo visivo per l'acquisizione corrisponde a un'immagine con 10-30 batteri immobili, tutti correttamente focalizzati (tutti i batteri sono sullo stesso piano). Se sei interessato a estrarre dati FLIM-FRET a singole cellule, assicurati che i batteri siano ben individualizzati (la segmentazione delle immagini sarà molto più semplice).
- Aprire il software SPCM (software commerciale per l'acquisizione dei dati) e verificare che la velocità di conteggio dei fotoni non sia troppo elevata per evitare effetti di accumulo che possono influire sulle misurazioni della durata. Se necessario, abbassare l'intensità del laser per mantenere bassa la velocità di conteggio dei fotoni (circa l'1% del tasso di ripetizione del laser).
NOTA: l'effetto Accumulo descrive gli effetti dei fotoni persi ad alti tassi di conteggio dei fotoni a causa del tempo morto dei dispositivi TCSPC (Time Correlated Single Photon Counting). Se si verifica pile-up, la durata media misurata diventa artificialmente più breve con possibilmente un componente più corto aggiuntivo che può apparire nel decadimento a causa del sovracampionamento dei fotoni che emettono rapidamente. - Regolare i parametri di acquisizione, incluso il tempo di raccolta dell'acquisizione (in genere sono necessari da 60 a 180 s per raccogliere abbastanza fotoni).
- Premere il pulsante Start e attendere il completamento dell'acquisizione.
- Salvare i dati.
- Interrompere la scansione del campione e spegnere il rilevatore.
- Selezionare un altro campo visivo nell'esempio e ripetere i passaggi 6.14-6.22 o visualizzare una nuova diapositiva di microscopia ripetendo i passaggi da 6.1 a 6.22.
NOTA: P. aeruginosa può vivere e dividersi fino a 6-8 ore a temperatura ambiente sul tampone di agarosio (corrispondente ad un tempo di raddoppio di almeno ~4 a 20 °C). Idealmente, non aspettare troppo a lungo per eseguire la misurazione FLIM-FRET per evitare di osservare un pad completamente coperto di batteri.
7. Analisi dei dati
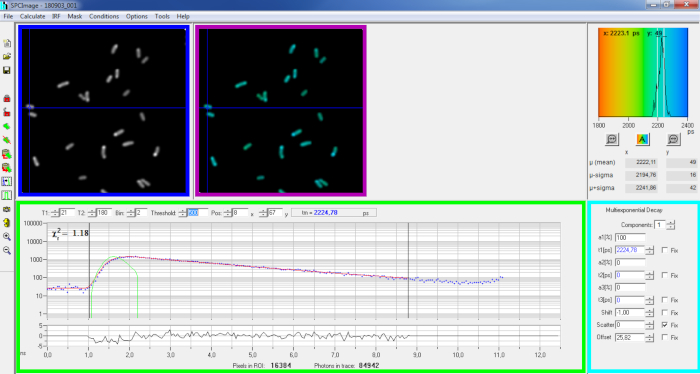
Figura 6: Pannello principale della finestra di analisi dei dati del software SPCImage. Immagine di intensità (scatola blu), immagine a vita (scatola viola), istogramma a vita (in alto a destra), curva di decadimento in posizione selezionata (scatola verde) e parametri di decadimento in posizione selezionata (scatola ciano) di un decadimento rappresentativo PvdA-eGFP registrato in P. aeruginosa dal vivo utilizzando una scheda di acquisizione bh SPC830 su una configurazione a eccitazione a due fotoni fatta in casa-FLIM-FRET. La curva sperimentale di decadimento del pixel puntato nell'immagine precedente, la sua vestibilità mono-esponenziale (curva rossa) che deconvoluzza il decadimento dalla sua funzione di risposta strumentale calcolata (curva verde) può essere vista nel pannello verde. Clicca qui per visualizzare una versione più grande di questa figura.
- Analisi di base
- Eseguire il software SPCImage.
- Importare il file SPCM salvato. L'immagine di intensità viene visualizzata sul pannello superiore sinistro del software(figura 6 casella blu).
- Esaminate la finestra della curva didecadimento (figura 6 casella verde) che visualizza i dati di decadimento corrispondenti al pixel selezionato nell'immagine di intensità(figura 6 casella blu). I numeri di fotoni di ogni canale di volta sono mostrati come punti blu e l'adattamento del decadimento viene disegnato come una linea rossa. Si noti che dopo aver caricato i dati il software visualizza il pixel più luminoso dell'immagine. Spostare la croce blu sull'immagine per esaminare i pixel con intensità inferiore. La finestra di decadimento verrà aggiornata automaticamente in ogni nuova posizione pixel.
NOTA: Misurare la funzione di risposta strumentale (IRF) di un sistema di scansione laser è molto difficile. Un IRF calcolato dal bordo crescente delle curve di decadimento della fluorescenza nei dati FLIM può essere usato per la deconvoluzione del decadimento. Questa è l'opzione eseguita per impostazione predefinita in SPCimage(Figura 6 curva verde). - Regolate l'intervallo di raccordi spostando i canali iniziale e finale della scatola di raccordo (T1 e T2 nella casella verde). T1 dovrebbe iniziare dai primi canali del decadimento crescente e T2 definire l'ultimo canale alla fine del decadimento e può essere scelto come uno degli ultimi canali del decadimento con un numero di fotoni sopra l'offset del conteggio dei fotoni (cioè, i livelli di fotoni contati prima che il decadimento aumenti).
- Scegliere l'binning modificando il valore Collocazione. Il decadimento della curva integra il conteggio dei fotoni del pixel selezionato insieme a un'area di i pixel intorno alla posizione del cursore definita dal parametro bin (l'aumento del binning aumenterà il numero di fotoni nel decadimento e può essere utile per raggiungere i conteggi dei fotoni necessari per i modelli multi-esponenziali).
- Regolare il valore Soglia. I pixel che non hanno almeno un canale con un numero di fotoni superiore al valore di soglia non verranno inclusi nella procedura di adattamento. Naturalmente maggiore è il numero di pixel da adattare, più lunga è l'analisi.
NOTA: i dati FLIM possono contenere un numero enorme di pixel e canali di tempo. Le ultime versioni del software consentono di utilizzare GPU (Graphics Processor Unit) per elaborare un gran numero di pixel in parallelo, riducendo enormemente i tempi di elaborazione. Può essere interessante regolare i parametri di binning e soglia utilizzando immagini corrispondenti alle costruzioni batteriche che mostrano la più bassa intensità di fluorescenza (ad esempio, con ceppi batterici con i livelli di espressione più bassi). Ciò garantirà che i decadimenti pertinenti osservati in questi campioni soddisfino i criteri di filtraggio e siano inclusi nell'analisi. Questi parametri possono quindi essere utilizzati per tutte le immagini. - Regolare, se necessario, i parametri di decadimento (scatola ciano). Che lo spostamento vari, la maggior parte della dispersione e dell'offset del tempo può essere fissata a zero se uno sguardo alle funzioni di decadimento mostra che il loro contributo è trascurabile. L'offset può essere stimato guardando i primi canali del decadimento - si noti che l'imaging per lungo tempo a causa della bassa fluorescenza nel campione di solito si traduce in offset non zero. Lo scattering avviene principalmente in campioni spessi e può essere considerato trascurabile altrimenti.
- Prima di eseguire l'adattamento, selezionate l'algoritmo di raccordo. Aprire la finestra delle impostazioni dell'algoritmo in Mostra/Nascondi opzioni modello. Selezionare l'algoritmo di stima della massima verosimiglianza (MLE)( Figura 7A).
- Eseguite il raccordo dell'immagine facendo clic su Calcola (Calculate) | matrice decadimento (Decay matrix). Una volta completata, l'immagine FLIM codificata a vita viene visualizzata nel pannello immagine a vita(Figura 6 casella viola).
NOTA: Nella finestra della curva didecadimento (figura 6 casella verde) è possibile visualizzare il valore di durata corrispondente a ciascun pixel dell'immagine spostando la croce blu.
NOTA: per elaborare automaticamente un gran numero di file di dati FLIM simili, è possibile utilizzare una modalità di elaborazione batch. - Controllare la qualità della vestibilità guardando i residui (idealmente distribuiti casualmente intorno a 0) e un valore quadrato Chi vicino a 1.
- I dati montati possono essere esportati in diversi formati. Per esportare file in file txt, passare a File | Esportazione. Nella finestra Opzioni di esportazione (Figura 7B) scegliere Seleziona tutto e quindi fare clic su Esporta.
- Salvare infine il file di analisi. I file di analisi vengono salvati come file *.img e possono essere riaperti direttamente in SPCImage.
NOTA: In particolari casi di quantità non bilanciate di donatori/accettori, FLIM-FRET può rivelare sotto-popolazioni in una miscela di complessi proteici interagenti, in particolare quando le concentrazioni dei due partner sono molto diverse, risultando così in miscele di specie complesse e libere. Le specie non interagenti (caratterizzate da un decadimento molto simile all'unico decadimento del donatore) possono essere discriminate da quelle interagenti assumendo un'invarianza spaziale dei componenti di vita del donatore attraverso il set di dati. Allo stesso modo, possono formarsi complessi interagenti non stechiometrici con più donatori o più accettori di fluorescenza. I decadimenti della fluorescenza di tali complessi sono solitamente difficili da interpretare. Un diagramma FLIM può essere utilizzato per fornire informazioni critiche sulla stechiometria e sulla modalità di associazione degliIPP 20,23. Il diagramma FLIM è una rappresentazione grafica della componente a vita più breve in funzione della sua ampiezza. Può essere utilizzato per visualizzare pixel con firme di decadimento simili. Per disegnare tali rappresentazioni, i decadimenti sperimentali della fluorescenza devono essere dotati di un modello esponenziale. I passaggi seguenti possono essere una guida attraverso questo processo. - Inizia analizzando i dati dell'unica costruzione del donatore. Consentirà di determinare il valore di vita del donatore. Idealmente, misurare questo valore su diverse immagini registrate nelle stesse condizioni delle costruzioni donatore /accettore per recuperare un robusto valore di vita per il donatore.
- Una volta determinato, adattarsi ai decadimenti della fluorescenza delle costruzioni donatore/accettore con un modello a due esponenziali. Nella casella parametro decadimento ciano(Figura 6) impostare il numero di componenti su 2. Fissare il parametro t2(ps) al robusto valore di durata del donatore determinato nel passaggio 1 e selezionare la casella per correggere questo parametro.
NOTA: È importante fissare la durata di vita lunga τ2 al fine di limitare l'over-fitting, migliorare la convergenza dei raccordi e ottenere parametri di adattamento due esponenzialipiù affidabili 24,25,26. - Salvare il file *img ed esportare i dati come file *.asc come nel passaggio 7.1.11.
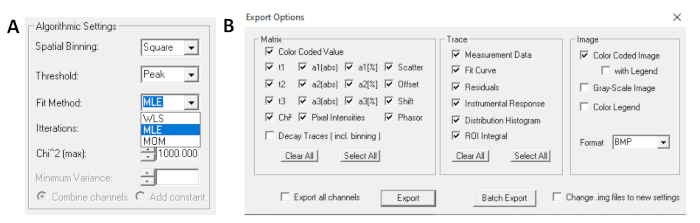
Figura 7: (A) Impostazioni dell'algoritmo per adattare i decadimenti con modelli esponenziali. Selezione di MLE (algoritmo di massima verosimiglianza o stima della massima verosimiglianza, MLE) come modello di adattamento e (B) finestra delle opzioni di esportazione. Clicca qui per visualizzare una versione più grande di questa figura.
- Analisi avanzata delle immagini FLIM in R
- Installare R (https://cran.r-project.org) e RStudio (https://rstudio.com) se necessario.
- Aprire RStudio e creare un nuovo progetto.
- Spostare tutto il file di analisi *.asc in una cartella denominata "data" nella cartella principale del progetto.
- Aprire un nuovo file script (o aprire lo script aggiuntivo FLIM_analysis. R).
- Installare il pacchetto flimDiagRam dedicato per l'analisi dei dati flim https://github.com/jgodet/flimDiagRam. Chiamare il pacchetto nell'area di lavoro. (Cfr. avviso HowTo_FlimDiagRam)
NOTA: L'installazione dei pacchetti deve essere eseguita una sola volta. Una volta installati, i pacchetti possono essere chiamati da qualsiasi nuova sessione R. Il download di pacchetti R da github richiede l'installazione del pacchetto "devtools". L'installazione di "devtools" può richiedere diversi minuti. Il pacchetto flimDiagRam può essere utilizzato per rappresentare i parametri e le distribuzioni dei dati FLIM, per estrarre dati FLIM a livello di singole celle individualizzate, per confrontare i risultati FLIM tra condizioni o ceppi e per esplorare i dati FLIM utilizzando strumenti di visualizzazione avanzati come il grafico FLIM. - Utilizzare il codice commentato passo-passo e i dati vengono resi disponibili per riprodurre in modo indipendente tutte le sottomaschere presentate nella sezione Risultati rappresentativi riportata di seguito. Questa esercitazione è disponibile nell'avviso HowTo_FlimDiagRam all'https://github.com/jgodet/flimDiagRam/blob/master/HowTo.pdf. Il codice può essere facilmente trasposto per analizzare i dati.
Risultati
Le funzioni di distribuzione cumulativa empirica (ecdf) delle durate di fluorescenza misurate per i diversi ceppi batterici sono mostrate nella figura 8. Se si verifica fret, gli ecdf vengono spostati verso le vite di durata più breve (Figura 8A,8B). Si noti che quando l'interazione delle due proteine si traduce in una lunga distanza tra i due fluorofori, non può verificarsi alcun FRET (Figura 8C). Questa situazio...
Discussione
FLIM-FRET offre alcuni vantaggi chiave rispetto all'imaging FRET basato sull'intensità. La durata della fluorescenza è un parametro intrinseco del fluoroforo. Di conseguenza, non dipende dalle concentrazioni locali di fluorofori né dall'intensità dell'eccitazione della luce. La durata della fluorescenza è inoltre scarsamente influenzata dallo sbiancamento fotografico. È particolarmente interessante evidenza di IPP in cellule in cui le concentrazioni di proteine locali possono essere altamente eterogenee in tutti i ...
Divulgazioni
Gli autori non hanno nulla da rivelare.
Riconoscimenti
Riconosciamo il Dott. Ludovic Richert per la sua preziosa assistenza nell'acquisizione dei dati FLIM e per la manutenzione tecnica e lo sviluppo della configurazione FLIM. Questo lavoro è stato finanziato da sovvenzioni della Fondation pour la Recherche en Chimie (https://icfrc.fr/). VN è finanziato dalla Fondation pour la Recherche Médicale (FRM‐SPF201809006906). YM è grata all'Institut Universitaire de France (IUF) per il sostegno e per aver fornito ulteriore tempo per dedicarsi alla ricerca. IJS e JG riconoscono l'Istituto per la distribuzione di droga di Strasburgo per il suo sostegno finanziario.
Materiali
| Name | Company | Catalog Number | Comments |
| 525/50 nm band-pass filter | F37-516, AHF, Germany | ||
| 680 nm short pass filter | F75-680, AHF, Germany | ||
| Agarose | Sigma-Aldrich | A9539 | |
| Ammonium Sulfate (NH4)2SO4 | Sigma-Aldrich | A4418 | |
| DreamTaq DNA polymerase 5U/μL | ThermoFisher Scientific | EP0714 | |
| E. coli TOP10 | Invitrogen | C404010 | |
| Fiber-coupled avalanche photo-diode | SPCM-AQR-14- FC, Perkin Elmer | ||
| Glass coverslips (Thickness No. 1.5, 20×20mm | Knitel glass | MS0011 | |
| High-Fidelity DNA polymerase Phusion 2U/μL | ThermoFisher Scientific | F530S | |
| Lysogeny broth (LB) | Millipore | 1.10285 | |
| Magnesium Sulfate Heptahydrate (MgSO4 . 7H2O) | Sigma-Aldrich | 10034-99-8 | |
| Microscope slides (25×75mm) | Knitel glass | MS0057 | |
| NucleoSpin Gel and PCR Clean-up | Macherey-Nagel | 740609.50 | |
| NucleoSpin Plasmid | Macherey-Nagel | 740588.10 | |
| Potassium Phosphate Dibasic (K2HPO4) | Sigma-Aldrich | RES20765 | |
| Potassium Phosphate Monobasic (KH2PO4) | Sigma-Aldrich | P5655 | |
| Sodium Succinate (Disodium) | Sigma-Aldrich | 14160 | |
| SPCImage, SPCM software | Becker & Hickl | ||
| Sterile inoculating loop | Nunc | 7648-1PAK | |
| T4 DNA ligase 1U/μL | ThermoFisher Scientific | 15224017 | |
| TCSPC module | SPC830, Becker & Hickl, Germany | ||
| Ti:Sapphire laser | Insight DeepSee, Spectra Physics | ||
| Tubes 50mL | Falcon | 352070 |
Riferimenti
- Braun, P., Gingras, A. C. History of protein-protein interactions: From egg-white to complex networks. Proteomics. 12, 1478-1498 (2012).
- Nooren, I. M. A., Thornton, J. M. Structural characterisation and functional significance of transient protein-protein interactions. Journal of Molecular Biology. 325, 991-1018 (2003).
- Hayes, S., Malacrida, B., Kiely, M., Kiely, P. A. Studying protein-protein interactions: Progress, pitfalls and solutions. Biochemical Society Transactions. 44, 994-1004 (2016).
- Guillon, L., Altenburger, S., Graumann, P. L., Schalk, I. J. Deciphering protein dynamics of the siderophore pyoverdine pathway in Pseudomonas aeruginosa. PLoS ONE. 8, 1-9 (2013).
- Ringel, M. T., Brüser, T. The biosynthesis of pyoverdines. Microbial Cell. 5, 424-437 (2018).
- Schalk, I. J., Rigouin, C., Godet, J. An overview of siderophore biosynthesis among fluorescent Pseudomonads and new insights into their complex cellular organization. Environmental Microbiology. 22, 1447-1466 (2020).
- Cui, Y., et al. Techniques for detecting protein-protein interactions in living cells: principles, limitations, and recent progress. Science China Life Sciences. , (2019).
- Day, R. N., Mazumder, N., Sun, Y., Christopher, K. G. FRET microscopy: Basics, issues and advantages of FLIM-FRET imaging. Springer Series in Chemical Physics. 111, 249-276 (2015).
- Bastiaens, P. I. H., Squire, A. Fluorescence lifetime imaging microscopy: Spatial resolution of biochemical processes in the cell. Trends in Cell Biology. 9, 48-52 (1999).
- Yasuda, R. Imaging spatiotemporal dynamics of neuronal signaling using fluorescence resonance energy transfer and fluorescence lifetime imaging microscopy. Current Opinion in Neurobiology. 16, 551-561 (2006).
- Tramier, M., Zahid, M., Mevel, J. -. C., Masse, M. -. J., Coppey-Moisan, M. Sensitivity of CFP/YFP and GFP/mCherry Pairs to Donor Photobleaching on FRET Determination by Fluorescence Lifetime Imaging Microscopy in Living Cells. Microscopy Research and Technique. 71, 146-157 (2008).
- Bajar, B. T., Wang, E. S., Zhang, S., Lin, M. Z., Chu, J. A guide to fluorescent protein FRET pairs. Sensors (Switzerland). 16, 1-24 (2016).
- Piston, D. W., Kremers, G. J. Fluorescent protein FRET: the good, the bad and the ugly. Trends in Biochemical Sciences. 32, 407-414 (2007).
- Leray, A., et al. Optimized protocol of a frequency domain fluorescence lifetime imaging microscope for fret measurements. Microscopy Research and Technique. 72, 371-379 (2009).
- Elson, D. S., et al. Real-time time-domain fluorescence lifetime imaging including single-shot acquisition with a segmented optical image intensifier. New Journal of Physics. 6, 1-13 (2004).
- Rajoria, S., Zhao, L., Intes, X., Barroso, M. FLIM-FRET for Cancer Applications. Current Molecular Imaging. 3, 144-161 (2014).
- Drobizhev, M., Makarov, N. S., Tillo, S. E., Hughes, T. E., Rebane, A. Two-photon absorption properties of fluorescent proteins. Nature Methods. 8, 393-399 (2011).
- Visca, P., Ciervo, A., Orsi, N. Cloning and nucleotide sequence of the pvdA gene encoding the pyoverdin biosynthetic enzyme L-ornithine N5-oxygenase in Pseudomonas aeruginosa. Journal of Bacteriology. 176, 1128-1140 (1994).
- Imperi, F., Visca, P. Subcellular localization of the pyoverdine biogenesis machinery of Pseudomonas aeruginosa: A membrane-associated 'siderosome'. FEBS Letters. 587, 3387-3391 (2013).
- Gasser, V., et al. In cellulo FRET-FLIM and single molecule tracking reveal the supra-molecular organization of the pyoverdine bio-synthetic enzymes in Pseudomonas aeruginosa. Quarterly Reviews of Biophysics. , 1-11 (2019).
- Rietsch, A., Mekalanos, J. J. Metabolic regulation of type III secretion gene expression in Pseudomonas aeruginosa. Molecular Microbiology. 59, 807-820 (2006).
- Herrero, M., De Lorenzo, V., Timmis, K. N. Transposon vectors containing non-antibiotic resistance selection markers for cloning and stable chromosomal insertion of foreign genes in gram-negative bacteria. Journal of Bacteriology. 172, 6557-6567 (1990).
- Godet, J., Mély, Y. Exploring protein-protein interactions with large differences in protein expression levels using FLIM-FRET. Methods and Applications in Fluorescence. 8, 014007 (2019).
- El Meshri, S. E., et al. Role of the nucleocapsid domain in HIV-1 gag oligomerization and trafficking to the plasma membrane: A fluorescence lifetime imaging microscopy investigation. Journal of Molecular Biology. 427, 1480-1494 (2015).
- Becker, W. The bh TCSPC Handbook. Scanning. , 1 (2010).
- Richert, L., Didier, P., de Rocquigny, H., Mély, Y. Monitoring HIV-1 protein oligomerization by FLIM FRET microscopy. Springer Series in Chemical Physics. , 111 (2015).
- Fereidouni, F., Blab, G. A., Gerritsen, H. C. Phasor based analysis of FRET images recorded using spectrally resolved lifetime imaging. Methods and Applications in Fluorescence. 2, (2014).
- Fereidouni, F., Gorpas, D., Ma, D., Fatakdawala, H., Marcu, L. Rapid fluorescence lifetime estimation with modified phasor approach and Laguerre deconvolution: a comparative study. Methods and Applications in Fluorescence. 5, 035003 (2017).
- Margineanu, A., et al. Screening for protein-protein interactions using Förster resonance energy transfer (FRET) and fluorescence lifetime imaging microscopy (FLIM). Scientific Reports. 6, (2016).
- Guzmán, C., Oetken-Lindholm, C., Abankwa, D. Automated High-Throughput Fluorescence Lifetime Imaging Microscopy to Detect Protein-Protein Interactions. Journal of Laboratory Automation. 21, 238-245 (2016).
- Liu, W., Cui, Y., Ren, W., Irudayaraj, J. Epigenetic biomarker screening by FLIM-FRET for combination therapy in ER+ breast cancer. Clinical Epigenetics. 11, 1-9 (2019).
- Liu, X., et al. Fast fluorescence lifetime imaging techniques: A review on challenge and development. Journal of Innovative Optical Health Sciences. 12, 1-27 (2019).
- Padilla-Parra, S., Auduge, N., Coppey-Moisan, M., Tramier, M. Non fitting based FRET-FLIM analysis approaches applied to quantify protein-protein interactions in live cells. Biophysical Reviews. 3, 63-70 (2011).
- Padilla-Parra, S., Audugé, N., Coppey-Moisan, M., Tramier, M. Quantitative FRET analysis by fast acquisition time domain FLIM at high spatial resolution in living cells. Biophysical Journal. 95, 2976-2988 (2008).
- Stringari, C., et al. Phasor approach to fluorescence lifetime microscopy distinguishes different metabolic states of germ cells in a live tissue. Proceedings of the National Academy of Sciences of the United States of America. 108, 13582-13587 (2011).
- Digman, M. A., Caiolfa, V. R., Zamai, M., Gratton, E. The phasor approach to fluorescence lifetime imaging analysis. Biophysical Journal. 94, 14-16 (2008).
- Liang, Z., Lou, J., Scipioni, L., Gratton, E., Hinde, E. Quantifying nuclear wide chromatin compaction by phasor analysis of histone Förster resonance energy transfer (FRET) in frequency domain fluorescence lifetime imaging microscopy (FLIM) data. Data in Brief. 30, 105401 (2020).
- Grimm, J. B., Heckman, L. M., Lavis, L. D. The chemistry of small-molecule fluorogenic probes. Progress in Molecular Biology and Translational Science. 113, (2013).
- Li, L., Sun, H. Next Generation of Small-Molecule Fluorogenic Probes for Bioimaging. Biochemistry. 59, 216-217 (2020).
- Yao, R., Ochoa, M., Yan, P., Intes, X. Net-FLICS: fast quantitative wide-field fluorescence lifetime imaging with compressed sensing - deep learning approach. Light: Science and Applications. 8, 1-7 (2019).
- Smith, J. T., et al. Fast fit-free analysis of fluorescence lifetime imaging via deep learning. Proceedings of the National Academy of Sciences of the United States of America. 116, 24019-24030 (2019).
- Yao, R., Ochoa, M., Intes, X., Yan, P. Deep compressive macroscopic fluorescence lifetime imaging. Proceedings - International Symposium on Biomedical Imaging. 2018, 908-911 (2018).
Ristampe e Autorizzazioni
Richiedi autorizzazione per utilizzare il testo o le figure di questo articolo JoVE
Richiedi AutorizzazioneThis article has been published
Video Coming Soon