È necessario avere un abbonamento a JoVE per visualizzare questo. Accedi o inizia la tua prova gratuita.
Method Article
Valutazione della distribuzione spaziale delle γH2AX seguenti radiazioni ionizzanti
In questo articolo
Riepilogo
Analisi al microscopio di γH2AX focolai, che formano in seguito alla fosforilazione di H2AX a livello della serina-139 in risposta a DNA a doppio filamento, è diventato uno strumento prezioso nel campo della biologia delle radiazioni. Qui abbiamo usato un anticorpo mono-istone H3 metilato in lisina 4 come marker epigenetici attivamente eucromatina di trascrizione, di valutare la distribuzione spaziale della radiazione indotta formazione γH2AX all'interno del nucleo.
Abstract
Una risposta precoce molecolare a DNA a doppio filamento (DSB) è la fosforilazione della Ser-139 residui all'interno del motivo terminale SQEY del 1,2 istone H2AX. Questa fosforilazione di H2AX è mediato dalla fosfatidil-inosito 3-chinasi (PI3K) famiglia di proteine, atassia telangiectasia mutati (ATM), DNA-proteina chinasi subunità catalitiche e ATM e RAD3-correlate (ATR) 3. La forma fosforilata di H2AX, denominato γH2AX, si estende alle regioni limitrofe della cromatina dal sito del DSB, formando focolai distinti, che sono facilmente visibili al microscopio immunofluorecence 3. L'analisi e la quantificazione di γH2AX focolai è stato ampiamente utilizzato per valutare la formazione di DSB e la riparazione, in particolare in risposta alle radiazioni ionizzanti e per valutare l'efficacia di radiazione modificando vari composti e composti citotossici 4.
Data la specificità e la squisita sensibilità di questo marcatore de novo di DSB, ha fornito nuove informazioni sui processi di danno e riparazione del DNA nel contesto della cromatina. Ad esempio, nella biologia radiazione il paradigma centrale è che il DNA nucleare è l'obiettivo fondamentale rispetto alla sensibilità alle radiazioni. Infatti, il consenso generale del settore è stato in gran parte per visualizzare cromatina come un modello omogeneo per danno e riparazione del DNA. Tuttavia, con l'uso di γH2AX come marcatore molecolare di DSB, una disparità di γ-irradiazione indotto la formazione di focolai in γH2AX eucromatina ed eterocromatina è stato osservato 5-7. Recentemente, abbiamo utilizzato un pannello di anticorpi sia mono-, di-o tri-istone H3 metilato in lisina 9 (H3K9me1, H3K9me2, H3K9me3) che sono impronte epigenetiche di eterocromatina costitutiva e silenziamento trascrizionale e lisina 4 (H3K4me1, H3K4me2, H3K4me3 ), che sono strettamente correlate attivamente trascrizione regioni eucromatiche, per studiare la distribuzione spaziale delle γH2AX seguenti radiazioni ionizzanti 8. In conformità con le idee prevalenti quanto riguarda la biologia della cromatina, i nostri risultati indicato una stretta correlazione tra la formazione e la trascrizione γH2AX attivo 9. Qui mostriamo il nostro metodo di immunofluorescenza per la rilevazione e quantificazione di γH2AX focolai in cellule non-aderenti, con un focus particolare sulla co-localizzazione con altri marcatori epigenetici, analisi di immagine e di modellazione 3D.
Protocollo
Preparazione delle cellule
- Umano erythroleukemic K562 cellule sono coltivate in RPMI-1640 medium supplementato con 10% (v / v) di siero fetale bovino e 20 mg / ml di gentamicina in un umidificata al 5% CO 2 ambiente a 37 ° C.
- Circa 18 ore prima della colorazione lavare cellule in crescita esponenziale (ottimale a 5 x 10 5 cellule / ml) con tampone fosfato (PBS), risospendere in mezzi freschi e ritorno a 37 ° C, 5% di CO 2.
- Lavare le cellule due volte con PBS per centrifugazione a circa 1500 rpm per 5 minuti e risospendere in mezzi freschi.
- Contare le celle e regolare la densità cellulare di circa 5 x 10 5 cellule / ml.
Le celle possono essere contati o con un metodo automatico (ad esempio Sysmex o Coulter contatore con dimensioni filtro tra 5 e 20 micron) oppure utilizzando il metodo trypan esclusione blu e un emocitometro. Il numero in eccesso di cellule (> 800 cellule / cm 2) può portare a non uniforme colorazione.
Irradiazione e colorazione di immunofluorescenza
- Assemblare clip cytospin, carte di filtro e diapositive con un'etichettatura appropriata.
Durante il montaggio dell'apparato cytospin garantire che il foro nella carta filtro coincide con l'imbuto.
- Esporre le cellule a uno γ-radiazioni o raggi X (2Gy in questo esempio).
Per prevenire la formazione di focolai durante l'irradiazione, mantenere le cellule in ghiaccio per 5 a 10 minuti prima e durante l'irradiazione.
A seguito di irradiazione incubare le cellule a 37 ° C, 5% di CO 2 per il tempo necessario (per i livelli di picco γH2AX 30 minuti a 1 ora, 1 ora in questo esempio).
- Lavare le cellule due volte con PBS freddo per centrifugazione e risospendere in PBS fresco.
- Dispensare 100-150 microlitri della sospensione cellulare in ogni imbuto cytospin e far girare a 500 giri per 5 minuti.
- Separare le diapositive da cytospins e permettono alle cellule di aria moderatamente asciutto per circa 15 minuti.
Non lasciare che le cellule asciugare completamente la morfologia cellulare potrebbe disintegrarsi.
- Disegna un cerchio intorno alle cellule con una penna idrofobo.
- Fissare le cellule con paraformaldeide 4% per 5 minuti a temperatura ambiente (RT).
Nella nostra esperienza, la fissazione con paraformaldeide è superiore a fissare con etanolo o metanolo. Cellule in sospensione richiedono la fissazione per 5 minuti a temperatura ambiente, mentre i tempi di fissazione più lunghi (15 a 20 minuti) sono necessari per il fissaggio ottimale di cellule aderenti.
Troviamo che produce colorazione immediatamente risultati migliori rispetto la memorizzazione dei vetrini fissati a -20 ° C che si traduce di solito in fluorescenza di fondo elevato.
- Lavare le cellule due volte con PBS per 5 minuti ciascuno.
- Permeabilize le cellule con 0,1% Triton X-100 a temperatura ambiente per 5 minuti.
Rispetto al Tween-80 abbiamo tipicamente osservare meglio i segnali nelle cellule permeabilizzate con Triton X-100. A seconda del tipo di cellula che può essere necessario modificare il tempo di permeabilizzazione. Per esempio, in cellule K562 permeabilization per 5 minuti a temperatura ambiente è sufficiente, mentre in cellule aderenti uno permeabilization 15 minuti è richiesto.
- Lavare le cellule per tre volte con PBS per 5 minuti ciascuno.
- Bloccare le cellule in 1% BSA per 30 minuti (3 x 10 minuti) a temperatura ambiente.
Abbiamo confrontato l'efficienza di bloccaggio utilizzando siero (derivati da specie come gli anticorpi secondari) e di BSA in PBS. BSA è stato più efficace nel ridurre al minimo legame non specifico degli anticorpi secondari rispetto al siero. Abbiamo confrontato diverse volte il blocco, il blocco durante la notte a 4 ° C rispetto a 3 x 10 minuti e 3 x 20 passi minuto blocco a temperatura ambiente, e stabilito che il blocco di 3 x 10 minuti è stato ottimale.
- Aggiungere 50 ml di anticorpo primario (1: 500 diluito in 1% BSA) per ogni diapositiva.
Incubazione con anticorpo primario a temperatura ambiente per 60 minuti consente di ottenere la colorazione di fondo ridotto rispetto a una notte di incubazione a 4 ° C.
- Per colorare marcatori diversi contemporaneamente utilizzare anticorpi cresciuto a specie diverse (ad esempio anti-topo γH2AX e di coniglio anti-metilato H3K4 in questo esempio).
- Incubare con anticorpi primari per 90 minuti a temperatura ambiente su una piattaforma oscillante a 250 giri al minuto.
Tutte le incubazioni vengono eseguite in una vasca colorazione umidificato.
- Lavare le cellule per tre volte con PBS per 5 minuti ciascuno.
- Aggiungere 50 ml di (1: 1000 diluito in 1% BSA), l'anticorpo secondario per ogni diapositiva.
A macchia di diversi marcatori sulla stessa diapositiva uso anticorpi secondari con dspecie specificità ifferent (secondo l'anticorpo primario) e fluorescenza d'onda di emissione (ad esempio anti-topo Alexa 488 (verde) e anti-coniglio Alexa-546 (rosso) in questo esempio).
- Incubare le cellule per 60 minuti a temperatura ambiente su una piattaforma oscillante a 250 giri al minuto.
Si raccomanda di incubare le cellule in una camera buia umida per evitare che sbiadiscano e asciugatura dell'anticorpo secondario.
- Lavare le cellule per tre volte con PBS per 5 minuti ciascuno.
- Aggiungere 50 ml di (diluito 1:500 in PBS) TOPRO-3 per ogni diapositiva.
- Lavare i vetrini con PBS per 3 volte 5 minuti.
- Rimuovere l'umidità in eccesso da diapositive e aggiungere l'anti-fade soluzione.
Si consiglia di non asciugare completamente prima di aggiungere le cellule anti-sbiadimento soluzione.
- Vetrino, sigillare con smalto e lasciare diapositive notte al buio a temperatura ambiente.
Si raccomanda di usare confocale vetrini specifici # 1.5 (0,16-19 micron di spessore) e smalto trasparente.
Pur ponendo il coprioggetto sul vetrino, occorre prestare attenzione per evitare la formazione di bolle d'aria.
Acquisizione di immagini
- Un LSM510 microscopio confocale Zeiss Meta è utilizzato per acquisire immagini utilizzando lo standard GFP (verde, 488 nm), PI (rosso 543 nm) e laser molto rosso (blu 633 nm).
Immagini acquisite utilizzando un microscopio confocale rivelare migliore risoluzione spaziale.
Dal momento che le dimensioni del γH2AX focolai può essere inferiore a 0,5 μicrons, si raccomanda di acquisire immagini con almeno una dimensione di 0,5 μicron passo lungo l'asse Z.
Un olio 63 x lente ad immersione obiettivo è preferita rispetto a uno ingrandimento superiore o inferiore. Per le celle di imaging per molti focolai conteggio è preferibile utilizzare una velocità di scansione di 8 e le dimensioni dell'immagine di 1024 x 1024 pixel. Per illustrare la relazione spaziale tra le diverse proteine nucleari, si raccomanda di utilizzare una velocità di scansione di 6 con dimensioni immagine di 2048 x 2048 pixel.
Quando l'imaging da più canali una acquisizione di scansione linea è consigliato rispetto ad una scansione telaio. Questo evita lo sbiancamento e una migliore risoluzione si ottiene. Scansione sequenziale è utilizzato per prevenire sanguinare tramite tra i canali.
Foci conteggio
Analisi delle immagini e il conteggio focolai possono essere eseguite utilizzando vari pacchetti software (inlcuding Metamorph, immagine-J e Imaris). La procedura per il conteggio delle foci utilizzando Metamorph è descritto qui.
- Aprire stack immagine γH2AX direttamente dal menu file o utilizzando il numero di build stack di opzione.
- Vai al processo del menu e scegliere aritmetica stack.
- Ha scelto massima di proiezione e selezionare gli aerei da inserire e fare clic su OK.
- Salvare l'immagine risultante come Tiff γH2AX. file e aprire il TOPRO-3 (blu) dell'immagine.
- Vai a regioni e selezionare uno strumento di disegno regione e disegnare le regioni intorno nuclei.
- Clicca sull'immagine γH2AX Tiff e andare al menu di processo e selezionare i filtri morfologici.
- Ha scelto Top-Hat e selezionare l'immagine γH2AX come immagine di origine.
- Applicare Top-Hat filtro e l'immagine risultante sarà una immagine binaria con meno rumore e ridotta variazione di intensità fuochi.
Si deve prestare attenzione nella scelta di Top-Hat valori. Valori ottimali può essere ottenuta con confronto visivo delle immagini prima e dopo l'applicazione di filtri.
- Vai al menu di processo e selezionare l'immagine soglia.
- Scegli soglia inclusiva e selezionare i valori di soglia inferiori e superiori.
E 'tipicamente i valori di soglia che influenzano il numero focolai. Pertanto, molta attenzione è necessaria quando la selezione di valori di soglia. Questo può essere meglio raggiunto il numero focolai conteggio utilizzando diversi valori di soglia in cellule esposte a inferiore a 2 Gy di radiazione γ-e poi confrontare i numeri con focolai conteggio manuale (ad occhio). Il valore di soglia ottimale è quella che riduce al minimo l'inserimento di sfondo e massimizza inclusione di fuochi.
- Selezionare la TOPRO-3 immagine e andare al menu regioni e selezionare l'opzione di trasferimento regioni. Selezionare l'immagine sorgente come TOPRPO-3 e la destinazione come Top-Hat e fare clic su OK.
- Una volta che le regioni nucleari vengono trasferiti al Top-Hat immagine andare al menu misurare e selezionare Analisi integrata morfometria.
- Nella finestra pop-up selezionare la sorgente dell'immagine-Top-Hat e misura per zona.
- Seleziona misura tutte le regioni e cliccare su misura per ottenere il numero di focolai in tutti i nuclei corrispondente nella Top-Hat immagine.
- I numeri focolai di ogni regione può essere esportato in un file di MS-Excel utilizzando i dati di log-opzione.
3Dricostruzione delle immagini
Per creare un'immagine 3D da una pila utilizzando Metamorph:
- Vai al menu stack e scegliete la ricostruzione 3D.
- Selezionare l'angolo di rotazione (ad esempio 160 °, 320 °).
- Selezionare la ricostruzione 3D di tipo massimo.
- Scelto il piano di rotazione (orizzontale o verticale).
- Ha scelto la Z-calibrazione distanza.
E 'preferibile utilizzare l'utente specificato Z-distanza (ottimale è di 0,5 μicrons).
- Scegli OK per creare una ricostruzione 3D.
Linea di scansione analisi
Per l'analisi linea di scansione utilizzando Metamorph:
- Aperto sia una singola immagine aereo o un file impilati.
- Vai alla misura strumento barre dei comandi e selezionare la scansione analisi di linea.
- Tracciare una linea attraverso il cellulare e questo verrà automaticamente rivela l'intensità di fluorescenza e la distanza di ogni marcatore lungo il percorso della linea.
Per chiarire la relazione spaziale della cromatina marcatori diversi, per esempio, γH2AX foci e la metilazione degli istoni, è preferibile disegnare linee che si estendono su regioni che sono entrambi densi e poveri di questi marcatori.
Dati rappresentante
Ai fini di questa dimostrazione abbiamo utilizzato anticorpi γH2AX foci e H3K4me, per valutare la distribuzione spaziale delle DSB ad un determinante epigenetico di trascrivere attivamente eucromatina. Come mostrato in figura 1, a seguito di irradiazione con 2Gy, γH2AX foci formata prevalentemente nelle regioni che sono stati spenti per entrambi colorazione H3K4me e per il DNA macchia TOPRO-3 (regioni di DNA vivacemente colorati sono indicativi di eterocromatina).
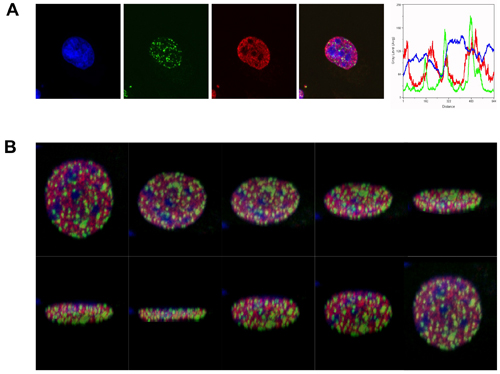
Figura 1. ΓH2AX focolai forma prevalentemente in eucromatina, in risposta alle radiazioni ionizzanti. (A) la visualizzazione di immunofluorescenza γH2AX focolai (verde) in cellule umane erythroleukemic K562, 1 ora dopo γ-irradiazione (2 Gy). γH2AX focolai sono riportati nella relazione al H3K4me, che rappresenta attivamente trascrivere eucromatina (rosso). Il DNA è marcato con TOPRO-3 (blu). L'immagine fusa (blu, rosso e verde) dimostra l'esclusione γH2AX focolai dalle regioni eterocromatici. L'analisi di scansione di linea (fluorescenza distanza vs intensità) indica la distribuzione relativa dei marker in un unico piano. (B) Fette a piani diversi dalla ricostruzione 3D delle immagini fuse descritto in precedenza.
Divulgazioni
Riconoscimenti
Il sostegno della Australian Institute of Nuclear Science and Engineering è riconosciuto. TCK è stato il destinatario di premi AINSE. Lab Medicina epigenomic è supportata dal National Health and Medical Research Council of Australia (566.559). Questo lavoro è finanziato dal CRC for Biomedical Imaging Development Ltd, con sede e supportato in Centri di Ricerca Cooperativa il governo australiano s (CRC) del programma. LM è supportato da Melbourne di ricerca (Università di Melbourne) e borse di studio integrative Biomedical Imaging CRC. Il supporto di Monash Micro Imaging (Dr. Stephen Cody e Iska Carmichael) è stata preziosa per questo lavoro.
Materiali
| Name | Company | Catalog Number | Comments | |
| Roswell Park Memorial Institute -1640 (RPMI-1640) | Growth medium | Invitrogen | 22400071 | RPMI-1640, pH 7.4 medium supplemented with 10% (v/v) fetal bovine, 2mM L-glutamine, 20μg/ml gentamicin, 20mM (HEPES) N-2-Hydroxyethylpiperazine-N’-2-Ethanesulfonic Acid |
| Fetal Bovine Serum (FBS) | Sigma-Aldrich | F2442 | ||
| Bovine Serum Albumin (BSA) | Sigma-Aldrich | A7906 | BSA (1%) is used to block any non-specific antibody binding. Primary and secondary antibodies are diluted in BSA. | |
| PBS (without Ca2+ and Mg2+) | Invitrogen | 17-517Q | ||
| Trypan blue | Sigma-Aldrich | T6146 | Used to distinguish between live and dead cells. | |
| Triton X-100 | Reagent | Sigma-Aldrich | T8787 | Triton X-100 (0.1%) used to permeabilise cells. |
| Paraformaldehyde | Reagent | Sigma-Aldrich | 158127 | Paraformaldehyde (4%) used to fix cells. |
| Mouse monoclonal anti-phospho histone-H2AX antibody | Primary Antibody | EMD Millipore | 16193 | Dilution of primary antibody (1:500), in 1% BSA. |
| HistoneH3 (Mono-methyl K4) | Primary Antibody | Abcam | AB8895 | |
| Alexa Fluor 488 goat anti-mouse IgG (H+L) | Secondary Antibody | Invitrogen | 11029 | Dilution of secondary antibody (1:500), in 1% BSA. |
| Alexa Fluor 546 goat anti-rabbit IgG (H+L) | Secondary Antibody | Invitrogen | 11035 | |
| TOPRO3 | DNA Stain | Invitrogen | T3605 | TOPRO3 is a DNA dye with an Abs/Em of 642/661 nm. DAPI could be used if the confocal microscope is equipped with a 405 nm laser. |
| ProLong Gold | Anti-fade solution | Invitrogen | P36930 | This glycerol based mounting medium must be used with an oil based lense that matches its refractive index. |
| Polylysine slides | Menzel-Glaser | |||
| Coverslips (22x50mm) | Coverslips | Menzel-Glaser | CS2250100 | |
| Tissue Culture Flask, Vented Cap | Culture Flask | BD Biosciences | 353112 | |
| Shandon Cytospin 4 | Thermo Fisher Scientific, Inc. | |||
| Cytofunnels | Shandon, Inc. | |||
| Filter Cards | Shandon, Inc. | 353025 | ||
| Coplin Jar, glass | Grale Scientific P/L | 1771-OG | ||
| Staining Trough | Grale Scientific P/L | V1991.99 | ||
| PAP Pen | Zymed Laboratories, Inc. | 008877 | ||
| Gammacell 1000 Elite Irradiator | Gamma Irradiator | Nordion International Inc. | ||
| Zeiss LSM 510 Meta Confocal | Confocal Microscope | Equipped with 3 lasers: 488 nm, 543 nm and 633 nm. | ||
| Metamorph | Software for Imaging analysis | Molecular Devices |
Riferimenti
- Rogakou, E. P., Boon, C., Redon, C., Bonner, W. M. Megabase chromatin domains involved in DNA double-strand breaks in vivo. J Cell Biol. 146 (5), 905-916 (1999).
- Rogakou, E. P., Pilch, D. R., Orr, A. H., Ivanova, V. S., Bonner, W. M. DNA double-stranded breaks induce histone H2AX phosphorylation on serine 139. J Biol Chem. 273 (10), 5858-5868 (1998).
- Bonner, W. M. . Nat Rev Cancer. 8 (12), 957-967 (2008).
- Dickey, J. S. H2AX: functional roles and potential applications. Chromosoma. 118 (6), 683-692 (2009).
- Kim, J. A., Kruhlak, M., Dotiwala, F., Nussenzweig, A., Haber, J. E. Heterochromatin is refractory to gamma-H2AX modification in yeast and mammals. J Cell Biol. 178 (2), 209-218 (2007).
- Cowell, I. G. gammaH2AX foci form preferentially in euchromatin after ionising-radiation. PLoS One. 2 (10), e1057-e1057 (2007).
- Kinner, A., Wu, W., Staudt, C., &, I. l. i. a. k. i. s., G, . Gamma-H2AX in recognition and signaling of DNA double-strand breaks in the context of chromatin. Nucleic Acids Res. 36 (17), 5678-5694 (2008).
- Vasireddy, R. S., Karagiannis, T. C., El-Osta, A. gamma-radiation-induced gammaH2AX formation occurs preferentially in actively transcribing euchromatic loci. Cell Mol Life Sci. 67 (2), 291-294 (2010).
- Goodarzi, A. A. ATM signaling facilitates repair of DNA double-strand breaks associated with heterochromatin. Mol Cell. 31 (2), 167-177 (2008).
Ristampe e Autorizzazioni
Richiedi autorizzazione per utilizzare il testo o le figure di questo articolo JoVE
Richiedi AutorizzazioneThis article has been published
Video Coming Soon