É necessária uma assinatura da JoVE para visualizar este conteúdo. Faça login ou comece sua avaliação gratuita.
Method Article
Avaliação da Distribuição Espacial da γH2AX seguintes Radiação Ionizante
Neste Artigo
Resumo
Análise microscópica de γH2AX focos, que formam após a fosforilação de H2AX em Ser-139 em resposta ao DNA dupla fita-breaks, tornou-se uma ferramenta valiosa na biologia da radiação. Aqui usamos um anticorpo mono-metilado histona H3 em lisina 4 como um marcador epigenético ativamente de eucromatina transcrever, para avaliar a distribuição espacial da radiação induzida por formação γH2AX dentro do núcleo.
Resumo
Uma resposta rápida molecular de DNA de fita dupla-breaks (LAP) é a fosforilação da Ser-139 resíduos dentro do tema SQEY terminal do 1,2 H2AX histonas. Esta fosforilação de H2AX é mediada pela fosfatidil-inosito 3-quinase (PI3K) família de proteínas, ataxia telangiectasia mutantes (ATM), subunidade proteína-quinase DNA catalítico e ATM e Rad3 relacionados (ATR) 3. A forma fosforilada da H2AX, referido como γH2AX, se espalha para regiões adjacentes da cromatina a partir do site do DSB, formando focos discretos, que são facilmente visualizados por microscopia immunofluorecence 3. Análise e quantificação de γH2AX focos tem sido amplamente utilizada para avaliar DSB formação e reparação, especialmente em resposta à radiação ionizante e para avaliar a eficácia da radiação modificando vários compostos e compostos citotóxicos 4.
Dada a especificidade e sensibilidade requintada deste marcador de novo de LAP, tem fornecido novos insights sobre os processos de danos no DNA e reparo no contexto da cromatina. Por exemplo, na biologia da radiação, o paradigma central é que o DNA nuclear é o alvo crítico com relação à sensibilidade à radiação. Na verdade, o consenso geral no campo tem sido em grande parte para ver cromatina como um modelo homogêneo para danos no DNA e reparo. No entanto, com o uso de γH2AX como marcador molecular de LAP, uma disparidade de γ-induzida por irradiação formação γH2AX focos em eucromatina e heterocromatina foi observada 5-7. Recentemente, foi utilizado um painel de anticorpos para mono-, di-ou tri-metilados histona H3 em lisina 9 (H3K9me1, H3K9me2, H3K9me3), que são marcas epigenéticas de heterocromatina constitutiva e silenciamento transcricional e lisina 4 (H3K4me1, H3K4me2, H3K4me3 ), que estão fortemente correlacionados ativamente transcrever regiões eucromáticos, para investigar a distribuição espacial da radiação ionizante γH2AX seguintes 8. De acordo com as idéias predominantes em relação à biologia da cromatina, nossos resultados indicaram uma estreita correlação entre a formação γH2AX e transcrição ativa 9. Aqui nós demonstramos o nosso método de imunofluorescência para detecção e quantificação de γH2AX focos em células não aderentes, com um foco particular em co-localização com outros marcadores epigenéticos, análise de imagens e modelagem 3D.
Protocolo
Preparação celular
- Humana erythroleukemic K562 células são cultivadas em RPMI-1640 meio suplementado com 10% (v / v) de soro fetal bovino e 20 mg / ml de gentamicina em um 5% CO 2 umidificado ambiente a 37 ° C.
- Aproximadamente 18 horas antes da coloração lavar exponencialmente células de crescimento (ideal a 5 x 10 5 células / ml) com tampão fosfato salino (PBS), ressuspender em novas mídias e voltar para 37 ° C, 5% CO 2.
- Lave as células duas vezes com PBS por centrifugação a aproximadamente 1500 rpm por 5 minutos e ressuspender em mídia fresco.
- Contagem de células e ajustar a densidade da célula para aproximadamente 5 x 10 5 células / ml.
As células podem ser contadas, quer por um método automatizado (por exemplo, Sysmex ou Coulter contador com o tamanho do filtro entre 5 a 20 microns) ou usando o método de exclusão pelo azul de tripan e um hemocitómetro. Número excessivo de células (> 800 células / cm 2) pode levar a coloração não uniforme.
Irradiação e imunofluorescência
- Montar clips cytospin, cartões de filtro e slides com uma rotulagem adequada.
Ao montar o aparelho cytospin garantir que o buraco na placa de filtro coincide com o funil.
- Expor as células a qualquer radiação γ ou raios-X (2Gy neste exemplo).
Para evitar a formação de focos durante a irradiação, manter as células no gelo por 5 a 10 minutos antes e durante a irradiação.
Irradiação seguintes incubar as células a 37 ° C, 5% CO 2 durante o tempo necessário (para níveis de pico γH2AX 30 minutos a 1 hora; uma hora, neste exemplo).
- Lave as células duas vezes com frio PBS por centrifugação e ressuspender em PBS fresco.
- Dispense 100-150 mL da suspensão de células em cada funil cytospin e girar a 500 rpm por 5 minutos.
- Separar os slides de cytospins e permitir que as células a moderadamente o ar seco por aproximadamente 15 minutos.
Não deixe secar completamente as células como a morfologia celular pode se desintegrar.
- Desenhar um círculo em torno das células com uma caneta hidrofóbica.
- Fix células com paraformaldeído 4% por 5 minutos em temperatura ambiente (RT).
Em nossa experiência, a fixação com paraformaldeído é superior à fixação com etanol ou metanol. Células em suspensão necessidade de fixação por 5 minutos em temperatura ambiente enquanto que a fixação vezes mais (15 a 20 minutos) são necessários para a fixação ideal de células aderentes.
Nós achamos que a coloração imediatamente produz melhores resultados do que armazenar a lâminas a -20 ° C, que normalmente resulta em alta fluorescência de fundo.
- Lave as células duas vezes com PBS por 5 minutos cada.
- Permeabilizar as células com 0,1% Triton X-100 na RT por 5 minutos.
Em comparação com Tween-80 que normalmente observam sinais de melhor em células permeabilizadas com Triton-X 100. Dependendo do tipo de célula que pode ser necessário alterar a hora permeabilisation. Por exemplo, em células K562 permeabilização por 5 minutos em temperatura ambiente é suficiente enquanto que em células aderentes uma permeabilização 15 minutos é necessário.
- Lave as células três vezes com PBS por 5 minutos cada.
- Bloco de células em BSA 1% por 30 minutos (3 x 10 minutos) em temperatura ambiente.
Nós comparamos a eficiência de bloqueio usando soro (derivado de mesma espécie que os anticorpos secundários) e soro albumina bovina em PBS. BSA foi mais eficiente na redução de ligação não específica de anticorpos secundário em comparação com soro. Nós comparamos diferentes tempos de bloqueio, bloqueio durante a noite a 4 ° C em comparação com 3 x 10 minutos e 3 x 20 minutos passos bloqueio à temperatura ambiente, e determinou que o bloqueio de 3 x 10 minutos, foi ótimo.
- Adicionar 50 mL de anticorpo primário (1: 500 diluído em BSA 1%) para cada slide.
Incubação com anticorpo primário em RT por 60 minutos resulta em coloração de fundo reduzido em comparação com uma incubação overnight a 4 ° C.
- A mancha diferentes marcadores usar simultaneamente anticorpos produzidos em diferentes espécies (por exemplo, anti-rato e coelho γH2AX H3K4 anti-metilado, neste exemplo).
- Incube com anticorpos primários por 90 minutos em temperatura ambiente em uma plataforma de balanço de 250 rpm.
Todas as incubações são realizadas em uma calha de coloração umidificado.
- Lave as células três vezes com PBS por 5 minutos cada.
- Adicionar 50 mL de (1: 1000 diluído em BSA 1%) anticorpo secundário para cada slide.
A mancha de marcadores diferentes no mesmo slide utilizam anticorpos secundários com despecificidade de espécie IFERENTES (de acordo com o anticorpo primário) e fluorescência de emissão de comprimento de onda (por exemplo, anti-mouse Alexa 488 (verde) e anti-coelho Alexa-546 (vermelho), neste exemplo).
- Incubar as células durante 60 minutos a RT em uma plataforma de balanço de 250 rpm.
Recomenda-se incubar as células em uma câmara escura úmido para evitar o desbotamento e secagem do anticorpo secundário.
- Lave as células três vezes com PBS por 5 minutos cada.
- Adicionar 50 mL de (1:500 diluídos em PBS) TOPRO-3 para cada slide.
- Lavagem das lâminas com PBS para 3X de 5 minutos.
- Retire o excesso de umidade a partir de slides e adicionar anti-fade solução.
Recomenda-se não secar completamente antes de adicionar células anti-fade solução.
- Lamela, vedação com esmalte de unha e deixar slides durante a noite no escuro à temperatura ambiente.
É recomendado o uso de confocal lamínulas específica # 1.5 (0,16-19 mícrons de espessura) e unha polonês claro.
Ao colocar a lamela no slide, os cuidados devem ser tomados para evitar a formação de bolhas de ar.
Aquisição de imagem
- A Zeiss Microscópio Confocal LSM510 Meta é usado para adquirir imagens usando o padrão GFP (verde, 488 nm), PI (vermelho 543 nm) e lasers vermelho distante (azul 633 nm).
Imagens obtidas utilizando um microscópio confocal revelar melhor resolução espacial.
Desde o tamanho do γH2AX focos pode ser inferior a 0,5 μicrons, recomenda-se para adquirir imagens com pelo menos um tamanho de passo 0,5 μicron ao longo do eixo Z.
A 63 de óleo x lente objetiva de imersão é o preferido em comparação com qualquer ampliação maior ou menor. Para as células de imagem para muitos focos de contagem é preferível usar uma velocidade de digitalização de 8 e tamanho da imagem de 1024 x 1024 pixels. Para ilustrar a relação espacial entre diferentes proteínas nucleares, é recomendado o uso de uma velocidade de digitalização de 6 com o tamanho da imagem de 2048 x 2048 pixels.
Quando imagem em diversos canais de uma aquisição scan line é recomendado em comparação com uma varredura frame. Isto evita o branqueamento e melhor resolução é alcançada. A varredura sequencial é usado para impedir sangrar por entre os canais.
Contagem de focos
Análise de imagem e contando focos pode ser realizada usando vários pacotes de software (inlcuding Metamorph, Image-J e Imaris). O procedimento para a contagem de focos usando Metamorph é descrito aqui.
- Abra γH2AX pilhas de imagens diretamente no menu arquivo ou usando o número de compilação opção stacks.
- Ir para Processo menu e escolher a aritmética pilha.
- Escolheu máxima de projeção e selecione os aviões a serem incluídos e clique em OK.
- Salvar a imagem resultante como um Tiff γH2AX. arquivo e abra a imagem TOPRO-3 (azul).
- Ir para regiões e selecione uma ferramenta de desenho e desenhar região regiões ao redor do núcleo.
- Clique na imagem γH2AX Tiff e vá para o menu de processo e selecionar os filtros morfológicos.
- Top Hat escolheu e selecione imagem γH2AX como uma imagem de origem.
- Aplicar Top Hat-filtro e imagem resultante será uma imagem binária com menos ruído e redução da variação na intensidade de focos.
Cuidados devem ser tomados ao escolher Top Hat-valores. Valores ótimos podem ser obtidos por comparação visual de imagens de antes e depois da aplicação de filtros.
- Vá para o menu de processo e selecionar a imagem limiar.
- Escolha limiar inclusiva e selecionar os valores limite inferior e superior.
É tipicamente os valores limite que afetam o número de focos. Portanto, muita atenção é necessária quando a seleção de valores limite. Isto pode ser melhor alcançada pelo número de focos de contagem utilizando limiares diferentes nas células expostas à inferior a 2 Gy de radiação γ-e então comparar os números de focos com a contagem manual (por olho). O limiar ideal é aquele que minimiza a inclusão de fundo e maximiza a inclusão de focos.
- Selecione a imagem TOPRO-3 e ir para o menu regiões e selecione a opção de transferência de regiões. Selecione a imagem de origem como TOPRPO-3 e do destino como Top Hat e clique em OK.
- Uma vez que as regiões nuclear são transferidos para a imagem Top Hat-ir para o menu e selecione medida Morfometria Análise Integrada.
- Na janela pop-up selecione fonte de imagem-Top Hat e medida pela área.
- Selecione medir todas as regiões e clique em medida para obter o número de focos em todos os núcleos correspondentes na imagem Top Hat.
- Os números de focos de cada região podem ser exportados para um arquivo MS-Excel usando a opção de log de dados.
3Dreconstrução de imagens
Para criar uma imagem 3D a partir de uma pilha usando Metamorph:
- Vá para o menu pilha e escolha de reconstrução 3D.
- Selecionar o ângulo de rotação (por exemplo, 160 °, 320 °).
- Selecione o tipo de reconstrução 3D máximo.
- Escolheu o plano de rotação (horizontal ou vertical).
- Escolheu a distância Z-calibração.
É preferível utilizar o usuário especificado Z-distância (ideal é de 0,5 μicrons).
- Escolheu OK para criar uma reconstrução 3D.
Linha de varredura análise
Para a análise de linha de varredura usando Metamorph:
- Abra uma imagem qualquer plano único ou um arquivo empilhados.
- Ir para a medida de comando barras de ferramentas e selecione a análise scan line.
- Desenhar uma linha sobre a célula e esta será automaticamente revelam a intensidade de fluorescência e distância de cada marcador ao longo do trajeto da linha.
Para elucidar a relação espacial dos marcadores de cromatina vários, por exemplo, γH2AX focos e metilação das histonas, é preferível para desenhar linhas que abrangem regiões que são densas e pobres destes marcadores.
Dados representativos
Para os fins desta demonstração foram utilizados anticorpos para γH2AX focos e H3K4me, para avaliar a distribuição espacial da LAP para um determinante de epigenética ativamente transcrever eucromatina. Como mostrado na figura 1, após irradiação com 2Gy, γH2AX focos formado predominantemente em regiões que foram maçante tanto para coloração H3K4me e para o DNA da mancha TOPRO-3 (brilhantemente regiões do DNA são indicativos manchadas de heterocromatina).
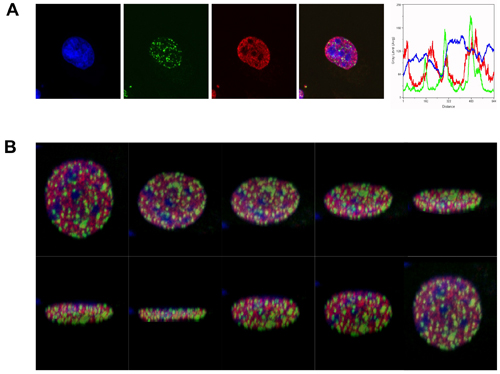
Figura 1. ΓH2AX forma focos predominantemente em eucromatina em resposta à radiação ionizante. (A) visualização de imunofluorescência γH2AX focos (verde) em humanos erythroleukemic K562 células, uma hora após a irradiação γ-(2 Gy). γH2AX focos são mostrados em relação ao H3K4me, representando ativamente transcrever eucromatina (vermelho). DNA é rotulado com TOPRO-3 (azul). A imagem mesclada (azul, vermelho e verde) demonstra a exclusão γH2AX focos de regiões heterocromáticas. A análise varredura de linha (distância vs intensidade de fluorescência) indica a distribuição relativa dos marcadores em um único plano. (B) Slices em diferentes planos da reconstrução 3D a imagem mesclada descrito acima.
Divulgações
Agradecimentos
O apoio do Instituto Australiano de Ciência Nuclear e Engenharia é reconhecido. TCK foi o ganhador de prêmios AINSE. Lab Medicine epigenômico é suportado pelo National Health and Medical Research Council of Australia (566.559). Este trabalho é financiado pelo CRC para Biomédica imagem Development Ltd, criado e apoiado em Centros de o Governo australiano de Pesquisa Cooperativa (CRC) do programa. LM é apoiada por Melbourne Research (University of Melbourne) e Biomedical Imaging bolsas complementares CRC. O apoio de Monash Micro Imaging (Drs. Stephen Cody e ISKA Carmichael) foi inestimável para este trabalho.
Materiais
| Name | Company | Catalog Number | Comments | |
| Roswell Park Memorial Institute -1640 (RPMI-1640) | Growth medium | Invitrogen | 22400071 | RPMI-1640, pH 7.4 medium supplemented with 10% (v/v) fetal bovine, 2mM L-glutamine, 20μg/ml gentamicin, 20mM (HEPES) N-2-Hydroxyethylpiperazine-N’-2-Ethanesulfonic Acid |
| Fetal Bovine Serum (FBS) | Sigma-Aldrich | F2442 | ||
| Bovine Serum Albumin (BSA) | Sigma-Aldrich | A7906 | BSA (1%) is used to block any non-specific antibody binding. Primary and secondary antibodies are diluted in BSA. | |
| PBS (without Ca2+ and Mg2+) | Invitrogen | 17-517Q | ||
| Trypan blue | Sigma-Aldrich | T6146 | Used to distinguish between live and dead cells. | |
| Triton X-100 | Reagent | Sigma-Aldrich | T8787 | Triton X-100 (0.1%) used to permeabilise cells. |
| Paraformaldehyde | Reagent | Sigma-Aldrich | 158127 | Paraformaldehyde (4%) used to fix cells. |
| Mouse monoclonal anti-phospho histone-H2AX antibody | Primary Antibody | EMD Millipore | 16193 | Dilution of primary antibody (1:500), in 1% BSA. |
| HistoneH3 (Mono-methyl K4) | Primary Antibody | Abcam | AB8895 | |
| Alexa Fluor 488 goat anti-mouse IgG (H+L) | Secondary Antibody | Invitrogen | 11029 | Dilution of secondary antibody (1:500), in 1% BSA. |
| Alexa Fluor 546 goat anti-rabbit IgG (H+L) | Secondary Antibody | Invitrogen | 11035 | |
| TOPRO3 | DNA Stain | Invitrogen | T3605 | TOPRO3 is a DNA dye with an Abs/Em of 642/661 nm. DAPI could be used if the confocal microscope is equipped with a 405 nm laser. |
| ProLong Gold | Anti-fade solution | Invitrogen | P36930 | This glycerol based mounting medium must be used with an oil based lense that matches its refractive index. |
| Polylysine slides | Menzel-Glaser | |||
| Coverslips (22x50mm) | Coverslips | Menzel-Glaser | CS2250100 | |
| Tissue Culture Flask, Vented Cap | Culture Flask | BD Biosciences | 353112 | |
| Shandon Cytospin 4 | Thermo Fisher Scientific, Inc. | |||
| Cytofunnels | Shandon, Inc. | |||
| Filter Cards | Shandon, Inc. | 353025 | ||
| Coplin Jar, glass | Grale Scientific P/L | 1771-OG | ||
| Staining Trough | Grale Scientific P/L | V1991.99 | ||
| PAP Pen | Zymed Laboratories, Inc. | 008877 | ||
| Gammacell 1000 Elite Irradiator | Gamma Irradiator | Nordion International Inc. | ||
| Zeiss LSM 510 Meta Confocal | Confocal Microscope | Equipped with 3 lasers: 488 nm, 543 nm and 633 nm. | ||
| Metamorph | Software for Imaging analysis | Molecular Devices |
Referências
- Rogakou, E. P., Boon, C., Redon, C., Bonner, W. M. Megabase chromatin domains involved in DNA double-strand breaks in vivo. J Cell Biol. 146 (5), 905-916 (1999).
- Rogakou, E. P., Pilch, D. R., Orr, A. H., Ivanova, V. S., Bonner, W. M. DNA double-stranded breaks induce histone H2AX phosphorylation on serine 139. J Biol Chem. 273 (10), 5858-5868 (1998).
- Bonner, W. M. . Nat Rev Cancer. 8 (12), 957-967 (2008).
- Dickey, J. S. H2AX: functional roles and potential applications. Chromosoma. 118 (6), 683-692 (2009).
- Kim, J. A., Kruhlak, M., Dotiwala, F., Nussenzweig, A., Haber, J. E. Heterochromatin is refractory to gamma-H2AX modification in yeast and mammals. J Cell Biol. 178 (2), 209-218 (2007).
- Cowell, I. G. gammaH2AX foci form preferentially in euchromatin after ionising-radiation. PLoS One. 2 (10), e1057-e1057 (2007).
- Kinner, A., Wu, W., Staudt, C., &, I. l. i. a. k. i. s., G, . Gamma-H2AX in recognition and signaling of DNA double-strand breaks in the context of chromatin. Nucleic Acids Res. 36 (17), 5678-5694 (2008).
- Vasireddy, R. S., Karagiannis, T. C., El-Osta, A. gamma-radiation-induced gammaH2AX formation occurs preferentially in actively transcribing euchromatic loci. Cell Mol Life Sci. 67 (2), 291-294 (2010).
- Goodarzi, A. A. ATM signaling facilitates repair of DNA double-strand breaks associated with heterochromatin. Mol Cell. 31 (2), 167-177 (2008).
Reimpressões e Permissões
Solicitar permissão para reutilizar o texto ou figuras deste artigo JoVE
Solicitar PermissãoThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Todos os direitos reservados