È necessario avere un abbonamento a JoVE per visualizzare questo. Accedi o inizia la tua prova gratuita.
Method Article
Live Cell Imaging della segregazione cromosomica durante la mitosi
In questo articolo
Riepilogo
Questo protocollo descrive un metodo facile e conveniente per etichettare e visualizzare live cromosomi nelle cellule mitotiche utilizzando etichetta Histone2B-GFP BacMam 2.0 e un sistema di microscopia confocal di filatura disco.
Abstract
Cromosomi devono essere uniformemente e in modo affidabile suddivise in cellule della figlia durante la divisione cellulare mitotica. Fedeltà della segregazione cromosomica è controllata da meccanismi multipli che includono il mandrino Assembly Checkpoint (SAC). Il SAC è parte di un sistema di feedback complessi che è responsabile per la prevenzione di un progresso delle cellule attraverso la mitosi, a meno che tutti i cinetocori cromosomici hanno attaccato per mandrino microtubuli. Cromosomiche in ritardo e cromosoma anormale segregazione è un indicatore di checkpoint di controllo del ciclo cellulare disfunzionale e può essere usata per misurare la stabilità genomica di divisione delle cellule. Deregolamentazione del SAC può causare la trasformazione di una cellula normale in una cellula maligna attraverso l'accumulo di errori durante la segregazione cromosomica. Implementazione del SAC e la formazione del cinetocore complesse sono strettamente regolati dalle interazioni tra chinasi e fosfatasi come proteina fosfatasi 2A (PP2A). Questo protocollo descrive live cell imaging di in ritardo di cromosomi in fibroblasti embrionali di topo isolate da topi che avevano un knockout della subunità regolatoria di PP2A-B56γ. Questo metodo sormonta le imperfezioni di altri ciclo cellulare controllo tecniche di imaging quali la citometria a flusso o immunocitochimica che forniscono solo un'istantanea di uno stato di citochinesi cellulare, invece di una visualizzazione dinamica spazio-temporale dei cromosomi durante la mitosi.
Introduzione
Nel seguente protocollo, descriviamo un metodo conveniente per visualizzare la segregazione cromosomica e mitotica progressione durante il ciclo cellulare in fibroblasti embrionali del mouse utilizzando istone 2B-GFP, BacMam 2.0 etichettatura e live cell imaging.
I checkpoint di controllo del ciclo cellulare monitorare la segregazione del cromosoma e svolgono un ruolo importante nel mantenimento dell'integrità genetica delle cellule 1,2,3. Accumulo di mis-segregati cromosomi può portare a aneuploide, che è un segno dei più solidi tumori 4. Quindi, rilevamento di in ritardo cromosomi utilizzabile come un metodo per studiare l'instabilità cromosomica.
L'etichetta fluorescente proteine possono essere utilizzato per visualizzare la segregazione del cromosoma dal vivo e cromosomiche in ritardo ma la generazione di mCherry-etichetta o proteina H2B-GFP etichettate richiede conoscenza sostanza di gene consegna e biologia molecolare 5. Qui descriviamo l'uso del reagente CellLight Histone2B-GFP BacMam 2.0, chiamato in seguito reggente di CL-HB, per ragioni di semplicità. Questo reagente può essere utilizzato immediatamente e quindi Elimina le preoccupazioni circa la qualità di vettore e l'integrità. Inoltre, questo reagente non richiede l'uso di trattamenti potenzialmente dannosi o di lipidi e di tintura-caricamento prodotti chimici. A differenza di etichette fluorescenti convenzionali, il reggente di CL-HB macchie indipendentemente dalla funzione (cioè., potenziale di membrana). Il reggente di CL-HB può essere semplicemente aggiunti alle cellule e incubato durante la notte per l'espressione della proteina. Il reggente di CL-HB non replica in cellule di mammiferi e può essere utilizzato nelle regolazioni del laboratorio 1 (BSL) livelli di biosicurezza. Inoltre, la trasfezione transiente può essere rilevata dopo incubazione overnight per fino a 5 giorni, tempo sufficiente per svolgere analisi cellulari più dinamiche.
In alternativa, le anomalie cromosomiche potrebbero essere studiate mediante varie tecniche quali la citometria a flusso, immunohistochemistry o fluorescenza in situ di ibridazione (pesce) 6. Citometria a flusso può essere utilizzato per studiare l'aneuploidia, che può essere misurato basato sul contenuto del DNA e la fase delle cellule nel ciclo cellulare. Anche se la citometria a flusso può essere utilizzato per misurare l'aneuploidia, non fornisce informazioni sulla mis segregazione cromosomica. Tecniche di immunoistochimica e pesce utilizzano sonde fluorescenti per associare al DNA o cromosomi. Mentre queste tecniche forniscono un'istantanea dello stato di una popolazione di cellule, non consentono live cell imaging quindi manca qualsiasi informazione ottenuta attraverso la visualizzazione spaziotemporali della citochinesi in cellule specifiche seguiti per un periodo di tempo.
Questo protocollo è stato usato per studiare i cromosomi in ritardo o mis-segregazione cromosomica in fibroblasti embrionali del mouse di nocodazole trattati (MEFs) isolate da topi-B56γ-PP2A. In aggiunta ai sopra applicazione, questo protocollo fornisce un semplice strumento per etichettare e visualizzare segregazione cromosomica in vari tipi di cellule che può essere usato per studiare la regolazione del ciclo cellulare o instabilità cromosomica in cellule del tumore. Inoltre, può essere utilizzato anche per studiare l'instabilità cromosomica causata da vari trattamenti farmacologici o per studiare gli effetti di gene knock out conseguente mis segregazione cromosomica.
Protocollo
Tutti gli esperimenti condotti in questi studi sono stati effettuati secondo protocolli approvati dal comitato di uso e cura degli animali istituzionali presso la struttura di ricerca di Food and Drug Administration (FDA).
1. isolamento e coltura di fibroblasti embrionali del Mouse (MEFs)
- Isolare i fibroblasti embrionali del mouse (MEFs) da un ceppo di PP2A-B56γ-mouse e wild-type littermates da protocollo standard 7,8,9.
- ExpandMEFs per 3 passaggi, congelare e conservare fino a quando necessario per esperimenti 8.
2. coltura Mefs in vetro di copertura Chambered 2-pozzo per vivere Imaging
- Preparare 500 mL di mezzi di sviluppo MEF contenente Dulbecco per volta Eagle Medium (DMEM/F12) con 10% siero bovino fetale (FBS), 1 X penicillina/streptomicina antibiotici, L-Glutammina (200 mM) e 1 X gli aminoacidi non essenziali (NEAA) in una bottiglia di 500 mL media.
- Scongelare i flaconcini di MEFs congelati da wild type e PP2A-B56γ-topi al passaggio 3 in bagno di acqua pre-riscaldata a 37 ° C.
- Trasferimento scongelati MEFs di provette da 15 mL e lentamente aggiungere goccia a goccia 20 mL di DMEM/F12 media ai tubi 15 mL utilizzando una pipetta di 10 mL.
- Trasferimento MEFs insieme con 20 mL di coltura DMEM/F12 in boccette T75 ed espanderli fino a quando le cellule sono 70% confluenti. Confluency è stimato utilizzando un microscopio inverso a 4x o ingrandimento 10x.
- Aspirare il terreno di coltura utilizzando un pascolo di vetro Pipettare collegato ad un sistema di vuoto nella cappa, aggiungere 3 mL di tripsina/EDTA 0.25% e incubare a 37 ° C per 5 min. aggiungere 3 mL di terreno di coltura DMEM/F12 per arrestare la reazione.
- Centrifugare le cellule a bassa velocità (300 x g) per 5 min a temperatura ambiente. Con attenzione rimuovere il supernatante e risospendere il pellet cellulare in 1 mL di terreno di coltura DMEM/F12 pre-riscaldato a 37 ° C in un bagno d'acqua.
- Enumerare le celle utilizzando il metodo di esclusione del blu di trypan o altre cella appropriata contando metodo 8.
- Seme circa 20.000 MEFs/pozzetto in un vetro di copertura chambered 2-pozzo. Aggiungere 200 µ l/pozzetto di coltura DMEM/F12 e permettono alle cellule di allegare incubando una notte a 37 ° C e 5% CO2.
3. sincronizzazione
- Sincronizzare MEFs in fase G0/G1 incubando le cellule in coltura DMEM/F12 contenente 0,1% FBS per 24 h, per ottenere un numero massimo di cellule in fase G0/G1.
Nota: Sebbene inedia del siero è stato utilizzato come metodo di preferenza, vari altri metodi possono essere utilizzati a seconda della fase mitotica in cui le cellule sono necessarie per essere arrestato 10.
4. etichettatura
- Preparare 1,5 mg/mL di soluzione madre di nocodazole in DMSO. Nocodazole arresti le cellule in fase G2/M inibendo la formazione di microtubuli 10.
- Tre giorni dopo la sincronizzazione, aggiungere 200 µ l di terreno di coltura (con 10% FCS), 200 ng/mL nocodazole e CL-HB regent (istone 2B-GFP BacMam 2.0).
- Calcolare il reggente di CL-HB particelle Per cella (PPC) da aggiungere alle cellule come segue:
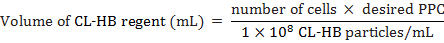
Dove il numero delle cellule è il numero totale stimato delle cellule al momento dell'etichettatura, PPC è il numero di particelle Per cella, e 1 × 108 è il numero di particelle per mL di reagente. Ad esempio, alle cellule di etichetta 20.000 con un PPC di 30
Nota: Regent CL-HB funziona bene con molti tipi di cellule tra 10 e 50 PPC. Tuttavia, 30 PPC ha funzionato meglio per questo studio. - Incubare MEFs per 18 h a 37 ° C e 5% CO2.
Nota: Per l'esperimento corrente, visualizzazione di cromosomi nelle cellule mitotiche sfuggire il checkpoint di montaggio del mandrino si è verificato 18 h dopo l'arresto del ciclo cellulare con nocodazole.
5. visualizzazione di in ritardo di cromosomi
- Visualizzare fibroblasti embrionali di topo (MEFs) utilizzando un sistema di microscopio confocale di filatura disco dotato di una camera climatica e un olio da immersione, obiettivo obiettivo 63X.
- Utilizzare lunghezza d'onda di eccitazione di 488 nm ed emissione lunghezza d'onda 450 nm per acquisizione di immagini di canale GFP.
- Utilizzare un sistema di microscopio confocal di filatura disco con la capacità di fasi motore di scansione, Z-Piezo inserti, incubazione di palcoscenico-top e recupero di fluorescenza diretta dopo foto lo sbiancamento per questa tecnica.
- Un giorno prima di formazione immagine, accendere l'unità di camera ambientale e riscaldare l'intera camera a 37 ° C durante la notte.
- Accendere per il basamento del microscopio, macchina fotografica, unità disco di filatura, illuminatore, laser ad argon, computer e tavolino motorizzato.
- Lasciare che il sistema riscaldare per 3 min; avviare il laser di argon attivando la chiave di accensione. L'interruttore toggle per argon laser dalla modalità "standby" per "esecuzione di laser".
- Avviare il software di acquisizione ed elaborazione di dati.
- Avviare il controller di CO2 per l'incubatrice di fase superiore e impostare la concentrazione di CO2 al 5%. Questo deve essere fatto prima dell'inizio dell'imaging.
- Rimuovere il vetro di copertura incamerato dall'incubatore e posto sul palco per la visualizzazione. Visualizzare le cellule tramite un obiettivo ad immersione 63 x obiettivo (NA1.4).
- Vista attraverso le lenti oculari, mettere a fuoco l'immagine e identificare una cella in fase di ripartizione (NEBD) involucro nucleare.
- Avviare adeguate laser (laser di argon 488 nm per visualizzare Histone2B-GFP).
- Aprire la finestra acquisizione del controllo e impostare il tempo di esposizione per il canale GFP. Identificare una cella che si trova in NEBD.
- Manualmente determinare la parte superiore della cella di destinazione e il piano focale inferiore e immettere impostazioni di sezionamento ottico xyz.
- Osservarlo per 20 min; Se la cella non procede attraverso la divisione cellulare, interrompere l'acquisizione di immagini dopo 20 min e passare alla cella successiva che è in NEBD.
- Circa 1 h per ogni cella è necessaria ai cromosomi in ritardo immagine durante la mitosi in PP2A-B56γ-cellule che fuggiti dal SAC. Per ottenere i dati per un film, scattare foto ogni 3 min.
Nota: cellule Wild type arrestano e non progredire oltre NEBD quando trattati con nocodazole. - Salvare le immagini nel formato di file di zvi per ulteriori analisi.
6. analisi ed elaborazione di immagini
Nota: Eseguire l'elaborazione immagine tridimensionale e analisi utilizzando qualsiasi software disponibile come Axiovision versione 4.8.2 o Imaris versione 8.2. Per questo studio il ImageJ software è stato utilizzato.
- Aprire la sequenza di immagini. Se le immagini sono già in un formato di pila, procedere al passaggio successivo. Se non, combinare tutte le immagini pertinenti in una pila utilizzando ' immagine > Pile > immagini allo Stack' nella barra dei menu.
- Eseguire eventuali regolazioni come richiesto per luminosità/contrasto e livelli.
- Per aggiungere un timestamp al film:
- Vai alla ' immagine > Pile > etichetta... ' nella barra dei menu.
- Selezionare il formato appropriato, il valore di tempo iniziale e intervallo di tempo tra ogni immagine.
- Selezionare la casella di 'Anteprima' e regolare le impostazioni di posizione e formato. Premere 'OK' per applicare il timbro di tempo.
- Per aggiungere una barra di scala per il film:
- Impostare l'immagine ridimensionamento sotto ' analisi > Imposta scala ' nella barra dei menu.
- Nel campo 'Distanza in pixel', immettere un numero di pixel di cui è nota la distanza e nel campo 'Distanza noto' immettere la distanza. Impostare la corretta unità di lunghezza per la distanza, per esempio, in µm. Premere 'OK' per applicare la scala allo stack.
- Vai a ' Analyze > Strumenti > scala bar... ' per aggiungere la barra della scala. Impostare la dimensione della barra come 'Larghezza in µm' e regolare le restanti opzioni come appropriato di formattazione. Selezionare la casella 'Etichettare tutte le fette' per aggiungere la barra della scala per l'intero stack.
- Visualizzare in anteprima il filmato facendo clic sul pulsante triangolare play in basso a sinistra del bordo della finestra immagine. Regolare il frame rate tramite ' immagine > Pile > Animazione > Opzioni di animazione nella barra dei menu.
- Esportare il file del filmato selezionando ' File > Salva con nome > AVI... ' e selezionare il frame rate e compressione.
- Premere 'OK' per scegliere Salva percorso e nome file. Premere 'Salva' per salvare il file del filmato.
Risultati
MEFs da wild type e PP2A-B56γ-topi sono stati seminati in un vetro di copertura 2-pozzo camera e permesso di allegare. Il giorno 2, MEFs sono stati sincronizzati utilizzando 0,1% FBS per 24 h. Il giorno 3, MEFs nei media con 200 ng/mL nocodazole e 30 PPC CL-HB regentwere incubati per 18 h 37° C e 5% CO2. Il giorno 4, le cellule erano imaged utilizzando un sistema di microscopio confocal di filatura disco (Figura 1). Live imaging cellul...
Discussione
I checkpoint di controllo del ciclo cellulare che assicurano la segregazione del cromosoma accurata prevenire aneuploidia e cella trasformazione 1,2,3. Nello studio presente, abbiamo trovato che l'inattivazione di PP2A-B56γ ha provocato un checkpoint di montaggio mandrino indebolito. Live cell imaging ci ha permesso di osservare la mis-segregazione cromosomica durante la mitosi in MEFs di PP2A-B56γ trattati con nocodazole
Divulgazioni
Gli autori dichiarano di non avere nessun concorrenti interessi finanziari.
Riconoscimenti
Vorremmo ringraziare il Dr. Guo-Sabrina Hung e Dr. Bharatkumar Joshi per i preziosi commenti che il manoscritto è migliorato.
Materiali
| Name | Company | Catalog Number | Comments |
| DMEM/F12 | Gibco | 11320082 | |
| L-Glutamine | Gibco | 25030081 | |
| MEM Non-Essential amino acids solution (100X) | Gibco | 11140050 | |
| Penicillin-Streptomycin (10,000 U/mL) | Gibco | 15140122 | |
| Dulbecco’s Phosphate Buffered saline | Gibco | 14190144 | |
| Trypsin-EDTA | Gibco | 25300054 | |
| Fetal bovine serum | Atlanta Biologicals | S11150 | |
| DMSO | EMD Millipore | MX 1458-6 | |
| Cryogenic vial storage boxes | Fisherbrand | 10-500-28 | |
| Cryogenic vials | Corning/costar | 431416 | |
| T75 flasks | Cellstar | 658175 | |
| 2 well chambered cover glass | Nunc | 155380PK | |
| Cellometer Vision Cell Profiler | Nexcelom Bioscience LLC | Cellometer Vision Trio | |
| Nocodazole | Sigma | M1404 | |
| CellLight Histone 2B-GFP, BacMam 2.0 | Thermo Fisher Scientific Inc. | C10594 | |
| Zeiss Cell Observer Spinning Disk Confocal Microscope system | Carl Zeiss Microscopy | Zeiss Cell Observer SD | |
| Water Bath | Thermoscientific | 280 series | |
| Incubator | Sanyo commercial solutions | MCO-18AIC (UV) | |
| Class II Biological safety cabinet | The Baker Company | SterilGard | |
| Axiovision software | Zeiss | Ver.4.8.2 | |
| ImageJ software | National Institute of Health | Ver. 1.51r |
Riferimenti
- Funk, L. C., Zasadil, L. M., Weaver, B. A. Living in CIN: Mitotic Infidelity and Its Consequences for Tumor Promotion and Suppression. Dev Cell. 39, 638-652 (2016).
- Etemad, B., Kops, G. J. Attachment issues: kinetochore transformations and spindle checkpoint silencing. Curr Opin Cell Biol. 39, 101-108 (2016).
- Lara-Gonzalez, P., Westhorpe, F. G., Taylor, S. S. The spindle assembly checkpoint. Curr Biol. 22, 966-980 (2012).
- Jallepalli, P. V., Lengauer, C. Chromosome segregation and cancer: cutting through the mystery. Nat Rev Cancer. (2), 109-117 (2001).
- Zhu, L., et al. Mitotic protein CSPP1 interacts with CENP-H protein to coordinate accurate chromosome oscillation in mitosis. J Biol Chem. 290 (45), 27053-27066 (2015).
- Pikor, L., Thu, K., Vucic, E., Lam, W. The detection and implication of genome instability in cancer. Cancer Metastasis Rev. 32 (3-4), 341-352 (2013).
- Varadkar, P., Despres, D., Kraman, M., Lozier, J., Phadke, A., Nagaraju, K., McCright, B. The protein phosphatase 2A B56γ regulatory subunit is required for heart development. Dev Dyn. 243 (6), 778-790 (2014).
- Jozefczuk, J., Drews, K., Adjaye, J. Preparation of Mouse Embryonic Fibroblast Cells Suitable for Culturing Human Embryonic and Induced Pluripotent Stem Cells. J. Vis. Exp. (64), e3854 (2012).
- Varadkar, P., Abbasi, F., Takeda, K., Dyson, J. J., McCright, B. PP2A-B56γ is required for an efficient spindle assembly checkpoint. Cell Cycle. 18 (12), 1210-1219 (2017).
- Rosner, M., Schipany, K., Hengstschläger, M. Merging high-quality biochemical fractionation with a refined flow cytometry approach to monitor nucleocytoplasmic protein expression throughout the unperturbed mammalian cell cycle. Nat Protoc. 8 (3), 602-626 (2013).
- Jonkman, J., Brown, C. M. Any Way You Slice It-A Comparison of Confocal Microscopy Techniques. J Biomol Tech. 26 (2), 54-65 (2015).
- Versaevel, M., Braquenier, J. B., Riaz, M., Grevesse, T., Lantoine, J., Gabriele, S. Super-resolution microscopy reveals LINC complex recruitment at nuclear indentation sites. Sci Rep. 8 (4), 7362 (2014).
- Wild, T., Larsen, M. S., Narita, T., Schou, J., Nilsson, J., Choudhary, C. The Spindle Assembly Checkpoint Is Not Essential for Viability of Human Cells with Genetically Lowered APC/C Activity. Cell Rep. 1 (8), 1829-1840 (2016).
- Iuso, D., et al. Exogenous Expression of Human Protamine 1 (hPrm1) Remodels Fibroblast Nuclei into Spermatid-like Structures. Cell Rep. 1 (9), 1765-1771 (2015).
- Ratcliffe, E., Glen, K. E., Naing, M. W., Williams, D. J. Current status and perspectives on stem cell-based therapies undergoing clinical trials for regenerative medicine: case studies. Br Med Bull. 108, 73-94 (2013).
Ristampe e Autorizzazioni
Richiedi autorizzazione per utilizzare il testo o le figure di questo articolo JoVE
Richiedi AutorizzazioneThis article has been published
Video Coming Soon