È necessario avere un abbonamento a JoVE per visualizzare questo. Accedi o inizia la tua prova gratuita.
Method Article
Nanosensori per rilevare attività della proteasi In Vivo per la diagnostica non invasiva
In questo articolo
Riepilogo
Proteasi sono strettamente regolamentati enzimi coinvolti in processi biologici fondamentali e dysregulated proteasi attività unità progressione di malattie complesse come il cancro. Obiettivo di questo metodo consiste nel creare nanosensori che misurano proteasi attività in vivo producendo un segnale di fenditura che è rilevabile dall'urina di host e discrimina la malattia.
Abstract
Proteasi sono enzimi multifunzionali che si specializzano nell'idrolisi di legami peptidici e controllano ampi processi biologici, compreso l'omeostasi e Allostasi. Inoltre, l'attività della proteasi dysregulated unità patogenesi ed è un biomarcatore funzionale di malattie come il cancro; Pertanto, la capacità di rilevare attività della proteasi in vivo può fornire informazioni clinicamente rilevanti per la diagnostica biomedica. L'obiettivo del presente protocollo è quello di creare nanosensori sonda per attività della proteasi in vivo producendo un segnale quantificabile nelle urine. Queste proteasi nanosensori costituiti da due componenti: una nanoparticella e substrato. Le funzioni di nanoparticelle per aumentare la consegna di Half-Life e substrato di circolazione malattia nei siti di destinazione. Il substrato è una sequenza di brevi peptidi (6-8 AA), che è progettata per essere specifici di una proteasi di destinazione o un gruppo di proteasi. Il substrato è coniugato alla superficie della nanoparticella e viene terminato da un giornalista, ad esempio un marker fluorescente, per il rilevamento. Come proteasi dysregulated fendono il substrato peptide, il reporter viene filtrato in urina per quantificazione come biomarcatore di attività della proteasi. Qui descriviamo la costruzione di un nanosensore per proteinasi metallica della tabella 9 (MMP9), che è associato con la progressione del tumore e metastasi, per la rilevazione di cancro colorettale in un modello murino.
Introduzione
Proteasi sono enzimi multifunzionali che si specializzano nell'idrolisi di legami peptidici e hanno il controllo significativo rispetto a molti processi biologici, compreso l'omeostasi, Allostasi e malattia1. Uno stato alterato di attività della proteasi è stato correlato ad una varietà di malattie, compreso cancro e la malattia cardiovascolare, rendendo i candidati attraenti di proteasi per sviluppo in biomarker clinici2,3. Inoltre, l'attività della proteasi è funzionalmente collegati a distinte pathogeneses, gli esiti dei pazienti e la prognosi di malattia4. In generale, biosensori sono state sviluppate per rilevare vari fenomeni biologici e malattie, quali cancro, malattie neurodegenerative e trasferimento di elettroni elabora5,6,7,8 , 9. più specificamente, sensori basati su substrato della proteasi sono stati sviluppati per rilevare l'attività della proteasi e includono sonde fluorogenic per diagnostica per immagini10 e isotopicamente etichettati substrati del peptide per in vitro rilevamento di spettrometria di massa11. Inoltre, sonde basate su attività sono state sviluppate, che contengono regioni di substrato-come che legano o modificare la destinazione della proteasi12. Con questo metodo, la proteasi di destinazione è irreversibilmente inibita quando viene modificato il sito attivo e analisi richiedono la raccolta di tessuto, che limita le applicazioni in vivo . Tuttavia, è importante rilevare proteasi attività in vivo, perché il regolamento di attività della proteasi è fortemente dipendente dal contesto di altre attività biologiche quali la presenza di inibitori endogeni.
L'obiettivo di questo lavoro è di descrivere la formulazione dei nanosensori basati su attività che rilevano attività della proteasi in vivo producendo un segnale misurabile nelle urine. Questa piattaforma è utilizzata come una diagnostica non invasiva per discriminare complesse malattie come il cancro utilizzando l'attività della proteasi dysregulated come biomarcatore funzionale. La nostra piattaforma nanosensore è costituito da nanoparticelle di ossido di ferro (IONP) coniugate a substrati di proteasi. Questi substrati vengono terminati da un reporter fluorescente che viene rilasciato quando proteasi fendono il substrato. Questi IONPs circolare in vivo, siti di malattia si localizzano ed esporre substrati alle proteasi attive di malattia-collegati. Dopo la scissione, reporter fluorescenti vengono rilasciati e, grazie alle loro piccole dimensioni, vengono filtrati in urina, mentre substrati ApoAlert sulla IONP rimangono nel corpo. Pertanto, un aumento di attività della proteasi in vivo si tradurrà in concentrazioni più elevate di reporter nelle urine (Figura 1). Poiché la nostra piattaforma è un test delle urine, nessuna piattaforma di imaging è obbligatorio e segnali diagnostici sono arricchiti in urina.
Questa piattaforma può essere progettata per rilevare una varietà di malattie compreso il cancro, fibrosi e trombosi13,14. Qui descriviamo il design dei nanosensori per rilevare le elevazioni in Matrix metallopeptidase 9 (MMP9) attività come biomarcatore di cancro colorettale. Il cancro colorettale è la seconda causa principale di morte per cancro negli Stati Uniti, con circa 136.800 nuovi casi e 50.300 morti nel 2014 solo15. Le cellule del tumore del colon-retto producono MMP9, che ha dimostrato di guidare la progressione maligna, la degradazione della matrice, così come metastasi16. Inoltre, abbiamo identificato un substrato adatto peptide (PLGVRGK) per MMP9 dalla letteratura17. Questa piattaforma può essere utilizzata per la diagnosi precoce del cancro e diagnostica point-of-care basso costo13,14,18,19,20,21.
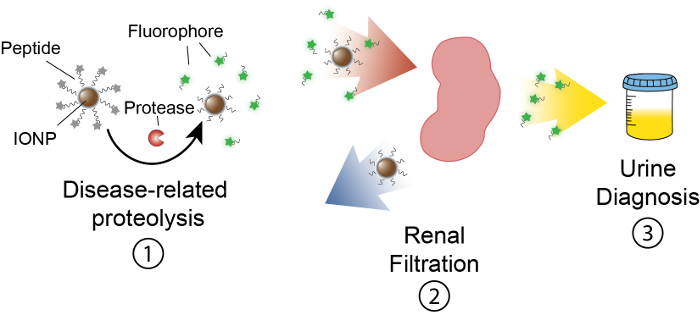
Figura 1: schematico di nanosensori attività In vivo. Nanosensori circolano attraverso il corpo e si localizzano siti di malattia. Quindi, correlati alla malattia proteasi fendono substrati del peptide presentati da IONPs. Le dimensioni dei frammenti fenduti consente la clearance renale, causando loro di localizzare nelle urine. Dopo che l'animale urina, questi frammenti peptidici possono essere analizzati dalla loro molecola reporter. Clicca qui per visualizzare una versione più grande di questa figura.
Protocollo
È necessario effettuare i seguenti esperimenti animali istituzionale approvazione istituzionale Animal Care ed uso Committee (IACUC) alla istituzione del ricercatore. Inoltre, strutture di standard di cura degli animali (ad es., alloggiamento chambers, cappe animale sterile, surrenalectomia alloggiamenti per amputate e CO2 alloggiamenti per euthanization etico endpoint) sono necessarie per svolgere correttamente queste esperimenti. Speciale formazione e assistenza con queste strutture può essere forniti da laboratorio di ricerca fisiologica (PRL) alla istituzione di uno. Tutto il lavoro animale è stato approvato dal IACUC al Georgia Tech (protocollo: A14100).
1. sintesi di nanoparticelle (IONP) di ossido di ferro
Nota: Sicurezza: l'intera sintesi di nanoparticelle di ossido di ferro devono essere effettuate utilizzando dispositivi di protezione individuale e sotto un prodotto chimico cappa aspirante.
- In una provetta conica da 15 mL, chill 1 mL di idrossido di ammonio di 30% in un secchio di ghiaccio per 30 min.
- Lavare un matraccio di Erlenmeyer con acqua deionizzata da 250 mL e aggiungere 10 mL di acqua distillata doppia (ddH2O). Immergere l'esterno del pallone in un bagno di ghiaccio che riposa su una piastra stir/calore. Utilizzare una pipetta di plastica per gas2 flusso N nei ddH2O nel pallone. Bolla con N2 a deoxygenate per 15 minuti.
- Pesare µmol 224 (4,5 grammi) di destrano (peso molecolare (MW) = 20 kDa) in una provetta conica da 50 mL. Tale che il volume finale della miscela, compreso il destrano, è di 20 mL, aggiungere H2O. Vortice vigorosamente per sciogliere il destrano.
- Pesare 290 µmol (78,5 grammi) di ferro (III) cloruro esaidrato, aggiungere alla soluzione del dextrano e vortex per sciogliere.
- Filtrare la soluzione attraverso un filtro da 0,2 µm.
- Trasferire 1 mL refrigerati di idrossido di ammonio in 9 mL di acqua deossigenata. Tornare a 4 ° C.
- Pesare µmol 367 (73,0 grammi) di ferro (II) cloruro tetraidrato, sciogliere in 1 mL di ossigeno-libero ddH2O e filtrare attraverso un filtro da 0,2 µm.
- Trasferire la soluzione di dextran-iron(III) nella beuta da 250 mL e deoxygenate con N2 per 15 minuti sul ghiaccio. Mescolare con un ancoretta magnetica di filatura a 1600 giri/min.
- Per creare un'atmosfera di azoto omogenea, tappo la beuta con un setto di gomma. Puntura del setto con un 18 ago a flusso N2 del manometro nel pallone. Inserire un ago 18 calibro separato come una presa di flusso.
- Aggiungere alla soluzione di dextran-iron(III) 467 µ l della soluzione di ferro (II) con una siringa da 1 mL, mescolando con un ancoretta magnetica a 1600 giri/min. Questo rapporto di ferro (II) di ferro (III) provoca una reazione equilibrata che produrrà magnetite o Fe3O4.
- Aggiungere che il refrigerate diluire idrossido di ammonio goccia a goccia nella soluzione del dextrano del ferro (II) - ferro (III) - per avviare il processo di nucleazione21. Assicurarsi che ogni goccia mescola bene prima di cadere in un altro. Finire la reazione dopo l'aggiunta di 1-2 mL di idrossido di ammonio.
- Arrestare il flusso di N2 e rimuovere il setto di gomma. Il bagno di ghiaccio di rimuovere e sostituire con una vasca di acqua calda, pur continuando a mescolare la soluzione. Assicurarsi che la temperatura della soluzione raggiunge i 75 ° C ed incubare per 75 minuti.
- Togliere il matraccio dalla piastra calda mescolare e doppiamente filtrare la soluzione attraverso 0,2 µm quindi 0,1 µm filtri per rimuovere le particelle grossolane.
- Cambio di buffer le particelle in ddH2O, utilizzando il peso molecolare di 100 kDa tagliate con concentratori (m.w.c.o.) per rimuovere l'eccesso destrano dalla soluzione, che dovrebbe essere molto viscosa. Sostituire i filtri, se il flusso attraverso rimane oscuro anche dopo 2-3 giri, che indica che il filtro potrebbe essersi rotto.
- Nel buffer di scambio con filtri spin, centrifugare a 4 ° C a 4800 X g per 15 minuti, scartare il flusso continuo e aggiungere nuovo buffer alla soluzione IONP. Ripetere 3 - 5 volte.
- Determinare la concentrazione delle nanoparticelle utilizzando un lettore di spettrofotometro/piastra. Prendere misure di assorbanza a 400 nm e uso l'assorbività molare di IONP a questa lunghezza d'onda (ε = 2.07 x 106 cm-1 M-1) per determinare la concentrazione di particelle IO.
- Risospendere il IONPs a 10 mg/mL in ddH2O (volume totale è solitamente ~ 3 mL) e assicurarsi di utilizzare tubi in polipropilene per questo passaggio per compatibilità chimica. Aggiungere 1,6 volumi di 5 M NaOH, quindi aggiungere 0,65 volumi di Epicloridrina. Rigorosamente mescolare su un agitatore per piastre a temperatura ambiente per 12 ore per crosslink destrano.
- Utilizzare una siringa da 20 mL con un ago calibro 18 per trasferire la soluzione IONP nella membrana di dialisi di m.w.c.o. di 50 kDa. Posizionare la membrana di dialisi in 4L ddH2O e sostituire H2O un paio di volte (ogni 2-3 ore). Incubare per una notte.
- Misurare la concentrazione e portare IONPs a 5 mg/mL (Vedi punto 1.15). Aggiungere idrossido di ammonio per raggiungere il 20% (v/v) ed agitare a temperatura ambiente per > 12 ore per laminato con la superficie del IONPs. Cambio di buffer utilizzando 30 kDa m.w.c.o. filtri (Vedi 1.14).
- Regolare il volume fino a 2,5-3,5 mL prima purificazione finale di Fast Protein Liquid Chromatography (FPLC; Vedi Tabella materiali per colonna di gel filtrazione).
- Utilizzare diffusione dinamica della luce (DLS) per determinare il raggio idrodinamico di IONPs (intervallo previsto 10-100 nm, dimensione media 40-50 nm). A tale scopo aliquotare 1 mL del campione diluito IONP in una cuvetta: 100x, posizionare in macchina e utilizzare il software del produttore per prendere la misura.
Nota: La popolazione totale di IONPs sarà tra 10 e 100 nm mediante misure di liste di distribuzione, ma la maggior parte della popolazione è intorno al diametro medio, che vanno da 40-50 nm. Se si vuole limitare la gamma di dimensioni, si può usare ulteriormente cromatografia di esclusione di formato per isolare frazioni con diametri diversi. IONPs deve essere conservato a 4 ° C.
2. peptide Design, coniugazione IONP, e In Vitro di convalida
- Sintetizzare un substrato peptide (ad es., da una struttura di nucleo o commercialmente) per una proteasi di destinazione con un reporter fluorescente del N-terminale come ad esempio isotiocianato di fluorescina (FITC) e un residuo di cisteina terminale C per permettere l'aggancio del tiolo-mediata.
Nota: Nel caso di questo studio per MMP9, il peptide utilizzato era FITC-PLGVRGK-C, con kgatto/km ~ 2.0 x 105 M-1s-1. - Aliquota 0,5 mg di IONP (a 1 mg/mL) e lo scambio di buffer nel buffer di accoppiamento (50mm borato di sodio con 1 mM EDTA, pH = 8,5) utilizzando un giro di m.w.c.o. di kDa 10 filtro (Vedi punto 1.14).
- Sciogliere Succinimidyl iodoacetato (SIA) in dimetilformammide (DMF) per raggiungere una concentrazione di ~ 30 mg/mL e aggiungere SIA IONP in un rapporto di mole di 500 SIA:IONP.
Nota: SIA è un eterobifunzionali Croce del linker che media l'accoppiamento delle ammine su IONPs ai gruppi solfidrilici sul substrato del peptide. - Incubare per 1-2 ore a temperatura ambiente al buio. Se lasciato durante la notte, incubare a 4 ° C.
- Buffer di exchange utilizzando un filtro di spin m.w.c.o. 10 kDa in tampone di accoppiamento per rimuovere SIA non reagito (Vedi punto 1.14).
- Portare la soluzione di prodotto finale a 1 mg/mL (0,5 mL di soluzione). Mescolare il peptide di interesse con un rapporto molare di 90:1 (peptide: IONP) con 20 kDa con terminazione del tiolo polietilenglicole (PEG) con un rapporto molare di 20:1 (PEG: IONP). Mescolare questa soluzione peptide-PEG con IONP.
- Incubare per una notte a temperatura ambiente su un agitatore per piastre sulla velocità più alta, copertura tubi in carta stagnola per evitare che photobleaching di molecole fluorescenti.
- Aggiungere L-cisteina in un rapporto molare di 500: 1 (C:IONP) per neutralizzare eventuali molecole SIA non reagite. Incubare per 1 ora a 25 ° C in un agitatore a velocità massima a temperatura ambiente.
- Purificare tramite FPLC (Vedi 1.1.19). Utilizzare uno spettrofotometro per misurare l'assorbanza della soluzione del campione e calcolare il rapporto di Peptide: IONP secondo le seguenti. Dopo la purificazione, conservare il prodotto a 1 mg/mL a 4 ° C in PBS.
- Utilizzare uno spettrofotometro per misurare l'assorbanza della soluzione del campione a 400 nm (uncampione, 400) e la lunghezza d'onda di massima assorbenza per il fluoroforo utilizzato, che è 488 nm per FITC (uncampione, 488). Misurare l'assorbanza di una soluzione IONP di ammina stock presso la stesse due lunghezze d'onda (400 nm e 488 nm), che sono rappresentati rispettivamente come unNP, 400 e unNP, 488.
- Utilizzare le equazioni 1 e 2 per calcolare il normalizzato A400 e un valori di488 .
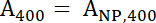 Equazione 1
Equazione 1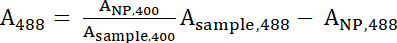 Equazione 2
Equazione 2 - Un400 e un488 rappresentano i valori normalizzati del campione. Utilizzare questi valori per calcolare il rapporto di Peptide: IONP con equazione 3.
 Equazione 3
Equazione 3 - Dove
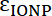 e
e 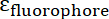 sono i absorptivities molare il IONP e il fluoroforo, rispettivamente. Per queste particelle
sono i absorptivities molare il IONP e il fluoroforo, rispettivamente. Per queste particelle 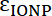 = 2,06 x 106 M-1cm-1 e per FITC
= 2,06 x 106 M-1cm-1 e per FITC 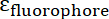 = 72.000 M-1cm-1, quindi il valore di
= 72.000 M-1cm-1, quindi il valore di  dovrebbe essere 28,75. Rapporti tipici Peptide: IONP dovrebbero essere nel range di 20:1 a 50: 1.
dovrebbe essere 28,75. Rapporti tipici Peptide: IONP dovrebbero essere nel range di 20:1 a 50: 1.
- Convalidare la funzionalità delle sonde eseguendo un'analisi di clivaggio in vitro .
- Con PBS (1% BSA), rendere una nanoparticella di 18 µ l di soluzione con una concentrazione di 200 nM del peptide. Mescolare con 2 µ l di proteasi di interesse (MMP9; 0,1 - 1 mg/mL).
- Incubare in un lettore di piastre a 37 ° C per 1 ora, prendendo misure di fluorescenza (utilizzando l'appropriato eccitazione e lunghezze d'onda di emissione, che sono 485 nm e 528 nm per FITC, rispettivamente) ogni 1-2 minuti per monitorare la fenditura.
3. amministrazione di nanosensori e urina rilevazione di cancro
Nota: Per maggiori dettagli sul modello di esempio, vedere la nostra precedente relazione13.
- Creare una gabbia metabolica per la raccolta dell'urina fissando un manicotto cilindrico nella parte superiore di una piastra ben 96. Durante la raccolta delle urine, posizionare il mouse all'interno della manica e la copertura con una copertura di Petri per impedire la fuga dell'animale.
- Preparare una soluzione per iniezione (200 µ l di volume massima) contenente nanosensori ad una concentrazione di 3000 mg/kg (~ 50 µmol) dal peptide in soluzione fisiologica sterile.
- Nei topi femminili, nudi, 8 - settimana-vecchi cuscinetto dello xenotrapianto tumori colorettali LS174T a un peso di ~ 100-300 mm3, che dovrebbe verificarsi circa il giorno 10, amministrare nanosensore soluzione tramite iniezione della vena della coda. Immediatamente dopo l'iniezione, posizionare il mouse in gabbie metaboliche e prendere nota del tempo di iniezione per ogni mouse.
- A 60-90 minuti dopo l'iniezione, rimuovere il manicotto cilindrico da piastra a 96 pozzetti. Reprimere il mouse ed applicare una leggera pressione sulla vescica per indurre topi di annullare qualsiasi restante urina sulla piastra. Raccogliere tutte le urine (200-500 µ l).
- Analizzare i campioni di urina mediante immunoprecipitazione per purificare FITC dall'urina e aumentare la sensibilità. Biglie magnetiche uso accoppiato con gli anticorpi anti-FITC.
- In primo luogo, lavare 25mg di biglie magnetiche 3 volte con tampone di rivestimento e portare il volume finale a 225 µ l.
Nota: È importante rendere fresca che ogni volta e circa 20 mL di ciascuno è necessario il tampone di rivestimento (borato di sodio 0,1 M, pH 9.5), tampone bloccante (PBS, 0.5% di BSA, 0,05% di Tween-20, pH 7.4) e tampone di lavaggio (PBS, 0.1% BSA, 0,05% di Tween-20, pH 7.4). - Aggiungere 200 µ l di anti-FITC (5 mg/mL) e 200 µ l di solfato di ammonio (3 M). Incubare per 16-24 ore a 37 ° C in un rotatore.
- Aggiungere la soluzione a un separatore magnetico, eliminare il surnatante, sostituire con 625
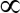 Λ blocco buffer e incubare a 37 ° C per una notte. Quindi lavare 3 volte con tampone di lavaggio e memorizzare in 1,25 mL di tampone di lavaggio.
Λ blocco buffer e incubare a 37 ° C per una notte. Quindi lavare 3 volte con tampone di lavaggio e memorizzare in 1,25 mL di tampone di lavaggio. - Incubare 2 µ l di urina con 5 µ l di biglie magnetiche (20 mg/mL) e portare a un volume totale di 50 µ l con PBS (0.01% Tween 20). Incubare per 60 min.
- Lavare due volte con 50 µ l PBS (0.01% Tween 20) utilizzando un separatore magnetico per raccogliere perle magnetiche dopo ogni lavaggio.
- Eluire due volte con 32,5 µ l di acido acetico glaciale di 5%. Neutralizzare l'eluizione in pool (70 µ l) con 35 µ l di 2 M Tris per ottenere un pH finale di ~ 7.
- Leggere su un lettore di piastra (Vedi Tabella materiali) alle lunghezze d'onda eccitazione e di emissione appropriato per quantificare la fluorescenza di urina. Calcolare la concentrazione di reporter fluorescente contro una scala di concentrazioni note di fluoroforo gratis.
- In primo luogo, lavare 25mg di biglie magnetiche 3 volte con tampone di rivestimento e portare il volume finale a 225 µ l.
Risultati
La maggior parte della popolazione della IONPs è intorno al diametro medio, che vanno da 40-50 nm. Dopo PEGilazione, questo intervallo di grandezza ha un'emivita di circolazione di circa 6 ore13 in vivo (Vedi Figura 2a). Se si vuole selezionare per una gamma di particolari dimensioni, uno può utilizzare cromatografia di esclusione di formato per isolare le frazioni IONP con differenti diametri. Le nanoparticelle di TEM appa...
Discussione
Questo metodo viene descritto lo sviluppo di Nanosensori basati su attività costituito da proteasi substrati coniugati ad un nucleo di nanoparticelle. L'evento di clivaggio proteolitico è soprannominato "switch farmacocinetiche," perché prodotti fenduti peptidici sono più piccoli rispetto al limite di filtrazione renale dimensione di 5 nm23 e filtro in urina per produrre un segnale non invadente. Pertanto, è importante utilizzare le nanoparticelle o vettori con un raggio idrodinamico è maggi...
Divulgazioni
Dr. Kwong è co-fondatore e serve come consulente a Glympse Bio, che sta sviluppando prodotti legate alla ricerca descritta in questo documento. Questo studio potrebbe influire sulla sua situazione finanziaria personale. I termini di questo accordo sono stati esaminati e approvati dal Georgia Tech secondo le proprie politiche di conflitto di interessi
Riconoscimenti
Questo lavoro è stato finanziato da un direttore di NIH nuovo Innovator Award (premio No. DP2HD091793). Q.D.M. è supportato dal programma di borse di ricerca laureato NSF (Grant No. DGE-1650044). B.A.H è supportato dalla nazionale istituti di salute GT BioMAT formazione sovvenzione premio numero 5T32EB006343 così come il Presidente di Georgia Tech Fellowship. G.A.K. tiene un premio alla carriera all'interfaccia scientifica del fondo benvenuto di Burroughs. Il contenuto è di esclusiva responsabilità degli autori e non rappresentano necessariamente il punto di vista ufficiale del National Institutes of Health.
Materiali
| Name | Company | Catalog Number | Comments |
| 0.2 µm syringe filters | VWR | 4652 | |
| 18G needle | VWR | 89134-024 | |
| 15 mL conicals | VWR | 89039-670 | |
| 250 mL Erlenmeyer flask | VWR | 89000-362 | |
| Stir bar | VWR | 58949-006 | |
| Hot Plate/Magnetic Stirrer | VWR | 97042-634 | |
| Glacial acetic acid | VWR | 97064-482 | |
| Albumin from Bovine Serum (BSA) | Thermo Fisher | A13100 | |
| Iron (III) chloride hexahydrate | Sigma | 236489 | |
| Iron (II) chloride tetrahydrate | Sigma | 44939 | |
| Epichlorohydrin | Sigma | 45340-500ML-F | |
| DMF | Sigma | D4551 | |
| Ammonium Hydroxide | Sigma | 320145-500ML | |
| Sodium Hydroxide pellets | Sigma | 221465-500G | |
| EDTA | Sigma | E9884 | |
| Sodium Borate | Sigma | B9876 | |
| L-Cysteine | Sigma | 168149-100G | |
| Tris-HCl | Sigma | T5941 | |
| Tris base | Sigma | T6066 | |
| PBS tablets | Sigma | P4417 | |
| Dextran | Pharmacosmos | 5510 0020 9006 | |
| Amicon 15 mL 10k filters, 24 pk | Millipore | UFC901024 | |
| Amicon 15 mL 30k filters, 24 pk | Millipore | UFC903024 | |
| Amicon 15 mL 100k filters, 24 pk | Millipore | UFC910024 | |
| Zetasizer Nano ZS | Malvern Panalytical | NanoZS | |
| Slide-A-Lyzer Dialysis Cassette | LifeTech | 66130 | |
| Dynabeads MyOne Tosylactivated | LifeTech | 65501 | |
| SIA | Life Tech | 22349 | |
| PEG 20k | Laysan Bio | MPEG-SH-20K-1g | |
| Fluorescein Antibody [2A3] | GeneTex | GTX10257 | |
| Hiload 16/600 superdex 200 | GE Healthcare | 45-002-490 | |
| Plate Reader | Fisher | BTCYT5M | |
| BD Insulin Syringes | Fisher | NC0872854 |
Riferimenti
- Lopez-Otin, C., Bond, J. S. Proteases: Multifunctional enzymes in life and disease. Journal of Biological Chemistry. 283 (45), 30433-30437 (2008).
- Hua, Y., Nair, S. Proteases in cardiometabolic diseases: Pathophysiology, molecular mechanisms and clinical applications. Biochimica et Biophysica Acta. 1852 (2), 195-208 (2015).
- Koblinski, J. E., Ahram, M., Sloane, B. F. Unraveling the role of proteases in cancer. Clinica Chimica Acta. 291 (2), 113-135 (2000).
- Lilja, H., Vickers, A., Scardino, P. Measurements of proteases or protease system components in blood to enhance prediction of disease risk or outcome in possible cancer. Journal of Clinical Oncology. 25 (4), 347-348 (2007).
- Jin, H., et al. Flexible surface acoustic wave resonators built on disposable plastic film for electronics and lab-on-a-chip applications. Scientific Reports. 3, 2140 (2013).
- Ma, W., Liu, H. -. T., Long, Y. -. T. Monitoring dopamine quinone-induced dopaminergic neurotoxicity using dopamine functionalized quantum dots. ACS Applied Materials & Interfaces. 7 (26), 14352-14358 (2015).
- Zhang, W. -. H., Ma, W., Long, Y. -. T. Redox-mediated indirect fluorescence immunoassay for the detection of disease biomarkers using dopamine-functionalized quantum dots. Analytical Chemistry. 88 (10), 5131-5136 (2016).
- Ma, W., et al. Investigating electron-transfer processes using a biomimetic hybrid bilayer membrane system. Nature Protocols. 8 (3), 439-450 (2013).
- Holt, B. A., et al. Fc microparticles can modulate the physical extent and magnitude of complement activity. Biomaterials science . 5, 463-474 (2017).
- Edgington, L. E., Verdoes, M., Bogyo, M. Functional imaging of proteases: Recent advances in the design and application of substrate-based and activity-based probes. Current Opinion in Chemical Biology. 15 (6), 798-805 (2011).
- Kleifeld, O., et al. Identifying and quantifying proteolytic events and the natural N terminome by terminal amine isotopic labeling of substrates. Nature Protocols. 6 (10), 1578-1611 (2011).
- Sanman, L. E., Bogyo, M. Activity-based profiling of proteases. Annual Review of Biochemistry. 83 (1), 249-273 (2014).
- Kwong, G. A., et al. Mass-encoded synthetic biomarkers for multiplexed urinary monitoring of disease. Nature Biotechnology. 31 (1), 63-70 (2013).
- Dudani, J. S., Buss, C. G., Akana, R. T. K., Kwong, G. A., Bhatia, S. N. Sustained-release synthetic biomarkers for monitoring thrombosis and inflammation using point-of-care compatible readouts. Advanced Functional Materials. 26 (17), 2919-2928 (2016).
- Meester, R. G. S., et al. Public health impact of achieving 80% colorectal cancer screening rates in the United States by 2018. Cancer. 121 (13), 2281-2285 (2015).
- Mehner, C., et al. Tumor cell-produced matrix metalloproteinase 9 (MMP-9) drives malignant progression and metastasis of basal-like triple negative breast cancer. Oncotarget. 5 (9), 2736-2749 (2014).
- Bremer, C., Tung, C. H., Weissleder, R. In vivo molecular target assessment of matrix metalloproteinase inhibition. Nature Medicine. 7 (6), 743-748 (2001).
- Warren, A. D., et al. Disease detection by ultrasensitive quantification of microdosed synthetic urinary biomarkers. Journal of the American Chemical Society. 136 (39), 13709-13714 (2014).
- Warren, A. D., Kwong, G. A., Wood, D. K., Lin, K. Y., Bhatia, S. N. Point-of-care diagnostics for noncommunicable diseases using synthetic urinary biomarkers and paper microfluidics. Proceedings of the National Academy of Sciences of the United States of America. 111 (10), 3671-3676 (2014).
- Kwong, G. A., et al. Mathematical framework for activity-based cancer biomarkers. Proceedings of the National Academy of Sciences of the United States of America. 112 (41), 12627-12632 (2015).
- Dudani, J. S., Jain, P. K., Kwong, G. A., Stevens, K. R., Bhatia, S. N. Photoactivated spatiotemporally-responsive nanosensors of in vivo protease activity. ACS Nano. 9 (12), 11708-11717 (2015).
- Palmacci, S., Josephson, L. Synthesis of polysaccharide covered superparamagnetic oxide colloids. US patent. , (1991).
- Choi, H. S., et al. Renal clearance of nanoparticles. Nature Biotechnology. 25 (10), 1165-1170 (2007).
- Harris, T. J., von Maltzahn, G., Derfus, A. M., Ruoslahti, E., Bhatia, S. N. Proteolytic actuation of nanoparticle self-assembly. Angewandte Chemie (International edition in English). 45 (19), 3161-3165 (2006).
- Kwon, E. J., Dudani, J. S., Bhatia, S. N. Ultrasensitive tumour-penetrating nanosensors of protease activity. Nature Biomedical Engineering. 1, (2017).
- Villanueva, J., Nazarian, A., Lawlor, K., Tempst, P. Monitoring Peptidase activities in complex proteomes by MALDI-TOF mass spectrometry. Nature Protocols. 4 (8), 1167-1183 (2009).
- Villanueva, J., et al. Differential exoprotease activities confer tumor-specific serum peptidome patterns. The Journal of Clinical Investigation. 116 (1), 271-284 (2006).
Ristampe e Autorizzazioni
Richiedi autorizzazione per utilizzare il testo o le figure di questo articolo JoVE
Richiedi AutorizzazioneThis article has been published
Video Coming Soon