Method Article
Colorazione e imaging ad alta risoluzione di modelli organoidi e sferoidi tridimensionali
* Questi autori hanno contribuito in egual misura
In questo articolo
Riepilogo
Qui, forniamo protocolli dettagliati, robusti e complementari per eseguire la colorazione e l'imaging a risoluzione subcellulare di modelli di coltura cellulare tridimensionale fissi che vanno da 100 μm a diversi millimetri, consentendo così la visualizzazione della loro morfologia, composizione del tipo di cellula e interazioni.
Abstract
I modelli di coltura cellulare tridimensionale (3D) in vitro, come organoidi e sferoidi, sono strumenti preziosi per molte applicazioni, tra cui lo sviluppo e la modellazione di malattie, la scoperta di farmaci e la medicina rigenerativa. Per sfruttare appieno questi modelli, è fondamentale studiarli a livello cellulare e subcellulare. Tuttavia, la caratterizzazione di tali modelli di coltura cellulare 3D in vitro può essere tecnicamente impegnativa e richiede competenze specifiche per eseguire analisi efficaci. Qui, questo documento fornisce protocolli dettagliati, robusti e complementari per eseguire la colorazione e l'imaging con risoluzione subcellulare di modelli di coltura cellulare 3D fissi in vitro che vanno da 100 μm a diversi millimetri. Questi protocolli sono applicabili a un'ampia varietà di organoidi e sferoidi che differiscono per cellula di origine, morfologia e condizioni di coltura. Dalla raccolta della struttura 3D all'analisi delle immagini, questi protocolli possono essere completati entro 4-5 giorni. In breve, le strutture 3D vengono raccolte, fissate e possono quindi essere elaborate attraverso l'incorporazione di paraffina e colorazione istologica / immunoistochimica, o direttamente immunomarcate e preparate per la pulizia ottica e la ricostruzione 3D (profondità 200 μm) mediante microscopia confocale.
Introduzione
Negli ultimi decenni, i progressi nella biologia delle cellule staminali e nelle tecnologie di coltura 3D in vitro hanno annunciato una rivoluzione nella biologia e nella medicina. I modelli cellulari di maggiore complessità in 3D sono diventati molto popolari in quanto consentono alle cellule di crescere e interagire con una struttura extracellulare circostante, ricapitolando da vicino gli aspetti dei tessuti viventi tra cui la loro architettura, l'organizzazione e le interazioni cellulari o persino le caratteristiche di diffusione. Pertanto, i modelli di coltura cellulare 3D possono fornire informazioni uniche sul comportamento delle cellule nei tessuti in via di sviluppo o malati in vitro. Gli organoidi e gli sferoidi sono entrambe strutture 3D multicellulari, che vanno da diversi micrometri a millimetri, e sono le strutture 3D in vitro più importanti. Entrambi possono essere coltivati all'interno di un'impalcatura di supporto che include (i) idrogel derivati da animali (estratto di membrana basale, collagene), piante (alginato / agarosio) o sintetizzati da sostanze chimiche, o (ii) matrici inerti contenenti pori per promuovere la proliferazione e la crescita cellulare.
Organoidi e sferoidi possono anche svilupparsi senza la presenza di un'impalcatura di supporto facendo affidamento sulle cellule per auto-assemblarsi in cluster. Ciò si basa su diverse tecniche come l'uso di materiali non adesivi per inibire l'attaccamento cellulare, la tensione superficiale e la forza gravitazionale (ad esempio, tecniche di caduta sospesa) o la rotazione circolare costante dei vasi (ad esempio, coltura di spinner). In tutti i casi, queste tecniche facilitano le interazioni cellula-cellula e cellula-matrice per superare i limiti della tradizionale coltura cellulare monostrato1. I termini "organoidi" e "sferoidi" sono stati usati in modo intercambiabile in passato, ma ci sono differenze chiave tra questi due modelli di coltura cellulare 3D. Gli organoidi sono cluster cellulari 3D in vitro derivati da cellule staminali pluripotenti o cellule staminali tessuto-specifiche, in cui le cellule si auto-organizzano spontaneamente in progenitori e tipi cellulari differenziati e che ricapitolano almeno alcune funzioni dell'organo di interesse2. Gli sferoidi comprendono una gamma più ampia di strutture 3D multicellulari formate in condizioni non aderenti e possono derivare da una grande varietà di tipi di cellule come linee cellulari immortalizzate o cellule primarie3. Quindi, inerente alle loro origini intrinseche delle cellule staminali, gli organoidi hanno una maggiore propensione all'autoassemblaggio, alla vitalità e alla stabilità rispetto agli sferoidi.
Tuttavia, in sostanza, questi due modelli sono strutture 3D composte da più cellule e le tecniche sviluppate per studiarle sono quindi molto simili. Ad esempio, potenti approcci di imaging a livello di risoluzione di una singola cellula sono necessari per sondare la complessità cellulare di organoidi e sferoidi. Qui, riassumendo l'esperienza di questo gruppo e quella dei leader nel campo degli organoidi4, questo articolo descrive procedure dettagliate per eseguire colorazioni bidimensionali (2D) e 3D a montaggio intero, imaging e analisi della composizione cellulare e subcellulare e dell'organizzazione spaziale di organoidi e sferoidi che vanno da 100 μm a diversi millimetri. In effetti, questa procedura presenta due tipi diversi e complementari di colorazione e acquisizione di immagini per analizzare una grande varietà di dimensioni e tipi di modelli di coltura cellulare 3D in vitro. L'uso dell'uno (analisi 3D a montaggio intero) o dell'altro (analisi di sezione 2D) dipenderà dal modello studiato e dalle risposte ricercate. L'analisi 3D a montaggio intero mediante microscopia confocale può, ad esempio, essere applicata per visualizzare cellule in coltura 3D fino a 200 μm di profondità, indipendentemente dalle dimensioni complessive della struttura 3D, mentre l'analisi di sezioni 2D fornisce approfondimenti su campioni di qualsiasi dimensione, anche se a livello 2D. Questa procedura è stata applicata con successo su una varietà di organoidi4,5 e sferoidi derivati da cellule umane e murine, provenienti da diversi strati germinali embrionali. La panoramica della procedura è illustrata nella Figura 1. Sono indicate le fasi principali, le relazioni tra loro, i passaggi decisivi e i tempi previsti.
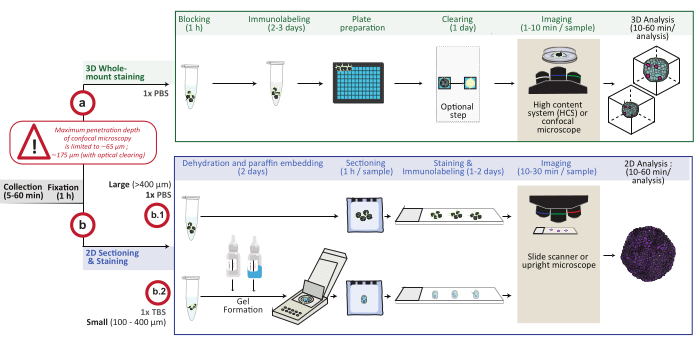
Figura 1: Panoramica schematica della procedura. I modelli di coltura cellulare 3D in vitro vengono raccolti e fissati, quindi preparati per la colorazione 3D a montaggio intero (opzione a) o incorporati in paraffina per il sezionamento e la colorazione 2D (opzione b). Per gli esperimenti di colorazione 3D a montaggio intero, le strutture 3D fisse sono immunomarcate seguendo la fase di fissazione. È possibile eseguire una fase di pulizia ottica opzionale per migliorare la qualità dell'imaging e la profondità della microscopia ottica riducendo la diffusione della luce durante l'elaborazione delle immagini. Le immagini vengono catturate su un microscopio confocale invertito o su un sistema confocale ad alto contenuto e analizzate utilizzando il software appropriato. Per l'incorporazione di paraffina, le strutture 3D vengono elaborate direttamente (opzione b.1 per strutture di grandi dimensioni ≥ 400 μm) o incluse in un gel (b.2; piccole strutture ≤ 400 μm) per la disidratazione e l'incorporazione di paraffina. I blocchi di paraffina vengono quindi tagliati e colorati (colorazione istologica o immunochimica). Le immagini delle sezioni 2D sono ottenute su uno scanner digitale o un microscopio verticale e analizzate su una piattaforma di analisi delle immagini utilizzando un'analisi quantitativa digitale veloce. Fare clic qui per visualizzare una versione ingrandita di questa figura.
Protocollo
NOTA: una perdita del ≤25% del numero iniziale di strutture 3D dovrebbe essere prevista durante le fasi che comportano la sostituzione del reagente e il lavaggio nella seguente procedura. Pianificare l'utilizzo di un numero finale di almeno dieci strutture 3D, con una dimensione compresa tra 100 e 500 μm, per condizione testata per eseguire analisi qualitative e quantitative delle immagini. Se necessario, per le strutture più grandi, tagliare le estremità delle punte delle pipette da 1 mL per evitare di rompere le strutture. Per tutte le fasi, se la sedimentazione della struttura 3D è troppo lunga, le cellule possono essere fatte girare delicatamente a 50 × g per 5 minuti a temperatura ambiente (RT). A seconda del problema esaminato, è necessario considerare i vantaggi / svantaggi di tale fase di rotazione, in quanto la centrifugazione può compromettere la forma delle strutture 3D. Evitare di girare a >100 × g.
1. Raccolta e fissazione di modelli di coltura cellulare 3D
NOTA: Fare attenzione a non aspirare le strutture 3D, che saranno solo attaccate liberamente alla parete del tubo.
- Raccolta di modelli di coltura cellulare 3D incorporati in una matrice
NOTA: Questa sezione descrive il recupero di strutture 3D cresciute in gocce di un estratto di membrana basale dal sarcoma murino di Engelbreth-Holm-Swarm (BME), ma può essere adattato ad altre matrici. Vedi la discussione per i punti cruciali riguardanti l'ECM.- Rimuovere il terreno di coltura dai pozzetti senza interrompere la matrice 3D. Precoat l'interno e l'esterno di una punta di pipetta da 1 mL con proteine (di seguito chiamate punta prerivestita da 1 ml ) immergendo l'intera lunghezza della punta in albumina sierica bovina allo 0,1% (BSA) in soluzione salina tamponata fosfato (PBS) (di seguito denominata soluzione PBS-BSA allo 0,1%) e pipettando 1 mL di questa soluzione su e giù due volte.
NOTA: Questo prerivestimento impedirà alle cellule di attaccarsi alla punta e ridurrà al minimo qualsiasi perdita. - Precovestire l'interno di un tubo di centrifuga (15 ml) con proteine (chiamato di seguito tubo di centrifuga prerivestito ) riempiendo ripetutamente con soluzione PBS-BSA allo 0,1% e svuotando il tubo.
NOTA: Ciò impedirà alle cellule di aderire al tubo e ridurrà al minimo qualsiasi perdita. - Utilizzando la punta preverniciata da 1 ml, risospendere con cautela le strutture 3D del pozzetto utilizzando 1 mL di 1x PBS ghiacciato e trasferire delicatamente la sospensione contenente le strutture 3D al tubo da centrifuga preverniciato.
- Aggiungere delicatamente 13 ml di 1x PBS ghiacciato e lasciare che le strutture 3D sedimentino sul ghiaccio per almeno 10 minuti.
NOTA: Se necessario, centrifugare per 5 minuti a 50 × g a 4 °C. Evitare di girare >100 × g, in quanto ciò comprometterebbe la forma delle strutture 3D. - Rimuovere il surnatante. Utilizzando una punta prepatinata da 1 ml, risospendere delicatamente le strutture 3D in 1 mL di 1x PBS ghiacciato. Ripetere i passaggi da 1.1.4 a 1.1.5 per ottenere un pellet omogeneo senza residui di matrice 3D.
NOTA: la rimozione efficiente della matrice è influenzata dal tipo di matrice, dal numero e dalle dimensioni delle strutture 3D e richiede l'ottimizzazione per le diverse condizioni di coltura. Per le strutture 3D coltivate in BME, il recupero dalla rimozione della matrice richiede in genere 45-60 minuti. - Utilizzando una punta preverniciata da 1 ml, trasferire la sospensione 1x PBS da 1 mL contenente le strutture 3D in una provetta da centrifuga preverniciata da 1,5 mL e procedere con la sezione 1.3.
- Rimuovere il terreno di coltura dai pozzetti senza interrompere la matrice 3D. Precoat l'interno e l'esterno di una punta di pipetta da 1 mL con proteine (di seguito chiamate punta prerivestita da 1 ml ) immergendo l'intera lunghezza della punta in albumina sierica bovina allo 0,1% (BSA) in soluzione salina tamponata fosfato (PBS) (di seguito denominata soluzione PBS-BSA allo 0,1%) e pipettando 1 mL di questa soluzione su e giù due volte.
- Raccolta di modelli di coltura cellulare 3D galleggianti
- Utilizzando una punta preverniciata da 1 ml, raccogliere con cura e trasferire le strutture 3D in una provetta da centrifuga preverniciata da 1,5 ml. Lasciare sedimentare le strutture 3D o ruotare per 5 minuti a 50 × g a RT.
- Rimuovere il surnatante. Utilizzando una punta prepatinata da 1 ml, risospendere le strutture 3D in 1 mL di 1x PBS. Procedere con la sezione 1.3.
- Fissazione di modelli di coltura cellulare 3D
- Consentire alle strutture 3D di sedimentare. Rimuovere con attenzione il surnatante; sotto una cappa aspirante, risospendere delicatamente le strutture 3D in 1 mL di formalina utilizzando una punta preverniciata da 1 ml.
NOTA: La formalina contiene formaldeide, che è pericolosa. Manipolare la sostanza chimica in una cappa chimica. Indossare guanti di gomma e occhiali protettivi. - Incubare le strutture 3D per 30 minuti a RT.
NOTA: Una fase di fissazione di 30 minuti con formalina è necessaria per l'immunocolorazione di una vasta gamma di strutture 3D (variabili per dimensioni, forma e origine). Tuttavia, in generale, tempi di fissazione più lunghi (>3 ore) sono più adatti a preservare la fluorescenza delle proteine reporter. - Lasciare sedimentare le strutture 3D o ruotare per 5 minuti a 50 × g a RT. Rimuovere delicatamente la formalina e sostituirla con 1 mL di 1x PBS. Ripetere questa fase di lavaggio in 1x PBS due volte. Conservare i campioni a 4 °C e procedere con la sezione 2 o la sezione 3.
NOTA: Il protocollo può essere messo in pausa qui e le celle possono essere mantenute a 4 °C per la conservazione a lungo termine (>1 anno).
- Consentire alle strutture 3D di sedimentare. Rimuovere con attenzione il surnatante; sotto una cappa aspirante, risospendere delicatamente le strutture 3D in 1 mL di formalina utilizzando una punta preverniciata da 1 ml.
2.3D Colorazione a montaggio completo, imaging e analisi di modelli di coltura cellulare 3D
NOTA: Poiché gli organoidi sono fissati liberamente alla parete del tubo, maneggiarli delicatamente poiché tutti i successivi cambiamenti del reagente possono causare la perdita del campione. Prima di iniziare, assicurarsi della disponibilità dei controlli corretti per la colorazione. I controlli positivi e negativi possono essere cellule, in cui la proteina di interesse è nota per essere sovraespressa o assente, rispettivamente. Incubare campioni senza l'anticorpo primario per determinare se il segnale osservato è dovuto al legame non specifico dell'anticorpo secondario. Poiché alcune cellule tendono a mostrare alti livelli di autofluorescenza, utilizzare controlli privi di anticorpi secondari per determinare se la fluorescenza osservata proviene dall'autofluorescenza di fondo. L'immunolabeling e la visualizzazione fluorescente del reporter possono essere combinati.
- Colorazione 3D dell'intera montatura
- Preparare la soluzione che blocca la permeabilizzazione (PB) integrando 1x PBS con 0,1%-1% di tensioattivo non ionico (vedere la tabella dei materiali), 1% dimetilsolfossido, 1% BSA e 1% siero d'asino (o dall'animale in cui sono stati allevati gli anticorpi secondari).
NOTA: Ottimizzare attentamente la concentrazione del tensioattivo non ionico in base alla localizzazione del bersaglio: membrana (0-0,5%), citoplasma (0,5-1%) e nucleo (1%). Questa soluzione può essere conservata a 4°C per un massimo di 1 mese. BSA di solito funziona bene per la fase di blocco, ma in caso di rumore di fondo elevato, eseguire un test empirico per ottenere i migliori risultati possibili per una data combinazione di anticorpi. - Trasferire gli organoidi dalla provetta da centrifuga da 1,5 mL a una provetta da 0,5 mL utilizzando una punta prerivestita da 1 ml. Lasciare sedimentare gli organoidi, rimuovere delicatamente il PBS 1x e sostituirlo con 0,5 ml di soluzione PB. Incubare gli organoidi con delicata agitazione orizzontale (30-50 rpm) per 1 ora a RT.
- Lasciare sedimentare gli organoidi, rimuovere delicatamente la soluzione PB e lavare due volte in 1 mL PBS-BSA 0,1% per 3 minuti.
NOTA: L'attesa di 3 minuti consente alle strutture di sedimentarsi sul fondo del tubo. - Rimuovere delicatamente il PBS-BSA 0,1% e aggiungere 250 μL di anticorpo primario diluito alla concentrazione appropriata nella soluzione PB:1x PBS (1:10). Per preparare 10 mL di soluzione PB:1x PBS (1:10), diluire 1 mL di soluzione PB in 9 mL di 1x PBS. Incubare per 2-3 giorni agitando delicatamente l'orizzontale (30-50 giri/min) a 4 °C.
NOTA: Un tempo di incubazione anticorpale appropriato è fondamentale per una penetrazione anticorpale adeguata poiché le strutture 3D possono talvolta raggiungere grandi dimensioni. - Lasciare sedimentare gli organoidi e rimuovere delicatamente la soluzione anticorpale primaria. Lavare 5 volte in PBS-BSA 0,1% per 3 minuti per lavaggio e poi 2 volte in 1 mL PBS-BSA 0,1% per 15 minuti per lavaggio con delicata agitazione orizzontale.
- Aggiungere 250 μL di anticorpo secondario diluito a 1:250 in soluzione PB:1x PBS (1:10). Incubare per 24 h a 4°C con delicata agitazione orizzontale (30-50 giri/min). Per questo passaggio, proteggere i campioni dalla luce.
- Aggiungere 250 μL di Hoechst 33342 (soluzione madre da 20 μM) diluito a 1:1000 in soluzione PB:1x PBS (1:10) e incubare per altre 2 ore a 4 °C con delicata agitazione orizzontale (30-50 giri/min).
- Lasciare sedimentare gli organoidi e rimuovere delicatamente la soluzione contenente anticorpi secondari + Hoechst 33342. Lavare gli organoidi 5x in 1 mL di 1x PBS per 3 min per lavaggio e poi 2x in 1 mL di 1x PBS per 15 min per lavaggio con delicata agitazione orizzontale (30-50 rpm).
NOTA: è fondamentale lavare ampiamente i campioni per evitare rumori di fondo o perdita di segnale. - Conservare i campioni in PBS a 4 °C fino all'acquisizione dell'immagine. Procedere con la sezione 2.2.
NOTA: Il protocollo può essere messo in pausa qui e i campioni possono essere conservati a 4 °C per diversi mesi, al riparo dalla luce.
- Preparare la soluzione che blocca la permeabilizzazione (PB) integrando 1x PBS con 0,1%-1% di tensioattivo non ionico (vedere la tabella dei materiali), 1% dimetilsolfossido, 1% BSA e 1% siero d'asino (o dall'animale in cui sono stati allevati gli anticorpi secondari).
- Preparazione del campione per l'imaging confocale
- Utilizzando una punta prepatinata da 1 ml, trasferire con cura gli organoidi in 50 μL di 1x PBS per pozzetto in una micropiastra di polistirene nero da 96 pozzetti. Procedere con il punto 2.2.3 o la sezione 2.3.
NOTA: In questa fase, il campione può essere protetto dalla luce e conservato a 4 °C per molte settimane. - Spurgo
NOTA: La fase di compensazione è facoltativa e può essere utilizzata per immunomarcare organoidi o per rilevare fluorescenza endogena. La pulizia può causare il restringimento della struttura 3D, ma non modifica la morfologia generale ad eccezione degli organoidi sferici monostrato con grandi lumen4. Per questi organoidi cistici, saltare la fase di compensazione ed eseguire l'imaging dei tessuti profondi6.- Preparare una soluzione di eliminazione glicerolo-fruttosio 2,5 M contenente glicerolo al 50% v/v, 11% v/v di acqua distillata e fruttosio al 45% p/v miscelando su un agitatore magnetico almeno per una notte fino a completa solubilizzazione e omogeneità della soluzione. Conservare a 4 °C al buio per un massimo di 1 mese.
- Rimuovere il più possibile 1x PBS senza toccare gli organoidi. Aggiungere 200 μL della soluzione di pulizia utilizzando una punta di pipetta da 1 mL dopo aver rimosso l'estremità e risospendere delicatamente per evitare la formazione di bolle. Incubare a RT per almeno 12 ore e procedere con la sezione 3.
NOTA: poiché la soluzione di compensazione è viscosa, i piccoli volumi sono difficili da gestire. Per facilitare la manipolazione, assicurarsi che la soluzione sia a RT e pipettare lentamente. Per una pulizia ottimale, lasciare sedimentare il campione nella soluzione di pulizia per almeno 24 ore prima dell'imaging. Se le strutture 3D galleggiano al momento dell'acquisizione, eseguire una rotazione opzionale per 10 minuti a <100 × g a RT o concedere più tempo (da uno a diversi giorni) per lasciarle sedimentare. Il protocollo può essere messo in pausa in questa fase prima di procedere all'imaging se è protetto dalla luce e conservato a 4 °C (per settimane) o -20 °C (per mesi).
- Utilizzando una punta prepatinata da 1 ml, trasferire con cura gli organoidi in 50 μL di 1x PBS per pozzetto in una micropiastra di polistirene nero da 96 pozzetti. Procedere con il punto 2.2.3 o la sezione 2.3.
- Acquisizione e analisi delle immagini
NOTA: la tecnologia di sezionamento delle immagini sarà necessaria per l'immagine di strutture 3D.- Utilizzare microscopi confocali e favorire obiettivi di immersione con apertura numerica (NA) più elevata rispetto all'aria. Scegli gli obiettivi di ingrandimento (10x, 20x, 40x) in base alle dimensioni delle strutture 3D, alla ricostruzione dell'immagine (stitching) e alle soluzioni utilizzate per l'analisi.
- Quando si seleziona la modalità di acquisizione, prendere in considerazione la profondità di messa a fuoco dell'obiettivo utilizzato per definire il passo per lo stacking Z; consentono un rendering 3D ottimale.
NOTA: le soluzioni di analisi delle immagini variano e l'analisi dovrà essere adattata al software utilizzato. Ad esempio, questo protocollo di analisi è stato stabilito su un software di analisi ad alto contenuto (vedere la tabella dei materiali e la figura supplementare 1 per i dettagli) e fornisce dati sulla segmentazione degli oggetti, sul calcolo delle proprietà e sulla selezione della popolazione cellulare all'interno di un oggetto ricostruito in 3D.
3. Sezionamento 2D, colorazione, imaging e analisi di modelli di coltura cellulare 3D
NOTA: i modelli di coltura cellulare 3D variano in dimensioni. Procedere con la sezione 3.1 o 3.2 per un'efficace incorporazione della paraffina (Figura 2). Lasciare tempo sufficiente per la sedimentazione della struttura 3D prima di eventuali lavaggi e cambi di reagente. Fare attenzione a non aspirare gli organoidi che galleggiano sul fondo del tubo. Per l'incorporamento di paraffina, fare riferimento alla Figura 2 per istruzioni.
- Incorporamento di paraffina di modelli di coltura cellulare 3D di grandi dimensioni (Ø ≥ 400 μm)
- Il giorno prima dell'incorporazione, preriscaldare due palloni da 150 ml riempiti con paraffina (bagni di paraffina), un piccolo stampo metallico per incorporazione per campione e una pinza fine a 65 °C.
- Utilizzando una punta prepatinata da 1 ml, trasferire con attenzione gli organoidi in 1x PBS in un tubo di vetro a fondo piatto con un tappo di bottiglia rivestito in politetrafluoroetilene. Lasciare sedimentare gli organoidi, rimuovere con cura il PBS 1x e sostituirlo con etanolo al 70%. Incubare per almeno 30 min.
- Lasciare sedimentare gli organoidi e rimuovere con cura l'etanolo al 70%. Sostituirlo con 1 mL di soluzione eosina Y pronta all'uso. Sfiorare il tubo e colorare per almeno 30 minuti. Rimuovere con cura la soluzione di eosina e disidratare gli organoidi in tre lavaggi successivi con 1 ml di etanolo al 100% per ~ 30 minuti ciascuno.
NOTA: L'etanolo, un liquido infiammabile e volatile, provoca gravi irritazioni agli occhi e alle vie respiratorie. Manipolalo in una cappa aspirante e indossa occhiali protettivi per gli occhi. - Rimuovere con cura l'etanolo al 100% e, sotto un cappuccio chimico, eliminare gli organoidi in 3 lavaggi successivi con 1 ml di xilene per ~ 30 minuti ciascuno.
NOTA: Lo xilene è un liquido tossico, infiammabile i cui vapori possono causare irritazione. Manipolarlo in una cappa aspirante. Evitare il contatto diretto con la pelle e indossare guanti di gomma e occhiali protettivi. - Sotto un cappuccio chimico, preparare una cassetta di tessuto microgemello bianco posizionando un pezzo di tampone per biopsia (precedentemente imbevuto di xilene) all'interno di uno degli scomparti della cassetta. Trasferire con cautela le strutture 3D utilizzando una pipetta Pasteur in plastica prerivestita da 2 ml sul tampone per biopsia. Coprirli con un altro tampone per biopsia imbevuto di xilene per evitare che gli organoidi si muovano e chiudere la cassetta.
- Se vengono elaborati più campioni, posizionare la cassetta in un bagno di xilene in attesa di ulteriori elaborazioni. Una volta che tutti i campioni sono stati trasferiti in cassette, posizionare le cassette in un bagno di paraffina preriscaldato per 30 minuti a 65 °C. Trasferire le cassette in un bagno di paraffina preriscaldato fresco durante la notte.
- Dopo l'impregnazione della paraffina, prendere uno stampo di incorporazione preriscaldato e aggiungere la paraffina riscaldata ad esso. Posizionare il tampone per biopsia contenente le strutture 3D nello stampo e agitarlo delicatamente fino a quando tutti gli organoidi cadono sul fondo dello stampo. Posizionare con molta attenzione le strutture 3D al centro dello stampo utilizzando una pinza fine preriscaldata. Procedere con la sezione 3.3.
NOTA: Fare attenzione a non disturbare le strutture 3D con la pinza; Spingere, ma non pizzicarli.
- Incorporamento di paraffina di piccoli (Ø ≤ 400 μm) modelli di coltura cellulare 3D
- Il giorno prima dell'incorporazione, preriscaldare due palloni da 150 ml riempiti con paraffina (bagni di paraffina), un piccolo stampo metallico per incorporazione per campione e una pinza fine a 65 °C.
- Rimuovere con cautela 1x PBS dalla sospensione organoide. Eseguire delicatamente 3 lavaggi in 1 mL di 1x soluzione salina tamponata Tris (TBS). Rimuovere il più possibile 1x TBS senza toccare gli organoidi.
NOTA: Fare attenzione a non aspirare il campione. Se necessario, eseguire una rotazione di 5 minuti a 50 x g a RT. Le tracce rimanenti di fosfato interferiranno con i seguenti passaggi, in particolare impedendo la polimerizzazione del gel. Pertanto, non utilizzare soluzioni PBS durante qualsiasi fase di elaborazione. Per questa fase, è stato utilizzato un kit commerciale, contenente cassette, Reagente #1 (fluido trasparente) e Reagente #2 (fluido colorato), per facilitare la procedura incorporata in paraffina senza potenzialmente perdere piccoli frammenti (vedi Tabella dei materiali). Seguire le istruzioni del kit. Le cassette sono preassemblate con carta di supporto e inserti di cartone già in posizione. - Aggiungere 2 gocce di Reagente #2 nel tubo e mescolare delicatamente toccando il tubo. Aggiungere 2 gocce di Reagente #1 e mescolare nuovamente picchiettando per far solidificare il gel. Usando la pinza fine, rimuovere il gel dal tubo e posizionarlo nel pozzetto della cassetta.
- Sotto la cappa aspirante, disidratare il campione ponendo la cassetta in bagni successivi come segue (utilizzare i palloni da 150 ml e utilizzare etanolo o xilene fresco per ogni bagno): etanolo 70%, 30 min; etanolo 96%, 30 min; etanolo 100%, tre lavaggi, 30 min ciascuno; xilene, tre lavaggi, 30 minuti ciascuno.
- Mettere le cassette in un bagno di paraffina preriscaldato per 30 minuti a 65 °C e trasferirle in un bagno di paraffina fresco preriscaldato durante la notte. Dopo l'impregnazione della paraffina, prendere uno stampo di incorporamento preriscaldato e aggiungere paraffina riscaldata in esso. Aprire la cassetta, rimuovere con cura il gel con una pinza fine e posizionare il gel contenente le strutture 3D al centro dello stampo incorporante. Procedere con la sezione 3.3.
- Passaggi comuni per l'incorporazione di paraffina
- Trasferire delicatamente lo stampo in una zona fredda per lasciare che la paraffina si solidifichi in uno strato sottile, che manterrà le strutture 3D nella posizione appropriata. Aggiungere una cassetta di carta di carta sopra lo stampo e aggiungere paraffina calda per coprire questa cassetta di plastica. Rimuovere lo stampo una volta che è completamente solidificato e procedere con la sezione 3.4.
NOTA: I blocchi di paraffina possono essere conservati a temperatura ambiente per anni.
- Trasferire delicatamente lo stampo in una zona fredda per lasciare che la paraffina si solidifichi in uno strato sottile, che manterrà le strutture 3D nella posizione appropriata. Aggiungere una cassetta di carta di carta sopra lo stampo e aggiungere paraffina calda per coprire questa cassetta di plastica. Rimuovere lo stampo una volta che è completamente solidificato e procedere con la sezione 3.4.

Figura 2: Panoramica della procedura per l'incorporamento di paraffina di modelli di coltura cellulare 3D in vitro grandi e piccoli.
(A) Procedura standard per l'incorporazione di paraffina. Dopo la fissazione e la disidratazione, le strutture 3D vengono colorate con eosina per facilitare la loro visualizzazione (in alto e in basso a sinistra). Le strutture 3D vengono accuratamente posizionate sul tampone bioptico (blu) nella cassetta utilizzando un pipet Pasteur da 2 ml (al centro). Dopo l'impregnazione della paraffina, le strutture 3D vengono delicatamente lasciate cadere nella paraffina liquida usando una pinza e agitate delicatamente nel tampone bioptico. Piccole strutture 3D vengono perse durante questo passaggio in quanto non possono essere rilasciate dal pad (in basso a destra: incorporamento non riuscito). Verranno incorporate solo strutture 3D di grandi dimensioni (in alto a destra: incorporamento riuscito). Le punte di freccia puntano a culture 3D. (B) Alternativa al protocollo standard di incorporazione della paraffina. Dopo aver fissato piccole strutture 3D, viene utilizzato un kit commerciale per mantenere le cellule in un gel e facilitare il loro trasferimento nello stampo dopo l'impregnazione della paraffina (a destra: incorporazione riuscita). Fare clic qui per visualizzare una versione ingrandita di questa figura.
- Sezionamento e colorazione dei blocchi
- Tagliare sezioni da 4 μm utilizzando un microtomo standard ed eseguire tecniche istologiche e immunoistochimiche standard. Procedere con la sezione 3.5.
NOTA: Sono state utilizzate diapositive specifiche (vedi Tabella dei materiali) per una migliore adesione delle sezioni. I vetrini possono essere conservati a temperatura ambiente o a 4 °C per anni.
- Tagliare sezioni da 4 μm utilizzando un microtomo standard ed eseguire tecniche istologiche e immunoistochimiche standard. Procedere con la sezione 3.5.
- Acquisizione e analisi delle immagini
- Eseguire l'imaging utilizzando uno scanner digitale a vetrini o un microscopio verticale e analizzare i dati utilizzando una piattaforma per un'analisi quantitativa digitale rapida che riporta i dati morfologici e di espressione multiplex su base cellula per cellula in intere sezioni della struttura 3D (vedere la Figura supplementare 2 per i dettagli).
NOTA: l'obiettivo 20x viene utilizzato di routine da questo gruppo.
- Eseguire l'imaging utilizzando uno scanner digitale a vetrini o un microscopio verticale e analizzare i dati utilizzando una piattaforma per un'analisi quantitativa digitale rapida che riporta i dati morfologici e di espressione multiplex su base cellula per cellula in intere sezioni della struttura 3D (vedere la Figura supplementare 2 per i dettagli).
Risultati
Questo protocollo fornisce una panoramica delle fasi critiche per la colorazione 2D e 3D a montaggio intero, nonché l'imaging e le analisi quantitative dei modelli di coltura cellulare 3D (Figura 3 e Figura 4). È applicabile a una vasta gamma di modelli di coltura cellulare 3D, dagli sferoidi agli organoidi di diverse specie o tessuti ospiti, e consente l'acquisizione di informazioni accurate e quantitative sull'architettura, l'organizzazione cellulare e le interazioni a livello cellulare e subcellulare (Figura 3 e Figura 4). I laboratori potrebbero aver bisogno di ottimizzare le tecniche istologiche e immunoistochimiche 2D e le concentrazioni di anticorpi in base alle proprie esigenze.
Entrambi i metodi forniscono preziose informazioni biologiche. La colorazione 3D a montaggio intero e la microscopia confocale forniscono informazioni visive sulla composizione cellulare e sulla posizione spaziale con un campo di profondità fino a 200 μm (Figura 3B). Tuttavia, il sezionamento 2D è conveniente per le strutture 3D più grandi per rivelare tratti morfologici cellulari dettagliati nell'intera sezione delle strutture 3D che possono essere altrimenti difficili da osservare in situ a causa della diffusione della luce che compromette la risoluzione in campioni più grandi. Inoltre, entrambe le tecniche possono fornire dati quantitativi. Infatti, la risoluzione ottenuta consente l'applicazione di algoritmi di segmentazione cellulare e subcellulare per la quantificazione del numero di cellule e la rilevazione della presenza di vari marcatori cellulari in diversi sottotipi cellulari (Figura 3F e Figura 4). In sintesi, le tecniche di imaging qui descritte sono riproducibili, semplici e complementari e rappresentano strumenti preziosi per studiare l'eterogeneità cellulare.
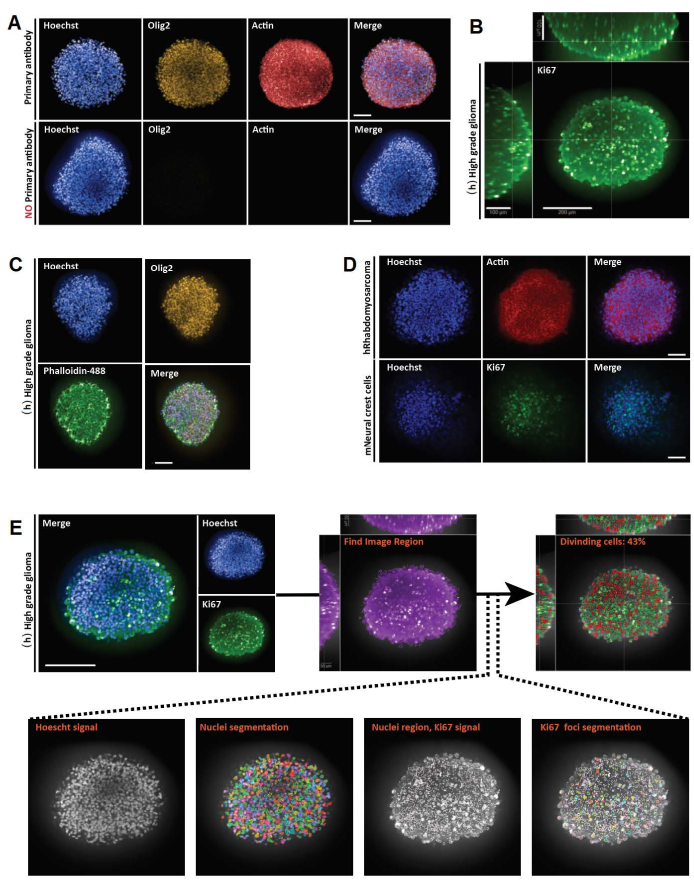
Figura 3: Risultati rappresentativi per l'intera montatura 3D, l'imaging e l'analisi di sezioni ottiche 3D e 2D. (A) Immagini confocali di (h) sferoide glioma umano di alto grado coltivato per una settimana ed etichettato con Hoechst (blu), Olig2 (giallo) e Actina (rosso) (obiettivo 20x acqua). Per tutte le immagini acquisite, le impostazioni del microscopio sono state stabilite utilizzando un controllo positivo (in alto), e quindi il controllo negativo è stato ripreso utilizzando impostazioni identiche per controllare la mancanza di fluorescenza in assenza di anticorpi primari (in basso). (B) Rappresentazione ortogonale 3D a montaggio intero della colorazione Ki67 eseguita in (h) sferoide glioma di alto grado coltivato per una settimana (eliminazione glicerolo-fruttosio; obiettivo acqua 20x, confocale). (C) Immagini confocali di (h) sferoide glioma di alto grado coltivato per una settimana ed etichettato con Hoechst (blu), Olig2 (giallo) e Phalloidine-488 (verde) (eliminazione glicerolo-fruttosio; obiettivo acqua 20x). (D) Immagini confocali di sferoidi umani (h) rabdomiosarcoma (in alto) e di topo (m) (in basso) coltivati per una settimana ed etichettati rispettivamente con Hoechst (blu), actina (rosso) e Ki67 (verde) (eliminazione glicerolo-fruttosio; obiettivo secco 20x). (E) Immagini confocali di (h) sferoide glioma di alto grado coltivato per una settimana ed etichettato con Hoechst (blu) e Ki67 (verde) (eliminazione glicerolo-fruttosio; obiettivo acqua 40x) (in alto a sinistra). Le immagini segmentate sul canale di Hoechst e le regioni nucleari Ki67-positive (+) sul canale verde sono state generate utilizzando un software di analisi ad alto contenuto (vedi Figura supplementare 1 e Tabella dei materiali) (in basso). L'output dato è la percentuale di nuclei Ki67+ per struttura 3D segmentata (in alto a destra). Barra di scala = 100 μm. Fare clic qui per visualizzare una versione ingrandita di questa figura.
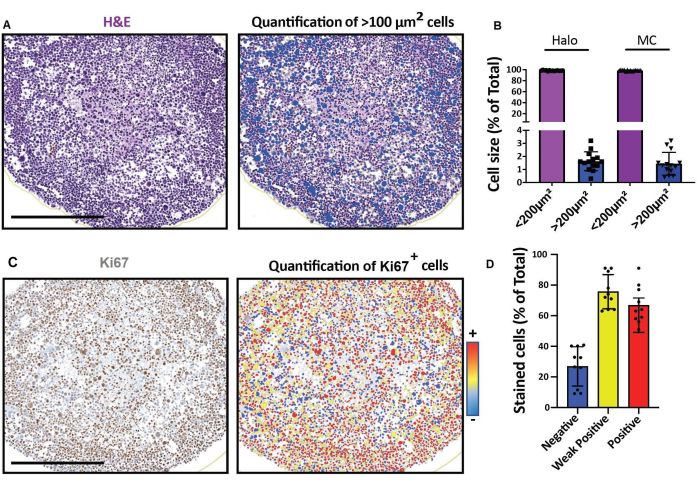
Figura 4: Risultati rappresentativi per l'imaging e l'analisi di sezioni ottiche 2D. (A, D) Immagini di sezione 2D di un modello cellulare 3D (sferoidi rabdomiosarcoma umani coltivati per un mese) ottenute con un scanner digitale e analizzate su una piattaforma per un'analisi quantitativa digitale veloce. (A) Colorazione H&E e rilevamento delle cellule in base alle loro dimensioni. Barra di scala = 500 μm. (B) L'istogramma mostra la percentuale di cellule < 100 μm 2 e > 100 μm2 rilevate utilizzando un software per l'analisi quantitativa digitale veloce (a sinistra: Halo) o il conteggio manuale (a destra: MC). (C) Colorazione di Ki67 e rilevamento delle cellule in funzione dell'intensità del loro segnale 3,3'-diaminobenzidina (DAB). Negativo (blu), debolmente positivo (giallo), positivo (rosso). Barra della scala = 500 μm. (D) L'istogramma mostra la percentuale di cellule Ki67-negative, debolmente positive e positive. Abbreviazioni: H&E = ematossilina ed eosina; MC = conteggio manuale. Fare clic qui per visualizzare una versione ingrandita di questa figura.
Figura supplementare 1: Panoramica delle fasi del software di analisi per immagini. Le analisi si basano sull'associazione di elementi costitutivi. Ogni blocco di costruzione corrisponde a una funzione - segmentazione, calcolo, associazione, definizione dell'output - e offre più algoritmi e selezioni di variabili per abbinare il campione biologico visualizzato. Il software fornisce più protocolli di analisi RMS (Ready Made Solution) che possono essere facilmente utilizzati e modificati. I protocolli di analisi delle immagini integrati possono essere salvati, applicati a diversi set di dati e condivisi tra gli utenti. In breve, il protocollo di analisi implica la segmentazione sequenziale degli oggetti: sferoide, nuclei e, infine, tasche Ki67 (A488). Quindi, l'intensità media delle sacche Ki67 viene calcolata per discriminare ulteriormente gli eventi positivi. Infine, i nuclei che comprendono le tasche positive Ki67 sono selezionati positivamente. Clicca qui per scaricare questo file.
Figura supplementare 2: Panoramica delle fasi procedurali del software di analisi quantitativa. Passo 1. Carica i file utilizzando la scheda Studi . I file verranno aperti nella sezione Azioni immagine . Passo 2. Aprite la scheda Annotazioni , quindi fate clic su Azioni livello per progettare un nuovo livello attorno alla struttura utilizzando lo strumento cerchio della barra degli strumenti. Per le strutture non circolari, è possibile utilizzare lo strumento penna . Passo 3. La barra degli strumenti può essere utilizzata per progettare annotazioni e visualizzare la quantificazione con lo  strumento. Passo 4. Aprire la scheda Analisi e selezionare le condizioni migliori per l'analisi del campione (potrebbero essere necessarie diverse prove qui). Passo 4.1. Utilizzare la sezione Selezione macchia per impostare la condizione di colorazione. In caso di più macchie, queste possono essere aggiunte e rinominate e il colore virtuale può essere modificato. Il rilevamento della localizzazione può essere specificato: colorazione nucleare o citoplasmatica. Passo 4.2. Utilizzare la sezione Rilevamento celle per configurare il rilevamento delle celle. Questa sezione sarà la più importante per l'analisi. La sezione Soglia di contrasto nucleare consentirà il rilevamento di tutti i nuclei. È necessario prestare attenzione nel caso in cui ci siano più dimensioni della popolazione, il software può rilevare diverse celle invece di una grande unica. Le sezioni Dimensione nucleare e Segmentazione nucleare aggressività possono essere utilizzate per quantificare gli intervalli di popolazione delle dimensioni delle cellule. Passo 5. Descrizione su come eseguire l'analisi dei campioni. Seguire i passaggi illustrati nella figura. La sezione Layer di annotazione eseguirà l'impostazione solo in questa diapositiva. La quantificazione può essere visualizzata utilizzando lo
strumento. Passo 4. Aprire la scheda Analisi e selezionare le condizioni migliori per l'analisi del campione (potrebbero essere necessarie diverse prove qui). Passo 4.1. Utilizzare la sezione Selezione macchia per impostare la condizione di colorazione. In caso di più macchie, queste possono essere aggiunte e rinominate e il colore virtuale può essere modificato. Il rilevamento della localizzazione può essere specificato: colorazione nucleare o citoplasmatica. Passo 4.2. Utilizzare la sezione Rilevamento celle per configurare il rilevamento delle celle. Questa sezione sarà la più importante per l'analisi. La sezione Soglia di contrasto nucleare consentirà il rilevamento di tutti i nuclei. È necessario prestare attenzione nel caso in cui ci siano più dimensioni della popolazione, il software può rilevare diverse celle invece di una grande unica. Le sezioni Dimensione nucleare e Segmentazione nucleare aggressività possono essere utilizzate per quantificare gli intervalli di popolazione delle dimensioni delle cellule. Passo 5. Descrizione su come eseguire l'analisi dei campioni. Seguire i passaggi illustrati nella figura. La sezione Layer di annotazione eseguirà l'impostazione solo in questa diapositiva. La quantificazione può essere visualizzata utilizzando lo  strumento. Ripetere i punti 4.1-5 fino a ottenere un'adeguata quantificazione. Passi 6-6.1. Questi passaggi consentono di disegnare una figura utilizzando il software. Passo 7. I grafici di quantificazione ottenuti tramite software possono essere salvati. Passo 8. I dati possono essere esportati. Clicca qui per scaricare questo file.
strumento. Ripetere i punti 4.1-5 fino a ottenere un'adeguata quantificazione. Passi 6-6.1. Questi passaggi consentono di disegnare una figura utilizzando il software. Passo 7. I grafici di quantificazione ottenuti tramite software possono essere salvati. Passo 8. I dati possono essere esportati. Clicca qui per scaricare questo file.
Discussione
La coltura cellulare è uno strumento indispensabile per scoprire i meccanismi biologici fondamentali coinvolti nello sviluppo, nella funzione, nella rigenerazione e nella distruzione dei tessuti e degli organi, e nella malattia. Sebbene la coltura cellulare 2D monostrato abbia predominato, la ricerca recente si è spostata verso colture che generano strutture 3D più riflettenti delle risposte cellulari in vivo, in particolare a causa dell'organizzazione spaziale aggiuntiva e dei contatti cellula-cellula che influenzano l'espressione genica e il comportamento cellulare e potrebbero quindi fornire dati più predittivi7. Tuttavia, rimangono molte sfide, tra cui la necessità di tecniche di colorazione e imaging user-friendly per la visualizzazione microscopica dettagliata e la valutazione di strutture 3D complesse a livello cellulare e subcellulare. In tale contesto, sono stati forniti protocolli dettagliati, robusti e complementari per eseguire la colorazione e l'imaging con risoluzione cellulare e subcellulare di modelli fissi di colture cellulari 3D in vitro di dimensioni comprese tra 100 μm e diversi millimetri.
Questa procedura presenta due diverse strategie per affrontare una grande varietà di dimensioni e tipi di modelli di coltura cellulare 3D in vitro. La scelta dell'uno (analisi 3D a montaggio intero) o dell'altro (analisi di sezionamento 2D) dipenderà dal modello utilizzato e dal problema esaminato. L'analisi 3D a montaggio intero mediante microscopia confocale consente la visualizzazione di cellule con un campo di profondità fino a 200 μm, indipendentemente dalle dimensioni complessive della struttura 3D, mentre il sezionamento 2D è applicabile a campioni di qualsiasi dimensione, ma la visualizzazione rimane dimensionale 2D. Di seguito sono riportati alcuni suggerimenti per la risoluzione dei problemi e considerazioni tecniche.
La perdita di strutture 3D durante il flusso di lavoro è lo svantaggio più comune. Possono rimanere aderenti alle punte e ai tubi, motivo per cui è fondamentale preverniciare punte e tubi con soluzione PBS-BSA allo 0,1%. Inoltre, è fondamentale lasciare che le strutture 3D sedimentino tra i cambiamenti del reagente ed eseguire tutto il pipettaggio con molta attenzione. Come menzionato nella procedura, per tutte le fasi, se la sedimentazione della struttura 3D è troppo lunga, le cellule possono essere fatte girare delicatamente a 50 × g per 5 minuti a RT. A seconda dello scopo dello studio, i vantaggi / svantaggi di tale fase di rotazione dovrebbero essere considerati in quanto la centrifugazione può compromettere la forma delle strutture 3D. Inoltre, bisogna fare attenzione a preservare questa morfologia durante la fase di fissazione perché gli organoidi cistici tendono a collassare. Il fissaggio di strutture di dimensioni inferiori a 400 μm dovrebbe impedire cambiamenti strutturali.
Per una marcatura immunologica ottimale, il recupero degli organoidi dalle loro matrici 3D è un passo cruciale. La matrice 3D può impedire un'adeguata penetrazione degli anticorpi o portare a un'elevata colorazione dello sfondo a causa del legame non specifico alla matrice. La rimozione della ECM può alterare la morfologia dei segmenti esterni degli organoidi (in particolare nel caso di piccole sporgenze cellulari che si estendono da strutture 3D studiate) e ostacolare parzialmente le analisi. Per tali strutture 3D, la matrice può essere mantenuta durante tutta la procedura; Tuttavia, le condizioni di coltura devono essere attentamente adattate per far crescere le cellule in una quantità minima di matrice per evitare una penetrazione insufficiente di soluzioni e anticorpi ed evitare fasi di lavaggio successive volte a ridurre l'eccessivo rumore di fondo 6,8.
La fase di pulizia ottica descritta in questo protocollo nella sezione di colorazione 3D dell'intero montaggio è pertinente per l'imaging di strutture 3D fino a 150-200 μm di profondità anziché 50-80 μm senza clearing. Rispetto ad altre metodologie di compensazione che spesso richiedono diverse settimane e utilizzano agenti di compensazione tossici, in questo protocollo 4,9 è stata utilizzata una fase di compensazione rapida e sicura precedentemente pubblicata. Inoltre, questa fase di pulizia è reversibile e nuovi anticorpi possono essere aggiunti alla colorazione iniziale senza perdita di risoluzione o luminosità4. Tuttavia, a seconda del modello di coltura cellulare 3D studiato, una profondità di 150-200 μm potrebbe non essere sufficiente per visualizzare la struttura 3D in modo informativo e questo protocollo di compensazione può causare cambiamenti nella morfologia generale degli organoidi sferici monostrato con grandi lumen4. Gli utenti dovrebbero progettare attentamente il loro esperimento e, se necessario, ottimizzare i tempi della fase di permeabilizzazione / blocco (per consentire la penetrazione degli anticorpi e della soluzione), la fase di compensazione (per penetrare più in profondità di 200 μm, i campioni dovrebbero essere completamente eliminati) e l'acquisizione delle immagini. Le due tecnologie più diffuse disponibili nelle strutture principali sarebbero il foglio leggero e la microscopia confocale. Gli utenti dovranno scegliere attentamente una tecnologia in base alle dimensioni delle loro strutture 3D e alla loro domanda biologica10. Tuttavia, rispetto alla microscopia confocale, la risoluzione al microscopio a foglio di luce ottenuta per strutture così profonde rimane subottimale per ottenere la risoluzione subcellulare.
Qui, è stato riportato un processo dettagliato e robusto dedicato all'incorporazione di paraffina di singoli campioni. È interessante notare che Gabriel et al. hanno recentemente sviluppato un protocollo che incorpora colture cellulari 3D in paraffina con una maggiore produttività. Hanno usato uno stampo in polidimetilsilossano (PDMS) per confinare 96 strutture 3D in un modello di microarray in un unico blocco, fornendo nuove prospettive per studi su modelli tumorali 3D che comprendono più gruppi, punti temporali, condizioni di trattamento e repliche11. Tuttavia, questo metodo richiede competenze e macchinari approfonditi, in particolare per la fabbricazione del prestampo utilizzato per creare stampi PDMS.
In sintesi, questo articolo descrive due approcci diversi, complementari e adattabili che consentono l'acquisizione di informazioni accurate e quantitative sulla composizione architettonica e cellulare dei modelli cellulari 3D. Entrambi i parametri sono cruciali per lo studio di processi biologici come l'eterogeneità cellulare intratumorale e il suo ruolo nella resistenza ai trattamenti.
Divulgazioni
Gli autori non hanno nulla da rivelare.
Riconoscimenti
Questo lavoro è stato sostenuto dal St Baldrick's Robert J. Arceci Innovation Award #604303.
Materiali
| Name | Company | Catalog Number | Comments |
| Equipment | |||
| Biopsy pad Q Path blue | VWR | 720-2254 | |
| Cassettes macrostar III Blc couv. Char. x1500 | VWR | 720-2233 | |
| Cassette microtwin white | VWR | 720-2183 | |
| Chemical hood | Erlab | FI82 5585-06 | |
| Filter tips 1000 µL | Star lab Tip-One | S1122-1730 | |
| Fine forceps | Pyramid innovation | R35002-E | |
| Flat-bottom glass tubes with PTFE lined 2 mL | Fisher Scientific | 11784259 | Excellent for environmental samples, pharmaceuticals and diagnostic reagents. PTFE is designed for the ultimate in product safety. PTFE provides totally inert inner seal and surface facing the sample or product. |
| Glass bottom dish plate 35 mm | Ibedi | 2018003 | |
| Horizontal agitation | N-BIOTEK | NB-205 | |
| Incubator prewarmed to 65 °C | Memmert Incubator | LAB129 | |
| Inox molds 15x15 | VWR | 720-1918 | |
| Microscope Slides Matsunami TOMO-11/90 | Roche diagnostics | 8082286001 | these slides are used for a better adhesion of sections |
| Microtome | Microm Microtech France | HM340E | |
| Panoramic scan II | 3dhistech | 2397612 | |
| Paraffin embedding equipment | Leica | EG1150C | |
| Plastic pipette Pasteur 2 mL | VWR | 612-1681 | |
| Q Path flacon 150mL cape blanc x250 | VWR | 216-1308 | Good for environmental samples, pharmaceuticals and diagnostic reagents. Polypropylene (PP) are rigid, solid, provide excellent stress crack and impact resistance and have a good oil and alcohol barrier and chemical resistance. PE-lined cap is stress crack resistant and offers excellent sealing characteristics. |
| Set of micropipettors (p200, p1000) | Thermo Scientific | 11877351 (20-200) 11887351(p1000) | |
| OPERA PHENIX | PerkinElmer | HH14000000 | |
| SP5 inverted confocal microscope | Leica | LSM780 | |
| Tissue cassette | VWR | 720-0228 | |
| Zeiss Axiomager microscope | Leica | SIP 60549 | |
| Reagent | |||
| Bovine Serum Albumin (BSA) | Sigma-Aldrich | A7030-100G | |
| Cytoblock (kit) | Thermofisher Scientific | 10066588 | |
| Dimethyl sulfoxide (DMSO) | Sigma-Aldrich | 57648266 | CAUTION: toxic and flammable. Vapors may cause irritation. Manipulate in a fume hood. Avoid direct contact with skin. Wear rubber gloves, protective eye goggles. |
| Eosin aqueous 1% | Sigma-Aldrich | HT110316 | |
| Ethanol 96% | VWR | 83804.360 | CAUTION: Causes severe eye irritation. Flammable liquid and vapor. Causes respiratory tract irritation. Manipulate in a fume hood. Wear protective eye goggles. |
| Ethanol 100% | VWR | 20821.365 | CAUTION: Causes severe eye irritation. Flammable liquid and vapor. Causes respiratory tract irritation. Manipulate in a fume hood. Wear protective eye goggles. |
| Formalin 4% | Microm Microtech France | F/40877-36 | CAUTION: Formalin contains formaldehyde which is hazardous. Manipulate in a fume hood. Avoid direct contact with skin. Wear rubber gloves and protective eye goggles. |
| Fructose | Sigma-Aldrich | F0127 | |
| Gill hematoxylin type II | Microm Microtech France | F/CP813 | |
| Glycerol | Sigma-Aldrich | G5516 | 500 mL |
| Hoechst 33342 | Life Technologies | H3570 | CAUTION: Suspected of causing genetic defects. Avoid direct contact with skin. Wear rubber gloves and protective eye goggles. |
| Normal donkey serum | Sigma-Aldrich | D9663 | 10 mL |
| Paraffin Wax tek III | Sakura | 4511 | |
| Phosphate Buffer Saline (PBS) 1 X | Gibco | 14190-094 | |
| Tris-Buffered Saline (TBS) 10X | Microm Microtech France | F/00801 | 100 mL |
| Triton X-100 | Sigma-Aldrich | T8532 | CAUTION: Triton X100 is hazardous. Avoid contact with skin and eyes. |
| Xylene | Sigma-Aldrich | 534056 | CAUTION: Xylene is toxic and flammable. Vapors may cause irritation. Manipulate in a fume hood. Avoid direct contact with skin. Wear rubber gloves, protective eye goggles. |
| Solutions | |||
| Clearing solution | Glycerol-Fructose clearing solution is 60% (vol/w) glycerol and 2.5 M fructose. To prepare 10 mL of this solution, mix 6 mL of glycerol and 4.5 g of fructose. Complete to 10 mL with dH2O. Use a magnetic stirrer overnight. Refractive index = 1.4688 at room temperature (RT: 19–23 °C). Store at 4 °C in dark for up to 1 month. | ||
| PBS-BSA 0,1% solution | To prepare 0,1% (vol/wt) PBS-BSA 0,1% solution, dissolve 500 mg of BSA in 50 mL of PBS-1X (store at 4°C for up to 2 weeks). And dilute 1mL of this solution into 9mL of PBS-1X. This solution can be used to precoat the tip and centrifugation tube. | ||
| Permeabilisation-blocking solution (PB solution) | The PBSDT blocking solution is PBS-1X supplemented with 0.1% – 1% Tritonx-100 (depending on the protein localization membrane/nucleus), 1% DMSO, 1% BSA and 1% donkey serum (or from the animal in which the secondary antibodies were raised). This solution can be stored at 4°C for up to 1 month. | ||
| PB:PBS-1X (1:10) solution | PB:PBS-1X (1:10) solution is a 10 time diluted PB solution. To prepare 10 mL of this solution dilute 1 mL of PB solution in 9 mL of PBS-1X. | ||
| Software | |||
| Halo software | Indicalabs | NM 87114 | |
| Harmony software | PerkinElmer | HH17000010 |
Riferimenti
- Ryu, N. E., Lee, S. H., Park, H. Spheroid culture system methods and applications for mesenchymal stem cells. Cells. 8 (12), 1-13 (2019).
- Bartfeld, S., Clevers, H. Stem cell-derived organoids and their application for medical research and patient treatment. Journal of Molecular Medicine. 95 (7), 729-738 (2017).
- Cui, X., Hartanto, Y., Zhang, H. Advances in multicellular spheroids formation. Journal of the Royal Society, Interface. 14 (127), (2017).
- Dekkers, J. F., et al. High-resolution 3D imaging of fixed and cleared organoids. Nature Protocols. 14, 1756-1771 (2019).
- Broutier, L., et al. Culture and establishment of self-renewing human and mouse adult liver and pancreas 3D organoids and their genetic manipulation. Nature Protocols. 11 (9), 1724-1743 (2016).
- Rezanejad, H., Lock, J. H., Sullivan, B. A., Bonner-Weir, S. Generation of pancreatic ductal organoids and whole-mount immunostaining of intact organoids. Current Protocols in Cell Biology. 83 (1), 82 (2019).
- Edmondson, R., Broglie, J. J., Adcock, A. F., Yang, L. Three-dimensional cell culture systems and their applications in drug discovery and cell-based biosensors. Assay and Drug Development Technologies. 12 (4), 207-218 (2014).
- McCray, T., Richards, Z., Marsili, J., Prins, G. S., Nonn, L. Handling and assessment of human primary prostate organoid culture. Journal of Visualized Experiments: JoVE. (143), e59051 (2019).
- Ueda, H. R., et al. Tissue clearing and its applications in neuroscience. Nature Reviews: Neuroscience. 21 (2), 61-79 (2020).
- Lazzari, G., et al. Light sheet fluorescence microscopy versus confocal microscopy: in quest of a suitable tool to assess drug and nanomedicine penetration into multicellular tumor spheroids. European Journal of Pharmaceutics and Biopharmaceutics. 142, 195-203 (2019).
- Gabriel, J., Brennan, D., Elisseeff, J. H., Beachley, V. Microarray embedding/sectioning for parallel analysis of 3D cell spheroids. Scientific Reports. 9, 16287 (2019).
Ristampe e Autorizzazioni
Richiedi autorizzazione per utilizzare il testo o le figure di questo articolo JoVE
Richiedi AutorizzazioneThis article has been published
Video Coming Soon