このコンテンツを視聴するには、JoVE 購読が必要です。 サインイン又は無料トライアルを申し込む。
Method Article
X -線結晶構造のためのタンパク質の結晶化
要約
分子の3次元構造がどのように分子の機能のユニークな理解を提供します。ニア原子分解能で構造決定のための主要な方法は、X線結晶です。ここで、我々はX線結晶構造決定に適している任意の高分子の三次元結晶を得るための現在の方法を示しています。
要約
彼らがどのように機能するか推測する生体高分子の三次元構造を使用すると、現代生物学の最も重要な分野の一つである。原子分解能構造の可用性は、タンパク質の機能の深さとユニークな理解を提供し、そして生きている細胞の内部の仕組みを解明するのに役立ちます。これまでに、蛋白質構造データバンク(RCSB - PDB)のエントリの86%がX線結晶学を用いて決定された巨大分子の構造です。
結晶解析に適した結晶を得るために、高分子(例えばタンパク質、核酸、タンパク質 - タンパク質複合体やタンパク質 - 核酸複合体)が均一にまで精製する必要がある、またはできるだけ近い均一になるまで。準備の均一性は高解像度(;マクファーソン、1999 Bergfors、1999)に回折する結晶を得るための重要な要素です。
結晶は、過飽和に高分子をもたらす必要があります。サンプルは、したがって、高分子の凝集または沈澱(通常は20〜50 mg / mL)を起こすことなく可能な限り高い濃度に濃縮する必要があります。沈殿剤にサンプルを導入することは、溶液から成長する大型の三次元結晶をもたらすことができます溶液中でのタンパク質結晶の核形成を促進することができます。蒸気拡散とバッチの結晶化:結晶を取得する2つの主なテクニックがあります。蒸気拡散、沈殿剤と蛋白溶液の混合物を含むドロップで純粋な沈殿剤とチャンバー内に封入されている。ドロップと沈殿剤の浸透圧は(図1A)等しくなるまで水蒸気は、ドロップから拡散する。均衡が達成されるまで滴の脱水は、理想的に相図の結晶核のゾーンで、タンパク質と沈殿剤の両方の遅い濃度を引き起こします。バッチ法は、沈殿剤の適切な量(図1B)とを混合タンパク質によって核のゾーンに直接タンパク質をもたらすに依存しています。このメソッドは、通常ドロップのうち水の拡散を防ぐために、パラフィン/鉱物油の混合物の下に実行されます。
ここでは、石油のもとでのバッチ結晶化に加えて、ドロップやシッティングドロップを吊り下げ、蒸気の拡散のための実験装置の二種類のデモンストレーションを行います。
プロトコル
材料:
- タンパク質試料 - リゾチーム(50 mg / ml)を
- ドロップ24ウェルトレーハンギング
- シッティングドロップ24ウェルトレー
- Microbatchの結晶化96ウェルトレー
- 結晶化ソリューション(どちらか市販または自家製)
- シリコングリース
- ルアーロックせずに5 mLの注射器
- シリコーンカバースライド
- 光学シールテープ
- パラフィン
- 低保持のヒントと0.1〜2μLマイクロピペット
- ピンセット
- プロのワイプ
1。ハンギング/ドロッププロシージャを座って。
- 初期画面のために一つの試験条件のスパース行列不完全な階乗のメソッドを利用する市販のスクリーンを使用することができます。スパース行列の不完全要因加算法は、電圧値と高分子のための既知の結晶化条件(カーターとカーター、1979; Cudney ら、1994;。Jancarikと金、1991;。Jancarikら 、1991)から選択される。結晶化条件がすでにわかっている場合は、狭い画面はオリジナルのヒットの周りに沈殿剤濃度とpHを変化させること、組み立てることができます。最も一般的に成功した沈殿は硫安が続くポリエチレングリコール(PEG)、である。結晶化に使用したすべての記録された高分子の沈殿剤の約60%をこれら2つの沈殿剤は、アカウント(。ギリランドら 、1994;キンバーら、2003;。。らページ、2003)。
- あなたの結晶化溶液を調製。通常、これらの条件は、沈殿剤、ある種の塩とpHを設定するには、バッファから結合されます。場合によっては添加剤は、目的のタンパク質のために特別に追加される可能性があります。すべてのストック溶液を0.22μmのフィルターを使用してフィルタリングする必要があります。ストック溶液の一部は、(50%PEGは、グリセリン、PEG - MME、など)非常に粘性のあるかもしれない。
- 0.6 1.5 mLの、0.8、1.0、1.2、1.4、0.1 M NaOAc pHは4.0、4.3、4.6と4.9の1.6 M NaClをそれぞれを(24ソリューションの合計)の準備
- あなたのタンパク質サンプルを解凍し、氷上に置きます。目的のタンパク質は、(もしそうなら、4℃で保存)/解凍サイクルを凍結する敏感でない限り、タンパク質の株式は、-80℃で保存してください。典型的なタンパク質の濃度は高濃度が通常より良い結果を与えることで、50〜50 mg / mLの範囲内である。
- 000xg / 4、サンプル15min/18をスピン° C - いくつかのタンパク質は、解凍工程の後に部分的に集約し、沈殿する傾向がある。これらの凝集体は、したがって、完全なスピンがダウンして、あなたのサンプル中の唯一の可溶性分子を持って確保するタンパク質分子の残りの非晶析出を促進することができます。トレイを設定するまで、氷上に蛋白質を保つ。
- UV分光光度計を用いて280nmでの吸光度からタンパク質濃度を決定する。タンパク質濃度は、吸光度測定値と蛋白質の吸光係数から計算することができます。 :吸光係数はExpasyウェブサイト上のProtParamツールを使用して計算することができますhttp://ca.expasy.org/tools/protparam.html
- 24ウェルハング/トレイの設定のスキーム(図2B)によると沈殿剤溶液500μLをドロップトレイに座っての井戸を埋める。ウェルの縁の周りにシリコーングリースのリングを作成します。リングは良くはシールされているように空気の圧力上昇を防ぐための小さなギャップを持つ必要があります。座っ滴で、よくは(図2A)テープで封印されているため、グリースの必要はありません。
- カバースライドを取り、凝縮した空気スプレーで掃除、またはワイププロ - ダストが結晶化ドロップの解釈を容易にし、汚染を除去する避ける。ドロップ、カバースライドを座ってするために使用されていません。その代わり、よくは、タンパク質と沈殿剤とを混合されている棚を含んでいます。
- カバースライドでタンパク質試料とリザーバー溶液の等量を置く。タンパク質と沈殿剤の異なるボリュームを最適化プロセスの一部として試されることがあります。希薄なタンパク質サンプルのために望ましいことができ、結晶の核生成や成長を遅らせるために平衡後降下、でタンパク質の正味濃度の沈殿剤の結果よりも多くのタンパク質を使用する。ドロップソリューション内の他の不揮発性の物質も、同じ要因によって濃縮される。カバースライド上にタンパク質とリザーバー溶液をピペッティングするときに、気泡の形成を避けるために非常に注意してください。これは多くの場合空気がピペットから吹き出される場合に発生します。
- カバーのスライドの中央に0.02 M NaOAcのpHを4.9に50 mg / mLのリゾチームのロード2μLをし、そのためにリザーバー溶液2μLを加える。懸滴。
- ドロップ座り:荷重棚の中心から50 mg / mLのリゾチーム溶液2μLをし、そのためにリザーバー溶液2μLを加える。
- 静かにカバーをスライドして反転し、井戸の上にグリースをリングの上に下に置く。 prにグリースにやさしく空気が溝に沿って井戸から脱出できることを押し下げイベントの空気の圧力の井戸で蓄積し、よく密封してください。ウェル内の空気の圧力上昇は、よくシール性を損なう、カバースライドを上げることができます。シッティングドロップ法では、光学的に透明な透明テープは井戸をカバーするために使用されます。したがって、このメソッドはシールですべての行/列に行われます。
- トレイが完了するまで次のウェルに移ります。
- セットアップ時にトレイを確認してください - ダスト粒子や他の汚染を排除するタンパク質結晶の偽陽性の識別を減らすことができます。
- あなたの実験を文書化するスコアリングシートを使用してください。
- 一般的に4 ° Cと室温との間で、目的の培養温度でトレイを置きます。 20 ° Cは、最も一般的に成功しています。静かにトレイを処理し、どんな揺れを避けるために注意してください。インキュベーターの扉を開いたり閉じたりしながら注意してください。移転やインキュベーション中に、振動や温度変化は、結晶成長を防止または否定的に結晶品質に影響を与える可能性があります。物質を吸収するショック(例えばパッキンの泡)が結晶トレイの下に詰め物として使用することができます。
- 結晶のトレイは、次の日を確認して、数日ごとに、常に注意してトレイを処理。トレイ特定のスコアリングシート内の任意の調査結果とドロップの形態を文書化します。結晶が時折はるかに早く(ほとんどすぐに)以降(最大で数ヶ月に!)表示されますが、結晶は、通常、表示に2-5日かかる。結晶化条件が特定されており、結晶の成長速度がわかれば、それは結晶が出現し、彼らの最終的な大きさの少なくとも半分に成長するまでトレイをそっとしておくのが最善の方法です。トレーが邪魔されないままにしておくと、それ故に大きな結晶の小さい数字を生成、結晶核生成のイベントの数を減らすことができます。
2。手順をMicrobatch:
- タンパク質試料と沈殿剤の調製は、前述の通りである。
- エアスプレー、ほこりや他の大きな粒子を避けるために、新しいmicrobatchトレイ。
- microbatchトレイで、高3ミリメートル(井戸をカバーするだけの十分な)にパラフィン油をいっぱいに。油のアクセスを削除します。
- よく充填済み油にタンパク質溶液(50 mg / mLのリゾチーム)の1μLをロードする - 井戸の底(図2A)に直接ソリューションをピペッティングすることを確認してください。
- ドロップは、蛋白質の液滴で十分とヒューズの底に沈むことを確認 - よくトレイのセットアップ方式(図2B)によると中に沈殿剤溶液の1μLをロードする。
- トレイが完了するまで次のウェルに移動します。
- 吊り下げ/シッティングドロップの手順のステップ11から14に従ってください。
3。代表的な結果:
結晶化は通常、X線結晶構造解析のボトルネックと呼ばれています。沈殿条件のスパース行列不完全な階乗の画面は、通常、それらの大きな単結晶の中で、タンパク質の凝集と沈殿の多くの異なったタイプを生成します。タンパク質や沈殿剤濃度が高すぎる場合、1つは、明確な形と大きさ(アモルファス降水量)と茶色の問題を見ることができます。解決策が不飽和のされている場合、ドロップはしばしば降水量のあらゆる種類の完全に明確で欠いているだろう。図3は、降水現象と結晶(デッサウら 、2006)のためのいくつかの例を示しています。より詳細な解釈を持つより多くの降水現象は、次のURLで参照することができますhttp://xray.bmc.uu.se/terese/tutorials.html 。
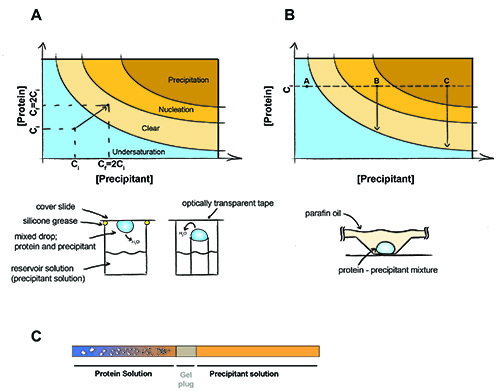
図1。タンパク質の結晶化の原理。
タンパク質の結晶化の原理。蒸気拡散実験()で沈殿剤と蛋白質の等容量はドロップに存在しています。水が出て拡散すると平衡はドロップとリザーバー溶液との間で達成されるまで、沈殿剤やタンパク質濃度の両方が倍増されます。バッチ晶析では(B)沈殿剤と蛋白質の濃度は、実験中に変化しません。指して - タンパク質は不飽和のまま、結晶が、ポイントBを形成することができます - タンパク質の核生成が起こり、結晶が形成し始めたと溶液中のタンパク質の濃度は飽和状態に低下します。ポイントC - タンパク質は沈殿するが、結晶は依然として大きくなることがあります。キャピラリー差し込ま-ゲルを使用して(C)透析結晶化。塩は、ゲルのプラグを介して(および/または沈殿剤拡散タンパク質試料に)タンパク質試料から拡散するように結晶が現れます。
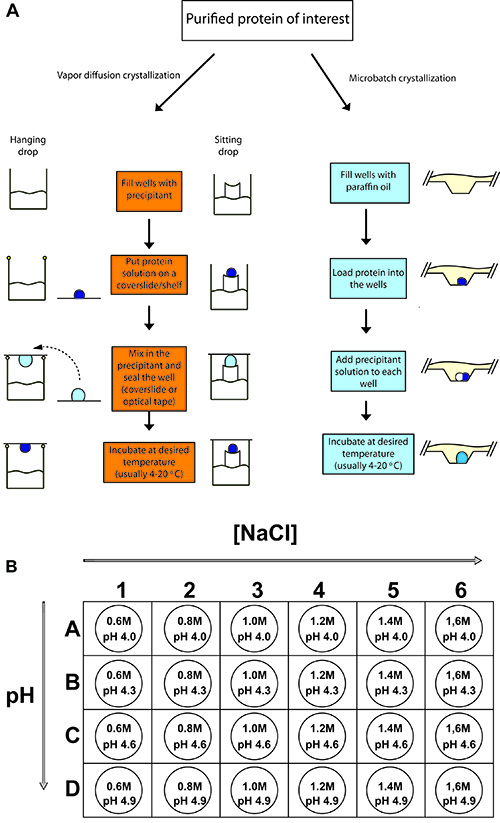
図2。典型的なタンパク質の結晶化実験の概要。
ハンギングドロップ蒸気拡散、シッティングドロップ蒸気拡散、およびmicrobatchタンパク質の結晶化のための(A)の手順。それぞれのケースでは、濃縮されたタンパク質試料の少量をprecipitanの等しいまたはより小さいボリュームと混合され、T溶液と平衡化させた。 (B)鶏リゾチーム、24ウェルトレーの結晶は塩化ナトリウムの様々な濃度(沈殿剤)とし、種々のpHにおいて緩衝剤として0.1 M酢酸ナトリウム( - 4.9 4.0)と設定されています。

図3。タンパク質結晶化実験の典型的な成果。
(A)アモルファス沈殿。タンパク質や沈殿剤(または両方)を高濃度にある時。 (B)相分離。高濃度で一定の沈殿剤と混合したときにタンパク質や洗剤は、別のフェーズに分けることができます。 (C)AtCSN7のロッド状のタンパク質の結晶は、ポリエチレングリコール8000及び酢酸マグネシウム(デッサウら 、2006)で得られた。 (D)リゾチームの結晶は、1.0 M NaClと酢酸ナトリウムのpHを4.9に得られた。 (E)飽和滴の下で、通常はクリアのまま。モシェデッサウによる写真撮影。
結果
Crystallization is usually referred to as the bottleneck of X-ray crystallography. A sparse matrix incomplete factorial screen of precipitating conditions typically produces many different types of protein aggregation and precipitation, among them large single crystals. If the protein or precipitant concentrations are too high one can see brown matter with no distinct shape and size (amorphous precipitation). When the solution is undersaturated, the drop will often be completely clear and devoid of any kind of precipitat...
ディスカッション
この記事では、タンパク質の結晶化のための一般的な現在のプロトコルを説明し、実証する。マルチステップの手順でそれ以来、いくつかの考慮事項は次のことに注意一つのニーズがあります。非常に少量(0.5-2μL)で作業する場合、蒸発による減少の乾燥は大きな懸念事項です。したがって、それは十分に制御された環境(低空気の流れと、高湿度と厳しい温度制御)で動作するように、?...
開示事項
謝辞
この作品は、YMにバロウズウェルカムの研究者賞で、エール大学からMDへのブラウン - コックスポストドクトラルフェローシップでサポートされていました。
資料
| Name | Company | Catalog Number | Comments |
| Lysozyme | Sigma-aldrich | L6876-1G | |
| 24 well VDX Plate | Hampton research | HR3-142 | |
| 24 well Cryschem Plate | Hampton research | HR3-158 | |
| Dow Corning Vacuum Grease | Hampton research | HR3-510 | |
| Siliconized glass circle coverslides | Hampton research | HR3-231 | |
| 100% paraffin oil | Hampton research | HR3-411 | |
| 1.88 inch wide Crystal Clear Sealing Tape | Hampton research | HR3-511 | |
| 96 Well Imp@ct Plate (Microbatch plate) | Hampton research | HR3-098 |
参考文献
- Bergfors, T. M. . Protein crystallization : techniques, strategies, and tips : a laboratory manual. , (1999).
- Carter, C. W., Carter, C. W. Protein crystallization using incomplete factorial experiments. J Biol Chem. 254, 12219-12223 (1979).
- Cudney, R., Patel, S., Weisgraber, K., Newhouse, Y., McPherson, A. Screening and optimization strategies for macromolecular crystal growth. Acta Crystallogr D Biol Crystallogr. 50, 414-423 (1994).
- Dessau, M., Chamovitz, D. A., Hirsch, J. A. Expression, purification and crystallization of a PCI domain from the COP9 signalosome subunit 7 (CSN7). Acta Crystallogr Sect F Struct Biol Cryst Commun. 62, 1138-1140 (2006).
- Gilliland, G. L., Tung, M., Blakeslee, D. M., Ladner, J. E. Biological Macromolecule Crystallization Database, Version 3.0: new features, data and the NASA archive for protein crystal growth data. Acta Crystallogr D Biol Crystallogr. 50, 408-413 (1994).
- Jancarik, J., Kim, S. H. Sparse matrix sampling: a screening method for crystallization of proteins. J Appl Cryst. 23, 409-411 (1991).
- Jancarik, J., Scott, W. G., Milligan, D. L., Koshland, D. E., Kim, S. H. Crystallization and preliminary X-ray diffraction study of the ligand-binding domain of the bacterial chemotaxis-mediating aspartate receptor of Salmonella typhimurium. J Mol Biol. 221, 31-34 (1991).
- Kimber, M. S., Vallee, F., Houston, S., Necakov, A., Skarina, T., Evdokimova, E., Beasley, S., Christendat, D., Savchenko, A., Arrowsmith, C. H. Data mining crystallization databases: knowledge-based approaches to optimize protein crystal screens. Proteins. 51, 562-568 (2003).
- Lorber, B., Sauter, C., Theobald-Dietrich, A., Moreno, A., Schellenberger, P., Robert, M. C., Capelle, B., Sanglier, S., Potier, N., Giege, R. Crystal growth of proteins, nucleic acids, and viruses in gels. Prog Biophys Mol Biol. 101, 13-25 (2009).
- McPherson, A. . Crystallization of biological macromolecules. , (1999).
- McPherson, A. Introduction to protein crystallization. Methods. 34, 254-265 (2004).
- Page, R., Grzechnik, S. K., Canaves, J. M., Spraggon, G., Kreusch, A., Kuhn, P., Stevens, R. C., Lesley, S. A. Shotgun crystallization strategy for structural genomics: an optimized two-tiered crystallization screen against the Thermotoga maritima proteome. Acta Crystallogr D Biol Crystallogr. 59, 1028-1037 (2003).
転載および許可
このJoVE論文のテキスト又は図を再利用するための許可を申請します
許可を申請さらに記事を探す
This article has been published
Video Coming Soon
Copyright © 2023 MyJoVE Corporation. All rights reserved