このコンテンツを視聴するには、JoVE 購読が必要です。 サインイン又は無料トライアルを申し込む。
Method Article
ホット生物触媒:等温滴定熱量測定酵素反応を特徴づけるために
要約
熱流が化学反応で放出又は吸収等温滴定熱量測定する。この方法は、酵素触媒作用を定量化するために使用することができる。本論文では、楽器の設定、実験の実行、およびデータ分析のためのプロトコルは、一般的に説明されており、ナタマメウレアーゼによる酵素尿素加水分解の評価に適用。
要約
等温滴定熱量測定(ITC)は、ほぼすべての化学プロセスを特徴付けるために固有のプローブとして使用して、化学反応の間に放出または吸収される熱を測定する十分に記載された技術である。今日では、この技術は、広く生体分子の結合平衡の熱力学的パラメータを決定するために適用される。また、ITCは直接アプリケーションがまだunderexploitedであっても、酵素反応の動力学及び熱力学的パラメータ( の k cat、K M、ΔH)を測定できることが実証されている。熱変化は、自発的に酵素触媒反応中に発生するように、ITCは、分析中のシステムの任意の改変または標識を必要とせず、溶液中で行うことができる。さらに、この方法は、材料の少量を必要とする。これらのプロパティは、ITCのようないくつかのアプリケーションでは、例えば、薬剤の発見に酵素反応速度を研究するために、非常に貴重な、強力でユニークなツールとなっています。
、Canavalia ensiformis(ジャック豆)ウレアーゼによる尿素の酵素加水分解のK 猫とK Mを決定するために適用される。反応の固有のモルエンタルピー(ΔHの整数 )の計算が行われる。このようにして得られた値は、方法の信頼性を実証する、文献に報告され、以前のデータと一致している。
概要
生化学反応の定量は生命の基礎で生物学的プロセスへの洞察を提供しています。熱量測定を定量的に、溶液中のほぼすべての化学反応を特徴づけるラベルフリーの方法論を提供しています。この技術は、経時的に放出または吸収される熱を測定し、したがって、分子( 即ち、結合熱力学)を反応させるの量を定量化するため、ならびに反応速度( すなわち、反応速度)を測定するためのユニバーサル検出システムと非常に便利な方法である。特に、等温滴定熱量測定(ITC)は、タンパク質-リガンド、タンパク質-タンパク質、タンパク質-金属イオンおよびタンパク質-DNA相互作用1-6伴う、生体分子の熱力学平衡を特徴付けるための選択の方法として採用されている。本出願の電位がまだあるがまた、動力学的情報を提供するITCの能力は、その酵素触媒を測定するための非常に強力なシステムとなる7-9を過小評価していた。
ミカエリス定数(K M)および触媒速度定数(k cat)を :は、2つの動態パラメータに応じて、反応速度及び基質濃度との関係を提供するので、ミカエリス-メンテン式10は 、酵素反応の定量的記述である。 の k cat / K M比が酵素の触媒効率と呼ばれる。実際には、特定の反応のために、K、MとKの猫の決定は、触媒作用の完全な説明を提供しています。
典型的な酵素反応( 図1)において、基板(S)は、その後、遷移状態中に活性化される酵素-基質(ES)複合体を形成する酵素(E)、(ES *)と相互作用する。後者は、最終的には解離し、酵素生成物(EP)錯体に変換される。これらのステップsが、以下の反応により記載されている。
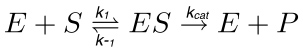 (1)
(1)
k 1は ES複合体の形成の速度定数である場合の k cat は触媒速度定数またはターンオーバー数であるが、kが-1、ES複合体の解離の速度定数である。
ミカエリス·メンテン式10によれば、反応速度は次のように計算できます。
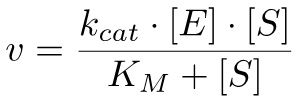 (2)
(2)
ここで、K、M =(K -1 + K 猫 )/ K 1とK 猫 = V maxは / [E]、V maxは 、すべての酵素が基質に結合されたときに到達した最大速度であることと。
等温滴定熱量計は、尿素の酵素的加水分解を特徴づけるためにこの研究で使用される機器である。この器具は、2つの造語状細胞( 図1)を含有する断熱シールドで形成されている。これらは、狭いアクセスチューブと外部に接続されている。基準セルは、一般に水または分析のために使用される溶媒で満たされている間に試料セル( 約 1.4ミリリットル)を、酵素溶液でロードされる。通常、基質溶液の約0.3 mlを含む長い針及び取り付けられた攪拌パドルを有する回転注射器は、試料セルに取り付けられている。熱電素子」とは、細胞フィードバックネットワーク」を用いて、試料と参照セルとの間の温度差を測定し、熱を加算または減算することによってゼロにあるこの差を維持する。実験中、基板は、一定の選択された温度で酵素溶液に注入される。ときはアンzymatic反応は、放出又は吸収される熱の量は、生成物分子に変換される基質分子の数に比例する、起こる。また、熱流の速度を直接反応速度に関係する。最初のベースラインからの熱痕跡のずれ( 図1)として現れる測定データは、試料セルで発生する熱流に比例した試料セルに機器によって供給される熱出力(μcal/秒)を表す時間をかけて。
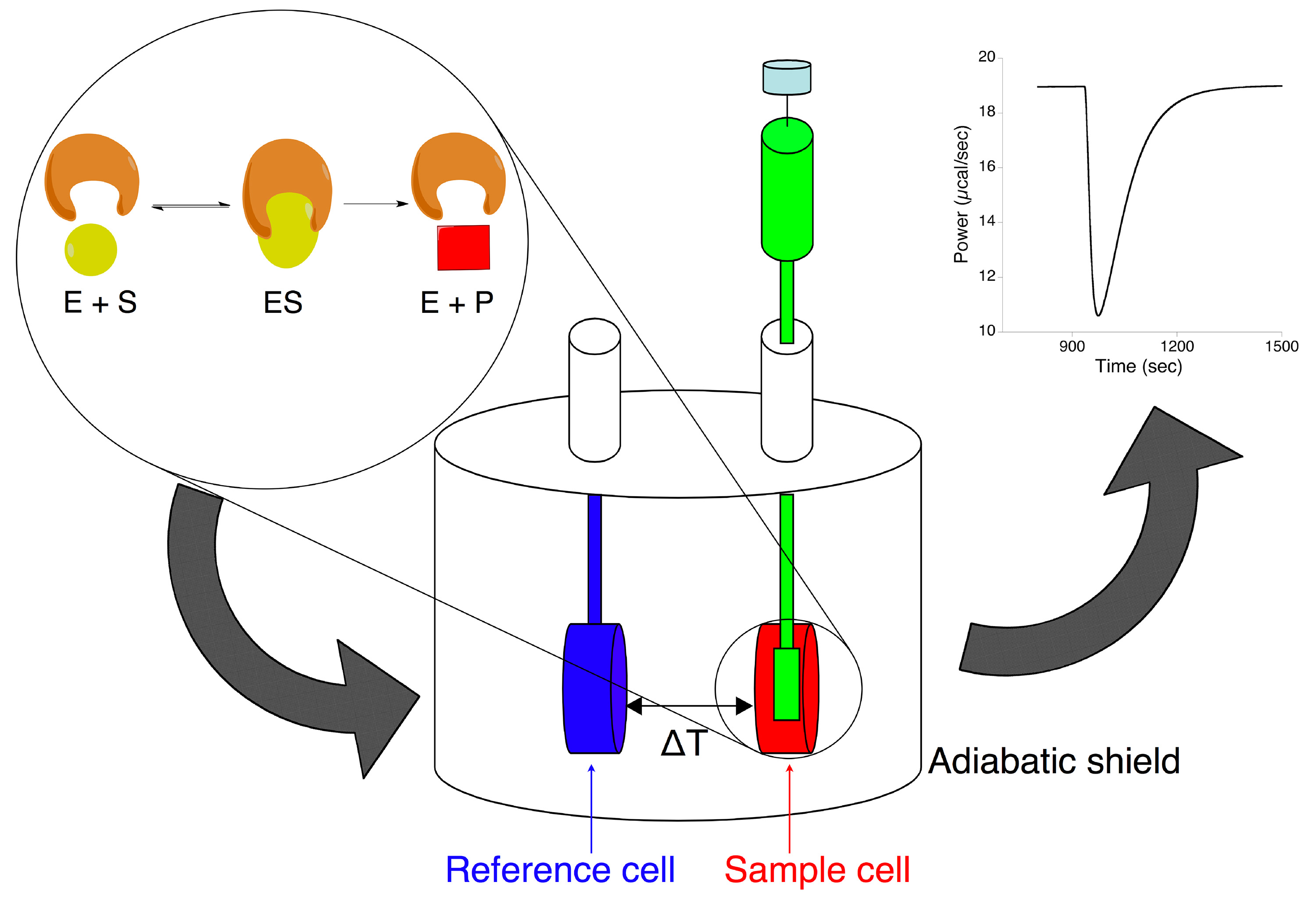
図1。酵素反応を研究するための等温滴定熱量計の概略図。酵素反応は、酵素溶液に(注射器)に基板の滴定の際に発生する(試料セル内)THERの変化をもたらすサンプルセルとリファレンスセル定数の温度差を維持するのに必要な熱量計によってリリースMALパワー、。 拡大画像を表示するにはここをクリックしてください。
全体的に、熱変化(Q)は、反応(ΔH)のモルエンタルピーひいては全体積倍の濃度で与えられる(n)が生成された生成物のモル数に比例する。
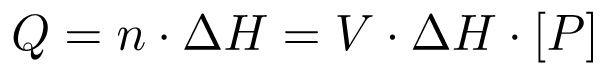 (3)
(3)
反応速度に対応する時間(のdP / dt)は上の生成物の形成は、このようにして関係を介して、同時に(dQ / dtは)上で発生した熱の量に関連することができる。
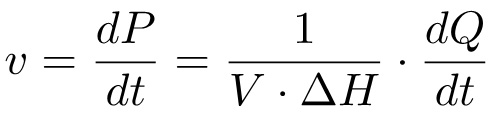 (4)
(4)
この式によれば、ミカエリス-メンテンプロットを得るためには、私を測定する必要がある)合計モルエンタルピーΔH、および異なる基質濃度でdQ / dtは ii)の熱流。通常、これは、2つの異なる実験で行われる:第一の実験( 方法1、M1)において、基質は、酵素溶液中に注入され、完全な基質変換のための熱を測定する;第二の実験( 方法2、M2)は、基板の複数回の注入が行われ、熱発生の速度は、異なる基質濃度で測定される。これら2つのデータセットは、動力学的パラメーターK Mおよびkの猫を導出するのに十分である。
本記事では、ITCを用いて行われる酵素反応のための速度論的パラメータを決定する一般的なプロトコルが記載されている。私たちは、Canavalia ensiformis尿素により尿素加水分解法を適用それ自体、基準システムとして。この方法を用いて得られた結果を、文献に報告されたデータとの間の良好な一致は、このアプローチの信頼性を実証する。
プロトコル
1。準備サンプル
- 酵素溶液2ミリリットル、各実験操作のための基質溶液0.5mlを準備します。基質添加時に希釈および混合の熱を最小限にするために同一の組成を有する緩衝液中で酵素と基質の濃縮ストック溶液を希釈する。
- 実験中にpHの変化を防ぐために適切である緩衝液の条件を選択してください。例えば、20mMのHEPES pH7の中性pHでの測定に適している。
注:プロトン交換が関与している場合、それらは反応の測定ΔHに影響するため、使用されるバッファのプロトン化エンタルピーは、考慮しなければならない。分析中のシステム上のバッファや添加物分子の可能な具体的な効果を考慮しなければなりません。有機溶媒( 例えば、DMSO)を酵素液に含まれている場合、基質溶液中に、正確に同じ濃度でそれらを追加する。 - M1の実験のために、開始nM範囲の酵素濃度( 例えば、1 nM)を有する、酵素濃度よりも上記とK M 3桁以上高い基質濃度を有する。
- M2の実験では、PM-nM範囲の酵素濃度( 例えば 15時)から開始します。シリンジ内の基質濃度は、10mMの範囲( 例えば、400 mM)の中にある。
注:単回注射のM1の実験では、濃度が、実験時間をかけて製品への総基質変換を達成するのに十分でなければなりません。そのため、酵素の濃度は、酵素レートによって異なります。低k 猫との酵素が検討されている場合、より高い酵素濃度(最大10μm)を使用する必要があります。一方、M2実験に使用される酵素濃度は、注入された基板はわずか(5%未満)が消費酵素反応が定常状態で進行することがあることを保証しなければならない。このため、より高い酵素の効率、必要な酵素濃度は低い。実験の終わりに、試料セル中の基質濃度は、K Mよりも高くあるべきである。
- 実験中にpHの変化を防ぐために適切である緩衝液の条件を選択してください。例えば、20mMのHEPES pH7の中性pHでの測定に適している。
- 慎重に280nmで、比色、BCAアッセイ11で適切な分析手順( 例えば吸光度、酵素と基質濃度をチェックしてください。これは、熱力学および動力学的パラメータの正確で信頼性の高い計算を得るために必要とされる。
- 溶液のpHを測定し、酵素と基質溶液の両方のpHの不一致が実験条件(±0.05 pH単位)の下で最小であることを確認してください。
2。実験を行う
注:同じ手順を両方次々に実行され、M1とM2の実験のために適用する必要があります。
- サンプルセルと注射器がメーカーのに従って洗浄されていることを確認し指示。蒸留水で機器に付属のローディングシリンジを記入し、静かに、試料セル内の針を挿入し、セルを満たし、同じ注射器を用いて液体を除去。この方法を使用して、緩衝液で2回蒸留水で2回試料セルを洗浄し。
- 慎重に気泡の形成を回避し、ローディングシリンジを用いて酵素液2mlで試料セルをロードする。それは細胞幹の上をこぼしまで、ゆっくり細胞に溶液を注入する。 CAのスパートを生成します。ソリューションの0.25ミリリットルのセルに。二回繰り返します。このステップは、試料セル内に混入した気泡を除去する。
- 細胞幹細胞とポートとの間の棚に積載注射器の針を配置し、余分な溶液を除去。
- VP-Viewerプログラムを起動し、コンピュータのインターフェイスから、目的の実験温度より低い温度3℃にITC装置を平衡化。これは、長い平衡化を避けるために必要である受動的な楽器の冷却装置による実験前の期間、。
- 上記と同様の手順で、蒸留水で参照セルを埋める。高イオン強度又は高浸透圧を有する緩衝液を試料溶液中にある場合に、同じ緩衝液は、基準として使用されるべきである。
- 薄いシリコンチューブを使用して、注射器の充填ポートにプラスチックシリンジをリンクします。水が注射器が満杯であることを示す、上部充填口から出てくるまで、水の中に針先端を配置し、汲み上げ注射器を充填する。その後、注射器を空にし、水から注射器の先端を移動させ、空気を引き込む。この手順では、緩衝液を注射器を洗浄し、システムを介して空気を引き込む。続いて、チューブの底に溶液の少量を残して、慎重に引き、完全に注射器を充填する、基質溶液0.5mlを含有する細管内に注射針を配置する。
- からコンピュータインターフェース、Enterキーを押し、「閉じる充填ポート」。シリコンローディングチューブを取り外します。を押して「パージとリフィル「気泡を取り除くために、バックバルク溶液にそれらを排出するために注射器を可能にするためのボタンを。二回繰り返します。
- 任意の水滴を除去し、サンプルセル中に注射器の針を配置するために脇に拭き、注射器を移動します。
- コンピュータ上で、適切な運転パラメータを設定します。実験的な酵素および基質濃度を示すことができる。
- M1実験では、再現性を確認するために、基板の少なくとも二つの加算(5〜30μl)を設定する。熱信号は、次の添加の前にベースラインに戻っていることを確認するのに十分な大きさごとに注射( 例えば千秒)の間の間隔の時間を設定します。
- M2の実験では、複数( 例えば 15×5〜10μL)の注射を設定します。注射の間隔を設定します( 例:180秒)sにシステムを可能にする各注射後に新しいベースラインに火力をtabilize。
NOTE:M2実験において注射の間の間隔時間は、測定値が定常状態条件下で行うことができるように、基質のかなりの量の変換を回避するのに十分に短くすべきである。 - M2の実験では、その対応する値、その後のデータ解析の際に廃棄されている最初の注射のために、少量( 例えば 2μL)を使用します。実際に、多くの場合、注射器の先端を介して初期基板拡散によるアーチファクトを提示し、注射針に捕捉された気泡の存在である。
- 基準パワー、反応が開始する前に、ベースラインが20の値で、配置されているおおよそのレベルを設定します。その後、実験的な酵素と基質濃度を定義し、テストの名前を選びました。
- 典型的には25℃で、実験温度を定義するITCは2間の作業温度を許可する ℃〜80℃、実験は、実行するための準備ができています。実験を開始するを押して「スタート」ボタンを押します。
- 実験が完了したら、製造業者の説明書に従って試料セルとシリンジをきれい。
- データの再現性を確認するための実験を少なくとも1つまたは2つ以上の回繰り返す。
3。データ解析
- 解析プログラムから、「データを読み込む」ボタンをクリックして、実施したM2の実験の。ITCファイルがあるフォルダに移動します。 「ファイルの種類」のスクロールダウン矢印をクリックし、「酵素アッセイ(それを?)」を選択します。その後、クリックして、M2の実験の。ITCファイルを開きます。
- 式5によると、M1の実験の曲線を統合からの反応のΔHを取得します。
"幅=" 126 "/>(5)- 「データの読み込み」ボタンをクリックすると、行わM1実験のファイル。ITCを選択します。原産地を使用して、一つのピークをそれぞれ含むさまざまな部分でのトレースを分割し。OPJファイルとして各ピークを保存します。単回注射のM1実験のサーモのベースラインのずれに起因する、最初のピークに対応したファイルを開き、それを統合し、得られた面積値を分割し、μcalで表し、試料セル中の最終基質濃度によって、μmで、および細胞容積によって式5の反応のΔHによると、決定、リットルで表した。
- M1の実験の第二のピークのために同じ手順を繰り返し、2ΔH測定の平均値を取得する。
- 各注射後の元のベースラインと新しいベースラインとの間の差を測定するのM2実験からDQ / DTを決定します。 Eを変換式4による反応速度への新実験データはM1の実験で得られたエンタルピー値を使用してミカエリス·メンテン式にデータをフィットします。
- 解析プログラムから、「データを読み込む」ボタンをクリックして、実施したM2実験のファイル。ITCを選択します。 「ファイルの種類」のスクロールダウン矢印をクリックし、「酵素アッセイ(それを?)」を選択します。
- 酵素アッセイダイアログボックスは4つのモデルのいずれかを選択できるように、オープンします。窓から - "基板のみ方法2」を選択します。
- ΔHウィンドウでは、ステップ4.2で得られたΔHの値を示している。
- 適合手順で使用される濃度値をチェックするために「集中」ボタンをクリックしてください。 「平均時間(P)」ボタンをクリックしてください。ダイアログボックスは、デフォルト値を変更するか、受け入れる機会を与えて開く。この値は、各注入前の時間を表す各基質濃度における電力レベルを決定するためのイオンで楽器平均電力信号。デフォルト値を確認して[OK]をクリックします。
- 「ゼロY軸」ボタンをクリックしてください。カーソルは、Y = 0で配置するために、ポイントをダブルクリックすることができ十字に変わります。ちょうど最初の注入点の前にダブルクリックします。
- 計算速度のボタンをクリックします。速度は、(アカウント希釈効果を考慮)試料セル容積のために滴定中に加え、基板を割った基質濃度に対してプロットする。この動作は、K Mおよびkの猫を得るために取り付けることができる典型的なミカエリス·メンテン·プロットを与える。
- いくつかのポイントを削除する場合は、「切り捨てデータ」ボタンを選択します。不正なデータポイントと入力するマーカーやプレスの1をダブルクリックを除外するためにデータマーカーを移動します。
- カーブにフィットし、するような「モデルに適合」機能を使用する速度定数を得る。
結果
ウレアーゼ(EC 3.5.1.5、尿素アミドヒドロラーゼ)は古細菌、バクテリア、単細胞真核生物および植物に見られるマルチサブニッケル含有酵素である。自然発生的にアンモニアと炭酸水素塩( 式6)12の第2の分子を与えるために分解し、アンモニアおよびカルバメートに尿素の加水分解を触媒する有機窒素無機化の最終段階で、このタンパク質が作用し、。
ディスカッション
既存の方法に関連して酵素活性を研究するITCの意義
結合平衡を研究するための古典的な用途に加えて、等温滴定熱量計は、システムの改変または標識を必要とせずに、プローブとして反応熱を用いて溶液中で酵素反応を特徴付けるため信頼性が高く、迅速な方法を提供する。速度論的パラメータは猫を kとK Mは 、通常、生成物?...
開示事項
著者らは、開示することは何もありません。
謝辞
スペシャリティ肥料製品会社(SFP)は、この研究のために必要な資金を提供するために認められている。
資料
| Name | Company | Catalog Number | Comments |
| HEPES | Sigma | H3375 | dissolving in water and adjusting pH with NaOH |
| TRIZMA-Base | Sigma | T1503 | dissolving in water and adjusting pH with HCl |
| Sodium dihydrogen phosphate | Riedel-de-Haen | 4270 | dissolving in water |
| Sodium phosphate dibasic | Riedel-de-Haen | 30427 | dissolving in water |
| Urea | Sigma | U4128 | dissolving in water at 40 °C |
| Canavalia ensiformis urease (type C-3) | Sigma | U0251 | dissolving in 20 mM HEPES pH 7 and stored at -80 °C |
| VP-ITC on Origin 7.0 | MicroCal (GE Healthcare) | SYS13901 | instrument |
| VPViewer2000 1.30.00 on Origin 7.0 | MicroCal (GE Healthcare) | data acquisition software supplied with the instrument |
参考文献
- Leavitt, S., Freire, E. Direct measurement of protein binding energetics by isothermal titration calorimetry. Curr. Opin. Struct. Biol. 11, 560-566 (2001).
- Ladbury, J. E. Application of isothermal titration calorimetry in the biological sciences: things are heating up! BioTechniques. 37, 885-887 (2004).
- Zambelli, B., Bellucci, M., Danielli, A., Scarlato, V., Ciurli, S. The Ni2+ binding properties of Helicobacter pylori NikR. Chem. Commun. , 3649-3651 (2007).
- Zambelli, B., et al. High-affinity Ni2+ binding selectively promotes binding of Helicobacter pylori NikR to its target urease promoter. J. Mol. Biol. 383, 1129-1143 (2008).
- Duff, M. R., Grubbs, J., Howell, E. E. Isothermal titration calorimetry for measuring macromolecule-ligand affinity. J. Vis. Exp. , (2011).
- Ghai, R., Falconer, R. J., Collins, B. M. Applications of isothermal titration calorimetry in pure and applied research--survey of the literature from 2010. J. Mol. Recognit. 25, 32-52 (2012).
- Todd, M. J., Gomez, J. Enzyme kinetics determined using calorimetry: a general assay for enzyme activity. Anal. Biochem. 296, 179-187 (2001).
- Bianconi, M. L. Calorimetry of enzyme-catalyzed reactions. Biophys. Chem. 126, 59-64 (2007).
- Demarse, N. A., Killian, M. C., Hansen, L. D., Quinn, C. F. Determining enzyme kinetics via isothermal titration calorimetry. Methods Mol. Biol. 978, 21-30 (2013).
- Michaelis, L., Menten, M. Die kinetik der invertinwirkung. Biochem. Z. 49, 333-369 (1913).
- Walker, J., Wilson, K., Walker, J. . Principle and techniques of practical biochemistry. , 312-356 (2000).
- Ciurli, S., Sigel, A., Sigel, H., Sigel, R. K. O. . Nickel and its surprising impact in nature. 2, 241-278 (2007).
- Zambelli, B., Musiani, F., Benini, S., Ciurli, S. Chemistry of Ni2+ in urease: sensing, trafficking, and catalysis. Acc. Chem. Res. 44, 520-530 (2011).
- Zonia, L. E., Stebbins, N. E., Polacco, J. C. Essential role of urease in germination of nitrogen-limited Arabidopsis thaliana seeds. Plant Physiol. 107, 1097-1103 (1995).
- Follmer, C. Insights into the role and structure of plant ureases. Phytochemistry. 69, 18-28 (2008).
- Sumner, J. B. The isolation and crystallization of the enzyme urease. J. Biol. Chem. 69, 435-441 (1926).
- Krajewska, B. Ureases I. Functional, catalytic and kinetic properties: A review. J. Mol. Cat. B. 59, 9-21 (2009).
- Callahan, B. P., Yuan, Y., Wolfenden, R. The burden borne by urease. J. Am. Chem. Soc. 127, 10828-10829 (2005).
- Krajewska, B., van Eldik, R., Brindell, M. Temperature- and pressure-dependent stopped-flow kinetic studies of jack bean urease. Implications for the catalytic mechanism. J. Biol. Inorg. Chem. 17, 1123-1134 (2012).
- Hausinger, R. P., Karplus, P. A., Huber, R., Poulos, T., Wieghardt, K. . Handbook of Metalloproteins. , 867-879 (2001).
- Goldberg, R., Kishore, N., Lennen, R. Thermodynamic quantities for the ionization reactions of buffers. J. Phys. Chem. Ref. Data. 31, 231-370 (2002).
- Baumann, M. J., et al. Advantages of isothermal titration calorimetry for xylanase kinetics in comparison to chemical-reducing-end assays. Anal. Biochem. 410, 19-26 (2011).
- Noske, R., Cornelius, F., Clarke, R. J. Investigation of the enzymatic activity of the Na+,K+-ATPase via isothermal titration microcalorimetry. Biochim. Biophys. Acta. 1797, 1540-1545 (2010).
- Harmon, K. M., Niemann, C. The competitive inhibition of the urease-catalyzed hydrolysis of urea by phosphate. J. Biol. Chem. 177, 601-605 (1949).
- Benini, S., Rypniewski, W. R., Wilson, K. S., Ciurli, S., Mangani, S. Structure-based rationalization of urease inhibition by phosphate: novel insights into the enzyme mechanism. J. Biol. Inorg. Chem. 6, 778-790 (2001).
- Segel, I. H. . Enzyme kinetics: behavior and analysis of rapid equilibrium and steady-state enzyme systems. , (1993).
転載および許可
このJoVE論文のテキスト又は図を再利用するための許可を申請します
許可を申請さらに記事を探す
This article has been published
Video Coming Soon
Copyright © 2023 MyJoVE Corporation. All rights reserved