Zum Anzeigen dieser Inhalte ist ein JoVE-Abonnement erforderlich. Melden Sie sich an oder starten Sie Ihre kostenlose Testversion.
Method Article
Hot Biologische Katalyse: Isotherme Titrationskalorimetrie zu Enzymreaktionen Charakterisieren
In diesem Artikel
Zusammenfassung
Isotherme Titrationskalorimetrie Maßnahmen Wärmestrom freigegeben oder in chemischen Reaktionen absorbiert. Dieses Verfahren kann verwendet werden, um Enzym-Katalyse zu quantifizieren. In diesem Papier, das Protokoll für die Instrumental Setup-Experiment läuft, und Datenanalyse wird allgemein beschrieben, und der Charakterisierung der enzymatischen Hydrolyse von Harnstoff Bohne Urease aufgebracht.
Zusammenfassung
Isotherme Titrationskalorimetrie (ITC) ist eine gut beschriebene Technik, die die Wärme freigesetzt oder während einer chemischen Reaktion aufgenommen, sie als eine intrinsische Sonde zur Charakterisierung praktisch jeden chemischen Prozess misst. Heutzutage wird dieses Verfahren weitgehend angewendet, um thermodynamische Parameter des biomolekularen Bindungsgleichgewichte bestimmen. Zusätzlich ITC wurde gezeigt, können direkt zu messen Kinetik und thermodynamischen Parameter (k cat, K M, &Dgr; H) von enzymatischen Reaktionen, obwohl diese Anwendung noch unzureichend genutzt werden. Als Wärme Änderungen spontan während der enzymatischen Katalyse kommen, die jedoch ITC keine Änderung oder die Kennzeichnung des Systems unter Analyse bedürfen und kann in Lösung durchgeführt werden. Darüber hinaus muss das Verfahren wenig Materialmenge. Diese Eigenschaften machen ITC eine unschätzbare, leistungsstarke und einzigartige Werkzeug zur Enzymkinetik in mehreren Anwendungen, wie zum Beispiel, Arzneimittelforschung studieren.
k cat und K M der enzymatischen Hydrolyse von Harnstoff durch Canavalia ensiformis (jack bean) Urease. Berechnung der intrinsischen molare Enthalpie (&Dgr; H int) der Reaktion durchgeführt. Die so erhaltenen Werte sind konsistent mit früheren Daten in der Literatur berichtet, was die Zuverlässigkeit der Methode.
Einleitung
Quantitative Bestimmung von biochemischen Reaktionen Einblicke in die biologischen Prozesse auf der Grundlage des Lebens. Kalorimetrie bietet eine markierungsfreie Methode zur quantitativen Charakterisierung praktisch jede chemische Reaktion in Lösung. Dieses Verfahren misst die Wärme freigegeben oder im Laufe der Zeit absorbiert wird, und ist daher eine universelle Erfassungssystem und eine sehr bequeme Methode, um die Menge der reagierenden Moleküle (dh Bindung Thermodynamik), als auch die Reaktionsgeschwindigkeit (dh Kinetik) messen zu quantifizieren. Insbesondere hat isothermen Titrationskalorimetrie (ITC) als Methode der Wahl, um die Thermodynamik der biomolekularen Gleichgewichte zu charakterisieren, mit Protein-Ligand-Protein-Protein, Protein-Metallionen und Protein-DNA-Wechselwirkungen 6.1 verabschiedet. Darüber hinaus ist die Fähigkeit des ITC, um kinetische Informationen ist es ein sehr leistungsfähiges System zur Enzymkatalyse zu messen, obwohl das Potenzial dieser Anwendung ist nochunterschätzt 09.07.
Die Michaelis-Menten-Gleichung 10 ist eine quantitative Beschreibung von enzymatischen Reaktionen, da es eine Beziehung zwischen der Reaktionsgeschwindigkeit und der Substratkonzentration, in Abhängigkeit von zwei kinetischen Parameter: die Michaelis-Konstante (K M) und die katalytische Geschwindigkeitskonstante (k cat) . Die k cat / K M-Verhältnis ist als das katalytische Effizienz eines Enzyms bezeichnet. In der Praxis Bestimmung von K M und k cat für eine bestimmte Reaktion eine vollständige Beschreibung der Katalyse.
In einem typischen Enzymreaktion (Abb. 1), einem Substrat (S) mit dem Enzym (E) die Enzym-Substrat-(ES)-Komplex gebildet wird, der anschließend in den Übergangszustand aktiviert wird (ES *). Letzteres wird in das Enzym-Produkt (EP)-Komplex dissoziiert, die schließlich umgewandelt. Diese Schritts werden durch die folgende Reaktion beschrieben.
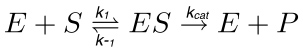 (1)
(1)
k 1 die Geschwindigkeitskonstante für die Bildung des Komplexes ES, k-1 die Geschwindigkeitskonstante für die Dissoziation des Komplexes ES, während k cat ist die katalytische Geschwindigkeitskonstante oder Umsatzzahl.
: Gemäß der Michaelis-Menten-Gleichung 10 kann die Geschwindigkeit der Reaktion berechnet werden
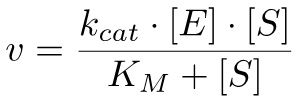 (2)
(2)
wobei K = M (k-1 + k cat) / k 1 und k cat = v max / [E], wobei V max die maximale Geschwindigkeit erreicht, wenn alle Enzyms an das Substrat gebunden.
Die isotherme Titrationskalorimeter ist das Instrument in dieser Studie verwendet, um die enzymatische Hydrolyse von Harnstoff zu charakterisieren. Dieses Instrument besteht aus einem adiabatischen Abschirmung, die zwei geprägte förmigen Zellen (Fig. 1) hergestellt. Diese sind nach außen mit schmalen Zugang Rohren verbunden. Die Probenzelle (ca. 1,4 ml) wird mit der Enzymlösung beladen, und die Referenzzelle wird in der Regel mit Wasser oder mit der für die Analyse verwendeten Lösungsmittel gefüllt. Eine rotierende Spritze mit langer Nadel und einem Rührstäbchen Paddel befestigt ist, die gewöhnlich ca. 0,3 ml Substratlösung wird auf die Probenzelle montiert. Eine thermoelektrische Vorrichtung mißt die Temperaturdifferenz zwischen der Probe und der Referenzzelle und mit einem "Zellrückkopplungsnetzwerk", behält er diese Differenz auf Null durch Addieren oder Subtrahieren Wärme. Während des Experiments wird das Substrat in der Enzymlösung bei einem konstanten gewählten Temperatur eingespritzt. Wenn die enenzymatische Reaktion stattfindet, die Menge an Wärme freigesetzt oder absorbiert wird, ist proportional zu der Anzahl von Substratmolekülen, die in Produktmoleküle umgewandelt werden. Darüber hinaus ist die Wärmestromrate direkt mit der Geschwindigkeit der Reaktion stehen. Die gemessenen Daten, wie eine Abweichung der Spur von der ersten Wärmegrundlinie (Fig. 1) erscheinen, (μcal / sec) durch das Instrument auf die Probenzelle, die proportional zu der in der Probenzelle auftretenden Wärmestrom versorgt sind der thermische Leistungs über die Zeit.
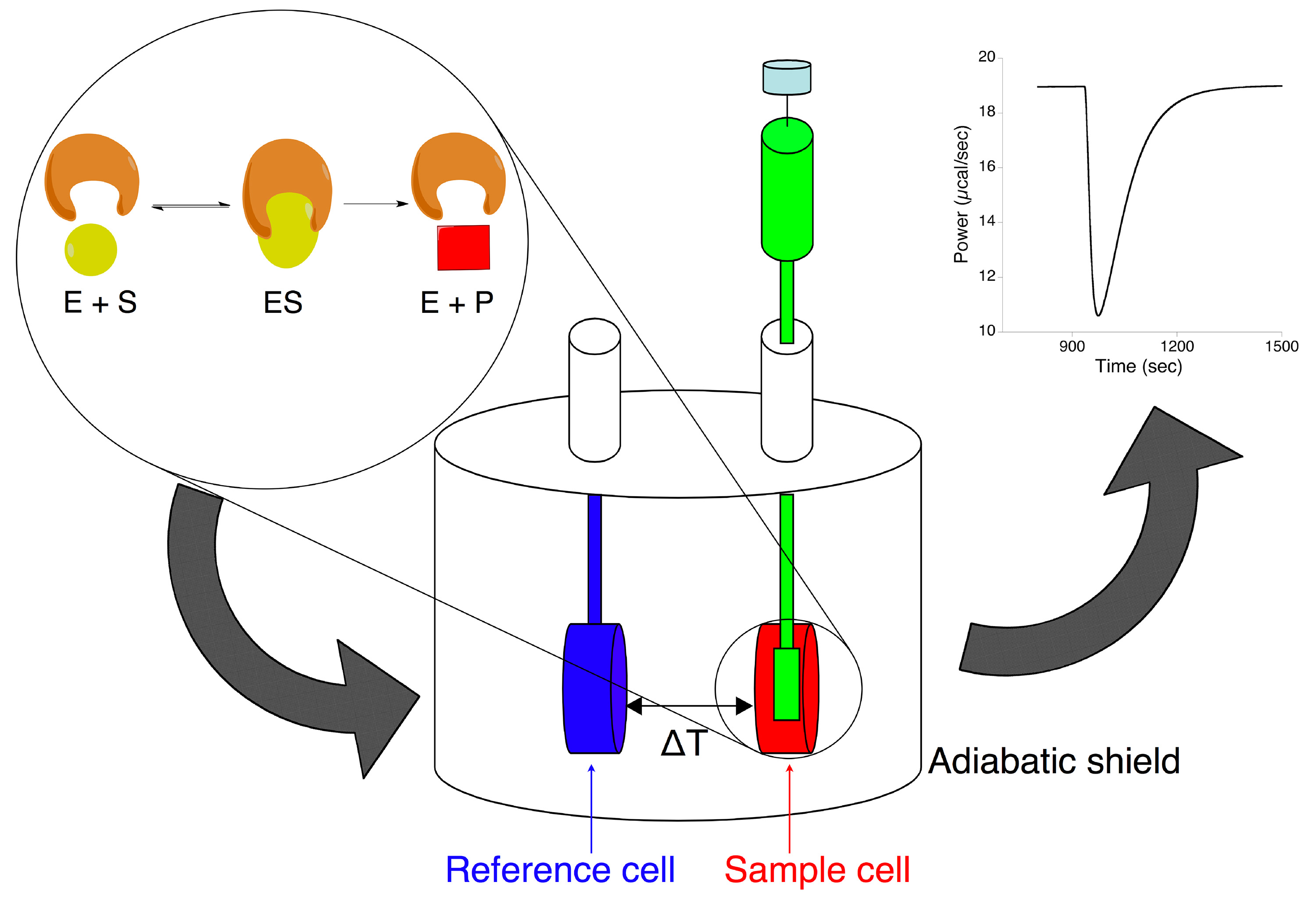
Fig. 1 ist. Schematische Darstellung der isothermen Titrationskalorimeter um enzymatische Reaktionen zu untersuchen. Einer enzymatischen Reaktion bei der Titration des Substrats (in der Injektionsspritze) auftritt in die Enzymlösung (in der Probenzelle) zu einer Änderung der thermoFehlleistung durch das Kalorimeter, notwendig, um die Temperaturdifferenz zwischen der Probenzelle und der Referenzzelle konstant zu halten veröffentlicht. Klicken Sie hier für eine größere Ansicht.
Insgesamt ist die Wärmeänderung (Q) proportional zum molaren Enthalpie der Reaktion (AH) und der Anzahl von Molen des Produkts erzeugt (n), die wiederum durch den Gesamtvolumen-fache der Konzentration gegeben:
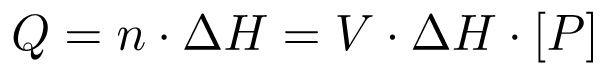 (3)
(3)
Die Produktbildung über die Zeit (dP / dt), die die Reaktionsgeschwindigkeit entspricht, kann somit der Wärmemenge über die gleiche Zeit (dQ / dt) durch die Beziehung erzeugt verwandt sind:
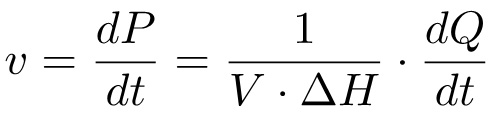 (4)
(4)
Nach dieser Gleichung um eine Michaelis-Menten-Plot zu erhalten ist es notwendig, zu messen i) die Gesamtstoff Enthalpie AH, und ii) der Wärmestrom dQ / dt bei verschiedenen Substratkonzentrationen. Gewöhnlich wird dies auf zwei verschiedene Experimente durchgeführt: Im ersten Versuch (Methode 1, M1), wird das Substrat in der Enzymlösung injiziert und die Wärme zur vollständigen Substratumsatz gemessen wird; in dem zweiten Experiment (Methode 2, M2), werden mehrere Injektionen des Substrats durchgeführt und die Rate der Wärmeerzeugung bei verschiedenen Substratkonzentrationen gemessen. Diese zwei Sätze von Daten ausreichend sind, um die kinetischen Parameter K m und k cat abzuleiten.
Im vorliegenden Artikel wird eine allgemeine Protokoll, um die kinetischen Parameter für enzymatische Reaktionen unter Verwendung von ITC bestimmen beschrieben. Wir wendeten die Methode zur Harnstoff-Hydrolyse durch Canavalia ensiformis Harnstoffse als Bezugssystem. Die gute Übereinstimmung zwischen den mit dieser Methode erzielten Ergebnisse sowie die in der Literatur berichtet Daten zeigen die Zuverlässigkeit dieses Ansatzes.
Protokoll
1. Vorbereitung von Proben
- Herstellung von 2 ml Enzymlösung und 0,5 ml der Substratlösung für jede Versuchsreihe. Konzentriertes Stammlösungen von Enzym und Substrat in Pufferlösungen mit identischer Zusammensetzung, um die Wärme der Verdünnung und Durchmischung während der Substratzugabe zu minimieren.
- Wählen Sie die Pufferbedingungen, die ausreichend sind, um pH-Änderung während Experiment zu verhindern. Zum Beispiel ist 20 mM HEPES pH 7 ausreichend für Messungen bei neutralem pH-Wert.
HINWEIS: Wenn Protonenaustausch beteiligt ist, muss die Protonierung Enthalpien der verwendeten Puffer betrachtet werden, da sie die gemessene &Dgr; H der Reaktion beeinflussen. Möglichen spezifischen Auswirkungen der Puffer oder Zusatzstoffe Moleküle auf dem System unter Analyse berücksichtigt werden. Werden organische Lösungsmittel (z. B. DMSO) werden in der Enzymlösung enthalten, fügen sie in genau der gleichen Konzentration in der Substratlösung. - Für die M1 Experiment startenmit Enzym-Konzentration im nM-Bereich (beispielsweise 1 nM) und mit einer Substratkonzentration von mindestens drei Größenordnungen höher ist als die Enzymkonzentration und oberhalb der K M.
- Für das Experiment M2, beginnen Sie mit der Enzymkonzentration in der pM-nM-Bereich (zB 15 Uhr). Substratkonzentration in der Spritze ist im mm-Bereich (z. B. 400 mm).
HINWEIS: In der einzigen Injektion M1 Versuch sollten die Konzentrationen ausreichend sein, um die Gesamtsubstratumwandlung in das Produkt über die Versuchszeit zu erreichen. Daher ist die Konzentration des Enzyms hängt von dem Enzym ab: wenn Enzyme mit niedrigem k cat werden noch untersucht, höhere Enzymkonzentrationen (bis zu 10 &mgr; M) verwendet werden. Andererseits muss Enzymkonzentrationen in der M2-Experiment verwendet sicherzustellen, daß die eingespritzte Substrat nur geringfügig (weniger als 5%) verbraucht wird und daß die Enzymreaktion schreitet im stationären Zustand. Aus diesem Grund wird, je höher dieEnzymeffizienz, desto geringer ist die benötigte Enzymkonzentration. Am Ende des Experiments sollte der Substratkonzentration in der Probenzelle über K M sein.
- Wählen Sie die Pufferbedingungen, die ausreichend sind, um pH-Änderung während Experiment zu verhindern. Zum Beispiel ist 20 mM HEPES pH 7 ausreichend für Messungen bei neutralem pH-Wert.
- Sorgfältig zu prüfen, Enzym-und Substratkonzentrationen mit einem geeigneten Analyseverfahren (zB Absorption bei 280 nm, kolorimetrisch, BCA-Assays 11. Dies ist erforderlich, um genaue und zuverlässige Berechnung der thermodynamischen und kinetischen Parameter zu erhalten.
- Der pH-Wert der Lösung und sicherzustellen, dass der pH-Fehlanpassung sowohl dem Enzym und dem Substrat Lösungen minimal ist unter den Versuchsbedingungen (± 0,05 pH-Einheiten).
2. Durchführung des Experiments
HINWEIS: Das gleiche Verfahren ist sowohl für die M1-und M2-Experiment, das einen nach dem anderen durchgeführt werden, angewandt werden.
- Stellen Sie sicher, dass die Probenzelle und die Injektionsspritze sind nach der Hersteller gereinigtAnweisungen. Füllen Sie die Spritze mit der Be-Messgerät mit destilliertem Wasser vorgesehen ist, vorsichtig die Nadel in der Probenzelle, füllen Sie die Zelle aus und entfernen Sie die Flüssigkeit mit der gleichen Spritze. Mit dieser Methode, wäscht den Probenzelle zweimal mit destilliertem Wasser und zweimal mit dem Puffer.
- Laden der Probenzelle mit 2 ml einer Enzymlösung mit der zu ladende Spritze sorgfältig vermeidet die Bildung von Luftblasen. Langsam spritzen die Lösung in die Zelle, bis sie heraus schwappt die Oberseite der Zelle Stamm. Produzieren Sie ein Spurt von ca. 0,25 ml Lösung in die Zelle. Zweimal wiederholen. Dieser Schritt entfernt von Luftblasen in der Probenzelle eingeschlossen.
- Setzen Sie die Nadel der Spritze Laden auf der Leiste zwischen der Zelle und dem Zellstamm Port und entfernen überschüssige Lösung.
- Starten des VP-Viewer-Programm und von der Computerschnittstelle, die ITC äquilibrieren Instrument auf eine Temperatur 3 ° C unter die gewünschte Versuchstemperatur. Dies ist erforderlich, um Gleichgewichtseinstellung zu vermeiden langPerioden vor dem Experiment durch die passive Kühleinrichtung instrumental.
- Füllen Sie die Referenzzelle mit destilliertem Wasser mit dem gleichen Verfahren oben. Wenn Puffer mit hoher Ionenstärke oder hohe Osmolalität in der Probenlösung, sollte derselbe Puffer als Referenzlösung verwendet werden.
- Verknüpfen einer Plastikspritze auf die Einfüllöffnung der Injektionsspritze, mit einem dünnen Silikonschlauch. Füllen der Injektionsspritze Platzierung der Nadelspitze in Wasser und Erstellung, bis Wasser aus dem oberen Einfüllöffnung, die anzeigt, dass die Injektionsspritze voll ist. Dann bewegen Sie die Spitze der Spritze aus dem Wasser und stellt Luft, um die Injektionsspritze zu entleeren. Mit diesem Verfahren, waschen Sie die Injektionsspritze mit Puffer und saugen Luft durch das System. Anschließend legen Sie die Injektionsnadel in einem engen Röhrchen mit 0,5 ml Substratlösung, sorgfältig auszuarbeiten und der Injektionsspritze vollständig zu füllen, so dass kleine Menge der Lösung an der Unterseite des Rohres.
- Von derComputer-Schnittstelle, drücken Sie auf "Schließen Einfüllöffnung". Entfernen Sie die Silizium Laderohr. Drücken Sie auf "Purge und Refill"-Taste, damit der Injektionsspritze, um Luftblasen zu entfernen und sie zurück in die Groß Lösung zu vertreiben. Zweimal wiederholen.
- Bewegen Sie die Injektionsspritze, wischen an den Seiten, alle Tropfen zu entfernen und die Nadel der Injektionsspritze in die Probenzelle.
- Auf dem Computer, legen Sie die entsprechenden Betriebsparameter. Die experimentelle Enzym und Substratkonzentrationen angegeben werden.
- In der M1-Experiment, müssen Sie mindestens zwei Zugänge (5-30 ul) Substrat, um die Reproduzierbarkeit zu überprüfen. Die Abstandszeit zwischen jeder Injektion (z. B. 1000 s) groß genug ist, um sicherzustellen, dass die Wärmesignal wieder auf die Grundlinie vor der nächsten Zugabe.
- In der M2 Experiment, setzen mehrere (z. B. 15 x 10.5 ul)-Injektionen. Das Intervall zwischen den Injektionen (z. B. 180 Sek.), so dass das System stabilize die Wärmeversorgung des neuen Baseline nach jeder Injektion.
HINWEIS: Der Zeitabstand zwischen den Injektionen in der M2 Experiment sollte kurz genug sein, um die Umwandlung einer bedeutenden Menge des Substrats zu vermeiden, so dass die Messungen unter stationären Bedingungen durchgeführt werden. - In der M2-Experiment verwendet kleine Mengen (z. B. 2 &mgr; l) für die erste Injektion, deren entsprechender Wert während der anschließenden Datenanalyse verworfen. Tatsächlich stellt es oft Artefakte aufgrund der anfänglichen Substratdiffusion durch die Spritzenspitze und das Vorhandensein von Luftblasen in der Spritze Nadel gefangen.
- Stellen Sie die Referenzleistung, die ungefähre Ebene, in der die Grundlinie wird vor der Umsetzung gelegt werden beginnt, bei einem Wert von 20. Dann definieren Sie die experimentelle Enzym-und Substratkonzentrationen und wählte einen Namen für das Experiment.
- Bestimmen die Versuchstemperatur, typischerweise bei 25 ° C. ITC ermöglicht Arbeitstemperaturen zwischen 2 ° C und 80 ° C. Das Experiment ist bereit für den Betrieb. Drücken Sie "Start"-Taste, um das Experiment zu starten.
- Nachdem das Experiment beendet ist, reinigen Sie die Probenzelle und die Spritze entsprechend den Anweisungen des Herstellers.
- Wiederholen Sie das Experiment, zumindest ein oder zwei Mal, um die Reproduzierbarkeit der Daten zu überprüfen.
3. Datenanalyse
- Aus der Analyse-Programm, klicken Sie auf die Schaltfläche "Daten lesen", und navigieren Sie zu dem Ordner, in dem die. Itc-Datei des Experiments durchgeführt M2 befindet. Klicken Sie auf den Pfeil nach unten scrollen der "Dateityp" und wählen Sie "Enzym-Assay (es?)". Anschließend klicken Sie auf und öffnen Sie die. Itc-Datei des M2-Experiment.
- Besorgen Sie sich die AH der Reaktion aus der Integration der Kurve der M1 Versuch gemäß Gleichung 5.
"Breite =" 126 "/> (5)- Klicken Sie auf die Schaltfläche "Daten lesen", und wählen Sie die Datei. Itc der durchgeführten Experiment M1. Mit Origin, teilen Sie die Kurve in verschiedenen Teilen enthalten jeweils einen Spitzenwert und speichern Sie jede Spitzen als. Opj Datei. Öffnen der Datei entsprechend dem ersten Peak Abweichung von der Grundlinie im Thermogramm der einzelnen Injektion M1 Experiment integrieren und verteilen die erhaltene Fläche resultierende Wert, ausgedrückt in μcal, durch die endgültige Substratkonzentration in der Probenzelle, ausgedrückt in uM, und durch die Zellenvolumen in Litern, Bestimmen, gemäß Gleichung 5, die &Dgr; H der Reaktion.
- Wiederholen Sie den Vorgang für den zweiten Höhepunkt der M1 Experiment und einen durchschnittlichen Wert für die beiden AH-Messungen zu erhalten.
- Bestimmen dQ / dt von der M2 Experiment Messen der Differenz zwischen der ursprünglichen und der neuen Basisgrundlinie nach der jeder Injektion. Konvertieren Sie die Experimental Daten an Reaktionsgeschwindigkeiten nach Gleichung 4, mit dem in der M1-Experiment erhalten Enthalpie-Wert und passen die Daten an die Michaelis-Menten-Gleichung.
- Aus der Analyse-Programm, klicken Sie auf die Schaltfläche "Daten lesen", und wählen Sie die Datei. Itc der durchgeführten Experiment M2. Klicken Sie auf den Pfeil nach unten scrollen der "Dateityp" und wählen Sie "Enzym-Assay (es?)".
- Das Dialogfeld Enzymassay öffnet, so dass die Auswahl einer der vier Modelle. Wählen Sie "Methode 2 - nur Erde" aus dem Fenster.
- In der AH-Fenster, geben Sie den Wert von AH in Schritt 4.2 erhalten.
- Klicken Sie auf die "Konzentration", um die Konzentrationswerte zu überprüfen, um in der Anpassungsprozedur verwendet werden. Klicken Sie auf den "Durchschnittszeit (P)"-Taste. Das Dialogfeld geöffnet, so dass die Möglichkeit zu ändern oder den Standardwert. Dieser Wert steht für die Zeit vor jeder spritzenIonen in der das Instrument mittelt das Leistungssignal an den Leistungspegel bei jeder Substratkonzentration zu bestimmen. Klicken Sie auf OK, um den Standardwert zu bestätigen.
- Klicken Sie auf die Schaltfläche "Zero-Y-Achse". Der Cursor verwandelt sich in ein Fadenkreuz so dass ein Doppelklick auf einen Punkt, bei y = 0 setzen. Doppelklicken Sie kurz vor dem ersten Einspritzstelle.
- Klicken Sie auf die Schaltfläche Rate berechnen. Betrag ist gegenüber der Substratkonzentration, durch Teilung der während der Titration der Probe Zellvolumen (unter Berücksichtigung von Verwässerungseffekten) aufgenommen Substrat abgeleitet aufgetragen. Diese Operation gibt eine typische Michaelis-Menten-Plot, ausgestattet können bis K und M k cat erhalten.
- Wenn einige Punkte sollten gelöscht werden, wählen Sie die Schaltfläche "Truncate Daten". Verschieben Sie die Daten Marker, um die schlechten Datenpunkte und klicken Sie doppelt auf eine der Markierungen oder drücken Sie die Eingabetaste auszuschließen.
- Mit der Funktion "Fit zu modellieren", um die Kurve zu passen underhalten die kinetischen Konstanten.
Ergebnisse
Urease (EC 3.5.1.5; amidohydrolase Harnstoff) ist ein Multiunternickelhaltige Enzym in Archaea, Bakterien, einzelligen Eukaryoten und Pflanzen gefunden. Dieses Protein wirkt in der letzten Stufe von organischen Stickstoffmineralisierung und katalysiert die Hydrolyse von Harnstoff zu Ammoniak und Carbamat, das spontan zerfällt, um ein zweites Molekül Ammoniak und Bicarbonat (Gleichung 6) 12 zu ergeben.
Diskussion
Bedeutung der ITC, um die enzymatische Aktivität gegenüber bestehenden Methoden zu studieren
Zusätzlich zu den klassischen Anwendungen, verbindliche Gleichgewichte zu untersuchen, bietet isothermen Titrationskalorimetrie eine zuverlässige und schnelle Methode, um enzymatische Reaktionen in Lösung charakterisieren, mit der Reaktionswärme als Sonde, ohne Systemänderung oder die Kennzeichnung erfordern. Die kinetischen Parameter k cat und K M...
Offenlegungen
Die Autoren haben nichts zu offenbaren.
Danksagungen
Der Spezialdünger Products Company (SFP) ist für die Bereitstellung der erforderlichen Mittel für diese Studie bestätigt.
Materialien
| Name | Company | Catalog Number | Comments |
| HEPES | Sigma | H3375 | dissolving in water and adjusting pH with NaOH |
| TRIZMA-Base | Sigma | T1503 | dissolving in water and adjusting pH with HCl |
| Sodium dihydrogen phosphate | Riedel-de-Haen | 4270 | dissolving in water |
| Sodium phosphate dibasic | Riedel-de-Haen | 30427 | dissolving in water |
| Urea | Sigma | U4128 | dissolving in water at 40 °C |
| Canavalia ensiformis urease (type C-3) | Sigma | U0251 | dissolving in 20 mM HEPES pH 7 and stored at -80 °C |
| VP-ITC on Origin 7.0 | MicroCal (GE Healthcare) | SYS13901 | instrument |
| VPViewer2000 1.30.00 on Origin 7.0 | MicroCal (GE Healthcare) | data acquisition software supplied with the instrument |
Referenzen
- Leavitt, S., Freire, E. Direct measurement of protein binding energetics by isothermal titration calorimetry. Curr. Opin. Struct. Biol. 11, 560-566 (2001).
- Ladbury, J. E. Application of isothermal titration calorimetry in the biological sciences: things are heating up! BioTechniques. 37, 885-887 (2004).
- Zambelli, B., Bellucci, M., Danielli, A., Scarlato, V., Ciurli, S. The Ni2+ binding properties of Helicobacter pylori NikR. Chem. Commun. , 3649-3651 (2007).
- Zambelli, B., et al. High-affinity Ni2+ binding selectively promotes binding of Helicobacter pylori NikR to its target urease promoter. J. Mol. Biol. 383, 1129-1143 (2008).
- Duff, M. R., Grubbs, J., Howell, E. E. Isothermal titration calorimetry for measuring macromolecule-ligand affinity. J. Vis. Exp. , (2011).
- Ghai, R., Falconer, R. J., Collins, B. M. Applications of isothermal titration calorimetry in pure and applied research--survey of the literature from 2010. J. Mol. Recognit. 25, 32-52 (2012).
- Todd, M. J., Gomez, J. Enzyme kinetics determined using calorimetry: a general assay for enzyme activity. Anal. Biochem. 296, 179-187 (2001).
- Bianconi, M. L. Calorimetry of enzyme-catalyzed reactions. Biophys. Chem. 126, 59-64 (2007).
- Demarse, N. A., Killian, M. C., Hansen, L. D., Quinn, C. F. Determining enzyme kinetics via isothermal titration calorimetry. Methods Mol. Biol. 978, 21-30 (2013).
- Michaelis, L., Menten, M. Die kinetik der invertinwirkung. Biochem. Z. 49, 333-369 (1913).
- Walker, J., Wilson, K., Walker, J. . Principle and techniques of practical biochemistry. , 312-356 (2000).
- Ciurli, S., Sigel, A., Sigel, H., Sigel, R. K. O. . Nickel and its surprising impact in nature. 2, 241-278 (2007).
- Zambelli, B., Musiani, F., Benini, S., Ciurli, S. Chemistry of Ni2+ in urease: sensing, trafficking, and catalysis. Acc. Chem. Res. 44, 520-530 (2011).
- Zonia, L. E., Stebbins, N. E., Polacco, J. C. Essential role of urease in germination of nitrogen-limited Arabidopsis thaliana seeds. Plant Physiol. 107, 1097-1103 (1995).
- Follmer, C. Insights into the role and structure of plant ureases. Phytochemistry. 69, 18-28 (2008).
- Sumner, J. B. The isolation and crystallization of the enzyme urease. J. Biol. Chem. 69, 435-441 (1926).
- Krajewska, B. Ureases I. Functional, catalytic and kinetic properties: A review. J. Mol. Cat. B. 59, 9-21 (2009).
- Callahan, B. P., Yuan, Y., Wolfenden, R. The burden borne by urease. J. Am. Chem. Soc. 127, 10828-10829 (2005).
- Krajewska, B., van Eldik, R., Brindell, M. Temperature- and pressure-dependent stopped-flow kinetic studies of jack bean urease. Implications for the catalytic mechanism. J. Biol. Inorg. Chem. 17, 1123-1134 (2012).
- Hausinger, R. P., Karplus, P. A., Huber, R., Poulos, T., Wieghardt, K. . Handbook of Metalloproteins. , 867-879 (2001).
- Goldberg, R., Kishore, N., Lennen, R. Thermodynamic quantities for the ionization reactions of buffers. J. Phys. Chem. Ref. Data. 31, 231-370 (2002).
- Baumann, M. J., et al. Advantages of isothermal titration calorimetry for xylanase kinetics in comparison to chemical-reducing-end assays. Anal. Biochem. 410, 19-26 (2011).
- Noske, R., Cornelius, F., Clarke, R. J. Investigation of the enzymatic activity of the Na+,K+-ATPase via isothermal titration microcalorimetry. Biochim. Biophys. Acta. 1797, 1540-1545 (2010).
- Harmon, K. M., Niemann, C. The competitive inhibition of the urease-catalyzed hydrolysis of urea by phosphate. J. Biol. Chem. 177, 601-605 (1949).
- Benini, S., Rypniewski, W. R., Wilson, K. S., Ciurli, S., Mangani, S. Structure-based rationalization of urease inhibition by phosphate: novel insights into the enzyme mechanism. J. Biol. Inorg. Chem. 6, 778-790 (2001).
- Segel, I. H. . Enzyme kinetics: behavior and analysis of rapid equilibrium and steady-state enzyme systems. , (1993).
Nachdrucke und Genehmigungen
Genehmigung beantragen, um den Text oder die Abbildungen dieses JoVE-Artikels zu verwenden
Genehmigung beantragenThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Alle Rechte vorbehalten