このコンテンツを視聴するには、JoVE 購読が必要です。 サインイン又は無料トライアルを申し込む。
Method Article
細菌感染や解析のためのキイロショウジョウバエ幼虫血球の抽出
要約
このメソッドは、三次元 (3 D) モデルを昆虫細胞に病原体の侵入を視覚化する方法を示します。ショウジョウバエ幼虫から血液細胞は、細菌やウイルスの病原体、体外か体内に感染していた。感染した血球、固定され共焦点顕微鏡とそれに続く細胞三次元イメージングのステンド グラスします。
要約
ショウジョウバエ、病原菌の感染中に血球は感染で免疫応答に重要な役割を果たします。したがって、このプロトコルの目標は、ハエ、すなわち血液細胞の特定の免疫のコンパートメントの病原体の侵入を可視化する手法を開発することです。最大 3 × 10、ここで提示法を用いた6ライブ血球は、 ex vivo感染症のため 30 分で 200ショウジョウバエ3rd齢幼虫から入手できます。また、血球は感染した体内感染後 24 時間を追った血球抽出による 3rd幼虫の注射を通してできます。これらの感染した一次電池は修正され、ステンド グラス、共焦点顕微鏡を用いたイメージングします。その後、3 D 表現は、病原体の侵入を決定的に表示する画像から生成されました。また、qRT PCR のための質の高い RNA は mRNA 次の病原体の検出のため得ることができる感染症、および十分な蛋白質、西部のしみの分析のためのこれらの細胞から抽出されます。病原体の侵入の明確な和解と細菌やウイルスの病原体の種類と血球抽出のための効率的な方法を使用して、ショウジョウバエから十分なライブの血球を取得する感染症の確認法を提案する一緒に取られて、ex vivoとin vivoの感染実験の幼虫。
概要
キイロショウジョウバエは免疫1の研究の確立されたモデル生物です。生得の免疫反応の間に血液細胞は病原体の挑戦への応答に重要な役割を果たします。血液細胞は、ウイルス、真菌や細菌感染2,3貪食作用を通じて病原体との闘いにおける重要な機能を持っていることと同様、寄生虫をカプセル化するために重要です。
最高病原微生物感染ホストの生得の免疫反応を理解し、病原体が伝染の間に宿主細胞に侵入する方法を視覚化することが重要です。この可視化は、侵入のメカニズムの理解に貢献しています。細胞応答し病原体の細胞内局在性の詳細と、これらのデータは感染症と微生物の相互作用、細胞細胞器官に対する宿主の反応についての手がかりを提供できます。したがって、顕微鏡によるイメージング後 3次元モデルの再構成は、宿主細胞に病原体の正確な位置を決定することができます。本研究ではコクシエラ中 (c.burnetii)、 Q 熱、プライマリショウジョウバエ血球に人間や動物の健康に深刻な脅威をもたらす人畜伝染病疾患の病原の侵略を可視化。最近、ショウジョウバエが 2 の 9 マイル相 II (NMII) クローンのc.burnetiiの 4 種、この株はショウジョウバエ4、複製することが、そのを示すバイオ セーフティ レベルに敏感であることを示したショウジョウバエ c.burnetii病因を研究するモデル生物として使用できます。
以前の研究では、ホストの生得の免疫反応を調べる血球を使用しています。血球は、形態学的観察5,6,7, 形態計測学的解析2,8、貪食分析2,3, qRT PCR2に使用されています。,9, 免疫沈降10,11、免疫蛍光分析10,12, 免疫染色13, イムノブロット3,10、 11および免疫組織化学の9,14。ショウジョウバエS2 細胞が様々 な体外実験の利用も、不死化および既存ウイルス感染の可能性は、動作15,16を変更します。S2 細胞などの不死化細胞ラインではなく一次電池の使用は、自然免疫の研究システムで全体の生物の代表的な。また、血液細胞の生体内での抽出の前に感染症は、他のホスト蛋白質および組織前のヴィヴォ感染前に血液細胞の抽出上の優位性と対話するセルをできます。血液細胞の生きている8,17,18,19を保つために時間の短い期間での血球の十分な数を取得するさまざまな方法の番号を利用されています。
本研究ではショウジョウバエ3rd病原微生物感染c.burnetii、リステリア菌(リステリア菌) や無脊椎動物の虹色の幼虫から血球を抽出する手法を提案します。ウイルス 6 (IIV6)。体内と体外の両方の血球感染症法について述べる。生体内でおよび前のヴィヴォ-感染した血液細胞の共焦点顕微鏡による可視化し、, c.burnetii侵略の 3 D モデルを構築するために使用します。また前のヴィヴォの抽出のプロトコルを使用して-感染した血液細胞は遺伝子および蛋白質の表現のために使用されたアッセイ。具体的には、IIV6 とリステリア感染症の程度を確認する総 RNA やタンパク質が分離された qRT PCR または西部のしみの分析のための細胞から。一緒に取られて、プロトコル 3rd幼虫と体内または体外に主な血液細胞が感染している証拠から急速に血球数を収集するメソッドを提供します、適切なプラットフォームの微生物病原体感染症研究および適用の下流解析顕微鏡、トランスクリプトミクス、プロテオミクスなど。
Access restricted. Please log in or start a trial to view this content.
プロトコル
1 感染前のヴィヴォ。
- 媒体および装置
- 生殖不能の条件の下で新鮮なショウジョウバエ血球分離培地 (DHIM) 75% シュナイダーのショウジョウバエを含む準備中] 25% で胎仔ウシ血清 (FBS)、フィルター殺菌それ。
- 顕微鏡下で 10 cm × 10 cm パラフィン フィルムの 2 〜 3 個をレイヤーします。
- ガラス毛細管を準備します。キャピラリーの引き手のヒーターを最大の 55% に設定します。キャピラリー チューブを約 10 μ m の鋭いポイントに引き出します。
- バックフィル鉱油と毛細血管。
- Nanoinjector (図 1A) に充填キャピラリー チューブを組み立てるし、先端から鉗子 (図 1B) を破壊して融合キャピラリー チューブの先端を開きます。先端の外径は、血球の簡単取り込みのため 100 μ m をする必要があります。
- キャピラリー チューブの先端からできるだけ多くのオイルを取り出し DHIM と入力します。アップ撮影の体液と油の境界線の容易な視覚化のため油と DHIM の気泡が含まれます (図 1B')。
- 体液抽出
- ピックアップ 3rd令ショウジョウバエ幼虫内部から食品の壁が優しく鉗子を使用バイアルし、100 μ m ストレーナー (図 1C) にそれらを置きます。3rdの幼虫は、大人の女性が肥沃な卵を次の 3-6 日を発見されています。
注: これらの実験使用遺伝子型、w1118;P {w+ mC= Hml GAL4.Δ} 2、P {w+ mC= UA 2xEGFP} AH2、のでこれらの動物からの血液細胞表現強化された緑の蛍光蛋白質 (EGFP) 顕微鏡による細胞の同定を支援します。 - 幼虫を 5 mL の滅菌水を注ぐし、余分な水分を除去する 5 s. 位ワイプ タスク上にストレーナー ストレーナーを振る (図 1C')。
- 1.5 mL 遠心チューブに幼虫を移します。それらの 5 の CO2ガス麻酔 s (図 1D)。
- 背側 (図 2A) に直面するいると、実体顕微鏡下でパラフィン フィルム上に幼虫を配置します。
- 場所幼虫の後方のボディに軽くガラスキャピラリの場所でそれを保持し、後部のキューティクルを破壊は、良い先の尖った鉗子 (図 2B) を使用して開きます。
- パラフィン膜 (図 2C) に流れる体液を許可します。
- パラフィン フィルム上で一度に 20 50 幼虫から血液を含む体液のプールを作る。
- Nanoinjector (図 2D) に毛細管ガラスを使用してプールの体液を取る。
注: 体液の約 10-20 μ L 必要があります。 - DHIM (図 2E) 500 μ L を含む 1.5 mL 遠心チューブに体液を取り出します。
- 1.2.4 の手順を繰り返します) - 1.2.9) 幼虫の各バッチの。
- ピックアップ 3rd令ショウジョウバエ幼虫内部から食品の壁が優しく鉗子を使用バイアルし、100 μ m ストレーナー (図 1C) にそれらを置きます。3rdの幼虫は、大人の女性が肥沃な卵を次の 3-6 日を発見されています。
- 血液細胞の数をカウントします。
- 死んだ細胞を染色する 0.6 mL 遠心チューブに 0.4% トリパン ブルー溶液 5 μ L をピペットします。優しく DHIM と血球、ピペットを使用して 1.5 mL チューブを混在させる DHIM 0.6 mL 遠心チューブに血液細胞を含むの 5 μ L を転送、軽く混ぜます。
- トリパン ブルー: 血球の 1:1 混合物からの細胞の 10 μ L をピペットの検定に。
- ライブ血液細胞がないトリパン ブルーと、hemocytomter の 4 コーナー フィールドの各青染色、計算式を使用しての 1 ミリリットルあたり血液の濃度の数をカウントします。
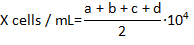
X; の 1 ミリリットルあたりのライブ血液の濃度は、a、b、c、および d は、検定でフィールドの各 4 (トリパン ブルー色素排除によって決定される) として生きているセルの数を数えます。細胞カウントの合計数の 2 で除算は、トリパン ブルー色素の細胞の 1:1 希釈によるものです。トリパン ブルー染色細胞は死んでいると見なされます。
-
前のヴィヴォ感染症
- 各実験に必要な縦長と生物学的に基づいたセルのシード処理する井戸の数をレプリケートを決定します。5.0 × 104血球が感染 RNA および蛋白質の浄化のために望ましい。
- DHIM で次の数式を使用して感染を希釈する病原体在庫量を計算します。
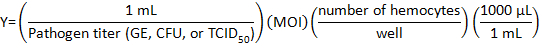
感染症 (MOI) の多様性は、ウイルスの数又は細菌セルあたり希望します。
注: 使用 MOI は個々 の実験、試金の実行に依存します。ここで、10 のゲノム同等 (GE)/ c.burnetii、リステリア菌の 10 CFU/セルまたは IIV6 の 1 の TCID50/cell のセルを使用しました。 - 24 ウェル プレートの各ウェルに DHIM 適量をチューブでウイルスや細菌のボリュームに追加することによって病原体の媒体の 500 μ L を準備します。
- 12 mm 丸いカバー ガラス (厚さ号 1) 24 ウェル プレートのウェルにします。
- 24 ウェル プレートのウェルに、血球を含む DHIM を分割します。
- よく血球に病原体の媒体の 500 μ L を追加します。
- 5 分間 1,000 x g でプレートを遠心します。
- 28 ° C で 1 時間のプレートを孵化させなさい15 分毎優しく後ろからプレートをフロント、し 5 の右に左に傾け手で s。
- H の後 1 侵略/添付ファイル手順 1.4.8 の)、優しくピペットの病原体中を洗い新鮮な DHIM と血球と新鮮な DHIM の 500 μ L でそれを補充します。
- 感染した血球を目的の時間にインキュベートします。これらの実験により, c.burnetii- または IIV6 に感染した血液細胞が 24 h とリステリアインキュベート-感染した血液細胞は、1、2、または 4 時間インキュベートします。
2体内感染。
- 感染症
- 暖かい室温 15 分板のショウジョウバエフルーツ ジュース寒天プレートは以前作られていますは、20をについて説明します。
- 寒天の 30 g を加える水のオートクレーブ 700 mL 40 分のためのそれ。
- 10 mL の無水エタノールにメチルパラベンの 0.5 g を溶解します。
- フルーツ ジュース濃縮液 300 mL に、メチル パラベン ソリューションを追加します。
- すぐにオートクレーブ寒天液に濃縮ジュースをミックスし、5 mL を 10 × 35 mm シャーレに分注します。
- プレートは、15 分間冷却後 4 ° C で保存します。
- 準備手順 1.2.1 3rd齢幼虫) - 1.2.3)。
- 寒天酵母ペーストを配置します。(図 3A) の乾燥を避けるために幼虫を移行できます寒天プレートに細かいカットを作る。パラフィン フィルム (図 3B C) を使用して鉗子をかざして 0.001 mm 先の尖ったタングステン針を組み立てます。
- 高価 mCherry の 50 μ L をピペット表現する-c.burnetii (5.95 × 109 GE/mL) パラフィンにステレオの顕微鏡の下で映画し、細菌のプールに幼虫を配置。
- 細菌のプールに幼虫を配置します。タングステン針 (図 3D) で幼虫を刺します。寒天プレート (図 3E) 上に幼虫を転送します。
- 寒天プレート上に残りの病原体媒質を転送し、パラフィン フィルム プレートをシールします。
- 希望の時間は感染後 (図 3F) までプレートに湿った空気中の幼虫を維持します。実験の結果, c.burnetii-感染した幼虫は 24 h のプレートに。
- 暖かい室温 15 分板のショウジョウバエフルーツ ジュース寒天プレートは以前作られていますは、20をについて説明します。
- 体液抽出と血球のめっき
- 準備中および次のステップ 1.1 装置)。
- 12 mm 丸いカバー ガラス (厚さ号 1) 24 ウェル プレートのウェルにします。井戸に DHIM の 500 μ L をピペットします。
- 体液から抽出手順 1.2.4 感染幼虫) - 1.2.8)。
- さて次に体液を取り出すステップ 2.2.2)。
- 2.2.3 を繰り返す) と 2.2.4) 幼虫の複数のバッチの。
- 5 分間 1,000 x g でプレートを遠心します。
3. 可視化
- 固定および汚損
- 井戸の丸いカバー ガラスで解決する血球を可能にした後軽く各ウェルからメディアを削除します。
- 優しく落ち着いた血球の各ウェルに 4% パラホルムアルデヒド (PFA) の 200 μ L を追加します。室温で 20 分間血球を孵化させなさい。
- 削除 4 %pfa と軽く 200 μ L の PBS の 0.1% を含むを追加トリトン X-100 と 1% ウシ血清アルブミン (BSA) を各ウェル。血球室温で 10 分間、インキュベートします。
- PBS を削除し、優しく 1 × 4', 6-diamidino-2-phenylindole (DAPI) を各ウェルの 200 μ L を追加します。血球室温で暗闇の中で 10 分間、インキュベートします。
- DAPI のソリューションを削除し、優しく PBS を各ウェルに追加します。室温で 5 分間血球を孵化させなさい。
- ガラス顕微鏡スライドに antifade 取り付け中の 10 μ L をドロップします。
- 各ウェルから PBS を除去した後細かい指摘の鉗子を用いた 24 ウェル プレートからカバー ガラスを削除します。スライド ガラスの antifade マウント媒体にカバーガラスを下向き血球とそっと置きます。
- 暗い一晩でそれを置くことによって乾燥するスライドを許可します。
- 共焦点レーザー顕微鏡
- DAPI、EGFP、および mCherry の 3 つのカラー イメージングの共焦点顕微鏡を構成します。次の設定を使用: DAPI の励起 (ex) 405 nm、発光 (em) 415 480 nm;488 ex EGFP nm、em 493 564 nm;587 ex mCherry nm、em 597-700 nm。
- 顕微鏡と 63 X を使用してサンプルを中心にサンプルを配置/1.4 開口数 (NA) の目的。イメージングのためのビューのフィールドで必要な血球を探します。
- サンプルの適切な露出を得るためにレーザー パワーと検出器ゲインを調整します。露出レベルはサンプル全体の厚さに適したように複数の z 平面を確認してください。
- 全血球の z 軸の上下の位置を見つけます。Z 区分のための開始および終了位置としてこれらの位置を設定します。
- 画像、血球を含む領域をスキャン ズームを使用のみです。3 X のズーム倍率が使われます。
- X − y 平面で 1024 x 1024 ピクセルなど適切な解像度で画像のシリーズと z 軸 0.3 μ m 間隔を収集します。
- 3次元モデルの再構成
注: オープン ソース ソフトウェアは、3次元モデルの再構成のための以下の機能の多くを実行する存在します。- Z 断面画像系ファイルを 3次元モデルの再構成のための共焦点顕微鏡に関連するソフトウェアにインポートします。
- 核染色 DAPI と血球を表現する、EGFP でc.burnetiiを表現する mCherry の共局在を示すセルを選択します。1 つのセルだけを格納する一連のイメージをトリミングします。
- ソフトウェアの事前包装されたアルゴリズムを使用して 3 D モデルを再構築 3 D ビューアーのオプションを選択します。ブレンド、表面、および混合のオプションの間での 3 D 表現の目的のタイプを選択します。この方法では血球, 核, とc.burnetiiはサーフェス モデルを使用して示されています。
- マウスのボタンを押し、画面の周りにカーソルをドラッグして様々 な視聴位置から 3 D 再構築されたセルを確認します。セル方向と、イメージを最適化するためにモデル化された光源の位置を調整します。不透明度、最小値と最大しきい値、鏡面、アンビエント、Shineness、およびガンマの他のオプションは、イメージを最適化するために存在します。
- 血球の内部の内容を視覚化するクリッピングし、断面のコマンドを使用してモデルを断面を取る。
4. 遺伝子/タンパク質分析用アプリケーション
- その後 qRT PCR のための細胞を溶解 IIV6 とリステリア菌、感染または西部のしみの分析は前述21次の製造元の指示をしました。
注: qRT PCR のためのプライマーおよび西部のしみのための抗体の材料表を参照してください。 - Agarose のゲルの電気泳動で増幅産物の適切な長さを確保する前述の22と PCR の製品を分析します。
Access restricted. Please log in or start a trial to view this content.
結果
収集ex vivo感染症、血球を 3 × 10 まで生きて6血球は 200ショウジョウバエ3rd幼虫から抽出されました。本手法を開発するには、さまざまなテクニックの数を行った。個々 の幼生解離 1.5 h までと 8000 ~ セルの平均を得たこの法18、その大半はコレクションの終わりまで生きてなかった。次に、我々 は、血液を含んだ体液を?...
Access restricted. Please log in or start a trial to view this content.
ディスカッション
宿主細胞が感染になる方法を理解、特に、以前テストされていない病原体および細胞型の組み合わせ4実験細胞、病原体の局在を明らかにすることが重要です。感染細胞反応カスケードを勉強して生産的な病原体の侵入を示すことができます、イメージングと細胞応答データの組み合わせ病原体の侵入、感染を示すことが欠かせません。一方、宿主細胞に病原体の侵入の 2D ?...
Access restricted. Please log in or start a trial to view this content.
開示事項
著者は、彼らは競合する金銭的な利益があることを宣言します。
謝辞
MCherry 表現コクシエラ ラベンダーの株を提供するため博士ロバート ・ Heinzen に感謝しております。博士ルイス ・ テイシェイラにありがとうはえの在庫を提供する無脊椎動物虹色ウイルス 6 およびブルーミントン ストック センターを提供します。このプロジェクトは、NIH グラント R00 AI106963 (A.G.G.) に、ワシントン州立大学によって一部で賄われていた。
Access restricted. Please log in or start a trial to view this content.
資料
| Name | Company | Catalog Number | Comments |
| Schneider's Drosophila Medium | Thermo Fisher Scientific (Gibco) | 21720024 | 1.1.1), 2.1.2) |
| Fetal Bovine Serum | GE Healthcare Life Sciences (HyClone) | SH30070.03HI | 1.1.1), 2.1.2) |
| Filter (0.22 µL) | RESTEK | 26158 | 1.1.1) |
| Strainer (100 µm) | Greiner bio-one | 542000 | 1.2.1), 2) |
| Stereo microscope | Amscope | SM-1BSZ-L6W | 1.2), 2) |
| Glass capillary | Fisher Scientific | 21-171-4 | 1.1), 1.2), 2) |
| Capillary puller | Narishige International USA, Inc. | PC-10 | 1.1.3) |
| Mineral oil | Snow River Products | 1.1.4) | |
| Nanoinjector | Drummond Scientific Company | 3-000-204 | 1.1), 1.2), 2.2) |
| Forceps | VWR | 82027-402 | 1.1.5), 1.2), 2), 3.1.7) |
| CO2 delivery apparatus | Genesee Scientific | 59-122BC | 1.2), 2) |
| Trypan Blue | Thermo Fisher Scientific (Gibco) | 15250061 | 1.3) |
| Hemocytometer | Hausser Scientific | 3100 | 1.3) |
| 24 well plate | Greiner bio-one | 662160 | 1.4), 2.2) |
| Coxiella burnetii - mCherry | Dr. Heinzen, R. | 1.4), 2.2) | |
| Drosophila fruit juice plates | Cold Spring Harbor Protocols | 2.1) http://cshprotocols.cshlp.org/content/2007/9/pdb.rec11113.full | |
| Agar | Fisher Bioreagents | BP1423-500 | 2.1.1.1) |
| Methyl paraben | Amresco | 0572-500G | 2.1.1.2) |
| Absolute ethanol | Fisher Bioreagents | BP2818-500 | 2.1.1.2) |
| Welch's 100% Grape juice frozen concentrate, 340 mL | Amazon | B0025UJVGM | 2.1.1.3) |
| Petri dishes, 10 x 35 mm | Fisher Scientific | 08-757-100A | 2.1.1.4) |
| Microscope cover glass | Fisher Scientific | 12-545-80 | 1.4.4), 2.2.2) |
| Yeast, Bakers Dried Active | MP Biomedicals | 0210140001 | 2.1) Add 2 parts of water to 1 part of yeast (v/v) |
| Tungsten needle | Fine Science Tools | 10130-20 | 2.1) |
| Holding forceps | VWR | HS8313 | 2.1) |
| Paraformaldehyde | Fisher Scientific | FLO4042-500 | 3.1.3) |
| Triton X-100 | Fisher Scientific | BP151-500 | 3.1.3) |
| Bovine Serum Albumin | Fisher Scientific | BP9706-100 | 3.1.3) |
| 4',6-diamidino-2-phenylindole | Thermo Fisher Scientific | 62247 | 3.1.4) |
| Antifade mounting medium | Thermo Fisher Scientific | P36930 | 3.1.6) |
| Confocal microsope | Leica | TCS SP8-X White Light Confocal Laser Scanning Microscope | 3.2) |
| 3D imaging reconstruction software | Leica | LASX with 3D visualization module | 3.3) |
| Microscope slides | Fisher Scientific | 12-552-3 | 3.1.6) |
| Invertebrate iridescent virus 6 (IIV6) | Dr. Teixeria, L. | 4) PLoS Biol, 6 (12), 2753-2763, doi: 10.1371/journal.pbio.1000002, (2008) | |
| Listeria monocytogenes | ATCC | strain: 10403S | 4) Listeria monocytogenes strain 10403S (Bishop and Hinrichs, 1987) was grown in Difco Brain-heart infusion (BHI) broth (BD Biosciences) containing 50 µg/ml streptomycin at 30 °C. |
| DNase I | Thermo Fisher Scientific(Invitrogen) | 18068015 | gDNA degradation |
| cDNA Synthesis Kit | Bio-Rad | 1708891 | cDNA synthesis |
| IIV6_193R_F | IDT | qRT-PCR, 5'- TCT TGT TTT CAG AAC CCC ATT -3' | |
| IIV6_193R_R | IDT | qRT-PCR, 5'- CAC GAA GAA TGA CCA CAA GG -3' | |
| RpII_qRTPCR_fwd | SIGMA-ALDRICH | qRT-PCR, 5'- GAA GCG TTT CTC CAA ACG -AG | |
| RpII_qRTPCR_rev | SIGMA-ALDRICH | qRT-PCR, 5'- TTG AGC GTA AGC ATC ACC -TG | |
| SYBR Green qRT-PCR reagent | Thermo Fisher Scientific | K0251, K0252, K0253 | qRT-PCR |
| Real-Time PCR System | Thermo Fisher Scientific | 4351107, 7500 Software v2.0 | qRT-PCR |
| Anti-Listeria monocytogenes antibody | abcam | ab35132 | Western blot |
| Anti-Actin antibody produced in rabbit | SIGMA-ALDRICH | A2066 | Western blot |
| Anti-Rabbit IgG (H+L), HRP Conjugate | Promega | W4011 | Western blot |
参考文献
- Hoffmann, J. A. The immune response of Drosophila. Nature. 426 (6962), 33-38 (2003).
- Regan, J. C., et al. Steroid hormone signaling is essential to regulate innate immune cells and fight bacterial infection in Drosophila. PLoS Pathog. 9 (10), 1003720(2013).
- Yano, T., et al. Autophagic control of listeria through intracellular innate immune recognition in Drosophila. Nat Immunol. 9 (8), 908-916 (2008).
- Bastos, R. G., Howard, Z. P., Hiroyasu, A., Goodman, A. G. Host and Bacterial Factors Control Susceptibility of Drosophila melanogaster to Coxiella burnetii Infection. Infect Immun. 85 (7), (2017).
- Kacsoh, B. Z., Schlenke, T. A. High hemocyte load is associated with increased resistance against parasitoids in Drosophila suzukii, a relative of D. melanogaster. PLoS One. 7 (4), 34721(2012).
- Tsuzuki, S., et al. Switching between humoral and cellular immune responses in Drosophila. is guided by the cytokine GBP. Nat Commun. 5, 4628(2014).
- Kurucz, E., et al. Definition of Drosophila. hemocyte subsets by cell-type specific antigens. Acta Biol Hung. 58, Suppl 95-111 (2007).
- Neyen, C., Bretscher, A. J., Binggeli, O., Lemaitre, B. Methods to study Drosophila immunity. Methods. 68 (1), 116-128 (2014).
- Arefin, B., et al. Apoptosis in Hemocytes Induces a Shift in Effector Mechanisms in the Drosophila. Immune System and Leads to a Pro-Inflammatory State. PLoS One. 10 (8), 0136593(2015).
- Rus, F., et al. Expression pattern of Filamin-240 in Drosophila blood cells. Gene Expr Patterns. 6 (8), 928-934 (2006).
- Kurucz, E., et al. Hemese, a hemocyte-specific transmembrane protein, affects the cellular immune response in Drosophila. P Natl Acad Sci USA. 100 (5), 2622-2627 (2003).
- Markus, R., et al. Sessile hemocytes as a hematopoietic compartment in Drosophila melanogaster. P Natl Acad Sci USA. 106 (12), 4805-4809 (2009).
- Bretscher, A. J., et al. The Nimrod transmembrane receptor Eater is required for hemocyte attachment to the sessile compartment in Drosophila melanogaster. Biol Open. 4 (3), 355-363 (2015).
- Zettervall, C. J., et al. A directed screen for genes involved in Drosophila blood cell activation. P Natl Acad Sci USA. 101 (39), 14192-14197 (2004).
- Flynt, A., Liu, N., Martin, R., Lai, E. C. Dicing of viral replication intermediates during silencing of latent Drosophila viruses. P Natl Acad Sci USA. 106 (13), 5270-5275 (2009).
- Jovel, J., Schneemann, A. Molecular characterization of Drosophila cells persistently infected with Flock House virus. Virology. 419 (1), 43-53 (2011).
- Stoepler, T. M., Castillo, J. C., Lill, J. T., Eleftherianos, I. A simple protocol for extracting hemocytes from wild caterpillars. J Vis Exp. (69), e4173(2012).
- Sampson, C. J., Williams, M. J. Protocol for ex vivo incubation of Drosophila primary post-embryonic haemocytes for real-time analyses. Methods Mol Biol. 827, 359-367 (2012).
- Nehme, N. T., et al. A model of bacterial intestinal infections in Drosophila melanogaster. PLoS Pathog. 3 (11), 173(2007).
- Drosophila fruit juice egg plates. Cold Spring Harbor Protocols. (9), pdb.rec11113 (2007).
- Ahlers, L. R., Bastos, R. G., Hiroyasu, A., Goodman, A. G. Invertebrate Iridescent Virus 6, a DNA Virus, Stimulates a Mammalian Innate Immune Response through RIG-I-Like Receptors. PLoS One. 11 (11), 0166088(2016).
- Lee, P. Y., Costumbrado, J., Hsu, C. Y., Kim, Y. H. Agarose gel electrophoresis for the separation of DNA fragments. J Vis Exp. (62), (2012).
- Petraki, S., Alexander, B., Bruckner, K. Assaying Blood Cell Populations of the Drosophila melanogaster Larva. J Vis Exp. (105), (2015).
- Figliozzi, R. W., Chen, F., Chi, A., Hsia, S. C. Using the inverse Poisson distribution to calculate multiplicity of infection and viral replication by a high-throughput fluorescent imaging system. Virol Sin. 31 (2), 180-183 (2016).
- Rizki, T. M., Rizki, R. M. Lamellocyte differentiation in Drosophila. larvae parasitized by Leptopilina. Dev Comp Immunol. 16 (2-3), 103-110 (1992).
- Markus, R., Kurucz, E., Rus, F., Ando, I. Sterile wounding is a minimal and sufficient trigger for a cellular immune response in Drosophila melanogaster. Immunol Lett. 101 (1), 108-111 (2005).
- McCormack, R., et al. Perforin-2 Protects Host Cells and Mice by Restricting the Vacuole to Cytosol Transitioning of a Bacterial Pathogen. Infect Immun. 84 (4), 1083-1091 (2016).
- Ozgen, A., et al. Construction and characterization of a recombinant invertebrate iridovirus. Virus Res. 189, 286-292 (2014).
- Jakob, N. J., Muller, K., Bahr, U., Darai, G. Analysis of the first complete DNA sequence of an invertebrate iridovirus: coding strategy of the genome of Chilo iridescent virus. Virology. 286 (1), 182-196 (2001).
- Ghigo, E., Colombo, M. I., Heinzen, R. A. The Coxiella burnetii parasitophorous vacuole. Adv Exp Med Biol. 984, 141-169 (2012).
- Liu, F., et al. Drosophila melanogaster prophenoloxidases respond inconsistently to Cu2+ and have different activity in vitro. Dev Comp Immunol. 36 (3), 619-628 (2012).
- De Gregorio, E., et al. An immune-responsive Serpin regulates the melanization cascade in Drosophila. Dev Cell. 3 (4), 581-592 (2002).
- Kari, B., et al. The raspberry Gene Is Involved in the Regulation of the Cellular Immune Response in Drosophila melanogaster. PLoS One. 11 (3), 0150910(2016).
- Wu, A. R., et al. Quantitative assessment of single-cell RNA-sequencing methods. Nat Methods. 11 (1), 41-46 (2014).
- Buettner, F., et al. Computational analysis of cell-to-cell heterogeneity in single-cell RNA-sequencing data reveals hidden subpopulations of cells. Nat Biotechnol. 33 (2), 155-160 (2015).
- Nevil, M., Bondra, E. R., Schulz, K. N., Kaplan, T., Harrison, M. M. Stable Binding of the Conserved Transcription Factor Grainy Head to its Target Genes Throughout Drosophila melanogaster Development. Genetics. 205 (2), 605-620 (2017).
- Yang, C. P., et al. Transcriptomes of lineage-specific Drosophila neuroblasts profiled by genetic targeting and robotic sorting. Development. 143 (3), 411-421 (2016).
- Jaitin, D. A., et al. Dissecting Immune Circuits by Linking CRISPR-Pooled Screens with Single-Cell RNA-Seq. Cell. 167 (7), 1883-1896 (2016).
- Karaiskos, N., et al. The Drosophila embryo at single-cell transcriptome resolution. Science. 358 (6360), 194-199 (2017).
Access restricted. Please log in or start a trial to view this content.
転載および許可
このJoVE論文のテキスト又は図を再利用するための許可を申請します
許可を申請さらに記事を探す
This article has been published
Video Coming Soon
Copyright © 2023 MyJoVE Corporation. All rights reserved