Un abonnement à JoVE est nécessaire pour voir ce contenu. Connectez-vous ou commencez votre essai gratuit.
Method Article
Extraction des hémocytes de larves de drosophile d’Infection microbienne et d’analyse
Dans cet article
Résumé
Cette méthode montre comment visualiser invasion de pathogènes dans les cellules d’insectes avec des modèles tridimensionnels (3D). Hémocytes de larves de drosophile étaient infectées par des pathogènes virus ou bactériens, ex vivo ou in vivo. Hémocytes infectés ont été ensuite fixe et teintés d’imagerie avec un microscope confocal et la reconstruction cellulaire 3D ultérieure.
Résumé
Au cours de l’infection pathogène de Drosophila melanogaster, hemocytes jouent un rôle important dans la réponse immunitaire tout au long de l’infection. Ainsi, l’objectif du présent protocole est de développer une méthode pour visualiser l’invasion d’agents pathogènes dans un compartiment spécifique immunitaire des mouches, à savoir les hémocytes. À l’aide de la méthode présentée ici, jusqu'à 3 × 106 hémocytes vivants peuvent provenir de 200 larves de stade Drosophila 3rd en 30 min pour infection ex vivo . Hémocytes peuvent également être infecté en vivo par injection 3rd des larves de stade suivi par extraction hémocytes d’infection après 24h. Ces cellules primaires infectés ont été fixés, colorés et imagés à l’aide de la microscopie confocale. Ensuite, des représentations 3D ont été générées à partir des images pour montrer définitivement invasion de pathogènes. En outre, ARN de haute qualité pour qRT-PCR peut être obtenue pour la détection des suivants d’ARNm pathogène infection et suffisamment de protéines peuvent être extraites de ces cellules pour analyse par Western blot. Ensemble, nous présentons une méthode pour la réconciliation définitive de l’invasion de l’agent pathogène et la confirmation de l’infection à l’aide de types d’agents pathogènes bactériens et viraux et une méthode efficace pour l’extraction de hémocytes d’obtenir assez hémocytes direct de drosophile larves d’expériences d’infection ex vivo et in vivo .
Introduction
Drosophila melanogaster est un organisme modèle bien établi pour l’étude de l’immunité innée1. Au cours de la réponse immunitaire innée, hemocytes jouent un rôle important dans la réponse au défi de l’agent pathogène. Hémocytes sont critiques pour encapsuler des parasites, comme ayant une fonction importante dans la lutte contre l’agent pathogène par l’action phagocytaire au cours de l’infection fongique, virale et bactérienne2,3.
Afin de mieux comprendre réponse immunitaire innée de l’hôte à l’infection microbienne pathogène, il est important de visualiser comment l’agent pathogène envahit les cellules de l’hôte au cours de l’infection. Cette visualisation contribue à la compréhension du mécanisme d’invasion. Ainsi que les détails de la localisation intracellulaire pathogène et la réponse cellulaire, ces données peuvent fournir des indices sur la réponse de l’hôte à l’infection et les organites cellulaires avec lequel interagit le microbe. Ainsi, reconstruction 3D modèle après l’imagerie par microscopie peut être utile pour déterminer l’emplacement précis d’agents pathogènes dans les cellules hôtes. Dans cette étude, nous avons visualisé l’invasion de Coxiella burnetii (c. burnetii), l’agent causal de la fièvre Q, une zoonose qui constitue une menace grave pour la santé humaine et animale, en primaires hémocytes de drosophile . Récemment, il a été démontré que les Drosophila sont susceptibles d’être du niveau de biosécurité 2 phase de Nine Mile II (NMII) cloner 4 souches de c. burnetii et que cette souche est capable de se répliquer dans drosophile4, indiquant que Drosophile peut être utilisé comme un organisme modèle pour étudier la pathogenèse de c. burnetii .
Des études antérieures ont utilisé les hémocytes pour étudier la réponse immunitaire innée chez l’hôte. Hémocytes ont été utilisés pour des observations morphologiques5,6,7, morphométriques analyse2,8, phagocytose analyse2,3, qRT-PCR2 , 9, immunoprécipitation10,11, analyse par immunofluorescence10,12, immunomarquage13, immunoblotting3,10, 11 et immunohistochimie9,14. Bien que les cellules de drosophile S2 sont également disponibles pour diverses expériences in vitro , immortalisation et infection virale préexistante potentielle changent leur comportement15,16. L’utilisation de cellules primaires par opposition à une lignée de cellules immortalisées, telles que les cellules S2, permet pour l’étude du système immunitaire inné dans un système plus représentative de l’ensemble de l’organisme. En outre, l’infection des hémocytes in vivo, avant l’extraction, permet aux cellules d’interagir avec d’autres protéines de l’hôte et le tissu, un avantage sur l’extraction de hemocytes avant l’ex vivo de l’infection. Un certain nombre de méthodes différentes ont été utilisé pour obtenir un nombre suffisant d’hémocytes dans un court laps de temps pour garder les hémocytes vivant8,17,18,19.
Dans cette étude, nous présentons une méthode pour extraire les hémocytes de drosophile 3rd stade larvaire pour infection microbienne pathogène avec c. burnetii, monocytogenes de Listeria (Listeria) ou invertébré irisé Virus 6 (IIV6). Nous décrivons les méthodes pour les infections hémocytes fois in vivo et ex vivo . In vivoet ex vivo-hémocytes infectés ont été visualisés à l’aide de la microscopie confocale et utilisées pour construire des modèles 3D de l’invasion de c. burnetii . En outre, en utilisant le protocole d’extraction, ex vivo-hémocytes infectées ont été utilisées pour l’expression génique et protéique des essais. Plus précisément, afin d’examiner l’étendue de l’infection avec IIV6 et Listeria, total ARN ou protéine a été isolé des cellules pour qRT-PCR ou analyse par Western blot. Pris ensemble, le protocole fournit des méthodes pour collecter rapidement un nombre élevé d’hémocytes de 3rd stade larvaire et les éléments de preuve que les hémocytes primaires, infecté soit in vivo ou ex vivo, sont une plate-forme appropriée pour microbienne études d’infection pathogène et des analyses en aval il y a lieu, comme la microscopie, transcriptomique et protéomique.
Access restricted. Please log in or start a trial to view this content.
Protocole
1. ex vivo de l’infection
- Moyens et équipements
- Dans des conditions stériles, préparer fraîches Drosophila hémocytes isolant moyen (DHIM) contenant de 75 % Schneider Drosophila moyen avec 25 % sérum bovin fœtal (SVF) et filtre à stérilisent.
- Couche 2-3 morceaux de pellicule de paraffine de 10 cm x 10 cm sous un stéréomicroscope.
- Préparer le capillaire de verre. Régler le chauffage extracteur capillaire à 55 % du maximum. Tirez le tube capillaire d’une pointe acérée d’environ 10 µm.
- Remblayer le capillaire à l’huile minérale.
- Assembler le tube capillaire rempli sur la nanoinjector (Figure 1A), puis ouvrez le bout de tube capillaire fusionnés en brisant l’extrémité avec une pince (Figure 1B). Le diamètre extérieur de la pointe devrait être de 100 µm pour une absorption facile des hémocytes.
- Éjecter autant de pétrole que possible de l’extrémité du tube capillaire, puis remplir avec Simon. Pour faciliter la visualisation de la frontière de l’hémolymphe aspiré vers le haut et l’huile, inclure une bulle d’air entre le pétrole et le DHIM (Figure 1B").
- Extraction de l’hémolymphe
- Choisissez 3rd stades de larves de drosophile par l’intérieur mur d’aliments fioles délicatement à l’aide de pinces et rangez-les dans un filtre à 100 µm (Figure 1C). 3rd stade larvaire se trouvent 3-6 jours après la ponte fertile par une femelle adulte.
Remarque : Ces expériences utilisé le génotype, w1118; P {w+ mC= Hml-GAL4.Δ} 2, P {w+ mC= SAMU-2xEGFP} AH2, puisque les hémocytes provenant de ces animaux expriment la protéine fluorescente verte améliorée (EGFP) pour faciliter l’identification des cellules au microscope. - Verser 5 mL d’eau stérile sur les larves et agiter la passoire pendant 5 s. Place la crépine sur la lingette spéciale enlever l’excès d’eau (Figure 1C').
- Transférer les larves dans un tube de microtubes de 1,5 mL. Les anesthésier avec CO2 gaz pendant 5 s (Figure 1D).
- Placez les larves sur la pellicule de paraffine sous le stéréomicroscope, face dorsale-vers le haut (Figure 2A).
- Lieu le verre capillaire légèrement sur le corps postérieur larvaire de maintenir en place et de perturber la cuticule postérieure ouvert à l’aide de fines pinces pointues (Figure 2B).
- Permettre l’hémolymphe s’écouler sur le film de paraffine (Figure 2C).
- Faire un bassin de l’hémolymphe, y compris les hémocytes de 20-50 larves à la fois sur le film de paraffine.
- Prenez hémolymphe regroupée en utilisant le verre capillaire sur le nanoinjector (Figure 2D).
Remarque : Il devrait y avoir environ 10-20 µL de l’hémolymphe. - Éjectez l’hémolymphe dans un tube de microtubes de 1,5 mL contenant 500 µL de DHIM (Figure 2E).
- Répétez les étapes 1.2.4) - 1.2.9) pour chaque lot de larves.
- Choisissez 3rd stades de larves de drosophile par l’intérieur mur d’aliments fioles délicatement à l’aide de pinces et rangez-les dans un filtre à 100 µm (Figure 1C). 3rd stade larvaire se trouvent 3-6 jours après la ponte fertile par une femelle adulte.
- Dénombrer les hémocytes.
- Pipetter 5 µL de solution de bleu de Trypan 0,4 % dans un tube de microcentrifuge de 0,6 mL de colorer les cellules mortes. Doucement, mélanger la DHIM et les hémocytes dans le tube de 1,5 mL à l’aide d’une pipette et transférer 5 µL de DHIM y compris les hémocytes dans le tube de microcentrifuge de 0,6 mL et mélanger doucement.
- Dans l’hémocytomètre, déposer 10µl de cellules à partir du mélange de bleu : hémocytes Trypan 1:1.
- Dénombrer les hémocytes vivants qui ne sont pas souillées avec Trypan blue dans chacun des domaines 4 coin de la hemocytomter et de calculer la concentration des hémocytes par millilitre, à l’aide de la formule :
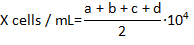
où X est la concentration de hemocytes direct par millilitre ; a, b, c et d sont le nombre de cellules vivantes (tel que déterminé par le bleu Trypan exclusion) dans chaque 4 des champs compté dans l’hémocytomètre. Division par 2 du nombre total de cellules comptées est due à la dilution de 1:1 des cellules avec le bleu Trypan. Les cellules colorées au Trypan bleus sont considérés comme morts.
-
Ex vivo Infections
- Déterminer le nombre de puits pour l’ensemencement avec cellules basées sur des intervalles et biologiques réplique nécessaire pour chaque expérience. 5,0 × 104 hémocytes sont souhaitables pour la purification d’ARN et de protéine après l’infection.
- Calculer le volume de stock pathogène à diluer avec Simon pour l’infection à l’aide de la formule suivante :
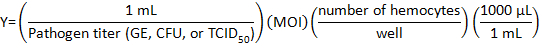
où la multiplicité d’infection (MOI) est le nombre de virus ou bactéries désiré par cellule.
Remarque : MOI utilisé dépend de l’expérience individuelle et les analyses exécutées. Ici, 10 génomes (GE) / c. burnetii, 10 UFC/cellule de Listeriaou 1 TCID50virus de IIV6 a été utilisé. - Préparer 500 µL de milieu pathogène pour chaque puits d’une plaque 24 puits en ajoutant le volume approprié de DHIM au volume viral ou bactériens dans un tube.
- Place un 12 mm ronde couvercle en verre (épaisseur n ° 1) dans un puits d’une plaque 24 puits.
- Diviser la DHIM y compris les hémocytes dans des puits d’une plaque 24 puits.
- Ajouter 500 µL de milieu pathogène à hémocytes dans un puits.
- Centrifuger la plaque à 1 000 x g pendant 5 min.
- Incuber la plaque pendant 1 h à 28 ° C. Toutes les 15 min, incliner délicatement la plaque de l’arrière à l’avant, puis de gauche à droite pour 5 s à la main.
- Après 1 h d’étape de l’invasion/pièces jointes 1.4.8), doucement Pipetter hors milieu pathogène laver les hémocytes avec DHIM fraîche et remplissez-le avec 500 µL de DHIM fraîche.
- Incuber les hémocytes infectés pendant le temps désiré. Dans ces expériences, c. burnetii- ou hémocytes IIV6-infectés sont incubées pendant 24h et Listeria-hémocytes infectés sont incubés pendant 1, 2 ou 4 h.
2. in vivo de l’infection
- Infection
- Chaud, la plaque de gélose de jus de fruit drosophile à température ambiante pendant 15 min. plaques sont faites comme précédemment décrit20.
- Ajouter 30 g d’agar dans 700 mL d’eau et stériliser pendant 40 min.
- Dissoudre 0,5 g de méthyl paraben dans 10 mL d’éthanol absolu.
- Ajouter la solution de méthyl paraben à 300 mL de concentré de jus de fruits.
- Mélanger le concentré de jus dans la solution d’agar autoclavé et diluer 5 mL dans 10 × 35 mm Pétri rapidement.
- Une fois que les plaques ont refroidi pendant 15 min, les stocker à 4 ° C.
- Préparer des larves de stade 3rd suivant étapes 1.2.1) - 1.2.3).
- Placer la pâte de levure sur une gélose. Faites une coupe fine dans la gélose où les larves peuvent migrer pour éviter le séchage (Figure 3A). Assembler une aiguille de pointes de tungstène de 0,001 mm avec holding pince à l’aide de paraffine film (Figure 3B, C).
- Distribuer 50 µL de haut-titre mCherry exprimant -c. burnetii (5,95 × 109 GE/mL) sur la paraffine du film sous le microscope stéréo et placer les larves dans le pool des bactéries.
- Placer les larves dans le pool des bactéries. Piquer les larves avec une aiguille de tungstène (Figure 3D). Transférer les larves sur une gélose (Figure 3E).
- Transférer le milieu pathogène restants sur la gélose et sceller la plaque avec le film de paraffine.
- Garder les larves sur la plaque dans l’air humide jusqu'à ce que l’infection après heure désirée (Figure 3-F). Dans cette expérience, c. burnetii-larves infectées sont sur la plaque pendant 24 h.
- Chaud, la plaque de gélose de jus de fruit drosophile à température ambiante pendant 15 min. plaques sont faites comme précédemment décrit20.
- Extraction de l’hémolymphe et électrodéposition d’hémocytes
- Préparer le support et l’équipement suivant étape 1.1).
- Place un 12 mm ronde couvercle en verre (épaisseur n ° 1) dans un puits d’une plaque 24 puits. Pipeter 500 µL de Simon dans le puits.
- Extrait l’hémolymphe des larves infectées suivant étapes 1.2.4) - 1.2.8).
- L’hémolymphe d’éjection dans la suite bien étape 2.2.2).
- 2.2.3 le répète) et 2.2.4) pour plusieurs lots de larves.
- Centrifuger la plaque à 1 000 x g pendant 5 min.
3. visualisation
- Fixation et coloration
- Après avoir laissé les hémocytes de s’installer sur le couvercle en verre rond dans le puits, retirez doucement le milieu de chaque puits.
- Doucement ajouter 200 µL de paraformaldéhyde à 4 % (PFA) dans chaque puits d’hémocytes sédentarisés. Incuber les hémocytes pendant 20 min à température ambiante.
- Enlever les 4 % PFA et doucement ajouter 200 µL de PBS contenant 0,1 % Triton X-100 et 1 % Bovine Serum Albumin (BSA) dans chaque puits. Incuber les hémocytes pendant 10 min à température ambiante.
- Retirez le PBS et doucement ajouter 200 µL de 1 × 4', 6-diamidino-2-phénylindole (DAPI) dans chaque puits. Incuber les hémocytes pendant 10 min à l’obscurité à température ambiante.
- Enlever la solution DAPI et ajouter doucement les PBS dans chaque puits. Incuber les hémocytes pendant 5 min à température ambiante.
- Déposer 10 µL de la milieu de montage antifade sur une lame de microscope.
- Après avoir retiré les PBS de chaque puits, retirer le couvercle en verre de la plaque de 24 puits à l’aide de pinces à pointe fine. Placez délicatement le couvercle en verre sur le support de montage antifade sur la lame de verre, avec les hémocytes vers le bas.
- Laisser la lame sécher en le plaçant dans la nuit sombre.
- Imagerie confocale
- Configurer le microscope confocal pour l’imagerie couleur trois DAPI EGFP et mCherry. Utilisez le paramètre suivant : excitation DAPI (ex) 405 nm, émission (em) 415-480 nm ; EGFP ex 488 nm, em 493-564 nm ; mCherry ex 587 nm, em 597-700 nm.
- Placez l’échantillon sur le microscope et l’accent mis sur l’échantillon à l’aide d’un 63 X / 1.4 objectif ouverture numérique (NA). Localiser les hémocytes souhaitées dans le champ de vision pour l’imagerie.
- Ajuster le laser détecteur de puissance et de gains pour réaliser une exposition appropriée de l’échantillon. Vérifier plusieurs z-avions pour assurer que le niveau d’exposition est approprié pour l’épaisseur de l’échantillon entier.
- Trouver la position haut et en bas sur l’axe z d’un hémocytes entiers. Définir ces postes comme les positions de début et de fin pour le z-sectionnement.
- Utilisez uniquement le balayage zoom d’image de la zone contenant les hémocytes. Les facteurs de zoom 3 X sont souvent utilisés.
- Recueillir les séries d’images à résolution appropriée, par exemple 1024 x 1024 pixels dans le plan x-y et 0,3 µm espacement dans la dimension z.
- Reconstruction du modèle 3D
Remarque : Logiciel Open source existe qui effectue la plupart des fonctions décrites ci-dessous pour la reconstruction du modèle 3D.- Importez le fichier de série z-sectionné image dans le logiciel associé à la microscopie confocale pour la reconstruction du modèle 3D.
- Sélectionnez une cellule présentant la co-localisation des noyaux colorées au DAPI et mCherry exprimant c. burnetii dans un EGFP exprimant les hémocytes. Récolte des séries d’images pour contenir uniquement la cellule unique.
- Sélectionnez l’option visionneuse 3D qui reconstitue le modèle 3D à l’aide d’algorithme pré-packagées du logiciel. Choisissez le type de représentation 3D parmi les options mixtes, Surface et mélange désiré. Dans cette méthode, hemocytes, noyaux et c. burnetii sont indiquées à l’aide de modèles de surface.
- Observer la cellule 3D reconstituée à partir de différentes positions de Regarde un en tenant le bouton de la souris et en faisant glisser le curseur autour de l’écran. Ajustez l’orientation de la cellule et la position de la source lumineuse modélisée pour optimiser l’image. Autres options pour l’opacité, Minimum et Maximum Threshold, spéculaire, Ambient, Shineness et Gamma existent pour optimiser l’image.
- Prendre des coupes à travers le modèle à l’aide de commandes de coupure et de sectionnement pour visualiser le contenu intérieur des hémocytes.
4. la demande d’analyse de gènes ou de protéines
- Suite à l’infection avec IIV6 et Listeria, lyser les cellules pour ultérieur qRT-PCR ou analyse par Western blot décrite précédemment21 et suivants du fabricant.
Remarque : Consultez la Table des matières pour les amorces pour qRT-PCR et les anticorps pour la tache occidentale. - Analyser les produits PCR par électrophorèse sur gel d’agarose, comme précédemment décrit22, pour s’assurer de la bonne longueur du produit amplifié.
Access restricted. Please log in or start a trial to view this content.
Résultats
Pour recueillir la hauteur hémocytes d’infection ex vivo , de 3 × 106 hémocytes ont été extraites de 200 larves de stade Drosophila 3rd . Pour développer notre méthode, un certain nombre de techniques différentes ont été tenté. Dissection de larve individuelle prendrait jusqu'à 1,5 h, et une moyenne de ~ 8000 cellules ont été obtenues à l’aide de cette méthode18, dont la plupart ne vivait pas avant la fin ...
Access restricted. Please log in or start a trial to view this content.
Discussion
Pour mieux comprendre comment sont infectent les cellules de l’hôte, il est important de préciser la localisation de l’agent pathogène dans les cellules, surtout quand l’expérimentation sur les pathogènes préalablement testés et cellule type combinaisons4. Alors qu’étudie la cascade de réaction cellulaire suite à une infection peut indiquer l’invasion pathogène productive, la combinaison des données de réponse cellulaire et d’imagerie est essentielle pour démontrer l’in...
Access restricted. Please log in or start a trial to view this content.
Déclarations de divulgation
Les auteurs déclarent qu’ils n’ont aucun intérêt financier concurrentes.
Remerciements
Nous sommes reconnaissants à m. Robert Heinzen pour fournir des stocks d’exprimant le mCherry Coxiella burnetii. Nous remercions le Dr Luis Teixeira prévoyant la fourniture stocks de mouche de virus iridescent d’invertébrés 6 et le centre de Stock de Bloomington. Ce projet a été financé en partie par les NIH grant R00 AI106963 (à A.G.G.) et de la Washington State University.
Access restricted. Please log in or start a trial to view this content.
matériels
| Name | Company | Catalog Number | Comments |
| Schneider's Drosophila Medium | Thermo Fisher Scientific (Gibco) | 21720024 | 1.1.1), 2.1.2) |
| Fetal Bovine Serum | GE Healthcare Life Sciences (HyClone) | SH30070.03HI | 1.1.1), 2.1.2) |
| Filter (0.22 µL) | RESTEK | 26158 | 1.1.1) |
| Strainer (100 µm) | Greiner bio-one | 542000 | 1.2.1), 2) |
| Stereo microscope | Amscope | SM-1BSZ-L6W | 1.2), 2) |
| Glass capillary | Fisher Scientific | 21-171-4 | 1.1), 1.2), 2) |
| Capillary puller | Narishige International USA, Inc. | PC-10 | 1.1.3) |
| Mineral oil | Snow River Products | 1.1.4) | |
| Nanoinjector | Drummond Scientific Company | 3-000-204 | 1.1), 1.2), 2.2) |
| Forceps | VWR | 82027-402 | 1.1.5), 1.2), 2), 3.1.7) |
| CO2 delivery apparatus | Genesee Scientific | 59-122BC | 1.2), 2) |
| Trypan Blue | Thermo Fisher Scientific (Gibco) | 15250061 | 1.3) |
| Hemocytometer | Hausser Scientific | 3100 | 1.3) |
| 24 well plate | Greiner bio-one | 662160 | 1.4), 2.2) |
| Coxiella burnetii - mCherry | Dr. Heinzen, R. | 1.4), 2.2) | |
| Drosophila fruit juice plates | Cold Spring Harbor Protocols | 2.1) http://cshprotocols.cshlp.org/content/2007/9/pdb.rec11113.full | |
| Agar | Fisher Bioreagents | BP1423-500 | 2.1.1.1) |
| Methyl paraben | Amresco | 0572-500G | 2.1.1.2) |
| Absolute ethanol | Fisher Bioreagents | BP2818-500 | 2.1.1.2) |
| Welch's 100% Grape juice frozen concentrate, 340 mL | Amazon | B0025UJVGM | 2.1.1.3) |
| Petri dishes, 10 x 35 mm | Fisher Scientific | 08-757-100A | 2.1.1.4) |
| Microscope cover glass | Fisher Scientific | 12-545-80 | 1.4.4), 2.2.2) |
| Yeast, Bakers Dried Active | MP Biomedicals | 0210140001 | 2.1) Add 2 parts of water to 1 part of yeast (v/v) |
| Tungsten needle | Fine Science Tools | 10130-20 | 2.1) |
| Holding forceps | VWR | HS8313 | 2.1) |
| Paraformaldehyde | Fisher Scientific | FLO4042-500 | 3.1.3) |
| Triton X-100 | Fisher Scientific | BP151-500 | 3.1.3) |
| Bovine Serum Albumin | Fisher Scientific | BP9706-100 | 3.1.3) |
| 4',6-diamidino-2-phenylindole | Thermo Fisher Scientific | 62247 | 3.1.4) |
| Antifade mounting medium | Thermo Fisher Scientific | P36930 | 3.1.6) |
| Confocal microsope | Leica | TCS SP8-X White Light Confocal Laser Scanning Microscope | 3.2) |
| 3D imaging reconstruction software | Leica | LASX with 3D visualization module | 3.3) |
| Microscope slides | Fisher Scientific | 12-552-3 | 3.1.6) |
| Invertebrate iridescent virus 6 (IIV6) | Dr. Teixeria, L. | 4) PLoS Biol, 6 (12), 2753-2763, doi: 10.1371/journal.pbio.1000002, (2008) | |
| Listeria monocytogenes | ATCC | strain: 10403S | 4) Listeria monocytogenes strain 10403S (Bishop and Hinrichs, 1987) was grown in Difco Brain-heart infusion (BHI) broth (BD Biosciences) containing 50 µg/ml streptomycin at 30 °C. |
| DNase I | Thermo Fisher Scientific(Invitrogen) | 18068015 | gDNA degradation |
| cDNA Synthesis Kit | Bio-Rad | 1708891 | cDNA synthesis |
| IIV6_193R_F | IDT | qRT-PCR, 5'- TCT TGT TTT CAG AAC CCC ATT -3' | |
| IIV6_193R_R | IDT | qRT-PCR, 5'- CAC GAA GAA TGA CCA CAA GG -3' | |
| RpII_qRTPCR_fwd | SIGMA-ALDRICH | qRT-PCR, 5'- GAA GCG TTT CTC CAA ACG -AG | |
| RpII_qRTPCR_rev | SIGMA-ALDRICH | qRT-PCR, 5'- TTG AGC GTA AGC ATC ACC -TG | |
| SYBR Green qRT-PCR reagent | Thermo Fisher Scientific | K0251, K0252, K0253 | qRT-PCR |
| Real-Time PCR System | Thermo Fisher Scientific | 4351107, 7500 Software v2.0 | qRT-PCR |
| Anti-Listeria monocytogenes antibody | abcam | ab35132 | Western blot |
| Anti-Actin antibody produced in rabbit | SIGMA-ALDRICH | A2066 | Western blot |
| Anti-Rabbit IgG (H+L), HRP Conjugate | Promega | W4011 | Western blot |
Références
- Hoffmann, J. A. The immune response of Drosophila. Nature. 426 (6962), 33-38 (2003).
- Regan, J. C., et al. Steroid hormone signaling is essential to regulate innate immune cells and fight bacterial infection in Drosophila. PLoS Pathog. 9 (10), 1003720(2013).
- Yano, T., et al. Autophagic control of listeria through intracellular innate immune recognition in Drosophila. Nat Immunol. 9 (8), 908-916 (2008).
- Bastos, R. G., Howard, Z. P., Hiroyasu, A., Goodman, A. G. Host and Bacterial Factors Control Susceptibility of Drosophila melanogaster to Coxiella burnetii Infection. Infect Immun. 85 (7), (2017).
- Kacsoh, B. Z., Schlenke, T. A. High hemocyte load is associated with increased resistance against parasitoids in Drosophila suzukii, a relative of D. melanogaster. PLoS One. 7 (4), 34721(2012).
- Tsuzuki, S., et al. Switching between humoral and cellular immune responses in Drosophila. is guided by the cytokine GBP. Nat Commun. 5, 4628(2014).
- Kurucz, E., et al. Definition of Drosophila. hemocyte subsets by cell-type specific antigens. Acta Biol Hung. 58, Suppl 95-111 (2007).
- Neyen, C., Bretscher, A. J., Binggeli, O., Lemaitre, B. Methods to study Drosophila immunity. Methods. 68 (1), 116-128 (2014).
- Arefin, B., et al. Apoptosis in Hemocytes Induces a Shift in Effector Mechanisms in the Drosophila. Immune System and Leads to a Pro-Inflammatory State. PLoS One. 10 (8), 0136593(2015).
- Rus, F., et al. Expression pattern of Filamin-240 in Drosophila blood cells. Gene Expr Patterns. 6 (8), 928-934 (2006).
- Kurucz, E., et al. Hemese, a hemocyte-specific transmembrane protein, affects the cellular immune response in Drosophila. P Natl Acad Sci USA. 100 (5), 2622-2627 (2003).
- Markus, R., et al. Sessile hemocytes as a hematopoietic compartment in Drosophila melanogaster. P Natl Acad Sci USA. 106 (12), 4805-4809 (2009).
- Bretscher, A. J., et al. The Nimrod transmembrane receptor Eater is required for hemocyte attachment to the sessile compartment in Drosophila melanogaster. Biol Open. 4 (3), 355-363 (2015).
- Zettervall, C. J., et al. A directed screen for genes involved in Drosophila blood cell activation. P Natl Acad Sci USA. 101 (39), 14192-14197 (2004).
- Flynt, A., Liu, N., Martin, R., Lai, E. C. Dicing of viral replication intermediates during silencing of latent Drosophila viruses. P Natl Acad Sci USA. 106 (13), 5270-5275 (2009).
- Jovel, J., Schneemann, A. Molecular characterization of Drosophila cells persistently infected with Flock House virus. Virology. 419 (1), 43-53 (2011).
- Stoepler, T. M., Castillo, J. C., Lill, J. T., Eleftherianos, I. A simple protocol for extracting hemocytes from wild caterpillars. J Vis Exp. (69), e4173(2012).
- Sampson, C. J., Williams, M. J. Protocol for ex vivo incubation of Drosophila primary post-embryonic haemocytes for real-time analyses. Methods Mol Biol. 827, 359-367 (2012).
- Nehme, N. T., et al. A model of bacterial intestinal infections in Drosophila melanogaster. PLoS Pathog. 3 (11), 173(2007).
- Drosophila fruit juice egg plates. Cold Spring Harbor Protocols. (9), pdb.rec11113 (2007).
- Ahlers, L. R., Bastos, R. G., Hiroyasu, A., Goodman, A. G. Invertebrate Iridescent Virus 6, a DNA Virus, Stimulates a Mammalian Innate Immune Response through RIG-I-Like Receptors. PLoS One. 11 (11), 0166088(2016).
- Lee, P. Y., Costumbrado, J., Hsu, C. Y., Kim, Y. H. Agarose gel electrophoresis for the separation of DNA fragments. J Vis Exp. (62), (2012).
- Petraki, S., Alexander, B., Bruckner, K. Assaying Blood Cell Populations of the Drosophila melanogaster Larva. J Vis Exp. (105), (2015).
- Figliozzi, R. W., Chen, F., Chi, A., Hsia, S. C. Using the inverse Poisson distribution to calculate multiplicity of infection and viral replication by a high-throughput fluorescent imaging system. Virol Sin. 31 (2), 180-183 (2016).
- Rizki, T. M., Rizki, R. M. Lamellocyte differentiation in Drosophila. larvae parasitized by Leptopilina. Dev Comp Immunol. 16 (2-3), 103-110 (1992).
- Markus, R., Kurucz, E., Rus, F., Ando, I. Sterile wounding is a minimal and sufficient trigger for a cellular immune response in Drosophila melanogaster. Immunol Lett. 101 (1), 108-111 (2005).
- McCormack, R., et al. Perforin-2 Protects Host Cells and Mice by Restricting the Vacuole to Cytosol Transitioning of a Bacterial Pathogen. Infect Immun. 84 (4), 1083-1091 (2016).
- Ozgen, A., et al. Construction and characterization of a recombinant invertebrate iridovirus. Virus Res. 189, 286-292 (2014).
- Jakob, N. J., Muller, K., Bahr, U., Darai, G. Analysis of the first complete DNA sequence of an invertebrate iridovirus: coding strategy of the genome of Chilo iridescent virus. Virology. 286 (1), 182-196 (2001).
- Ghigo, E., Colombo, M. I., Heinzen, R. A. The Coxiella burnetii parasitophorous vacuole. Adv Exp Med Biol. 984, 141-169 (2012).
- Liu, F., et al. Drosophila melanogaster prophenoloxidases respond inconsistently to Cu2+ and have different activity in vitro. Dev Comp Immunol. 36 (3), 619-628 (2012).
- De Gregorio, E., et al. An immune-responsive Serpin regulates the melanization cascade in Drosophila. Dev Cell. 3 (4), 581-592 (2002).
- Kari, B., et al. The raspberry Gene Is Involved in the Regulation of the Cellular Immune Response in Drosophila melanogaster. PLoS One. 11 (3), 0150910(2016).
- Wu, A. R., et al. Quantitative assessment of single-cell RNA-sequencing methods. Nat Methods. 11 (1), 41-46 (2014).
- Buettner, F., et al. Computational analysis of cell-to-cell heterogeneity in single-cell RNA-sequencing data reveals hidden subpopulations of cells. Nat Biotechnol. 33 (2), 155-160 (2015).
- Nevil, M., Bondra, E. R., Schulz, K. N., Kaplan, T., Harrison, M. M. Stable Binding of the Conserved Transcription Factor Grainy Head to its Target Genes Throughout Drosophila melanogaster Development. Genetics. 205 (2), 605-620 (2017).
- Yang, C. P., et al. Transcriptomes of lineage-specific Drosophila neuroblasts profiled by genetic targeting and robotic sorting. Development. 143 (3), 411-421 (2016).
- Jaitin, D. A., et al. Dissecting Immune Circuits by Linking CRISPR-Pooled Screens with Single-Cell RNA-Seq. Cell. 167 (7), 1883-1896 (2016).
- Karaiskos, N., et al. The Drosophila embryo at single-cell transcriptome resolution. Science. 358 (6360), 194-199 (2017).
Access restricted. Please log in or start a trial to view this content.
Réimpressions et Autorisations
Demande d’autorisation pour utiliser le texte ou les figures de cet article JoVE
Demande d’autorisationThis article has been published
Video Coming Soon