Для просмотра этого контента требуется подписка на Jove Войдите в систему или начните бесплатную пробную версию.
Method Article
Извлечение Hemocytes из личинки Drosophila melanogaster микробной инфекции и анализа
В этой статье
Резюме
Этот метод демонстрируется визуализировать возбудителя вторжения в насекомое клетки с трехмерной (3D) модели. Hemocytes от дрозофилы личинки были инфицированы вирусных или бактериальных патогенов, ex vivo или в естественных условиях. Зараженных hemocytes были затем фиксированной и витражи для изображений с конфокального микроскопа и последующих клеточных 3D реконструкции.
Аннотация
Во время патогенной инфекции Drosophila melanogaster, hemocytes играют важную роль в иммунной реакции на протяжении инфекции. Таким образом цель настоящего Протокола заключается в разработке метода для визуализации возбудителя вторжения в конкретных иммунной отсеке мух, а именно hemocytes. С помощью метода, представленные здесь, до 3 × 106 живой hemocytes можно получить из 200 дрозофила 3rd instar личинок в 30 минут для ex vivo инфекции. Кроме того hemocytes может быть зараженных в естественных условиях путем инъекций личинок 3rd instar следуют кровяные добычи 24 ч после инфекции. Эти зараженные клетки первичной были исправлены, витражи и образы с помощью конфокальной микроскопии. Затем 3D представлений были созданы из изображений, чтобы окончательно показать возбудителя вторжения. Кроме того, можно получить РНК высокого качества для qRT ПЦР для выявления возбудителя мРНК следующие инфекции и достаточно белка могут быть извлечены из этих клеток для Западный анализ помаркой. Взятые вместе, мы представляем метод определенное согласование возбудителя вторжения и подтверждения инфекции, используя типы бактериальных и вирусных патогенов и эффективный метод для извлечения кровяные для получения достаточно живой hemocytes от дрозофилы Личинки для ex vivo , так и в естественных условиях инфекции экспериментов.
Введение
Drosophila melanogaster это организм устоявшихся моделей для изучения врожденного иммунитета1. Во время врожденный иммунный ответ hemocytes играют важную роль в ответ на вызов возбудителя. Hemocytes являются критическими для инкапсуляции паразитов, а также важную роль в борьбе против возбудителя через фагоцитарной действий во время грибковые, вирусные и бактериальные инфекции2,3.
Для того чтобы лучше понять врожденный иммунный ответ хозяина патогенные микробные инфекции, важно для визуализации, как возбудитель поражает клетки хозяина во время инфекции. Эта визуализация способствует пониманию механизма вторжения. Вместе с детали внутриклеточной локализации возбудителя и клеточного ответа эти данные могут предоставить подсказки о принимающей реакции на инфекции и клеточных органелл, с которыми взаимодействует микроба. Таким образом 3D-модель восстановления после съемки по микроскопии может быть полезно определить точное местоположение патогенов в клетки хозяина. В этом исследовании мы визуализирована вторжения Coxiella burnetii (C. burnetii), возбудитель Ку-лихорадки, зоонозные болезни, которая представляет собой серьезную угрозу для здоровья человека и животных, в первичной Drosophila hemocytes. Недавно было продемонстрировано, что дрозофилы восприимчивы к биобезопасности уровня 2 9 км фаза II (NMII) клон 4 штамм C. burnetii и что этот штамм имеет возможность реплицировать в дрозофилы4, указанием что Дрозофила может использоваться в качестве модельного организма для изучения патогенеза C. burnetii .
Предыдущие исследования использовали hemocytes для изучения врожденный иммунный ответ хозяина. Hemocytes были использованы для морфологических замечания5,6,7, морфометрический анализ2,8, фагоцитоз анализ2,3, qRT ПЦР2 , 9, иммунопреципитация10,11, immunofluorescent анализ10,12, иммуноокрашивания13,, immunoblotting3,10 11 и иммуногистохимии9,14. Хотя дрозофилы S2 клетки также доступны для различных экспериментов в пробирке , увековечении и потенциальных существующей вирусной инфекции изменить их поведение15,16. Использование первичных элементов увековечен клеток линии, таких как S2 клетки, в отличие от позволяет для изучения врожденной иммунной функции в системе более представителя всего организма. Кроме того инфекции hemocytes в естественных условиях, до извлечения, позволяет клетки взаимодействуют с другими принимающей белков и ткани, преимущество над добычей hemocytes до ex vivo инфекции. Получить достаточное количество hemocytes в течение короткого времени, чтобы сохранить hemocytes жив8,,1718,19были использованы несколько различных методов.
В этом исследовании мы представляем метод для извлечения hemocytes из дрозофила 3rd instar личинки патогенные микробные инфекции с C. burnetii, листерий (листериоз) или беспозвоночных радужные вирус 6 (IIV6). Мы описываем методы как в естественных условиях , так и ex vivo кровяные инфекций. В естественных условиях- и ex vivo-зараженных hemocytes были визуализируется с помощью конфокальной микроскопии и используется для создания 3D-моделей C. burnetii вторжения. Кроме того, используя протокол извлечения, ex vivo-зараженных hemocytes были использованы для выражения генов и белков анализов. В частности изучить масштабы инфицирования с IIV6 и Listeria, всего RNA или протеина был изолирован от клеток для qRT ПЦР или Западный анализ помаркой. Взятые вместе, протокол предоставляет методы быстро собрать большое количество hemocytes от 3rd instar личинок и доказательства того, что основной hemocytes, инфицированных в vivo или ex vivo, являются подходящей платформой для микробной возбудитель инфекции исследования и применимым течению анализы микроскопии, transcriptomics и протеомики.
Access restricted. Please log in or start a trial to view this content.
протокол
1. ex vivo инфекции
- Средний и оборудование
- В стерильных условиях, подготовить Свежая дрозофилы кровяные изоляции среднего (DHIM) содержащие 75% Шнайдер дрозофилы средних с 25% плода Bovine сыворотки (ФБС) и фильтр стерилизовать его.
- Слой 2-3 куска пленки парафин 10 см х 10 см под стереомикроскопом.
- Подготовьте стеклянный капилляр. Установите нагреватель капиллярного съемник до 55% от максимума. Вытяните капиллярной трубки острием приблизительно 10 мкм.
- Засыпки капилляра с минеральным маслом.
- Соберите заполненные капилляр на nanoinjector (рис. 1А) и открыть плавленого капилляр наконечник, облом кончик с щипцами (рис. 1Б). Наружный диаметр кончика должно быть 100 мкм для легко поглощения hemocytes.
- Извлечь столько нефти как можно дальше от кончика капиллярной трубки, а затем заполнить с DHIM. Для легкой визуализации границы до получения гемолимфа и масла, включают пузырек воздуха между нефтью и DHIM (рис. 1Б').
- Гемолимфа добыча
- Выберите 3rd Инстар дрозофилы личинки изнутри стена пищи флакон осторожно с помощью щипцов и поместите их в 100 мкм фильтр (рис. 1C). 3rd instar личинки находятся 3-6 дней после плодородные откладки взрослой самки.
Примечание: Эти эксперименты используемые генотип, w1118; P {w+ mC= Hml-GAL4.Δ} 2, P {w+ mC= бла 2xEGFP} AH2, так как hemocytes от этих животных Экспресс расширенной зеленого флуоресцентного белка (EGFP) для оказания помощи в идентификации клеток при микроскопии. - Залить 5 мл стерильной воды личинки и встряхните сетчатый фильтр для 5 s. место сетчатый фильтр на задачи очистки, чтобы удалить излишки воды (рис. 1C').
- Переноса личинок в пробки microcentrifuge 1,5 мл. Анестезировать их с газом CO2 для 5 s (рис. 1D).
- Место личинки на парафин фильм под стереомикроскопом, с спинной стороне, обращенной вверх(рис. 2).
- Место стеклянный капилляр слегка на личиночной задней тела чтобы удерживать его на месте и сорвать задняя кутикулы открыть, с помощью тонкой острые щипцы (рис. 2B).
- Разрешить гемолимфа поступать на фильм парафина (рис. 2C).
- Чтобы пул гемолимфа, включая hemocytes от 20-50 личинок одновременно на фильм парафина.
- Занять до пула гемолимфа, используя стекло капилляров на nanoinjector (рис. 2D).
Примечание: Там должно быть примерно 10-20 мкл гемолимфа. - Извлечь гемолимфа в 1,5 мл microcentrifuge трубка, содержащая 500 мкл DHIM (2 рисунокE).
- Повторите шаги 1.2.4) - 1.2.9) для каждого пакета личинок.
- Выберите 3rd Инстар дрозофилы личинки изнутри стена пищи флакон осторожно с помощью щипцов и поместите их в 100 мкм фильтр (рис. 1C). 3rd instar личинки находятся 3-6 дней после плодородные откладки взрослой самки.
- Подсчитать количество hemocytes.
- Пипетка 5 мкл раствора Трипановый синий 0,4% в пробки microcentrifuge 0,6 мл пятно мертвые клетки. Осторожно смешать DHIM и hemocytes в 1,5 мл трубку с помощью пипетки и передача 5 мкл DHIM, включая hemocytes пробки microcentrifuge 0,6 мл и осторожно перемешать.
- Пипетка 10 мкл клеток от 1:1 смесь Трипановый синий: кровяные в Горяева.
- Подсчитать количество живой hemocytes, которые не запятнаны с Трипановый синий в каждом из 4 угла поля hemocytomter и рассчитать концентрацию hemocytes на миллилитр, используя формулу:
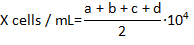
где X — концентрация живой hemocytes на миллилитр; а, b, c и d являются количество живых клеток (как определено исключение Трипановый синий) в каждом 4 полей учитываются в Горяева. Деление на 2 общего числа клеток насчитал объясняется разбавления 1:1 клеток с Трипановый синий. Клетки окрашивали Трипановый синий считается мертвым.
-
Ex vivo Инфекции
- Определите количество скважин для заполнения с клетками, основанные на timepoints и биологического реплицирует необходимые для каждого эксперимента. 5,0 × 104 hemocytes желательны для очистки РНК и белка, после инфицирования.
- Рассчитайте объем запасов возбудителя быть разведен с DHIM для инфекции, с использованием следующей формулы:
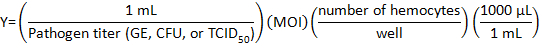
где обилие инфекции (мои) — это количество вирусных или желаемого бактерии в клетку.
Примечание: MOI используется зависит от индивидуальных эксперимент и выполненных анализов. Здесь, 10 генома эквиваленты (GE) / клеток C. burnetii, 10 кое/ячейки листерийили 1 TCID50/cell от IIV6 было использовано. - Подготовьте 500 мкл возбудителя среднего для каждой скважины 24-ну плиты, добавив надлежащего объема DHIM вирусной или бактериальной громкости в трубке.
- Место 12 мм круглые Стекло покровное (№ 1 толщина) в хорошо 24-ну плиты.
- Разделение DHIM, включая hemocytes в скважины 24-ну плиты.
- Добавьте 500 мкл возбудителя среднего hemocytes в колодец.
- Центрифуга пластину на 1000 x g за 5 мин.
- Инкубировать пластину за 1 час на 28 ° C. Каждые 15 мин, нежно наклона пластины от задней стойки, а затем слева направо для 5 s вручную.
- После 1 h вторжения/вложение шаг 1.4.8) аккуратно Пипетка от возбудителя среднего и мыть hemocytes с свежими DHIM и пополнить его с 500 мкл свежих DHIM.
- Инкубируйте зараженных hemocytes на требуемое время. В этих экспериментах, C. burnetii- или IIV6-инфицированных hemocytes инкубируют 24 h, и Listeria-зараженных hemocytes инкубируют для 1, 2 или 4 h.
2. в естественных условиях инфекции
- Инфекции
- Теплый, агар сок дрозофилы фрукты при комнатной температуре 15 мин пластины сделаны ранее описал20.
- Добавьте 30 г агара 700 мл воды и автоклав для 40 мин.
- Растворите 0,5 г метил paraben в 10 мл абсолютного этанола.
- Метил парабена решение добавьте 300 мл фруктовый сок.
- Быстро смешать сок концентрат в раствор ячеистого агар и распределить 5 мл на 10 × 35 мм Петри.
- После того, как пластины остыли 15 мин, храните их на 4 ° C.
- Подготовка 3rd instar личинки следующие шаги 1.2.1) - 1.2.3).
- Место вставки дрожжей на плите агар. Сделайте прекрасный разрез в пластину агар, где личинки можно перенести во избежание высыхания (рисA). Соберите иглы указал вольфрама 0,001 мм с проведением щипцы с использованием парафина фильмов (Рисунок 3B, C).
- Пипетка 50 мкл высокого титра mCherry выражая -C. burnetii (5,95 × 109 GE/мл) на парафин фильм под стерео микроскоп и личинки в бассейн бактерий.
- Поместите личинки в бассейн бактерий. Укол личинки с вольфрамовой иглой (рис. 3D). Переноса личинок на агаре пластину (рис. 3E).
- Переносить оставшиеся среды патогена на пластину агар и печатью пластины с парафином фильм.
- Держите личинки на плите в влажный воздух до желаемого времени послеоперационные инфекции (рис. 3F). В этом эксперименте, C. burnetii-зараженные личинки находятся на табличке на 24 часа.
- Теплый, агар сок дрозофилы фрукты при комнатной температуре 15 мин пластины сделаны ранее описал20.
- Гемолимфа добыча и покрытием hemocytes
- Подготовка среднего и оборудование после шага 1.1).
- Место 12 мм круглые Стекло покровное (№ 1 толщина) в хорошо 24-ну плиты. Пипетка 500 мкл DHIM в колодец.
- Извлечение гемолимфа из зараженные личинки, следующие шаги 1.2.4) - 1.2.8).
- Извлечь гемолимфа в хорошо после шаг 2.2.2).
- Повторить 2.2.3) и 2.2.4) для нескольких пакетов личинок.
- Центрифуга пластину на 1000 x g за 5 мин.
3. Визуализация
- Фиксации и окраски
- После позволяя hemocytes поселиться на круглые Стекло покровное в колодец, аккуратно извлеките носитель из каждой скважины.
- Аккуратно добавьте 200 мкл параформальдегида 4% (PFA) для каждой скважины поселились hemocytes. Инкубируйте hemocytes 20 минут при комнатной температуре.
- Удалите 4% PFA и аккуратно 200 мкл PBS, содержащие 0,1% тритон X-100 и 1% бычьим сывороточным альбумином (БСА) для каждой скважины. Инкубируйте hemocytes 10 мин при комнатной температуре.
- Удаление PBS и аккуратно добавить 200 мкл 1 × 4', 6-diamidino-2-phenylindole (DAPI) для каждой скважины. Инкубируйте hemocytes 10 мин в темноте при комнатной температуре.
- Удаление решения DAPI и осторожно добавить PBS в каждой скважине. Инкубируйте hemocytes 5 минут при комнатной температуре.
- Падение 10 мкл среднего antifade монтажа на стекло микроскопа.
- После удаления PBS из каждой скважины, удалите крышку стекла из 24-ну пластину, используя штраф отметил пинцет. Осторожно поместите крышку стекла на носитель antifade монтажа на слайде стекла, с hemocytes вниз.
- Разрешить на слайд, чтобы высушить, поместив его в темной ночи.
- Конфокальная томография
- Настройка Конфокальный микроскоп для трех изображений цвета DAPI, EGFP, и mCherry. Используйте следующие настройки: DAPI возбуждения (бывших) 405 нм, выбросов (ЭМ) 415-480 Нм; EGFP ex 488 нм, Эм 493-564 Нм; mCherry ex 587 Нм, Эм 597-700 Нм.
- Поместите образец на Микроскоп и сосредоточиться на образце с помощью 63 X / 1.4 Цель числовой апертуры (NA). Найдите нужную hemocytes в поле зрения для воображения.
- Отрегулируйте лазер мощности и детектор выгоды для достижения соответствующей экспозиции образца. Проверьте несколько z самолеты, чтобы убедиться, что уровень воздействия подходит для толщины весь пример.
- Найти верхней и нижней позиции по оси z весь кровяные. Задайте эти позиции как начальную и конечную позиции для z резании.
- Используйте только сканирование масштаб изображение области, содержащей кровяные. Часто используются факторы зум 3 X.
- Соберите изображения серии в соответствующей резолюции, например, 1024 x 1024 пикселей в плоскости x-y и 0,3 мкм интервалов в измерении z.
- 3D модель восстановления
Примечание: С открытым исходным кодом существует, выполняет многие из описанных ниже функций 3D модель восстановления.- Импортируйте файл серии z секционного изображения в программное обеспечение, связанное с Конфокальный микроскоп для 3D модель восстановления.
- Выберите показаны совместно локализации ядер, окрашенных с DAPI и mCherry, выражая C. burnetii в EGFP, выражая кровяные клетки. Обрезка изображения серии содержат только одну ячейку.
- Выберите параметр просмотра 3D, который реконструирует 3D-модели с использованием алгоритма расфасованных программного обеспечения. Выберите требуемый тип 3D представительства среди смесь, поверхности и смешанные варианты. В этом методе hemocytes, ядер и C. burnetii указаны с использованием моделей поверхности.
- Наблюдать за 3D реконструированный ячейки с различных позиций, просмотра, удерживая кнопку мыши и перетащив курсор вокруг экрана. Измените ячейки ориентации и положение моделируется источника света для оптимизации изображения. Существуют и другие варианты для непрозрачности, минимального и максимального порога, зеркальные, Ambient, Shineness и гамма для оптимизации изображения.
- Возьмите сечения с помощью модели, с помощью команды срезания и резания для визуализации интерьера содержимое кровяные.
4. Заявка на ген и/или белка анализа
- После инфекции с IIV6 и Listeria, лизировать клетки для последующего qRT ПЦР или Западный анализ помаркой как описано ранее21 и следующими инструкциями.
Примечание: Приведена Таблица материалов для грунтовки для qRT-PCR и антитела для западной помарки. - Анализ продуктов ПЦР электрофорезом геля агарозы, как описано выше22, для обеспечения надлежащей длины амплифицированного продукта.
Access restricted. Please log in or start a trial to view this content.
Результаты
Собирать жить hemocytes ex vivo инфекции, до 3 × 106 hemocytes были извлечены из 200 дрозофила 3rd instar личинки. Развивать наш метод, были попытки ряда различных методов. Индивидуальные личиночной рассечение примет до 1,5 ч, и в среднем ~ 8000 клетки были получены с помощ?...
Access restricted. Please log in or start a trial to view this content.
Обсуждение
Чтобы лучше понять, как инфицированных клеток хозяина, важно уточнить локализации возбудителя в клетках, особенно когда эксперименты на ранее непроверенных возбудителя и ячейки типа комбинации4. Во время учебы Каскад клеточный ответ, после инфицирования может указывать п...
Access restricted. Please log in or start a trial to view this content.
Раскрытие информации
Авторы заявляют, что они не имеют никаких финансовых интересов.
Благодарности
Мы благодарны д-р Роберт Heinzen для обеспечения запасов выражая mCherry Coxiella burnetii. Мы благодарим д-р Луис Тейшейра за предоставление беспозвоночных радужные вирус 6 и фондового центра Bloomington для предоставления летать запасов. Этот проект частично финансировался NIH Грант R00 AI106963 (A.G.G.) и университета штата Вашингтон.
Access restricted. Please log in or start a trial to view this content.
Материалы
| Name | Company | Catalog Number | Comments |
| Schneider's Drosophila Medium | Thermo Fisher Scientific (Gibco) | 21720024 | 1.1.1), 2.1.2) |
| Fetal Bovine Serum | GE Healthcare Life Sciences (HyClone) | SH30070.03HI | 1.1.1), 2.1.2) |
| Filter (0.22 µL) | RESTEK | 26158 | 1.1.1) |
| Strainer (100 µm) | Greiner bio-one | 542000 | 1.2.1), 2) |
| Stereo microscope | Amscope | SM-1BSZ-L6W | 1.2), 2) |
| Glass capillary | Fisher Scientific | 21-171-4 | 1.1), 1.2), 2) |
| Capillary puller | Narishige International USA, Inc. | PC-10 | 1.1.3) |
| Mineral oil | Snow River Products | 1.1.4) | |
| Nanoinjector | Drummond Scientific Company | 3-000-204 | 1.1), 1.2), 2.2) |
| Forceps | VWR | 82027-402 | 1.1.5), 1.2), 2), 3.1.7) |
| CO2 delivery apparatus | Genesee Scientific | 59-122BC | 1.2), 2) |
| Trypan Blue | Thermo Fisher Scientific (Gibco) | 15250061 | 1.3) |
| Hemocytometer | Hausser Scientific | 3100 | 1.3) |
| 24 well plate | Greiner bio-one | 662160 | 1.4), 2.2) |
| Coxiella burnetii - mCherry | Dr. Heinzen, R. | 1.4), 2.2) | |
| Drosophila fruit juice plates | Cold Spring Harbor Protocols | 2.1) http://cshprotocols.cshlp.org/content/2007/9/pdb.rec11113.full | |
| Agar | Fisher Bioreagents | BP1423-500 | 2.1.1.1) |
| Methyl paraben | Amresco | 0572-500G | 2.1.1.2) |
| Absolute ethanol | Fisher Bioreagents | BP2818-500 | 2.1.1.2) |
| Welch's 100% Grape juice frozen concentrate, 340 mL | Amazon | B0025UJVGM | 2.1.1.3) |
| Petri dishes, 10 x 35 mm | Fisher Scientific | 08-757-100A | 2.1.1.4) |
| Microscope cover glass | Fisher Scientific | 12-545-80 | 1.4.4), 2.2.2) |
| Yeast, Bakers Dried Active | MP Biomedicals | 0210140001 | 2.1) Add 2 parts of water to 1 part of yeast (v/v) |
| Tungsten needle | Fine Science Tools | 10130-20 | 2.1) |
| Holding forceps | VWR | HS8313 | 2.1) |
| Paraformaldehyde | Fisher Scientific | FLO4042-500 | 3.1.3) |
| Triton X-100 | Fisher Scientific | BP151-500 | 3.1.3) |
| Bovine Serum Albumin | Fisher Scientific | BP9706-100 | 3.1.3) |
| 4',6-diamidino-2-phenylindole | Thermo Fisher Scientific | 62247 | 3.1.4) |
| Antifade mounting medium | Thermo Fisher Scientific | P36930 | 3.1.6) |
| Confocal microsope | Leica | TCS SP8-X White Light Confocal Laser Scanning Microscope | 3.2) |
| 3D imaging reconstruction software | Leica | LASX with 3D visualization module | 3.3) |
| Microscope slides | Fisher Scientific | 12-552-3 | 3.1.6) |
| Invertebrate iridescent virus 6 (IIV6) | Dr. Teixeria, L. | 4) PLoS Biol, 6 (12), 2753-2763, doi: 10.1371/journal.pbio.1000002, (2008) | |
| Listeria monocytogenes | ATCC | strain: 10403S | 4) Listeria monocytogenes strain 10403S (Bishop and Hinrichs, 1987) was grown in Difco Brain-heart infusion (BHI) broth (BD Biosciences) containing 50 µg/ml streptomycin at 30 °C. |
| DNase I | Thermo Fisher Scientific(Invitrogen) | 18068015 | gDNA degradation |
| cDNA Synthesis Kit | Bio-Rad | 1708891 | cDNA synthesis |
| IIV6_193R_F | IDT | qRT-PCR, 5'- TCT TGT TTT CAG AAC CCC ATT -3' | |
| IIV6_193R_R | IDT | qRT-PCR, 5'- CAC GAA GAA TGA CCA CAA GG -3' | |
| RpII_qRTPCR_fwd | SIGMA-ALDRICH | qRT-PCR, 5'- GAA GCG TTT CTC CAA ACG -AG | |
| RpII_qRTPCR_rev | SIGMA-ALDRICH | qRT-PCR, 5'- TTG AGC GTA AGC ATC ACC -TG | |
| SYBR Green qRT-PCR reagent | Thermo Fisher Scientific | K0251, K0252, K0253 | qRT-PCR |
| Real-Time PCR System | Thermo Fisher Scientific | 4351107, 7500 Software v2.0 | qRT-PCR |
| Anti-Listeria monocytogenes antibody | abcam | ab35132 | Western blot |
| Anti-Actin antibody produced in rabbit | SIGMA-ALDRICH | A2066 | Western blot |
| Anti-Rabbit IgG (H+L), HRP Conjugate | Promega | W4011 | Western blot |
Ссылки
- Hoffmann, J. A. The immune response of Drosophila. Nature. 426 (6962), 33-38 (2003).
- Regan, J. C., et al. Steroid hormone signaling is essential to regulate innate immune cells and fight bacterial infection in Drosophila. PLoS Pathog. 9 (10), 1003720(2013).
- Yano, T., et al. Autophagic control of listeria through intracellular innate immune recognition in Drosophila. Nat Immunol. 9 (8), 908-916 (2008).
- Bastos, R. G., Howard, Z. P., Hiroyasu, A., Goodman, A. G. Host and Bacterial Factors Control Susceptibility of Drosophila melanogaster to Coxiella burnetii Infection. Infect Immun. 85 (7), (2017).
- Kacsoh, B. Z., Schlenke, T. A. High hemocyte load is associated with increased resistance against parasitoids in Drosophila suzukii, a relative of D. melanogaster. PLoS One. 7 (4), 34721(2012).
- Tsuzuki, S., et al. Switching between humoral and cellular immune responses in Drosophila. is guided by the cytokine GBP. Nat Commun. 5, 4628(2014).
- Kurucz, E., et al. Definition of Drosophila. hemocyte subsets by cell-type specific antigens. Acta Biol Hung. 58, Suppl 95-111 (2007).
- Neyen, C., Bretscher, A. J., Binggeli, O., Lemaitre, B. Methods to study Drosophila immunity. Methods. 68 (1), 116-128 (2014).
- Arefin, B., et al. Apoptosis in Hemocytes Induces a Shift in Effector Mechanisms in the Drosophila. Immune System and Leads to a Pro-Inflammatory State. PLoS One. 10 (8), 0136593(2015).
- Rus, F., et al. Expression pattern of Filamin-240 in Drosophila blood cells. Gene Expr Patterns. 6 (8), 928-934 (2006).
- Kurucz, E., et al. Hemese, a hemocyte-specific transmembrane protein, affects the cellular immune response in Drosophila. P Natl Acad Sci USA. 100 (5), 2622-2627 (2003).
- Markus, R., et al. Sessile hemocytes as a hematopoietic compartment in Drosophila melanogaster. P Natl Acad Sci USA. 106 (12), 4805-4809 (2009).
- Bretscher, A. J., et al. The Nimrod transmembrane receptor Eater is required for hemocyte attachment to the sessile compartment in Drosophila melanogaster. Biol Open. 4 (3), 355-363 (2015).
- Zettervall, C. J., et al. A directed screen for genes involved in Drosophila blood cell activation. P Natl Acad Sci USA. 101 (39), 14192-14197 (2004).
- Flynt, A., Liu, N., Martin, R., Lai, E. C. Dicing of viral replication intermediates during silencing of latent Drosophila viruses. P Natl Acad Sci USA. 106 (13), 5270-5275 (2009).
- Jovel, J., Schneemann, A. Molecular characterization of Drosophila cells persistently infected with Flock House virus. Virology. 419 (1), 43-53 (2011).
- Stoepler, T. M., Castillo, J. C., Lill, J. T., Eleftherianos, I. A simple protocol for extracting hemocytes from wild caterpillars. J Vis Exp. (69), e4173(2012).
- Sampson, C. J., Williams, M. J. Protocol for ex vivo incubation of Drosophila primary post-embryonic haemocytes for real-time analyses. Methods Mol Biol. 827, 359-367 (2012).
- Nehme, N. T., et al. A model of bacterial intestinal infections in Drosophila melanogaster. PLoS Pathog. 3 (11), 173(2007).
- Drosophila fruit juice egg plates. Cold Spring Harbor Protocols. (9), pdb.rec11113 (2007).
- Ahlers, L. R., Bastos, R. G., Hiroyasu, A., Goodman, A. G. Invertebrate Iridescent Virus 6, a DNA Virus, Stimulates a Mammalian Innate Immune Response through RIG-I-Like Receptors. PLoS One. 11 (11), 0166088(2016).
- Lee, P. Y., Costumbrado, J., Hsu, C. Y., Kim, Y. H. Agarose gel electrophoresis for the separation of DNA fragments. J Vis Exp. (62), (2012).
- Petraki, S., Alexander, B., Bruckner, K. Assaying Blood Cell Populations of the Drosophila melanogaster Larva. J Vis Exp. (105), (2015).
- Figliozzi, R. W., Chen, F., Chi, A., Hsia, S. C. Using the inverse Poisson distribution to calculate multiplicity of infection and viral replication by a high-throughput fluorescent imaging system. Virol Sin. 31 (2), 180-183 (2016).
- Rizki, T. M., Rizki, R. M. Lamellocyte differentiation in Drosophila. larvae parasitized by Leptopilina. Dev Comp Immunol. 16 (2-3), 103-110 (1992).
- Markus, R., Kurucz, E., Rus, F., Ando, I. Sterile wounding is a minimal and sufficient trigger for a cellular immune response in Drosophila melanogaster. Immunol Lett. 101 (1), 108-111 (2005).
- McCormack, R., et al. Perforin-2 Protects Host Cells and Mice by Restricting the Vacuole to Cytosol Transitioning of a Bacterial Pathogen. Infect Immun. 84 (4), 1083-1091 (2016).
- Ozgen, A., et al. Construction and characterization of a recombinant invertebrate iridovirus. Virus Res. 189, 286-292 (2014).
- Jakob, N. J., Muller, K., Bahr, U., Darai, G. Analysis of the first complete DNA sequence of an invertebrate iridovirus: coding strategy of the genome of Chilo iridescent virus. Virology. 286 (1), 182-196 (2001).
- Ghigo, E., Colombo, M. I., Heinzen, R. A. The Coxiella burnetii parasitophorous vacuole. Adv Exp Med Biol. 984, 141-169 (2012).
- Liu, F., et al. Drosophila melanogaster prophenoloxidases respond inconsistently to Cu2+ and have different activity in vitro. Dev Comp Immunol. 36 (3), 619-628 (2012).
- De Gregorio, E., et al. An immune-responsive Serpin regulates the melanization cascade in Drosophila. Dev Cell. 3 (4), 581-592 (2002).
- Kari, B., et al. The raspberry Gene Is Involved in the Regulation of the Cellular Immune Response in Drosophila melanogaster. PLoS One. 11 (3), 0150910(2016).
- Wu, A. R., et al. Quantitative assessment of single-cell RNA-sequencing methods. Nat Methods. 11 (1), 41-46 (2014).
- Buettner, F., et al. Computational analysis of cell-to-cell heterogeneity in single-cell RNA-sequencing data reveals hidden subpopulations of cells. Nat Biotechnol. 33 (2), 155-160 (2015).
- Nevil, M., Bondra, E. R., Schulz, K. N., Kaplan, T., Harrison, M. M. Stable Binding of the Conserved Transcription Factor Grainy Head to its Target Genes Throughout Drosophila melanogaster Development. Genetics. 205 (2), 605-620 (2017).
- Yang, C. P., et al. Transcriptomes of lineage-specific Drosophila neuroblasts profiled by genetic targeting and robotic sorting. Development. 143 (3), 411-421 (2016).
- Jaitin, D. A., et al. Dissecting Immune Circuits by Linking CRISPR-Pooled Screens with Single-Cell RNA-Seq. Cell. 167 (7), 1883-1896 (2016).
- Karaiskos, N., et al. The Drosophila embryo at single-cell transcriptome resolution. Science. 358 (6360), 194-199 (2017).
Access restricted. Please log in or start a trial to view this content.
Перепечатки и разрешения
Запросить разрешение на использование текста или рисунков этого JoVE статьи
Запросить разрешениеСмотреть дополнительные статьи
This article has been published
Video Coming Soon
Авторские права © 2025 MyJoVE Corporation. Все права защищены