Zum Anzeigen dieser Inhalte ist ein JoVE-Abonnement erforderlich. Melden Sie sich an oder starten Sie Ihre kostenlose Testversion.
Method Article
Extraktion von Hemocytes aus Drosophila Melanogaster Larven für mikrobielle Infektionen und Analyse
In diesem Artikel
Zusammenfassung
Diese Methode zeigt, wie Erreger Invasion in Insektenzellen mit dreidimensionalen (3D) Modellen zu visualisieren. Hemocytes von Drosophila Larven infiziert wurden mit viralen oder bakteriellen Erregern, Ex Vivo oder in Vivo. Infizierte Hemocytes wurden dann fixiert und für die Bildgebung mit einem confocal Mikroskop und anschließende zelluläre 3D-Rekonstruktion befleckt.
Zusammenfassung
Während die pathogenen Infektion von Drosophila Melanogaster, Rolle Hemocytes eine wichtige bei der Immunantwort in die Infektion. Ziel dieses Protokolls ist es daher, Entwicklung eine Methode um die Invasion der Erreger in einem bestimmten immun Fach fliegen, nämlich Hemocytes zu visualisieren. Mit der Methode, die hier vorgestellten, bis zu 3 × 106 live Hemocytes von 200 Drosophila 3rd Instar Larven in 30 min für Ex-Vivo -Infektion erhalten. Alternativ kann die Hemocytes infizierte in Vivo durch Injektion von 3rd Instar Larven gefolgt von Hemocyte Extraktion bis zu 24 h nach der Infektion. Diese infizierten Primärzellen gefixt, gebeizt und abgebildet mit der konfokalen Mikroskopie. Dann wurden aus den Bildern endgültig Erreger Invasion zeigen 3D-Darstellungen generiert. Darüber hinaus hochwertige RNA für die qRT-PCR für den Nachweis des Erregers mRNA folgenden erhalten werden Infektionen und ausreichend Protein aus diesen Zellen für Western-Blot Analyse extrahiert werden können. Zusammengenommen, präsentieren wir Ihnen eine Methode zur definitiven Versöhnung der Erreger Invasion und Bestätigung der Infektion mit bakterieller und viraler Erreger-Typen und eine effiziente Methode für die Extraktion von Hemocyte genug live Hemocytes von Drosophila zu erhalten Larven für ex-Vivo - und in-Vivo -Infektions-Experimente.
Einleitung
Drosophila Melanogaster ist eine gut etablierte Modellorganismus zur Erforschung der angeborenen Immunität1. Im Rahmen der angeborenen Immunantwort spielen Hemocytes eine wichtige Rolle bei der Reaktion auf Krankheitserreger Herausforderung. Hemocytes sind entscheidend für die Kapselung von Parasiten, sowie eine wichtige Funktion bei der Bekämpfung des Erregers durch phagocytic Aktion während Pilz-, Virus- und bakterielle Infektion2,3.
Um den Host angeborene Immunantwort auf pathogene mikrobielle Infektion am besten zu verstehen, ist es wichtig, wie der Erreger dringt in Wirtszellen während der Infektion zu visualisieren. Diese Visualisierung trägt zum Verständnis des Mechanismus der Invasion. Zusammen mit Details der Erreger intrazelluläre Lokalisation und die zelluläre Antwort können diese Daten Aufschluss über der Wirtsantwort zur Infektion und die zellularen Organellen die Mikrobe interagiert. 3D-Modell Wiederaufbau nach Bildgebung durch Mikroskopie kann hilfreich, um die genaue Lage von Krankheitserregern in Wirtszellen zu bestimmen. In dieser Studie visualisieren wir die Invasion von Coxiella Burnetii (C. Burnetii), dem Erreger der q-Fieber, eine Zoonose, die eine ernsthafte für die menschliche und tierische Gesundheit in primären Drosophila Hemocytes Bedrohung. Vor kurzem wurde gezeigt, dass Drosophila sind anfällig für die Sicherheitsstufe 2 Nine Mile Phase II (NMII) Klon 4 Stamm von C. Burnetii und, dass diese Sorte in Drosophila4replizieren kann darauf hinweist, dass Drosophila einsetzbar als Modellorganismus C. Burnetii Pathogenese zu studieren.
Frühere Studien haben Hemocytes verwendet, um angeborene Immunantwort des Wirtes zu untersuchen. Hemocytes wurden verwendet für morphologische Beobachtungen5,6,7, morphometrische Analyse2,8, Phagozytose Analyse2,3, qRT-PCR-2 , 9, Immunopräzipitation10,11, immunofluorescent Analyse10,12, Immunostaining13, Immunoblotting3,10, 11 und Immunohistochemistry9,14. Obwohl Drosophila S2 Zellen auch für verschiedene in-vitro- Experimente zur Verfügung stehen, ändern Immortalisierung und potenzielle bereits bestehende Virusinfektion ihr Verhalten15,16. Die Verwendung von Primärzellen im Gegensatz zu einer immortalisierte Zelllinie wie S2-Zellen, kann für das Studium der angeborene immune Funktion in einem System repräsentativer für den gesamten Organismus. Darüber hinaus kann die Infektion von Hemocytes in Vivo, vor der Extraktion, die Zellen, die Interaktion mit anderen Host Proteine und Gewebe, einen Vorteil gegenüber der Gewinnung von Hemocytes vor der ex-Vivo -Infektion. Eine Reihe von verschiedenen Methoden wurden genutzt, um eine ausreichende Anzahl von Hemocytes in kurzer Zeit zu den Hemocytes lebendig8,17,18,19zu erhalten.
In dieser Studie stellen wir Ihnen eine Methode zur Hemocytes von Drosophila 3rd Instar Larven für pathogene mikrobielle Infektion mit C. Burnetii, Listeria Monocytogenes (Listerien) oder Wirbellosen schillernden extrahieren Virus 6 (IIV6). Wir beschreiben die Methoden zur in Vivo und ex Vivo Hemocyte Infektionen. In Vivound ex Vivo-infizierten Hemocytes waren mit der konfokalen Mikroskopie visualisiert und verwendet zum Erstellen von 3D Modellen von C. Burnetii Invasion. Darüber hinaus verwenden die Extraktion Protokoll, ex-Vivo-infizierten Hemocytes dienten für gen- und Protein-Expression Assays. Insbesondere war um zu prüfen, inwieweit der Infektion mit IIV6 und Listeria, total RNA oder Protein isoliert aus den Zellen zur qRT-PCR oder Western-Blot Analyse. Zusammen genommen, das Protokoll bietet Methoden, um schnell hohe Zahl von Hemocytes von 3rd Instar Larven und Beweise, dass primäre Hemocytes infiziert, in Vivo und Ex Vivosammeln, sind eine geeignete Plattform für mikrobielle Erreger Infektion Studien und anwendbaren nachgeschalteten Analysen wie Mikroskopie, Transkriptom und Proteomik.
Access restricted. Please log in or start a trial to view this content.
Protokoll
1. ex Vivo -Infektion
- Medium und Ausrüstung
- Unter sterilen Bedingungen bereiten frische Drosophila Hemocyte isolieren Medium (DHIM) mit 75 % Schneider Drosophila mit 25 % mittlere fetalen Bovine Serum (FBS) und Filter Sterilisieren sie.
- Layer-2-3 Stücke von 10 x 10 cm Paraffin Film unter einem Stereomikroskop.
- Bereiten Sie die Glas-Kapillare. Legen Sie die Kapillare Puller-Heizung auf 55 % des Maximums. Ziehen Sie das Kapillarrohr zu einer scharfen Spitze von ca. 10 µm.
- Abgleich der Kapillare mit Mineralöl.
- Montieren Sie gefüllte Kapillarröhrchen auf der Nanoinjector (Abb. 1A), und öffnen Sie verschmolzen Kapillarrohr Tipp durch Abbruch der Tipp mit der Pinzette (Abbildung 1B). Der Außendurchmesser der Spitze sollte 100 µm für die einfache Aufnahme von den Hemocytes.
- Werfen Sie so viel Öl wie möglich von der Spitze des Kapillarrohr aus, dann füllen Sie mit DHIM. Für einfache Visualisierung von der Grenze bis genommen Hämolymphe und das Öl, enthalten eine Luftblase zwischen Öl und DHIM (Abbildung 1B").
- Hämolymphe Extraktion
- Wählen Sie 3rd instar Drosophila Larven von innen Wand von Lebensmitteln Fläschchen vorsichtig mit Pinzette und legen Sie sie in einem 100 µm-Sieb (Abbildung 1C). 3rd Instar Larven befinden sich ca. 3-6 Tage nach fruchtbaren Eiablage durch eine erwachsene Frau.
Hinweis: Diese Experimente verwendet den Genotyp, w1118; P {w+ mC= Hml-GAL4.Δ}, 2P {w+ mC= FH-2xEGFP} AH2, da Hemocytes von diesen Tieren erhöhte grünes fluoreszierendes Protein (EGFP) zur Identifizierung der Zellen durch Mikroskopie Unterstützung zum Ausdruck bringen. - Larven 5 mL sterilem Wasser übergießen und schütteln Sie das Sieb 5 S. Ort das Sieb auf Aufgabe wischen, um das überschüssige Wasser entfernen (Abbildung 1C").
- Übertragen Sie die Larven in einem 1,5 mL Microcentrifuge Schlauch. Betäuben sie mit CO2 -Gas für 5 s (Abbildung 1D).
- Legen Sie die Larven auf Paraffin-Film unter dem Stereomikroskop mit Dorsalseite nach oben (Abb. 2A).
- Stelle das Glas leicht auf die Larven hinteren Körpers Kapillare zu befestigen und zu stören die hintere Nagelhaut öffnen mit feinen Spitzen Pinzette (Abbildung 2B).
- Lassen Sie die Hämolymphe auf Paraffin-Folie (Abbildung 2C) fließen.
- Machen Sie einen Pool von Hämolymphe einschließlich Hemocytes von 20-50 Larven zu einem Zeitpunkt auf dem Paraffin-Film.
- Nehmen Sie gepoolten Hämolymphe mit dem Glas Kapillare auf den Nanoinjector (Abb. 2D).
Hinweis: Sollte ca. 10-20 µL der Hämolymphe. - Werfen Sie die Hämolymphe zu einem 1,5 mL Microcentrifuge Schlauch mit 500 µL DHIM (Abb. 2E).
- Wiederholen Sie die Schritte 1.2.4) - 1.2.9) für jede Charge der Larven.
- Wählen Sie 3rd instar Drosophila Larven von innen Wand von Lebensmitteln Fläschchen vorsichtig mit Pinzette und legen Sie sie in einem 100 µm-Sieb (Abbildung 1C). 3rd Instar Larven befinden sich ca. 3-6 Tage nach fruchtbaren Eiablage durch eine erwachsene Frau.
- Die Anzahl der Hemocytes.
- Pipette 5 µL 0,4 % Trypan blau-Lösung in einem 0,6 mL Microcentrifuge Schlauch, die toten Zellen zu beflecken. Vorsichtig mischen Sie, DHIM und Hemocytes in der 1,5 mL Tube mit einer Pipette und übertragen Sie 5 µL DHIM einschließlich Hemocytes 0,6 mL Microcentrifuge Schlauch und mischen Sie vorsichtig.
- Pipette 10 µL Zellen aus der 1:1 Trypan blau: Hemocyte Mischung in der Hemocytometer.
- Die Anzahl der Leben Hemocytes, die sind nicht befleckt mit Trypan blau in jeder der 4 Ecken Felder von der Hemocytomter und die Konzentration von Hemocytes pro Milliliter Formel zu berechnen:
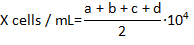
wo X ist die Konzentration von live Hemocytes pro Milliliter; a, b, C und d sind die Anzahl der lebenden Zellen (wie von Trypan blau Ausgrenzung bestimmt) in je 4 der Felder in der Hemocytometer gezählt. Division durch 2 von der Gesamtzahl der Zellen gezählt ist aufgrund der Verdünnung von 1:1 der Zellen mit Trypan blau. Mit Trypan blau gefärbten Zellen gelten als tot.
-
Ex-vivo Infektionen
- Bestimmen Sie die Anzahl der Vertiefungen mit Zellen basierend auf Zeitpunkte und biologische gesät werden repliziert benötigt für jedes Experiment. 5.0 × 104 Hemocytes sind wünschenswert, dass RNA und Protein Reinigung nach Infektion.
- Berechnen Sie das Volumen der Erreger Lager mit DHIM verdünnt werden, für eine Infektion mit der folgenden Formel:
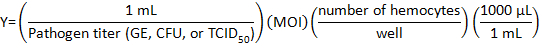
wo Multiplizität der Infektion (MOI) ist die Anzahl der Viren oder Bakterien auf Wunsch pro Zelle.
Hinweis: MOI verwendet richtet sich nach den einzelnen Experiment und Tests durchgeführt. Hier, 10 Genom-äquivalente (GE) / Zelle von C. Burnetii, 10 KBE/Zelle von Listerienoder 1 TCID50/cell von IIV6 diente. - Bereiten Sie 500 µL des Erregers Medium für jede Vertiefung einer 24-Well-Platte durch virale oder bakterielle Volumen in einem Rohr das richtige Volumen der DHIM hinzufügen.
- Platz 12 mm Runde Deckglas (Nr. 1-Dicke) in einen Brunnen von einer 24-Well-Platte.
- Die DHIM einschließlich der Hemocytes in Vertiefungen von einer 24-Well-Platte aufgeteilt.
- Hemocytes in einem gut 500 µL des Erregers Medium hinzufügen.
- Zentrifugieren Sie die Platte bei 1.000 x g für 5 min.
- Inkubieren Sie die Platte für 1 h bei 28 ° c Alle 15 min leicht kippen die Platte von hinten, vorne, dann links nach rechts für 5 s von Hand.
- Nach 1 h Einmarsch/Anlage Schritt 1.4.8) sanft pipette aus dem Erreger Medium waschen die Hemocytes mit frischen DHIM und füllen Sie ihn mit 500 µL des frischen DHIM.
- Die gewünschte Zeit inkubieren Sie der infizierten Hemocytes. In diesen Experimenten, C. Burnetii- oder IIV6-infizierten Hemocytes sind für 24 h und Listeriainkubiert-infizierten Hemocytes sind für 1, 2 oder 4 h inkubiert.
2. in-Vivo -Infektion
- Infektion
- Warm Drosophila Obst Saft nährbodenplatte bei Raumtemperatur für 15 min. Platten erfolgt wie oben beschrieben20.
- 700 mL Wasser und Autoklaven 30 g Agar-Agar hinzufügen es 40 Minuten.
- 0,5 g Methylparaben in 10 mL von absoluten Ethanol auflösen.
- 300 mL Fruchtsaftkonzentrat Methyl-Paraben-Lösung hinzufügen.
- Schnell die Saft-Konzentrat in die autoklaviert Agar Lösung mischen und 5 mL in 10 × 35 mm Petrischalen zu verzichten.
- Nachdem die Platten für 15 min abgekühlt haben, speichern sie bei 4 ° C.
- Bereiten Sie 3rd Instar Larven folgende Schritte 1.2.1) - 1.2.3).
- Legen Sie die Hefe-Paste auf einer nährbodenplatte. Machen Sie einen feinen Schnitt in der Agarplatte, wo die Larven migrieren können, um zu vermeiden, Trocknen(Abbildung 3). Montieren Sie eine 0,001 mm Spitzen Wolfram Nadel mit Zange mit Paraffin-Film (Abbildung 3B, C) zu halten.
- Hohe Titer mCherry 50 µL Pipette auszudrücken -C. Burnetii (5,95 × 109 GE/mL) auf das Paraffin unter dem Stereo-Mikroskop und Auflegen der Larven in den Pool von Bakterien.
- Legen Sie die Larven in den Pool von Bakterien. Stechen Sie die Larven mit einer Wolfram-Nadel (Abbildung 3D). Übertragen Sie die Larven auf einer nährbodenplatte (Abbildung 3E).
- Die verbleibenden Erreger Medium auf die Agarplatte zu übertragen und die Dichtplatte mit Paraffin Film.
- Halten Sie die Larven auf der Platte in feuchter Luft bis die gewünschte Zeit nach der Infektion (Abbildung 3F). In diesem Experiment, C. Burnetii-infizierten Larven sind auf dem Teller für 24 h.
- Warm Drosophila Obst Saft nährbodenplatte bei Raumtemperatur für 15 min. Platten erfolgt wie oben beschrieben20.
- Hämolymphe Extraktion und Beschichtung von hemocytes
- Bereiten Sie das Medium und die Ausrüstung nach Schritt 1.1).
- Platz 12 mm Runde Deckglas (Nr. 1-Dicke) in einen Brunnen von einer 24-Well-Platte. Pipette 500 µL DHIM in den Brunnen.
- Auszug der Hämolymphe aus infizierten Larven, die folgenden Schritte 1.2.4) - 1.2.8).
- Werfen Sie die Hämolymphe in den nun folgenden Schritt 2.2.2).
- 2.2.3) wiederholen und 2.2.4) für mehrere Batches von Larven.
- Zentrifugieren Sie die Platte bei 1.000 x g für 5 min.
(3) Visualisierung
- Fixierung und Färbung
- Entfernen Sie nach Abzug der Hemocytes an die Runde Abdeckung Glas in den Brunnen zu begleichen vorsichtig das Medium aus jedem Brunnen.
- Fügen Sie sanft 200 µL 4 % Paraformaldehyd (PFA) in jede Vertiefung der sesshaften Hemocytes. Inkubieren Sie die Hemocytes für 20 min bei Raumtemperatur.
- Entfernen Sie die 4 % PFA und sanft fügen Sie 200 µL PBS mit 0,1 % Triton X-100 und 1 % Bovine Serum Albumin (BSA) in jede Vertiefung. Inkubieren Sie die Hemocytes für 10 min bei Raumtemperatur.
- Entfernen Sie die PBS und fügen Sie sanft 200 µL 1 × 4', 6-Diamidino-2-Phenylindole (DAPI) in jede Vertiefung. Inkubieren Sie die Hemocytes für 10 Minuten im Dunkeln bei Raumtemperatur.
- Entfernen Sie die DAPI-Lösung, und fügen Sie sanft PBS in jede Vertiefung. Inkubieren Sie die Hemocytes für 5 min bei Raumtemperatur.
- Fallen Sie 10 µL des Mediums antifade Montage auf einen Glas-Objektträger.
- Nach dem Entfernen der PBS aus jedem Brunnen, entfernen Sie das Deckglas aus den 24-Well-Platte mit feinen Spitzen Pinzette. Sanft lege das Deckglas auf den antifade Eindeckmittel auf den Objektträger mit dem Hemocytes nach unten.
- Lassen Sie die Folie trocken indem man sie in die dunkle Nacht.
- Confocal Imaging
- Konfigurieren Sie die confocal Mikroskop für drei Farbe Imaging DAPI, EGFP und mCherry. Verwenden Sie die folgende Einstellung: DAPI Erregung (ex) 405 nm, Emission (Em) 415-480 nm; EGFP ex 488 nm, Em 493-564 nm; mCherry ex 587 nm, Em 597-700 nm.
- Legen Sie die Probe auf das Mikroskop und den Fokus auf die Probe mit einem 63 X / 1.4 numerische Apertur (NA) Ziel. Suchen Sie nach gewünschten Hemocytes in das Sichtfeld für die Bildgebung.
- Stellen Sie Laser Power und Detektor Gewinne um angemessene Belichtung der Probe zu erreichen. Überprüfen Sie mehrere Z-Flugzeuge, um sicherzustellen, dass die Belichtung für die gesamte Probendicke geeignet ist.
- Die oberen und unteren Position auf der z-Achse eine ganze Hemocyte zu finden. Legen Sie diese Positionen als die Anfangs- und Endpositionen für Z-Schnitt.
- Verwenden Sie nur Scan Zoom Bild des Bereichs, in den Hemocyte. Zoom-Faktoren von 3 X werden häufig verwendet.
- Sammeln Sie die Bildserien mit geeigneten Auflösung z. B. 1024 x 1024 Pixel in der XY-Ebene und 0,3 µm-Abstand in der Z-Dimension.
- 3D Modell-Rekonstruktion
Hinweis: Open-Source-Software ist vorhanden, die viele der unten beschriebenen Funktionen für 3D Modell Rekonstruktion ausführt.- Importieren Sie die Z-geschnittene Serie Bilddatei in die Software zugeordnete confocal Mikroskop für 3D-Modell Wiederaufbau.
- Wählen Sie eine Zelle zeigt Co Lokalisierung der Kerne gebeizt mit DAPI und mCherry mit dem Ausdruck C. Burnetii in einem EGFP Hemocyte zum Ausdruck zu bringen. Schneiden Sie die Bildserie, um nur die einzelne Zelle enthalten.
- Die Option 3D Viewer die mit abgepackten Softwarealgorithmus 3D-Modell rekonstruiert. Wählen Sie die gewünschte Art der 3D Darstellung unter Blend, Oberfläche und gemischte Optionen. Bei dieser Methode werden Hemocytes, Kerne und C. Burnetii Oberflächenmodelle mit angezeigt.
- Beobachten Sie die 3D rekonstruierte Zelle aus verschiedenen Positionen anzeigen durch die Maustaste gedrückt halten und ziehen den Cursor über den Bildschirm. Passen Sie die Zelle Ausrichtung und Position der modellierten Lichtquelle um das Bild zu optimieren. Andere Optionen für Deckkraft, Minimum und maximale Schwelle, Specular, Ambient, Shineness und Gamma bestehen, um das Bild zu optimieren.
- Nehmen Sie Querschnitte durch das Modell mit Clipping und speziellen Befehlen, um den inneren Inhalt der Hemocyte zu visualisieren.
4. Antrag auf gen und/oder Protein-Analyse
- Nach der Infektion mit IIV6 und Listeria, lösen Sie Zellen für nachfolgende qRT-PCR oder Western-Blot Analyse wie zuvor beschrieben,21 und folgende Angaben des Herstellers.
Hinweis: Siehe die Tabelle der Materialien für die Primer für qRT-PCR und die Antikörper für Western-Blot. - Analysieren Sie PCR-Produkte durch Agarose-Gelelektrophorese, wie zuvor beschrieben22, um die richtige Länge des verstärkten Produktes zu gewährleisten.
Access restricted. Please log in or start a trial to view this content.
Ergebnisse
Zu sammeln Hemocytes für Ex-Vivo -Infektion, Leben bis zu 3 × 10 wurden 200 Drosophila 3rd Instar Larven6 Hemocytes entzogen. Um unsere Methode zu entwickeln, wurden eine Reihe von verschiedenen Techniken versucht. Einzelne Larven Dissektion bis zu 1,5 Stunden dauern würde wurden, und durchschnittlich ~ 8000 Zellen mit dieser Methode18, von denen meisten nicht bis zum Jahresende Sammlung noch am Leben waren. Als nächstes ...
Access restricted. Please log in or start a trial to view this content.
Diskussion
Um besser zu verstehen, wie Wirtszellen infiziert werden, ist es wichtig, die Lokalisation des Erregers in den Zellen zu klären, vor allem, wenn zuvor noch nicht getesteten Erreger und Zelle Typ Kombinationen4experimentieren. Während Studium der zellulären Antwort Kaskade nach Infektion produktive Erreger Invasion anzeigen kann, gilt die Kombination von bildgebenden und zellulären Antwortdaten Erreger Invasion und Infektion nachweisen. Während Berichte zeigen 2D-Bilder Erreger Invasion in die...
Access restricted. Please log in or start a trial to view this content.
Offenlegungen
Die Autoren erklären, dass sie keine finanziellen Interessenkonflikte.
Danksagungen
Wir sind dankbar, Dr. Robert Heinzen für die Bereitstellung von Aktien von mCherry exprimierenden Coxiella Burnetii. Wir danken Dr. Luis Teixeira für Bereitstellung von Wirbellosen irisierende Virus 6 und Bloomington Stock Center für die Bereitstellung fliegen Bestände. Dieses Projekt wurde teilweise von NIH Grant R00 AI106963 (A.G.G) und der Washington State University finanziert.
Access restricted. Please log in or start a trial to view this content.
Materialien
| Name | Company | Catalog Number | Comments |
| Schneider's Drosophila Medium | Thermo Fisher Scientific (Gibco) | 21720024 | 1.1.1), 2.1.2) |
| Fetal Bovine Serum | GE Healthcare Life Sciences (HyClone) | SH30070.03HI | 1.1.1), 2.1.2) |
| Filter (0.22 µL) | RESTEK | 26158 | 1.1.1) |
| Strainer (100 µm) | Greiner bio-one | 542000 | 1.2.1), 2) |
| Stereo microscope | Amscope | SM-1BSZ-L6W | 1.2), 2) |
| Glass capillary | Fisher Scientific | 21-171-4 | 1.1), 1.2), 2) |
| Capillary puller | Narishige International USA, Inc. | PC-10 | 1.1.3) |
| Mineral oil | Snow River Products | 1.1.4) | |
| Nanoinjector | Drummond Scientific Company | 3-000-204 | 1.1), 1.2), 2.2) |
| Forceps | VWR | 82027-402 | 1.1.5), 1.2), 2), 3.1.7) |
| CO2 delivery apparatus | Genesee Scientific | 59-122BC | 1.2), 2) |
| Trypan Blue | Thermo Fisher Scientific (Gibco) | 15250061 | 1.3) |
| Hemocytometer | Hausser Scientific | 3100 | 1.3) |
| 24 well plate | Greiner bio-one | 662160 | 1.4), 2.2) |
| Coxiella burnetii - mCherry | Dr. Heinzen, R. | 1.4), 2.2) | |
| Drosophila fruit juice plates | Cold Spring Harbor Protocols | 2.1) http://cshprotocols.cshlp.org/content/2007/9/pdb.rec11113.full | |
| Agar | Fisher Bioreagents | BP1423-500 | 2.1.1.1) |
| Methyl paraben | Amresco | 0572-500G | 2.1.1.2) |
| Absolute ethanol | Fisher Bioreagents | BP2818-500 | 2.1.1.2) |
| Welch's 100% Grape juice frozen concentrate, 340 mL | Amazon | B0025UJVGM | 2.1.1.3) |
| Petri dishes, 10 x 35 mm | Fisher Scientific | 08-757-100A | 2.1.1.4) |
| Microscope cover glass | Fisher Scientific | 12-545-80 | 1.4.4), 2.2.2) |
| Yeast, Bakers Dried Active | MP Biomedicals | 0210140001 | 2.1) Add 2 parts of water to 1 part of yeast (v/v) |
| Tungsten needle | Fine Science Tools | 10130-20 | 2.1) |
| Holding forceps | VWR | HS8313 | 2.1) |
| Paraformaldehyde | Fisher Scientific | FLO4042-500 | 3.1.3) |
| Triton X-100 | Fisher Scientific | BP151-500 | 3.1.3) |
| Bovine Serum Albumin | Fisher Scientific | BP9706-100 | 3.1.3) |
| 4',6-diamidino-2-phenylindole | Thermo Fisher Scientific | 62247 | 3.1.4) |
| Antifade mounting medium | Thermo Fisher Scientific | P36930 | 3.1.6) |
| Confocal microsope | Leica | TCS SP8-X White Light Confocal Laser Scanning Microscope | 3.2) |
| 3D imaging reconstruction software | Leica | LASX with 3D visualization module | 3.3) |
| Microscope slides | Fisher Scientific | 12-552-3 | 3.1.6) |
| Invertebrate iridescent virus 6 (IIV6) | Dr. Teixeria, L. | 4) PLoS Biol, 6 (12), 2753-2763, doi: 10.1371/journal.pbio.1000002, (2008) | |
| Listeria monocytogenes | ATCC | strain: 10403S | 4) Listeria monocytogenes strain 10403S (Bishop and Hinrichs, 1987) was grown in Difco Brain-heart infusion (BHI) broth (BD Biosciences) containing 50 µg/ml streptomycin at 30 °C. |
| DNase I | Thermo Fisher Scientific(Invitrogen) | 18068015 | gDNA degradation |
| cDNA Synthesis Kit | Bio-Rad | 1708891 | cDNA synthesis |
| IIV6_193R_F | IDT | qRT-PCR, 5'- TCT TGT TTT CAG AAC CCC ATT -3' | |
| IIV6_193R_R | IDT | qRT-PCR, 5'- CAC GAA GAA TGA CCA CAA GG -3' | |
| RpII_qRTPCR_fwd | SIGMA-ALDRICH | qRT-PCR, 5'- GAA GCG TTT CTC CAA ACG -AG | |
| RpII_qRTPCR_rev | SIGMA-ALDRICH | qRT-PCR, 5'- TTG AGC GTA AGC ATC ACC -TG | |
| SYBR Green qRT-PCR reagent | Thermo Fisher Scientific | K0251, K0252, K0253 | qRT-PCR |
| Real-Time PCR System | Thermo Fisher Scientific | 4351107, 7500 Software v2.0 | qRT-PCR |
| Anti-Listeria monocytogenes antibody | abcam | ab35132 | Western blot |
| Anti-Actin antibody produced in rabbit | SIGMA-ALDRICH | A2066 | Western blot |
| Anti-Rabbit IgG (H+L), HRP Conjugate | Promega | W4011 | Western blot |
Referenzen
- Hoffmann, J. A. The immune response of Drosophila. Nature. 426 (6962), 33-38 (2003).
- Regan, J. C., et al. Steroid hormone signaling is essential to regulate innate immune cells and fight bacterial infection in Drosophila. PLoS Pathog. 9 (10), 1003720(2013).
- Yano, T., et al. Autophagic control of listeria through intracellular innate immune recognition in Drosophila. Nat Immunol. 9 (8), 908-916 (2008).
- Bastos, R. G., Howard, Z. P., Hiroyasu, A., Goodman, A. G. Host and Bacterial Factors Control Susceptibility of Drosophila melanogaster to Coxiella burnetii Infection. Infect Immun. 85 (7), (2017).
- Kacsoh, B. Z., Schlenke, T. A. High hemocyte load is associated with increased resistance against parasitoids in Drosophila suzukii, a relative of D. melanogaster. PLoS One. 7 (4), 34721(2012).
- Tsuzuki, S., et al. Switching between humoral and cellular immune responses in Drosophila. is guided by the cytokine GBP. Nat Commun. 5, 4628(2014).
- Kurucz, E., et al. Definition of Drosophila. hemocyte subsets by cell-type specific antigens. Acta Biol Hung. 58, Suppl 95-111 (2007).
- Neyen, C., Bretscher, A. J., Binggeli, O., Lemaitre, B. Methods to study Drosophila immunity. Methods. 68 (1), 116-128 (2014).
- Arefin, B., et al. Apoptosis in Hemocytes Induces a Shift in Effector Mechanisms in the Drosophila. Immune System and Leads to a Pro-Inflammatory State. PLoS One. 10 (8), 0136593(2015).
- Rus, F., et al. Expression pattern of Filamin-240 in Drosophila blood cells. Gene Expr Patterns. 6 (8), 928-934 (2006).
- Kurucz, E., et al. Hemese, a hemocyte-specific transmembrane protein, affects the cellular immune response in Drosophila. P Natl Acad Sci USA. 100 (5), 2622-2627 (2003).
- Markus, R., et al. Sessile hemocytes as a hematopoietic compartment in Drosophila melanogaster. P Natl Acad Sci USA. 106 (12), 4805-4809 (2009).
- Bretscher, A. J., et al. The Nimrod transmembrane receptor Eater is required for hemocyte attachment to the sessile compartment in Drosophila melanogaster. Biol Open. 4 (3), 355-363 (2015).
- Zettervall, C. J., et al. A directed screen for genes involved in Drosophila blood cell activation. P Natl Acad Sci USA. 101 (39), 14192-14197 (2004).
- Flynt, A., Liu, N., Martin, R., Lai, E. C. Dicing of viral replication intermediates during silencing of latent Drosophila viruses. P Natl Acad Sci USA. 106 (13), 5270-5275 (2009).
- Jovel, J., Schneemann, A. Molecular characterization of Drosophila cells persistently infected with Flock House virus. Virology. 419 (1), 43-53 (2011).
- Stoepler, T. M., Castillo, J. C., Lill, J. T., Eleftherianos, I. A simple protocol for extracting hemocytes from wild caterpillars. J Vis Exp. (69), e4173(2012).
- Sampson, C. J., Williams, M. J. Protocol for ex vivo incubation of Drosophila primary post-embryonic haemocytes for real-time analyses. Methods Mol Biol. 827, 359-367 (2012).
- Nehme, N. T., et al. A model of bacterial intestinal infections in Drosophila melanogaster. PLoS Pathog. 3 (11), 173(2007).
- Drosophila fruit juice egg plates. Cold Spring Harbor Protocols. (9), pdb.rec11113 (2007).
- Ahlers, L. R., Bastos, R. G., Hiroyasu, A., Goodman, A. G. Invertebrate Iridescent Virus 6, a DNA Virus, Stimulates a Mammalian Innate Immune Response through RIG-I-Like Receptors. PLoS One. 11 (11), 0166088(2016).
- Lee, P. Y., Costumbrado, J., Hsu, C. Y., Kim, Y. H. Agarose gel electrophoresis for the separation of DNA fragments. J Vis Exp. (62), (2012).
- Petraki, S., Alexander, B., Bruckner, K. Assaying Blood Cell Populations of the Drosophila melanogaster Larva. J Vis Exp. (105), (2015).
- Figliozzi, R. W., Chen, F., Chi, A., Hsia, S. C. Using the inverse Poisson distribution to calculate multiplicity of infection and viral replication by a high-throughput fluorescent imaging system. Virol Sin. 31 (2), 180-183 (2016).
- Rizki, T. M., Rizki, R. M. Lamellocyte differentiation in Drosophila. larvae parasitized by Leptopilina. Dev Comp Immunol. 16 (2-3), 103-110 (1992).
- Markus, R., Kurucz, E., Rus, F., Ando, I. Sterile wounding is a minimal and sufficient trigger for a cellular immune response in Drosophila melanogaster. Immunol Lett. 101 (1), 108-111 (2005).
- McCormack, R., et al. Perforin-2 Protects Host Cells and Mice by Restricting the Vacuole to Cytosol Transitioning of a Bacterial Pathogen. Infect Immun. 84 (4), 1083-1091 (2016).
- Ozgen, A., et al. Construction and characterization of a recombinant invertebrate iridovirus. Virus Res. 189, 286-292 (2014).
- Jakob, N. J., Muller, K., Bahr, U., Darai, G. Analysis of the first complete DNA sequence of an invertebrate iridovirus: coding strategy of the genome of Chilo iridescent virus. Virology. 286 (1), 182-196 (2001).
- Ghigo, E., Colombo, M. I., Heinzen, R. A. The Coxiella burnetii parasitophorous vacuole. Adv Exp Med Biol. 984, 141-169 (2012).
- Liu, F., et al. Drosophila melanogaster prophenoloxidases respond inconsistently to Cu2+ and have different activity in vitro. Dev Comp Immunol. 36 (3), 619-628 (2012).
- De Gregorio, E., et al. An immune-responsive Serpin regulates the melanization cascade in Drosophila. Dev Cell. 3 (4), 581-592 (2002).
- Kari, B., et al. The raspberry Gene Is Involved in the Regulation of the Cellular Immune Response in Drosophila melanogaster. PLoS One. 11 (3), 0150910(2016).
- Wu, A. R., et al. Quantitative assessment of single-cell RNA-sequencing methods. Nat Methods. 11 (1), 41-46 (2014).
- Buettner, F., et al. Computational analysis of cell-to-cell heterogeneity in single-cell RNA-sequencing data reveals hidden subpopulations of cells. Nat Biotechnol. 33 (2), 155-160 (2015).
- Nevil, M., Bondra, E. R., Schulz, K. N., Kaplan, T., Harrison, M. M. Stable Binding of the Conserved Transcription Factor Grainy Head to its Target Genes Throughout Drosophila melanogaster Development. Genetics. 205 (2), 605-620 (2017).
- Yang, C. P., et al. Transcriptomes of lineage-specific Drosophila neuroblasts profiled by genetic targeting and robotic sorting. Development. 143 (3), 411-421 (2016).
- Jaitin, D. A., et al. Dissecting Immune Circuits by Linking CRISPR-Pooled Screens with Single-Cell RNA-Seq. Cell. 167 (7), 1883-1896 (2016).
- Karaiskos, N., et al. The Drosophila embryo at single-cell transcriptome resolution. Science. 358 (6360), 194-199 (2017).
Access restricted. Please log in or start a trial to view this content.
Nachdrucke und Genehmigungen
Genehmigung beantragen, um den Text oder die Abbildungen dieses JoVE-Artikels zu verwenden
Genehmigung beantragenThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Alle Rechte vorbehalten