È necessario avere un abbonamento a JoVE per visualizzare questo. Accedi o inizia la tua prova gratuita.
Method Article
Estrazione di emociti da larve di Drosophila melanogaster per infezione microbica e l'analisi
In questo articolo
Riepilogo
Questo metodo viene illustrato come visualizzare l'invasione del patogeno nelle cellule di insetto con modelli tridimensionali (3D). Emociti da larve di Drosophila sono stati infettati con agenti patogeni virali o batterici, ex vivo o in vivo. Emociti infetti erano quindi fissi e macchiati per l'imaging con un microscopio confocale e la successiva ricostruzione cellulare 3D.
Abstract
Durante l'infezione patogena di Drosophila melanogaster, emociti gioca un ruolo importante nella risposta immunitaria durante l'infezione. Così, l'obiettivo del presente protocollo è di sviluppare un metodo per visualizzare l'invasione di agente patogeno in un apposito comparto immune di mosche, vale a dire emociti. Utilizzando il metodo presentato qui, fino a 3 × 106 live emociti sono ottenibili da 200 drosofila 3rd instar larve in 30 min per infezione ex vivo . In alternativa, emociti possono essere infetti in vivo attraverso l'iniezione di 3rd instar larve seguita da estrazione di emociti post-all'infezione 24 h. Queste cellule primarie infette sono stati corretti, macchiato e imaging utilizzando la microscopia confocale. Quindi, rappresentazioni in 3D sono stati generati da immagini per mostrare definitivamente l'invasione dell'agente patogeno. Inoltre, può essere ottenuto RNA di alta qualità per qRT-PCR per la rilevazione delle seguenti mRNA patogeno infezione e sufficienti proteine possono essere estratte da queste cellule per l'analisi Western blot. Presi insieme, presentiamo un metodo per la riconciliazione definitiva dell'invasione di agente patogeno e conferma dell'infezione utilizzando tipi di agente patogeno batterico e virale e un metodo efficiente per l'estrazione di emociti per ottenere abbastanza emociti dal vivo dalla drosofila larve per esperimenti di infezione ex vivo ed in vivo .
Introduzione
Drosophila melanogaster è un organismo modello consolidata per lo studio della immunità innata1. Durante la risposta immunitaria innata, emociti gioca un ruolo importante nella risposta alla sfida di agente patogeno. Emociti sono critici per incapsulare i parassiti, oltre ad avere una funzione importante nella lotta contro l'agente patogeno attraverso azione fagocitica durante infezione fungine, virali e batteriche2,3.
Al fine di meglio comprendere la risposta immunitaria innata dell'ospite all'infezione microbica patogena, è importante visualizzare come l'agente patogeno invade le cellule dell'ospite durante l'infezione. Questa visualizzazione contribuisce alla comprensione del meccanismo di invasione. Insieme ai dettagli della localizzazione intracellulare di agente patogeno e la risposta cellulare, questi dati possono fornire indizi circa la risposta dell'ospite all'infezione ed organelli cellulari con cui interagisce il microbo. Così, la ricostruzione del modello 3D dopo formazione immagine di microscopia può essere utile per determinare l'esatta posizione di agenti patogeni e cellule ospiti. In questo studio, abbiamo visualizzato l'invasione della Coxiella burnetii (c. burnetii), l'agente eziologico della febbre Q, una malattia zoonotica che rappresenta una grave minaccia per la salute umana e animale, in primarie emociti di Drosophila . Recentemente, è stato dimostrato che Drosophila sono sensibili al livello di biosicurezza 2 fase di Nine Mile II (NMII) clonare 4 ceppo di c. burnetii e che questo ceppo è in grado di replicare in drosofila4, che indica che Drosofila può essere utilizzato come organismo modello per studiare la patogenesi da c. burnetii .
Gli studi precedenti hanno usato emociti di esaminare la risposta immunitaria innata dell'ospite. Emociti sono stati utilizzati per osservazioni morfologiche5,6,7, analisi morfometrica2,8, fagocitosi analisi2,3, qRT-PCR2 , 9, immunoprecipitazione10,11, analisi immunofluorescente10,12, immunostaining13, immunoblotting3,10, 11 e immunohistochemistry9,14. Anche se le cellule della drosofila S2 sono anche disponibile per vari esperimenti in vitro , immortalizzazione e potenziale pre-esistente infezione virale cambiare loro comportamento15,16. L'utilizzo di cellule primarie al contrario di una linea di cellule immortalizzate, quali cellule S2, consente lo studio della funzione immune innata in un sistema più rappresentativo dell'intero organismo. Inoltre, l'infezione di emociti in vivoprima dell'estrazione, permette alle cellule di interagire con altre proteine ospite ed il tessuto, un vantaggio sopra estrazione di emociti prima dell'infezione ex vivo . Un numero di metodi differenti è stato utilizzato per ottenere un numero sufficiente di emociti in un breve periodo di tempo per mantenere l'emociti vivo8,17,18,19.
In questo studio, presentiamo un metodo per estrarre emociti da Drosophila 3rd instar larve per infezione microbica patogena con da c. burnetii, Listeria monocytogenes (Listeria) o invertebrato iridescente Virus 6 (IIV6). Descriviamo i metodi per le infezioni di emociti sia in vivo ed ex vivo . In vivo- ed ex vivo-emociti infetti sono stati visualizzati con microscopia confocale e utilizzati per creare modelli 3D di invasione da c. burnetii . Inoltre, utilizzando il protocollo di estrazione, ex vivo-emociti infetti sono stati utilizzati per l'espressione genica e proteica saggi. In particolare, per esaminare l'estensione dell'infezione con IIV6 e Listeria, RNA o la proteina totale è stata isolata dalle cellule per qRT-PCR o analisi Western blot. Presi insieme, il protocollo fornisce i metodi per raccogliere rapidamente un numero elevato di emociti da 3rd instar larve e prove che primari emociti, infetto in vivo o ex vivo, sono una piattaforma adatta per microbica agente patogeno infezione studi e analisi applicabili a valle quali microscopia, trascrittomica e proteomica.
Access restricted. Please log in or start a trial to view this content.
Protocollo
1. infezione ex vivo
- Medium e attrezzature
- In condizioni di sterilità, preparare fresco Drosophila emociti isolamento medio (DHIM) contenente di 75% Schneider Drosophila medie con 25% siero bovino fetale (FBS) e filtro sterilizzarlo.
- Strato 2-3 pezzi di pellicola di paraffina di 10 x 10 cm sotto un microscopio stereoscopico.
- Preparare il capillare di vetro. Impostare la stufa capillare estrattore al 55% del massimo. Tirare il tubo capillare di una punta acuminata di circa 10 µm.
- Recupero di informazioni del capillare con olio minerale.
- Assemblare il tubo capillare riempito sul nanoinjector (Figura 1A) e aprire punta fuso tubo capillare rompendo fuori la punta con il forcipe (Figura 1B). Il diametro esterno della punta dovrebbe essere 100 µm per facile assorbimento degli emociti.
- Espellere tanto olio quanto possibile dalla punta tubo capillare, quindi riempire con DHIM. Per una visualizzazione semplice del bordo dell'emolinfa up-preso e l'olio, includono una bolla d'aria tra l'olio e DHIM (Figura 1B').
- Estrazione di emolinfa
- Scegli 3rd instar larve di Drosophila dall'interno muro di cibo fiale delicatamente usando il forcipe e inserirli in un colino di 100 µm (Figura 1C). 3rd instar larve si trovano 3-6 giorni successivi fertile uova da una femmina adulta.
Nota: Questi esperimenti usato il genotipo, w1118; P {w+ mC= Hml-GAL4.Δ} 2, P {w+ mC= UAS-2xEGFP} AH2, poiché emociti da questi animali esprimono la proteina fluorescente verde avanzata (EGFP) per aiutare nella identificazione delle cellule mediante microscopia. - Versare 5 mL di acqua sterile sopra larve e scuotere il filtro per 5 s. posto il filtro sulla cancellazione di attività eliminare l'acqua in eccesso (Figura 1C').
- Trasferire le larve in una microcentrifuga da 1,5 mL. Anestetizzare li con CO2 gas per 5 s (Figura 1D).
- Posizionare le larve sulla pellicola di paraffina sotto lo stereomicroscopio, con il dorso rivolto verso l'alto (Figura 2A).
- Luogo il vetro capillare leggermente sul corpo posteriore larvale per tenerlo in posizione e disturbare la cuticola posteriore aperto utilizzando una pinzetta appuntita (Figura 2B).
- Consentire l'emolinfa di fluire sulla pellicola paraffina (Figura 2C).
- Rendere un pool di emolinfa compreso emociti da larve di 20-50 alla volta sul film di paraffina.
- Prendere l'emolinfa pool utilizzando il vetro capillare su nanoinjector (Figura 2D).
Nota: Ci dovrebbe essere di circa 10-20 µ l di emolinfa. - Espellere l'emolinfa in una provetta da microcentrifuga da 1,5 mL contenente 500 µ l di DHIM (Figura 2E).
- Ripetere i passaggi da 1.2.4) - 1.2.9) per ogni batch di larve.
- Scegli 3rd instar larve di Drosophila dall'interno muro di cibo fiale delicatamente usando il forcipe e inserirli in un colino di 100 µm (Figura 1C). 3rd instar larve si trovano 3-6 giorni successivi fertile uova da una femmina adulta.
- Contare il numero di emociti.
- Pipettare 5 µ l di soluzione di blu di Trypan 0,4% in un tubo del microcentrifuge 0,6 mL a macchiare le cellule morte. Delicatamente mescolare il DHIM ed emociti nel tubo da 1,5 mL utilizzando una pipetta e trasferire 5 µ l di DHIM tra cui emociti al tubo del microcentrifuge 0,6 mL e mescolare delicatamente.
- Pipettare 10 µ l di cellule da miscela 1:1 di Trypan blu: emociti nell'emocitometro.
- Contare il numero di emociti dal vivo che non sono macchiati con Trypan blu in ogni campo 4 angolo dell'hemocytomter e calcolare la concentrazione di emociti per millilitro usando la formula:
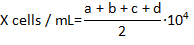
dove X è la concentrazione di emociti live per millilitro; a, b, c e d sono contato il numero di cellule vive (come determinato dall'esclusione del Trypan blu) in ogni 4 dei campi nell'emocitometro. Divisione per 2 il numero totale di cellule contate è dovuto alla diluizione di 1:1 delle cellule con Trypan blue. Le cellule colorate con Trypan blue sono considerate morte.
-
Ex vivo Infezioni
- Determinare il numero di pozzetti di essere seminato con celle basate su punti temporali e biologico replica necessario per ogni esperimento. 5.0 × 104 emociti sono desiderabili per la purificazione di RNA e proteine dopo l'infezione.
- Calcolare il volume di stock di agente patogeno essere diluiti con DHIM per infezione utilizzando la seguente formula:
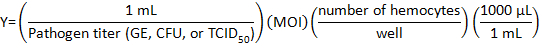
dove molteplicità di infezione (MOI) è il numero di virali o batteri desiderato per ogni cella.
Nota: MOI utilizzato dipende dal singolo esperimento e analisi eseguite. Qui, 10 equivalenti di genoma (GE) / cellula di c. burnetii, 10 CFU/di Listeriao 1 TCID50valore di IIV6 è stato usato. - Preparare 500 µ l di terreno di agente patogeno per ciascun pozzetto di una piastra a 24 pozzetti aggiungendo un volume adeguato di DHIM al volume virale o batterico in un tubo.
- Posto un 12 millimetri rotonda copertura in vetro (spessore n ° 1) in un pozzetto di una piastra a 24 pozzetti.
- Dividere il DHIM compreso gli emociti nei pozzetti di una piastra a 24 pozzetti.
- Aggiungere 500 µ l di terreno di agente patogeno di emociti in un pozzo.
- Centrifugare la piastra a 1.000 x g per 5 min.
- Incubare la piastra per 1 h a 28 ° C. Ogni 15 min, inclinare delicatamente la piastra dalla parte posteriore a anteriore, quindi da sinistra a destra per 5 s a mano.
- Dopo 1 h di passaggio invasione/allegato 1.4.8), delicatamente Pipettare fuori il mezzo di agente patogeno e lavare gli emociti con DHIM fresco e riempirlo con 500 µ l di DHIM fresca.
- Incubare gli emociti infetti per il tempo desiderato. In questi esperimenti, da c. burnetii- o IIV6-infettati emociti vengono incubati per 24 h e Listeria-emociti infetti vengono incubate per 1, 2 o 4 h.
2. infezione in vivo
- Infezione
- Caldo il piatto di agar succo frutta Drosophila a temperatura ambiente per 15 min. piastre sono realizzati come precedentemente descritto20.
- Aggiungere 30 g di agar a 700 mL di acqua e autoclave per 40 min.
- Sciogliere 0,5 g di paraben metilico in 10 mL di etanolo assoluto.
- Aggiungere la soluzione di paraben metilico in 300 mL di succo di frutta concentrato.
- Mescolare il succo concentrato nella soluzione agar sterilizzato nell'autoclave e pipettare 5 mL in 10 × 35 mm piastre Petri rapidamente.
- Dopo le piastre sono raffreddati per 15 min, conservarli a 4 ° C.
- Preparare 3rd instar larve come segue 1.2.1) - 1.2.3).
- Posizionare la pasta di lievito su una piastra di agar. Fare un taglio sottile nella piastra di agar dove le larve possono migrare per evitare la disidratazione (Figura 3A). Montare un ago di tungsteno appuntito di 0,001 mm con che tiene il forcipe usando la pellicola di paraffina (Figura 3B, C).
- Pipettare 50 µ l di alto-titolo mCherry esprimenti -da c. burnetii (5.95 × 109 GE/mL) sulla paraffina della pellicola sotto il microscopio stereo e posizionare le larve nella piscina di batteri.
- Posizionare le larve nella piscina di batteri. Pungere le larve con un ago di tungsteno (Figura 3D). Trasferire le larve su una piastra di agar (Figura 3E).
- Trasferire il restante mezzo agente patogeno sulla piastra di agar e sigillare la piastra con la pellicola di paraffina.
- Mantenere le larve sulla piastra in aria umida fino a quando post-l'infezione tempo desiderato (Figura 3F). In questo esperimento, da c. burnetii-larve infette sono sul piatto per 24 h.
- Caldo il piatto di agar succo frutta Drosophila a temperatura ambiente per 15 min. piastre sono realizzati come precedentemente descritto20.
- Estrazione di emolinfa e placcatura di emociti
- Preparare il mezzo e attrezzatura seguendo passo 1.1).
- Posto un 12 millimetri rotonda copertura in vetro (spessore n ° 1) in un pozzetto di una piastra a 24 pozzetti. Pipettare 500 µ l di DHIM nel pozzo.
- Estrarre l'emolinfa dalle larve infette come segue 1.2.4) - 1.2.8).
- Espellere l'emolinfa in seguito di ben punto 2.2.2).
- Ripetere 2.2.3) e 2.2.4) per più gruppi di larve.
- Centrifugare la piastra a 1.000 x g per 5 min.
3. visualizzazione
- Fissazione e colorazione
- Dopo aver consentito l'emociti di stabilirsi sul vetro di copertura rotondo nel pozzo, rimuovere delicatamente il mezzo da ogni pozzetto.
- Delicatamente aggiungere 200 µ l di paraformaldeide al 4% (PFA) in ciascun pozzetto di emociti depositati. Incubare gli emociti per 20 min a temperatura ambiente.
- Rimuovere il 4% PFA e delicatamente aggiungere 200 µ l di PBS contenente 0,1% Triton X-100 e 1% albumina di siero bovino (BSA) in ciascun pozzetto. Incubare gli emociti per 10 min a temperatura ambiente.
- Rimuovere il PBS e delicatamente aggiungere 200 µ l di 1 × 4', 6-diamidino-2-phenylindole (DAPI) in ciascun pozzetto. Incubare gli emociti per 10 min al buio a temperatura ambiente.
- Rimuovere la soluzione DAPI e aggiungere delicatamente PBS in ciascun pozzetto. Incubare gli emociti per 5 min a temperatura ambiente.
- Goccia 10 µ l del mezzo di montaggio antifade su un vetrino per microscopio.
- Dopo aver rimosso il PBS da ogni pozzetto, è necessario rimuovere il vetro di copertura dalla piastra 24 pozzetti utilizzando forcipe ammenda-aguzzo. Posizionare delicatamente il vetro di copertura sul supporto montaggio antifade su vetrino, con gli emociti rivolto verso il basso.
- Lasciar asciugare inserendolo nella notte scura.
- Formazione immagine confocal
- Configurare il microscopio confocale per formazione immagine di DAPI, EGFP e mCherry tre di colore. Utilizzare la seguente impostazione: eccitazione DAPI (ex) 405 nm, emissione (em) 415-480 nm; EGFP ex 488 nm, em 493-564 nm; mCherry ex 587 nm, em 597-700 nm.
- Posizionare il campione sul microscopio e messa a fuoco sul campione utilizzando un 63 X / 1.4 obiettivo apertura numerica (NA). Individuare gli emociti desiderati nel campo di vista per l'imaging.
- Regolare i guadagni di potenza e rilevatore laser per ottenere esposizione appropriata del campione. Verifica z-piani multipli per garantire che il livello di esposizione è appropriato per lo spessore dell'intero campione.
- Trovare la posizione superiore e inferiore sull'asse z di un intero emociti. Impostare queste posizioni come le posizioni di inizio e di fine per il sezionamento di z.
- Utilizzare solo scansione zoom all'immagine l'area che contiene l'emociti. Spesso vengono utilizzati fattori di zoom di 3x.
- Raccogliere la serie di immagini a risoluzione appropriata come 1024 x 1024 pixel nel piano x-y e 0,3 µm spaziatura nella dimensione z.
- Ricostruzione del modello 3D
Nota: Il software Open source esistente che esegue molte delle funzioni descritte di seguito per la ricostruzione del modello 3D.- Importare il file di serie z-sezionato immagine nel software associato con il microscopio confocale per la ricostruzione del modello 3D.
- Selezionare una cella risultati co-localizzazione dei nuclei macchiato con DAPI e mCherry esprimendo da c. burnetii in un EGFP esprimendo emociti. Ritagliare l'immagine in serie per contenere solo la singola cella.
- Selezionare l'opzione Visualizzatore 3D che ricostruisce il modello 3D utilizzando pre-confezionati algoritmo del software. Scegliere il tipo di rappresentazione 3D tra Blend, superficie e opzioni miste desiderato. In questo metodo, emociti, nei nuclei e da c. burnetii sono illustrati utilizzando modelli di superficie.
- Osservare la cella ricostruita 3D da diverse angolazioni di visualizzazione tenendo premuto il pulsante del mouse e trascinando il cursore sullo schermo. Regolare l'orientamento della cella e la posizione della sorgente luminosa modellata per ottimizzare l'immagine. Esistono altre opzioni per opacità, minimo e massimo soglia, speculare, Ambient, Shineness e Gamma per ottimizzare l'immagine.
- Prendere le sezioni trasversali attraverso il modello utilizzando i comandi di clipping e sezionamento per visualizzare il contenuto interno degli emociti.
4. l'applicazione per l'analisi genica e/o proteine
- Dopo l'infezione con IIV6 e Listeria, lisare cellule per successive qRT-PCR o analisi Western blot come precedentemente descritto21 e le istruzioni del produttore riportato di seguito.
Nota: Consultare la Tabella dei materiali per i primers per qRT-PCR e gli anticorpi per la macchia occidentale. - Analizzare i prodotti di PCR di elettroforesi del gel dell'agarosi, come descritto in precedenza22, per garantire la corretta lunghezza del prodotto amplificato.
Access restricted. Please log in or start a trial to view this content.
Risultati
Per raccogliere vivere emociti per infezione ex vivo , fino a 3 × 106 emociti sono stati estratti da 200 drosofila 3rd instar larve. Per sviluppare il nostro metodo, una serie di diverse tecniche sono stata tentata. Dissezione larvale individuale sarebbe prendere fino a 1,5 h, e una media di ~ 8000 cellule sono state ottenute utilizzando questo metodo18, la maggior parte dei quali non erano viva alla fine della raccolta. Suc...
Access restricted. Please log in or start a trial to view this content.
Discussione
Per comprendere meglio come cellule dell'ospite infettate, è importante chiarire la localizzazione dell'agente patogeno nelle cellule, soprattutto quando si sperimenta il patogeno non precedentemente testato e cella tipo combinazioni4. Mentre studiando la cascata di risposta cellulare dopo l'infezione può indicare invasione produttivo dell'agente patogeno, la combinazione dei dati di risposta cellulare e imaging è essenziale per dimostrare l'infezione e l'invasione dell'agente patogeno. Mentre ...
Access restricted. Please log in or start a trial to view this content.
Divulgazioni
Gli autori dichiarano di non avere nessun concorrenti interessi finanziari.
Riconoscimenti
Siamo grati al Dr. Robert Heinzen per fornire scorte di mCherry-esprimendo il burnetii della coxiella. Ringraziamo il Dr. Luis Teixeira per la fornitura di invertebrati marini iridescente virus 6 e il centro di Stock di Bloomington per fornire scorte volare. Questo progetto è stato finanziato in parte dalla concessione di NIH R00 AI106963 (al A.G.G) e Washington State University.
Access restricted. Please log in or start a trial to view this content.
Materiali
| Name | Company | Catalog Number | Comments |
| Schneider's Drosophila Medium | Thermo Fisher Scientific (Gibco) | 21720024 | 1.1.1), 2.1.2) |
| Fetal Bovine Serum | GE Healthcare Life Sciences (HyClone) | SH30070.03HI | 1.1.1), 2.1.2) |
| Filter (0.22 µL) | RESTEK | 26158 | 1.1.1) |
| Strainer (100 µm) | Greiner bio-one | 542000 | 1.2.1), 2) |
| Stereo microscope | Amscope | SM-1BSZ-L6W | 1.2), 2) |
| Glass capillary | Fisher Scientific | 21-171-4 | 1.1), 1.2), 2) |
| Capillary puller | Narishige International USA, Inc. | PC-10 | 1.1.3) |
| Mineral oil | Snow River Products | 1.1.4) | |
| Nanoinjector | Drummond Scientific Company | 3-000-204 | 1.1), 1.2), 2.2) |
| Forceps | VWR | 82027-402 | 1.1.5), 1.2), 2), 3.1.7) |
| CO2 delivery apparatus | Genesee Scientific | 59-122BC | 1.2), 2) |
| Trypan Blue | Thermo Fisher Scientific (Gibco) | 15250061 | 1.3) |
| Hemocytometer | Hausser Scientific | 3100 | 1.3) |
| 24 well plate | Greiner bio-one | 662160 | 1.4), 2.2) |
| Coxiella burnetii - mCherry | Dr. Heinzen, R. | 1.4), 2.2) | |
| Drosophila fruit juice plates | Cold Spring Harbor Protocols | 2.1) http://cshprotocols.cshlp.org/content/2007/9/pdb.rec11113.full | |
| Agar | Fisher Bioreagents | BP1423-500 | 2.1.1.1) |
| Methyl paraben | Amresco | 0572-500G | 2.1.1.2) |
| Absolute ethanol | Fisher Bioreagents | BP2818-500 | 2.1.1.2) |
| Welch's 100% Grape juice frozen concentrate, 340 mL | Amazon | B0025UJVGM | 2.1.1.3) |
| Petri dishes, 10 x 35 mm | Fisher Scientific | 08-757-100A | 2.1.1.4) |
| Microscope cover glass | Fisher Scientific | 12-545-80 | 1.4.4), 2.2.2) |
| Yeast, Bakers Dried Active | MP Biomedicals | 0210140001 | 2.1) Add 2 parts of water to 1 part of yeast (v/v) |
| Tungsten needle | Fine Science Tools | 10130-20 | 2.1) |
| Holding forceps | VWR | HS8313 | 2.1) |
| Paraformaldehyde | Fisher Scientific | FLO4042-500 | 3.1.3) |
| Triton X-100 | Fisher Scientific | BP151-500 | 3.1.3) |
| Bovine Serum Albumin | Fisher Scientific | BP9706-100 | 3.1.3) |
| 4',6-diamidino-2-phenylindole | Thermo Fisher Scientific | 62247 | 3.1.4) |
| Antifade mounting medium | Thermo Fisher Scientific | P36930 | 3.1.6) |
| Confocal microsope | Leica | TCS SP8-X White Light Confocal Laser Scanning Microscope | 3.2) |
| 3D imaging reconstruction software | Leica | LASX with 3D visualization module | 3.3) |
| Microscope slides | Fisher Scientific | 12-552-3 | 3.1.6) |
| Invertebrate iridescent virus 6 (IIV6) | Dr. Teixeria, L. | 4) PLoS Biol, 6 (12), 2753-2763, doi: 10.1371/journal.pbio.1000002, (2008) | |
| Listeria monocytogenes | ATCC | strain: 10403S | 4) Listeria monocytogenes strain 10403S (Bishop and Hinrichs, 1987) was grown in Difco Brain-heart infusion (BHI) broth (BD Biosciences) containing 50 µg/ml streptomycin at 30 °C. |
| DNase I | Thermo Fisher Scientific(Invitrogen) | 18068015 | gDNA degradation |
| cDNA Synthesis Kit | Bio-Rad | 1708891 | cDNA synthesis |
| IIV6_193R_F | IDT | qRT-PCR, 5'- TCT TGT TTT CAG AAC CCC ATT -3' | |
| IIV6_193R_R | IDT | qRT-PCR, 5'- CAC GAA GAA TGA CCA CAA GG -3' | |
| RpII_qRTPCR_fwd | SIGMA-ALDRICH | qRT-PCR, 5'- GAA GCG TTT CTC CAA ACG -AG | |
| RpII_qRTPCR_rev | SIGMA-ALDRICH | qRT-PCR, 5'- TTG AGC GTA AGC ATC ACC -TG | |
| SYBR Green qRT-PCR reagent | Thermo Fisher Scientific | K0251, K0252, K0253 | qRT-PCR |
| Real-Time PCR System | Thermo Fisher Scientific | 4351107, 7500 Software v2.0 | qRT-PCR |
| Anti-Listeria monocytogenes antibody | abcam | ab35132 | Western blot |
| Anti-Actin antibody produced in rabbit | SIGMA-ALDRICH | A2066 | Western blot |
| Anti-Rabbit IgG (H+L), HRP Conjugate | Promega | W4011 | Western blot |
Riferimenti
- Hoffmann, J. A. The immune response of Drosophila. Nature. 426 (6962), 33-38 (2003).
- Regan, J. C., et al. Steroid hormone signaling is essential to regulate innate immune cells and fight bacterial infection in Drosophila. PLoS Pathog. 9 (10), 1003720(2013).
- Yano, T., et al. Autophagic control of listeria through intracellular innate immune recognition in Drosophila. Nat Immunol. 9 (8), 908-916 (2008).
- Bastos, R. G., Howard, Z. P., Hiroyasu, A., Goodman, A. G. Host and Bacterial Factors Control Susceptibility of Drosophila melanogaster to Coxiella burnetii Infection. Infect Immun. 85 (7), (2017).
- Kacsoh, B. Z., Schlenke, T. A. High hemocyte load is associated with increased resistance against parasitoids in Drosophila suzukii, a relative of D. melanogaster. PLoS One. 7 (4), 34721(2012).
- Tsuzuki, S., et al. Switching between humoral and cellular immune responses in Drosophila. is guided by the cytokine GBP. Nat Commun. 5, 4628(2014).
- Kurucz, E., et al. Definition of Drosophila. hemocyte subsets by cell-type specific antigens. Acta Biol Hung. 58, Suppl 95-111 (2007).
- Neyen, C., Bretscher, A. J., Binggeli, O., Lemaitre, B. Methods to study Drosophila immunity. Methods. 68 (1), 116-128 (2014).
- Arefin, B., et al. Apoptosis in Hemocytes Induces a Shift in Effector Mechanisms in the Drosophila. Immune System and Leads to a Pro-Inflammatory State. PLoS One. 10 (8), 0136593(2015).
- Rus, F., et al. Expression pattern of Filamin-240 in Drosophila blood cells. Gene Expr Patterns. 6 (8), 928-934 (2006).
- Kurucz, E., et al. Hemese, a hemocyte-specific transmembrane protein, affects the cellular immune response in Drosophila. P Natl Acad Sci USA. 100 (5), 2622-2627 (2003).
- Markus, R., et al. Sessile hemocytes as a hematopoietic compartment in Drosophila melanogaster. P Natl Acad Sci USA. 106 (12), 4805-4809 (2009).
- Bretscher, A. J., et al. The Nimrod transmembrane receptor Eater is required for hemocyte attachment to the sessile compartment in Drosophila melanogaster. Biol Open. 4 (3), 355-363 (2015).
- Zettervall, C. J., et al. A directed screen for genes involved in Drosophila blood cell activation. P Natl Acad Sci USA. 101 (39), 14192-14197 (2004).
- Flynt, A., Liu, N., Martin, R., Lai, E. C. Dicing of viral replication intermediates during silencing of latent Drosophila viruses. P Natl Acad Sci USA. 106 (13), 5270-5275 (2009).
- Jovel, J., Schneemann, A. Molecular characterization of Drosophila cells persistently infected with Flock House virus. Virology. 419 (1), 43-53 (2011).
- Stoepler, T. M., Castillo, J. C., Lill, J. T., Eleftherianos, I. A simple protocol for extracting hemocytes from wild caterpillars. J Vis Exp. (69), e4173(2012).
- Sampson, C. J., Williams, M. J. Protocol for ex vivo incubation of Drosophila primary post-embryonic haemocytes for real-time analyses. Methods Mol Biol. 827, 359-367 (2012).
- Nehme, N. T., et al. A model of bacterial intestinal infections in Drosophila melanogaster. PLoS Pathog. 3 (11), 173(2007).
- Drosophila fruit juice egg plates. Cold Spring Harbor Protocols. (9), pdb.rec11113 (2007).
- Ahlers, L. R., Bastos, R. G., Hiroyasu, A., Goodman, A. G. Invertebrate Iridescent Virus 6, a DNA Virus, Stimulates a Mammalian Innate Immune Response through RIG-I-Like Receptors. PLoS One. 11 (11), 0166088(2016).
- Lee, P. Y., Costumbrado, J., Hsu, C. Y., Kim, Y. H. Agarose gel electrophoresis for the separation of DNA fragments. J Vis Exp. (62), (2012).
- Petraki, S., Alexander, B., Bruckner, K. Assaying Blood Cell Populations of the Drosophila melanogaster Larva. J Vis Exp. (105), (2015).
- Figliozzi, R. W., Chen, F., Chi, A., Hsia, S. C. Using the inverse Poisson distribution to calculate multiplicity of infection and viral replication by a high-throughput fluorescent imaging system. Virol Sin. 31 (2), 180-183 (2016).
- Rizki, T. M., Rizki, R. M. Lamellocyte differentiation in Drosophila. larvae parasitized by Leptopilina. Dev Comp Immunol. 16 (2-3), 103-110 (1992).
- Markus, R., Kurucz, E., Rus, F., Ando, I. Sterile wounding is a minimal and sufficient trigger for a cellular immune response in Drosophila melanogaster. Immunol Lett. 101 (1), 108-111 (2005).
- McCormack, R., et al. Perforin-2 Protects Host Cells and Mice by Restricting the Vacuole to Cytosol Transitioning of a Bacterial Pathogen. Infect Immun. 84 (4), 1083-1091 (2016).
- Ozgen, A., et al. Construction and characterization of a recombinant invertebrate iridovirus. Virus Res. 189, 286-292 (2014).
- Jakob, N. J., Muller, K., Bahr, U., Darai, G. Analysis of the first complete DNA sequence of an invertebrate iridovirus: coding strategy of the genome of Chilo iridescent virus. Virology. 286 (1), 182-196 (2001).
- Ghigo, E., Colombo, M. I., Heinzen, R. A. The Coxiella burnetii parasitophorous vacuole. Adv Exp Med Biol. 984, 141-169 (2012).
- Liu, F., et al. Drosophila melanogaster prophenoloxidases respond inconsistently to Cu2+ and have different activity in vitro. Dev Comp Immunol. 36 (3), 619-628 (2012).
- De Gregorio, E., et al. An immune-responsive Serpin regulates the melanization cascade in Drosophila. Dev Cell. 3 (4), 581-592 (2002).
- Kari, B., et al. The raspberry Gene Is Involved in the Regulation of the Cellular Immune Response in Drosophila melanogaster. PLoS One. 11 (3), 0150910(2016).
- Wu, A. R., et al. Quantitative assessment of single-cell RNA-sequencing methods. Nat Methods. 11 (1), 41-46 (2014).
- Buettner, F., et al. Computational analysis of cell-to-cell heterogeneity in single-cell RNA-sequencing data reveals hidden subpopulations of cells. Nat Biotechnol. 33 (2), 155-160 (2015).
- Nevil, M., Bondra, E. R., Schulz, K. N., Kaplan, T., Harrison, M. M. Stable Binding of the Conserved Transcription Factor Grainy Head to its Target Genes Throughout Drosophila melanogaster Development. Genetics. 205 (2), 605-620 (2017).
- Yang, C. P., et al. Transcriptomes of lineage-specific Drosophila neuroblasts profiled by genetic targeting and robotic sorting. Development. 143 (3), 411-421 (2016).
- Jaitin, D. A., et al. Dissecting Immune Circuits by Linking CRISPR-Pooled Screens with Single-Cell RNA-Seq. Cell. 167 (7), 1883-1896 (2016).
- Karaiskos, N., et al. The Drosophila embryo at single-cell transcriptome resolution. Science. 358 (6360), 194-199 (2017).
Access restricted. Please log in or start a trial to view this content.
Ristampe e Autorizzazioni
Richiedi autorizzazione per utilizzare il testo o le figure di questo articolo JoVE
Richiedi AutorizzazioneThis article has been published
Video Coming Soon