Method Article
非正規の開いたリーディング ・ フレームから翻訳された蛋白質を発表する OpenProt データベースを利用した質量分析を用いたプロテオミクス解析
要約
OpenProt は、真核生物ゲノムのコードするモデルを適用自由にアクセス可能なデータベースです。ここで、質量データセットを問い合わせるときの OpenProt データベースを使用するためのプロトコルを紹介します。プロテオーム解析のためのデータベース OpenProt を使用して小説や以前検出できないタンパク質の探索できます。
要約
ゲノムの注釈はプロテオーム風景の輪郭を描画として今日のプロテオーム研究の中心です。オープンの伝統的なモデル フレーム (ORF) 注釈を読んで任意の 2 つの条件を課す: 100 コドンとトラン スクリプトごと単一 ORF の最小の長さ。ただし、成長の多くの研究報告の容疑者以外のコーディングから蛋白質の表現領域、現在のゲノムの注釈の精度に挑戦します。蛋白質が発見されたこれらの小説エンコードか以内非コード Rna、5' 3' 非翻訳領域 (UTRs) の Mrna、重なり合ったり ORF の代わりに知られているコーディング シーケンス (CD)。OpenProt は、トラン スクリプトあたり複数の ORFs の注釈を許可する真核生物のゲノムにコードするモデルを適用する最初のデータベースです。OpenProt が自由にアクセスできる 10 種のタンパク質配列のカスタム ・ ダウンロードを提供しています。OpenProt プロテオーム実験用データベース新規タンパク質の探索を有効にして真核生物遺伝子のコードする性質を強調します。(全ての予測蛋白質) OpenProt データベースのサイズは、実質的な分析のアカウントに取られる必要があります。ただし、適切な偽の発見率 (FDR) 設定または制限された OpenProt データベースを使用して、ユーザーは、プロテオーム風景のより現実的なビューを得ることが。全体的にみて、OpenProt、プロテオームの発見を促進する自由に利用できるツールです。
概要
過去十年にわたって質量分析法 (MS-) 基づくプロテオミクス真核細胞1,2,3,4、5のプロテオームを解読する黄金の技術となっています。このメソッドは、可能性6,7,8の範囲を示す参照タンパク質シーケンス データベースを生成する現在のゲノムの注釈に依存します。しかし、ゲノムの注釈は、ORF 注釈、100 コドンとトラン スクリプト9,10につき単一 ORF の最小の長さなど任意の条件を保持します。研究数の増加は、現在のアノテーション モデルに挑戦し、真核生物ゲノム8,11,12,13、unannotated 機能 ORFs の発見を報告 14。これらの新規タンパク質でエンコードされた容疑者は非コード Rna がある、非翻訳領域 (UTR) の Mrna、や代替フレームの標準的なコーディング シーケンス (Ccd) の重複を 5' または 3' で。これらの発見のほとんどは、偶然されているが、彼らは現在のゲノムの注釈の注意点、および真核生物遺伝子8のコードする性質を示しています。
ここでは、MS ベース プロテオミクス OpenProt データベースの使用を強調表示します。OpenProt は、真核生物のトランスクリプトームのコードするアノテーション モデルを保持するために最初のデータベースです。Www.openprot.org15で自由に利用可能です。これらの割合だろうと予言した Orf ランダムで非機能的な理由 OpenProt 累積信頼を高めるための実験的で機能的な証拠であります。実験的な証拠には、(MS) による蛋白質の表現および翻訳の証拠 (リボソームプロファイリング) の15が含まれます。(アプローチのように妄想) とタンパク質 orthology と機能ドメイン予測15機能的な証拠が含まれます。
OpenProt では、カスタムメイドのデータベースにのみよくサポートされている蛋白質を含んでいるから、複数のデータベースをダウンロードする可能性を提供しています。ここでは、OpenProt データベースを使用するためのパイプラインを紹介します、実験の目的を考慮した選択するデータベースへの洞察を提供します。ここで紹介するプロテオミクス解析パイプラインは、オープン アクセスと利用簡単だが、データベースは任意のワークフロー16,17,18を扱うことができます銀河フレームワークによってサポートされます。プロテオーム風景の包括的なビューを提供するさんによる OpenProt データベースで検出された新規タンパク質に関するさらなる情報の収集、プロテオミクスとバイオ マーカーの発見を促進 OpenProt ウェブサイトを使用する方法を紹介します現在の方法よりもより体系的な方法です。
このプロトコルは MS データセットを尋問するとき OpenProt データベース15の使用を強調表示します。それは設計を見直さない実験自体は、徹底的にされている見直し他20,21,22。完全にオープン ソースを維持するために、プロトコルは自由に利用できる (補足材料 S1-S4) です。読みやすくするため OpenProt とこここのプロトコルで使用されるすべての用語は、表 1に定義されます。
プロトコル
1. OpenProt データベースのダウンロード
注: RNA シーケンス データに基づくカスタム データベースなども取得でき、このプロトコルの 2 番目のセクションでの手順を詳しく説明。カスタム データベースが必要な場合は、次のセクションにスキップしてください。
- OpenProt のウェブサイトに行く: www.openprot.org し、トップページ メニューからのリンクを使用してダウンロード ページを開きます。
- 分析実験データに基づく興味の種をクリックします。
- 目的タンパク質の型をクリックします。
注: OpenProt は、3 つの分類をご利用いただけます: RefProt、アイソ フォームと AltProt。図 1のように、このパラメーターは研究目的に応じて異なります。- RefProt 一人でのみ知られている蛋白質を含んでいるファイルを生成するをクリックします。
- AltProt とアイソ フォームのみ新規蛋白質 - 知られている蛋白質 (アイソ フォーム) のいずれかの新規アイソ フォームを含んでいるまたは代替 ORF (AltProts) のコード ファイルを生成するをクリックします。OpenProt が 30 コドン15の最小の ORF 長さを強制することに注意してください。
- AltProts、アイソ フォームと RefProts OpenProt データベース - 既知および新規のタンパク質に存在すべてのタンパク質の型を含むファイルを生成するをクリックします。
- 利用可能な場合は、シーケンスを描画するタンパク質から注釈をクリックします。
注: OpenProt は複数のアノテート アイテムを組み合わせることで包括的なプロテオーム風景を提供しています。トランスクリプトーム注釈最小限の重複; があります。したがって、選択したアノテーションは大幅に可視化プロテオミクス プロファイル15,23に影響を与えます。 - タンパク質の考察に必要な証拠をサポートのレベルをクリックします。図 1のように、このパラメーターは研究目的に応じて異なります。
- 最小検出された 2 つのユニークなペプチドの一番自信のタンパク質だけを含むファイルを生成するをクリックします。
注: 2 つのユニークなペプチドの基準は現在プロテオミクス蛋白質の表現のためのゴールド スタンダードと見なされます。実験の目的を知られており、よくサポートされているタンパク質を検出する場合は、このパラメーターの使用をお勧めします。 - 検出された 1 つのユニークなペプチドの最小OpenProt によって再分析質量分析実験の間で少なくとも 1 回に見られている蛋白質を含んでいるファイルを生成するをクリックします。
注: これにより、AltProts とそれらのいくつかが 1 つだけユニークなトリプシン ペプチド8,11に含まれている確率の短い長さの検討のため。 - すべての予測OpenProt 予測のすべてを含むファイルを生成するをクリックします。
注: この設定はのみお勧め場合は実験目的蛋白質 (図 1) を発見することです。7,15以下の説明に従って適応解析パイプラインの検索空間の呼び出し後の実質的な増加。
- 最小検出された 2 つのユニークなペプチドの一番自信のタンパク質だけを含むファイルを生成するをクリックします。
- 目的のファイル形式をダウンロードするをクリックします。プロテオーム解析 (タンパク質) Fasta ファイルを選択します。Readme ファイルには、ファイル形式にすべての必要な情報が含まれています。
2. カスタム OpenProt データベースのダウンロード
注: このセクションは、カスタム データベースを取得する方法を説明します。カスタム データベースが必要ない場合は、次のセクションに進んでください。
- OpenProt ウェブサイト (www.openprot.org) に移動し、ページのトップ メニューからのリンクを使用して検索ページを開きます。
- 分析した実験的データに基づく興味の種をクリックします。
- 遺伝子または関心のトラン スクリプトのリストを入力します。
- 遺伝子のリストを使用する場合は、遺伝子のクエリ ボックスに入力します。
- 成績証明書のリストを使用する場合は、トラン スクリプトのクエリ ボックスに入力します。
- 目的のデータベースに適用するボックスをチェックします。
- 蛋白 OpenProt でサポートされているすべての型を含むテーブルを取得するための任意のボックスをクリックしてしないで: RefProt、アイソ フォームと AltProts。
- MS によって少なくとも 1 回検出されているタンパク質 (RefProts、アイソ フォームと AltProts) のすべてのタイプを含むテーブルを取得する実験的証拠が付いている蛋白質のみを表示をクリックしておよび/または翻訳の証拠は、リボソームから収集されています。データをプロファイリングします。
- 同様に、地図をMS で検出したタンパク質だけまたは MS またはそれぞれのプロファイリング リボソームによって少なくとも 1 回検出されているタンパク質のすべてのタイプを含むテーブルを取得するリボソーム プロファイリングによって検出されたタンパク質だけを表示をクリックします。
- 地図を表示AltProts のみまたはそれぞれ AltProts またはのみアイソ フォームのみを含むテーブルを取得するアイソ フォームだけを表示をクリックします。
- 両方AltProts のみを示しアイソ フォームのみ両方の種類のタンパク質が含まれているテーブルを取得するをクリックします。
注: すべてのフィルターの組み合わせが可能です。
- すべての必要なパラメーターを設定すると、検索をクリックします。表出力は、検索クエリのフィールドの下に表示されます。
- 出力テーブルの右上隅にあるFasta のダウンロードボタンをクリックします。遺伝子または成績証明書の照会されたリストから生じるすべての蛋白質を含んでいる Fasta ファイルが生成されます。
- OpenProt が計算上の理由から、する 2,000 の要素の最大値を保持することに注意してください、時に (遺伝子または成績証明書) を照会します。いくつかの fasta の生成し、(下記のとおり) 連結; が発生した場合、その制限を超えるリストまたは単に OpenProt データベース全体をダウンロードし、必要に応じて、得られたファイル フィルターします。
- 遺伝子または成績証明書の全一覧を bin 以下 2,000 エントリのリストに。各サブのリスト (手順 3.3 から 3.6) 上記 Fasta ファイルをダウンロードしてください。
- Https://usegalaxy.eu/ 欧州銀河インスタンス (またはプロテオミクス ツールが利用可能な他のインスタンス) にログインします。
- 新たな歴史を作成し、画面の左上にアップロードのロゴをクリックしてすべてのダウンロード OpenProt データベース (1 つの遺伝子や成績証明書の下位リスト) をインポートします。
- Fasta ファイルの結合とフィルターの一意のシーケンスツールを使用しては、GalaxyP 開発者 (https://github.com/galaxyproteomics/) によって開発されました。すべて Fasta をマージオプションを選択し、すべてのインポートされた OpenProt データベースを入力します。
注: 各ツールは画面の左側にある検索ボックスを使用して検索することができます。 - シーケンスの単一性を評価し、OpenProt の識別子の解析ルールをコピーする加盟のみオプションを選択 (>(.*) \ |)、[実行] をクリックします。
- 冗長でない今、画面の右側にある [ヒストリ] パネルに表示されるすべてのファイルが一意の Fasta ファイルに連結されたことに注意してください。これは、作業データベースを構成します。
3. データベース処理
注: さあ今から銀河のプラットフォームを使用するが、プロテオームの他のソフトウェアに同じ原則を適用できます。
- Https://usegalaxy.eu/ 欧州銀河インスタンス (またはプロテオミクス ツールが利用可能な他のインスタンス) にログインします。
- 新たな歴史を作成し、画面の左上にアップロードのロゴをクリックしてダウンロードした OpenProt のデータベースをインポートします。
- [ワークフロー] ページに移動し、中央のパネルの左上にアップロードのロゴをクリックしてデータベース処理ワークフロー (補足材料 S1) をインポートします。
- ワークフローを実行をクリックし、入力としてインポートする OpenProt データベースを選択します。
注: このワークフローでは、OpenProt fasta を CRAPome リポジトリが追加され、おとりシーケンス (逆シーケンス)24を生成します。シャッフルおとりリストが必要な場合は、DecoyDatabase ツールでこのパラメーターを変更することによって行うことができます。 - 得られた Fasta ファイルを変更すると、何かを意味します。データベースはプロテオミクス解析に使用する準備ができています。
4. 質量分析ファイルの準備
注: 銀河インスタンスで使用可能のプロテオミクス ツールのほとんどは、mzML 形式を使用、ペプチド検索エンジン重心モードでデータを好みます。
- ProteoWizard スイートから自由に利用可能な MSConvert ツールを開き、分析25にデータ ファイルをアップロードします。
- 出力と mzML を目的のファイル形式のディレクトリを選択します。
- MS1 と MS2 レベルのウェーブレット ベースのアルゴリズム (CWT) を使用してフィルターを選ぶピークを設定し、変換26を開始します。
5. ペプチドおよびタンパク質の同定/数量
注: パイプラインのこの部分は、汎用性と使いやすいフレームワーク18OpenMS 組曲ツールを使用します。
- Https://usegalaxy.eu/ 欧州銀河インスタンス (またはプロテオミクス ツールが利用可能な他のインスタンス) にログインします。
- 新しい履歴を作成し、以前に作成したデータベース (ステップ 3.5) をドラッグ アンド ドロップでこの新たな歴史に転送します。
- アップロード画面の左上にあるロゴをクリックして、変換された mzML データ ファイル (手順 4.3) をインポートします。
- [ワークフロー] ページに移動し、中央のパネルの左上にアップロードのロゴをクリックして、目的のワークフローをインポートします。
注: MS の実験は、異なる目的の最終的な出力に基づいて設計されます。ワークフローは、2 つの頻繁にデザイン、ここに表示されます: 安定同位体標識 (SIL) に基づくタンパク質の同定とプロテインの定量。しかし、銀河のインスタンスには、プロテオーム解析27,28の他の種類をサポートする他の多くのツールが含まれています。- タンパク質同定設計の補助材料 S2で提供されるワークフローをインポートします。このワークフローを使用する場合くださいファイル (手順 4.2) 変換時の zlip 圧縮を使用しません。
- 安定同位体ラベル デザインに基づくタンパク質定量、補足材料 S3で提供されるワークフローをインポートします。
- ワークフローを実行を選択し、別のパラメーターを確認します。
- 入力、およびデータベース Fasta ファイルとして以前に作成したデータベース (ステップ 3.5) としてインポートされた mzML データ ファイルを選択します。
- ワークフローは、X を使用して!タンデムの検索エンジン、X のインポート!タンデム デフォルト構成ファイル (補足材料 S4で提供される)29画面の左上にアップロードのロゴをクリックしています。
- ワークフローは、複数の検索エンジンを使用する (MS-GF + と X!タンデム)。他の検索エンジンを追加または単に追加またはワークフロー30,31からツールを削除して 1 つを選択します。
注: 複数の検索エンジンを使用してお勧めします感性と分析32の感度を増加すると。 - OpenProt データベース全体を使用する場合、サイズの大幅な増加を対応するために、厳格な FDR15を使用します。既定では、指定されたワークフローは 0.001% の設定 FDR、OpenProt データベース全体の使用のために十分な。他のデータベースは目的の値にすることで編集できます。
注: 必ず使用される質量分析計によってさまざまなツールや実験的プロトコル (前駆物質イオンとフラグメントのエラー、修正、変数の変更、酵素など) のパラメーターを適応してください。
- 必要に応じて、[ヒストリ] パネルから選択したステップをクリックし、下に表示される保存のロゴをクリックしてストレージまたは品質管理分析のワークフローの各ステップの出力をダウンロードします。
6. 品質管理
注: MS ベース プロテオミクス各ステップが再現可能な結果を生成するように最適化する必要がある複雑なプロセスの結果なので、品質管理は33のワークフローに必要な手続きです。
- いくつかの指標は、パフォーマンス、ペプチド スペクトル マッチ (PSM)、識別されたペプチッドおよび蛋白質の数の数などの一般的なベンチマークです。このようなメトリックを提供する (図 2の緑色で示される) IDFilter 出力ファイル情報ツールを実行します。
- 特に大規模なデータセットの場合と、すべての識別に該当しない新規タンパク質のレポートは慎重に評価常にする必要があります。タンパク質スコア、シーケンス カバレッジ、および発見を支援するスペクトルの検査は重要です。OpenMS フレームワークから TOPPview ツールを使用してこれを行うそれは自由に利用できるとも18,34,35です。
7. OpenProt データベースのマイニング
注: OpenProt (新規アイソ フォームの AltProts と II_ の IP_ から始まる加盟番号) によって予想される新規蛋白質の自信を持って同定が行われた後生物学的詳細は OpenProt ウェブサイト15から収集できます。
- OpenProt のウェブサイトに行く: www.openprot.org とトップ メニューでリンクを使用して検索ページを開く。
- (1 つの蛋白質が識別されたと同じ) の興味の種をクリックし、[クエリ ボックスで蛋白質蛋白質加盟番号を入力します。
- 検索をクリックし、照会されたタンパク質に関する基本的な情報を含むテーブルが表示されます。テーブル機能: ドメインとタンパク質の予測 (アミノ酸) で蛋白質の長さ、その分子量 (kDa) と等電点、MS やリボソーム プロファイリング (翻訳証拠, TE)、機能予測に基づく実験的証拠をよう支援orthology (OpenProt、支え 10 種を渡って v1.3)。テーブルには、関連遺伝子とトラン スクリプトおよびトラン スクリプト内で蛋白質のローカリゼーションに関する情報も含まれています。
- さらに情報を収集する詳細]リンクをクリックします。新しくオープンしたページには、照会されたタンパク質と、ゲノムとトランスクリプトーム座標とコザックの高効率変換開始サイト (TIS) モチーフ36、プレゼンスなどの情報を中心とするゲノムのブラウザーが含まれています 37。
- タンパク質やDNAのタンパク質または DNA 配列をそれぞれ取得する [情報] タブからリンクをクリックします。
- MS の証拠、リボソーム15上部のタブをクリックして検出、保護、識別された蛋白質の領域をプロファイルの詳細についてを参照します。
結果
上記のワークフローは、プライド リポジトリ38,39で利用できる MS のデータセットに適用されました。元の研究法を開発 (iMixPro) の細胞培養 (SILAC) のアミノ酸の安定同位体標識、アフィニ ティー精製 MS からの偽陽性を排除するために (AP ・ MS) 実験38。簡単に言えば、AP MS 実験関心 (餌) とその対話者 (餌) の蛋白質をフェッチするためビーズ結合抗体を使用で構成されます。収集されたタンパク質は、消化され、MS のために準備します。サンプル調製法、機器の設定は、プライド リポジトリ (PXD004246)、元の研究では、説明します。そのような実験の課題は、特にビーズがなく、餌に結合する蛋白質からの偽陽性の豊富です。本当の捕食と偽陽性の間異なる同位体比を生成する SILAC を使用私たちここでは、: 3 コントロール サンプル (いいえベイト) 光媒体、1 試料光培地中で培養餌を表現して重い培地中で培養餌を表現する 1 つのサンプルの培養は、ビーズとさらに質量分析が処理されます。このようなデザインで、ビーズに結合する無指定蛋白質が重・軽の割合は 1:4;とき真餌が 1:138の比率があります。
OpenProt データベースを使用して AP MS データ再分析餌に (PTPN14、JIP3、IQGAP1) 3 つの内因性のタンパク質が含まれている、2 つは過剰蛋白質 (RAF1 および RNF41) を表現しました。プロテインの定量のためのギャラクシー ワークフローが使用された実験 SILAC を使用するので (補足資料 S3図 2)。ワークフローは、OpenProt データベース全体 (OpenProt_all) または制限された OpenProt データベース (OpenProt_2pep、以前は 2 つのユニークなペプチドの最小検出蛋白質のみを含む) を使用して実行されました。
タンパク質同定と定量異なる使用データベース間で良いと再現をしました。図 3に示すとおり、元のペーパーで識別されるほとんどの蛋白質は、OpenProt_2pep または OpenProt_all のいずれかのデータベース (詳細なリストは補足材料 S5で利用可能) を使用してかをまた識別されました。この結果は、ここで説明したパイプラインとデータベース、タンパク質同定と定量に匹敵する UniProtKB データベース40に基づく現在の手続を生成することが OpenProt を示します。ただし、OpenProt データベースの使用研究ここで示されているように小説や以前検出できないタンパク質の検出を許可するユニークな利点があります。
11、よくサポートされている蛋白質 (1 アイソ フォームおよび 10 AltProts)、まだ現在は注釈付きのデータベースは、自信を持ってペプチド、OpenProt_2pep データベース (すべて蛋白質系統、およびサポートの数を使用して、すべてのデータセットにまたがる識別されました。ペプチドは補足材料 S5で利用可能) です。このデータベースは使用できます伝統的な 1% の検索スペースの増加として FDR の適度なまま。これらの 11 のタンパク質は、不在だった、元の研究では認められなかったデータベースから。
自信を持ってペプチド、OpenProt_all データベースを使用してすべてのデータセットにまたがる 29 蛋白質 (16 アイソ フォームと 13 AltProts) が発見された (番号と共に、すべての蛋白質系統ペプチドをサポートするので利用できる補足材料 S6).図 3のように、識別された蛋白質の総数を減少したが、最も自信を持ってタンパク質同定は推奨される厳しい FDR に影響しなかった.比較的 OpenProt_2pep データベースに新規タンパク質の高い番号は自信を持って識別できます。これらの新規タンパク質のすべてが存在しない OpenProt_2pep データベースから。これは MS に基づくプロテオミクスの選択したデータベースの重要な役割を強調表示します。
1 つの新規タンパク質は、RAF1 蛋白質 (IP_637643) の相互作用として発見されました。OpenProt のウェブサイトを使用して、1 つは MS も今までリボソームプロファイリングでこのタンパク質が検出されていない見ることができる (OpenProt v1.3)。蛋白質は長い 46 アミノ酸をトリプシン消化時に 2 つのユニークなペプチドを与えることができるだけ。検出されたペプチド RAF1 AP ・ MS のデータセット (分数 18)図 4に示すように、質の良いスペクトルを持っていたし、1, 09 の重・軽の比率を表示します。タンパク質は、 NANOGNBの偽遺伝子であるNANOGNBP1遺伝子にエンコードされます。成績証明書 (ENST00000448444)、現在非コーディングとアノテーションが GTEx ポータル40によるといくつかの組織で検出されました。タンパク質には、DNA 結合 (遺伝子オントロジー行く: 0003677)41に関連付けられている予測機能ドメインが含まれています。
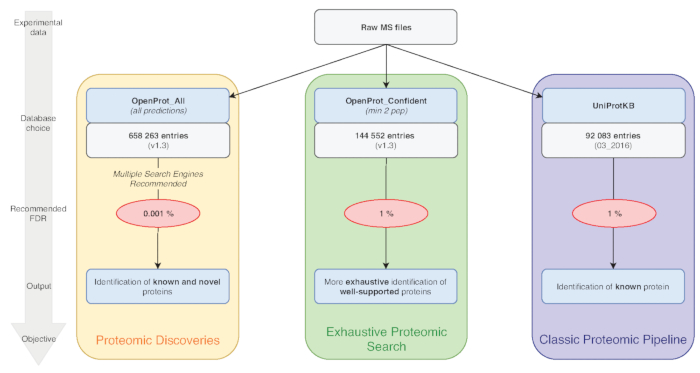
図 1: データベースのプロテオミクス解析グラフ選択します。特に、データベースの選択、MS データの解析は、研究目的に依存します。3 つの共通の目標は、青 (古典的なプロテオーム パイプライン)、緑 (網羅的プロテオーム検索)、オレンジ (プロテオミクス探索) で概説されます。それぞれの目的は、適切なデータベースとパイプラインに依存します。単一識別ツールを網羅的かつ古典的なプロテオミクス使うパイプライン。プロテオミクス探索パイプラインは、複数の識別エンジンを使用を強くお勧めします。推奨 Fdr は赤で示され、タンパク質データベースのサイズは、灰色の箱に示されています。この図の拡大版を表示するのにはここをクリックしてください。
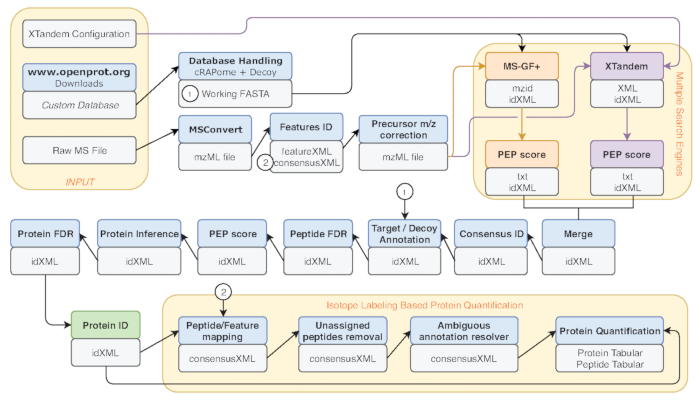
図 2: 使用される銀河ワークフローのグラフィカルな表現です。38Eyckerman らデータの再分析で使用するプロテオーム解析ワークフローのステップ バイ ステップの表現。プロテインの定量、ペプチドの検索、入力ファイルは、オレンジ色のボックスで示されます。使用するツールに対応する青色のボックスと、灰色の箱は、生成された出力ファイルに対応します。別の検索エンジン (MS-GF + と X!タンデム)、必要な入力と出力を示す矢印と同様に異なる色 (それぞれ赤と紫) で示されます。緑のボックスは、タンパク質同定のリストを生成するツールを強調表示します。後工程に使用するものは最も近いとして示される複数の出力が生成されるときの矢印を。このワークフローは、自由に利用できる補助材料 S2です。X!タンデム既定パラメーター構成ファイルは補足材料 S4で利用可能です。この図の拡大版を表示するのにはここをクリックしてください。
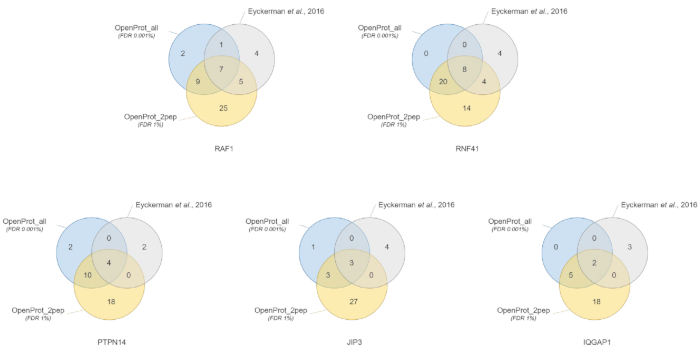
図 3: 別のデータベースを使用して餌あたり入力/終端] 識別の比較。最も自信を持って OpenProt を用いたタンパク質同定のベン図データベース 1% (オレンジ、最小 2 ユニークなペプチド、OpenProt_2pep の証拠) の FDR、または全体の OpenProt 0.001% (ブルー、OpenProt_all) でデータベース FDR、または報告元の紙 (グレー) で38。各ダイアグラムは、述べられた餌に識別されたインターアクターに対応: RAF1、RNF41、PTPN14、JIP3、IQGAP1。この図の拡大版を表示するのにはここをクリックしてください。
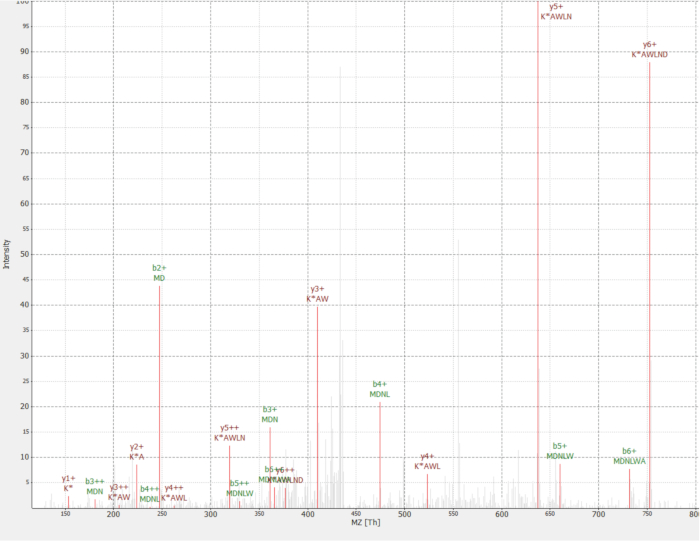
図 4: の MS/MS スペクトル識別 MDNLWAK(13 6) IP_637643 蛋白質由来ペプチド。(0 ~ 100%) の強度は相対パスです。選択したピークは、y イオン注釈は、暗い赤 b イオンで、注釈と緑赤で示されます。TOPPview ソフトウェア34から抽出されます。前駆体エラー = 2.70 ppm、PEP スコア = 0.12。この図の拡大版を表示するのにはここをクリックしてください。
| 用語 | 定義 | 参照 |
| 代替 ORF (AltORF) | 現在はゲノムの注釈は、注釈が付けられたが、OpenProt の注釈の非正規の ORF。 | 15 |
| 参照 ORF (RefORF) | 正規の ORF がゲノムの注釈および OpenProt の注釈付き。 | 15 |
| 代わりとなる蛋白質 (AltProt) | RefProt と有意な類似性がないと、AltORF で符号化された蛋白質。加盟プレフィックス: IP_。 | 15 |
| 参照タンパク質 (RefProt) | 現在 UniProtKB、Ensembl NCBI RefSeq など蛋白質シーケンス データベースで、OpenProt を注釈する蛋白質。 | 15 |
| 新規アイソ フォーム | RefProt と有意な類似性と、AltORF で符号化された蛋白質。加盟プレフィックス: II_。 | 15 |
| OpenProt_2pep データベース | すべての RefProts と OpenProt、既に 2 ユニークなペプチドの最小検出によって予測された蛋白質シーケンスが含まれています。 | 15 |
| OpenProt_1pep データベース | すべての RefProts と OpenProt、既に 1 のユニークなペプチドの最小検出によって予測された蛋白質シーケンスが含まれています。 | 15 |
| OpenProt_all データベース | すべての RefProts と OpenProt によって予測された蛋白質シーケンスが含まれています。 | 15 |
表 1: OpenProt とプロトコルの中で使用される用語の定義
補足材料 S1: データベースを処理するため銀河ワークフロー 。これは、CRAPome とおとりのシーケンスを入力データベースに (逆方向) を追加します。出力は Fasta ファイルです。をダウンロードするここをクリックしてください。
補足材料 S2: タンパク質の同定のためのギャラクシー ワークフロー 。これは 2 つの検索エンジンを使用して質量分析データ ファイルからの蛋白質を識別する (MS-GF + と X!タンデム)。各パラメーターを調整することができますに応じてワークフローを実行する前に。をダウンロードするここをクリックしてください。
補足材料 S3: 安定同位体標識 (SIL) を用いたタンパク質定量銀河ワークフロー 。これは識別し、2 つの検索エンジンを使用して質量分析データ ファイルから蛋白質を定量化 (MS-GF + と X!タンデム)。各パラメーターを調整することができますに応じてワークフローを実行する前に。をダウンロードするここをクリックしてください。
補足材料 S4: X!タンデム既定パラメーターの構成ファイル。この XML ファイル X を実行するために必要です!銀河プラットフォーム上の TandemAdapter ツールです。をダウンロードするここをクリックしてください。
補足材料 S5: iMixPro データセットからの蛋白質を定量化します。Eyckerman ら 201638からのデータ ファイルは、OpenProt データベースを使用して処理された、定量化された蛋白質は条件ごとに表示されます。餌は、PTPN14、JIP3、IQGAP1、RAF1、RNF41.緑色の遺伝子名は蛋白質のまた元の紙38で識別に対応します。オレンジ色で示す遺伝子名は、オリジナルの論文で報告された:npo によると既知のインターアクターに対応します。水色で示されている遺伝子名は、インターアクター (対応するタンパク質の加盟数はかっこで示される) として識別された蛋白質に対応します。薄いグレーで示される遺伝子名、斜体は可能性が高い汚染物質 (ケラチン蛋白質) に対応します。をダウンロードするここをクリックしてください。
補足材料 S6: iMixPro データセットから蛋白質を識別します。OpenProt データベースを使用した Eyckerman ら 201638からデータ ファイルが処理され、識別された蛋白質は、条件ごとに一覧表示されます。餌は、PTPN14、JIP3、IQGAP1、RAF1、RNF41.タンパク質受入番号のとおり、知られていた蛋白質の新規アイソ フォームの II_ と IP_ 蛋白質代替 ORF (AltProt) からの開始です。数ペプチドをサポート ブラケットに示されています。をダウンロードするここをクリックしてください。
ディスカッション
質量分析計からのデータを分析する場合、タンパク質同定の品質は部分的使用データベース6,20の精度に依存します。現在のアプローチは、伝統的 UniProtKB データベースを使用して、まだこれら議事録につき単一 ORF のゲノムのアノテーション モデルと 100 (以前に実証例) を除いてコドン40の最小の長さをサポートします。複数の研究は、容疑者以外のコーディングから機能 ORFs の発見とそのようなデータベースの欠点を関連付ける領域8,11,12,13。今、OpenProt より網羅的なタンパク質の同定と複数のトランスクリプトーム注釈からタンパク質配列を描画します。OpenProt 取得 NCBI RefSeq (GRCh38.p7) と Ensembl (GRCh38.83) トランスクリプトームと UniProtKB の注釈 (UniProtKB SwissProt、2017-09-27)40,42,43。現在のコメントは現在ほとんど重複、OpenProt は従ってときに 1 つの注釈15に制限よりも潜在的なプロテオーム風景の包括的なビューを表示します。
さらに、OpenProt は、コードするモデルを適用とトラン スクリプトあたり複数の蛋白質のアノテーションをできます。統計と計算上の理由から、OpenProt はまだ 30 コドン15の最小の長さのしきい値を保持します。しかし、それによりタンパク質の同定の可能性の範囲を拡大、新規蛋白質シーケンスの何千もを予測します。このアプローチでは、OpenProt より体系的な方法でプロテオームの発見をサポートします。
タンパク質同定の品質は、使用されるパラメーターによっても影響を。MS ベースのプロテオミクス解析は通常 1% 蛋白質 FDR を保持します。ただし、全体の OpenProt データベースには、約 6 倍以上のエントリ (図 1) が含まれています。検索スペースの相当な増加を考慮、0.001% のより厳格な FDR を使用をお勧めします。このパラメーターは、ベンチマーク研究とランダムに選択されたスペクトル15のマニュアル評価を使用して最適化されていました。しかし、偽陽性がまだ可能性、徹底的な検査とサポートする新規蛋白質のための証拠の検証をお勧め。バック グラウンド データや誤データセット15間で異なる推奨される標準的な 2 つの異なる MS 実行から蛋白質の同定可能性があります。
同様に、実験的なデザインとパラメーターに合わせて嬉しいパイプラインここ提供、事例研究で使用に変更できます。感性とペプチド同定32の感度を増加すると、複数の検索エンジンを使用して私たちをお勧めします。さらに、(図 1) の実験の目的に最適に対応するデータベースを使用してお勧めします。厳しい FDR が付属してデータベース全体の OpenProt を使用して、真の身分が失われます。したがって、データベース全体は、小さい OpenProt データベース (上記事例で使用される OpenProt_2pep) などに使用する必要が古典的なプロテオミクス プロファイリングしながら新規タンパク質の探索が意図されるべき。
いくつかの研究は、他コドン44,45翻訳開始を強調したに対し、OpenProt は、現在 ATG コドンから始まるシーケンスを予測しています。新規タンパク質は、1 つまたはいくつかのユニークなペプチドによって識別される、本当の開始コドンは推定 ATG ではない可能性です。ユーザーは、OpenProt ウェブサイト上の翻訳の証拠を見ることができます。現在、OpenProt は、全体の予測された蛋白質シーケンス (100% 重複)15にかかわる場合にのみ翻訳イベントを報告します。したがって、翻訳の証拠の不在というタンパク質が翻訳されていないが、その疑惑の ATG 開始コドンありません可能性があります。
その現在の制限にもかかわらず OpenProt は、真核生物のゲノムのコーディング可能性の包括的なビューを提供しています。OpenProt データベースは、プロテオーム発見とプロテオームの機能と相互作用の理解を促進します。他の種の注釈が含まれます OpenProt データベースの今後の展開、非 ATG から証拠を翻訳開始コドンと新規タンパク質は、ゲノム、エキソーム配列研究するためのパイプラインの開発。
開示事項
著者は利害の衝突を宣言しません。
謝辞
ビビアン Delcourt は、彼の助けやディスカッション、この作品についてのアドバイスを感謝いたします。フォン ・ ド ・ ルシェルシュ ・ デュ ・ ケベック州健康 FRQS サポート センター ・ デ ・凝ったデュのメンバーである X.R. センター病院ユニヴェルシテール ・ デ ・ シャーブ ルック。この研究は、X.R. と機構の付与モップ 137056 機能・発現プロテオミクスと小説タンパク質発見カナダ研究の椅子によって支えられました。天秤 Québec そして計算カナダ シャーブ ルック大学からスーパー コンピューター mp2 の使用と彼らのサポートのためにチームに感謝いたします。Mp2 のスーパー コンピューターの操作によって、カナダの基盤の革新 (CFI)、ル ミニステール資金を供給される de l' 仏、デ ラ科学 et ・ デ ・ l'innovation ・ デュ ・ ケベック (MESI) とレ ・ フォン ・ デ ・凝ったケベック - 自然 et 技術 (周波数 NT)。いくつかのプロテオミクスの計算に使用された銀河サーバー共同研究センター 992 医療エピジェネティクス (DFG グラント SFB 992/1 2012) とドイツ連邦教育省と研究によって資金を供給される一部 (BMBF 付与 031 RBC A538A/A538C、031L0101B/031L0101C ド。NBI エピ、031 L 0106 デ。階段 (de。NBI))。
資料
| Name | Company | Catalog Number | Comments |
| OpenProt website | open source | n/a | www.openprot.org |
| Galaxy Server | open source | n/a | https://usegalaxy.eu/ |
| TOPPview software | open source | n/a | www.openms.de |
参考文献
- Kim, M. S., et al. A draft map of the human proteome. Nature. 509 (7502), 575-581 (2014).
- Wilhelm, M., et al. Mass-spectrometry-based draft of the human proteome. Nature. 509 (7502), 582-587 (2014).
- Hein, M. Y., et al. A human interactome in three quantitative dimensions organized by stoichiometries and abundances. Cell. 163 (3), 712-723 (2015).
- Huttlin, E. L., et al. The BioPlex Network: A Systematic Exploration of the Human Interactome. Cell. 162 (2), 425-440 (2015).
- Huttlin, E. L., et al. Architecture of the human interactome defines protein communities and disease networks. Nature. 545 (7655), 505-509 (2017).
- Kumar, D., Yadav, A. K., Dash, D. Choosing an Optimal Database for Protein Identification from Tandem Mass Spectrometry Data. Proteome Bioinformatics. , 17-29 (2017).
- Jeong, K., Kim, S., Bandeira, N. False discovery rates in spectral identification. BMC Bioinformatics. 13 (Suppl 16), (2012).
- Brunet, M. A., Levesque, S. A., Hunting, D. J., Cohen, A. A., Roucou, X. Recognition of the polycistronic nature of human genes is critical to understanding the genotype-phenotype relationship. Genome Research. , (2018).
- Brent, M. R. Genome annotation past, present, and future: how to define an ORF at each locus. Genome Research. 15 (12), 1777-1786 (2005).
- Harrow, J., et al. GENCODE: The reference human genome annotation for The ENCODE Project. Genome Research. 22 (9), 1760-1774 (2012).
- Samandi, S., et al. Deep transcriptome annotation enables the discovery and functional characterization of cryptic small proteins. eLife. 6, e27860 (2017).
- Saghatelian, A., Couso, J. P. Discovery and characterization of smORF-encoded bioactive polypeptides. Nature Chemical Biology. 11 (12), 909-916 (2015).
- Delcourt, V., Staskevicius, A., Salzet, M., Fournier, I., Roucou, X. Small Proteins Encoded by Unannotated ORFs are Rising Stars of the Proteome, Confirming Shortcomings in Genome Annotations and Current Vision of an mRNA. Proteomics. , (2017).
- Plaza, S., Menschaert, G., Payre, F. In Search of Lost Small Peptides. Annual Review of Cell and Developmental Biology. 33 (1), (2017).
- Brunet, M. A., et al. OpenProt: a more comprehensive guide to explore eukaryotic coding potential and proteomes. Nucleic Acids Research. , (2018).
- Afgan, E., et al. The Galaxy platform for accessible, reproducible and collaborative biomedical analyses: 2016 update. Nucleic Acids Research. 44 (W1), W3-W10 (2016).
- Afgan, E., et al. The Galaxy platform for accessible, reproducible and collaborative biomedical analyses: 2018 update. Nucleic Acids Research. 46, W537-W544 (2018).
- Sturm, M., et al. OpenMS – An open-source software framework for mass spectrometry. BMC Bioinformatics. 9 (1), 163 (2008).
- Carithers, L. J., et al. A Novel Approach to High-Quality Postmortem Tissue Procurement: The GTEx Project. Biopreservation and Biobanking. 13 (5), 311-319 (2015).
- Aebersold, R., Mann, M. Mass spectrometry-based proteomics. Nature. 422 (6928), 6928 (2003).
- Domon, B., Aebersold, R. Mass Spectrometry and Protein Analysis. Science. 312 (5771), 212-217 (2006).
- Hu, J., Coombes, K. R., Morris, J. S., Baggerly, K. A. The importance of experimental design in proteomic mass spectrometry experiments: Some cautionary tales. Briefings in Functional Genomics. 3 (4), 322-331 (2005).
- Wu, P. Y., Phan, J. H., Wang, M. D. Assessing the impact of human genome annotation choice on RNA-seq expression estimates. BMC Bioinformatics. 14 (11), S8 (2013).
- Mellacheruvu, D., et al. The CRAPome: a contaminant repository for affinity purification-mass spectrometry data. Nature Methods. 10 (8), 730-736 (2013).
- Adusumilli, R., Mallick, P. Data Conversion with ProteoWizard msConvert. Proteomics: Methods and Protocols. , 339-368 (2017).
- French, W. R., et al. Wavelet-Based Peak Detection and a New Charge Inference Procedure for MS/MS Implemented in ProteoWizard’s msConvert. Journal of Proteome Research. 14 (2), 1299-1307 (2015).
- Kuenzi, B. M., et al. APOSTL: An Interactive Galaxy Pipeline for Reproducible Analysis of Affinity Proteomics Data. Journal of Proteome Research. 15 (12), 4747-4754 (2016).
- Hoekman, B., Breitling, R., Suits, F., Bischoff, R., Horvatovich, P. msCompare: a framework for quantitative analysis of label-free LC-MS data for comparative candidate biomarker studies. Molecular & Cellular Proteomics: MCP. 11 (6), (2012).
- Bjornson, R. D., et al. X!!Tandem, an improved method for running X!tandem in parallel on collections of commodity computers. Journal of Proteome Research. 7 (1), 293-299 (2008).
- Kim, S., Pevzner, P. A. MS-GF+ makes progress towards a universal database search tool for proteomics. Nature Communications. 5, 5277 (2014).
- Vaudel, M., Barsnes, H., Berven, F. S., Sickmann, A., Martens, L. SearchGUI: An open-source graphical user interface for simultaneous OMSSA and X!Tandem searches. Proteomics. 11 (5), 996-999 (2011).
- Shteynberg, D., Nesvizhskii, A. I., Moritz, R. L., Deutsch, E. W. Combining results of multiple search engines in proteomics. Molecular & Cellular Proteomics: MCP. 12 (9), 2383-2393 (2013).
- Bittremieux, W., et al. Quality control in mass spectrometry-based proteomics. Mass Spectrometry Reviews. 37 (5), 697-711 (2018).
- Bertsch, A., Gröpl, C., Reinert, K., Kohlbacher, O. OpenMS and TOPP: Open Source Software for LC-MS Data Analysis. Data Mining in Proteomics: From Standards to Applications. , 353-367 (2011).
- Pfeuffer, J., et al. OpenMS – A platform for reproducible analysis of mass spectrometry data. Journal of Biotechnology. 261, 142-148 (2017).
- Kozak, M. Pushing the limits of the scanning mechanism for initiation of translation. Gene. 299 (1-2), 1-34 (2002).
- Noderer, W. L., et al. Quantitative analysis of mammalian translation initiation sites by FACS-seq. Molecular Systems Biology. 10, 748 (2014).
- Eyckerman, S., et al. Intelligent Mixing of Proteomes for Elimination of False Positives in Affinity Purification-Mass Spectrometry. Journal of Proteome Research. 15 (10), 3929-3937 (2016).
- Vizcaíno, J. A., et al. 2016 update of the PRIDE database and its related tools. Nucleic Acids Research. 44 (D1), D447-D456 (2016).
- Bateman, A., et al. UniProt: the universal protein knowledgebase. Nucleic Acids Research. 45 (D1), D158-D169 (2017).
- The Gene Ontology Consortium Expansion of the Gene Ontology knowledgebase and resources. Expansion of the Gene Ontology knowledgebase and resources. Nucleic Acids Research. 45 (D1), D331-D338 (2017).
- O’Leary, N. A., et al. Reference sequence (RefSeq) database at NCBI: current status, taxonomic expansion, and functional annotation. Nucleic Acids Research. 44, D733-D745 (2016).
- Zerbino, D. R., et al. Ensembl 2018. Nucleic Acids Research. 46 (D1), D754-D761 (2018).
- Andreev, D. E., et al. Translation of 5’ leaders is pervasive in genes resistant to eIF2 repression. eLife. 4, e03971 (2015).
- Jackson, R., et al. The translation of non-canonical open reading frames controls mucosal immunity. Nature. 564, 434-438 (2018).
転載および許可
このJoVE論文のテキスト又は図を再利用するための許可を申請します
許可を申請This article has been published
Video Coming Soon
Copyright © 2023 MyJoVE Corporation. All rights reserved