É necessária uma assinatura da JoVE para visualizar este conteúdo. Faça login ou comece sua avaliação gratuita.
Method Article
Localização de cor multi microscopia de proteínas de membrana única em organelas de células de mamíferos vivas
Neste Artigo
Resumo
Aqui, apresentamos um protocolo para multi cor localização das proteínas de membrana única em organelas de células vivas. Para anexar fluorophores, auto rotulação de proteínas são utilizadas. Proteínas, localizadas em compartimentos diferentes membranas da organela a mesma, podem ser localizadas com precisão de ~ 18 nm.
Resumo
Conhecimento sobre a localização de proteínas no celulares subcompartments é crucial para entender a sua função específica. Aqui, apresentamos uma técnica de super-resolução que permite a determinação de microcompartments que são acessíveis para proteínas, gerando a localização e rastreamento mapas destas proteínas. Além disso, pela microscopia de localização de várias cores, perfis de proteínas em diferentes subcompartments de rastreamento e a localização são obtidas simultaneamente. A técnica é específica para as células vivas e baseia-se a imagem repetitiva de proteínas da membrana celular único. Proteínas de interesse são geneticamente fundidas com tags Self rotulagem específicas, so-called. Essas marcas são enzimas que reagem com um substrato de maneira covalente. Conjugados para estes substratos são corantes fluorescentes. Reação das proteínas com a enzima-tag com a fluorescência rotulado resultados de substratos em proteínas etiquetadas. Aqui, diversos (TMR) e silício rodamina (Sr) são utilizados como corantes fluorescentes anexados para os substratos das enzimas. Usando concentrações de substrato a pM a gama nM, rotulagem sub estequiométrica é alcançado que resulta em sinais distintos. Estes sinais são localizados com ~ 15 – 27 precisão nm. A técnica permite a imagem multi cor de moléculas simples, segundo o qual o número de cores é limitado pelas tinturas de membrana permeável disponíveis e o repertório de auto-rotulantes enzimas. Vamos mostrar a viabilidade da técnica, determinando a localização da enzima (Pten) controle de qualidade-induzida da quinase 1 (PINK1) em diferentes compartimentos mitocondriais durante seu processamento em relação a outras proteínas de membrana. O teste para o verdadeiros interações físicas entre as proteínas única diferente etiquetadas por molécula FRET ou rastreamento co é restrito, porém, porque os graus de rotulagem baixos diminuem a probabilidade de ter duas proteínas adjacentes rotuladas ao mesmo tempo. Enquanto a técnica é forte para a imagem latente proteínas nos compartimentos de membrana, na maioria dos casos não é apropriado determinar a localização de proteínas solúveis altamente móveis.
Introdução
O objetivo do presente protocolo é fornecer um método de imagem para localizar e acompanhar as proteínas de membrana única dentro de células vivas. Chamamos esse método de rastreamento e localização de microscopia (TALM)1,2. Como estocástico microscopia de reconstrução óptica (Tempestade)3 e microscopia de fluorescência Photoactivation localização (PALM (F))4,5, TALM é uma técnica de localização de fluorescência único baseado na molécula. No entanto, é distinta da forma que a mobilidade das proteínas de membrana em combinação com imagem repetitiva do mesmo rotulado molécula em diferentes posições revela o microcompartment que é acessível para a proteína móvel. Em outras palavras, as possíveis localizações da proteína são definidas pela arquitetura da organela e pela mobilidade da proteína1. O método é complementar vários outros super-resolução técnicas6,7,8 , porque ele revela a trajetória e localização mapas por proteínas móveis de imagem. A rotulagem é baseada no uso de proteínas de fusão geneticamente modificados que são em si não-fluorescente. Estas proteínas de fusão são auto-rotulantes enzimas que reagem covalentemente com um substrato conjugado com um corante. Este procedimento tem a vantagem que o grau de rotulagem pode ser controlada pela quantidade de substrato adicionado. Além disso, permite para variar a cor da fluorescência, dependendo o corante escolhido conjugado. Vários auto rotulagem enzima-tags são disponíveis9. Outra vantagem de usar auto-rotulantes enzima-etiquetas, que os corantes conjugados são geralmente mais estável e mais brilhante do que proteínas fluorescentes1 e proteínas individuais, portanto, podem ser gravadas mais e mais precisamente até que eles são branqueados. Isto permite a gravação de trajetórias de proteínas móveis e a extração de coeficientes de difusão10,11.
Aqui, podemos demonstrar a viabilidade da TALM com proteínas da membrana mitocondrial, mas também pode ser aplicado para outras proteínas de membrana cellular intra e extra, incluindo células de diferentes tipos de12,13. Mostramos que TALM multi cor adicional permite a distinção simultânea das proteínas em diferentes subcompartments em complementação a existente Super-resolução fluorescência microscopia técnicas14,15, 16. TALM é compatível com células vivas de imagem17. A foto-física dos escolhido rhodamines diversos (TMR) e o silício-Rhodamien (Senhor), em particular seu brilho e estabilidade, permite que as proteínas de membrana único registro sobre vários quadros fornecendo mapas trajetória (e localização). No entanto, TALM é limitado para localização de proteínas solúveis com coeficientes de difusão de alta desde o motion blur é muito alto e os fótons coletados por quadro são demasiado baixos para a localização apropriada. Além disso, TALM requer menos energia de excitação do que por exemplo tempestade ou depleção de emissão estimulada (STED) microscopia6,7, reduzindo efeitos fototóxicos. Isto é importante, pois o estresse fototóxico frequentemente afeta a morfologia organellar18 e, portanto, de análise de mobilidade19. Em suma, apresentamos multi cor TALM em células vivas como uma técnica que preenche uma lacuna entre os métodos de microscopia de localização tempestade/STED/PALM (F) e técnicas que analisam a mobilidade de proteína, como a recuperação de fluorescência após fotobranqueamento (FRAP)20 ,21, fluorescência correlação espectroscopia (FCS)22e fluorescência cruzam correlação espectroscopia (FCCS)11,23.
Access restricted. Please log in or start a trial to view this content.
Protocolo
O seguinte protocolo segue as diretrizes do Comitê de ética de pesquisa instituição local.
1. métodos
-
Cultura celular
- Cultive células, por exemplo as células HeLa (carcinoma do colo do útero humano), em um frasco de cultura de célula T25 contendo 5 mL do meio de crescimento em 37 ° C e 5% de CO2.
Nota: Para a imagem latente, dividir as células em lamelas preparadas (consulte as etapas 1.3 e 1.4) e manter-se em média por imagens.
- Cultive células, por exemplo as células HeLa (carcinoma do colo do útero humano), em um frasco de cultura de célula T25 contendo 5 mL do meio de crescimento em 37 ° C e 5% de CO2.
-
Transfecção celular
Nota: Utilize as linhas celulares que expressam estàvel as proteínas marcadas sempre que possível24 para evitar superexpressão forte. Para transfeccao transitório, adaptar-se a quantidade de plasmídeo usado para transfeccao. Por exemplo, quando Ca2 + fosfato de transfeccao25 é usado, transfect células (80 – 90% confluência) em um prato de cultura de célula de 3,5 cm com 2,5 – 5 µ g de DNA de plasmídeo. Ao executar duplo transfeccao, use 2,5 µ g por cada construção do plasmídeo.- Para experiências de cor dupla, use uma linha celular com expressao de uma proteína Self rotulagem e transitoriamente transfect com o plasmídeo codificação a outras proteínas Self rotulagem17.
Nota: Aqui, para experimentos de cor dupla, as células HeLa foram usadas que expressa estàvel as proteínas Self rotulagem PINK1-Halo-Tag e Tag-fSNAP-Tom20.
- Para experiências de cor dupla, use uma linha celular com expressao de uma proteína Self rotulagem e transitoriamente transfect com o plasmídeo codificação a outras proteínas Self rotulagem17.
-
Limpeza de lamelas
- Lugar de lamelas num copo. Adicionar 30 mL de H2O para o béquer contendo as lamelas e agite suavemente para remover poeira de sua superfície.
- Reunir as lamelas com uma pinça e secá-los com um fluxo de nitrogênio.
- Remova qualquer contaminação orgânica na superfície das lamelas, por exemplo, por limpeza de plasma.
Nota: Para evitar mais contaminação do material vidro, use luvas durante o manuseio das lamelas.
Atenção: Quando as lamelas são limpos pelo plasma limpeza, somente a parte superior das lamelas é limpo; Use este lado para revestimento com poli-L-lisina-polietileno glicol-glicina-aspartato de arginina (PLL-PEG-RGD) (seção 1.4) e célula de semeadura (seção 1.5).
-
Revestimento da lamela com PLL-PEG-RGD
Nota: PLL-PEG-RGD é um derivado de poli-L-lisina (PLL) anexado com um glicol de polietileno (3.000 Da) e um peptídeo (CGRGDS) cysteine-glycine-arginine-glycine-aspartate-serine. PLL vincula-se à superfície de vidro carregado negativamente e dá forma a um pincel de PEG. Isto reduz drasticamente a vinculação inespecífica de corantes fluorescentes carregadas. Além disso, o motivo da RGD imita o peptídeo sinal do receptor integrina e desse modo promove a integrina-mediada da aderência das células que caso contrário não facilmente aderir.- Prepare o PLL-PEG-RGD como descrito anteriormente,26. Em suma, dissolva 0,8 mg de PLL-PEG-RGD em 1 mL de PBS. Adicione 10 µ l da solução de PLL-PEG-RGD no lado superior de uma lamela limpo.
- Pegue uma segunda lamela e coloque-a com sua superfície limpa de cabeça para baixo sobre a primeira lamela (que tem a queda de PLL-PEG-RGD na parte superior); Isso resulta na imprensa a solução de PLL-PEG-RGD entre duas lamelas.
- Cuidadosamente coloque as lamelas imprensadas numa proveta e incubar por 1h à temperatura ambiente em um ambiente livre de pó seco.
- Após 1 h, adicione 30 mL de H2O para o béquer para cobrir totalmente as lamelas com água.
- Agite o copo até que as lamelas separar uns dos outros.
- Use uma pinça para reunir as lamelas fora da água e secá-los sob um fluxo de gás nitrogênio.
Nota: As lamelas revestidas podem ser armazenadas em um vidro seco, estéril, placa de Petri com tampa por um par de dias.
-
Preparação da amostra para a imagem latente
- Transferir as única lamelas revestidas para numa placa de cultura de células de 35 mm, com a superfície revestida de PLL-PEG-RGD virado para cima e adicionar 2 mL de meio em cima de imagem.
- Adicione ~ 500.000 células trypsinized (200-500 µ l) que expressam as tags Self rotulagem para as proteínas de membrana respectivos para o mL 2 médio a placa de cultura de células com a lamela revestida de imagem. Agite delicadamente à mão para garantir uma distribuição homogénea das células para obter uma camada uniforme de célula.
- Incube as células a 37 ° C e 5% de CO2 até 80% de confluência é alcançada.
Nota: Amostras de células devem ser semeadas 3 dias antes de imagem e 1 dia antes do transfection. Células, que estàvel expressam a proteína de interesse, podem ser semeadas 2 dias antes da imagem latente. Mais tarde, apenas células cultivadas em lamela são criação da imagem.
-
Rotulagem de proteínas etiquetadas
Nota: Substratos mais fluorescentes tem que ser dissolvido em DMSO isento de água. Aconselhamos a utilização de soluções estoque de substrato fluorescente de 1 µM quando a concentração de rotulagem final é de 0,2 a 30 nM17. Para a imagem latente proteínas de membrana no interior das células, use substratos fluorescentes permeável de membrana.- Aquecer o meio da imagem latente para 37 ° C em banho-maria.
- Pipete 1 mL do meio de geração de imagens previamente aquecido em um tubo de 2 mL com tampa. Adicionar 0,2 – 30 µ l de substratos fluorescentes de soluções estoque de 1 µM para preparar a solução de rotulagem final (concentração final: 0,2 a 30 nM).
- Vórtice a solução de etiquetagem para 10 s.
- Substituir o meio da placa de cultura de 35 milímetros com as células em uma lamela (ver passo 1.5) 1 mL do preparado por solução de rotulagem.
- Incube as celulas na solução de etiquetagem em 37 ° C e 5% CO2 por 20 a 30 min.
- Lave as células com 2 mL de PBS uma vez e, em seguida, com 2 mL de meio de imagem duas vezes. Finalmente, Pipetar 1 mL de meio fresco de imagem para o prato de célula e colocar a amostra de volta para a incubadora a 37 ° C e 5% de CO2 pelo menos 1 h. Antes de imagem, troca a imagem médio mais uma vez.
Nota: Ao executar o experimento pela primeira vez, confirmam o direcionamento correto das proteínas Self rotuladas de membranas organellar manchando as organelas organela comercialmente disponível corantes específicos27,28. Neste caso, também use 100 – 200 nM de substrato para as enzimas Self rotulagem para produzir sinais fortes.
-
Preparação de uma amostra de grânulo fluorescente
Nota: Para determinar o desvio óptico e alinhar imagens dos diferentes canais, multi cores fluorescentes grânulos (0.1 µm) são utilizados. Com as imagens gravadas, será gerada uma matriz de transformação afim para os canais de emissão de dois.- Diluir a solução de grânulos de 1% com pura H2O.
- Lugar 5 gotas da solução preparada com os grânulos de fluorescência em cinco posições diferentes em uma lamela limpada (consulte a etapa 1.3).
- Seque o exemplo do grânulo fluorescente sobre uma bancada limpa.
Nota: A amostra pode ser re-utilizada; Portanto, cobrir a amostra com papel de alumínio para evitar a contaminação e mantê-lo em um 4 ° C.
2. microscopia
-
Instalação experimental
Nota: Um sistema de microscopia básica para a imagem latente de duplo-cor única molécula é baseado em um microscópio invertido: é equipado com dois lasers acoplados através de uma polarização multi-mode-óptica mantendo fibra monomode em uma única reflexão interna total (TIR) condensador, um objectivo de imersão de óleo projetado para TIRF, filtros de emissão um polyband, um divisor de imagem e uma câmera altamente sensível (Figura 1). Um condensador TIR é necessária que permite o ajuste contínuo do ângulo incidente para alternar entre o epi-, altamente inclinados e folha laminada óptica (iluminação fino altamente inclinado (HILO)29) e o modo de excitação TIRF com otimizado profundidade de penetração. Imagens são adquiridas com um sistema de refrigeração detector altamente sensíveis, por exemplo, um back-iluminado multiplicando elétrons carregados câmera dispositivo acoplado (EMCCD) (eficiência quântica QE > 90%) ou uma câmara de sCMOS (QE > 80 – 90%).- Determine o desvio ótico por imagem fluorescentes grânulos (consulte a etapa 2.2) nas mesmas condições como as que será posteriormente usado para o experimento, por exemplo, quando 10.000 frames são registrados no experimento, gravar também 10.000 quadros com a amostra do grânulo. Para a determinação da deriva óptica, compare a posição dos grânulos no primeiro frame e o último quadro adquirido (figura 1B). Se necessário, mais tarde corrigir a série de imagem óptico deriva30 e/ou usar deriva de ambientes estáveis.
- Equipe o cubo do filtro com o divisor de feixe dicroicas apropriado, por exemplo, para fluorescência laranja e vermelha, além disso, os filtros de emissão adequada para fluorescência laranja e fluorescência vermelha. Equipe o divisor de imagem com os filtros adequados. Verifique a possível fuga de sinais de um canal para outro canal gravando-as amostras de cor única em ambos os canais (Figura 1).
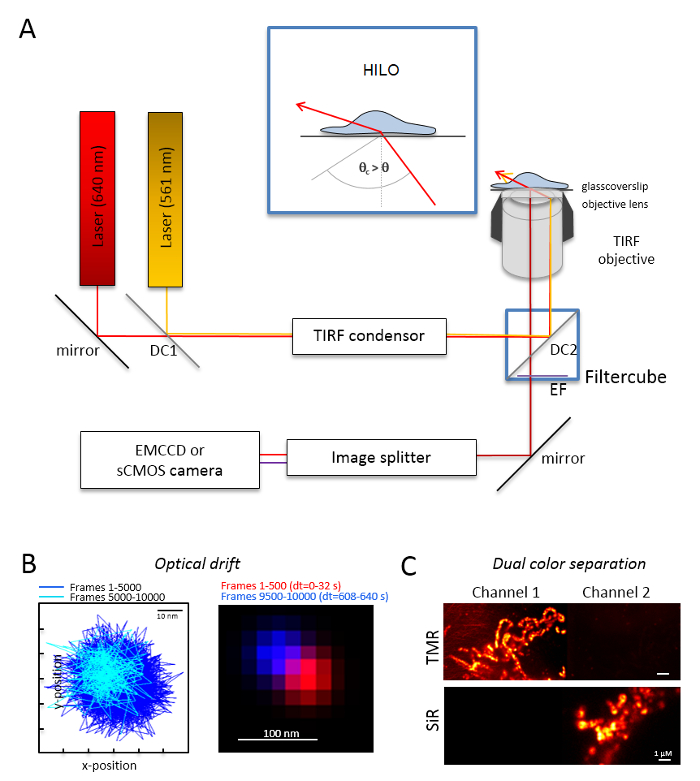
Figura 1 : Layout óptico para microscopia rastreamento e localização multi cor (TALM) com emissores de laranja e vermelhos. (A) instalação de microscópio invertido pelo menos dois lasers de excitação, um condensador TIRF, um objectivo adequado TIRF, um divisor de imagem e uma câmera sensível. Baixo-relevo: para excitar organelas no interior das células, o ângulo do feixe incidente deve ser definido menor do que o ângulo crítico para TIRF alcançar altamente inclinados e laminado iluminação folha óptica (HILO). DC1: Espelho dicroico 1; DC2: Espelho dicroico 2. EF: filtro de emissão. (B) teste na óptica drift por posições de um grânulo fluorescente para 10.000 quadros com a mesma taxa de quadros a partir dos seguintes experimentos de imagem (aqui: 15 Hz). Conectado posições dos primeiros 500 frames e os últimos 500 quadros mostram a deriva. Também, uma imagem mesclada com a posição do primeiro e o último quadro em vermelho e azul mostram um desvio mínimo. A deriva é que a distância entre o centro dos sinais dividido pelo tempo total de gravação, aqui 125 pm/s. (C) checar a separação clara dos sinais, aqui TMR e senhor. Para ambos os canais, foram geradas imagens de soma cumulativa de 3.000 quadros (TMR no canal 1) e o senhor no canal 2. SenhorHTL foi anexado para Tom20-HaloTag e TMRHTL para OxPhos complexo V-HaloTag. Cores são cores falsas. Barras de escala = 100 nm (B) e 1 µm (C). Clique aqui para ver uma versão maior desta figura.
-
O alinhamento físico do divisor de imagem imagens geradas
Nota: Para a montagem da amostra preparada em uma lamela, um self-made-porta-amostras e pode ser usado (Figura 2A). Para evitar a poeira, etc. , caindo a amostra, coloque a tampa do prato cultura frouxamente no topo da câmara, quando montado. O mesmo porta-amostras pode ser usado para montar a lamela com grânulos fluorescentes ou células; Quando as células são imaginadas, adicionar 0,5-0,8 mL de meio de imagem. O divisor de imagem divide a imagem em dois ou mais espectralmente separaram canais e projeta-los lado a lado sobre a mesma câmera. Este processo potencialmente introduz distorções sistemáticas entre os canais devido a distintos ópticos caminhos percorridos e dificulta a análise colocalization direto. Portanto, primeiro executar o alinhamento físico e segundo, pós-correção alinhamento com uma matriz de transformação. Para ambos os processos de alinhamento, grânulos fluorescentes devem ser distribuídos homogênea em todo o campo de visão.- Montar a amostra preparada com os grânulos fluorescentes no porta-amostra entre o politetrafluoroetileno (PTFE)-anel e o anel de borracha (Figura 2A).
- Inicie o microscópio, todos os componentes de hardware e todo o software necessário para microscopia.
- Limpe o objectivo e a parte inferior da lamela com uma limpeza de fiapos de tecido molhada com isopropanol. Depois seque os dois itens com um tecido fresco de fiapos. Coloque uma gota de óleo de imersão sobre o pupilo da lente objetiva.
- Coloque o porta-amostras com a amostra do grânulo para o palco do microscópio para que o fundo da lamela entra em contato com o óleo. Concentre-se em grânulos por transmissão da luz ou uma linha de laser.
- Ajuste a potência dos dois lasers excitação para atingir a intensidade de sinal semelhante nos dois canais de fluorescência. Procurar uma área com muitos distintos sinais fluorescentes.
- Gere uma exibição mesclada de canais de fluorescente usando o software de controle de câmera. Em seguida, use os parafusos no separador imagem manualmente inclinar os espelhos internos do separador imagem para atingir a melhor sobreposição dos sinais dos dois canais fluorescentes (Figura 2B).
Nota: atenção! Não exceda o intervalo dinâmico da câmera.
-
Alinhamento dos canais espectralmente separados pelo software executando transformação espacial
Nota: A parte seguinte mostra a pós-correção alinhamento e procedimento de localização com nosso software plugin (disponível mediante pedido).- Iniciar o microscópio TIRF software de controlo e escolha Exibir canais individuais no modo de transmissão ao vivo. Leve uma fluorescência emocionante imagem de instantâneo em todos os canais (Figura 2).
- Use esta imagem de instantâneo para produzir a matriz de transformação (ver Figura 2).
Nota: A matriz de transformação é usada para uma transformação espacial, normalmente um afim, que corrige para tradução (divergência de sinais de uma única fonte de ponto entre os dois canais). - Iniciar o plugin de análise de software (pode ser obtido mediante pedido do nosso laboratório, veja Figura 2).
- Carrega as imagens de cor dupla gravada anteriormente (ver passo 2.2) dos grânulos fluorescentes para o software. Escolha a orientação usada dos canais fluorescentes. Em seguida, clique em 'Sim' quando pediu 'calibrar imagens' e selecione o instantâneo anteriormente.
- Abra o "Gerenciador de unidade" para definir fatores de conversão de unidade (tamanho do pixel, taxa de quadros, fator de conversão de fótons).
- Abra o "Gerenciador de localização". Determine que o ponto de função (PSF) de espalhar primeiro. Pressione o botão: "Raio de PSF". Na janela "Estimador de PSF" que se abre, defina a abertura numérica e a máximo de emissão. Começar a "estimativa PSF raio" clicando. Aceite o obtidos experimental do PSF. definir a caixa de avaliação, o número de loops de deflação, e quantos núcleos do computador são utilizados para o cálculo. Pressione "localizar" para se adaptar a distribuição de intensidade de partículas única por uma função gaussiana simétrica 2D (Figura 2).
- "Aceite" o obtidos experimental do PSF. definir a caixa de avaliação, o número de loops de deflação, e quantos núcleos do computador são utilizados para o cálculo. Pressione "localizar" para se adaptar a distribuição de intensidade de partículas única por uma função gaussiana simétrica 2D (Figura 2).
- Abra o "Gerenciador de calibração". Na imagem renderizada mesclada de dois canais, são mostrados os sinais originais e os centros localizados. Escolha o modo "afim". Conecte manualmente os pares correspondentes dos centros localizados em dois canais que se originaram do grânulo fluorescente mesmo desenhando uma linha de conexão.
- Conecte os sinais correspondentes espalhados em todo o campo de visão. Após isto, pressione "aceitar". Salve a calibragem.
Nota: A transformação espacial é amostrada em cada grânulo fluorescente e interpolada entre. A função de transformação extraído representa um Δr(x,y) de campo de deslocamento que é usado para corrigir posteriormente as localizações experimental duplo-cor única molécula para que eles sobreposição dentro de sua precisão de localização. A matriz de transformação espacial é tipicamente um afim que corrige para tradução, escala e rotação entre canais com precisão de nanômetros, e pode ser inferida a partir deste mapeamento manual (Figura 2).
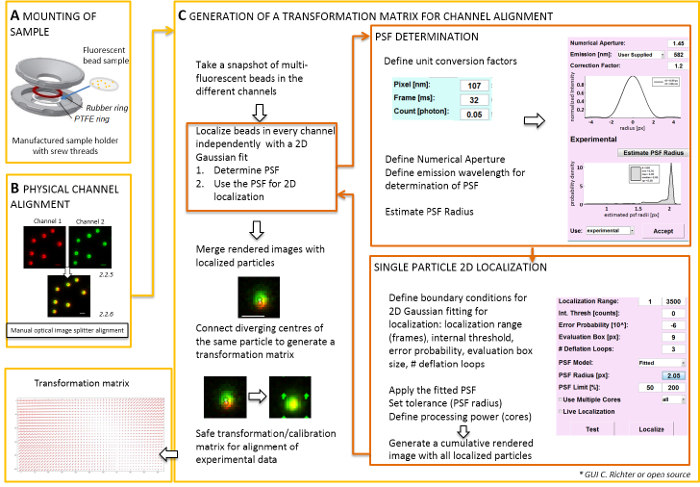
Figura 2 : Fluxo de trabalho para o alinhamento de dupla cor. (A), a lamela com os grânulos fluorescentes é montado em um suporte de amostra entre um PTFE e um anel de borracha. Em seguida, a parte superior e inferior da câmara são aparafusados. (B) alinhamento físico das vistas de canal que são gerados com o divisor de imagem. Gravou os sinais fluorescentes de missanga (0.1 µm) em dois canais (verde e vermelho, cores falsas) são mescladas. Os parafusos correspondentes no separador imagem óptica são virados manualmente até que seja alcançada a melhor sobreposição dos sinais diferentes (cor amarela, painel inferior). (C) geração de uma matriz de transformação para o alinhamento do canal post-processive. Para localização exata de uma partícula, é necessário determinar que o ponto de espalhar função (PSF) na dependência do comprimento de onda de emissão e a abertura numérica do objectivo. O centro de um PSF pode ser determinado pelo seu perfil de intensidade analisado por uma gaussiana bidimensional simétrica se encaixam. A localização resultante do pico do sinal é então projetada nos sinais originais, turva. Em uma imagem mesclada, os centros localizados dos sinais dos dois canais são conectados para gerar uma matriz de transformação que é usada mais tarde para o alinhamento post-processive dos dados experimentais. Barras de escala = 1 µm (B, C). Clique aqui para ver uma versão maior desta figura.
-
Imagem de molécula de proteínas de membrana mitocondrial
Nota: Todos os experimentos são realizados à temperatura ambiente. Células T ou células não-aderentes devem ser imobilizadas em agarose antes31de imagem.- Monte a amostra com células aderentes em lamela entre a borracha e anéis PTFE (Figura 3A). Encha a câmara com 0,5-0,8 mL de meio de imagem.
- Repita as etapas 2.2.2–2.2.5.
- Ajuste o microscópio TIRF de ângulo de iluminação controlando o software para criar um incidente ângulo menor do que o ângulo crítico para modo de TIR excitar a região específica de interesse através de uma folha de HILO32 (modo de HILO, figura 1A).
Atenção: Evite o contacto directo com o feixe de laser! - Definir o ganho EM e escolha um tempo de exposição adequado para o experimento que recolhe suficientes fotões por quadro.
- Defina a potência do laser para atingir um alto sinal-ruído (S/N) rácio (Figura 3B), desde que a precisão de localização corresponde diretamente ao S/N33 (Figura 3).
- Encontrar uma área na periferia de células com mitocôndrias não-sobreposição, alongadas e sinais de molécula (Figura 3D; Vídeo complementar 1). Se não há sinais de molécula são visíveis, espere até descoramento resultados no aparecimento dos sinais de molécula (Figura 3E).
- Registro até que o número de sinais é demasiado baixo para continuação razoável (geralmente de 1.000-10.000 quadros dependendo do comportamento de descoramento do corante fluorescente, Figura 3F).
- Começa a imagem processamento software e verificação de estruturas mitocondriais, gerando uma imagem de soma cumulativa de processado de pelo menos 1.000 quadros gravados (Figura 3).
Nota: O mais rápido possível quadro taxa é ditado pela área de leitura. O campo de visão para um canal é reduzido por um divisor de imagem dual-cores (512 x 512 pixels) para 256 x 512 pixels e para um Quad-cores para 256 x 256 pixels. Assim, para o uso de um divisor de imagem para duas cores, conjuntode 30 Hz. o modo de transferência de quadro para conseguir o menor tempo possível de leitura. - Inicie o software análise plugin e carga de dados brutos. Selecione o canal de orientação e carga de imagens. Use a matriz de transformação da etapa 2.3.9 quando perguntado para "Calibrar imagens". Canais serão exibidas separadamente.
- Abra o "Gerenciador de unidade" como antes para definir fatores de conversão de unidade para cada canal. Abra o "Gerenciador de localização" para cada canal. Definir a caixa de avaliação, o número de loops de deflação, em seguida, adicione o PSF teórico para as condições utilizadas e definir quantos núcleos do computador são utilizados para o cálculo. Finalmente, pressione "localizar" para obter partículas única localizadas (Figura 3 H; Vídeo complementar 2).
- Nota que o programa finalmente irá gerar uma imagem de superresolution cumulativa mostrando todos localizados partículas (Figura 3I).
- Realizar a análise, por exemplo, pelo software de fonte aberta ou nosso software disponível mediante solicitação.
- Rastrear as moléculas simples em ambos os canais localizados, por exemplo, com o rastreamento de alvos múltiplos de10
Nota: Passo 2.4.13 precisa de preliminar (experimental) conhecimento sobre a difusibilidade das proteínas de interesse para definir as condições de contorno corretamente. Geralmente, encontrar as condições de contorno corretas é um processo iterativo.
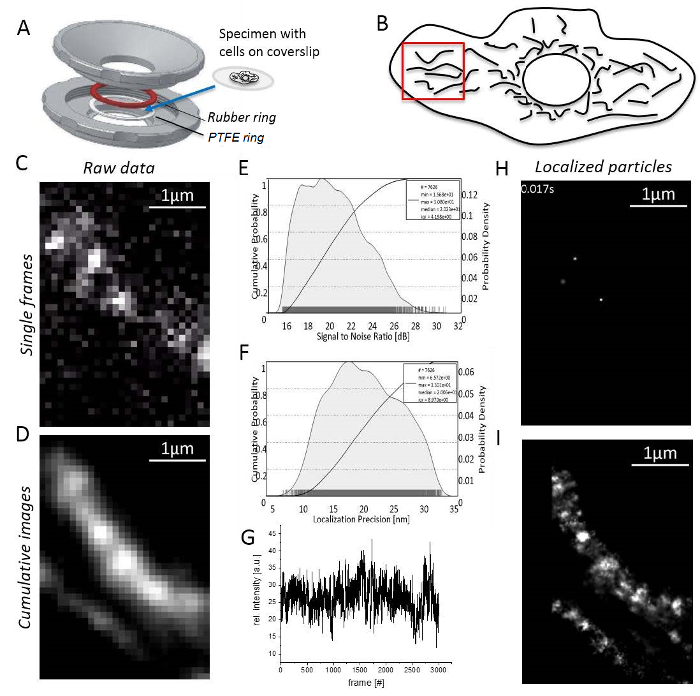
Figura 3 : Passos durante a microscopia de localização única molécula. (A), A lamela com o espécime é montada entre a parte superior e inferior (cinza) do porta-amostras caseiro (projetado por J. Bereiter-Hahn). Um anel de borracha (vermelho) e um anel PTFE (branco) do selo do sistema de acima e abaixo da lamela, quando as partes do porta-amostras são parafuso juntos. (B) relação sinal-ruído do sinal TMR. (C) calculado histograma de precisão de localização de partículas está localizadas. (D) escolha de uma região razoável de imagem, aqui, na periferia de células com mitocôndrias claramente separadas. (E) gravação e processamento de imagem: um único quadro com sinais distintos única molécula é mostrado (aqui, moléculas simples de CV-HaloTag/TMRHTL foram gravadas). (F) intensidade de TMR ao longo do tempo de gravação. Imagem de soma cumulativa (G) de 3.000 quadros, não transformados. (H) partículas de CV-HaloTag/TMRHTL localizados com uma função gaussiana 2D de um único quadro. (eu) cumulativa, processado sum imagem mostrando todas as partículas de CV-HaloTag/TMRHTL localizadas de 3.000 quadros. Clique aqui para ver uma versão maior desta figura.
Access restricted. Please log in or start a trial to view this content.
Resultados
Análise de imagem e colocalization multi cor pode ajudar a determinar a localização sub-organellar das proteínas. Temos demonstrado isso antes com a fosfatase citosólica e homólogo tensin, PINK1, que tem diferentes submitocondrial locais devido ao seu processamento por proteases mitocondrial17. PINK1 é um fator importante, garantindo a funcionalidade mitocondrial34,35. Para determinar que a localiza?...
Access restricted. Please log in or start a trial to view this content.
Discussão
Aqui, uma técnica para localização de dupla cor única molécula de proteínas da membrana celular foi apresentada. Seguindo o protocolo, proteínas de membrana são fundidos para auto-rotulantes proteínas que reagem com as tinturas de rodamina TMR e senhor conjugada com seus respectivos substratos. Rodamina corantes são brilhante e fotoestável e assim permitir a imagem repetitiva1. Para o desempenho bem sucedido, vários tópicos críticos e condições devem ser mantidas em mente.
Access restricted. Please log in or start a trial to view this content.
Divulgações
Os autores não têm nada para divulgar.
Agradecimentos
Os autores gostaria de agradecer o grupo de Biofísica e Jacob Piehler na Universidade de Osnabrück, para suporte contínuo, Wladislaw Kohl para assistência técnica e preparação de material e diretoria CellNanOs para fornecimento de microscópios para uso. O projeto foi financiado pelo SFB 944.
Access restricted. Please log in or start a trial to view this content.
Materiais
| Name | Company | Catalog Number | Comments |
| (2-(4-(2-hydroxyethyl)-1-piperazinyl)-ethanesulfonic acid, 1 M) (HEPES) | Biochrom | #1104E | |
| DC1: Dichroid beam splitter | Chroma | 640 dcxr | NC506031 |
| DC2: Polychroic Mirror, beamsplitter | Chroma | zt405/488/561/640rpc | discontinued |
| Dulbecco´s Phosphate-Buffered Saline (PBS) 1x (w/o Ca & Mg) | Sigma-Aldrich & Co. | #RNBF8311 | |
| Earle´s MEM without phenol red, without L-Glutamine and without NaHCO3 containing 1% FBS, 0.1% HEPES, 0.1% NEAA, 0.1% Alanyl-L-Glutamine and 34.78% sodium hydrogen carbonate (NaHCO3 0.75g/l) | Imaging medium | ||
| Earle´s minimum essential medium (MEM) with phenol red, containing 1% Fetal Bovine Serum Superior (FBS), 0.1% HEPES (2-(4-(2-hydroxyethyl)-1-piperazinyl)-ethanesulfonic acid, 1 M), and 0.1% non-essential amino acids (NEAA) | Growth medium | ||
| EF: Emission filter quadbandpass | AHF analysentechnik | F72-866 | Brightline HC 446 nm/523 nm/600 nm/677 nm |
| EMCCD camera | Andor | Andor iXON 897 | EMCCD camera |
| Emission filter QuadView filter cubes, orange | AHF analysentechnik | F39-637 | bandpass 582 - 619 nm |
| Emission filter QuadView filter cubes, red | Chroma | bandpass 655 - 725 nm (HQ 690/70) | |
| FBS (Fetal bovine serum) superior | Biochrom | S0615 | |
| Fluorescent beads: TetraSpeck™ Microspheres, 0.1 µm, fluorescent blue/green/orange/dark red | Thermo Fisher Scientific | T7279 | fluorescent microspheres |
| Glutamine | Biochrom | #0951C | |
| HeLa cells | DSMZ | ACC-57 | Cervical carcinoma cells from patient Henrietta Lacks |
| Hela cells CI::paGFP, stable | Muster et al., PLOSOne 2010 | ||
| Hela cells CV g::Halo7-Tag, stable | Appelhans et al., NanoLett 2012 | ||
| Hela cells Tom20::Halo7-Tag, stable | Appelhans et al., NanoLett 2012 | ||
| Image splitter | Photometrics | Dual-View QV2 | image splitter emission |
| Imaging processing software | ImageJ2 / Fiji | freeware | |
| Immersion Oil - ImmersolTM 518 F (ne = 1.518, ve = 45) | Carl Zeiss Jena GmbH | 444960-0000-000 | |
| Inverted epifluorescence microscope | Olympus IX-71/73/83 | ||
| Laser 561 nm, 200 mW | CrystaLaser | CL-561-200 | 561 nm emission |
| Laser 642 nm, 140 mW | Omicron | Luxx-642-140 | 642 nm emission |
| MATLAB | MathWorks | version R2013a | |
| MEM with Earle's Balanced Salt Solution 2.2 g/L NaHCO3, stable glutamine w/o PR | Biochrom | FG-0385 | |
| MEM with Earle's Balanced Salt Solution with 2.2 g/L NaHCO3, stable glutamine, Phenolred | Biochrom | FG-0325 | |
| MitoTracker® Deep Red FM | Thermo Fisher Scientific | M22426 | dye |
| MitoTracker® Green FM | Thermo Fisher Scientific | M7514 | dye |
| Multi-mode-optical polarization maintaining monomode fiber | Pointsource/Qioptiq | KineFLEX | |
| NHS-PEG-MAL, Rapp Polymer | Rapp Polymere GmbH Tübingen | coverslip coating | |
| non-essential amino acids (NEAA) | Biochrom | #0802E | |
| PEG 800 (Polyethylene glycol) 10 % | Carl Roth GmbH | Art No. 0263.1 | coverslip coating |
| Penicillin/Streptomycin | Biochrom | #0122E | |
| Plasmid for PINK1-Halo7-Tag expression | Beinlich et al., ACS Chemical Biology 2015 | ||
| Poly-L-lysine (1.2 mg/ml) | Sigma-Aldrich & Co. | Cat. No.P9155 | coverslip coating |
| RGD Peptide (Ac-CGRGDS-COOH) | Coring System Diagnostix GmbH, Gernsheim | coverslip coating / Intergrin receptor motif | |
| Silicon Rhodamine linked to HaloTag®-Ligand (SiRHTL) | personal gift from Kai Johnson | dye | |
| Software analysis plugin | self-written C. P. Richter, Biophysik Osnabrück | SLIMFAST 16g | |
| Tetramethylrhodamine / SNAP-Cell® TMR-Star linked to SNAP-Ligand (TMRstar) | New England Biolab® | S9105S | dye |
| Tetramethylrhodamine linked to HaloTag®-Ligand (TMRHTL) | Promega | G8251 | dye |
| TIRF condensor | Olympus | Cell^TIRF MITICO System | TIRF condensor |
| TIRF microscope controlling software | Olympus cellSens 1.12 | ||
| TIRF objective | Olympus | 150x oil objective (N.A. 1.45; Olympus UAPO) | |
| Trypsin/EDTA 10x | Biochrom | #0266 | |
| Water H2O 99,5 % Rotipuran® Low organic | Carl Roth GmbH | Art. No. HN57.1 |
Referências
- Appelhans, T., Richter, C., Wilkens, V., Hess, S., Piehler, J., Busch, K. Nanoscale organization of mitochondrial microcompartments revealed by combining tracking and localization microscopy. Nano Letters. 12 (2), 610-616 (2012).
- Appelhans, T., Busch, K. Single Molecule Tracking and Localization of Mitochondrial Protein Complexes in Live Cells. Methods Mol Biol. 1567, 273-291 (2017).
- Rust, M. J., Bates, M., Zhuang, X. Sub-diffraction-limit imaging by stochastic optical reconstruction microscopy (STORM). Nat Methods. 3 (10), 793-795 (2006).
- Gould, T. J., Verkhusha, V. V., Hess, S. T. Imaging biological structures with fluorescence photoactivation localization microscopy. Nat Protoc. 4 (3), 291-308 (2009).
- Pennacchietti, F., Gould, T. J., Hess, S. T. The Role of Probe Photophysics in Localization-Based Superresolution Microscopy. Biophys J. 113 (9), 2037-2054 (2017).
- Wegel, E., Göhler, A., Lagerholm, B. C., Wainman, A. Imaging cellular structures in super-resolution with SIM, STED and Localisation Microscopy: A practical comparison. Scientific reports. , Available from: https://www.nature.com/articles/srep27290?WT.feed_name=subjects_physical-sciences (2016).
- Pellett, P., et al. Two-color STED microscopy in living cells. Biomedical Optics Express. 2 (8), (2011).
- Ishigaki, M., et al. STED super-resolution imaging of mitochondria labeled with TMRM in living cells. Mitochondrion. 28, 79(2016).
- Liss, V., Barlag, B., Nietschke, M., Hensel, M. Self-labelling enzymes as universal tags for fluorescence microscopy, super-resolution microscopy and electron microscopy. Scientific Reports. 5, 17740(2015).
- Sergé, A., Bertaux, N., Rigneault, H., Marguet, D. Dynamic multiple-target tracing to probe spatiotemporal cartography of cell membranes. Nat Methods. 5 (8), (2008).
- Appelhans, T., Busch, K. B. Dynamic imaging of mitochondrial membrane proteins in specific sub-organelle membrane locations. Biophysical reviews. 9 (4), 345-352 (2017).
- Wilmes, S., et al. Triple-color super-resolution imaging of live cells: resolving submicroscopic receptor organization in the plasma membrane. Angewandte Chemie. 51 (20), 4868-4871 (2012).
- Niewidok, B., et al. Single-molecule imaging reveals dynamic biphasic partition of RNA-binding proteins in stress granules. J Cell Biol. , (2018).
- Wurm, C. A., Jakobs, S. Differential protein distributions define two sub-compartments of the mitochondrial inner membrane in yeast. FEBS Lett. 580 (24), 5628-5634 (2006).
- Schmidt, R., Wurm, C. A., Punge, A., Egner, A., Jakobs, S., Hell, S. W. Mitochondrial cristae revealed with focused light. Nano Lett. 9 (6), 2508-2510 (2009).
- Kukat, C., Wurm, C. A., Spahr, H., Falkenberg, M., Larsson, N. G., Jakobs, S. Super-resolution microscopy reveals that mammalian mitochondrial nucleoids have a uniform size and frequently contain a single copy of mtDNA. Proceedings of the National Academy of Sciences of the United States of America. 108 (33), 13534-13539 (2011).
- Beinlich, F., Drees, C., Piehler, J., Busch, K. Shuttling of PINK1 between Mitochondrial Microcompartments Resolved by Triple-Color Superresolution Microscopy. ACS chemical biology. 10 (9), 1970-1976 (2015).
- Shim, S., et al. Super-resolution fluorescence imaging of organelles in live cells with photoswitchable membrane probes. Proceedings of the National Academy of Sciences. 109 (35), 13978-13983 (2012).
- Sbalzarini, I., Mezzacasa, A., Helenius, A., Koumoutsakos, P. Effects of Organelle Shape on Fluorescence Recovery after Photobleaching. Biophysical Journal. 89 (3), 1482-1492 (2005).
- Reits, E., Neefjes, J. From fixed to FRAP: measuring protein mobility and activity in living cells. Nature Cell Biology. 3 (6), E145-E147 (2001).
- Goehring, N., Chowdhury, D., Hyman, A., Grill, S. FRAP Analysis of Membrane-Associated Proteins: Lateral Diffusion and Membrane-Cytoplasmic Exchange. Biophysical Journal. 99 (8), 2443-2452 (2010).
- Bacia, K., Haustein, E., Schwille, P. Fluorescence correlation spectroscopy: principles and applications. Cold Spring Harbor protocols. 2014 (7), 709-725 (2014).
- Sukhorukov, V., Dikov, D., Busch, K., Strecker, V., Wittig, I., Bereiter-Hahn, J. Determination of protein mobility in mitochondrial membranes of living cells. Biochimica et biophysica acta. 1798 (11), 2022-2032 (2010).
- Kim, T. K., Eberwine, J. H. Mammalian cell transfection: the present and the future. Anal Bioanal Chem. 397 (8), 3173-3178 (2010).
- Graham, F. L., van der Eb, A. J. A new technique for the assay of infectivity of human adenovirus 5 DNA. Virology. 52 (2), 456-467 (1973).
- Wedeking, T., et al. Spatiotemporally Controlled Reorganization of Signaling Complexes in the Plasma Membrane of Living Cells. Small. 11 (44), 5912-5918 (2015).
- Mironov, S. L., Ivannikov, M. V., Johansson, M. [Ca2+]i signaling between mitochondria and endoplasmic reticulum in neurons is regulated by microtubules. From mitochondrial permeability transition pore to Ca2+-induced Ca2+ release. J Biol Chem. 280 (1), 715-721 (2005).
- Poot, M., et al. Analysis of mitochondrial morphology and function with novel fixable fluorescent stains. J Histochem Cytochem. 44 (12), 1363-1372 (1996).
- Tokunaga, M., Imamoto, N., Sakata-Sogawa, K. Highly inclined thin illumination enables clear single-molecule imaging in cells (vol 5, pg 159). Nature Methods. 5 (5), 455(2008).
- Elmokadem, A., Yu, J. Optimal Drift Correction for Superresolution Localization Microscopy with Bayesian Inference. Biophys J. 109 (9), 1772-1780 (2015).
- Barlag, B., et al. Single molecule super-resolution imaging of proteins in living Salmonella enterica using self-labelling enzymes. Sci Rep. 6, 31601(2016).
- Tokunaga, M., Imamoto, N., Sakata-Sogawa, K. Highly inclined thin illumination enables clear single-molecule imaging in cells. Nature Methods. 5 (2), 159-161 (2008).
- Mortensen, K. I., Churchman, L. S., Spudich, J. A., Flyvbjerg, H. Optimized localization analysis for single-molecule tracking and super-resolution microscopy. Nat Methods. 7 (5), 377-381 (2010).
- Jin, S., Youle, R. J. PINK1- and Parkin-Mediated Mitophagy at a Glance. Journal of Cell Science. 125, 795-799 (2013).
- Yamano, K., Youle, R. J. PINK1 is degraded through the N-end rule pathway. Autophagy. 9 (11), 1758-1758 (2013).
- Wiedemann, N., et al. Machinery for protein sorting and assembly in the mitochondrial outer membrane. Nature. 424 (6948), 565-571 (2003).
- Thompson, R., Larson, D., Webb, W. Precise Nanometer Localization Analysis for Individual Fluorescent Probes. Biophysical Journal. 82 (5), 2775-2783 (2002).
Access restricted. Please log in or start a trial to view this content.
Reimpressões e Permissões
Solicitar permissão para reutilizar o texto ou figuras deste artigo JoVE
Solicitar PermissãoThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Todos os direitos reservados