Method Article
Tempo real RT-PCR do Pan-lyssavirus para o diagnóstico da raiva
Neste Artigo
Resumo
Este RT-PCR tempo real usando a tintura intercalando de dsDNA é apropriado diagnosticar infecções do lyssavirus. O método começa com o RNA extraído da raiva suspeita de amostras ante mortem ou post-mortem, detalhando a preparação da mistura mestra, adição de RNA, configuração da máquina em tempo real e interpretação correta dos resultados.
Resumo
Os ensaios moleculares são rápidos, sensíveis e específicos, e tornaram-se centrais para diagnosticar a raiva. Ensaios baseados em PCR têm sido utilizados há décadas para confirmar o diagnóstico de raiva, mas só recentemente foram aceitos pela OIE (Organização Mundial para a saúde animal) como método primário para detectar a infecção da raiva. Os ensaios de RT-PCR em tempo real fornecem dados em tempo real e são sistemas de tubos fechados, minimizando o risco de contaminação durante a instalação. Os ensaios de RT-PCR em tempo real intercalados com fluorocromo do DNA não necessitam de sondas caras, minimizando o custo por amostra, e quando os primers são projetados em regiões conservadas, os ensaios que são específicos em gêneros de vírus em vez de específicos a apenas um vírus espécies são possíveis. Aqui nós descrevemos um ensaio em tempo real do RT-PCR do Pan-lyssavirus SYBR que detecte lyssavirus através do género de lyssavírus , incluindo os vírus os mais DIVERGENTES ikov, WCBV e llebv. Em conjunto com a análise da curva de dissociação, este ensaio é sensível e específico, com a vantagem de detectar todas as espécies de lyssavirus. O ensaio foi adotado em muitos laboratórios diagnósticos com ambientes assegurados de qualidade, possibilitando diagnóstico robusto, rápido e sensível de casos de raiva animal e humana.
Introdução
O diagnóstico de raiva por meio de metodologias moleculares foi aceito pelo OIE em 20181, reconhecendo as vantagens dessas técnicas na confirmação de casos de raiva, particularmente em situações em que as amostras são subideais, ou para o diagnóstico ante mortem, como não há nenhuma exigência para o vírus vivo ou amostras frescas. Os ensaios do PCR para lyssavirus exigem uma transcrição reversa (RT) antes que o PCR possa começar porque o genoma é RNA. Ensaios de RT-PCR que detectam a 3 ' região proximal do genoma são considerados os mais sensíveis, pois os gradientes transcricionais ocorrem durante a replicação do lissavirus. Os ensaios de RT-PCR comumente usados podem ser divididos amplamente em duas categorias, ponto final (ou em gel) e em tempo real. Ambas as abordagens são sensíveis e específicas; no entanto, o ensaio em tempo real tem alguns benefícios adicionais, como a obtenção de resultados em "tempo real" e sendo realizado em um sistema de tubos totalmente fechados, reduzindo assim o potencial de contaminação do operador. Existem duas abordagens principais para detectar os amplicões específicos de lyssavirus obtidos por meio de ensaios em tempo real. O primeiro utiliza sondas de hidrólise (tais como sondas TaqMan) que contêm um fluoróforo e um quencher. Quando a sonda liga-se à região-alvo durante a amplificação, a atividade da de do polymerase resulta na dissociação do fluoróforo e do quencher, permitindo que a fluorescência resultante seja medida. O segundo utiliza um DNA intercalating corante (fluorocromo como SYBR verde) que se liga ao DNA encalhado duplo durante a amplificação. Os fluorochromes acoplados emitem a fluorescência que é detectada em cada ciclo, permitindo a deteção e a quantificação em tempo real do produto. Devido à natureza não específica da ligação a qualquer dsDNA, uma análise de curva de dissociação é realizada para confirmar a especificidade da reação. Os RT-PCRs tempo real são rápidos devido aos tamanhos pequenos do amplicon, tipicamente menos de 200 BP do comprimento; no entanto, identificar regiões adequadas para projetar primers e sondas em regiões conservadas, pode revelar-se desafiador, portanto, remover a exigência de uma sonda é uma vantagem distinta.
Um número de RT-PCRs em tempo real foram projetados para detectar especificamente cepas individuais ou linhagens de rabv2 e também para detectar lyssavirus em todo o gênero3,4,5,6, 7,8,9. Todos os ensaios terão um limite de detecção dependente de como conservou a cartilha (e, se necessário, a sonda) seqüências são em todo o gênero. Com efeito, as estirpes emergentes ou novas de vírus podem tornar ineficazes os ensaios baseados em sonda altamente específicos. A escolha da deteção do tempo real (tintura contra a ponta de prova) dependerá da aplicação pretendida. Para um laboratório que conduz a fiscalização no material localmente originado do cérebro e que espera números elevados de amostras negativas, o uso da tintura intercalating mais barata é uma escolha sensata. A abordagem SYBR Green também seria ideal ao realizar a vigilância de varredura, onde a presença de lyssavirus novos ou divergentes permaneceria sem ser detectada por ensaios mais restritos baseados em sonda.
Todos os membros do gênero lyssavirus causam a raiva da doença, que é fatal uma vez que os sintomas aparecem. A grande maioria dos casos de raiva humana e animal é devido ao vírus da raiva (RABV), o reservatório dominante para o qual é o cão doméstico10. Os morcegos são importantes reservatórios hospedeiros para os lissavírus e todas as espécies de lyssavirus, mas duas caracterizadas, foram identificadas diretamente em morcegos — Ikoma lyssavirus (IKOV) e Mokola Virus (MOKV) — e destes dois, o IKOV tem sido especulado para ter um reservatório de hospedeiro morcego 11. além do que as 16 espécies reconhecidas do lyssavirus12, há dois lyssavirus que foram descritos recentemente: lyssavírus do bastão de Formosa (twblv)13 e lyssavirus do bastão de KOTALAHTI (KBLV)14. Os lissavírus podem ser geneticamente divididos em três fitógrupos, com a maioria dos lissavírus, incluindo o RABV, pertencente ao phylogroup I. Entretanto, os lyssavirus os mais divergentes pertencem ao phylogroup III e são improváveis ser detectados por RT-PCRs projetados alvejar as seqüências do vírus de RABV ou de phylogroup I.
O ensaio descrito aqui utiliza o par de primers em tempo real JW12-N165, descrito pela primeira vez em 20053. Os primers foram projetados para ser Pan-lyssavirus embora a aplicação original foi como um ensaio TaqMan com sondas para diferenciar espécies de lyssavirus. A confirmação subseqüente que o par da primeira demão era Pan-lyssavirus na especificidade foi conseguida utilizando um teste do tempo real de SYBR de 2 etapas em todas as espécies do lyssavirus disponíveis que incluem WCBV15. O par da primeira demão é descrito aqui em um ensaio em tempo real do RT-PCR de uma etapa que utiliza um fluorochrome de intercalação, validado usando representantes de todas as 16 espécies reconhecidas do lyssavirus. Este ensaio em tempo real de uma etapa é um ensaio rápido, sensível, de lissavirus-específico e demonstra que a robustez da primeira demão ajustou-se para identificar mesmo a espécie altamente divergente do lyssavirus.
Protocolo
Amostras de material de diagnóstico recebidos na APHA após infecção natural, ou obtidas por camundongos inoculantes utilizando protocolos avaliados pelo Comitê de ética e estatística da APHA o estatuto de Home Office do Reino Unido licença 70/7394.
1. quantificação do RNA usando um Espectrofotômetro de microvolume
- Assegure-se de que os ajustes do espectrofotômetro estejam ajustados ao RNA.
- Use 1-2 μL de água de grau molecular para inicializar a máquina e definir uma linha de base.
- Use 1-2 μL de cada amostra de RNA de teste para avaliar a quantidade de RNA.
- Guarde as leituras e o documento.
- Ajuste o RNA para 1 μg/μL, se necessário.
Nota: O RNA deve ser mantido no gelo (ou em um bloco fresco) em todas as vezes. Se o RNA é obtido usando uma coluna, ou o método grânulo-baseado o RNA é geralmente menos de 1 μg/μL. Nesta situação use o RNA puro.
2. preparação da série de diluição de RNA para determinar a sensibilidade do ponto final
-
Faça uma diluição em série de 10 vezes do RNA.
- Os tubos da etiqueta com a série da diluição (por exemplo, 10-1, 10-2, etc.) e os detalhes do RNA.
- Adicionar 45 μL de água de grau molecular a cada tubo.
- Adicionar 5 μL do RNA (previamente diluído a 1 μg/μL) e misturar bem.
- Elimine a ponta da pipeta no desinfetante apropriado e substitua-a por uma ponta fresca.
- Tome 5 μL do 10-1 e adicione ao tubo 10-2 e misture bem.
- Repetir de 2.1.3 a 2.1.5 com as restantes diluições.
Nota: O RNA deve ser mantido no gelo (ou em um bloco fresco) em todas as vezes.
3. preparação de reações de RT-PCR em tempo real
- Usando uma planilha, planeje o layout da placa de acordo com o número de amostras de teste e amostras de controle, tanto para os ensaios de lyssavirus quanto de ß-actina.
Nota: Se 4 amostras devem ser testadas em duplicado com um controle positivo e negativo, isso equivale a 10 reações para ambos os ensaios. - Em uma "sala limpa" ou área separada do modelo de RNA, limpe as superfícies com um desinfetante apropriado antes de usar ou preparar uma estação de trabalho de PCR (se estiver usando). Para preparar a estação de trabalho, limpe a superfície do armário com um apropriado desinfectar e coloc os artigos exigidos na estação de trabalho e feche as portas. Ligue a luz UV durante 10 minutos
- Remover regentes e iniciadores do freezer e descongelar (reagentes listados na tabela 1 e primers na tabela 2).
Nota: A mistura enzimática é armazenada em glicerol por isso não requer descongelar e deve ser mantido no gelo (ou em um bloco fresco) em todos os momentos. Todos os outros reagentes podem ser descongelados à temperatura ambiente. - Uma vez descongelados, misture os reagentes e centrifugue brevemente para recolher o líquido.
Nota: Não vórtice a mistura enzimática, basta centrifugar brevemente. - Prepare misturas mestras separadas para o lyssavirus e o ß-Actin. Para cada reacção, adicionar 7,55 μL de água de grau molecular, 10 μL de 2x mistura de reacção verde universal SYBR, 0,6 μL de primer para a frente, 0,6 μL de primer reverso, 0,25 μL de mistura enzimática iTaq RT.
- Use uma planilha para calcular os volumes corretos para evitar erros no cálculo manual. Assegure-se de que o Master-Mix esteja preparado para compensar o erro de pipetagem. Portanto, se forem necessárias 10 reacções (ver nota em 3,1), prepare 12 reacções
- Prepare o mestre-misture no gelo (ou em um bloco fresco) e permaneça no gelo até que coloc na máquina do tempo real.
- Misture os Master-Mixes preparados, centrifugue brevemente e dispense 19 μL nos poços relevantes de tubos de strip ou uma placa de 96 poços compatível com a máquina em tempo real em uso.
Nota: Minimize a produção de bolhas nos poços enquanto pipetagem. - Em um quarto separado, ou em um armário UV preparado como descrito em 3,2, adicione com cuidado 1 μL do RNA ajustado previamente a 1 μg/μL (veja a etapa 1,5) abaixo da superfície do mestre-mistura apropriado bem e misture delicadamente. Descarte a ponta da pipeta em desinfetante diretamente após o uso (sob a superfície).
- Adicione os controles após as amostras de teste, com o controle positivo adicionado a seguir e o controle sem modelo (NTC – água de grau molecular) adicionado por último. A quantidade de RNA usada pode ser alterada dependendo do tipo de amostra, e a extração de RNA usada. A quantidade usada deve ser validada para garantir que a reação seja otimizada.
- Placa do selo usando as tampas ou o aferidor de tira que tomam o cuidado para assegurar-se de que todas as tampas estejam firmemente fechadas e rotuladas suficientemente para orientar as amostras. Rotule a borda dos tubos da placa/tira.
- Gire para baixo as amostras usando uma centrífuga para recolher todo o líquido na parte inferior dos poços.
- Placa de transferência à máquina do PCR do tempo real, abra a porta e coloc no suporte assegurando a posição/orientação corretas das amostras de acordo com a disposição da placa.
Nota: Se as tiras do tubo forem usadas, assegure-se de que o suporte esteja no lugar. - Abra o programa da máquina do PCR do tempo real e escolha a opção para o experimento em tempo real de SYBR com curva da dissociação. Programe a máquina de PCR em tempo real usando as condições de ciclagem térmica especificadas na tabela 3, incluindo os pontos de coleta de dados.
- Selecione SYBR como o corante fluorescente e selecione desconhecido como o tipo de amostra e insira um nome na caixa de nome de amostra correta.
Nota: Diferenciar entre as repetições e também entre os poços de lyssavirus e ß-actina. - Escolha um local de arquivo para salvar os dados experimentais, certifique-se de que a lâmpada será desligada no final da corrida e inicie a execução.
Nota: Como o primeiro passo é um estágio RT, nenhum dado é coletado durante esse tempo, portanto, se a lâmpada exigir um período de aquecimento, isso pode ocorrer durante o estágio RT. A máquina e o software em tempo real exibirão as curvas de amplificação em tempo real, enquanto a curva de fusão será gerada no final do ciclo.
4. análise de dados
-
Depois que a execução tiver sido concluída, execute a análise de dados da seguinte maneira.
- Primeiro analise os resultados do gráfico de amplificação das amostras de teste ao lado das amostras de controle. As amostras positivas exibem rampas exponenciais, geralmente seguidas de platô e um valor de Ct . Amostras negativas exibem parcelas de amplificação planas sem valores de Ct (Figura 1a). O valor de Ct é calculado automaticamente pelo software, embora isso deva ser verificado e alterado manualmente, se necessário.
- Em segundo lugar, analisar os resultados da curva de dissociação das amostras de teste ao lado das amostras de controle. Uma amostra positiva terá uma temperatura de derretimento (Tm) 77 – 80 ° c, e sobrepõem-se com o controle positivo Figura 1b).
- Obtenha o resultado diagnóstico geral assegurando que os controles são válidos. Use a tabela 4 para interpretar os resultados em relação ao controle interno do ß-Actin. Se as amostras de controle positivo forem negativas e/ou as amostras negativas forem positivas, a corrida deve ser ignorada.
- Registre os valores de Ct e tm obtidos para o RNA de controle em um "cartão de controle" para permitir a análise de tendência e ajudar a identificar desvios na sensibilidade do ensaio.
Resultados
Seguindo o protocolo descrito acima, a sensibilidade do Pan-lyssavirus RT-PCR foi demonstrada em uma série da diluição do vírus padrão do controle (CVS) (Figura 1) e uma gama de outros lissavírus (A Figura 2EA Figura 3). O corante verde I de SYBR foi utilizado como um RT-PCR universal de uma etapa, onde a síntese do cDNA e a amplificação do PCR são executadas em um único tubo. Como a amplificação do alvo específico ocorre, mais corante é ligado, resultando em tempo real níveis aumentados de fluorescência. Todos os ensaios intercalating da tintura em tempo real devem ser interpretados em duas fases: amplificação e dissociação. A fase de amplificação é idêntica a qualquer amplificação em tempo real (Figura 1A). Há uma "fase inicial" linear durante os ciclos iniciais onde a amplificação do ADN não pode ser calculada devido ao sinal insuficiente em relação ao fundo. O comprimento disso está diretamente relacionado à quantidade de destino na amostra. Subseqüentemente, há uma fase exponencial onde a duplicação de moléculas do ADN está detectada e gravada. Finalmente, a fase de platô é alcançada (além das amostras altamente diluídas que não podem chegar a esta fase antes do término do programa). Nesta fase, a intensidade dos níveis de fluorescência para fora, devido ao esgotamento dos reagentes. As parcelas de amplificação observadas por meio de uma diluição serial de 10 vezes do CVS, conformadas com as parcelas esperadas (Figura 1AC) em que o ensaio de lyssavirus demonstrou uma sensibilidade mais elevada do que o ensaio ß-actina. A curva de dissociação foi calculada após a amplificação, onde o dsDNA foi dissociado em ssDNA por um aumento incremental na temperatura e a fluorescência monitorada em função da temperatura (Figura 1B, D-F). A temperatura limiar na qual o amplicon específico se dissocia em ssDNA, causou uma liberação de fluorescência, que foi medido pelo software termocicatrador (TM). Esta fase de dissociação forneceu dados sobre o tamanho do amplicon, permitindo ao usuário interpretar o resultado em comparação com um controle positivo, resultando em uma probabilidade insignificante de resultados falso-positivos. O TMobservado para CVS utilizando o ensaio de Pan-lyssavirus (Figura 1B) e ensaio ß-actina (Figura 1D) são distintos, e auxiliou o usuário a confirmar a análise correta do ensaio observando o TMobtidas daFigura 1E). Além disso, o CTe TMvalores entre as corridas e os operadores foi avaliado e demonstrou ser reprodutível (Tabela 5). O limiar utilizado para calcular o CTvalor foi calculado automaticamente pelo software e dependente de muitos fatores, incluindo o mix de reação ou instrumento utilizado. Sobre os 12 funcionamentos independentes a média CTfoi de 20,66 (DP 0,63) para o ensaio de lyssavirus e 27,5 (DP 1,13) para o ensaio de ß-actina. Em contraste, a variação observada no TMvalores foi marcadamente menor, devido à falta de influências externas sobre essa medida. Por exemplo, a média de TMpara o ensaio do CVS lyssavirus foi 78,92 (DP 0,16) (Tabela 5), quando comparado com a média de todos os lissavírus 78,81 ° c (DP 0,531) (Tabela 6EFigura 1F). Esta falta de variação no TMem toda a Lyssavirusgênero é vantajoso, pois o mesmo RNA controle pode ser utilizado independentemente do lyssavirus na amostra, porém diferenciando-se entre as espécies de lyssavirus usando o TMNão é possível, especialmente porque diferentes sublinhagens RABV estendeu a gama de TMvalores observados (Tabela 6). As parcelas não específicas da amplificação são observadas raramente com este ensaio; no entanto, são necessários parâmetros específicos para definir um resultado positivo versus um resultado negativo não específico. O DP observado em todos os lissavírus (0,531) foi aplicado ao menor (77,34-LBVa) e maior (79,67-IKOV) observou TMvalores para definir um intervalo de 76,8 ° c-80,2 ° c para bandas específicas positivas. Portanto, TMvalores fora desse intervalo foram considerados não específicos e, portanto, um resultado negativo. Ocasionalmente, vários picos são observados para uma amostra. Se o pico dominante está no T corretoM(para cada repetição) então a amostra é considerada positiva. A razão mais comum de um pico não-específico é observado é devido a primer-dímeros, o ensaio foi otimizado para minimizar dimers primer. Dímeros primer tipicamente resultam em um amplicon menor do que a seqüência de destino, portanto, teria um TMinferior ao produto específico.
Uma diluição em série de 10 vezes de três RNAs da amostra de cérebro positivo de lyssavirus extraída usando TRIzol, foi executada paralelamente e plotada (Figura 2). O limite de detecção para os três lissavírus variou, mas nenhum ultrapassou Ct 36. Os valores de coeficiente de R2 para os vírus plotados na Figura 2 variaram de 0,9637 e 0,996. Para todos os vírus analisados (tabela 6) o intervalo não excedeu isso, além disso, 7 dos 29 lyssavirus apresentaram R2 > 0,99 (dados não mostrados). Tendo em conta que a preparação da série de diluição é proveniente de extrações de RNA total, a linearidade observada fornece evidências de que o ensaio é robusto. Finalmente, a detecção de todas as espécies de lyssavirus (particularmente os mais diversos vírus do phylogroup III) foi investigada usando um painel de RNAs que mede todos os três phylogroups no género de lyssavirus . O RNA extraído de camundongos originais ou experimentalmente infectados, material cerebral, foi utilizado utilizando os protocolos descritos acima. Os resultados confirmam que os primers amplificam todas as espécies de lyssavirus, incluindo os lissavírus do fitófilo divergente III IKOV, WBCV e LLEBV (tabela 6 e Figura 3). Um painel diversificado de Rhabdovírus não-lissavírus, originalmente coletado e analisado antigenicamente16, e mais recentemente17 geneticamente foram rastreados e não-cross reatividade foi detectada, indicando que os primers são específicos para Apenas os membros do género lyssavirus (dados não mostrados). O ensaio em tempo real do Pan-lyssavirus foi incluído nos esquemas de proficiência interlaboratorial do EURL (laboratório de referência da UE) desde 2013, demonstrando 100% de concordância com outros ensaios moleculares, como o ensaio TaqMan do Pan-lyssavirus e o ensaio convencional de RT-PCR, além da FAT (teste de anticorpo fluorescente).
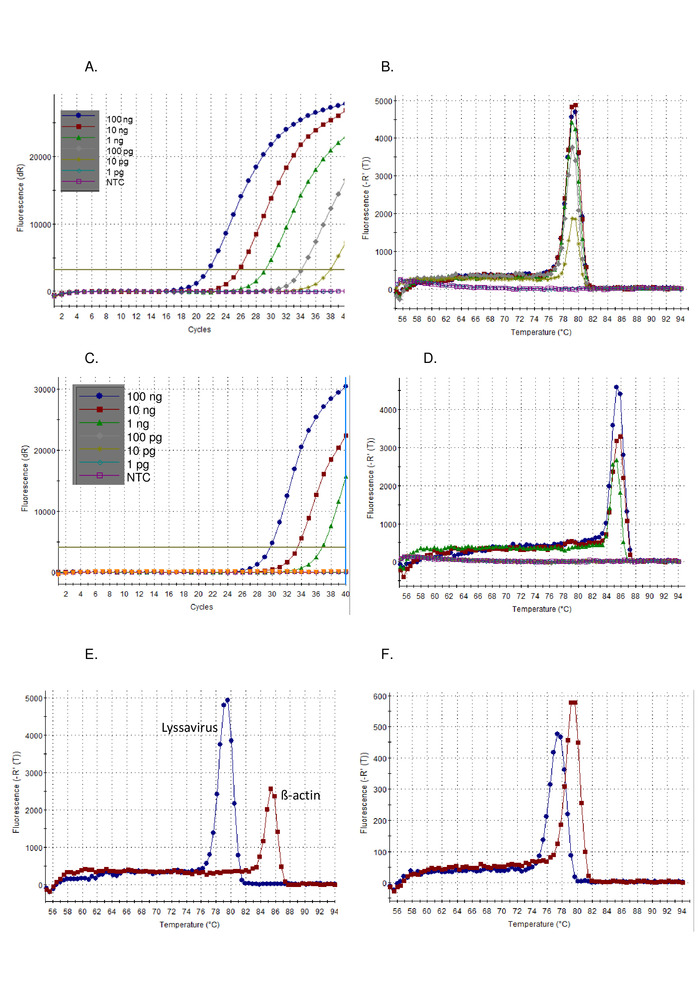
A figura 1:10-dobra a diluição em série do RNA de controle positivo de CVS, funcione no ensaio do RT-PCR do Pan-lyssavirus, visualizado como a parcela da amplificação (a), e a curva da dissociação (B), e funcione no ensaio do RT-PCR do ß-Actin visualizado como o lote da amplificação (C), e curva de dissociação (D). NTC = nenhum controle de modelo. A comparação do RNA de controle do CVS é executada no ensaio de RT-PCR do Pan-lyssavirus (azul) e no ensaio de RT-PCR de ß-actina (vermelho) demonstrando a diferença nas curvas de dissociação (E) — ver tabela 5 para valores médios; e, finalmente, curvas de dissociação para LBVa (azul) e IKOV (vermelho) demonstrando a amplitude dos valores de Tm observados em todo o gênero lyssavirus (F). Por favor clique aqui para ver uma versão maior desta figura.
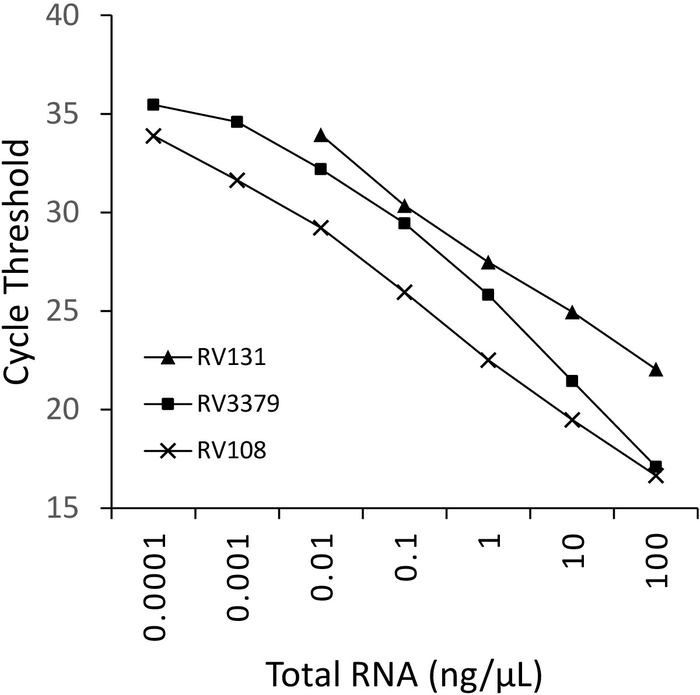
Figura 2:10-dobra diluições seriadas para três espécies de lyssavirus: RABV (RV108), DUVV (RV131) e ARAV (RV3379). R2 = 0,996, 0,9962 e 0,9637 respectivamente. Os pontos de dados em 10-7 (0, 1 ng/μL) atingiram o limite de detecção (onde um valor foi obtido). Por favor clique aqui para ver uma versão maior desta figura.
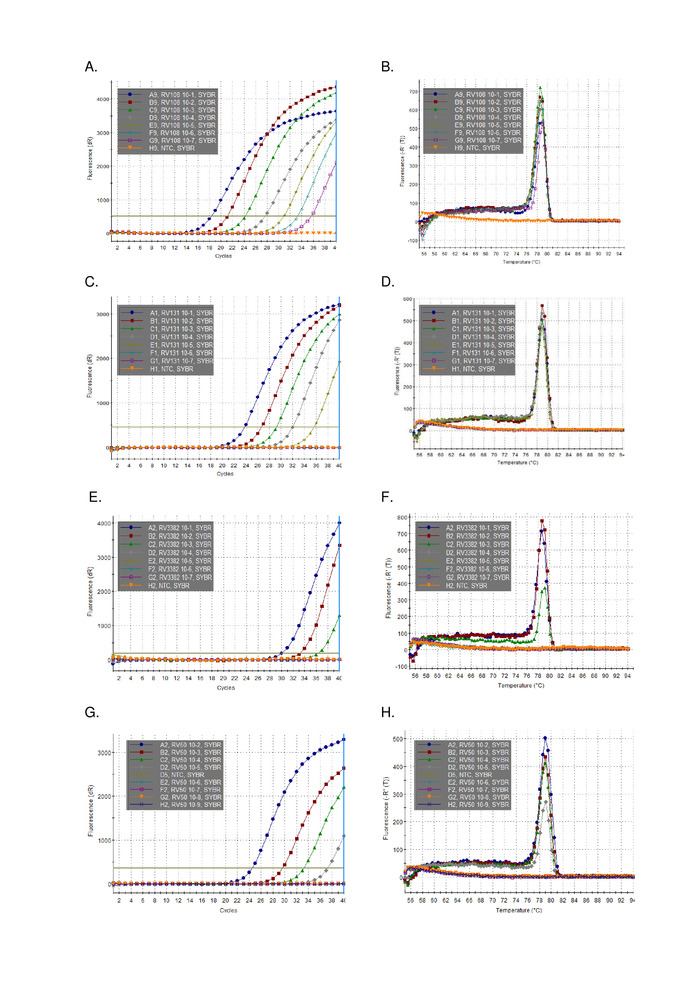
Figura 3: dados de diluição serial representativos de 10 vezes de todo o gênero lyssavirus (ver lendas individuais para a identidade de lyssavirus e tabela 6 para resultados tabulados em comparação com outros lissavírus). Painéis a, c, e, g , parcelas de amplificação e B, D, F, H curvas de dissociação para A, c, e e g, respectivamente. Por favor clique aqui para ver uma versão maior desta figura.
| Reagente | μL/reacção |
| Água da classe molecular | 7,55 |
| 2x mistura de reacção de PCR universal RT | 10 |
| Primer Forward [20 μM] | 0,6 |
| Primer reverso [20 μM] | 0,6 |
| Mistura enzimática RT | 0,25 |
| Total por reação | 19 |
Tabela 1: reagentes de mistura mestre de RT-PCR em tempo real do Pan-lyssavirus.
| Ensaio | Nome da cartilha | Papel primer | Sequência 5 '-3 ' | Posição1 |
| Lyssavirus | JW12 | RT-PCR | ATG TAA CAC CYC TAC AAT G | 53-73 |
| N165 | Pcr | GCA GGG TAY TTR TAC TCA TA | 165-146 | |
| ß-actina | ß-actina intronic | Pcr | CGA TGA AGA TCA AGA TCA TTG | 1051-1072 |
| ß-Actin reverso | RT-PCR | AAG CAT TTG CGG TGG AC | 1204-1188 | |
| As posições da primeira demão são dadas com relação à seqüência do vírus de Pasteur (M13215) e à seqüência do gene do ß-Actin do rato (NM_007393) | ||||
Tabela 2: detalhes do primer RT-PCR em tempo real do Pan-lyssavirus.
| Palco | Ciclos | Temperatura | Tempo | Coleta de dados |
| Transcrição reversa | 1 | 50 ° c | 10 minutos | |
| Inactivação de RT/desnaturação inicial | 1 | 95 ° c | 5 minutos | |
| Amplificação | 40 | 95 ° c | 10 de s | |
| 60 ° c | de 30 s | ponto final | ||
| Análise da curva de dissociação | 1 | 9 ° c | 1 minuto | |
| 55 ° c | 1 minuto | |||
| 55-95 ° c | 10 de s | todos os pontos |
Tabela 3: condições de ciclagem de RT-PCR em tempo real do Pan-lyssavirus.
| Resultado do teste | Controle interno do ß-Actin | Resultado geral |
| Negativo | Negativo1 | Inválido. Repita a extração e o ensaio2 |
| Negativo | Positivo | Resultado negativo relatado |
| Positivo | Positivo | Resultado positivo relatado |
| Positivo | Negativo1 | Repita a extração e o ensaio3 |
| 1. º O uso do controle externo heterólogo seria benéfico durante a extração da repetição. | ||
| 2. º Se um segundo resultado negativo for obtido para o controle interno, a amostra será relatada como não testável por este ensaio. | ||
| 3. º Se um segundo resultado negativo for obtido para o controle interno, ao lado de um resultado positivo do teste uma raiva secundária | ||
| o teste de diagnóstico deve ser empreendido para confirmar este resultado. | ||
Tabela 4: Resumo dos desfechos e resultados gerais do Pan-lyssavirus em tempo real RT-PCR. O negativo é designado a uma amostra sem o valor de Ct (amplificação) e nenhuma temperatura do derretimento (dissociação), ou uma temperatura do derretimento que seja fora da escala de tm para lyssavirus positivos (° c 76,8-80,2 ° c). O positivo é designado a uma amostra com um valor de Ct (amplificação) e uma temperatura do derretimento (dissociação) que seja dentro da escala de tm para lyssavirus positivos.
| Ensaio de lyssavirus | ensaio de ß-actina | |||
| Ct | Tm | Ct | Tm | |
| Significa | 20,66 | 78,92 | 27,5 | 85,26 |
| Sd | 0,63 | 0,16 | 1,13 | 0,35 |
| LCL (95%) | 19,39 | 78,59 | 25,23 | 84,56 |
| UCL (95%) | 21,93 | 79,25 | 29,76 | 85,96 |
Tabela 5: análise inter-Run do controle positivo de CVS através de 12 funcionamentos independentes que incluem operadores múltiplos.
| Phylogroup | Espécie | ID de vírus | Linhagem | Limite de detecção | Tm |
| Eu | RABV | RV50 | Morcego dos EUA | 10-5 | 79,5 |
| Eu | RABV | RV51 | Fox dos E.U. | 10-7 | 77,6 |
| Eu | RABV | RV108 | Bastão do Chile | 10-7 | 78,63 |
| Eu | RABV | RV313 | Fox Europeu | 10-9 | 78,53 |
| Eu | RABV | RV437 | RacDog Europeu | 10-7 | 78, 9 |
| Eu | RABV | RV1237 | Cervos europeus | 10-8 | 78,76 |
| Eu | RABV | RV334 | Vacina chinesa | 10-8 | 79, 3 |
| Eu | RABV | RV102 | África 2 | 10-7 | 78,58 |
| Eu | RABV | RV995 | África 3a | 10-8 | 79,66 |
| Eu | RABV | RV410 | África 3B | 10-7 | 79, 3 |
| Eu | RABV | RV2324 | África do Sul 4 | 10-7 | 79,17 |
| Eu | RABV | RV2417 | Cão de Sri Lanka | 10-9 | 78,71 |
| Eu | RABV | CVS-11 | 10-7 | 79,17 | |
| Eu | EBLV-1 | RV20 | Alemanha | 10-6 | 79, 5 |
| Eu | EBLV-2 | RV1787 | Reino unido | 10-7 | 78,76 |
| Eu | O BBLV | RV2507 | Alemanha | 10-9 | 78,71 |
| Eu | ABLV | RV634 | 10-8 | 78,25 | |
| Eu | O DUVV | RV131 | 10-5 | 79, 3 | |
| Eu | O GBLV | RV3269 | 10-7 | 79,15 | |
| Eu | Arav | RV3379 | 10-7 | 79,46 | |
| Eu | O KHUV | RV3380 | 10-7 | 78,97 | |
| Eu | A SHIBV | RV3381 | 10-7 | 78,59 | |
| Eu | O IRKV | RV3382 | 10-3 | 78,59 | |
| Ii | O LBVa | RV767 | 10-5 | 77,34 | |
| Ii | O LBVd | RV3383 | 10-7 | 78,59 | |
| Ii | O MOKV | RV4 | 10-3 | 78,81 | |
| Iii | IKOV | RV2508 | 10-5 | 79,67 | |
| Iii | O LLEBV | RV3208 | 10-4 | 79,15 | |
| Iii | A WCBV | RV3384 | 10-3 | 79 |
Tabela 6: síntese da especificidade do RT-PCR em tempo real do Pan-lyssavirus, sensibilidade e Tm para os lissavírus representativos em todos os três grupos de filosgroups. A média de Tm através de lyssavirus foi 78,81 (DP 0,531).
Discussão
O ensaio de RT-PCR em tempo real do Pan-lyssavirus descrito é um ensaio de tubo fechado, de uma etapa, que detecta os lissavírus em todos os três grupos de filosgroups. O teste foi validado para o diagnóstico animal e humano da raiva, incluindo o tecido de cérebro post-mortem (idealmente brainstem), e as amostras ante-mortem tais como a biópsia da pele, a saliva serialmente coletada, ou o líquido espinal cerebral (CSF). Os primers utilizados neste ensaio foram primeiro concebidos e utilizados para um ensaio à base de sonda para diferenciar entre RABV, EBLV-1 e EBLV-23, que tem sido utilizado em muitos laboratórios de raiva OIE e executa consistentemente nos esquemas de proficiência eurl. Subseqüentemente a natureza do ' Pan-lyssavirus ' destes primers foi confirmada usando um ensaio em tempo real de 2 etapas15. O ensaio descrito aqui utilizou os primers para otimizar ainda mais a RT-PCR em um ensaio SYBR de uma etapa, permitindo um sistema de tubo fechado, rápido. Além disso, o treinamento em países endêmicos de raiva usando este ensaio confirmou a adequação para implementar em qualquer laboratório com EPI básicos e sistemas de qualidade para reduzir a contaminação cruzada e amostras de rastreamento, instalações para armazenar os reagentes e um tempo real máquina com detecção SYBR. O ensaio é extremamente robusto e tem correlação de 100% com a FAT, com sensibilidade melhorada para amostras decompostas3. Uma das principais vantagens de um ensaio de DNA intercalante à base de corante em comparação com um ensaio baseado em sonda é o custo relativo. Um benefício adicional é que o ensaio só utiliza dois primers, portanto, há menos risco de detecção falhou devido à divergência de seqüência, que tem sido uma fraqueza em ensaios previamente publicados com base em sonda. De fato, representantes de todas as espécies de lyssavirus (além de TWBLV e KBLV) são detectados usando este ensaio, e a análise sequencial de TWBLV e KBLV nos sítios primários não revela nenhuma divergência significativa sugerindo fortemente que eles também serão detectados usando Esse método. O intervalo de limite de detecção observado em todos os vírus analisados pode ser considerado devido a dois fatores principais. O primeiro é que o RNA foi isolado do material clínico do cérebro, conseqüentemente a quantidade de cópias do genoma em cada amostra não diluída não é diretamente comparável. O RNA foi "normalizado" ajustando o RNA total a 1 μg/μL; Entretanto, a proporção de RNA do genoma viral dentro dessa amostra variará. A segunda é a diversidade de sequências de lyssavirus, apesar dos sítios primários estarem localizados em regiões conservadas, permanecem posições de variação. Portanto, não é surpreendente que a maioria dos lissavírus com menor sensibilidade para o ensaio sejam vírus phylogroup II e III. A análise da curva de dissociação representa um parâmetro essencial, minimizando um resultado falso positivo que, de outra forma, poderia ocorrer devido à formação de dímeros de primer, ou amplificação de uma região não específica no genoma hospedeiro. Na realidade, esta é uma ocorrência rara e a análise da curva da dissociação é equivalente a funcionar um gel do agarose para visualizar os amplicons convencionais corretamente feitos medida do RT-PCR. O intervalo de valores aceitáveis de Tm foi fornecido (77-80 ° c), com base nos dados coletados em nosso laboratório. Recomenda-se vivamente que os laboratórios individuais agrupem os dados em casa para garantir que a gama seja transferível e altere em conformidade. A interpretação dos resultados das parcelas de amplificação e dissociação, juntamente com os controles positivo e negativo e os resultados da ß-actina, possibilita resultados diagnósticos robustos e reprodutíveis.
Fora do escopo deste protocolo está o método de extração de RNA usado para obter RNA de alta qualidade. Todos os RNA analisados neste protocolo foram preparados utilizando-se TRIzol; Entretanto, há muitos protocolos apropriados da extração do RNA das extrações baseadas em guanidium disponíveis, incluindo os jogos coluna e grânulo-baseados da extração. A manipulação de amostras positivas (ou suspeitas positivas) do lyssavirus deve estar dentro das facilidades licenciadas da biocontenção aprovadas dentro do país. No entanto, o RNA total extraído é não infeccioso, portanto manuseado em laboratórios de baixa contenção. Dependendo do método de extração utilizado, a necessidade de quantificar e diluir o RNA precisaria ser avaliada. Para extrações à base de fenóis, incluindo TRIzol, esta etapa é necessária e impede a inibição do ensaio de contaminar gDNA; no entanto, as extrações baseadas em coluna e talão (particularmente aquelas com estágio de depleção de DNA) não necessitam de diluição antes do teste.
Durante todo o protocolo, é essencial que o cuidado e a diligência sejam usados para impedir a contaminação cruzada e a adição exata da amostra aos poços corretos. Uma planilha com os cálculos de reagentes e layout da placa está disponível para download como um arquivo suplementar. Boas práticas de laboratório, incluindo superfícies de trabalho limpas, mudanças regulares de luvas, uso de pontas de barreira e diferentes salas/armários UV para separar cada estágio irá minimizar a chance de contaminação. Para garantir que o teste está realizando com parâmetros esperados, controles positivos e negativos devem ser incluídos e todas as amostras de teste são executadas em duplicado (ou triplicado).
A inclusão de controles é uma característica essencial de qualquer PCR, particularmente para diagnósticos. O RNA de controle positivo foi preparado a partir de cérebros de mouse infectados pelo CVS (padrão de vírus de desafio) em lotes e validado e calibrado para garantir a consistência entre os lotes. O RNA controle foi quantificado e diluído em 1 μg/μL. o RNA para o qual um resultado positivo foi obtido em uma diluição em série até pelo menos 10-4 (igual a 100 PG/μL) foi considerado apto para a finalidade. O RNA de controle positivo foi armazenado em-80 ° c em 10-1 alíquotas. Quando necessário, uma alíquota foi diluída 1:100 para fornecer um estoque de trabalho em 1 ng/μL e armazenada em-80 ° c em 5 μL de alíquotas de uso único. O RNA de controle positivo diluído foi utilizado para representar amostras positivas de baixo nível, e para garantir que qualquer redução na sensibilidade do ensaio foi detectada. Um "cartão de controle" foi mantido para cada controle para monitorar os valores de Ct e identificar tendências (tabela 5). A tabela 5 demonstrou boa comparabilidade inter-Run para a amostra de controle positivo CVS Ct e tm valores em vários dias e operadores, proporcionando garantia de que o ensaio é robusto e reprodutível. Os resultados que se desviam dessas medições devem ser investigados e amostras de teste repetidas se necessário. A água da classe molecular foi incluída em cada funcionamento como um NTC para confirmar que os reagentes estavam livres da contaminação com RNA do lyssavirus e confirmam uma amostra negativa. Além disso, para assegurar a eficácia da extração do RNA, o ß-Actin foi testado ao lado das amostras de teste em um tubo separado. O RNA de controle positivo do lyssavirus foi usado igualmente para o controle positivo do ß-Actin. Outros genes endógenos ou sistemas de controle interno heterólogo podem ser utilizados. O uso desses controles garantiu que todas as etapas foram analisadas nas mesmas condições que as amostras de teste. Ocasionalmente, se a amostra foi altamente degradada ou não contiver RNA hospedeiro suficiente (como saliva ou LCR), o gene endógeno PCR pode falhar. Neste caso, onde o resultado de RT-PCR em tempo real do lyssavirus foi positivo, a confirmação de uma extração de RNA independente-para descartar a contaminação durante a extração do RNA no teste original ou por um teste secundário (ou molecular, como a RT-PCR convencional) , ou FAT. O uso da tabela 4 durante a análise de uma amostra diagnóstica garantiu a interpretação correta.
Independentemente do ensaio molecular utilizado para confirmar a infecção da raiva, as investigações de seguimento utilizando o sequenciamento de Sanger para determinar as espécies de lyssavirus e as técnicas clássicas em virologia como a FAT ou o isolamento de vírus também devem ser empreendidas para permitir caracterização dos vírus e apoiar a notificação de casos positivos ao OIE e à OMS.
Divulgações
Os autores não têm nada a revelar.
Agradecimentos
Os autores desejam agradecer a senhorita Emma Wise e Miss Megan Golding para obter ajuda para completar os experimentos. O desenvolvimento deste protocolo foi apoiado financeiramente pelo departamento do Reino Unido para o meio ambiente, alimentos e Assuntos rurais (DEFRA), Governo escocês e governo galês por subvenções [SV3500, e SE0431] e pelo projeto europeu de Virus Archive global (EVAg) que tem receberam financiamento do programa de investigação e inovação Horizon 2020 da União Europeia, a Convenção de subvenção n. o 653316.
Materiais
| Name | Company | Catalog Number | Comments |
| Art Barrier pipette tips (various sizes) | Thermofisher | various | |
| Centrifuge | Beckman | Allegra 21R | Rotor capable of holding 96 well plates required. Step 3.8. |
| Centrifuge (mico) | Sigma | ||
| Finnpipettes (to dispense 0.5-1,000 µL) | Thermofisher | various | |
| iTaq Universal SYBR Green One-Step RT-PCR kit | Bio-Rad | 172-5150 | Equivalent kits can be used if validated |
| MX3000P or MX3005P real-tme PCR system | Stratagene | N/A | Eqivalent machines can be used if validated |
| MicroAmp reaction plate base | Any suitable | Used to hold tube strip and plates securely. | |
| Optically clear flat clear strips (8) | ABgene | AB-0866 | |
| Perfect fit frame (if using tube strips) | Stratagene | N/A | Specific to machine |
| Primers: for primer details see Table 2. | Ordered at 0.05 µmole scale HPLC purified. | ||
| Thermo-Fast 96 well plares, non skirted | ABgene | AB-600 | |
| Thermo-Fast strips (8) Thermo-tubes | ABgene | AB-0452 | |
| Vortex machine / Whirlimixer | Fisons Scientific equipment | SGP-202-010J | |
| Unless stated, alternative equipment can be used |
Referências
- . OIE. OIE Terrestrial Manual 2018. , 8-14 (2018).
- Panning, M., et al. Comparative analysis of rabies virus reverse transcription-PCR and virus isolation using samples from a patient infected with rabies virus. Journal of Clinical Microbiology. 48 (8), 2960-2962 (2010).
- Wakeley, P. R., et al. Development of a real-time, TaqMan reverse transcription-PCR assay for detection and differentiation of lyssavirus genotypes 1, 5, and 6. Journal of Clinical Microbiology. 43 (6), 2786-2792 (2005).
- Wang, L., et al. A SYBR-green I quantitative real-time reverse transcription-PCR assay for rabies viruses with different virulence. Virologica Sinica. 29 (2), 131-132 (2014).
- Wadhwa, A., et al. A Pan-Lyssavirus Taqman Real-Time RT-PCR Assay for the Detection of Highly Variable Rabies virus and Other Lyssaviruses. PLOS Neglected Tropical Diseases. 11 (1), e0005258 (2017).
- Nadin-Davis, S. A., Sheen, M., Wandeler, A. I. Development of real-time reverse transcriptase polymerase chain reaction methods for human rabies diagnosis. Journal of Medical Virology. 81 (8), 1484-1497 (2009).
- Hoffmann, B., et al. Improved safety for molecular diagnosis of classical rabies viruses by use of a TaqMan real-time reverse transcription-PCR "double check" strategy. Journal of Clinical Microbiology. 48 (11), 3970-3978 (2010).
- Faye, M., et al. Development and validation of sensitive real-time RT-PCR assay for broad detection of rabies virus. Journal of Virological Methods. 243, 120-130 (2017).
- Dacheux, L., et al. Dual Combined Real-Time Reverse Transcription Polymerase Chain Reaction Assay for the Diagnosis of Lyssavirus Infection. PLOS Neglected Tropical Diseases. 10 (7), e0004812 (2016).
- Fooks, A. R., et al. Rabies. Nature Reviews Disease Primers. 3, 17091 (2017).
- Marston, D. A., et al. Ikoma lyssavirus, highly divergent novel lyssavirus in an african civet. Emerging Infectious Diseases. 18 (4), 664-667 (2012).
- . Virus Taxonomy: 2018 Release: Email ratification October 2018 (MSL #33) Available from: https://talk.ictvonline.org/taxonomy/ (2018)
- Hu, S. C., et al. Lyssavirus in Japanese Pipistrelle, Taiwan. Emerging Infectious Disease. 24 (4), 782-785 (2018).
- Nokireki, T., Tammiranta, N., Kokkonen, U. M., Kantala, T., Gadd, T. Tentative novel lyssavirus in a bat in Finland. Transboundary and Emerging Diseases. 65 (3), 593-596 (2018).
- Hayman, D. T., et al. A universal real-time assay for the detection of Lyssaviruses. Journal of Virological Methods. 177 (1), 87-93 (2011).
- Calisher, C. H., et al. Antigenic relationships among rhabdoviruses from vertebrates and hematophagous arthropods. Intervirology. 30 (5), 241-257 (1989).
- Aznar-Lopez, C., et al. Detection of rhabdovirus viral RNA in oropharyngeal swabs and ectoparasites of Spanish bats. Journal of General Virology. 94 (1), 69-75 (2013).
Reimpressões e Permissões
Solicitar permissão para reutilizar o texto ou figuras deste artigo JoVE
Solicitar PermissãoExplore Mais Artigos
This article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Todos os direitos reservados