É necessária uma assinatura da JoVE para visualizar este conteúdo. Faça login ou comece sua avaliação gratuita.
Method Article
Um modelo de cultura de órgãos proinflamatório e degenerativo para simular a doença do disco intervertebral em estágio inicial.
* Estes autores contribuíram igualmente
Neste Artigo
Resumo
Este protocolo apresenta um novo modelo experimental de cultura de órgãos bovinos proinflammatórios e degenerativos para simular a degeneração do disco intervertebral em estágio inicial.
Resumo
A degeneração do disco intervertebral sintomático (IVD) é uma grande carga socioeconômica e é caracterizada por inflamação e degradação tecidual. Devido à falta de terapias causais, há necessidade urgente de modelos inovadores de cultura de órgãos experimentais para estudar os mecanismos envolvidos na progressão da doença, encontrar metas terapêuticas e reduzir a necessidade de modelos animais. Apresentamos aqui um novo protocolo de modelo de cultura de órgãos tridimensional imitando o microambiente proinflammatório e catabólico, que está presente durante o IDD.
Inicialmente, foram dissecadas, limpas e cultivadas no meio de cultura de tecido. O carregamento fisiológico ou patológico dinâmico foi aplicado em um bioreator personalizado durante 2 horas por dia. Os IVDs foram atribuídos a um grupo controle (alto meio de glicose, carregamento fisiológico, injeção salina tamponada com fosfato) e um grupo patológico (baixo meio de glicose, carregamento patológico, injeção fator de necrose tumoral-alfa) por quatro dias. Foi realizada a análise da expressão genética das células pulposus do núcleo coletado dos IVDs e do ensaio imunosorbente ligado à enzima dos meios de cultura de órgãos condicionados.
Nossos dados revelaram maior expressão de marcadores inflamatórios e alturas de disco reduzidas após o carregamento no grupo patológico em comparação com o grupo controle. Este protocolo é confiável para simular inflamação e degeneração do IVD e pode ser expandido ainda mais para ampliar seu escopo de aplicação.
Introdução
Dor lombar (LBP) pode afetar indivíduos de todas as idades e é uma das principais causas de incapacidade em todo o mundo1,2,3. O custo total associado ao LBP excede US$ 100 bilhões por ano4,5. A degeneração do disco intervertebral sintomático (IVD), condição caracterizada pela inflamação e degradação tecidual, é uma das principais causas da LBP6,7. Especificamente, o IDD é caracterizado por uma gradual evolução da matriz extracelular do IVD (ECM), induzida e desencadeada por múltiplos fatores que levam a uma patologia acelerada, distúrbios neurológicos e eventualmente incapacidade. Além disso, o IDD está associado à liberação de citocinas proinflamatórias, biomecânica alterada da coluna vertebral, angiogênese e crescimento nervoso, o que aumenta a sensação de dor, causando totalmente LBP crônica (descoltidão ativa)6,8. Até o momento, as opções de tratamento incluem discectomia e posterior fusão das vértebras adjacentes, implantação de prótese intravenosa ou abordagens não cirúrgicas, como anti-inflamatórios não esteroides, opioides e relaxantes musculares para pacientes com IDD9. Ambas as opções terapêuticas padrão atuais, cirúrgicas e não cirúrgicas, são apenas parcialmente eficazes e não conseguem resolver o problema biológico subjacente9,10. A doença do disco degenerativo em estágio inicial é caracterizada por uma resposta inicial do tecido inflamatório, especialmente um aumento na expressão fator de necrose tumoral-alfa (TNF-alfa)11. Essas alterações iniciais do disco ocorrem principalmente no nível celular sem interromper a arquitetura do disco e poderiam ser anteriormente imitadas por deficiência nutricional sob condições pró-inflamatórias12. Portanto, a simulação precisa da situação in vivo para investigar esses mecanismos de degeneração e encontrar alvos terapêuticos adequados é crucial. Além disso, para essas simulações de propriedades moleculares, o ambiente de carregamento mecânico dos discos desempenha um papel fundamental nas alterações patológicas e fisiológicas do IVD. Consequentemente, a combinação dessas abordagens nos traria um passo à frente para imitar o complexo microambiente de IVDs in vivo. Atualmente, não há estudos considerando o aspecto do carregamento dinâmico, juntamente com o cenário pró-inflamatório e nutricional com o melhor de nosso conhecimento.
Embora grandes modelos animais permitam a investigação de potenciais interações in vivo relevantes, eles são caros e trabalham intensivos. Além disso, como o uso de modelos animais em pesquisas tem sido uma questão de controvérsia, a redução do número de animais necessários para responder a importantes questões de pesquisa é de grande interesse. Finalmente, atualmente não existe um modelo animal ideal para imitar o IDD na pesquisa ivd13,14. Portanto, é necessário estabelecer uma substituição econômica e confiável, como um modelo de cultura de órgãos para simular IDD e processos inflamatórios e degenerativos associados. Recentemente, a aplicação do presente protocolo sobre o estabelecimento de um modelo de cultura proinflamatória e degenerativa de órgãos para simular a doença do disco intervertebral em estágio inicial permitiu investigar o efeito de drogas anti-inflamatórias na cultura de órgãos IDD15.
Aqui, descrevemos como obter discos interverteberais bovinos e induzir o estado de IDD em estágio inicial através de um microambiente catabólico e proinflamatório causado pela injeção intradiscal direta de fator de necrose tumoral-alfa (TNF-α) e carregamento degenerativo em um bioreator sob baixas condições nutritivas médias. A Figura 1 ilustra o modelo experimental e mostra o bioreator usado para simular condições de carga degenerativa e fisiológica.
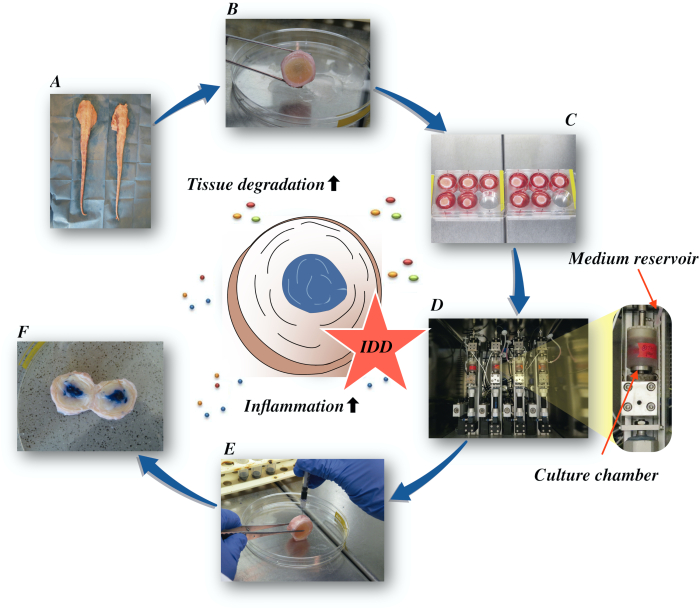
Figura 1: Ilustração da configuração experimental. A: cauda bovina; B: discos intervertebrales bovinos dissecados; C: transferência do disco para um bem-prato com meio de cultura; D: carregar a simulação em um bioreator; E: técnica de injeção intradiscal; F: IVD após injeção de corante azul PBS/trypan para revelar a distribuição. IDD: degeneração do disco intervertebral. Clique aqui para ver uma versão maior desta figura.
Protocolo
Os experimentos foram realizados utilizando-se caudas bovinas obtidas de matadouros locais. Os materiais biológicos utilizados no presente estudo são retirados da cadeia alimentar e não requerem aprovação ética no direito suíço e europeu.
1. Dissecção do disco intervertebral bovino
- Enxágüe toda a cauda completamente com água da torneira para remover sujeira e cabelo na superfície.
NOTA: Com extremidades distais intactas, um máximo de 9 IVDs (coccygeal 1-9) por cauda pode ser usado para os experimentos, dependendo do tamanho desejado dos IVDs. Considerando o diâmetro desejado entre 15-20 mm, utilizamos 12 caudas bovinas com 5 IVDs por cauda para os experimentos. - Mergulhe toda a cauda em uma caixa contendo 1% de solução betadina por 10 minutos. Seque brevemente a cauda com gaze estéril e coloque-a em uma cortina estéril.
NOTA: Durante a dissecção do disco, umidifice as caudas com a solução de Ringer molhada gaze para evitar a desidratação. Armazene as caudas (ou segmentos que sobram) envoltas em gaze molhada até que todo o procedimento de dissecção seja concluído. - Use um bisturi (nº 20) para remover o tecido mole o mais completamente possível da coluna caudal para facilitar a identificação dos IVDs. Remova os processos espinhosos e transversais das vértebras com alicate de remoção óssea.
NOTA: Selecione os IVDs com o diâmetro desejado. Foram utilizados ivDs com faixa de diâmetro de 15-20 mm no presente estudo. - Corte transversalmente com alicates ósseos no meio de cada corpo vertebral para obter segmentos individuais de movimento. Coloque segmentos de movimento em uma placa de Petri com gaze molhada com a solução de Ringer.
- Localize o IVD e a vértebra por palpação e movendo os segmentos de movimento suavemente. Faça dois cortes paralelos com a serra de banda na placa de crescimento dos IVDs, um em cada lado do IVD. Identifique a localização da placa de crescimento tocando e encontrando o local convexo da parte endplata óssea (dura) adjacente ao disco (macio) com uma distância de segurança de aproximadamente 0,5-1 mm do IVD em direção à vértebra. Certifique-se de que a lâmina da serra da banda seja resfriada com a solução de Ringer enquanto corta as vértebras.
- Transfira os IVDs em uma placa de Petri limpa com gaze limpa molhada com a solução de Ringer.
NOTA: A gaze deve ser umedecido e não muito molhado para evitar o inchaço dos IVDs, - Use a lâmina do bisturi para raspar o corpo vertebral (osso vermelho/rosa), placa de crescimento (cartilagem branca), deixar a placa de extremidade intacta (amarelo-rosa). Torne as duas superfícies planas e paralelas para o procedimento de carregamento. Transfira ivDs raspados para uma placa de Petri fresca com gaze molhada com a solução de Ringer.
NOTA: Use uma luva de corrente para proteger a mão enquanto segura o IVD e raspa. - Meça a altura e o diâmetro do disco com uma pinça. Limpe os coágulos sanguíneos no osso das vértebras com a solução de Ringer usando um sistema de lavagem de jatos.
- Transfira os IVDs para tubos plásticos de 50 mL, um IVD por tubo. Adicione 25 mL de Salina Tamponada de Fosfato (PBS) + 10% penicilina/estreptomicina (P/S) por IVD e deixe-a tremendo por 15 minutos em um agitador orbital à temperatura ambiente.
- Aspire o supernaspe e adicione 10 mL de PBS + 1% P/S por IVD por 2 min para enxaguar os IVDs.
2. Cultura e carregamento de IVD
- Transfira os discos para as câmaras ivd e adicione o meio de cultura IVD (Dulbecco's Modified Eagle Medium (DMEM, 4,5 g/L de alta glicose DMEM para o grupo fisiológico e 2 g/L de baixa glicose DMEM para o grupo patológico) + 1% P/S + 2% soro fetal de bezerro + 1% ITS (contém 5 μg/mL de insulina, 6 μg/mL transferrina, e 5 ng/mL ácido selenious) + 50 μg/mL ascorbate-2-fosfato + 1% aminoácido não essencial + 50 μg/mL agente antimicrobiano para células primárias) e coloque em uma incubadora a 37 °C, 85% de umidade e 5% CO2.
- Cultura os discos por 4 dias dentro de um sistema bioreator de acordo com os grupos experimentais16. No grupo patológico, mantenha as condições de carregamento degenerativas em 0,32-0,5 MPa, 5 Hz por 2 h/dia. No grupo de controle fisiológico, utilize um protocolo de carregamento de 0,02-0,2 MPa, 0,2 Hz para 2 h/dia.
NOTA: Posicione os IVDs em câmaras contendo 5 mL de meio IVD durante os procedimentos de carregamento. O volume depende do tamanho das câmaras de carregamento do bioreator. Entre os procedimentos de carregamento, coloque os IVDs em placas de seis poços com 7 mL de meio de cultura IVD para recuperação de inchaço livre. - Para analisar as alterações na altura do disco durante o período experimental, meça a altura do disco com uma pinça após a dissecção ivd (linha de base) e, em seguida, diariamente após o período de inchaço livre e após o carregamento dinâmico para a duração experimental.
3. Injeção de fator de necrose tumoral intradiscal (TNF-α) injeção
- Logo após o primeiro ciclo dinâmico de carregamento no primeiro dia, coloque os IVDs em uma placa de Petri em uma posição vertical e estabilize os IVDs com uma pinça.
- Injete tnf-α recombinante (100 ng em 70 μL de PBS por IVD) com uma agulha de insulina de 30 bitolas no tecido de pulposo do núcleo do grupo patológico17. Injete lentamente a uma velocidade de aproximadamente 70 μL em 1 min.
- Após a injeção, puxe a seringa no meio do caminho para trás dentro do IVD e puxe o êmbolo da seringa para criar um vácuo que impeça a solução injetada de vazar para trás, antes de remover a agulha e a seringa completamente do IVD.
NOTA: Realize um experimento piloto injetando PBS contendo corante azul trypan para avaliar a distribuição da solução injetada após o carregamento e a cultura durante a noite.
4. Expressão genética
- Colher os IVDs no dia 4. Colete o tecido de pulposo (NP) do núcleo (parte gelly no meio do IVD) com um soco de biópsia. Colete o annulus fibrosus externo (AF) com uma lâmina de bisturi (No.20).
NOTA: Para a referência da linha de base no dia 0, colete os tecidos imediatamente após a dissecação para extração de RNA. - Use a quantidade de tecido NP ou AF necessário para análise de expressão genética, dependendo do design experimental.
NOTA: Para os atuais experimentos, foram utilizados aproximadamente 150 mg de tecido. A razão da solução de isolamento de RNA para a massa tecidual deve ser de pelo menos 2 mL por tecido de 100-150 mgs para extração eficiente. - Digerir o tecido NP ou AF com a solução de digestão (0,2% de pronase no DMEM, filtro esterilizado) e incubar por 1h a 37 °C com agitação magnética18.
- Congele as amostras de tecido usando nitrogênio líquido e pulverize para um pó fino. Divida o pó de tecido pulverizado igualmente em dois tubos de 2 mL cada contendo 1 mL de tiocianato de guanidina e fenol em uma solução monofáfasa (solução de isolamento de RNA).
- Realize a homogeneização em tubos de 2 mL contendo a solução de isolamento de RNA e o pó de tecido pulverizado. Homogeneize o pó de tecido 5x com uma bola de aço inoxidável de 8 mm e um lyzer tecidual a 30 Hz por 3 min. Centrifugar a 12.000 x g,4 °C por 10 min e transferir o supernasal para um tubo fresco. Os supernantes podem ser armazenados a -80 °C por pelo menos um mês.
- Adicione 0,1 mL de 1-bromo-3-cloropropano (BCP) por 1 mL de solução de isolamento de RNA e agite vigorosamente por 15 s. Armazene a mistura resultante à temperatura ambiente em um agitador orbital por 15 minutos e centrífuga a 12.000 x g por 15 min a 4 °C.
NOTA: O RNA permanece exclusivamente na fase aquosa superior. - Transfira a fase aquosa para um tubo fresco e precipitar o RNA com 0,25 mL de isopropanol e 0,25 mL de solução de precipitação de sal elevado por 1 mL de solução de isolamento de RNA usada para homogeneização inicial. Armazene as amostras em temperatura ambiente por 15 minutos em um agitador orbital e centrífuga a 12.000 x g por 8 min a 4 °C.
NOTA: Alternativamente, use o método de extração de RNA baseado em coluna que geralmente leva a maior pureza do RNA, mas menor rendimento de RNA. - Remova o supernasce e lave a pelota de RNA com 1 mL de 75% de etanol por 1 mL de solução de isolamento de RNA usada para homogeneização inicial. Centrifugar a 7.500 x g por 5 min a 4 °C.
- Remova a lavagem do etanol e a pelota de RNA seca por 3-5 min. Dissolva o RNA em 20 μL de água tratada de dietilpiroto (DEPC) passando a solução algumas vezes através de uma ponta de pipeta e incubando por 10-15 min a 55-60 °C.
- Meça a absorvância em 230 nm, 260 nm e 280 nm (A230, A260 e A280, respectivamente). A260 de 1.0 corresponde a 40 μg/mL RNA. Espera-se uma razão A260/A280 de 1,6-1,9, enquanto a contaminação resulta em uma razão A260/A280 de <1,6.
- Prepare uma mistura de reação transcriptase reversa (RT) para um volume de reação de 20 μL. A mistura contém mistura de enzima RT, inibidor de ribonuclease, proteína auxiliar, primers, dNTPs, MgCl2, água livre de RNase e 0,4 μg de amostra de RNA.
- Centrifufique brevemente os tubos RT para misturar todos os componentes na parte inferior do tubo.
- Coloque as amostras no instrumento termocicr. Selecione o programa apropriado para o RT. Execute o RT por 10 min a 25 °C, seguido da etapa de transcrição reversa para 120 min a 42 °C e inativação da transcriptase reversa por 5 min a 85 °C, esfriando-a até 4 °C no final.
- Diluir o cDNA resultante com tris (hidroximetila)aminometano (Tris)-ethylenodiaminetetracetic (EDTA) (TE) tampão (10 mM Tris com 1 mM EDTA) para uma concentração final de 0,4 μg RNA usado para RT por solução de 100 μL cDNA. Armazene as amostras de cDNA a -20 °C.
- Realize a reação em cadeia de polimerase em tempo real (PCR) usando um volume de reação de 10 μL. O volume de reação contém a mistura mestre (contendo dna polimerase, uracil-DNA glycosylase, dNTPs com dUTP, referência passiva e componentes tampão otimizados), primer dianteiro 45 μM, primer reverso 45 μM, sonda 12,5 μM (contendo um corante repórter ligado à extremidade de 5' da sonda, um pequeno aglutinante de ranhura na extremidade 3' da sonda e um quencher não fluorescente no final da sonda de 3') , 2 μL cDNA e água tratada com DEPC.
- Execute um controle endógeno (RPLP0) para quantificação relativa com o método 2-ΔΔCT 19.
- Adicione amostras em duplicatas e execute um controle sem modelo adicionando te-buffer em vez de cDNA. Execute PCR em condições padrão (2 min a 50 °C, 10 min a 95 °C, 40 ciclos de 15 s a 95 °C e 1 min a 60 °C).
- Realize a quantificação relativa dos alvos mRNA seguindo o método de tomografia comparativa. A quantidade de mRNA normalizada para a amostra de linha de base é calculada como 2-ΔΔCT, enquanto ΔΔCT é a diferença entre o ΔCT (controle endógeno do alvo ct- CT) da amostra e ΔCT (alvo ct e controle endógeno ct) da amostra de linha de base.
5. Quantificação do teor proteico no meio IVD
- Colete o meio condicionado pelas amostras de IVD para medir o teor de proteína no meio. Realizar ensaio imunosorbente ligado à enzima (ELISA) de acordo com o protocolo da proteína alvo.
- O bovino interleukine-8 (IL-8) é quantificado pelo kit ANTI-bovino IL8 ELISA de acordo com a instrução do fabricante.
Resultados
A carga degenerativa em meio de baixa glicose combinada com a injeção de TNF-α causou um aumento significativo da expressão genética dos marcadores proinflamatórios interleucina 6 (IL-6) e interleucina 8 (IL-8) em comparação com o grupo de controle fisiológico em células NP após 4 dias de cultura(Figura 2). Em contraste, não observamos alterações significativas para os genes proinflamatórios interleucina 1β (IL-1β) e TNF-α em células NP (dados não mostrados). Além disso,...
Discussão
Nós fornecemos um protocolo detalhado para simular IVDs degenerativos e inflamatórios. Este protocolo pode ser aplicado para exames detalhados de vias inflamatórias que levam aos efeitos destrutivos no disco. Além disso, o protocolo pode ajudar a determinar alvos terapêuticos promissores envolvidos na progressão da doença.
Recentemente, mostramos que o α de α de recombinação humana poderia induzir inflamação nas células21bovinas e humanas, o que está de a...
Divulgações
Os autores não têm nada a revelar.
Agradecimentos
Este trabalho foi apoiado pela AO Foundation e pela AOSpine International. Babak Saravi recebeu apoio da Fundação Alemã da Coluna e da Fundação Alemã de Osteoartrite. Gernot Lang foi apoiado pelo Programa Berta-Ottenstein para Cientistas Clínicos Avançados, Faculdade de Medicina da Universidade de Freiburg, Alemanha.
Materiais
| Name | Company | Catalog Number | Comments |
| 1-Bromo-3-chloropropane(BCP) | Sigma-Aldrich, St. Louis, USA | B9673 | |
| Ascorbate-2-phosphate | Sigma-Aldrich, St. Louis, USA | A8960 | |
| Band saw | Exakt Apparatebau, Norderstedt, Germany | model 30/833 | |
| Betadine | Munndipharma, Frankfurt, Germany | ||
| Bovine IL-8 Do.it-Yourself ELISA | Kingfisher Biotech, St. Paul, USA | DIY1028B-003 | |
| Corning ITS Premix | Corning Inc., New York, USA | 354350 | |
| DMEM high glucose | Gibco by life technologies, Carlsbad, USA | 10741574 | |
| DMEM low glucose | Gibco by life technologies, Carlsbad, USA | 11564446 | |
| Ethanol for molecular biology | Sigma-Aldrich, St. Louis, USA | 09-0851 | |
| Fetal Bovine Serum (FBS) | Gibco by life technologies, Carlsbad, USA | A4766801 | |
| Non-essential amino acid solution | Gibco by life technologies, Carlsbad, USA | 11140050 | |
| Penicillin/Streptomycin(P/S) | gibco by life technologies, Carlsbad, USA | 11548876 | |
| Phosphate Buffer Solution, tablet | Sigma-Aldrich, St. Louis, USA | P4417 | |
| Pronase | Sigma-Aldrich, St. Louis, USA | 10165921001 | |
| Primocin | InvivoGen, Sandiego, USA | ant-pm-05 | |
| Pulsavac Jet Lavage System | Zimmer, IN,USA | ||
| TissueLyser II | Quiagen, Venlo, Netherlands | 85300 | |
| Streptavidinn-HRP | Kingfisher Biotech, St. Paul, USA | AR0068-001 | |
| Superscript VILO | Invitrogen by life Technologies, Carlsbad, USA | 10704274 | |
| cDNA Synthesis Kit | Applied Biosystems by life technologies | 10400745 | |
| TaqMan Universal Master Mix | Applied Biosystems by life technologies | ||
| TNF-alpha, recombinant human protein | R&D systems, Minnesota, USA | 210-TA-005 | |
| TRI Reagent | Molecular Research Center, Cincinnati, USA | TR 118 | |
| Tris-EDTA buffer solution | sigma-Aldrich, St. Louis, USA | 93283 | |
| Gene bIL-6 | Applied Biosystems by life technologies | Custom made probes | Primer fw (5′–3′) TTC CAA AAA TGG AGG AAA AGG A Primer rev (5′–3′) TCC AGA AGA CCA GCA GTG GTT Probe (5′FAM/3′TAMRA) CTT CCA ATC TGG GTT CAA TCA GGC GATT |
| Gene bIL8 | Applied Biosystems by life technologies | Bt03211906_m1 | |
| Gene bTNF-alpha | Applied Biosystems by life technologies | Custom made probes | Primer fw (5′–3′) CCT CTT CTC AAG CCT CAA GTA ACA A Primer rev (5′–3′) GAG CTG CCC CGG AGA GTT Probe (5′FAM/3′TAMRA) ATG TCG GCT ACA ACG TGG GCT ACC G |
| GENE bIL1beta | Applied Biosystems by life technologies | Custom made probes | Primer fw (5′–3′) TTA CTA CAG TGA CGA GAA TGA GCT GTT Primer rev (5′–3′) GGT CCA GGT GTT GGA TGC A Probe (5′FAM/3′TAMRA) CTC TTC ATC TGT TTA GGG TCA TCA GCC TCA A |
| RPLP0 | Applied Biosystems by life technologies | Bt03218086_m1 |
Referências
- Vos, T., et al. Global, regional, and national incidence, prevalence, and years lived with disability for 328 diseases and injuries for 195 countries, 1990-2016: a systematic analysis for the Global Burden of Disease Study 2016. The Lancet. 390 (10100), 1211-1259 (2017).
- Hoy, D., et al. Measuring the global burden of low back pain. Best Practice & Research Clinical Rheumatology. 24 (2), 155-165 (2010).
- Thiese, M. S., et al. Prevalence of low back pain by anatomic location and intensity in an occupational population. BMC Musculoskeletal Disorders. 15 (1), 283 (2014).
- Katz, J. N. Lumbar Disc Disorders and Low-Back Pain: Socioeconomic Factors and Consequences. The Journal of Bone and Joint Surgery (American). 88, 21 (2006).
- Vlaeyen, J. W. S., et al. Low back pain. Nature Reviews Disease Primers. 4 (1), 52 (2018).
- Khan, A. N., et al. Inflammatory biomarkers of low back pain and disc degeneration: a review: Biomarkers of disc degeneration and back pain. Annals of the New York Academy of Sciences. 1410 (1), 68-84 (2017).
- Kim, H. S., Wu, P. H., Jang, I. T. Lumbar Degenerative Disease Part 1: Anatomy and Pathophysiology of Intervertebral Discogenic Pain and Radiofrequency Ablation of Basivertebral and Sinuvertebral Nerve Treatment for Chronic Discogenic Back Pain: A Prospective Case Series and Review of Literature. International Journal of Molecular Sciences. 21 (4), 1483 (2020).
- Adams, M. A., Roughley, P. J. What is Intervertebral Disc Degeneration, and What Causes It. Spine. 31 (18), 2151-2161 (2006).
- Wu, P. H., Kim, H. S., Jang, I. T. Intervertebral Disc Diseases Part 2: A Review of the Current Diagnostic and Treatment Strategies for Intervertebral Disc Disease. International Journal of Molecular Sciences. 21 (6), 2135 (2020).
- Lurie, J. D., et al. Surgical Versus Nonoperative Treatment for Lumbar Disc Herniation: Eight-Year Results for the Spine Patient Outcomes Research Trial. Spine. 39 (1), 3-16 (2014).
- Risbud, M. V., Shapiro, I. M. Role of cytokines in intervertebral disc degeneration: pain and disc content. Nature Reviews Rheumatology. 10 (1), 44-56 (2014).
- Ponnappan, R. K., et al. An organ culture system to model early degenerative changes of the intervertebral disc. Arthritis Research & Therapy. 13 (5), 171 (2011).
- O'Connell, G. D., Vresilovic, E. J., Elliott, D. M. Comparison of Animals Used in Disc Research to Human Lumbar Disc Geometry. Spine. 32 (3), 328-333 (2007).
- Stannard, J. T., et al. Development of a whole organ culture model for intervertebral disc disease. Journal of Orthopaedic Translation. 5, 1-8 (2016).
- Li, Z., et al. Preclinical ex-vivo Testing of Anti-inflammatory Drugs in a Bovine Intervertebral Degenerative Disc Model. Frontiers in Bioengineering and Biotechnology. 8, 583 (2020).
- Li, Z., et al. Development of an ex vivo cavity model to study repair strategies in loaded intervertebral discs. European Spine Journal. 25 (9), 2898-2908 (2016).
- Kazezian, Z., Li, Z., Alini, M., Grad, S., Pandit, A. Injectable hyaluronic acid down-regulates interferon signaling molecules, IGFBP3 and IFIT3 in the bovine intervertebral disc. Acta Biomaterialia. 52, 118-129 (2017).
- Caprez, S., Menzel, U., Li, Z., Grad, S., Alini, M., Peroglio, M. Isolation of high-quality RNA from intervertebral disc tissue via pronase predigestion and tissue pulverization. JOR Spine. 1 (2), 1017 (2018).
- Lopa, S., Ceriani, C., Cecchinato, R., Zagra, L., Moretti, M., Colombini, A. Stability of housekeeping genes in human intervertebral disc, endplate and articular cartilage cells in multiple conditions for reliable transcriptional analysis. European Cells & Materials. 31, 395-406 (2016).
- Lang, G., et al. An intervertebral disc whole organ culture system to investigate proinflammatory and degenerative disc disease condition. Journal of Tissue Engineering and Regenerative Medicine. 12 (4), 2051-2061 (2018).
- Du, J., et al. Proinflammatory intervertebral disc cell and organ culture models induced by tumor necrosis factor alpha. JOR Spine. 3, 1104 (2020).
- Purmessur, D., Walter, B. A., Roughley, P. J., Laudier, D. M., Hecht, A. C., Iatridis, J. A role for TNFα in intervertebral disc degeneration: A non-recoverable catabolic shift. Biochemical and Biophysical Research Communications. 433 (1), 151-156 (2013).
- Walter, B. A., Likhitpanichkul, M., Illien-Junger, S., Roughley, P. J., Hecht, A. C., Iatridis, J. C. TNFα Transport Induced by Dynamic Loading Alters Biomechanics of Intact Intervertebral Discs. PLOS One. 10 (3), 0118358 (2015).
- Gullbrand, S. E., et al. A large animal model that recapitulates the spectrum of human intervertebral disc degeneration. Osteoarthritis and Cartilage. 25 (1), 146-156 (2017).
- Willems, N., et al. Safety of intradiscal injection and biocompatibility of polyester amide microspheres in a canine model predisposed to intervertebral disc degeneration: intradiscal application of pea microspheres. Journal of Biomedical Materials Research Part B: Applied Biomaterials. 105 (4), 707-714 (2017).
- Michalek, A. J., Buckley, M. R., Bonassar, L. J., Cohen, I., Iatridis, J. C. The effects of needle puncture injury on microscale shear strain in the intervertebral disc annulus fibrosus. The Spine Journal. 10 (12), 1098-1105 (2010).
- Illien-Jünger, S., et al. The combined effects of limited nutrition and high-frequency loading on intervertebral discs with endplates. Spine. 35 (19), 1744-1752 (2010).
- Gantenbein, B., et al. Organ culture bioreactors--platforms to study human intervertebral disc degeneration and regenerative therapy. Current Stem Cell Research & Therapy. 10 (4), 339-352 (2015).
- Boubriak, O. A., Watson, N., Sivan, S. S., Stubbens, N., Urban, J. P. G. Factors regulating viable cell density in the intervertebral disc: blood supply in relation to disc height. Journal of Anatomy. 222 (3), 341-348 (2013).
- Maroudas, A., Stockwell, R. A., Nachemson, A., Urban, J. Factors involved in the nutrition of the human lumbar intervertebral disc: cellularity and diffusion of glucose in vitro. Journal of Anatomy. 120, 113-130 (1975).
- Beckstein, J. C., Sen, S., Schaer, T. P., Vresilovic, E. J., Elliott, D. M. Comparison of Animal Discs Used in Disc Research to Human Lumbar Disc: Axial Compression Mechanics and Glycosaminoglycan Content. Spine. 33 (6), 166-173 (2008).
- Walter, B. A., Illien-Jünger, S., Nasser, P. R., Hecht, A. C., Iatridis, J. C. Development and validation of a bioreactor system for dynamic loading and mechanical characterization of whole human intervertebral discs in organ culture. Journal of Biomechanics. 47 (9), 2095-2101 (2014).
- Rajan, N. E., et al. Toll-Like Receptor 4 (TLR4) Expression and Stimulation in a Model of Intervertebral Disc Inflammation and Degeneration. Spine. 38 (16), 1343-1351 (2013).
- vanden Akker, G. G., Rorije, A. J., Davidson, E. N. B., vander Kraan, P. M. Phenotypic marker genes distinguish inner and outer annulus fibrosus from nucleus pulposus tissue in the bovine intervertebral disc. Osteoarthritis and Cartilage. 25, 402 (2017).
- Du, J., et al. Functional cell phenotype induction with TGF-β1 and collagen-polyurethane scaffold for annulus fibrosus rupture repair. European Cells & Materials. 39, 1-17 (2020).
- Risbud, M. V., et al. Defining the phenotype of young healthy nucleus pulposus cells: recommendations of the Spine Research Interest Group at the 2014 annual ORS meeting. Journal of Orthopaedic Research: Official Publication of the Orthopaedic Research Society. 33 (3), 283-293 (2015).
Reimpressões e Permissões
Solicitar permissão para reutilizar o texto ou figuras deste artigo JoVE
Solicitar PermissãoThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Todos os direitos reservados