É necessária uma assinatura da JoVE para visualizar este conteúdo. Faça login ou comece sua avaliação gratuita.
Method Article
Formulando e caracterizando nanopartículas lipídicas para entrega de genes usando uma plataforma de mistura microfluida
Neste Artigo
Resumo
As nanopartículas lipídicas são desenvolvidas usando uma abordagem de plataforma de mistura microfluida para encapsulamento de mRNA e DNA.
Resumo
Os portadores de medicamentos à base de lipídio têm sido usados para sistemas de entrega clinicamente e comercialmente disponíveis devido ao seu pequeno tamanho, biocompatibilidade e alta eficiência de encapsulamento. O uso de nanopartículas lipídicas (LNPs) para encapsular ácidos nucleicos é vantajoso para proteger o RNA ou DNA da degradação, ao mesmo tempo em que promove a absorção celular. Os LNPs geralmente contêm múltiplos componentes lipídesis, incluindo um lipídio ionizável, lipídio auxiliar, colesterol e lipídio conjugado de polietileno glicol (PEG). Os LNPs podem facilmente encapsular ácidos nucleicos devido à presença lipídica ionizável, que em pH baixo é cationic e permite a complexação com RNA ou DNA carregados negativamente. Aqui os LNPs são formados por encapsular o RNA mensageiro (mRNA) ou DNA plasmídeo (pDNA) usando a mistura rápida dos componentes lipídicos em uma fase orgânica e o componente ácido nucleico em uma fase aquosa. Esta mistura é realizada usando uma plataforma de mistura microfluida precisa, permitindo a automontagem de nanopartículas enquanto mantém o fluxo laminar. O tamanho hidrodinâmico e a polidispersidade são medidos usando dispersão dinâmica de luz (DLS). A carga de superfície eficaz no LNP é determinada medindo o potencial zeta. A eficiência de encapsulamento é caracterizada usando um corante fluorescente para quantificar o ácido nucleico preso. Os resultados representativos demonstram a reprodutibilidade deste método e a influência que diferentes formulações e parâmetros de processo têm sobre as LNPs desenvolvidas.
Introdução
Os portadores de drogas são usados para proteger e fornecer um terapêutico com propriedades favoráveis típicas, incluindo baixa citotoxicidade, aumento da biodisponibilidade e melhor estabilidade1,2,3. Nanopartículas poliméricas, micelas e partículas à base de lipídios foram previamente exploradas para encapsulamento e entrega de ácido nucleico4,5,6,7. Lipídios têm sido usados em diferentes tipos de sistemas de nanocarrier, incluindo lipossomos e nanopartículas lipídicas, pois são biocompatíveis com alta estabilidade8. Os LNPs podem facilmente encapsular ácidos nucleicos para a entrega de genes9,10. Protegem o ácido nucleico da degradação por proteases soro durante a circulação sistêmica11 e podem melhorar a entrega para locais específicos, pois a topografia superficial e as propriedades físicas dos LNPs influenciam sua biodistribução12. Os LNPs também melhoram a penetração de tecidos e a captação celular9. Estudos anteriores demonstraram o sucesso do encapsulamento do siRNA dentro de um LNP13, incluindo o primeiro LNP comercialmente disponível contendo siRNA terapêutico para o tratamento de poneuropatia de tratamento de amilidação mediada por transtoretinahereditária 14 tratamento que foi aprovado pela Food and Drug Administration (FDA) dos Estados Unidos e pela Agência Europeia de Medicamentos em 2018. Mais recentemente, lNPs estão sendo estudados para a entrega de maiores moieties de ácido nucleico, ou seja, mRNA e DNA9. Em 2018, havia ~ 22 sistemas de entrega de ácido nucleico à base de lipídios submetidos a testes clínicos14. Além disso, o mRNA contendo LNPs são atualmente os principais candidatos e foram empregados para uma vacina COVID-1915,16. O potencial sucesso dessas terapias genéticas não virais requer a formação de partículas pequenas (~100 nm), estáveis e uniformes com alto encapsulamento do ácido nucleico.
O uso de um lipídio ionizável como componente principal na formulação do LNP tem mostrado vantagens para a complexidade, encapsulamento e effciciency de entrega14. Lipídios ionizáveis normalmente têm uma constante de dissociação de ácido (pKa) < 7; por exemplo, dilinoleylmeethyl-4-dimethylaminobutyrate (D-Lin-MC3-DMA), o lipídio ionizável usado na formulação lnp aprovada pela FDA, tem um pKa de 6.4417. No pH baixo, os grupos de amina no lipídio ionizável tornam-se protoados e carregados positivamente, permitindo a montagem com grupos de fosfato carregados negativamente no mRNA e DNA. A razão de amina, "N", grupos para fosfato, "P", grupos é usado para otimizar a montagem. A razão N/P depende dos lipídios e ácidos nucleicos utilizados, que varia dependendo da formulação18. Após a formação, o pH pode ser ajustado a um pH neutro ou fisiológico para permitir a administração terapêutica. A esses valores de pH, o lipídio ionizável também é desprotoado que dá carga de superfície neutra ao LNP.
O lipídio ionizável também auxilia na fuga endossomal19,20. Os LNPs são submetidos à endocitose durante a captação celular e devem ser liberados do endosomhei para entregar a carga de mRNA no citoplasma celular ou carga de DNA para o núcleo21. Dentro do endosomho é tipicamente um ambiente mais ácido do que o meio extracelular, o que torna o lipídio ionizável carregado positivamente22,23. O lipídio ionizável carregado positivamente pode interagir com cargas negativas na membrana lipídica endossomal, o que pode causar desestabilização do endóssomo permitindo a liberação do LNP e do ácido nucleico. Diferentes lipídios ionizáveis estão sendo estudados para melhorar a eficácia tanto da distribuição do LNP, quanto para a fuga endossomal14.
Outros componentes típicos de um LNP incluem lipídios auxiliares, como um lipídio fosfatidylcholina (PC) ou fosfoetanolamina (PE). 1,2-Dioleoyl-sn -gliceo-3-fosfoethanolamina (DOPE), 1,2-distearoyl-sn-glycero-3-phos a foforolina (DSPC) e 1,2-dioleoyl-sn-gliceo-3-fosfocholina (DOPC) são lipídios auxiliares comumente usados24,25. A DOPE tem sido demonstrada para formar uma fase hexagonal ii invertida (HII) e melhorar a transfecção por fusão de membrana26,enquanto o DSPC foi pensado para estabilizar LNPs com sua geometria cilíndrica27. O colesterol também é incorporado na formulação, a fim de aumentar a rigidez da membrana, auxiliando posteriormente na estabilidade do LNP. Finalmente, o polietileno glicol conjugado lipídida (PEG) é incluído na formulação para fornecer a barreira estérica necessária para auxiliar na automontagem de partículas27. A PEG também melhora a estabilidade de armazenamento dos LNPs, impedindo a agregação. Além disso, o PEG é frequentemente usado como um componente furtivo e pode aumentar o tempo de circulação para os LNPs. No entanto, esse atributo também pode representar desafios para o recrutamento de LNPs para hepatócitos através de um mecanismo de segmentação endógeno impulsionado pela apolipoproteína E (ApoE)28. Assim, estudos têm investigado o comprimento da cadeia aciola para difusão de PEG do LNP, descobrindo que os comprimentos curtos (C8-14) se dissociam do LNP e são mais favoráveis ao recrutamento de ApoE em comparação com comprimentos de acicl mais longos28. Além disso, o grau de saturação da cauda lipídica a que o PEG é conjugado tem sido demonstrado para influenciar a distribuição tecidual de LNPs29. Recentemente, o Tween 20, que é um surfactante comumente usado em formulações de produtos biológicos de medicamentos e tem uma longa cauda lipídica insaturada, mostrou-se ter alta transfecção na drenagem de linfonodos em comparação com peg-DSPE, que transfetou em grande parte o músculo no local da injeção29. Este parâmetro pode ser otimizado para alcançar a biodistribução desejada do LNP.
Os métodos convencionais de formação de LNPs incluem o método de hidratação de filme fino e o método de injeção de etanol27. Embora estas sejam técnicas prontamente disponíveis, elas também são intensivas em mão-de-obra, podem resultar em baixa eficiência de encapsulamento e são desafiadoras para escalar27. Os avanços nas técnicas de mistura resultaram em métodos mais favoráveis à escala, ao mesmo tempo em que desenvolvem partículas mais uniformes27. Estes métodos incluem mistura de junção T, mistura de herringbone escalonado e foco hidrodinâmico microfluidic27. Cada método possui uma estrutura única, mas todos permitem a rápida mistura de uma fase aquosa contendo o ácido nucleico com uma fase orgânica contendo os componentes lipídicos, resultando em alto encapsulamento do ácido nucleico27. Neste protocolo é utilizada a mistura rápida e controlada através de um cartucho microfluido, que emprega o design de mistura de herringbone escalonado. Este protocolo descreve a preparação, montagem e caracterização do ácido nucleico contendo LNPs.
Protocolo
Um esquema do processo global é fornecido na Figura 1.
1. Preparação de tampões
NOTA: A filtragem estéril dos buffers é altamente sugerida aqui para remover quaisquer partículas que possam impactar o ácido nucleico e a qualidade do LNP.
- Salina Tamponada fosfato (PBS)
- Prepare 1x PBS usando 8 mM Na2HPO4, 2 mM KH2PO4, 137 mM NaCl e 2,7 mM KCl em água livre de nuclease e ajuste o pH para 7,4.
- Esterilize por filtragem a vácuo usando um filtro de tamanho de poro de 0,22 μm.
- Tampão de citrato
- Prepare o tampão citrato usando citrato de sódio de 5 mM, ácido cítrico de 5 mM e cloreto de sódio de 150 mM em água livre de nuclease e ajuste para pH 4,5.
- Esterilize por filtragem a vácuo usando um filtro de tamanho de poro de 0,22 μm.
NOTA: O tampão citrato só precisa ser preparado se o mRNA for o ácido nucleico que será encapsulado no LNP. Se o DNA for encapsulado pule 1.2 e prossiga para 1.3.
- Tampão de ácido malic
- Prepare o tampão de ácido málico usando ácido málico de 20 mM e cloreto de sódio de 30 mM em água livre de nuclease e ajuste ao pH 3.0.
- Esterilize por filtragem a vácuo usando um filtro de tamanho de poro de 0,22 μm.
NOTA: O tampão de ácido malic só precisa ser preparado se o DNA for o ácido nucleico que será encapsulado no LNP. Pule 1,3 se o mRNA for encapsulado. O tampão citrato é usado para encapsulamento de mRNA, pois o pH inferior de 3.0 com tampão de ácido málico pode levar a uma maior probabilidade de degradação do mRNA. O protocolo pode ser pausado aqui.
2. Preparação da mistura lipídica
- Se os lipídios de estoque estiverem em pó, solubilize em puro etanol à prova de 200.
- Calcule a mistura necessária de componentes lipídudos com base na razão molar desejada. Uma razão molar de 50:10:39:1 (lipídio ionizável:lipídio auxiliar:colesterol:PEG) será usado aqui como exemplo para uma concentração lipídica total de 10 mM. A Tabela 1 mostra as concentrações e volumes necessários para cada um desses componentes.
NOTA: Ao calcular o volume necessário para alcançar a concentração de mistura lipídica no etanol (EtOH) para o misturador microfluido, o volume total é contabilizado para garantir que a adição de EtOH não influencie as concentrações lipídicas. Por exemplo, um volume lipídetivo ionizável de 68,5 μL é calculado multiplicando-se a concentração de 5 mM no etanol por um volume total de mistura lipídica de 533 μL e, em seguida, dividindo-se pela concentração lipídica de 38,9 mM. - Adicione a quantidade apropriada de cada solução de caldo lipíduo a um frasco de vidro para permitir que os componentes se misturem com vórtices intermitentes. Adicione 200 etanol à prova para uma mistura total de 533 μL. Para o exemplo na Tabela 1,são 254 μL de etanol.
NOTA: Para uma única corrida para produzir 1 mL de LNPs, 342,5 μL de solução lipídica são necessários. Isso se deve a uma mistura 3:1 de ácido nucleico aquoso à solução lipídica orgânica com algum volume descartado antes e depois da coleta da amostra. Uma mistura de 533 μL é feita para compensar como excesso.
3. Preparação da solução de ácido nucleico
NOTA: A preparação e manuseio de soluções de ácido nucleico deve ser realizada em um ambiente estéril e livre de RNase sempre que possível. Trabalhe em um armário de biossegurança sempre que possível com o ácido nucleico.
- Calcule a razão N/P. A razão N/P é o número total de grupos lipídicos ionizáveis de amina (N) para o número total de grupos de fosfato nucleico carregados negativamente (P). A razão N/P é frequentemente um parâmetro que pode ser otimizado durante a formação do LNP. Siga os passos abaixo.
- Calcule o número de unidades N usando a fórmula abaixo:
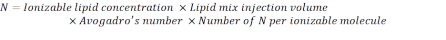
NOTA: A concentração lipídica ionizável(Tabela 1) é de 5 mM, o que equivale a 5 x 10-6 mol/mL. O volume de injeção lipídica necessário é de 0,3425 mL. Por exemplo, se o número de unidades N por molécula é 1, usando a equação acima, há 1,03 x 1018 N unidades na mistura lipídica. - Calcule as unidades P para a relação N/P desejada. Aqui um N/P = 36 é usado, por exemplo.
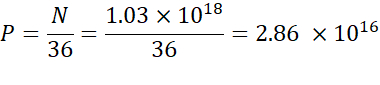
- Calcule o número de unidades N usando a fórmula abaixo:
- Calcule a concentração de ácido nucleico necessário para obter unidades de 2,86 x 1016 P utilizando a equação abaixo.
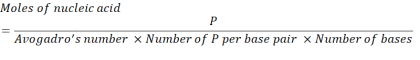
Onde, o número de unidades P por par base para mRNA é 1 e DNA é 2. Para um mRNA com 1.200 bases, a quantidade de mRNA necessária para um N/P = 36 é de 3,96 x 10-11 mols. - Calcule a concentração de massa de mRNA necessária para N/P = 36 usando a equação abaixo.
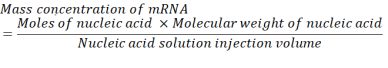
O peso molecular médio de uma unidade de monfosfato de ribonucleotídeo é de 322 g/mol30. Com mRNA de 1.200 bases, o peso molecular do mRNA é de 386.400 g/mol. O volume de injeção necessário da solução de ácido nucleico é de 1.028 mL. Assim, a concentração de mRNA necessária é de 1,488x10-5 g/mL, que é de 14,88 μg/mL. - Compor 1,5 mL de 14,88 μg/mL de mRNA no tampão citrato.
NOTA: Quando o DNA for encapsulado pelo ácido nucleico, use tampão de ácido málico para compor a solução de ácido nucleico.
4. Primindo os canais microfluidos
NOTA: Este protocolo é adaptado das diretrizes do fabricante do instrumento.
- Insira os parâmetros de escoramento no software de instrumento clicando nos campos apropriados(Tabela 2).
NOTA: Recomenda-se uma relação de fluxo de 3:1 e uma taxa de fluxo de 4-12 mL/min27,31 . Isso tem se mostrado ideal nos estudos aqui apresentados, bem como no fabricante. Isso pode ser variado se for de interesse para a aplicação. - Abra a tampa do instrumento e coloque um cartucho microfluido no bloco rotativo.
- Desenhe pelo menos 0,5 mL de etanol em uma seringa de 1 mL, garantindo que não haja bolhas ou aberturas de ar na ponta da seringa. Carregue esta seringa na entrada direita do cartucho.
- Encha uma seringa de 3 mL com 1,5 mL de tampão aquoso (citrato para RNA e ácido málico para DNA), garantindo que não haja bolhas de ar ou lacunas. Carregue esta seringa na entrada esquerda do cartucho.
- Insira dois tubos cônicos de 15 mL nos suportes do clipe para servir como recipientes de resíduos.
- Clique em Executar no software de instrumento para iniciar a mixagem, garantindo que os parâmetros estejam inseridos corretamente.
- Quando o instrumento parar de escorraçar, indicado pela luz azul inferior desligando, abra a tampa e descarte corretamente os tubos cônicos e seringas.
5. Formação do LNP
NOTA: Este protocolo é adaptado das diretrizes do fabricante do instrumento.
- Atualize o software com os parâmetros de formulação clicando nos campos apropriados(Tabela 2).
- Encha uma seringa de 1 mL com a mistura lipídica (preparada na etapa 2). Remova quaisquer lacunas de ar ou bolhas na ponta da seringa e insira a seringa no lado direito do cartucho.
- Desenhe a solução de ácido nucleico (preparada na etapa 3) em uma seringa de 3 mL, garantindo que não haja bolhas ou lacunas de ar na ponta da seringa. Insira a seringa na entrada esquerda do cartucho.
NOTA: Os volumes são fornecidos para fazer uma solução de 1 mL de LNPs. Este instrumento pode incorporar tamanhos de seringa de até 10 mL, e os volumes podem ser dimensionados de acordo sem influência no resultado. O volume máximo de LNPs que pode ser preparado em uma preparação é de 12 mL. - Rotule um tubo cônico sem RNase de 15 mL com o nome da amostra e insira no clipe do tubo esquerdo. Coloque um resíduo cônico de 15 mL no clipe do tubo direito.
- Feche a tampa do instrumento e clique em Executar,após confirmar a entrada correta dos parâmetros.
- Depois que o instrumento terminar de funcionar, descarte corretamente o recipiente de resíduos e o cartucho. Retenha o tubo cônico com a amostra do LNP.
- Diluir o LNP 5x com PBS para minimizar o etanol a <5% (v/v).
NOTA: É importante diluir os LNPs no PBS o mais rápido possível após a mistura microfluida para evitar a degradação. Sempre realize a diluição em um gabinete de biossegurança e continue trabalhando no gabinete de biossegurança durante as trocas de buffer.
6. Troca de buffer
NOTA: É fornecido protocolo para o uso de filtros ultra-centrífugas. Embora este método resulte em uma troca mais eficiente de buffers, a diálise pode ser substituída aqui.
- Pré-lave um filtro ultra-centrífugas (tamanho de poros de 100 kDa) com 2 mL de PBS por centrifugação a 1000 x g por 5 min. Esvazie o PBS do compartimento inferior.
NOTA: O PBS é escolhido para aumentar o pH para 7,4 ± 0,2, o que é fisiologicamente relevante e resultará na lipídio ionizável ter uma carga neutra. - Adicione LNPs diluídos ao compartimento superior do filtro ultracentrífugo pré-lavado e centrífuga a 1000 x g por 12 min.
- Descarte o fluxo do compartimento inferior. Execute mais duas lavagens adicionando 5 mL de PBS ao filtro ultra-centrífuga cada vez. Centrifugar nos mesmos parâmetros. Não há volume máximo que precisa ser mantido.
NOTA: Se um volume escalonado de LNPs foi preparado, aumente o volume de PBS para cada lavagem em conformidade. Por exemplo, se 2 mL de LNPs foram preparados em uma única execução, então 10 mL PBS por lavagem é sugerido. - Pipeta a solução LNP contra as paredes do filtro ultra-centrífuga algumas vezes para minimizar a perda do LNP. Remova a solução LNP do filtro ultra-centrífuga e armazene em um frasco livre de nuclease. Adicione PBS se necessário para obter um volume final da solução LNP de 1 mL.
- Filtre através de um filtro de seringa pré-molhado de 0,2 μm, se necessário.
NOTA: O protocolo pode ser pausado aqui.
7. Medir a eficiência do encapsulamento
- Prepare uma curva padrão fazendo diluições em série de 2 vezes da solução de ácido nucleico em funcionamento na PBS, começando com uma maior concentração de 500 ng/mL, e fazendo pelo menos cinco diluições. Use PBS como um espaço em branco.
- Prepare as diluições da amostra do LNP. Diluir amostras de LNP com PBS, para obter uma concentração teórica aproximada que fica ao redor do ponto médio da curva padrão (por exemplo, ~ 250 ng/mL de ácido nucleico estimado a partir da concentração inicial).
- Prepare uma solução do reagente de quantificação de RNA (para medições de mRNA) com TritonX-100 para interromper os LNPs e medir a quantidade total de ácido nucleico dentro e fora do LNP. Esta solução contém reagente 0,5% (v/v) RNA, 0,4% (v/v) TritonX-100 e 99,1% (v/v) PBS.
- Prepare uma solução do reagente sem TritonX-100 para medir a quantidade de ácido nucleico não encapsulado nos LNPs. Esta solução contém 0,5% (v/v) reagente de RNA e 99,5% (v/v) PBS.
NOTA: Se os LNPs encapsularem DNA duplo encalhado (dsDNA), como DNA plasmídeo, use o reagente dsDNA em 7.3 e 7.4 em vez disso, seguindo o mesmo procedimento. - Em uma placa capaz de fluorescência preta de 96 poços, carregue pelo menos quatro réplicas de cada uma das soluções padrão de ácido nucleico preparadas em 7.1 e 7.2.
- À metade das réplicas de padrões e amostras, adicione um volume igual do reagente contendo TritonX-100. Isso quantificará a quantidade total de ácido nucleico.
- Aos demais poços de normas e amostras, adicione um volume igual do reagente sem TritonX-100. Isso quantificará a quantidade de ácido nucleico não encapsulado dentro do LNP.
- Agite a placa por 5 minutos à temperatura ambiente para garantir a mistura completa de padrões e amostras com o reagente adicionado, tomando precauções para evitar a exposição à luz.
- Meça a fluorescência usando um leitor de microplaca, com um comprimento de onda de excitação de 480 nm e um comprimento de onda de emissão de 520 nm.
- Calcule a concentração de ácido nucleico fora do LNP usando a curva padrão feita com a adição do reagente sem TritonX-100. Multiplique pelo fator de diluição utilizado em 7,2.
- Calcule a concentração de ácido nucleico dentro e fora do LNP usando a curva padrão feita com a adição do reagente contendo TritonX-100. Multiplique pelo fator de diluição utilizado em 7,2.
- Calcule a concentração de ácido nucleico no interior subtraindo a concentração de ácido nucleico fora (calculado a partir da etapa 7.10) da concentração total de ácido nucleico dentro e fora (calculado a partir da etapa 7.11)
- Quantifique a eficiência de encapsulamento a partir da razão da concentração de ácido nucleico dentro do LNP (calculado a partir da etapa 7.12) e da concentração total de ácido nucleico (calculado a partir da etapa 7.11).
NOTA: O protocolo pode ser pausado aqui.
8. Ajustes de concentração
- Se necessário, ajuste a concentração de ácido nucleico dentro da solução LNP usando os resultados da eficiência de encapsulamento.
- Se for desejada uma solução menos concentrada, dilui a solução com PBS para alcançar a concentração desejada.
- Se uma solução mais concentrada for desejada, execute execuções adicionais de centrífugas usando um filtro ultra-centrífuga.
NOTA: O protocolo pode ser pausado aqui.
9. Medir o tamanho hidrodinâmico do LNP e a polidispersidade
- Diluir uma alíquota da solução LNP 40x com PBS para obter um volume final de 1 mL.
NOTA: Esta diluição pode ser alterada se necessário. Este valor de diluição é sugerido, pois utiliza um pequeno volume da solução de estoque LNP, ao mesmo tempo em que fornece resultados de qualidade. - Utilizando um cuvette semi-micro, meça o diâmetro hidrodinâmico e o índice de polidispersidade. Adicione a solução LNP no cuvette e insira no instrumento. Configure um procedimento operacional no software do instrumento para incluir o tipo de medição, detalhes da amostra (material, dispersante, temperatura e tipo celular) e instruções de medição (número de execuções). Clique em Iniciar quando estiver pronto para iniciar a aquisição da medição.
10. Medir o potencial de zeta do LNP
- Diluir uma alíquota da solução LNP 40x com água livre nuclease para obter um volume final de 1 mL.
NOTA: A água livre de nuclease é usada como solvente para medidas potenciais de zeta para minimizar a influência de tampões de sal elevados na condutividade. - Usando uma célula de zeta capilar dobrada, meça o potencial zeta.
- Adicione a solução LNP na cuvette até a linha de preenchimento. Insira-se no instrumento garantindo que os eletrodos estejam fazendo contato com o instrumento.
- Configure um procedimento operacional no software do instrumento para incluir o tipo de medição, detalhes da amostra (material, dispersante, temperatura e tipo celular) e instruções de medição (número de execuções). Clique em Iniciar quando estiver pronto para iniciar a aquisição da medição.
Resultados
Vários lotes de LNPs com a mesma formulação lipídica e razão N/P de 6 foram desenvolvidos em dias separados para demonstrar a reprodutibilidade da técnica. Lote 1 e 2 resultaram em distribuições de tamanho sobrepostas com polidispersidade semelhante(Figura 2A) Nenhuma diferença significativa foi observada no tamanho ou eficiência de encapsulamento entre os dois lotes diferentes(Figura 2B). A eficiência de encapsulament...
Discussão
A reprodutibilidade, a velocidade e a triagem de baixo volume são vantagens significativas do uso da mistura microfluida para formar LNPs em comparação com outros métodos existentes (por exemplo, hidratação de filme lipídico e injeção de etanol). Demonstramos a reprodutibilidade deste método sem impacto na eficiência de encapsulamento ou tamanho de partículas observada com diferentes lotes de LNP. Este é um critério essencial para que qualquer terapêutico, incluindo os LNPs, se torne clinicamente disponív...
Divulgações
Todos os autores são funcionários da Sanofi. Os autores declaram que não têm conflito de interesses ou interesses financeiros concorrentes.
Agradecimentos
Obrigado a Atul Saluja, Yatin Gokarn, Maria-Teresa Peracchia, Walter Schwenger e Philip Zakas por suas orientações e contribuições para o desenvolvimento do LNP.
Materiais
| Name | Company | Catalog Number | Comments |
| 1,2-dimyristoyl-rac-glycero-3-methoxypolyethylene glycol-2000 (C-14 PEG) | Avanti Polar Lipids | 880151P | |
| 10 µl Graduated Filter Tips (RNase-,DNase-, DNA-free) | USA Scientific | 1121-3810 | |
| 1000 µl Graduated Filter Tips (RNase-,DNase-, DNA-free) | USA Scientific | 1111-2831 | |
| 20 µl Beveled Filter Tips (RNase-,DNase-, DNA-free) | USA Scientific | 1120-1810 | |
| 200 µl Graudated Filter Tips (RNase-,DNase-, DNA-free) | USA Scientific | 1120-8810 | |
| 3β-Hydroxy-5-cholestene, 5-Cholesten-3β-ol (Cholesterol) | Sigma-Aldrich | C8667 | |
| BD Slip Tip Sterile Syringes (1 ml syringe) | Thermo Fisher Scientific | 14-823-434 | |
| BD Slip Tip Sterile Syringes (3 ml syringe) | Thermo Fisher Scientific | 14-823-436 | |
| BD Vacutainer General Use Syringe Needles (BD Blunt Fill Needle 18G) | Thermo Fisher Scientific | 23-021-020 | |
| Benchtop Centrifuge | Beckman coulter | ||
| Black 96 well plates | Thermo Fisher Scientific | 14-245-177 | |
| BrandTech BRAND BIO-CERT RNase-, DNase-, DNA-free microcentrifuge tubes (1.5mL) | Thermo Fisher Scientific | 14-380-813 | |
| Citric Acid | Fisher Scientific | 02-002-611 | |
| Corning 500ml Vacuum Filter/Storage Bottle System, 0.22 um pore | Corning | 430769 | |
| Disposable folded capillary cells | Malvern | DTS1070 | |
| Ethyl Alcohol, Pure 200 proof | Sigma-Aldrich | 459844 | |
| Fisher Brand Semi-Micro Cuvette | Thermo Fisher Scientific | 14955127 | |
| Invitrogen Conical Tubes (15 mL) (DNase-RNase-free) | Thermo Fisher Scientific | AM12500 | |
| MilliporeSigma Amicon Ultra Centrifugal Filter Units | Thermo Fisher Scientific | UFC901024 | |
| NanoAssemblr Benchtop | Precision Nanyosystems | ||
| Nuclease-free water | Thermo Fisher Scientific | AM9930 | |
| Phosphate Buffered Saline (PBS) | Thermo Fisher Scientific | AM9624 | |
| Quant-iT PicoGreen dsDNA Assay Kit | Thermo Fisher Scientific | P7589 | |
| Quant-iT RiboGreen RNA Assay Kit | Thermo Fisher Scientific | R11490 | |
| Sodium Chloride | Fisher Scientific | 02-004-036 | |
| Sodium Citrate, Dihydrate, granular | Fisher Scientific | 02-004-056 | |
| SpectraMax i3x | Molecular Devices | ||
| Zetasizer Nano | Malvern |
Referências
- Mitchell, M. J., Billingsley, M. M., Haley, R. M., Wechsler, M. E., Peppas, N. A., Langer, R., et al. Engineering precision nanoparticles for drug delivery. Nature Reviews Drug Discovery. , 1-24 (2020).
- Davis, M. E., Chen, Z., Shin, D. M. Nanoparticle therapeutics: an emerging treatment modality for cancer. Nanoscience and technology: A collection of reviews from nature journals. (239), 250 (2010).
- Patra, J. K., Das, G., Fraceto, L. F., et al. Nano based drug delivery systems: recent developments and future prospects. J Nanobiotechnol. 16 (71), (2018).
- Rai, R., Alwani, S., Badea, I. Polymeric nanoparticles in gene therapy: New avenues of design and optimization for delivery applications. Polymers. 11 (4), 745 (2019).
- Bailey, C. M., Nagarajan, R., Camesano, T. A. Designing polymer micelles of controlled size, stability, and functionality for siRNA delivery. ACS Symposium Series. 1271, 35-70 (2017).
- Yin, H., et al. Non-viral vectors for gene-based therapy. Nature Reviews Genetics. 15 (8), 541-555 (2014).
- Bailey-Hytholt, C. M., Nagarajan, R., Camesano, T. A. Förster resonance energy transfer probing of assembly and disassembly of short interfering RNA/Poly(ethylene glycol)-Poly-L-Lysine polyion complex micelles. Molecular Assemblies: Characterization and Applications. , 47-60 (2020).
- Puri, A., Loomis, K., Smith, B. Lipid-based nanoparticles as pharmaceutical drug carriers: from concepts to clinic. Crit Rev Ther Drug Carrier Syst. 26 (6), 523-580 (2009).
- Cullis, P. R., Hope, M. J. Lipid nanoparticle systems for enabling gene therapies. Molecular Therapy. 25 (7), 1467-1475 (2017).
- Munsell, E. V., Ross, N. L., Sullivan, M. O. Journey to the center of the cell: Current Nanocarrier design strategies targeting biopharmaceuticals to the cytoplasm an nucleus. Current Pharmaceutical Design. 22 (9), 1227-1244 (2016).
- Zhao, Y., Huang, L. Lipid nanoparticles for gene delivery. Advances in Genetics. 88, 13-36 (2014).
- Chen, S., et al. Influence of particle size on the in vivo potency of lipid nanoparticle formulations of siRNA. Journal of Controlled Release. 235, 236-244 (2016).
- Wan, C., Allen, T. M., Cullis, P. R. Lipid nanoparticle delivery systems for siRNA-based therapeutics. Drug Delivery and Translational Research. 4 (1), 74-83 (2014).
- Kulkarni, J. A., Cullis, P. R., Van Der Meel, R. Lipid nanoparticles enabling gene therapies: From concepts to clinical utility. Nucleic Acid Therapeutics. 28 (3), 146-157 (2018).
- Shin, M. D., et al. COVID-19 vaccine development and a potential nanomaterial path forward. Nature Nanotechnology. 15 (8), 646-655 (2020).
- Thanh Le, T., et al. The COVID-19 vaccine development landscape. Nature Reviews. Drug Discovery. 19 (5), 305-306 (2020).
- Tam, Y. Y. C., Chen, S., Cullis, P. R. Advances in lipid nanoparticles for siRNA delivery. Pharmaceutics. 5 (3), 498-507 (2013).
- Cayabyab, C., Brown, A., Tharmarajah, G., Thomas, A. mRNA lipid nanoparticles. Precision Nanosystems Application Note. , (2019).
- Gilleron, J., et al. Image-based analysis of lipid nanoparticle-mediated siRNA delivery, intracellular trafficking and endosomal escape. Nature Biotechnology. 31 (7), 638-646 (2013).
- Suzuki, Y., Ishihara, H. Structure, activity and uptake mechanism of siRNA-lipid nanoparticles with an asymmetric ionizable lipid. International Journal of Pharmaceutics. 510 (1), 350-358 (2016).
- Kowalski, P. S., Rudra, A., Miao, L., Anderson, D. G. Delivering the messenger: Advances in technologies for therapeutic mRNA delivery. Molecular Therapy. 27 (4), 710-728 (2019).
- Schmid, J. A. The acidic environment in endocytic compartments. Biochemical Journal. 303 (2), 679-680 (1994).
- Maugeri, M., et al. Linkage between endosomal escape of LNP-mRNA and loading into EVs for transport to other cells. Nature Communications. 10 (1), (2019).
- Kulkarni, J. A., Witzigmann, D., Leung, J., Tam, Y., Cullis, P. R. On the role of helper lipids in lipid nanoparticle formulations of siRNA. Nanoscale. (45), (2019).
- Hafez, I. M., Maurer, N., Cullis, P. R. On the mechanism whereby cationic lipids promote intracellular delivery of polynucleic acids. Gene Therapy. 8 (15), 1188-1196 (2001).
- Hafez, I. M., Culis, P. R. Roles of lipid polymorphism in intracellular delivery. Advanced Drug Delivery Reviews. 47 (2-3), 139-148 (2001).
- Evers, M. J. W., et al. State-of-the-art design and rapid-mixing production techniques of lipid nanoparticles for nucleic acid delivery. Small Methods. 2 (9), 1700375 (2018).
- Mui, B. L., et al. Influence of polyethylene glycol lipid desorption rates on pharmacokinetics and pharmacodynamics of siRNA lipid nanoparticles. Molecular Therapy - Nucleic Acids. 2 (139), (2013).
- Zukancic, D., et al. The importance of poly(Ethylene glycol) and lipid structure in targeted gene delivery to lymph nodes by lipid nanoparticles. Pharmaceutics. 12 (11), 1-16 (2020).
- NEBioCalculator. New England BioLabs Inc Available from: https://nebiocalculator.neb.com/#!/formulas (2020)
- Kastner, E., et al. High-throughput manufacturing of size-tuned liposomes by a new microfluidics method using enhanced statistical tools for characterization. International Journal of Pharmaceutics. 477 (1-2), 361-368 (2014).
- Zhigaltsev, I. V., et al. Bottom-up design and synthesis of limit size lipid nanoparticle systems with aqueous and triglyceride cores using millisecond microfluidic mixing. Langmuir. 28 (7), 3633-3640 (2012).
- Belliveau, N. M., et al. Microfluidic synthesis of highly potent limit-size lipid nanoparticles for in vivo delivery of siRNA. Molecular Therapy - Nucleic Acids. 1 (8), 37 (2012).
- Hassett, K. J., et al. Optimization of lipid nanoparticles for intramuscular administration of mRNA vaccines. Molecular Therapy - Nucleic Acids. 15, 1-11 (2019).
- Tanaka, H., et al. The delivery of mRNA to colon inflammatory lesions by lipid-nano-particles containing environmentally-sensitive lipid-like materials with oleic acid scaffolds. Heliyon. 4 (12), 00959 (2018).
- Singh, J., et al. Nucleic acid lipid nanoparticles. Precision Nanosystems Application Note. , (2018).
Reimpressões e Permissões
Solicitar permissão para reutilizar o texto ou figuras deste artigo JoVE
Solicitar PermissãoThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Todos os direitos reservados