È necessario avere un abbonamento a JoVE per visualizzare questo. Accedi o inizia la tua prova gratuita.
Method Article
Formulazione e caratterizzazione di nanoparticelle lipidiche per la somministrazione genica utilizzando una piattaforma di miscelazione microfluidica
* Questi autori hanno contribuito in egual misura
In questo articolo
Riepilogo
Le nanoparticelle lipidiche sono sviluppate utilizzando un approccio di piattaforma di miscelazione microfluidica per l'incapsulamento di mRNA e DNA.
Abstract
I vettori di farmaci a base lipidica sono stati utilizzati per i sistemi di somministrazione clinicamente e commercialmente disponibili grazie alle loro piccole dimensioni, alla biocompatibilità e all'elevata efficienza di incapsulamento. L'uso di nanoparticelle lipidiche (LNP) per incapsulare gli acidi nucleici è vantaggioso per proteggere l'RNA o il DNA dalla degradazione, promuovendo al contempo l'assorbimento cellulare. Gli LNP contengono spesso più componenti lipidici tra cui un lipide ionizzabile, lipide helper, colesterolo e lipide coniugato polietilenglicole (PEG). Gli LNP possono facilmente incapsulare gli acidi nucleici a causa della presenza lipidica ionizzabile, che a basso pH è cationica e consente la complessazione con RNA o DNA caricati negativamente. Qui gli LNP sono formati incapsulando l'RNA messaggero (mRNA) o il DNA plasmidico (pDNA) utilizzando una rapida miscelazione dei componenti lipidici in una fase organica e della componente dell'acido nucleico in una fase acquosa. Questa miscelazione viene eseguita utilizzando una precisa piattaforma di miscelazione microfluidica, che consente l'autoassemblaggio delle nanoparticelle mantenendo il flusso laminare. Le dimensioni idrodinamiche e la polidispersità sono misurate utilizzando la diffusione dinamica della luce (DLS). La carica superficiale effettiva sull'LNP è determinata misurando il potenziale zeta. L'efficienza di incapsulamento è caratterizzata utilizzando un colorante fluorescente per quantificare l'acido nucleico intrappolato. Risultati rappresentativi dimostrano la riproducibilità di questo metodo e l'influenza che i diversi parametri di formulazione e di processo hanno sui LNP sviluppati.
Introduzione
I vettori farmaceutici sono utilizzati per proteggere e fornire una terapia con proprietà favorevoli tipiche tra cui bassa citotossicità, aumento della biodisponibilità e miglioramento della stabilità1,2,3. Nanoparticelle polimeriche, micelle e particelle a base lipidica sono state precedentemente esplorate per l'incapsulamento e il rilascio di acidi nucleici4,5,6,7. I lipidi sono stati utilizzati in diversi tipi di sistemi nanocarrier, compresi i liposomi e le nanoparticelle lipidiche, in quanto sono biocompatibili con elevata stabilità8. Gli LNP possono facilmente incapsulare gli acidi nucleici per la consegnagenica 9,10. Proteggono l'acido nucleico dalla degradazione da parte delle proteasi sieriche durante la circolazione sistemica11 e possono migliorare la consegna a siti specifici, poiché la topografia superficiale e le proprietà fisiche delle LNP influenzano la loro biodistribuzione12. Gli LNP migliorano anche la penetrazione dei tessuti e l'assorbimento cellulare9. Studi precedenti hanno dimostrato il successo dell'incapsulamento di siRNA all'interno di un LNP13, incluso il primo LNP disponibile in commercio contenente siRNA terapeutico per il trattamento della polineuropatia del trattamento ereditario con amiloidosi14 mediata da transtiretina che è stato approvato dalla Food and Drug Administration (FDA) degli Stati Uniti e dall'Agenzia europea per i medicinali nel 2018. Più recentemente, gli LNP sono stati studiati per la consegna di porzioni di acido nucleico più grandi, vale a dire mRNA e DNA9. A partire dal 2018, c'erano ~ 22 sistemi di somministrazione di acidi nucleici a base lipidica sottoposti a studi clinici14. Inoltre, gli mRNA contenenti LNP sono attualmente candidati principali e sono stati impiegati per un vaccino COVID-1915,16. Il potenziale successo di queste terapie geniche non virali richiede la formazione di particelle piccole (~ 100 nm), stabili e uniformi con un elevato incapsulamento dell'acido nucleico.
L'uso di un lipide ionizzabile come componente principale nella formulazione LNP ha mostrato vantaggi per la complessazione, l'incapsulamento e l'efficienza di consegna14. I lipidi ionizzabili hanno tipicamente una costante di dissociazione acida (pKa) < 7; ad esempio, il dilinoleylmethyl-4-dimethylaminobutyrate (D-Lin-MC3-DMA), il lipide ionizzabile utilizzato nella formulazione LNP approvata dalla FDA, ha un pKa di 6,4417. A pH basso, i gruppi amminici sul lipide ionizzabile diventano protonati e caricati positivamente, consentendo l'assemblaggio con gruppi fosfato caricati negativamente su mRNA e DNA. Il rapporto tra ammina, "N", gruppi e fosfato, "P", gruppi viene utilizzato per ottimizzare l'assemblaggio. Il rapporto N/P dipende dai lipidi e dagli acidi nucleici utilizzati, che variano a seconda della formulazione18. Dopo la formazione, il pH può essere regolato su un pH neutro o fisiologico per consentire la somministrazione terapeutica. A questi valori di pH, il lipide ionizzabile viene anche deprotonato che conferisce carica superficiale neutra al LNP.
Il lipide ionizzabile aiuta anche nella fuga endosomiale19,20. Gli LNP subiscono endocitosi durante l'assorbimento cellulare e devono essere rilasciati dall'endosoma per consegnare il carico di mRNA nel citoplasma cellulare o nel carico di DNA al nucleo21. All'interno dell'endosoma c'è tipicamente un ambiente più acido rispetto al mezzo extracellulare, che rende il lipide ionizzabile caricato positivamente22,23. Il lipide ionizzabile caricato positivamente può interagire con cariche negative sulla membrana lipidica endosomiale, che può causare la destabilizzazione dell'endosoma consentendo il rilascio dell'LNP e dell'acido nucleico. Diversi lipidi ionizzabili sono attualmente in fase di studio per migliorare l'efficacia sia della distribuzione LNP, sia della fuga endosomiale14.
Altri componenti tipici di un LNP includono lipidi helper, come un fosfatidilcolina (PC) o fosfoetanolammina (PE) lipide. 1,2-dioleoil-sn-glicero-3-fosfoetanolammina (DOPE), 1,2-distearoil-sn-glicero-3-fosfocolina (DSPC) e 1,2-dioleoil-sn-glicero-3-fosfocolina (DOPC) sono lipidi helper comunemente usati24,25. DoPE ha dimostrato di formare una fase esagonale invertita II (HII) e migliorare la trasfezione mediante fusione di membrana26, mentre DSPC è stato pensato per stabilizzare LNP con la sua geometria cilindrica27. Il colesterolo è anche incorporato nella formulazione al fine di aumentare la rigidità della membrana, aiutando successivamente nella stabilità del LNP. Infine, il polietilenglicole coniugato con lipidi (PEG) è incluso nella formulazione per fornire la barriera sterica necessaria per aiutare nell'autoassemblaggio delle particelle27. PEG migliora anche la stabilità di archiviazione degli LNP impedendo l'aggregazione. Inoltre, PEG è spesso usato come componente stealth e può aumentare il tempo di circolazione per gli LNP. Tuttavia, questo attributo può anche porre sfide per il reclutamento di LNP negli epatociti attraverso un meccanismo di targeting endogeno guidato dall'apolipoproteina E (ApoE)28. Pertanto, gli studi hanno studiato la lunghezza della catena acilico per la diffusione del PEG dall'LNP, scoprendo che le lunghezze corte (C8-14) si dissociano dall'LNP e sono più suscettibili al reclutamento di ApoE rispetto alle lunghezze acilice più lunghe28. Inoltre, il grado di saturazione della coda lipidica a cui PEG è coniugato ha dimostrato di influenzare la distribuzione tissutale di LNP29. Recentemente, Tween 20, che è un tensioattivo comunemente usato nelle formulazioni di farmaci biologici e ha una lunga coda lipidica insatura, ha dimostrato di avere un'elevata trasfezione nei linfonodi drenanti rispetto a PEG-DSPE, che ha in gran parte trasfettato il muscolo nel sito di iniezione29. Questo parametro può essere ottimizzato per ottenere la biodistribuzione LNP desiderata.
I metodi convenzionali di formazione degli LNP includono il metodo di idratazione a film sottile e il metodo di iniezione di etanolo27. Mentre queste sono tecniche prontamente disponibili, sono anche ad alta intensità di lavoro, possono comportare una bassa efficienza di incapsulamento e sono difficili da scalare fino a27. I progressi nelle tecniche di miscelazione hanno portato a metodi più suscettibili di scalare, mentre sviluppano particelle più uniformi27. Questi metodi includono la miscelazione a giunzione T, la miscelazione a spina di pesce sfalsata e la messa a fuoco idrodinamica microfluidica27. Ogni metodo ha una struttura unica, ma tutti consentono una rapida miscelazione di una fase acquosa contenente l'acido nucleico con una fase organica contenente i componenti lipidici, con conseguente elevato incapsulamento dell'acido nucleico27. In questo protocollo, viene utilizzata una miscelazione rapida e controllata attraverso una cartuccia microfluidica, che impiega il design di miscelazione a spina di pesce sfalsato. Questo protocollo delinea la preparazione, l'assemblaggio e la caratterizzazione dell'acido nucleico contenente LNP.
Protocollo
Uno schema del processo complessivo è fornito nella Figura 1.
1. Preparazione dei buffer
NOTA: il filtraggio sterile dei tamponi è altamente consigliato qui per rimuovere eventuali particelle che possono influire sulla qualità dell'acido nucleico e dell'LNP.
- Soluzione salina tamponata con fosfato (PBS)
- Preparare 1x PBS utilizzando 8 mM Na2HPO4, 2 mM KH2PO4, 137 mM NaCl e 2,7 mM KCl in acqua priva di nucleasi e regolare il pH a 7,4.
- Sterilizzare mediante filtrazione sotto vuoto utilizzando un filtro a pori da 0,22 μm.
- Tampone citrato
- Preparare il tampone citrato utilizzando 5 mM di citrato di sodio, 5 mM di acido citrico e 150 mM di cloruro di sodio in acqua priva di nucleasi e regolare a pH 4,5.
- Sterilizzare mediante filtrazione sotto vuoto utilizzando un filtro a pori da 0,22 μm.
NOTA: il tampone citrato deve essere preparato solo se l'mRNA è l'acido nucleico che sarà incapsulato nell'LNP. Se il DNA sarà incapsulato saltare 1.2 e procedere a 1.3.
- Tampone acido malico
- Preparare il tampone di acido malico utilizzando 20 mM di acido malico e 30 mM di cloruro di sodio in acqua priva di nucleasi e regolare a pH 3,0.
- Sterilizzare mediante filtrazione sotto vuoto utilizzando un filtro a pori da 0,22 μm.
NOTA: il tampone dell'acido malico deve essere preparato solo se il DNA è l'acido nucleico che sarà incapsulato nel LNP. Saltare 1,3 se l'mRNA deve essere incapsulato. Il tampone citrato viene utilizzato per l'incapsulamento dell'mRNA, poiché il pH inferiore di 3,0 con tampone acido malico può portare ad una maggiore probabilità di degradazione dell'mRNA. Il protocollo può essere messo in pausa qui.
2. Preparazione della miscela lipidica
- Se i lipidi di riserva sono in polvere, solubilizzare in etanolo puro a prova di 200.
- Calcolare la miscela richiesta di componenti lipidici in base al rapporto molare desiderato. Un rapporto molare di 50:10:39:1 (lipide ionizzabile:lipide helper:colesterolo:PEG) sarà usato qui come esempio per una concentrazione lipidica totale di 10 mM. La tabella 1 mostra le concentrazioni e i volumi necessari per ciascuno di questi componenti.
NOTA: Quando si calcola il volume necessario per raggiungere la concentrazione della miscela lipidica in etanolo (EtOH) per il miscelatore microfluidico, il volume totale viene contabilizzato per garantire che l'aggiunta di EtOH non influenzi le concentrazioni lipidiche. Ad esempio, un volume lipidico ionizzabile di 68,5 μL viene calcolato moltiplicando la concentrazione di 5 mM in etanolo per un volume totale di miscela lipidica di 533 μL e quindi dividendo per la concentrazione lipidica di riserva di 38,9 mM. - Aggiungere la quantità appropriata di ciascuna soluzione di riserva lipidica a un flaconcino di vetro per consentire ai componenti di mescolarsi con vortici intermittenti. Aggiungere 200 etanolo di prova per una miscela totale di 533 μL. Per l'esempio nella Tabella 1, questo è 254 μL di etanolo.
NOTA: per una singola corsa per produrre 1 mL di LNP, sono necessari 342,5 μL di soluzione lipidica. Ciò è dovuto a una miscela 3:1 di acido nucleico acquoso in soluzione lipidica organica con un certo volume scartato prima e dopo la raccolta del campione. Una miscela di 533 μL è fatta per compensare come troppi.
3. Preparazione della soluzione di acido nucleico
NOTA: La preparazione e la manipolazione di soluzioni di acido nucleico deve essere eseguita in un ambiente sterile e privo di RNasi, ove possibile. Lavora in un armadio di biosicurezza quando possibile con l'acido nucleico.
- Calcola il rapporto N/P. Il rapporto N/P è il numero totale di gruppi lipidici amminici ionizzabili (N) rispetto al numero totale di gruppi fosfatici acidi nucleici caricati negativamente (P). Il rapporto N/P è spesso un parametro che può essere ottimizzato durante la formazione di LNP. Segui i passaggi seguenti.
- Calcola il numero di N unità utilizzando la seguente formula:
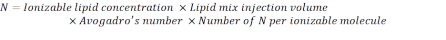
NOTA: La concentrazione lipidica ionizzabile (Tabella 1) è di 5 mM, che equivale a 5 x 10-6 mol/mL. Il volume di iniezione lipidico richiesto è di 0,3425 ml. Ad esempio, se il numero di N unità per molecola è 1, usando l'equazione di cui sopra, ci sono 1,03 x 1018 N unità nel mix lipidico. - Calcolare le unità P per il rapporto N/P desiderato. Qui viene utilizzato un N / P = 36 per esempio.
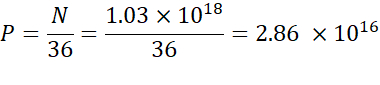
- Calcola il numero di N unità utilizzando la seguente formula:
- Calcolare la concentrazione di acido nucleico necessaria per ottenere unità 2,86 x 1016 P utilizzando l'equazione seguente.
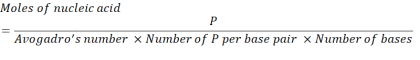
Dove, il numero di unità P per coppia di basi per mRNA è 1 e il DNA è 2. Per un mRNA con 1.200 basi, la quantità di mRNA richiesta per un N/P = 36 è 3,96 x 10-11 moli. - Calcola la concentrazione di massa di mRNA richiesta per N/P = 36 usando l'equazione seguente.
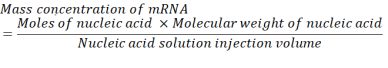
Il peso molecolare medio di un'unità di ribonucleotide monofosfato è 322 g/mol30. Con 1.200 basi di mRNA, il peso molecolare dell'mRNA è di 386.400 g/mol. Il volume di iniezione richiesto della soluzione di acido nucleico è di 1,028 ml. Pertanto, la concentrazione di mRNA necessaria è 1,488x10-5 g / mL, che è 14,88 μg / mL. - Costituiscono 1,5 mL di 14,88 μg/mL di mRNA nel tampone citrato.
NOTA: Quando il DNA sarà l'acido nucleico incapsulato, utilizzare il tampone dell'acido malico per covare la soluzione di acido nucleico.
4. Innesco dei canali microfluidici
NOTA: questo protocollo è adattato dalle linee guida del produttore dello strumento.
- Inserire i parametri di priming nel software dello strumento cliccando sugli appositi campi (Tabella 2).
NOTA: Si consiglia un rapporto di flusso di 3:1 e una portata di 4-12 ml/min27,31. Questo ha dimostrato di essere ottimale negli studi presentati qui, così come dal produttore. Questo può essere variato se è di interesse per l'applicazione. - Aprire il coperchio dello strumento e posizionare una cartuccia microfluidica nel blocco rotante.
- Aspirare almeno 0,5 ml di etanolo in una siringa da 1 mL, assicurandosi che non vi siano bolle o vuoti d'aria sulla punta della siringa. Caricare questa siringa nell'ingresso destro della cartuccia.
- Riempire una siringa da 3 mL con 1,5 mL di tampone acquoso (citrato per l'RNA e acido malico per il DNA), assicurandosi che non vi siano bolle d'aria o spazi vuoti. Caricare questa siringa nell'ingresso sinistro della cartuccia.
- Inserire due tubi conici da 15 ml nei porta clip per fungere da contenitori per i rifiuti.
- Fare clic su Esegui nel software dello strumento per iniziare il mixaggio, assicurandosi che i parametri siano inseriti correttamente.
- Quando lo strumento smette di innescare, indicato dalla luce blu inferiore che si spegne, aprire il coperchio e smaltire correttamente i tubi conici e le siringhe.
5. Formazione LNP
NOTA: questo protocollo è adattato dalle linee guida del produttore dello strumento.
- Aggiornare il software con i parametri di formulazione cliccando sugli appositi campi (Tabella 2).
- Riempire una siringa da 1 mL con la miscela lipidica (preparata al passaggio 2). Rimuovere eventuali spazi d'aria o bolle sulla punta della siringa e inserire la siringa sul lato destro della cartuccia.
- Aspirare la soluzione di acido nucleico (preparata al punto 3) in una siringa da 3 ml, assicurandosi che non vi siano bolle o vuoti d'aria nella punta della siringa. Inserire la siringa nell'ingresso sinistro della cartuccia.
NOTA: i volumi sono forniti per creare una soluzione da 1 mL di LNP. Questo strumento può incorporare siringhe di dimensioni fino a 10 ml e i volumi possono essere scalati di conseguenza senza alcuna influenza sul risultato. Il volume massimo di LNP che possono essere preparati in una preparazione è di 12 ml. - Etichettare un tubo conico privo di RNasi da 15 ml con il nome del campione e inserirlo nella clip del tubo sinistro. Posizionare un conico di scarico da 15 ml nella clip del tubo destro.
- Chiudere il coperchio dello strumento e fare clic su Esegui, dopo aver confermato il corretto inserimento dei parametri.
- Al termine del funzionamento dello strumento, scartare correttamente il contenitore e la cartuccia dei rifiuti. Conservare il tubo conico con il campione LNP.
- Diluire l'LNP 5x con PBS per ridurre al minimo l'etanolo a <5% (v/v).
NOTA: È importante diluire gli LNP in PBS il prima possibile dopo la miscelazione microfluidica per evitare la degradazione. Eseguire sempre la diluizione in un armadio di biosicurezza e continuare a lavorare nell'armadio di biosicurezza durante gli scambi tampone.
6. Scambio di buffer
NOTA: viene fornito il protocollo per l'utilizzo di filtri ultracentrghi. Mentre questo metodo si traduce in uno scambio di buffer più efficiente in termini di tempo, la dialisi può essere sostituita qui.
- Pre-lavare un filtro ultracentrghifugo (dimensione pori 100 kDa) con 2 mL di PBS centrifugando a 1000 x g per 5 min. Svuotare il PBS dal compartimento inferiore.
NOTA: PBS viene scelto per aumentare il pH a 7,4 ± 0,2, che è fisiologicamente rilevante e comporterà che il lipide ionizzabile abbia una carica neutra. - Aggiungere LNP diluiti al vano superiore del filtro ultracentrghino prelavato e della centrifuga a 1000 x g per 12 minuti.
- Scartare il flusso attraverso dal compartimento inferiore. Eseguire altri due lavaggi aggiungendo ogni volta 5 ml di PBS al filtro ultracentrghifugo. Centrifuga agli stessi parametri. Non esiste un volume massimo che deve essere mantenuto.
NOTA: se è stato preparato un volume di LNP in scala, aumentare di conseguenza il volume di PBS per ogni lavaggio. Ad esempio, se 2 mL di LNP sono stati preparati in una singola esecuzione, vengono suggeriti 10 mL PBS per lavaggio. - Pipettare la soluzione LNP contro le pareti del filtro ultracentrghifuga alcune volte per ridurre al minimo la perdita di LNP. Rimuovere la soluzione di LNP dal filtro ultracentrghifuga e conservare in un flaconcino privo di nucleasi. Aggiungere PBS se necessario per ottenere un volume finale della soluzione LNP di 1 mL.
- Filtrare attraverso un filtro a siringa pre-bagnato da 0,2 μm, se necessario.
NOTA: il protocollo può essere messo in pausa qui.
7. Misurare l'efficienza dell'incapsulamento
- Preparare una curva standard effettuando diluizioni seriali 2 volte della soluzione di acido nucleico funzionante in PBS, iniziando con una concentrazione massima di 500 ng / mL e facendo almeno cinque diluizioni. Utilizzare PBS come spazio vuoto.
- Preparare le diluizioni del campione LNP. Diluire campioni di LNP con PBS, per ottenere una concentrazione teorica approssimativa che si trova intorno al punto medio della curva standard (es. ~ 250 ng/mL di acido nucleico stimato dalla concentrazione iniziale).
- Preparare una soluzione del reagente di quantificazione dell'RNA (per le misurazioni dell'mRNA) con TritonX-100 per interrompere gli LNP e misurare la quantità totale di acido nucleico all'interno e all'esterno dell'LNP. Questa soluzione contiene lo 0,5% (v/v) di reagente RNA, lo 0,4% (v/v) di TritonX-100 e il 99,1% (v/v) di PBS.
- Preparare una soluzione del reagente senza TritonX-100 per misurare la quantità di acido nucleico non incapsulato negli LNP. Questa soluzione contiene reagente RNA 0,5% (v/v) e 99,5% (v/v) PBS.
NOTA: se gli LNP incapsulano DNA a doppio filamento (dsDNA), come il DNA plasmidico, utilizzare invece il reagente dsDNA in 7.3 e 7.4, seguendo la stessa procedura. - In una piastra a fluorescenza nera a 96 pozzetti, caricare almeno quattro repliche di ciascuna delle soluzioni standard di LNP e acido nucleico preparate in 7.1 e 7.2.
- Alla metà delle repliche di standard e campioni, aggiungere un volume uguale del reagente contenente TritonX-100. Questo quantificherà la quantità totale di acido nucleico.
- Ai restanti pozzeggi di standard e campioni, aggiungere un volume uguale del reagente senza TritonX-100. Questo quantificherà la quantità di acido nucleico non incapsulato all'interno del LNP.
- Agitare la piastra per 5 minuti a temperatura ambiente per garantire un'accurata miscelazione di standard e campioni con il reagente aggiunto, prendendo precauzioni per evitare l'esposizione alla luce.
- Misurare la fluorescenza utilizzando un lettore di micropiasse, con una lunghezza d'onda di eccitazione di 480 nm e una lunghezza d'onda di emissione di 520 nm.
- Calcolare la concentrazione di acido nucleico al di fuori dell'LNP utilizzando la curva standard realizzata con l'aggiunta del reagente senza TritonX-100. Moltiplicare per il fattore di diluizione utilizzato al punto 7.2.
- Calcolare la concentrazione di acido nucleico sia all'interno che all'esterno dell'LNP utilizzando la curva standard realizzata con l'aggiunta del reagente contenente TritonX-100. Moltiplicare per il fattore di diluizione utilizzato al punto 7.2.
- Calcolare la concentrazione di acido nucleico all'interno sottraendo la concentrazione di acido nucleico all'esterno (calcolata dal punto 7.10) dalla concentrazione totale di acido nucleico sia all'interno che all'esterno (calcolata dal punto 7.11)
- Quantificare l'efficienza di incapsulamento dal rapporto tra la concentrazione di acido nucleico all'interno del LNP (calcolata dal passo 7.12) e la concentrazione totale di acido nucleico (calcolata dal passo 7.11).
NOTA: il protocollo può essere messo in pausa qui.
8. Aggiustamenti della concentrazione
- Se necessario, regolare la concentrazione di acido nucleico all'interno della soluzione LNP utilizzando i risultati dell'efficienza di incapsulamento.
- Se si desidera una soluzione meno concentrata, diluire la soluzione con PBS per ottenere la concentrazione desiderata.
- Se si desidera una soluzione più concentrata, eseguire ulteriori cicli di centrifugazione utilizzando un filtro ultracentrghino.
NOTA: il protocollo può essere messo in pausa qui.
9. Misurare le dimensioni idrodinamiche LNP e la polidispersibilità
- Diluire un'aliquota della soluzione LNP 40x con PBS per ottenere un volume finale di 1 mL.
NOTA: questa diluizione può essere modificata se necessario. Questo valore di diluizione è suggerito in quanto utilizza un piccolo volume della soluzione stock LNP fornendo risultati di qualità. - Utilizzando una semi-micro cuvetta, misurare il diametro idrodinamico e l'indice di polidispersità. Aggiungere la soluzione LNP nella cuvetta e inserirla nello strumento. Impostare una procedura operativa nel software dello strumento per includere il tipo di misurazione, i dettagli del campione (materiale, disperdente, temperatura e tipo di cella) e le istruzioni di misurazione (numero di tirature). Fare clic su Avvia quando si è pronti per iniziare l'acquisizione della misurazione.
10. Misurare il potenziale zeta LNP
- Diluire un'aliquota della soluzione LNP 40x con acqua priva di nucleasi per ottenere un volume finale di 1 mL.
NOTA: l'acqua priva di nucleasi viene utilizzata come solvente per le misurazioni del potenziale zeta per ridurre al minimo l'influenza di tamponi salini elevati sulla conduttività. - Utilizzando una cellula zeta capillare piegata, misurare il potenziale zeta.
- Aggiungere la soluzione LNP nella cuvetta fino alla linea di riempimento. Inserire nello strumento assicurandosi che gli elettrodi entrino in contatto con lo strumento.
- Impostare una procedura operativa nel software dello strumento per includere il tipo di misurazione, i dettagli del campione (materiale, disperdente, temperatura e tipo di cella) e le istruzioni di misurazione (numero di tirature). Fare clic su Avvia quando si è pronti per iniziare l'acquisizione della misurazione.
Risultati
Più lotti di LNP con la stessa formulazione lipidica e rapporto N/P di 6 sono stati sviluppati in giorni separati per dimostrare la riproducibilità della tecnica. I lotti 1 e 2 hanno determinato distribuzioni dimensionali sovrapposte con polidispersità simile (Figura 2A) Non è stata osservata alcuna differenza significativa nelle dimensioni o nell'efficienza di incapsulamento tra i due diversi lotti (Figura 2B). L'efficienza ...
Discussione
Riproducibilità, velocità e screening a basso volume sono vantaggi significativi dell'utilizzo della miscelazione microfluidica per formare LNP rispetto ad altri metodi esistenti (ad esempio, idratazione del film lipidico e iniezione di etanolo). Abbiamo dimostrato la riproducibilità di questo metodo senza alcun impatto sull'efficienza di incapsulamento o sulla dimensione delle particelle osservate con diversi lotti LNP. Questo è un criterio essenziale affinché qualsiasi terapia, compresi gli LNP, diventi clinicamen...
Divulgazioni
Tutti gli autori sono dipendenti di Sanofi. Gli autori dichiarano di non avere conflitti di interesse o interessi finanziari concorrenti.
Riconoscimenti
Grazie ad Atul Saluja, Yatin Gokarn, Maria-Teresa Peracchia, Walter Schwenger e Philip Zakas per la loro guida e il loro contributo allo sviluppo di LNP.
Materiali
| Name | Company | Catalog Number | Comments |
| 1,2-dimyristoyl-rac-glycero-3-methoxypolyethylene glycol-2000 (C-14 PEG) | Avanti Polar Lipids | 880151P | |
| 10 µl Graduated Filter Tips (RNase-,DNase-, DNA-free) | USA Scientific | 1121-3810 | |
| 1000 µl Graduated Filter Tips (RNase-,DNase-, DNA-free) | USA Scientific | 1111-2831 | |
| 20 µl Beveled Filter Tips (RNase-,DNase-, DNA-free) | USA Scientific | 1120-1810 | |
| 200 µl Graudated Filter Tips (RNase-,DNase-, DNA-free) | USA Scientific | 1120-8810 | |
| 3β-Hydroxy-5-cholestene, 5-Cholesten-3β-ol (Cholesterol) | Sigma-Aldrich | C8667 | |
| BD Slip Tip Sterile Syringes (1 ml syringe) | Thermo Fisher Scientific | 14-823-434 | |
| BD Slip Tip Sterile Syringes (3 ml syringe) | Thermo Fisher Scientific | 14-823-436 | |
| BD Vacutainer General Use Syringe Needles (BD Blunt Fill Needle 18G) | Thermo Fisher Scientific | 23-021-020 | |
| Benchtop Centrifuge | Beckman coulter | ||
| Black 96 well plates | Thermo Fisher Scientific | 14-245-177 | |
| BrandTech BRAND BIO-CERT RNase-, DNase-, DNA-free microcentrifuge tubes (1.5mL) | Thermo Fisher Scientific | 14-380-813 | |
| Citric Acid | Fisher Scientific | 02-002-611 | |
| Corning 500ml Vacuum Filter/Storage Bottle System, 0.22 um pore | Corning | 430769 | |
| Disposable folded capillary cells | Malvern | DTS1070 | |
| Ethyl Alcohol, Pure 200 proof | Sigma-Aldrich | 459844 | |
| Fisher Brand Semi-Micro Cuvette | Thermo Fisher Scientific | 14955127 | |
| Invitrogen Conical Tubes (15 mL) (DNase-RNase-free) | Thermo Fisher Scientific | AM12500 | |
| MilliporeSigma Amicon Ultra Centrifugal Filter Units | Thermo Fisher Scientific | UFC901024 | |
| NanoAssemblr Benchtop | Precision Nanyosystems | ||
| Nuclease-free water | Thermo Fisher Scientific | AM9930 | |
| Phosphate Buffered Saline (PBS) | Thermo Fisher Scientific | AM9624 | |
| Quant-iT PicoGreen dsDNA Assay Kit | Thermo Fisher Scientific | P7589 | |
| Quant-iT RiboGreen RNA Assay Kit | Thermo Fisher Scientific | R11490 | |
| Sodium Chloride | Fisher Scientific | 02-004-036 | |
| Sodium Citrate, Dihydrate, granular | Fisher Scientific | 02-004-056 | |
| SpectraMax i3x | Molecular Devices | ||
| Zetasizer Nano | Malvern |
Riferimenti
- Mitchell, M. J., Billingsley, M. M., Haley, R. M., Wechsler, M. E., Peppas, N. A., Langer, R., et al. Engineering precision nanoparticles for drug delivery. Nature Reviews Drug Discovery. , 1-24 (2020).
- Davis, M. E., Chen, Z., Shin, D. M. Nanoparticle therapeutics: an emerging treatment modality for cancer. Nanoscience and technology: A collection of reviews from nature journals. (239), 250 (2010).
- Patra, J. K., Das, G., Fraceto, L. F., et al. Nano based drug delivery systems: recent developments and future prospects. J Nanobiotechnol. 16 (71), (2018).
- Rai, R., Alwani, S., Badea, I. Polymeric nanoparticles in gene therapy: New avenues of design and optimization for delivery applications. Polymers. 11 (4), 745 (2019).
- Bailey, C. M., Nagarajan, R., Camesano, T. A. Designing polymer micelles of controlled size, stability, and functionality for siRNA delivery. ACS Symposium Series. 1271, 35-70 (2017).
- Yin, H., et al. Non-viral vectors for gene-based therapy. Nature Reviews Genetics. 15 (8), 541-555 (2014).
- Bailey-Hytholt, C. M., Nagarajan, R., Camesano, T. A. Förster resonance energy transfer probing of assembly and disassembly of short interfering RNA/Poly(ethylene glycol)-Poly-L-Lysine polyion complex micelles. Molecular Assemblies: Characterization and Applications. , 47-60 (2020).
- Puri, A., Loomis, K., Smith, B. Lipid-based nanoparticles as pharmaceutical drug carriers: from concepts to clinic. Crit Rev Ther Drug Carrier Syst. 26 (6), 523-580 (2009).
- Cullis, P. R., Hope, M. J. Lipid nanoparticle systems for enabling gene therapies. Molecular Therapy. 25 (7), 1467-1475 (2017).
- Munsell, E. V., Ross, N. L., Sullivan, M. O. Journey to the center of the cell: Current Nanocarrier design strategies targeting biopharmaceuticals to the cytoplasm an nucleus. Current Pharmaceutical Design. 22 (9), 1227-1244 (2016).
- Zhao, Y., Huang, L. Lipid nanoparticles for gene delivery. Advances in Genetics. 88, 13-36 (2014).
- Chen, S., et al. Influence of particle size on the in vivo potency of lipid nanoparticle formulations of siRNA. Journal of Controlled Release. 235, 236-244 (2016).
- Wan, C., Allen, T. M., Cullis, P. R. Lipid nanoparticle delivery systems for siRNA-based therapeutics. Drug Delivery and Translational Research. 4 (1), 74-83 (2014).
- Kulkarni, J. A., Cullis, P. R., Van Der Meel, R. Lipid nanoparticles enabling gene therapies: From concepts to clinical utility. Nucleic Acid Therapeutics. 28 (3), 146-157 (2018).
- Shin, M. D., et al. COVID-19 vaccine development and a potential nanomaterial path forward. Nature Nanotechnology. 15 (8), 646-655 (2020).
- Thanh Le, T., et al. The COVID-19 vaccine development landscape. Nature Reviews. Drug Discovery. 19 (5), 305-306 (2020).
- Tam, Y. Y. C., Chen, S., Cullis, P. R. Advances in lipid nanoparticles for siRNA delivery. Pharmaceutics. 5 (3), 498-507 (2013).
- Cayabyab, C., Brown, A., Tharmarajah, G., Thomas, A. mRNA lipid nanoparticles. Precision Nanosystems Application Note. , (2019).
- Gilleron, J., et al. Image-based analysis of lipid nanoparticle-mediated siRNA delivery, intracellular trafficking and endosomal escape. Nature Biotechnology. 31 (7), 638-646 (2013).
- Suzuki, Y., Ishihara, H. Structure, activity and uptake mechanism of siRNA-lipid nanoparticles with an asymmetric ionizable lipid. International Journal of Pharmaceutics. 510 (1), 350-358 (2016).
- Kowalski, P. S., Rudra, A., Miao, L., Anderson, D. G. Delivering the messenger: Advances in technologies for therapeutic mRNA delivery. Molecular Therapy. 27 (4), 710-728 (2019).
- Schmid, J. A. The acidic environment in endocytic compartments. Biochemical Journal. 303 (2), 679-680 (1994).
- Maugeri, M., et al. Linkage between endosomal escape of LNP-mRNA and loading into EVs for transport to other cells. Nature Communications. 10 (1), (2019).
- Kulkarni, J. A., Witzigmann, D., Leung, J., Tam, Y., Cullis, P. R. On the role of helper lipids in lipid nanoparticle formulations of siRNA. Nanoscale. (45), (2019).
- Hafez, I. M., Maurer, N., Cullis, P. R. On the mechanism whereby cationic lipids promote intracellular delivery of polynucleic acids. Gene Therapy. 8 (15), 1188-1196 (2001).
- Hafez, I. M., Culis, P. R. Roles of lipid polymorphism in intracellular delivery. Advanced Drug Delivery Reviews. 47 (2-3), 139-148 (2001).
- Evers, M. J. W., et al. State-of-the-art design and rapid-mixing production techniques of lipid nanoparticles for nucleic acid delivery. Small Methods. 2 (9), 1700375 (2018).
- Mui, B. L., et al. Influence of polyethylene glycol lipid desorption rates on pharmacokinetics and pharmacodynamics of siRNA lipid nanoparticles. Molecular Therapy - Nucleic Acids. 2 (139), (2013).
- Zukancic, D., et al. The importance of poly(Ethylene glycol) and lipid structure in targeted gene delivery to lymph nodes by lipid nanoparticles. Pharmaceutics. 12 (11), 1-16 (2020).
- NEBioCalculator. New England BioLabs Inc Available from: https://nebiocalculator.neb.com/#!/formulas (2020)
- Kastner, E., et al. High-throughput manufacturing of size-tuned liposomes by a new microfluidics method using enhanced statistical tools for characterization. International Journal of Pharmaceutics. 477 (1-2), 361-368 (2014).
- Zhigaltsev, I. V., et al. Bottom-up design and synthesis of limit size lipid nanoparticle systems with aqueous and triglyceride cores using millisecond microfluidic mixing. Langmuir. 28 (7), 3633-3640 (2012).
- Belliveau, N. M., et al. Microfluidic synthesis of highly potent limit-size lipid nanoparticles for in vivo delivery of siRNA. Molecular Therapy - Nucleic Acids. 1 (8), 37 (2012).
- Hassett, K. J., et al. Optimization of lipid nanoparticles for intramuscular administration of mRNA vaccines. Molecular Therapy - Nucleic Acids. 15, 1-11 (2019).
- Tanaka, H., et al. The delivery of mRNA to colon inflammatory lesions by lipid-nano-particles containing environmentally-sensitive lipid-like materials with oleic acid scaffolds. Heliyon. 4 (12), 00959 (2018).
- Singh, J., et al. Nucleic acid lipid nanoparticles. Precision Nanosystems Application Note. , (2018).
Ristampe e Autorizzazioni
Richiedi autorizzazione per utilizzare il testo o le figure di questo articolo JoVE
Richiedi AutorizzazioneThis article has been published
Video Coming Soon