Method Article
Preparação de explantes de somita único a partir de embriões de peixe-zebra
Neste Artigo
Resumo
Apresentamos um protocolo para isolar somitos únicos de embriões de peixe-zebra, cuja dinâmica pode ser acompanhada em cultura por várias horas por microscopia de lapso de tempo de fluorescência, fornecendo assim uma metodologia para quantificar as mudanças na forma da escala do tecido na resolução de uma única célula.
Resumo
O eixo do corpo dos embriões de vertebrados é periodicamente subdividido em unidades multicelulares 3D chamadas somitos. Enquanto oscilações genéticas e pré-padrões moleculares determinam a escala de comprimento inicial dos somitos, processos mecânicos têm sido implicados na definição de seu tamanho e forma finais. Para entender melhor as propriedades materiais intrínsecas dos somitos, é desenvolvido um método para cultivar explante de somitos únicos a partir de embriões de peixe-zebra. Os somitos únicos são isolados removendo primeiro a pele dos embriões, seguida pela remoção da gema e excisão sequencial dos tecidos vizinhos. Usando embriões transgênicos, a distribuição de várias estruturas subcelulares pode ser observada por microscopia fluorescente de lapso de tempo. A dinâmica dos somitos explantados pode ser acompanhada por várias horas, fornecendo assim uma estrutura experimental para estudar as mudanças na forma da escala do tecido na resolução de uma única célula. Essa abordagem permite a manipulação mecânica direta de somitos, permitindo a dissecção das propriedades do material do tecido. Finalmente, a técnica descrita aqui pode ser prontamente estendida para o explante de outros tecidos, como a notocorda, a placa neural e o mesoderma da placa lateral.
Introdução
Grande parte do sistema musculoesquelético adulto vertebrado emerge de somitos embrionários, que se formam de maneira periódica e rítmica ao longo do eixo corporal dos embriões 1,2. Os somitos são unidades multicelulares tridimensionais (3D) que consistem tipicamente em um núcleo interno de células mesenquimais e uma camada epitelial periférica circundada por uma matriz extracelular rica em fibronectina3. A morfologia dos somitos, ou seja, seu tamanho e forma, é em parte determinada pelo relógio de segmentação e pré-padrões moleculares a jusante. No entanto, na última década, descobriu-se que pistas e forças mecânicas também desempenham um papel na regulação do relógio de segmentação4, além de facilitar a formação de somitos 5,6,7 e garantir maior precisão dos comprimentos dos somitos após a formação inicial de somitos8.
A mecânica dos tecidos pode ser estudada diretamente in vivo com a disponibilidade de novas ferramentas9, no entanto, para obter uma imagem completa subjacente aos processos físicos, as propriedades intrínsecas do material dos tecidos precisam ser estudadas simultaneamente. O protocolo descrito aqui fornece uma abordagem simples para preparar somitos únicos, cujas propriedades físicas das escalas celulares e teciduais podem ser estudadas isoladamente do embrião. Embora existam vários protocolos para preparar explantes em estágios de desenvolvimento semelhantes 10,11,12,13,14, até onde sabemos, este é o primeiro protocolo que descreve o isolamento de somites únicos. O protocolo é simples de implementar e requer apenas equipamentos básicos disponíveis na maioria dos laboratórios de peixe-zebra que trabalham com embriões.
Para ajudar a desvendar o papel da mecânica na formação morfológica de somitos, um método é desenvolvido para cultivar explante de somitos únicos a partir de embriões de peixe-zebra, que pode ser usado para sondar as propriedades intrínsecas do material dos somitos.
Protocolo
Este protocolo envolve o uso de embriões de vertebrados vivos com menos de 1 dia após a fertilização. Todos os experimentos foram realizados usando embriões derivados de adultos que acasalaram livremente e, portanto, estão cobertos pela licença geral de experimentos com animais da EPFL concedida pelo Service de la Consommation et des Affaires Vétérinaires do cantão de Vaud - Suíça (número de autorização VD-H23).
1. Antes da dissecação
- Obtenha embriões de um cruzamento de linhagens transgênicas heterozigóticas de interesse. Para explantar dois a três somitos, normalmente são necessários apenas alguns embriões. Neste protocolo, um embrião foi utilizado para explante de um somito.
- Prepare 1x tampão E3 de dois estoques 50x: o estoque 1 contém 0,458 mM Na2HPO4, 0,042 mM KH2PO4, 4,084 mM NaCl, 0,128 mM KCl e o estoque 2 contém 0,33 mM CaCl2, 0,33 mM MgSO4. Prepare 500 mL de 1x tampão E3 misturando 480 mL de água destilada com 10 mL cada um dos dois estoques 50x.
- Se dissecar no mesmo dia, levantar embriões em 25 mL de meio E3 em uma placa de Petri a 33 ° C até o estágio de três somitos. Se dissecar na manhã seguinte, criar embriões em meio E3 a 28 ° C até o estágio de escudo antes de transferir para 19 ° C para incubação durante a noite.
- Para identificar o estágio de blindagem ou os diferentes estágios de somito, use um estereomicroscópio de laboratório padrão com uma faixa de ampliação ajustável de aproximadamente 0,67x a 4,5x.
- Montar ferramentas e reagentes necessários para a dissecação.
- Meio L-15 da Alíquota Leibovitz em tubos de 50 mL e guardar na geladeira. Recomendamos usar a mesma alíquota da geladeira por no máximo duas rodadas.
- Preparar 50 ml de agarose a 2% em meio L-15 com um agitador magnético e manter a temperatura a 85 °C. Depois de dissolvida, armazene a solução em temperatura ambiente.
- Preparar 25 ml de agarose a 2% de baixo ponto de fusão em meio L-15 utilizando um agitador magnético e mantendo a temperatura a 85 °C. Uma vez dissolvido, alíquota da solução em tubos de 1,5 ml e conservar a 4 °C. Isso será usado para preparar a câmara de imagem.
- Despeje a agarose a 2% derretida em uma placa de Petri de 20 mm até que um terço da profundidade da placa seja preenchida e deixe a solução de agarose solidificar. As dissecções serão realizadas nessas placas de Petri. Preparar as placas de Petri com cobertura de agarose com bastante antecedência e conservar a 4 °C.
- Monte as seguintes ferramentas: pipeta Pasteur para transferência de embriões, um par de pinças para descorionar embriões, um par de pinças finas para remoção de pele de embriões, uma micro faca para fazer incisões em embriões, pipeta de vidro polido a fogo para transferir explantes para a câmara de imagem.
- Esterilize a pipeta de vidro em etanol 100% por 15 min e enxágue duas vezes pipetando 5 mL de meio L-15 para cima e para baixo.
- Prepare uma ferramenta de cílios colando um cílio ou cabelo da sobrancelha em um capilar de vidro.
- Use um estereoscópio fluorescente com uma faixa de ampliação ajustável de aproximadamente 0,63x a 6,3x e filtros GFP e RFP padrão para classificar embriões transgênicos positivos e transferir para uma placa de Petri separada com meio E3.
2. Preparação de explante de somito único
- Transfira 2 ou 3 embriões para uma placa revestida de agarose (feita na etapa 1.5.4) preenchida com 10 mL de meio L-15. Certifique-se de que o prato junto com o meio L-15 seja pré-aquecido a 28 ° C em uma incubadora por 30 minutos antes de transferir os embriões.
- Decorione cuidadosamente (Figura 1 A, B) um embrião com um par de pinças. Segure o córion com uma das pinças e use a segunda pinça para beliscar e puxar o córion perto de onde a primeira pinça está posicionada.
- Repita esta etapa mais uma vez para abrir o córion amplamente, após o que o embrião pode ser empurrado suavemente para fora. Certifique-se de que o embrião não seja danificado ou espremido durante a descorionação.
- Oriente o embrião em seu lado lateral e segure a pele entre a cabeça e a cauda do embrião com uma das pinças finas. Use a segunda pinça fina para beliscar a pele perto de onde a primeira pinça ainda está posicionada.
- Afaste as duas pinças uma da outra, enquanto segura a pele, criando assim uma grande abertura. Solte uma das pinças da pele e mova-a para perto de onde a segunda pinça é segurada e repita o processo de remoção da pele até que a pele seja removida nas regiões formadoras de somito.
NOTA: É fundamental remover completamente a pele ao redor dos somitos que serão explantados, caso contrário, fica difícil fazer dissecções mais finas nas etapas posteriores do protocolo. - Use uma microfaca para raspar a gema (Figura 1C - explante com a casca removida e a gema raspada) e faça uma série de incisões da seguinte forma:
- Corte o embrião com o primeiro corte anterior ao primeiro somito morfológico no embrião e o segundo corte posterior ao somito formado mais recentemente (Figura 1D). Corte mais uma vez posterior e anterior aos somitos de interesse (Figura 1E).
- Use a microfaca para raspar qualquer gema restante das regiões explantadas do embrião. Tente remover o máximo possível da gema para reduzir a possibilidade de explantes ficarem presos à microfaca. Se isso acontecer, use uma ferramenta de cílios para mover o explante de volta à solução, mantendo a microfaca imersa no meio.
- Posicione o explante dorsalmente e faça um corte ao longo do eixo anteroposterior através da notocorda, próximo aos somitos de um lado (Figura 1F, G).
- Pegue o explante sem a notocorda e corte a placa lateral do mesoderma (LPM) perto dos somitos.
NOTA: Esta etapa pode ser trocada com a etapa anterior, ou seja, funciona igualmente bem cortar o LPM primeiro, seguido de cortar a notocorda.
- Escolha o somito a ser explantado e faça dois cortes posterior e anterior ao somito, respectivamente (Figura 1H).
- Finalmente, gire o somito em 90° usando a ferramenta de cílios para visualizar a placa neural ainda presa ao somito. Faça uma fatia entre o somito e a placa neural, liberando assim um único somito ( Figura 1I ).
NOTA: A placa neural também pode ser removida imediatamente após o passo 2.6.4, ou seja, após a remoção da notocorda e do LPM e antes de dissecar um somite dos somitos vizinhos.
3. Imagem de explantes de somito único
NOTA: Aqui, o procedimento para imagens de explantes de somito único usando um microscópio de folha de luz de visão única é descrito. Como alternativa, os somitos únicos explantados também podem ser visualizados em um microscópio confocal, que está mais amplamente disponível ou mesmo em um microscópio de campo amplo, se estiver exclusivamente interessado em acompanhar a morfologia geral dos explantes.
- Prepare uma câmara de imagem da seguinte forma:
- Encha uma seringa de 1 mL com formulação de borracha de silicone. Corte uma ponta de pipeta de 200 μL a cerca de 1 cm de distância da boca e insira a seringa na ponta da pipeta.
- Enrole manualmente o suporte da membrana com uma tira de membrana de etileno propileno fluorado (FEP) pré-cortada (Figura 2A-C) e fixe a câmara de imagem no suporte da membrana, que se encaixam perfeitamente um no outro (Figura 2D).
- Adicione a formulação de borracha de silicone ao longo da interseção da câmara e da membrana e seque-a durante a noite, o que permite que a membrana grude na câmara. Remova o suporte da membrana 1 h antes do experimento de explante.
- Aquecer uma alíquota contendo 0,5 ml de agarose a 2% de baixo ponto de fusão feita com meio L-15 a 75 °C durante 10 min. Adicione 160 μL de agarose aquecida de baixo ponto de fusão ao centro da câmara e coloque um molde de imagem na agarose (Figura 2E, F) e segure-o na posição vertical até que a agarose solidifique.
- Adicione mais 160 μL de agarose aquecida de baixo ponto de fusão nas duas extremidades do molde e transfira toda a unidade para 4 ° C por 30 min.
- Adicione 600 μL de meio L-15 à câmara e remova o molde da câmara com uma pinça. Aguarde até que a câmara com meio L-15 retorne à temperatura ambiente.
- Transfira o somita explantado com uma pipeta de vidro polido pipetando para a câmara de imagem e use uma ferramenta de cílios para posicionar o explante no centro da câmara.
- Alinhe os lasers de imagem de acordo com as instruções do fabricante e transfira cuidadosamente a câmara de imagem para o microscópio equipado com uma câmara de incubação para manter os explantes a 28 °C.
- Realize uma segunda rodada de alinhamento a laser com regiões de interesse da linha transgênica, seguida pela configuração da pilha z desejada, intervalo de tempo e duração para imagens de lapso de tempo. Para este experimento, adquira 70 fatias z com espaçamento de 2 μm e intervalo de quadros de 2 min.
- Realize imagens de folha de luz de duas cores (Figura 1J) dos explantes com lasers de 561 nm (10% de potência do laser, 100 ms de exposição) e 488 nm (10% de potência do laser, 100 ms de exposição). Neste protocolo, o sinal foi coletado com uma objetiva de 25x/1,1 NA e através de filtros passa-banda de 561/25 nm e 525/50-25, respectivamente, em uma câmera sCMOS.
Resultados
Os explantes permitem a quantificação de mudanças de forma em grande escala em 3D. Para ilustrar isso, explantamos somitos quatro (N = 3) de embriões iniciais de peixe-zebra obtidos de um cruzamento entre linhagens heterozigóticas Utr::mCherry (Tg(actb2:mCherry-Hsa.UTRN); e119Tg) e H2B::GFP (Tg(h2az2a:h2az2a-GFP); kca6Tg). A utrofina é uma proteína de ligação à actina, e a linha Utr::mCherry mostra a distribuição de estruturas filamentosas de actina. Este transgene foi usado aqui como um marcador para contornos celulares. H2B::GFP é uma histona marcada com fluorescência e, consequentemente, marca a distribuição da cromatina, fornecendo uma descrição eficaz da localização e forma nuclear, bem como figuras mitóticas que destacam a divisão celular.
Avaliamos o sucesso do protocolo de explante durante a imagem. Observamos que em um somito danificado durante a dissecção, a integridade do tecido do somito é comprometida, com muitas células se dissociando e expulsando do somito explantado, e / ou muitas células morrendo, o que pode ser notado pela presença de núcleos fragmentados no canal nuclear. Os explantes bem-sucedidos permaneceram saudáveis por 4-6 h, após o que foram observadas alterações na integridade do somito com as células se dissociando do explante e morrendo.
No canal de utrofina, realizamos segmentação manual de explantes no MATLAB (R2018b) em z-slices espaçados a cada 10 μm. Desenvolvemos um algoritmo personalizado no MATLAB para segmentação, que está disponível gratuitamente para download (https://github.com/sundar07/SomSeg). No algoritmo, o arquivo, o quadro e o z-slice a serem segmentados podem ser definidos, seguidos por um prompt do usuário, que permite o desenho manual de um contorno em torno da região de interesse. Isso foi repetido para várias fatias z e plotado, que mostrou o arredondamento dos explantes ao longo do tempo em 3D (Figura 3A). Além disso, também foram obtidas informações complementares sobre a forma do tecido usando o canal nuclear. Para isso, utilizamos o Mastodon (versão 1.0.0-beta-19, https://github.com/mastodon-sc/mastodon), um plugin FIJI15 , para obter as posições dos núcleos centróides por meio da detecção de manchas. Primeiro, convertemos os arquivos tif do microscópio para o formato xml/hdf5 em FIJI, após o qual um novo projeto foi aberto usando o plug-in Mastodon. No plugin, escolhemos a opção de detecção de manchas, onde definimos uma região de interesse que cobria o explante e usamos a diferença do detector gaussiano com diâmetro de 5 μm e fator de qualidade de 25 para detecção de manchas. Em seguida, transferimos as posições dos núcleos centróides para o MATLAB e usamos uma função embutida (convhull) para obter um envoltório convexo (Figura 3B), que caracterizou a geometria do explante. Isso também mostrou arredondamento de explantes ao longo do tempo (Figura 3B). Os dados dos núcleos também permitem quantificar os movimentos das células rastreando em 3D e uma mudança no número de células ao longo do tempo. Por outro lado, o canal de utrofina permite a quantificação de mudanças nas formas celulares à medida que os explantes se tornam mais redondos. Juntos, esses parâmetros são valiosos para caracterizar as propriedades intrínsecas do material dos somitos, o que ajuda no desenvolvimento de descrições físicas eficazes das mudanças na forma da escala do tecido.
Os explantes também permitem caracterizar as tensões de contato com os tecidos vizinhos de maneira quantitativa. Para ilustrar isso, isolamos manualmente dois somitos e os colocamos próximos e observamos sua dinâmica (N = 2). Para este experimento, a orientação dos somitos em relação aos eixos do corpo in vivo não foi rastreada. Curiosamente, ao longo do tempo, os somitos explantados aderiram uns aos outros ao longo de uma superfície, enquanto as superfícies livres, ou seja, regiões distantes do local de contato, arredondaram (Figura 4). Isso sugere que as forças adesivas superam as tensões geradas pela tensão superficial nos locais de contato em explantes. Isso pode ser caracterizado pelas mudanças de forma descritas acima e pela quantificação dos ângulos de contato entre os dois tecidos ao longo do tempo em 3D. Assim, os explantes fornecem um sistema atraente para quantificar forças concorrentes em ação que levam a formas específicas de tecido, cujas implicações podem ser exploradas in vivo.
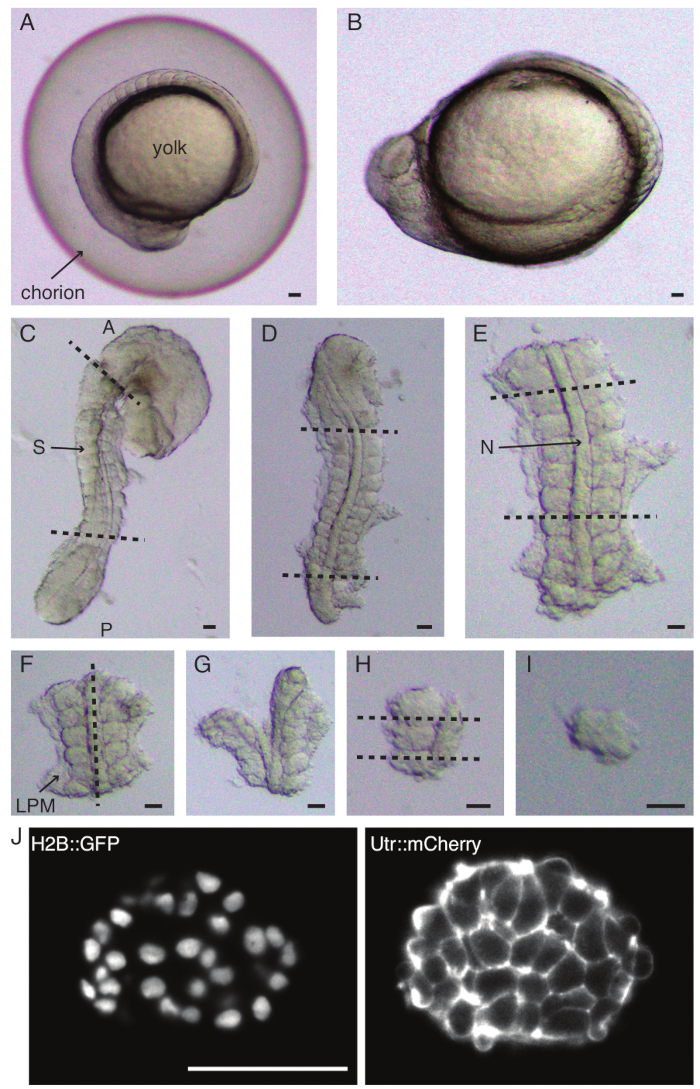
Figura 1: Preparação de explantes de somito único. O embrião de peixe-zebra (A) é primeiro descorionado (B), seguido pela remoção da pele e da gema (C). A região contendo somitos do embrião é então selecionada removendo o resto dos tecidos (D, E). As regiões ao redor do somito de interesse são então removidas em série (FH) para isolar um único somito (I), seguido por imagens de lapso de tempo usando um microscópio de folha de luz (seção z intermediária do somite mostrada aqui) (J). Abreviaturas: S = somito; N = notocorda; LPM = mesoderma da placa lateral; A = anterior; P = posterior. Linhas tracejadas indicam posições de corte. Barra de escala = 50 μm em todos os painéis. Clique aqui para ver uma versão maior desta figura.
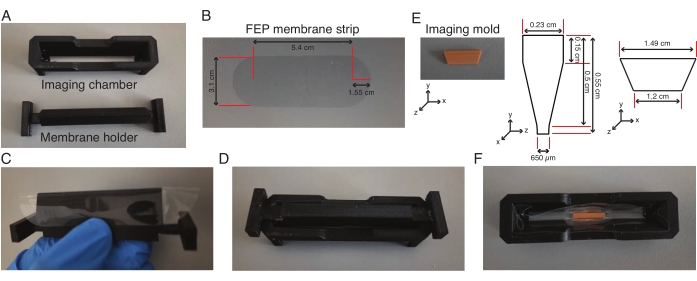
Figura 2: Montagem da câmara de imagem de folha de luz. (A-D) Uma tira de membrana FEP (dimensões em (B)) é enrolada em torno do suporte da membrana, encaixada na câmara de imagem e a membrana é colada à câmara de imagem. (E-F) No dia seguinte, o suporte da membrana é removido e um molde de imagem (dimensões em (E)) é colocado no centro da câmara de imagem em agarose de baixo ponto de fusão. Toda a unidade é mantida a 4 °C por 30 min, após o que o molde é removido e a câmara com a calha é usada para a imagem dos explantes. Clique aqui para ver uma versão maior desta figura.
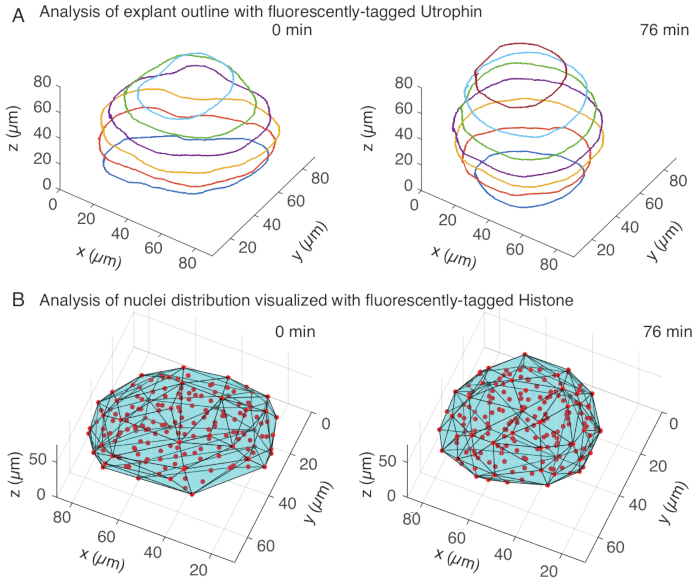
Figura 3: Análise 3D das formas dos tecidos. (A) Os contornos celulares foram visualizados com utrofina marcada com fluorescência (Utr::mCherry) e os núcleos foram visualizados com histona marcada com fluorescência (H2B::GFP). Os contornos do explante foram segmentados manualmente em várias profundidades usando o MATLAB e mostrados aqui. (B) Núcleos (vermelho) foram detectados no mesmo explante usando Mastodon, um plugin FIJI, e submetidos a um casco convexo (ciano), que informa sobre a geometria do explante. Nota: o arredondamento de explantes é evidente em ambas as análises. Clique aqui para ver uma versão maior desta figura.
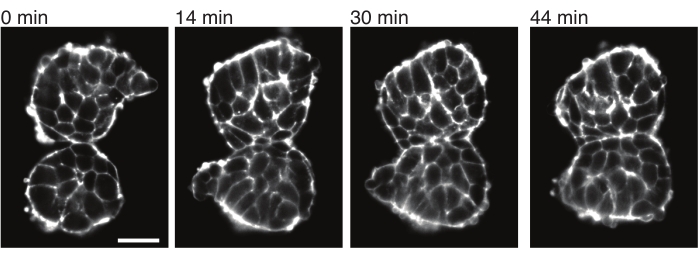
Figura 4: Somitos explantados aderem uns aos outros. Dois somitos isolados de embriões e colocados manualmente próximos tendem a aderir ao longo do tempo (N = 2). Várias fatias z com um intervalo de quadro de 2 min foram adquiridas usando um microscópio de folha de luz e seções intermediárias de somitos de pontos de tempo selecionados são mostradas aqui. Os contornos celulares foram visualizados com utrofina marcada com fluorescência (Utr::mCherry). Barra de escala = 25 μm. Clique aqui para ver uma versão maior desta figura.
Discussão
O campo da somitogênese tem sido dominado por estudos sobre o papel do relógio de segmentação na definição dos comprimentos dos segmentos durante o desenvolvimento embrionário. No entanto, é igualmente importante considerar o papel da mecânica dos tecidos na determinação das morfologias finais dos somitos. O protocolo aqui descrito permite o explante de somitos únicos, cujas propriedades físicas intrínsecas podem ser estudadas isoladamente do embrião. No entanto, a preparação manual limita o número de somitos que normalmente são preparados a quatro a seis por sessão de imagem. Como o protocolo envolve dissecações finas cuidadosas de vários tecidos em embriões sem o uso de enzimas para facilitar a remoção do tecido, pode levar algumas semanas de prática para dominar o processo de dissecação. Em nossas mãos, as etapas críticas do protocolo envolvem a remoção cuidadosa da pele e da gema dos explantes, o que evita que os explantes em diferentes estágios do protocolo grudem nas ferramentas usadas, o que, por sua vez, permite dissecções mais fáceis para remover em série os tecidos ao redor de um somito.
O protocolo de dissecção pode ser interrompido em etapas intermediárias para obter somitos ligados a apenas um dos tecidos circundantes ou para obter explantes de grupos de somitos. Isso fornece um método poderoso para remover tecidos em série e estudar o impacto dos tecidos vizinhos na facilitação de mudanças de forma em somitos ou nos tecidos vizinhos. Por outro lado, usando este protocolo, os tecidos explantados individuais podem aderir e se auto-organizar mecanicamente, como demonstrado pela colocação de dois somitos explantados próximos.
Os explantes preparados por este método não requerem quaisquer ingredientes adicionados no tampão ou restrições para garantir a sobrevivência por períodos de tempo até várias horas. No entanto, pode-se imaginar cultivá-los em hidrogéis e seguir sua dinâmica na presença de restrições externas, o que poderia servir de modelo para as tensões de contato que os somitos encontram in vivo. Além disso, os explantes permitem sondar diretamente as propriedades de seus materiais por meio de microscopia de força atômica, aspiração de pipetas ou usando ferramentas micro-robóticas16. Finalmente, esperamos que este método possa ser facilmente adaptado para cultivar e estudar outros tecidos de desenvolvimento em estágios semelhantes, como a notocorda, a placa neural e o mesoderma da placa lateral.
Divulgações
Os autores declaram não haver interesses conflitantes.
Agradecimentos
Agradecemos aos membros do laboratório Oates pelos comentários sobre o protocolo e as instalações pesqueiras da École polytechnique fédérale de Lausanne (EPFL). Em particular, agradecemos a Laurel Ann Rohde por dicas valiosas sobre remoção de pele e gema no protocolo de dissecção; Arianne Bercowsky Rama pela construção de um pipeline eficiente para o processamento de conjuntos de dados de folha de luz por meio do Mastodon; Jean-Yves Tinevez pela construção do software de código aberto Mastodon; Marko Popović por dicas sobre análise de dados; Chloé Jollivet, Guillaume Valentin e Florian Lang pelo amplo apoio nas instalações de pesca; Petr Strnad e Andrea Boni pela construção do microscópio de folha de luz e por dicas sobre imagens de folha de luz. Este trabalho foi apoiado pela EPFL e a SRN foi apoiada por uma bolsa de pós-doutorado do Programa de Ciência de Fronteira Humana de Longo Prazo (LT000078/2016).
Materiais
| Name | Company | Catalog Number | Comments |
| Agarose | Sigma | 9012-36-6 | For coating bottom of petri dishes |
| Agarose, low gelling temperature | Sigma | 39346-81-1 | For preparing Viventis imaging chamber |
| Camera | Andor | Andor Zyla 4.2 Plus | For image acquisition in the light-sheet microscope |
| Detection objective | Nikon | Nikon CFI75 Apo LWD 25x/1.1 NA | For imaging explants |
| FEP membrane strip | Lohmann Technologies UK Ltd | Dupont FEP Fluorocarbon film, 200A | For preparing Viventis imaging chamber |
| Fine forceps | Dumont | Dumont 5SF 11252-00 | For removal of skin of embryos |
| Forceps | Dumont | Dumont 55 | For dechorionating embryos |
| Leibovitz's L-15 medium | Gibco | 21083-027 | Explant culture medium |
| Light-sheet microscope | Viventis | LS1 live | For imaging explants |
| Micro knife | Fine Science Tools | 10318-14 | For making incisions in embryos |
| Silicone rubber formulation | Wacker Chemie AG | Silpuran 4200 | For preparing Viventis imaging chamber |
Referências
- Oates, A. C., Morelli, L. G., Ares, S. Patterning embryos with oscillations: structure, function and dynamics of the vertebrate segmentation clock. Development. 139 (4), Cambridge, England. 625-639 (2012).
- Pourquié, O. Segmentation of the vertebrate spine: From clock to scoliosis. Cell. 145 (5), 650-663 (2011).
- Naganathan, S. R., Oates, A. C. Patterning and mechanics of somite boundaries in zebrafish embryos. Seminars in Cell & Developmental Biology. 107, 170-178 (2020).
- Hubaud, A., Regev, I., Mahadevan, L., Pourquié, O. Excitable Dynamics and Yap-Dependent Mechanical Cues Drive the Segmentation Clock. Cell. 171 (3), 668-682 (2017).
- Dias, A. S., de Almeida, I., Belmonte, J. M., Glazier, J. A., Stern, C. D. Somites without a clock. Science. 343 (6172), New York, N.Y. 791-795 (2014).
- Nelemans, B. K. A., Schmitz, M., Tahir, H., Merks, R. M., Smit, T. H. Somite Division and New Boundary Formation by Mechanical Strain. iScience. 23 (4), 100976(2020).
- Grima, R., Schnell, S. Can tissue surface tension drive somite formation. Developmental Biology. 307 (2), 248-257 (2007).
- Naganathan, S. R., Popovic, M., Oates, A. C. Left–right symmetry of zebrafish embryos requires somite surface tension. Nature. 605, 516-521 (2022).
- Campàs, O. A toolbox to explore the mechanics of living embryonic tissues. Seminars in Cell & Developmental Biology. 55, 119-130 (2016).
- Langenberg, T., Brand, M., Cooper, M. S. Imaging brain development and organogenesis in zebrafish using immobilized embryonic explants. Developmental Dynamics: An Official Publication of The American Association of Anatomists. 228 (3), 464-474 (2003).
- Henry, C. A., Poage, C. T., McCarthy, M. B., Campos-Ortega, J., Cooper, M. S. Regionally autonomous segmentation within zebrafish presomitic mesoderm. Zebrafish. 2 (1), 7-18 (2005).
- Picker, A., Roellig, D., Pourquié, O., Oates, A. C., Brand, M. Tissue micromanipulation in zebrafish embryos. Methods in Molecular Biology. 546, Clifton, N.J. 153-172 (2009).
- Manning, A. J., Kimelman, D. Tbx16 and Msgn1 are required to establish directional cell migration of zebrafish mesodermal progenitors. Developmental Biology. 406 (2), 172-185 (2015).
- Simsek, M. F., Özbudak, E. M. A 3-D Tail Explant Culture to Study Vertebrate Segmentation in Zebrafish. Journal of Visualized Experiments:JoVE. (172), e61981(2021).
- Schindelin, J., et al. Fiji: an open-source platform for biological-image analysis. Nature Methods. 9 (7), 676-682 (2012).
- Özkale, B., et al. Modular soft robotic microdevices for dexterous biomanipulation. Lab on a Chip. 19 (5), 778-788 (2019).
Reimpressões e Permissões
Solicitar permissão para reutilizar o texto ou figuras deste artigo JoVE
Solicitar PermissãoExplore Mais Artigos
This article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Todos os direitos reservados