Method Article
Preparazione di espianti di somite singolo da embrioni di zebrafish
In questo articolo
Riepilogo
Presentiamo un protocollo per isolare singoli somiti da embrioni di zebrafish, la cui dinamica può essere seguita in coltura per diverse ore mediante microscopia time-lapse a fluorescenza, fornendo così una metodologia per quantificare i cambiamenti di forma su scala tissutale alla risoluzione di una singola cellula.
Abstract
L'asse corporeo degli embrioni di vertebrati è periodicamente suddiviso in unità multicellulari 3D chiamate somiti. Mentre le oscillazioni genetiche e i premodelli molecolari determinano la scala di lunghezza iniziale dei somiti, i processi meccanici sono stati implicati nel determinare la loro dimensione e forma finale. Per comprendere meglio le proprietà intrinseche dei somiti, è stato sviluppato un metodo per coltivare l'espiante di un singolo somite da embrioni di zebrafish. I singoli somiti vengono isolati rimuovendo prima la pelle degli embrioni, seguita dalla rimozione del tuorlo e dall'escissione sequenziale dei tessuti vicini. Utilizzando embrioni transgenici, la distribuzione di varie strutture subcellulari può essere osservata mediante microscopia time-lapse fluorescente. La dinamica dei somiti espiantati può essere seguita per diverse ore, fornendo così un quadro sperimentale per lo studio dei cambiamenti di forma su scala tissutale alla risoluzione di una singola cellula. Questo approccio consente la manipolazione meccanica diretta dei somiti, consentendo la dissezione delle proprietà del materiale del tessuto. Infine, la tecnica qui descritta può essere facilmente estesa per l'espianto di altri tessuti come la notocorda, la placca neurale e il mesoderma della placca laterale.
Introduzione
Gran parte del sistema muscolo-scheletrico adulto dei vertebrati emerge dai somiti embrionali, che si formano in modo periodico e ritmico lungo l'asse corporeo degli embrioni 1,2. I somiti sono unità multicellulari tridimensionali (3D) tipicamente costituite da un nucleo interno di cellule mesenchimali e da uno strato epiteliale periferico circondato da una matrice extracellulare ricca di fibronectina3. La morfologia dei somiti, cioè le loro dimensioni e forma, è in parte determinata dall'orologio di segmentazione e dai prepattern molecolari a valle. Tuttavia, nell'ultimo decennio, è emerso che anche i segnali e le forze meccaniche svolgono un ruolo nella regolazione dell'orologio di segmentazione4, oltre a facilitare la formazione di somite 5,6,7 e garantire una maggiore precisione delle lunghezze dei somiti dopo la formazione iniziale di somite8.
La meccanica dei tessuti può essere studiata direttamente in vivo con la disponibilità di nuovi strumenti9, tuttavia, per ottenere un quadro completo alla base dei processi fisici, è necessario studiare contemporaneamente le proprietà intrinseche dei materiali dei tessuti. Il protocollo qui descritto fornisce un approccio semplice per preparare singoli somiti, le cui proprietà fisiche da scala cellulare a scala tissutale possono essere studiate isolatamente dall'embrione. Sebbene esistano diversi protocolli per la preparazione di espianti in fasi di sviluppo simili 10,11,12,13,14, per quanto ne sappiamo, questo è il primo protocollo che descrive l'isolamento di singoli somiti. Il protocollo è semplice da implementare e richiede solo l'attrezzatura di base disponibile nella maggior parte dei laboratori di zebrafish che lavorano con gli embrioni.
Per aiutare a distinguere il ruolo della meccanica nella formazione morfologica del somite, è stato sviluppato un metodo per coltivare l'espiante di un singolo somite da embrioni di zebrafish, che può essere utilizzato per sondare le proprietà materiali intrinseche dei somiti.
Protocollo
Questo protocollo prevede l'uso di embrioni di vertebrati vivi di età inferiore a 1 giorno dopo la fecondazione. Tutti gli esperimenti sono stati condotti utilizzando embrioni derivati da adulti che si accoppiano liberamente e sono quindi coperti dalla licenza generale per esperimenti sugli animali dell'EPFL rilasciata dal Service de la Consommation et des Affaires Vétérinaires del cantone di Vaud - Svizzera (numero di autorizzazione VD-H23).
1. Prima della dissezione
- Ottenere embrioni da un incrocio di linee transgeniche eterozigoti di interesse. Per l'espianto di due o tre somiti, in genere sono necessari solo pochi embrioni. In questo protocollo, un embrione è stato utilizzato per l'espianto di un somite.
- Preparare 1 tampone E3 da due stock 50x: il grezzo 1 contiene 0,458 mM Na2HPO4, 0,042 mM KH2PO4, 4,084 mM NaCl, 0,128 mM KCl e il grezzo 2 contiene 0,33 mM CaCl2, 0,33 mM MgSO4. Preparare 500 mL di 1 tampone E3 mescolando 480 mL di acqua distillata con 10 mL ciascuno dei due stock 50x.
- In caso di dissezione lo stesso giorno, allevare gli embrioni in 25 mL di terreno E3 in una piastra di Petri a 33 °C fino allo stadio di tre somi. Se si disseziona la mattina seguente, allevare gli embrioni in terreno E3 a 28 °C fino allo stadio di scudo prima di trasferirli a 19 °C per l'incubazione notturna.
- Per identificare lo stadio di scudo o i diversi stadi di somite, utilizzare uno stereomicroscopio da laboratorio standard con un intervallo di ingrandimento regolabile da circa 0,67x a 4,5x.
- Assemblare gli strumenti e i reagenti necessari per la dissezione.
- Aliquotare il terreno L-15 di Leibovitz in provette da 50 mL e conservare in frigorifero. Si consiglia di utilizzare la stessa aliquota di frigo per un massimo di due giri.
- Preparare 50 mL di agarosio al 2% in terreno L-15 utilizzando un agitatore magnetico e mantenendo la temperatura a 85 °C. Una volta sciolta, conservare la soluzione a temperatura ambiente.
- Preparare 25 mL di agarosio a basso punto di fusione al 2% in terreno L-15 utilizzando un agitatore magnetico e mantenendo la temperatura a 85 °C. Una volta disciolta, aliquotare la soluzione in provette da 1,5 mL e conservare a 4 °C. Questo verrà utilizzato per preparare la camera di imaging.
- Versare l'agarosio fuso al 2% in una piastra di Petri da 20 mm fino a riempire un terzo della profondità della piastra e lasciare solidificare la soluzione di agarosio. Le dissezioni saranno eseguite in queste piastre di Petri. Preparare le piastre di Petri con rivestimento di agarosio con largo anticipo e conservare a 4 °C.
- Assemblare i seguenti strumenti: pipetta Pasteur per il trasferimento degli embrioni, un paio di pinze per dechorionare gli embrioni, un paio di pinze fini per la rimozione della pelle degli embrioni, un micro coltello per praticare incisioni negli embrioni, pipetta in vetro lucidato a fuoco per il trasferimento degli espianti nella camera di imaging.
- Sterilizzare la pipetta di vetro in etanolo al 100% per 15 minuti e risciacquare due volte pipettando 5 ml di terreno L-15 su e giù.
- Prepara uno strumento per ciglia attaccando una ciglia o dei peli dal sopracciglio a un capillare di vetro.
- Utilizzare uno stereoscopio fluorescente con un intervallo di ingrandimento regolabile da circa 0,63x a 6,3x e filtri GFP e RFP standard per selezionare gli embrioni transgenici positivi e trasferirli in una piastra di Petri separata con terreno E3.
2. Preparazione dell'espianto a singolo somite
- Trasferire 2 o 3 embrioni in un piatto rivestito di agarosio (realizzato nella fase 1.5.4) riempito con 10 mL di terreno L-15. Assicurarsi che la capsula insieme al terreno L-15 sia preriscaldata a 28 °C in un'incubatrice per 30 minuti prima di trasferire gli embrioni.
- Decorionato con attenzione (Figura 1 A,B) un embrione con un paio di pinze. Tenere il corion con una delle pinze e utilizzare la seconda pinza per pizzicare e tirare il corion vicino al punto in cui è posizionata la prima pinza.
- Ripeti questo passaggio ancora una volta per aprire completamente il corion, dopodiché l'embrione può essere espulso delicatamente. Assicurarsi che l'embrione non venga danneggiato o schiacciato durante la decorionazione.
- Orientare l'embrione sul lato laterale e tenerlo stretto alla pelle tra la testa e la coda dell'embrione con una delle pinze sottili. Usa la seconda pinza fine per pizzicare la pelle vicino a dove è ancora posizionata la prima pinza.
- Allontanare le due pinze l'una dall'altra, tenendosi aggrappate alla pelle, creando così un'ampia apertura. Rilascia una delle pinze dalla pelle e spostala vicino al punto in cui è tenuta la seconda pinza e ripeti il processo di rimozione della pelle fino a quando la pelle non viene rimossa nelle regioni che formano somiti.
NOTA: È fondamentale rimuovere completamente la pelle che circonda i somiti che verranno espiantati, altrimenti diventa difficile eseguire dissezioni più fini nelle fasi successive del protocollo. - Utilizzare un micro coltello per raschiare via il tuorlo (Figura 1C - espiantare con la pelle rimossa e il tuorlo raschiato via) e praticare una serie di incisioni come segue:
- Tagliare l'embrione con il primo taglio anteriore al primo somite morfologico nell'embrione e il secondo taglio posteriormente al somite di più recente formazione (Figura 1D). Affettare ancora una volta posteriormente e anteriormente ai somiti di interesse (Figura 1E).
- Usa il micro coltello per raschiare via il tuorlo rimasto dalle regioni espiantate dell'embrione. Cerca di rimuovere quanto più tuorlo possibile per ridurre la possibilità che le espianti si attacchino al micro coltello. In questo caso, utilizzare uno strumento per ciglia per riportare l'espiante in soluzione mantenendo il micro coltello immerso nel terreno.
- Posizionare l'espiante dorsalmente e fare una fetta lungo l'asse anteroposteriore attraverso la notocorda, vicino ai somiti su un lato (Figura 1F, G).
- Prendere l'espiante senza la notocorda e tagliare il mesoderma della placca laterale (LPM) vicino ai somiti.
NOTA: Questo passaggio può essere scambiato con il passaggio precedente, ad esempio, funziona altrettanto bene per tagliare prima l'LPM seguito dal taglio della notocorda.
- Scegliere il somite da espiantare e fare due fette rispettivamente posteriormente e anteriormente al somite (Figura 1H).
- Infine, ruota il somite di 90° usando lo strumento ciglia per visualizzare la piastra neurale ancora attaccata al somite. Fare una fetta tra il somite e la placca neurale, rilasciando così un singolo somite (Figura 1I).
NOTA: La placca neurale può anche essere rimossa immediatamente dopo il passaggio 2.6.4, cioè dopo la rimozione della notocorda e dell'LPM e prima di sezionare un somite dai somiti vicini.
3. Imaging di espianti di singolo somite
NOTA: Qui viene descritta la procedura per l'imaging di espianti di somite singolo utilizzando un microscopio a foglio di luce a vista singola. In alternativa, i singoli somiti espiantati possono anche essere ripresi in un microscopio confocale, che è più ampiamente disponibile, o anche in un microscopio a campo largo se interessati esclusivamente a seguire la morfologia complessiva degli espianti.
- Preparare una camera di imaging come segue:
- Riempire una siringa da 1 ml con una formulazione di gomma siliconica. Tagliare un puntale per pipetta da 200 μl a circa 1 cm di distanza dalla bocca e inserire la siringa nel puntale della pipetta.
- Avvolgere manualmente il supporto della membrana con una striscia di membrana in etilene propilene fluorurato (FEP) pretagliata (Figura 2A-C) e fissare la camera di imaging sul supporto della membrana, che si incastrano perfettamente l'una nell'altra (Figura 2D).
- Aggiungere una formulazione di gomma siliconica lungo l'intersezione della camera e della membrana e asciugarla durante la notte, il che consente alla membrana di aderire alla camera. Rimuovere il supporto della membrana 1 ora prima dell'esperimento di espianto.
- Riscaldare un'aliquota contenente 0,5 mL di agarosio a basso punto di fusione al 2% a base di terreno L-15 a 75 °C per 10 minuti. Aggiungere 160 μl di agarosio riscaldato a basso punto di fusione al centro della camera e posizionare uno stampo per immagini nell'agarosio (Figura 2E, F) e tenerlo in posizione verticale fino a quando l'agarosio non si solidifica.
- Aggiungere altri 160 μl di agarosio riscaldato a basso punto di fusione alle due estremità dello stampo e trasferire l'intera unità a 4 °C per 30 minuti.
- Aggiungere 600 μl di terreno L-15 alla camera e rimuovere lo stampo dalla camera con una pinza. Attendere che la camera con il mezzo L-15 ritorni a temperatura ambiente.
- Trasferire il somite espiantato con una pipetta di vetro lucidato pipettandolo nella camera di imaging e utilizzare uno strumento per ciglia per posizionare l'espiante al centro della camera.
- Allineare i laser per imaging secondo le istruzioni del produttore e trasferire con cura la camera di imaging al microscopio dotato di camera di incubazione per mantenere gli espianti a 28 °C.
- Eseguire un secondo ciclo di allineamento laser con le regioni di interesse dalla linea transgenica, seguito dall'impostazione dello z-stack, dell'intervallo di tempo e della durata desiderati per l'imaging time-lapse. Per questo esperimento, acquisire 70 z-slice con una spaziatura di 2 μm e un intervallo di fotogrammi di 2 minuti.
- Eseguire l'imaging a foglio luminoso bicolore (Figura 1J) degli espianti con laser a 561 nm (10% di potenza laser, 100 ms di esposizione) e 488 nm (10% di potenza laser, 100 ms di esposizione). In questo protocollo, il segnale è stato raccolto con un obiettivo 25x/1.1 NA e attraverso filtri passa-banda 561/25 nm e 525/50-25 rispettivamente su una fotocamera sCMOS.
Risultati
Gli espianti consentono di quantificare i cambiamenti di forma su larga scala in 3D. Per illustrare ciò, abbiamo espiantato somite quattro (N = 3) da embrioni precoci di zebrafish ottenuti da un incrocio tra linee eterozigoti Utr::mCherry (Tg(actb2:mCherry-Hsa.UTRN); e119Tg) e H2B::GFP (Tg(h2az2a:h2az2a-GFP); kca6Tg). L'utrofina è una proteina legante l'actina e la linea Utr::mCherry mostra la distribuzione delle strutture filamentose dell'actina. Questo transgene è stato utilizzato qui come marcatore per i contorni cellulari. H2B::GFP è un istone marcato in fluorescenza e di conseguenza segna la distribuzione della cromatina, fornendo una descrizione efficace della posizione e della forma nucleare, nonché figure mitotiche che evidenziano la divisione cellulare.
Abbiamo valutato il successo del protocollo di espianto durante l'imaging. Abbiamo osservato che in un somite danneggiato durante la dissezione l'integrità del tessuto somite è compromessa, con molte cellule che si dissociano ed estrudono dal somite espiantato e/o molte cellule che muoiono, il che può essere notato dalla presenza di nuclei frammentati nel canale nucleare. Gli espianti riusciti sono rimasti sani per 4-6 ore, dopodiché sono stati osservati cambiamenti nell'integrità del somite con le cellule che si dissociavano dall'espiante e morivano.
Nel canale dell'utrofina, abbiamo eseguito la segmentazione manuale degli espianti in MATLAB (R2018b) su z-slice distanziate ogni 10 μm. Abbiamo sviluppato un algoritmo personalizzato in MATLAB per la segmentazione, che è disponibile gratuitamente per il download (https://github.com/sundar07/SomSeg). Nell'algoritmo, è possibile impostare il file, il fotogramma e la z-slice da segmentare, seguiti da un prompt dell'utente, che consente di disegnare manualmente un contorno attorno alla regione di interesse. Questo è stato ripetuto per più z-slice e tracciato, che ha mostrato l'arrotondamento degli espianti nel tempo in 3D (Figura 3A). Inoltre, sono state ottenute anche informazioni complementari sulla forma dei tessuti utilizzando il canale nucleare. Per questo, abbiamo utilizzato Mastodon (versione 1.0.0-beta-19, https://github.com/mastodon-sc/mastodon), un plugin FIJI15 , per ottenere le posizioni del centroide dei nuclei attraverso il rilevamento dei punti. Per prima cosa abbiamo convertito i file tif dal microscopio al formato xml/hdf5 in FIJI, dopodiché è stato aperto un nuovo progetto utilizzando il plugin Mastodon. Nel plugin, abbiamo scelto l'opzione di rilevamento spot, in cui abbiamo definito una regione di interesse che copriva l'espianto e abbiamo utilizzato la differenza del rivelatore gaussiano con un diametro di 5 μm e un fattore di qualità di 25 per il rilevamento dei spot. Abbiamo quindi trasferito le posizioni del centroide dei nuclei a MATLAB e utilizzato una funzione incorporata (convhull) per ottenere un guscio convesso (Figura 3B), che ha caratterizzato la geometria dell'espianto. Questo ha mostrato in modo simile l'arrotondamento degli espianti nel tempo (Figura 3B). I dati dei nuclei consentono inoltre di quantificare i movimenti cellulari mediante il tracciamento in 3D e una variazione del numero di cellule nel tempo. D'altra parte, il canale dell'utrofina consente la quantificazione dei cambiamenti nelle forme cellulari man mano che gli espianti diventano più rotondi. Insieme, questi parametri sono preziosi per caratterizzare le proprietà intrinseche dei materiali dei somiti, che aiutano a sviluppare descrizioni fisiche efficaci dei cambiamenti di forma su scala tissutale.
Gli espianti consentono inoltre di caratterizzare in modo quantitativo gli stress da contatto con i tessuti vicini. Per illustrare ciò, abbiamo isolato manualmente due somiti e li abbiamo posizionati in stretta vicinanza e abbiamo osservato la loro dinamica (N = 2). Per questo esperimento, l'orientamento dei somiti rispetto agli assi del corpo in vivo non è stato tracciato. È interessante notare che, nel tempo, i somiti espiantati hanno aderito l'uno all'altro lungo una superficie, mentre le superfici libere, cioè le regioni lontane dal sito di contatto, si sono arrotondate (Figura 4). Ciò suggerisce che le forze adesive superano le sollecitazioni generate dalla tensione superficiale nei siti di contatto negli espianti. Questo può essere ulteriormente caratterizzato seguendo i cambiamenti di forma come descritto sopra e quantificando gli angoli di contatto tra i due tessuti nel tempo in 3D. Pertanto, gli espianti forniscono un sistema interessante per quantificare le forze concorrenti in azione che portano a specifiche forme di tessuto, le cui implicazioni possono poi essere esplorate in vivo.
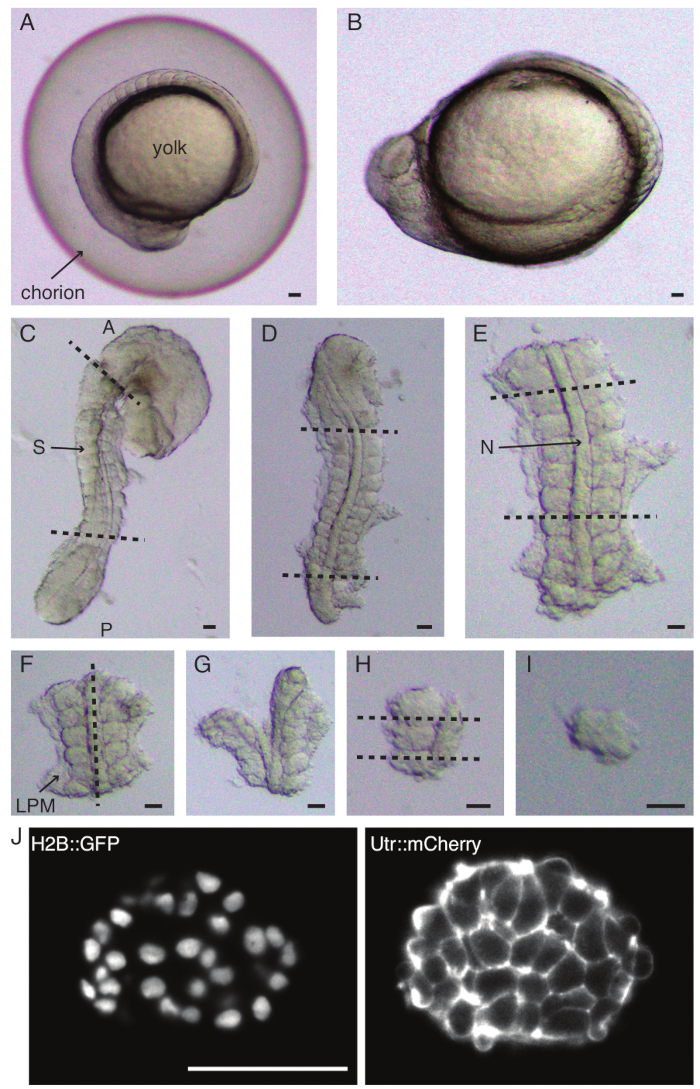
Figura 1: Preparazione di espianti di singolo somite. L'embrione di pesce zebra (A) viene prima dechorionato (B), seguito dalla rimozione della pelle e del tuorlo (C). La regione contenente somite dell'embrione viene quindi selezionata rimuovendo il resto dei tessuti (D, E). Le regioni intorno al somite di interesse vengono quindi rimosse in serie (F-H) per isolare infine un singolo somite (I), seguito da un'imaging time-lapse utilizzando un microscopio a foglio di luce (sezione z centrale del somite mostrata qui) (J). Abbreviazioni: S = somite; N = notocorda; LPM = mesoderma a placche laterali; A = anteriore; P = posteriore. Le linee tratteggiate indicano le posizioni di taglio. Barra di scala = 50 μm in tutti i pannelli. Clicca qui per visualizzare una versione più grande di questa figura.
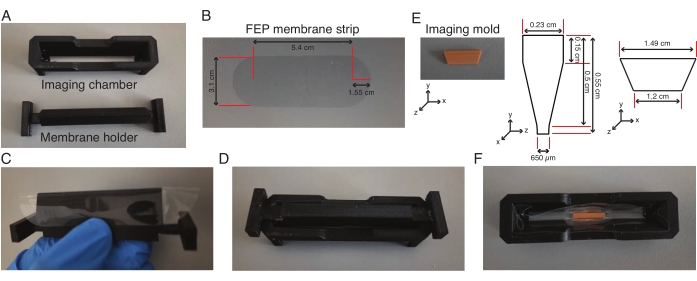
Figura 2: Assemblaggio della camera di imaging a foglio di luce. (A-D) Una striscia di membrana FEP (dimensioni in (B)) è avvolta attorno al supporto della membrana, montata sulla camera di imaging e la membrana è incollata alla camera di imaging. (E-F) Il giorno seguente, il supporto della membrana viene rimosso e uno stampo per immagini (dimensioni in (E)) viene posizionato al centro della camera di imaging in agarosio a basso punto di fusione. L'intera unità viene mantenuta a 4 °C per 30 minuti, dopodiché lo stampo viene rimosso e la camera con il trogolo viene utilizzata per l'imaging degli espianti. Clicca qui per visualizzare una versione più grande di questa figura.
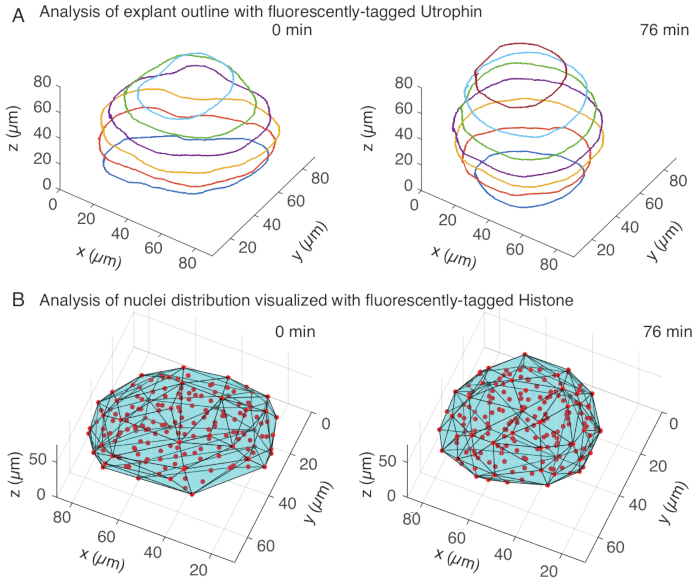
Figura 3: Analisi 3D delle forme dei tessuti. (A) I contorni delle cellule sono stati visualizzati con utrofina marcata in fluorescenza (Utr::mCherry) e i nuclei sono stati visualizzati con istone marcato in fluorescenza (H2B::GFP). I contorni dell'espianto sono stati segmentati manualmente a più profondità utilizzando MATLAB e mostrati qui. (B) I nuclei (rosso) sono stati rilevati nello stesso espiante utilizzando Mastodon, un plug-in FIJI, e sottoposti a un guscio convesso (ciano), che informa sulla geometria dell'espianto. Si noti che l'arrotondamento degli espianti è evidente in entrambe le analisi. Clicca qui per visualizzare una versione più grande di questa figura.
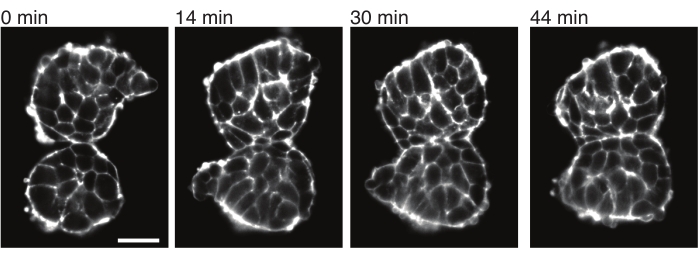
Figura 4: I somiti espiantati aderiscono l'uno all'altro. Due somiti isolati da embrioni e posti manualmente in stretta vicinanza tendono ad aderire nel tempo (N = 2). Più z-slice con un intervallo di fotogrammi di 2 minuti sono state acquisite utilizzando un microscopio a foglio luminoso e le sezioni centrali dei somiti provenienti da punti temporali selezionati sono mostrate qui. I contorni delle cellule sono stati visualizzati con Utrophin (Utr::mCherry) marcata in modo fluorescente. Barra della scala = 25 μm. Fare clic qui per visualizzare una versione più grande di questa figura.
Discussione
Il campo della somitogenesi è stato dominato dagli studi sul ruolo dell'orologio di segmentazione nell'impostazione della lunghezza dei segmenti durante lo sviluppo dell'embrione. Tuttavia, è altrettanto importante considerare il ruolo della meccanica tissutale nel determinare le morfologie finali del somite. Il protocollo qui descritto consente l'espianto di singoli somiti, le cui proprietà fisiche intrinseche possono essere studiate isolatamente dall'embrione. Tuttavia, la preparazione manuale limita il numero di somiti che vengono tipicamente preparati a quattro o sei per sessione di imaging. Poiché il protocollo prevede un'attenta dissezione fine di diversi tessuti negli embrioni senza l'uso di enzimi per facilitare la rimozione dei tessuti, potrebbero essere necessarie alcune settimane di pratica per padroneggiare il processo di dissezione. Nelle nostre mani, le fasi critiche del protocollo comportano un'attenta rimozione della pelle e del tuorlo dagli espianti, che impedisce agli espianti nelle diverse fasi del protocollo di attaccarsi agli strumenti utilizzati, il che a sua volta consente dissezioni più facili per rimuovere in serie i tessuti che circondano un somite.
Il protocollo di dissezione può essere interrotto in passaggi intermedi per ottenere somiti attaccati a uno solo dei tessuti circostanti o per ottenere espianti di gruppi di somiti. Ciò fornisce un metodo potente per spogliare in serie i tessuti e studiare l'impatto dei tessuti vicini nel facilitare i cambiamenti di forma nei somiti o nei tessuti vicini. D'altra parte, utilizzando questo protocollo, i singoli tessuti espiantati possono essere lasciati aderire e auto-organizzarsi meccanicamente, come dimostrato posizionando due somiti espiantati nelle immediate vicinanze.
Gli espianti preparati con questo metodo non richiedono l'aggiunta di ingredienti nel tampone o vincoli per garantire la sopravvivenza per periodi di tempo fino a diverse ore. Tuttavia, si può immaginare di coltivarli in idrogel e seguire le loro dinamiche in presenza di vincoli esterni, che potrebbero servire da modello per gli stress da contatto che i somiti incontrano in vivo. Inoltre, gli espianti consentono di sondare direttamente le proprietà del loro materiale attraverso la microscopia a forza atomica, l'aspirazione di pipette o l'utilizzo di strumenti micro-robotici16. Infine, ci aspettiamo che questo metodo possa essere facilmente adattato alla coltura e allo studio di altri tessuti dello sviluppo in stadi simili come la notocorda, la placca neurale e il mesoderma della placca laterale.
Divulgazioni
Gli autori dichiarano di non avere interessi concorrenti.
Riconoscimenti
Ringraziamo i membri del laboratorio Oates per i commenti sul protocollo e sulla struttura ittica dell'École polytechnique fédérale de Lausanne (EPFL). In particolare, ringraziamo Laurel Ann Rohde per i preziosi consigli sulla rimozione della pelle e del tuorlo nel protocollo di dissezione; Arianne Bercowsky Rama per la costruzione di una pipeline efficiente per l'elaborazione di set di dati light-sheet attraverso Mastodon; Jean-Yves Tinevez per la creazione del software open-source Mastodon; Marko Popović per suggerimenti sull'analisi dei dati; Chloé Jollivet, Guillaume Valentin e Florian Lang per l'ampio supporto nella struttura ittica; Petr Strnad e Andrea Boni per la costruzione del microscopio a foglio di luce e per i suggerimenti sull'imaging a foglio di luce. Questo lavoro è stato sostenuto dall'EPFL e dalla S.R.N. è stata sostenuta da una borsa di studio post-dottorato del Long-Term Human Frontier Science Program (LT000078/2016).
Materiali
| Name | Company | Catalog Number | Comments |
| Agarose | Sigma | 9012-36-6 | For coating bottom of petri dishes |
| Agarose, low gelling temperature | Sigma | 39346-81-1 | For preparing Viventis imaging chamber |
| Camera | Andor | Andor Zyla 4.2 Plus | For image acquisition in the light-sheet microscope |
| Detection objective | Nikon | Nikon CFI75 Apo LWD 25x/1.1 NA | For imaging explants |
| FEP membrane strip | Lohmann Technologies UK Ltd | Dupont FEP Fluorocarbon film, 200A | For preparing Viventis imaging chamber |
| Fine forceps | Dumont | Dumont 5SF 11252-00 | For removal of skin of embryos |
| Forceps | Dumont | Dumont 55 | For dechorionating embryos |
| Leibovitz's L-15 medium | Gibco | 21083-027 | Explant culture medium |
| Light-sheet microscope | Viventis | LS1 live | For imaging explants |
| Micro knife | Fine Science Tools | 10318-14 | For making incisions in embryos |
| Silicone rubber formulation | Wacker Chemie AG | Silpuran 4200 | For preparing Viventis imaging chamber |
Riferimenti
- Oates, A. C., Morelli, L. G., Ares, S. Patterning embryos with oscillations: structure, function and dynamics of the vertebrate segmentation clock. Development. 139 (4), Cambridge, England. 625-639 (2012).
- Pourquié, O. Segmentation of the vertebrate spine: From clock to scoliosis. Cell. 145 (5), 650-663 (2011).
- Naganathan, S. R., Oates, A. C. Patterning and mechanics of somite boundaries in zebrafish embryos. Seminars in Cell & Developmental Biology. 107, 170-178 (2020).
- Hubaud, A., Regev, I., Mahadevan, L., Pourquié, O. Excitable Dynamics and Yap-Dependent Mechanical Cues Drive the Segmentation Clock. Cell. 171 (3), 668-682 (2017).
- Dias, A. S., de Almeida, I., Belmonte, J. M., Glazier, J. A., Stern, C. D. Somites without a clock. Science. 343 (6172), New York, N.Y. 791-795 (2014).
- Nelemans, B. K. A., Schmitz, M., Tahir, H., Merks, R. M., Smit, T. H. Somite Division and New Boundary Formation by Mechanical Strain. iScience. 23 (4), 100976(2020).
- Grima, R., Schnell, S. Can tissue surface tension drive somite formation. Developmental Biology. 307 (2), 248-257 (2007).
- Naganathan, S. R., Popovic, M., Oates, A. C. Left–right symmetry of zebrafish embryos requires somite surface tension. Nature. 605, 516-521 (2022).
- Campàs, O. A toolbox to explore the mechanics of living embryonic tissues. Seminars in Cell & Developmental Biology. 55, 119-130 (2016).
- Langenberg, T., Brand, M., Cooper, M. S. Imaging brain development and organogenesis in zebrafish using immobilized embryonic explants. Developmental Dynamics: An Official Publication of The American Association of Anatomists. 228 (3), 464-474 (2003).
- Henry, C. A., Poage, C. T., McCarthy, M. B., Campos-Ortega, J., Cooper, M. S. Regionally autonomous segmentation within zebrafish presomitic mesoderm. Zebrafish. 2 (1), 7-18 (2005).
- Picker, A., Roellig, D., Pourquié, O., Oates, A. C., Brand, M. Tissue micromanipulation in zebrafish embryos. Methods in Molecular Biology. 546, Clifton, N.J. 153-172 (2009).
- Manning, A. J., Kimelman, D. Tbx16 and Msgn1 are required to establish directional cell migration of zebrafish mesodermal progenitors. Developmental Biology. 406 (2), 172-185 (2015).
- Simsek, M. F., Özbudak, E. M. A 3-D Tail Explant Culture to Study Vertebrate Segmentation in Zebrafish. Journal of Visualized Experiments:JoVE. (172), e61981(2021).
- Schindelin, J., et al. Fiji: an open-source platform for biological-image analysis. Nature Methods. 9 (7), 676-682 (2012).
- Özkale, B., et al. Modular soft robotic microdevices for dexterous biomanipulation. Lab on a Chip. 19 (5), 778-788 (2019).
Ristampe e Autorizzazioni
Richiedi autorizzazione per utilizzare il testo o le figure di questo articolo JoVE
Richiedi AutorizzazioneEsplora altri articoli
This article has been published
Video Coming Soon