Se requiere una suscripción a JoVE para ver este contenido. Inicie sesión o comience su prueba gratuita.
Method Article
Perfiles de Pre-micro ARN y microRNAs utilizando cuantitativa en tiempo real PCR (qPCR) Arrays
En este artículo
Resumen
Vamos a demostrar la configuración y el análisis de pre-microRNA-96 y las matrices de QPCR utilizando un robot, así como con la mano con una pipeta de Thermo Scientific multicanal Matrix.
Resumen
Cuantitativa en tiempo real PCR (QPCR) se ha convertido en una herramienta precisa y valiosa en los perfiles de los niveles de expresión génica. Una de sus muchas ventajas es un menor límite de detección en comparación con otros métodos de perfiles de expresión génica durante el uso de pequeñas cantidades de entrada para cada ensayo. Automatizado de configuración qPCR ha mejorado este campo, al permitir una mayor reproducibilidad. Su configuración permite una rápida y conveniente para experimentos de alto rendimiento, lo que permite determinar las características de muchos genes diferentes al mismo tiempo en cada experimento. Este método junto con los controles internos de la placa también reduce las variables experimentales comunes a otras técnicas. Recientemente hemos desarrollado un ensayo qPCR para la elaboración de perfiles de pre-microRNAs (pre-miRNAs) usando un conjunto de 186 pares de cebadores. Los microARNs se han convertido en una nueva clase de pequeños ARN no codificantes con la capacidad de regular muchas de las metas de mRNA en el nivel post-transcripcional. Estos ARN pequeños se transcribe primero por la ARN polimerasa II como un miRNA primaria (pri-miRNA) transcripción, que se escinde en el miARN precursor (pre-miRNA). Pre-miRNAs se exportan al citoplasma, donde Dicer corta la horquilla para producir miRNAs maduros. Los aumentos en los niveles de miRNA se puede observar tanto en el precursor y los niveles de miRNA maduros y el perfil de estas dos formas pueden ser útiles. Hay varias pruebas disponibles en el mercado de miRNAs maduros, sin embargo, su alto costo puede disuadir a los investigadores de esta técnica de perfiles. En este caso, hablamos de un costo-efectiva, confiable, basado en el método SYBR qPCR de los perfiles pre-miRNAs. Cambios en los niveles pre-miRNA maduros suelen reflejar los cambios miRNA y puede ser un indicador útil de la expresión de miRNA maduros. Sin embargo, el perfil simultánea de ambos pre-miRNAs y miRNAs maduros puede ser óptimo, ya que pueden aportar información redundante y proporcionar información sobre el procesamiento de microARN. Además, la técnica descrita aquí se puede ampliar para abarcar la elaboración de perfiles de grupos de otras bibliotecas de vías específicas o patógenos.
Protocolo
El qPCR pre-miRNA matrices de perfiles se pueden configurar como totalmente automatizada con un robot Tecan Libertad Evo (A) oa mano con la pipeta multicanal electrónica Matrix (B).
1) Prepare la mezcla maestra, placas de imprimación y muestras.
- Primer placas que contienen 186 pares de cebadores en formato de 96 pocillos (un total de 2 placas) en 24:05 deben ser almacenados a -80 ° C. Placas de descongelar a temperatura ambiente, mezclar y centrifugar brevemente antes de su uso.
- Para preparar la mezcla maestra, descongele SYBR Green 2x Mix PCR a temperatura ambiente. Cada reacción utiliza mezcla maestra 8ul con imprimación 2UL. La composición de la mezcla maestra por la reacción es la mezcla SYBR 4ul, 3UL agua grado PCR y 10-20 ng de ADN de muestras o de ADNc.
- Para la configuración manual de cada placa de 96 pozos, cuatro tubos de mezcla maestra se necesita. Las muestras se pueden ejecutar cuatro muestras por placa (singlicate), 2 muestras por placa (duplicado) o una sola muestra por cuadruplicado.
- Prepare la mezcla de maestro mediante la combinación de SYBR, el agua y la muestra en cada uno de 4 tubos Eppendorf de 2 ml. Cada tubo debe contener mezcla maestra suficiente para unas 100 reacciones, permitiendo que el exceso de residuos que permitan tomar. Vortex para mezclar.
A. La instalación de ensayo de pre-miRNA con la libertad Tecan Evo robot.
- Después de la inicialización del robot y la carga del programa Evoware, enjuague tres veces con cada uno de 30ml para limpiar el sistema de burbujas de aire que puedan interferir con la precisión de pipeteo.
- Configuración de la plataforma de robot para incluir lo siguiente:
- Etiquetado de 384 pocillos
- Placa de 96 pocillos de imprimación (1)
- Mezclas maestras
- Una cubeta con 2% de lejía
- Un sistema de recipiente lleno de líquido
- Contenedor de basura vacío
- Comience ejecutar robot automatizado seleccionando la opción "Ejecutar" en dos ocasiones.
- Al final del programa, quite la placa de 384 pocillos y sellado con una lámina de sellado LightCycler 480 y centrifugar brevemente. Lugar cerrado placa 384-y en la posición 1 del hotel.
- Ir al paso 2.2 y repetir para la placa de imprimación con mezclas de dos maestros nuevos y una nueva placa de 384 pocillos.
- Coloque la placa de sellado en la posición 2 del hotel.
- Programa abierto nuevas Evoware para la carga de la LightCycler del hotel, y seleccione "Ejecutar" en dos ocasiones.
- Libertad Evo se cargará automáticamente cada plato en el LightCycler y ejecute el siguiente SYBR verde I / HRM programa de ciclismo:
Pre (1 ciclo):
50 º durante 5 minutos a velocidad de rampa de 4,8 ° / seg
95 º durante 5 minutos a velocidad de rampa de 4,8 ° / seg
Amp (45 ciclos):
95 º durante 15 segundos a velocidad de rampa de 4,8 ° / seg
62 ° durante 30 segundos a velocidad de rampa de 2,5 ° / seg
(Adquisición de datos único en este paso)
La curva de fusión:
95 º durante 5 segundos a velocidad de rampa de 4,8 ° / seg
60 º C durante 1 minuto a velocidad de rampa de 2,5 ° / seg
95 ° continua en rampa de 0,11 ° / seg
con 5 ° C por adquisiciones
Cool:
50 º C durante 30 segundos a velocidad de rampa de 25 ° / seg
- Libertad Evo se cargará automáticamente cada plato en el LightCycler y ejecute el siguiente SYBR verde I / HRM programa de ciclismo:
B. La instalación de ensayo de pre-miRNA con la pipeta multicanal electrónica Matrix:
- Coloque el contenido del tubo de mezcla maestra 1 en un depósito.
- Establecer la pipeta electrónica para aspirar 16ul de la mezcla principal con 2 8ul prescindir de los pasos seguidos por un paso de purga.
- Para un maestro de la mezcla, distribuir 8ul en todos los otros y de la placa de 384 pocillos (establecido en 384-y espaciamiento de punta), empezando por el pocillo A1. (Es decir, los pozos A1, C1, E1, etc) Para el segundo 8ul prescindir, pasar a la columna 3 (es decir, pozos A3, C3, E3, etc) luego se purgan en un contenedor de residuos y consejos para eliminar.
- Siguen este patrón para el resto de 5 ciclos hasta llegar así A23.
- Repita el procedimiento para la mezcla principal 2, (a partir y A2, C2, E2, etc), Mix Master 3 (a partir de B1 así, D1, F1, etc); Master Mix 4 (a partir de B2, D2, F2, etc .).
- Para aliquotting de la placa de imprimación, establezca pipeta electrónica para aspirar y dispensar 2UL 2UL con un siguiente paso de purga. Prescindir 2UL de cebadores de la columna 1 de 96 y placa de imprimación (AH) en la placa de 384 pozos, pozos A1, C1, E1, etc purga en un contenedor de residuos y consejos para eliminar. Repita la operación para pozos A2, C2, E2, B1, D1, F1, y B2, D2, F2, etc
- Repetir las filas 2-12 de cebadores de 96 pozos, pasando por cada 2 columnas para cada columna de imprimación, hasta que en 384 y placa ha sido alícuotas.
- Placas de sellado con una lámina de sellado LightCycler y centrifugar brevemente.
- Coloque las placas en un LightCycler 480 y el ciclo de las placas de acuerdo con los siguientes valores con el SYBR Green I / formato de gestión de recursos humanos de detección:
Pre (1 ciclo):
50 º durante 5 minutos a velocidad de rampa de 4,8 ° / seg
95 º durante 5 minutos a velocidad de rampa de 4,8 ° / seg
Amp (45 ciclos):
95 º durante 15 segundos a velocidad de rampade 4,8 ° / seg
62 ° durante 30 segundos a velocidad de rampa de 2,5 ° / seg
(Adquisición de datos único en este paso)
La curva de fusión:
95 º durante 5 segundos a velocidad de rampa de 4,8 ° / seg
60 º C durante 1 minuto a velocidad de rampa de 2,5 ° / seg
95 ° continua en rampa de 0,11 ° / seg
con 5 ° C por adquisiciones
Cool:
50 º C durante 30 segundos a velocidad de rampa de 25 ° / seg - Repita con la placa de cebador 2, aliquotting en un plato fresco de 384 pozos con mezcla recién preparada maestro.
Secretos para el éxito:
Una característica importante en el diseño de matrices de la PCR es que los 186 pares de iniciadores tienen la misma temperatura de recocido, por lo que la placa se puede ejecutar en la misma configuración óptima PCR programa que se ejecuta.
Cada nueva matriz se debe comprobar con tres controles antes de la ejecución de ejemplos. Estos controles son los siguientes:
Un plazo de todos los primers funcionar con agua o 0,1 x TE descartar la contaminación de imprimación.
Un plazo de alternar el agua / TE y el control positivo, para garantizar que no arrastre.
3 funciona con el control positivo mismo, para asegurar la reproducibilidad entre carreras.
Mezclas maestro debe prepararse, y las placas se debe ejecutar dentro de las 24 horas para obtener resultados óptimos
Primer papel de la placa debe ser retirado lentamente para reducir el riesgo de contaminación entre los pocillos.
Cuando se utiliza un robot con puntas fijas, 2% lavado de cloro es necesario lavar las puntas de cada paso de pipeteo, seguida de un chorro de agua. Esto previene y elimina la prórroga, lo cual podría contaminar los datos y dar lugar a resultados no concluyentes.
Después de un plato que se ejecuta a través de la LightCycler, que no debe abrirse de nuevo en la sala de configuración de PCR. Esto ayuda a evitar la contaminación de PCR.
Los resultados representativos:
qPCR resultados son por lo general representado por los valores CT determinado por el software de análisis LightCycler. Una carrera exitosa por lo general consiste de una serie de TC de las muestras, por lo general entre 20 a 35 para las muestras que son positivas. Las muestras de agua en una buena racha siempre están en una tomografía computarizada de> 40 y las muestras o los pozos específicos con un TC más de 37 se consideran negativas o no detectados. Como regla general, las muestras de ceder un CT de <10 tampoco son fiables y están excluidos del análisis. Hay varias maneras de analizar los datos qPCR y la inclusión de los controles internos en nuestro ensayo ayuda a controlar la variación de la entrada de la muestra. La matriz pre-MIR incluye un manual de control de U6, que se utiliza a menudo como un gen de referencia para normalizar los valores de CT a partir de muestras diferentes. Este valor normalizado que se conoce como el valor delta CT (DCT). Los resultados suelen representarse como la materia prima CT, el valor de DCT o pueden ser analizados con los valores estandarizados.
En la Figura 1, el análisis de la totalidad de pre-microARN serie de cuatro muestras diferentes de un experimento timecourse se presenta en formato de mapa de calor. La expresión relativa de cada uno de microARN pre-que se muestra y surgieron tres grupos principales, distintas. La mayoría de los pre-MIR analizado experimentó cambios pequeños, insignificantes como se esperaba (Figura 1). Sin embargo, una pequeña parte de la pre-micro ARN se redujo significativamente (fig. 1B) o aumentado de manera espectacular (Figura 1 C) durante todo el experimento timecourse. Los niveles de expresión de microARN cada pre-se normalizaron con el punto de tiempo 0 h (muestra 1) como nivel de referencia. En algunos casos, es posible que tenga en cuenta que algunos de los pre-agrupados microARN también se agrupan en el análisis de mapa de calor (por ejemplo: let-7a, fig. 1B). Dependiendo del experimento, los grupos pueden surgir que dependen de la infección viral, la expresión de tipo de células específicas de pre-micro ARN o pre-MIR que están regulados por una molécula común o vía de señalización.
El ensayo previo de micro ARN que se han desarrollado también incluye una serie de conocidos viral antes de los microARN y los genes codificados por el sarcoma de Kaposi asociado herpes (HVSK) y virus de Epstein Barr (EBV), además de los humanos pre-MIR. Estos pueden ser usados como controles positivos o negativos, dependiendo de la línea celular y el estado viral de las muestras utilizadas. En la figura 2, los transformadores de intensidad media de una muestra de HVSK negativo ejecutar en cuatro se muestran. Es importante destacar que, HVSK LANA no se detectó en esta muestra por la matriz qPCR de alta sensibilidad. Sin embargo, U6 - nuestro control interno y dejar 7a - una alta expresión humana pre-microARN, se expresaron en niveles detectables. Finalmente, como se esperaba, el control negativo de PCR de calidad del agua no resulta un producto qPCR.
Otro indicador de una carrera exitosa qPCR es un análisis de la curva de fusión bien, lo que puede dar una idea de la posible contaminación en las muestras. Para establecer una temperatura de fusión, la placa se calienta lentamente después de la PCR se ha completado. Como las hebras de ADN separados, SYBR verde se libera, y disminuye la señal de fluorescencia. La temperatura a la que este proyecto de resoluciónop se produce la representación gráfica, y es conocida como la temperatura de fusión de la muestra. Dependiendo de la secuencia de ADN, las muestras se funden a temperaturas diferentes el uno del otro y del control negativo de agua, que no debe tener una temperatura de fusión del ADN, ya que no está presente. Los productos de PCR de cada par de cebadores que se funden a temperaturas similares. Por ejemplo, la Figura 3 se muestra un análisis de la curva de fusión de dos cebadores diferentes utilizando la misma muestra por duplicado. Está claro que la temperatura de fusión es diferente para los dos pares de primers, pero la muestra se está derritiendo a la misma temperatura exacta para cada réplica. Los signos de una muestra de mala o potencialmente contaminado puede incluir varios picos de fusión, lo que sugiere la presencia de dos diferentes fuentes de entrada.
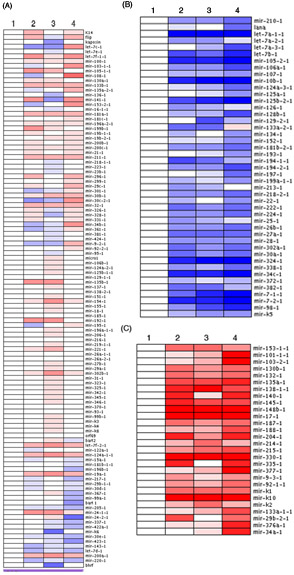
Figura 1. MicroARN Pre-emergen a través de perfiles de las firmas con un qPCR basada en la novela matriz. El promedio se calculó deltaCTU6 y valores normalizados fueron cargados en el software ArrayMinerTM, produciendo tres grupos distintos se muestra como mapas de calor. (A) Pre-microARN con pequeños cambios en la expresión se muestran. Pre-microARN cuyos niveles se redujeron significativamente (B) o mayor (C) también se muestran. Azul corresponde a los niveles más bajos de expresión mientras que el rojo representa el aumento de los niveles de expresión.
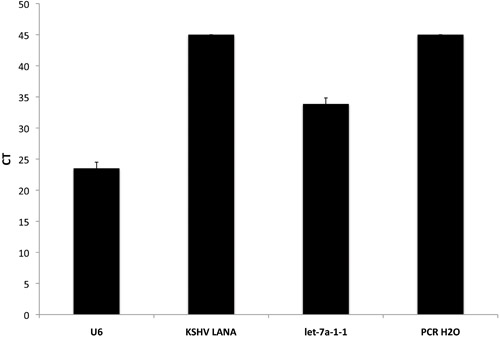
Figura 2. Inclusión de los controles internos en la qPCR basado en pre-microARN matriz. La prima valores CT para 3 genes diferentes y un control de plantilla no se muestran. U6 se utiliza como un control positivo interno, mientras que la PCR H2O nos sirve como control negativo. Vamos-7a-1-1 es un microARN normalmente se expresa en muchos tipos celulares diferentes, mientras que HVSK LANA se puede utilizar como un control positivo o negativo, según el estado viral de la línea de células o tejidos utilizados.
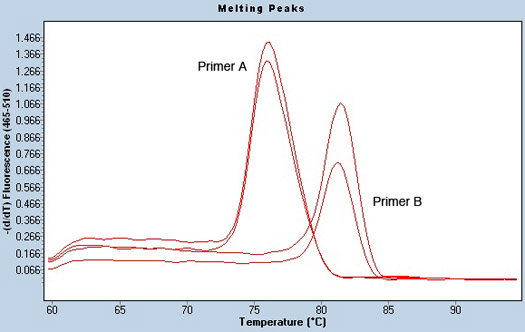
Figura 3. Fusión análisis de los picos de los cebadores pre-microARN y la falta de contaminación de la muestra. Las curvas de fusión se obtuvieron mediante el análisis de llamadas Tm en el software LightCycler. Dos cebadores diferentes (Primer A y B) para la misma muestra por duplicado se muestran. La temperatura de fusión de cada cebador es distinto. Sin embargo, la muestra tiene un solo pico, lo que demuestra la falta de contaminación de la muestra.
Discusión
QPCR es un ensayo muy sensible que puede ser utilizado para comparar los niveles de expresión genética entre las muestras en un estudio, así como para determinar la positividad en el caso de los virus. La ventaja de utilizar matrices de qPCR es la capacidad de ejecutar muchos primers (en nuestro caso, 186 pares de cebadores) para cada muestra en un corto período de tiempo. El uso de un robot de pipeteado como el Evo Tecan la libertad, la cantidad de tiempo necesario para establecer un experimento hasta pueden reduci...
Divulgaciones
Agradecimientos
Este trabajo fue apoyado por subvenciones del NIH DE018304, R01DE018281. KT es compatible con T32 GM07092-34 y por una beca de la Universidad de Carolina del Norte en Chapel Hill de Instituto Médico Howard Hughes (HHMI), a través del Mediterráneo en Grad Iniciativa. PC es compatible con T32 CA009156.
Materiales
| Material Name | Type | Company | Catalogue Number | Comment |
|---|---|---|---|---|
| Name | Company | Catalog Number | Comments | |
| Freedom Evo 150 | Tecan | 30017587 | ||
| Matrix Electronic Multichannel Pipette | Thermo Scientific | 2001-MTX | (or any comparable Thermo Scientific multichannel that can pipette the volumes described above) | |
| Matrix Integrity Filter Tips, Sterile | Thermo Scientific | 7435 | (or comparable tip for multichannel used) | |
| Lightcycler 480 SYBR Green 1 Master | Roche | 04 707 516 001 | ||
| Lightcycler 480 Instrument II | Roche | 05015243001 | 384-well version | |
| Lightcycler 480 Multiwell Plate 384, white | Roche | 04729749001 | ||
| LightCycler 480 Sealing Foil | Roche | 04729757001 | ||
| LightCycler 480 Multiple Plate Analysis Software | Roche | 05075122001 | ||
| Eliminase Decontaminant | Decon Laboratories | 04-355-31 |
Referencias
- Bustin, A. A. The MIQE Guidelines: Minimum Information for Publication of Quantitative Real-Time PCR Experiments. Clinical Chem. 55 (4), 611-622 (2009).
- Frégeau, C. J. Automated Processing of Forensic Casework Samples using Robotic Workstations Equipped with Nondisposable Tips - Contamination Prevention. J. Forensic Sci. 53 (3), 53-533 (2008).
- O'hara, A. J. Pre-micro RNA Signatures Delineate Stages of Endothelial Cell Transformation in Kaposi Sarcoma. PLoS Pathog. 5 (4), e1000389-e1000389 (2009).
Reimpresiones y Permisos
Solicitar permiso para reutilizar el texto o las figuras de este JoVE artículos
Solicitar permisoExplorar más artículos
This article has been published
Video Coming Soon
ACERCA DE JoVE
Copyright © 2025 MyJoVE Corporation. Todos los derechos reservados