Un abonnement à JoVE est nécessaire pour voir ce contenu. Connectez-vous ou commencez votre essai gratuit.
Method Article
Profilage de la pré-micro ARNs et micro-ARN en utilisant quantitative PCR en temps réel (qPCR) Arrays
Dans cet article
Résumé
Nous allons démontrer la configuration et l'analyse des pré-microARN de 96 puits pour les tableaux QPCR aide d'un robot ainsi que par la main avec une pipette multicanaux Thermo Scientific Matrix.
Résumé
Quantitative PCR en temps réel (QPCR) a émergé comme un outil précis et précieux dans le profilage des niveaux d'expression génique. Un de ses nombreux avantages est une limite inférieure de détection par rapport à d'autres méthodes de profilage de l'expression des gènes en utilisant de petites quantités d'entrée pour chaque dosage. Automatisé de configuration qPCR a amélioré ce domaine en permettant une plus grande reproductibilité. Sa mise en pratique et rapide permet d'expériences à haut débit, permettant le profilage de nombreux gènes différents simultanément dans chaque expérience. Cette méthode avec des contrôles plaque interne réduit également les variables expérimentales communes à d'autres techniques. Nous avons récemment développé un test de PCR quantitative pour le profilage de la pré-microARN (pré-miARN) en utilisant un ensemble de 186 paires d'amorces. Les microARN sont apparus comme une nouvelle classe de petits ARN non-codants avec la capacité de réguler les cibles ARNm nombreuses au niveau post-transcriptionnel. Ces petits ARN sont d'abord transcrit par l'ARN polymérase II comme un miARN primaires (PRI-miARN) transcription, qui est ensuite clivé dans le miARN précurseur (pré-miARN). Pré-miARN sont exportés vers le cytoplasme où Dicer clive la boucle en épingle à cheveux à céder miARN matures. L'augmentation des niveaux de miARN peuvent être observées à la fois le précurseur et le niveau miARN matures et de profilage de ces deux formes peuvent être utiles. Il existe plusieurs dosages disponibles commercialement pour miARN matures, mais leur coût élevé peut dissuader les chercheurs de cette technique de profilage. Ici, nous discutons d'un bon rapport coût-efficacité, la fiabilité, la méthode qPCR SYBR à base de profilage de pré-miARN. Les changements dans les niveaux de pré-miARN sont souvent le reflet des changements miARN mature et peut être un indicateur utile de l'expression du miARN mature. Toutefois, le profilage simultané des deux pré-miARN et miARN mature peut être optimale, car ils peuvent apporter des informations non redondantes et de donner un aperçu du traitement de microARN. Par ailleurs, la technique décrite ici peut être étendu pour englober le profilage des ensembles d'autres bibliothèques pour les voies spécifiques ou des agents pathogènes.
Protocole
Les tableaux qPCR profilage pré-miARN peuvent être mis en place comme entièrement automatisée avec un robot Tecan Freedom EVO (A) ou à la main avec la pipette multicanaux matrice électronique (B).
1) Préparer le mélange maître, plaques apprêt et échantillons.
- Plaques amorce contenant 186 paires d'amorces au format 96 puits (un total de 2 plaques) à 12h05 doit être stocké à -80 ° C. Plaques décongeler à température ambiante, vortex et centrifuger brièvement avant utilisation.
- Pour préparer le mélange maître, dégeler SYBR Green PCR 2x Mélanger à température ambiante. Chaque réaction utilise master mix 8ul avec un apprêt 2UL. La composition du mélange maître par réaction est un mélange SYBR 4UL, l'eau de qualité 3UL PCR et 10-20 ng d'échantillon d'ADN ou d'ADNc.
- Pour la configuration de chaque plaque d'amorce de 96 puits, quatre tubes master mix sera nécessaire. Les échantillons peuvent être exécutés 4 échantillons par plaque (singlicate), 2 échantillons par plaque (double) ou d'un seul échantillon en quatre exemplaires.
- Préparer le mélange maître en combinant SYBR, l'eau et l'échantillon dans chacun des tubes Eppendorf 4-2ml. Chaque tube doit contenir master mix assez pour environ 100 réactions, permettant l'excès pour le pipetage des déchets. Vortex pour mélanger.
Configuration A. de dosage pré-miARN utilisant la Liberté Tecan Evo robot.
- Après l'initialisation du robot et le chargement du programme EVOware, rincer trois fois avec chaque 30ml pour effacer le système de bulles d'air qui va interférer avec la précision de pipetage.
- Configuration de la plateforme de robot pour y inclure les éléments suivants:
- Étiqueté 384 puits
- Plaque de 96 puits d'amorces (1)
- Maître mêle
- Un bac contenant de l'eau de Javel à 2%
- Un récipient rempli de liquide du système
- Conteneur à déchets vide
- Commencez courir robot automatisé en sélectionnant "Exécuter" deux fois.
- À la fin du programme, retirez la plaque de 384 puits et sceller avec une feuille d'étanchéité LightCycler 480 et centrifuger brièvement. Placez scellées plaque de 384 puits dans la position 1 de l'hôtel.
- Passez à l'étape 2.2 et répéter pour 2 plaque d'amorce avec des mélanges nouveau maître et une nouvelle plaque de 384 puits.
- Placer la plaque scellée en position 2 de l'hôtel.
- Ouvrez nouveau programme EVOware pour le chargement des Lightcycler de l'hôtel et sélectionner "Exécuter" deux fois.
- Freedom EVO se charge automatiquement chaque plaque dans le LightCycler et exécutez la commande suivante SYBR-vert I / GRH programme de cyclisme:
Pré (1 cycle):
50 ° pendant 5 minutes à vitesse de rampe de 4,8 ° / sec
95 ° pendant 5 minutes à vitesse de rampe de 4,8 ° / sec
Amp (45 cycles):
95 ° pendant 15 secondes au taux de rampe de 4,8 ° / sec
62 ° pendant 30 sec à vitesse de rampe de 2,5 ° / sec
(Simple d'acquisition de données au cours de cette étape)
Courbe de fusion:
95 ° pendant 5 sec à vitesse de rampe de 4,8 ° / sec
60 ° pendant 1 minute à taux de rampe de 2,5 ° / sec
95 ° en continu au taux de rampe de 0,11 ° / sec
avec 5 acquisitions par ° C
Cool:
50 ° pendant 30 sec à vitesse de rampe de 25 ° / sec
- Freedom EVO se charge automatiquement chaque plaque dans le LightCycler et exécutez la commande suivante SYBR-vert I / GRH programme de cyclisme:
Installation de dosage B. miARN pré-utilisation de la pipette multicanaux matrice électronique:
- Placez le contenu du tube master mix 1 dans un réservoir.
- Régler la pipette pour aspirer électroniques 16ul de mélange maître avec 2-8ul passer les étapes suivies par une étape de purge.
- Pour Mix Master 1, passer dans chaque 8ul bien d'autres de la plaque de 384 puits (réglé sur 384 et l'espacement pointe), en commençant par le puits A1. (Ie puits A1, C1, E1, etc) Pour la seconde 8ul passer, passer à la colonne 3 (ie puits A3, C3, E3, etc) puis purger au cours d'un conteneur de déchets et des conseils céder.
- Suivez ce modèle pour les 5 autres cycles jusqu'à atteindre ainsi A23.
- Répétez l'opération pour Mix Master 2, (à commencer par godet A2, C2, E2, etc); master mix 3 (à partir de puits B1, D1, F1, etc); master mix 4 (à partir de B2, D2, F2, etc .).
- Pour aliquotting de plaque d'amorce, mettre une pipette pour aspirer 2UL électroniques et dispenser 2UL avec une étape de purge suivante. Distribuer 2UL d'amorces de la colonne 1 de la plaque de 96 puits d'amorces (AH) dans la plaque de 384 puits, puits A1, C1, E1, etc purge sur un conteneur à déchets et des conseils céder. Répétez l'opération pour puits A2, C2, E2, B1, D1, F1, et B2, D2, F2, etc
- Répétez l'opération pour les lignes 2-12 du amorces de 96 puits, se déplaçant sur tous les 2 colonnes pour chaque colonne d'apprêt, jusqu'à plein 384 puits ont été aliquotes.
- Sceau plaques avec un film d'étanchéité LightCycler et centrifuger brièvement.
- Plaques Placer dans un LightCycler 480 et le cycle des plaques en fonction des paramètres suivants en utilisant le SYBR Green I / format de détection de la GRH:
Pré (1 cycle):
50 ° pendant 5 minutes à vitesse de rampe de 4,8 ° / sec
95 ° pendant 5 minutes à vitesse de rampe de 4,8 ° / sec
Amp (45 cycles):
95 ° pendant 15 secondes au taux de rampede 4,8 ° / sec
62 ° pendant 30 sec à vitesse de rampe de 2,5 ° / sec
(Simple d'acquisition de données au cours de cette étape)
Courbe de fusion:
95 ° pendant 5 sec à vitesse de rampe de 4,8 ° / sec
60 ° pendant 1 minute à taux de rampe de 2,5 ° / sec
95 ° en continu au taux de rampe de 0,11 ° / sec
avec 5 acquisitions par ° C
Cool:
50 ° pendant 30 sec à vitesse de rampe de 25 ° / sec - Répétez l'aide de 2 plaque d'amorce, aliquotting dans une plaque fraîche de 384 puits en utilisant des mélanges maîtres fraîchement préparés.
Secrets de la réussite:
Une caractéristique importante lors de la conception des tableaux de PCR est que tous les 186 paires d'amorces ont la même température de recuit, donc la plaque peut être exécuté avec les mêmes paramètres de PCR optimales programme exécuté.
Chaque nouveau tableau doit être vérifiée en utilisant trois contrôles avant les échantillons. Ces contrôles sont:
1 piste de toutes les amorces fonctionner avec de l'eau ou 0,1 x TE d'exclure la contamination primaire.
1 piste de l'alternance eau / TE et de contrôle positif, afin de s'assurer qu'aucun report.
3 pistes en utilisant le même contrôle positif, afin d'assurer la reproductibilité entre les courses.
Master Mix doit être fraîchement préparée, et les plaques doivent être exécutés dans les 24 heures pour des résultats optimaux
Feuille plaque d'amorce doit être retiré lentement pour réduire le risque de contamination entre les puits.
Lors de l'utilisation d'un robot avec des conseils fixes, laver l'eau de Javel à 2% est nécessaire de laver les bouts entre chaque étape de pipetage, suivie d'un rinçage à l'eau. Cela évite et élimine le report, ce qui pourrait contaminer les autres données et conduire à des résultats concluants.
Après une plaque est géré par le LightCycler, il ne devrait pas être ouverte à nouveau dans la salle de configuration PCR. Cela permet d'éviter la contamination PCR.
Les résultats représentatifs:
résultats de qPCR sont généralement représentés par des valeurs de CT tel que déterminé par le logiciel d'analyse LightCycler. Une course réussie se compose généralement d'une gamme de TC pour les échantillons, généralement entre 20 à 35 pour les échantillons qui sont positifs. Les échantillons d'eau dans une bonne course sont toujours à un CT> 40 et tous les échantillons ou de puits spécifique avec un CT de plus de 37 sont considérés comme négatifs ou non détecté. En règle générale, les échantillons donnant une CT de <10 sont également peu fiables et sont exclus de l'analyse. Il ya plusieurs façons d'analyser les données qPCR et l'inclusion de contrôles internes dans notre test permet de contrôler les variations de l'entrée de l'échantillon. Le tableau pré-miR comprend un apprêt de contrôle U6, qui est souvent utilisé comme un gène de référence pour normaliser les valeurs CT à partir d'échantillons différents. Cette valeur normalisée est appelée la valeur delta CT (DCT). Les résultats sont souvent représentées comme le CT premières, la valeur DCT ou peut encore être analysés pour des valeurs normalisées.
Dans la figure 1, l'analyse de la pleine pré-microARN tableau de quatre échantillons différents d'une expérience timecourse est présenté en format carte de chaleur. L'expression relative de chaque pré-microARN est montré et trois majeurs, les clusters distincts ont émergé. La majorité des pré-mirs analysé subi de petites modifications insignifiantes comme prévu (figure 1A). Cependant, une petite portion de pré-microARN ont été significativement diminué (figure 1B) ou augmenté de façon spectaculaire (figure 1C) à travers l'expérience timecourse. Les niveaux d'expression de chacun de pré-microARN ont été normalisées à l'timepoint 0h (échantillon 1) comme un niveau de référence. Dans certains cas, vous pouvez noter que certains des clusters pré-microARN également se regrouper dans l'analyse heatmap (ex: laissez-7a Figure 1B,). Selon l'expérience, les clusters peuvent émerger qui dépendent de l'infection virale, l'expression type de cellule spécifique de pré-microARN ou pré-mirs qui sont réglementés par une molécule commune ou la voie de signalisation.
Le test pré-microARN que nous avons développé comprend également un certain nombre de pré-viraux connus microARN codés par des gènes et le sarcome de Kaposi herpèsvirus associé (KSHV) et le virus Epstein Barr (EBV), en plus de l'homme pré-mirs. Ceux-ci peuvent être utilisés comme contrôles positifs ou négatifs, selon la lignée cellulaire et statut viral des échantillons utilisés. Dans la figure 2, la CTS en moyenne à partir d'un échantillon de KSHV négatif exécuté en quatre exemplaires sont présentés. Surtout, KSHV LANA n'a pas été détectée dans cet échantillon par le tableau qPCR hautement sensible. Toutefois, U6 - notre contrôle interne et laissez-7a - hautement exprimé humaines pré-microARN, ont été exprimées à des niveaux détectables. Enfin, comme prévu, le contrôle négatif de la PCR-grade de l'eau n'a pas le rendement d'un produit qPCR.
Un autre indicateur d'une exécution réussie qPCR est une analyse fondre une bonne courbe, ce qui peut donner un aperçu de la contamination potentielle dans les échantillons. Pour établir une température de fusion, la plaque est chauffée lentement après la PCR est terminée. Comme les brins d'ADN séparés, SYBR green est libéré, et le signal de fluorescence diminue. La température à laquelle ce drop se produit est tracée, et est connue comme la température de fusion de l'échantillon. Selon la séquence d'ADN, les échantillons fondent à des températures différentes les unes des autres et de la maîtrise de l'eau négative, ce qui ne devrait pas avoir une température de fusion puisque aucun ADN est présent. Les produits de PCR de chaque paire d'amorces devraient fondre à des températures similaires. Par exemple, la figure 3 montre une analyse de la courbe de fusion de deux amorces différentes utilisant le même échantillon en double exemplaire. Il est clair que la température de fusion est différente pour les deux paires d'amorces, mais l'échantillon est fondant à la température exactement le même pour chaque réplique. Les signes d'un mauvais échantillon ou potentiellement contaminés peut inclure plusieurs pics de fusion, ce qui suggère la présence de deux sources différentes de l'entrée.
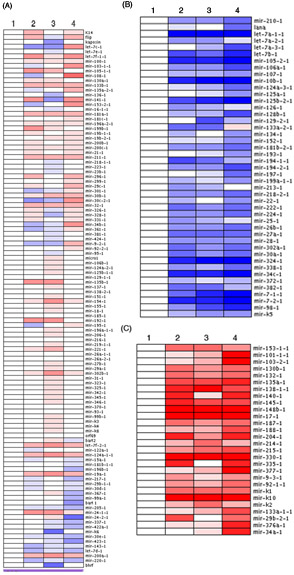
Figure 1. Pré-microARN signatures émerger à travers l'aide d'un profilage basé sur roman de qPCR tableau. Le deltaCTU6 moyenne a été calculée et les valeurs standardisées ont été chargés dans le logiciel ArrayMinerTM, produisant trois groupes distincts affiché comme heatmaps. (A) pré-microARN avec de petits changements dans l'expression sont affichées. Pré-microARN dont les niveaux étaient significativement diminué (B) ou augmenté (C) sont également représentées. Le bleu correspond à des niveaux inférieurs d'expression tandis que le rouge représente augmenté les niveaux d'expression.
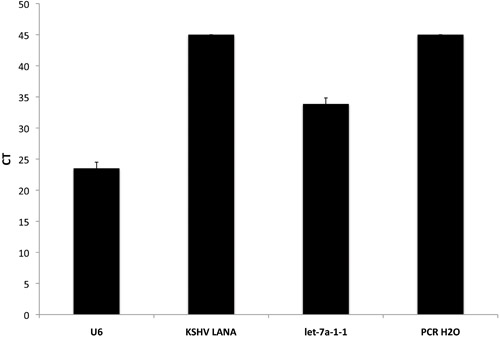
Figure 2. Inclusion des contrôles internes dans le qPCR à base de pré-microARN tableau. Les valeurs brutes CT pour 3 gènes différents et un contrôle de modèle ne sont affichés. U6 est utilisé comme un contrôle positif interne tandis PCR H2O nous sert de contrôle négatif. Laissez-7a-1-1 est un microARN normalement exprimé dans de nombreux types cellulaires différents tout KSHV LANA peut être utilisé soit comme un contrôle positif ou négatif, fondée sur le statut viral de la lignée cellulaire ou tissu utilisé.
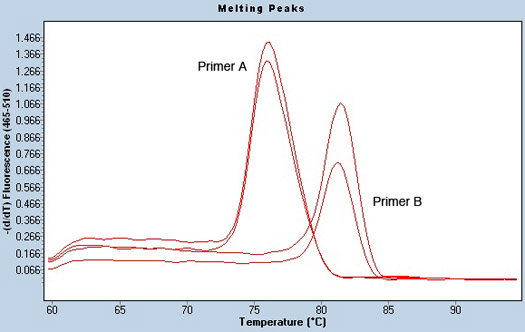
Figure 3. Fusion d'analyse de pointe de pré-microARN amorces et l'absence de contamination des échantillons. Courbes de fusion ont été obtenues en utilisant l'analyse appelant Tm dans le logiciel LightCycler. Deux amorces différentes (Primer A et B) pour le même échantillon analysés en double sont affichés. La température de fusion pour chaque amorce est distincte. Toutefois, l'échantillon a une seule pointe, démontrant l'absence de contamination des échantillons.
Access restricted. Please log in or start a trial to view this content.
Discussion
QPCR est un test très sensible qui peuvent être utilisés pour comparer les niveaux d'expression génique entre les échantillons dans une étude, ainsi que pour déterminer la positivité dans le cas des virus. L'avantage d'utiliser des tableaux qPCR est la capacité d'exécuter de nombreuses amorces (dans notre cas, 186 paires d'amorces) pour chaque échantillon dans un court laps de temps. L'utilisation d'un robot de pipetage tels que l'Evo Tecan Freedom, le montant de temps requis p...
Access restricted. Please log in or start a trial to view this content.
Déclarations de divulgation
Remerciements
Ce travail a été soutenu par des subventions du NIH DE018304, R01DE018281. KT est soutenu par T32 GM07092-34 et par une subvention à l'Université de Caroline du Nord à Chapel Hill de Howard Hughes Medical Institute (HHMI) à travers la Méditerranée en Grad Initiative. PC T32 est soutenu par CA009156.
Access restricted. Please log in or start a trial to view this content.
matériels
| Name | Company | Catalog Number | Comments |
| Freedom Evo 150 | Tecan Group Ltd. | 30017587 | |
| Matrix Electronic Multichannel Pipette | Thermo Fisher Scientific, Inc. | 2001-MTX | (or any comparable Thermo Scientific multichannel that can pipette the volumes described above) |
| Matrix Integrity Filter Tips, Sterile | Thermo Fisher Scientific, Inc. | 7435 | (or comparable tip for multichannel used) |
| Lightcycler 480 SYBR Green 1 Master | Roche Group | 04 707 516 001 | |
| Lightcycler 480 Instrument II | Roche Group | 05015243001 | 384-well version |
| Lightcycler 480 Multiwell Plate 384, white | Roche Group | 04729749001 | |
| LightCycler 480 Sealing Foil | Roche Group | 04729757001 | |
| LightCycler 480 Multiple Plate Analysis Software | Roche Group | 05075122001 | |
| Eliminase Decontaminant | Decon Laboratories | 04-355-31 |
Références
- Bustin, A. A. The MIQE Guidelines: Minimum Information for Publication of Quantitative Real-Time PCR Experiments. Clinical Chem. 55 (4), 611-622 (2009).
- Frégeau, C. J. Automated Processing of Forensic Casework Samples using Robotic Workstations Equipped with Nondisposable Tips - Contamination Prevention. J. Forensic Sci. 53 (3), 53-533 (2008).
- O'hara, A. J. Pre-micro RNA Signatures Delineate Stages of Endothelial Cell Transformation in Kaposi Sarcoma. PLoS Pathog. 5 (4), e1000389-e1000389 (2009).
Access restricted. Please log in or start a trial to view this content.
Réimpressions et Autorisations
Demande d’autorisation pour utiliser le texte ou les figures de cet article JoVE
Demande d’autorisationExplorer plus d’articles
This article has been published
Video Coming Soon