JoVE 비디오를 활용하시려면 도서관을 통한 기관 구독이 필요합니다. 전체 비디오를 보시려면 로그인하거나 무료 트라이얼을 시작하세요.
Method Article
정량 실시간 PCR (qPCR) 배열을 사용하여 사전 마이크로 RNAS과 microRNAs의 프로 파일링
요약
우리는 사전 microRNA의 설정 및 분석 서비스를 선보일 예정 써모 과학 매트릭스 멀티채널 피펫과 수작업으로뿐만 아니라 로봇을 사용하여 QPCR위한 96 잘 배열.
초록
정량 실시간 PCR (QPCR)는 유전자 발현 수준을 프로 파일링의 정확하고 가치있는 도구로 떠오르고있다. 의 많은 장점 중 하나는 각각의 분석에 대한 입력의 작은 금액을 사용하는 동안 유전자 발현 프로 파일링의 다른 방법에 비해 낮은 검출 한도입니다. 자동 qPCR 설정은 큰 재현성에 대한함으로써이 분야를 개선했다. 의 편리하고 신속한 설치는 각 실험에 동시에 여러 유전자의 프로 파일링을 지원, 높은 처리량 실험 수 있습니다. 내부 플레이트 컨트롤과 함께이 방법은 다른 기술에 공통 실험 변수를 감소시킵니다. 우리는 최근 186 프라이머 쌍 집합을 사용하여 사전 microRNAs의 프로 파일링을위한 qPCR 분석을 (사전 miRNAs) 개발. MicroRNAs는 포스트 transcriptional 수준에서 많은 mRNA 목표를 조절하는 능력을 가지고 작은 비 코딩 RNAS의 소설 클래스로 등장했습니다. 이러한 작은 RNAS 먼저 다음 miRNA 전구체 (미리 miRNA)로 죽습니다 기본 miRNA (PRI - miRNA) 성적표로 RNA 중합 효소 II에 의해 베꼈는데 있습니다. 사전 miRNAs는 주사위 놀이를하는 사람이 성숙한 miRNAs를 얻을 수있는 헤어핀 루프를 클리브스 세포질에 수출하고 있습니다. miRNA 수준의 증가가 유용할 수 있습니다 이러한 형태의 모두의 전구체와 성숙한 miRNA 수준 및 프로 파일링 모두에서 볼 수 있습니다. 성숙한 miRNAs에 대한 몇 가지 상용 assays가되지만, 높은 비용이 프로 파일링 기법에서 연구원을 억제 수도 있습니다. 여기서 우리는 사전 miRNAs을 프로파일 링의 비용 효율, 신뢰성, SYBR 기반 qPCR 방법을 논의합니다. 사전 miRNA 수준의 변화는 종종 성숙한 miRNA 변경 사항을 반영하고 성숙한 miRNA의 표현의 유용한 지표 수 있습니다. 그들이 nonredundant 정보를 기여하고 microRNA 처리에 대한 통찰력을 제공할 수 있습니다 그러나, 사전 miRNAs과 성숙한 miRNAs 모두 동시 프로파일 최적의 수 있습니다. 또한, 여기에 설명된 기술은 특정 경로 또는 병원균에 대한 다른 라이브러리 세트의 프로파일을 망라하는 확장하실 수 있습니다.
프로토콜
완전히 Tecan 자유 에보 (Evo) 로봇 (A) 또는 매트릭스 전자 멀티채널 피펫 (B)와 함께 손으로 자동으로 qPCR 사전 miRNA 프로 파일링 배열을 설정할 수 있습니다.
1) 마스터 믹스, 프라이머의 접시와 샘플을 준비합니다.
- 오후 12시 5분에서 96 - 웰 형식 (2 접시의 전체)에 186 프라이머 쌍을 포함하는 프라이머의 플레이트는 -80 ° C.에 보관해야 실내 온도, 소용돌이 및 사용하기 전에 원심 분리기의 간략에서 해동 플레이트 아웃.
- 마스터 믹스를 준비하려면, 실온에서 SYBR 그린 2X PCR 믹스를 누그 러 뜨 리다. 각 반응은 2ul 입문서로 8ul 마스터 믹스를 사용합니다. 반응 당 마스터 믹스의 구성은 4ul SYBR 믹스, 3ul PCR 등급 물 10-20 NG 샘플 DNA 또는 cDNA입니다.
- 각 96 - 웰 플레이트 뇌관의 설정을 위해, 넷 마스터 믹스 튜브가 필요합니다. 샘플은 접시 당 4 샘플 (singlicate), 2 플레이트 당 샘플 (중복) 또는 네 겹의에서 하나의 샘플을 실행할 수 있습니다.
- 4 2ml Eppendorf 튜브의 각 SYBR, 물 샘플을 결합하여 마스터 믹스를 준비합니다. 각각의 튜브는 폐기물을 pipetting에 대한 초과 수 있도록 약 100 반응 정도 마스터 믹스를 포함해야합니다. 섞어 와동.
자유 Tecan 에보 (Evo) 로봇을 사용하여 미리 miRNA 분석의 A. 설치.
- Evoware 프로그램의 로봇과 로딩의 초기화에 따라, 정확성을 pipetting에 영향을 줍니까 공기 방울의 시스템을 취소 30mls 각 세 번 플러시.
- 다음을 포함하는 로봇 플랫폼을 설정 :
- 라벨 384 자 판
- 96 - 웰 플레이트 프라이머 (1)
- 주인님은 믹스
- 2 % 표백제를 포함 트로프
- 가득 시스템 유체 컨테이너
- 빈 폐기물 컨테이너
- 두 번 "실행"을 선택하여 자동 로봇 실행을 시작합니다.
- 프로그램의 끝에, LightCycler 480 씰링 호일로 384 잘 판과 인감을 제거하고 간단히 원심 분리기. 장소는 호텔의 위치 1 384 잘 번호판을 봉인.
- 2.2 단계와 새로운 마스터 혼합하고 새로운 384 잘 플레이트와 프라이머 플레이트 2 반복으로 이동합니다.
- 호텔의 위치 2 밀폐 접시를 놓습니다.
- 호텔에서 Lightcycler를로드하고 두 번 "실행"을 선택하십시오를 위해 새로 문을 여는 Evoware 프로그램입니다.
- 자유 에보 (Evo)가 자동으로 LightCycler에 각 접시를로드 다음과 같은 SYBR 녹색 I / HRM 자전거 프로그램을 실행합니다 :
사전 (1 사이클)
50 4.8의 램프 속도로 5 분 ° ° / 초
95 4.8의 램프 속도로 5 분 ° ° / 초
앰프 (45 사이클) :
95 4.8의 램프 속도 15 초에 대한 ° ° / 초
62 2.5 램프 속도로 30 초에 대한 ° ° / 초
(이 단계에서 단일 데이터 수집)
용융 곡선 :
95 4.8의 램프 속도 5 초에 대한 ° ° / 초
60 2.5 램프 속도로 1 분 ° ° / 초
95 ° 램프 0.11의 속도 ° / 초에서 연속
° C 당 5 인수와 함께
쿨 :
50 25 램프 속도로 30 초에 대한 ° ° / 초
- 자유 에보 (Evo)가 자동으로 LightCycler에 각 접시를로드 다음과 같은 SYBR 녹색 I / HRM 자전거 프로그램을 실행합니다 :
매트릭스 전자 멀티채널 피펫을 사용하여 미리 miRNA 분석의 B. 설정 :
- 저수지에 마스터 믹스 튜브 1 플레이스 내용.
- 정화 단계에서 다음 단계를 분배 2 8ul과 마스터 믹스 16ul 대기음에 전자 피펫을 설정합니다.
- 마스터 믹스 1, 잘 A1부터 시작, 384 잘 플레이트 (384 자 팁 간격에 설정된)의 다른 모든 우물에 8ul 하는걸. (예 : 우물 A1, C1, E1 등) 두 번째 8ul가 폐기물 용기 및 폐기 팁 이상의 다음 열에서 3 (즉 우물 A3, C3, E3, 등) 정화로 이동, 분배하십시오.
- 당신이 잘 A23에 도달할 때까지 나머지 5주기 위해이 패턴을 따릅니다.
- 마스터 믹스 3 (잘 B1, D1, F1 등에서 시작), 마스터 믹스 4 (B2에서 시작, D2, F2, 등 (물론 A2, C2, E2, 등 시작), 마스터 믹스 2 반복 .).
- 프라이머 판의 aliquotting 들어, 2ul을 기음과 퍼지 단계 아래의 2ul 하는걸 전자 피펫을 설정합니다. 폐기물 컨테이너와 폐기 조언을 통해 384 자 판, 우물 A1, C1, E1 등 정리에 96 - 웰 플레이트 프라이머 (AH)의 열 1 primers의 2ul 하는걸. D2와 B2, F2, 등, B1, D1, F1, 우물 A2, C2, E2을 반복
- 전체 384 잘 플레이트가 aliquotted 때까지, 각 프라이머의 열 매 2 열 이상의 이동, 행 96 자 primers의 2-12에 대해 반복합니다.
- LightCycler의 씰링 호일과 간단히 원심 분리기와 인감 접시.
- SYBR 녹색 I / HRM 감지 형식을 사용하여 다음 설정에 따라 LightCycler 480 사이클 접시에 플레이스 플레이트 :
사전 (1 사이클)
50 4.8의 램프 속도로 5 분 ° ° / 초
95 4.8의 램프 속도로 5 분 ° ° / 초
앰프 (45 사이클) :
램프 속도 15 초에 대한 95 °4.8 ° / 초의
62 2.5 램프 속도로 30 초에 대한 ° ° / 초
(이 단계에서 단일 데이터 수집)
용융 곡선 :
95 4.8의 램프 속도 5 초에 대한 ° ° / 초
60 2.5 램프 속도로 1 분 ° ° / 초
95 ° 램프 0.11의 속도 ° / 초에서 연속
° C 당 5 인수와 함께
쿨 :
50 25 램프 속도로 30 초에 대한 ° ° / 초 - 신선한 마스터 믹스를 사용하여 신선한 384 자 판에 aliquotting, 뇌관 플레이트 2를 사용 반복합니다.
성공의 비밀 :
PCR의 배열을 설계 중요한 특성은 모든 186 프라이머 쌍은 동일한 어닐링 온도를 갖고있다, 접시가 동일한 최적의 PCR 실행 프로그램 설정에서 실행할 수 있도록.
각각의 새로운 배열은 이전 실행 샘플 세 가지 컨트롤을 사용하여 확인한다. 이러한 컨트롤은 다음과 같습니다
프라이머 오염을 배제하기 위해 물 또는 0.1 X TE와 함께 실행되는 모든 primers 1 실행합니다.
1도 carryover를 보장하지, 물 / TE와 긍정적인 통제를 교대의 실행합니다.
3 실행 사이의 재현성을 보장하기 위해, 같은 긍정적인 컨트롤을 사용하여 실행됩니다.
마스터 믹스는 신선한해야하고, 플레이트는 최적의 결과에 대해 24 시간 내에 실행되어야
프라이머 플레이트 박이 우물 사이의 오염의 위험을 줄이기 위해 천천히 제거한다.
고정 팁을 로봇을 사용할 때, 2 % 표백제 세척은 물 플러시 다음 각 pipetting 단계 사이의 도움말을 씻어 필요합니다. 이것은 달리 데이터를 오염시키고 결정적 결과를 초래할 수 carryover를 방지하고 제거합니다.
접시가 Lightcycler를 통해 실행되면, 그것은 PCR 설치 공간에 다시 열 수 없습니다. 이것은 PCR 오염을 방지하는 데 도움이됩니다.
대표 결과 :
qPCR 결과는 일반적으로 Lightcycler 분석 소프트웨어에서 결정 중부 표준시 값으로 표시됩니다. 성공적인 실행은 일반적으로 긍정적인 샘플 샘플, 일반적으로 20-35 사이의 CTS의 범위로 구성되어 있습니다. 좋은 실행의 물 샘플> 40 중부 표준시에 항상 37 이상의 모든 샘플 또는 중부 표준시로 특정 우물은 부정이나 검색되지 간주됩니다. 일반적으로, <10의 CT를 항복 샘플도 신뢰할 수 있으며, 추가 분석에서 제외됩니다. 우리의 분석에 qPCR 데이터 및 내부 컨트롤의 포함을 분석하는 방법에는 여러 가지 샘플 입력 변화에 대한 제어를하는 데 도움이 있습니다. 사전 미러 어레이는 종종 서로 다른 샘플에서 중부 표준시 가치를 정상화에 대한 참조 유전자로 사용 U6 제어 프라이머가 포함되어 있습니다. 이 정규화된 값은 델타 중부 표준시 값 (DCT)이라고합니다. 결과는 종종 원시 중부 표준시, DCT 값으로 그래프거나 더 표준화된 값으로 분석하실 수 있습니다.
그림 1에서 timecourse 실험에서 네 가지 샘플의 전체 사전 microRNA 배열의 분석 열지도 형식으로 제공됩니다. 각 사전 microRNA의 상대적인 표현은 그림과 세 대, 독특한 클러스터 등장합니다. (그림 1A) 예상대로 분석 사전 미르의 대다수는 작은, 사소한 변화를 받았습니다. 그러나, 사전 microRNAs의 일부가 크게 감소되었다 (그림 1B) 또는 timecourse 실험을 통해 (그림 1C) 급격하게 증가했다. 각 사전 microRNA의 표현 수준은 기본 수준으로 오 timepoint (예제 1) 정상화되었습니다. 어떤 경우에는, 당신은 클러스터된 사전 microRNAs의 일부도 (하자 - 7A, 그림 1B 예) 히트맵 분석에서 함께 클러스터 유의 있습니다. 실험에 따라 클러스터가 바이러스 감염에 의존하는 등장할 수 있습니다 일반적인 분자 또는 신호 경로에 의해 규제 사전 microRNAs 또는 사전 미르의 세포 유형의 구체적인 표현.
우리도 개발해야하는 사전 microRNA 분석은 인간의 사전 미르 이외에 Herpesvirus (KSHV)와 엡스타인 바 바이러스 (EBV)를 관련 Kaposi의 육종에 의해 인코딩된 알려진 바이러스 사전 microRNAs과 유전자의 숫자를 포함하고 있습니다. 이들은 세포 라인과 사용되는 샘플의 바이러스 상태에 따라 긍정적이거나 부정적인 컨트롤로 사용할 수 있습니다. 그림 2에서, 사통으로 작성된 실행 KSHV - 부정적인 샘플의 평균 CTS가 표시됩니다. 중요한 것은, KSHV라나는 매우 민감한 qPCR 배열하여이 샘플에서 검출되지 않았습니다. 그러나, U6 - 우리의 내부 통제 및하자 - 7A - 고도의 표현 인간 사전 microRNA는 감지 수준에서 표현되었다. 마지막으로, 예상대로, PCR - 등급 물의 부정적인 제어 qPCR 제품을 얻을하지 않았다.
성공 qPCR 실행의 또 다른 표시가 샘플에 잠재적인 오염에 대한 통찰력을 줄 수있는 좋은 용융 곡선 분석입니다. PCR이 완료된 후 용융 온도를 설정하려면, 접시가 천천히 가열됩니다. 별도의 DNA의 가닥으로 SYBR 녹색을 출시하고, 형광 신호는 감소됩니다. 온도되는이 박사조합이 그래프는 발생하고, 샘플의 용해 온도로 알려져 있습니다. DNA 순서에 따라 샘플을 서로 어떠한 DNA가 존재 없기 때문에 녹는 온도를하지 말았어야 물 부정적인 컨트롤, 다른 온도에서 녹기. 각각의 프라이머 쌍에서 PCR 제품과 비슷한 온도에서 용융한다. 예를 들어, 그림 3은 중복 같은 샘플을 사용하는 두 가지 다른 primers의 용융 곡선 분석을 보여줍니다. 그것은 녹는 온도가 두 뇌관 쌍을에 대해 서로 다른하지만 예제는 각 복제에 대해 동일한 온도에서 녹는 것은 분명합니다. 나쁜 또는 잠재적으로 오염된 시료의 흔적을 입력 두 가지 소스의 존재를 제안 몇 가지 용융 봉우리를 포함할 수 있습니다.
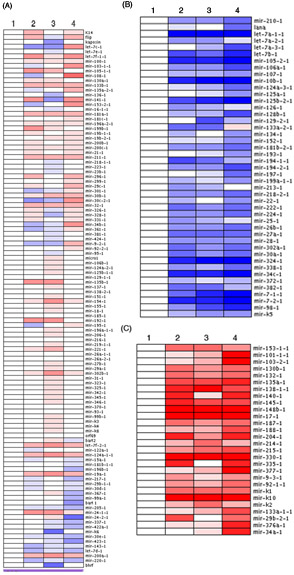
그림 1. 사전 microRNA의 서명이 소설 qPCR 기반의 배열을 사용하여 프로 파일링을 통해 등장. 평균 deltaCTU6을 계산하고 표준화된 값은 heatmaps로 표시 3 별개의 클러스터를 항복, ArrayMinerTM 소프트웨어에로드되었습니다. (A) 표현의 작은 변화를 사전 microRNAs이 표시됩니다. 그의 수준을 현저하게 감소되었다 사전 microRNAs는 (B) 또는 (C) 증가도 표시됩니다. 붉은 표현의 수준을 증가 나타냅니다 동안 블루 표현의 낮은 수준에 해당합니다.
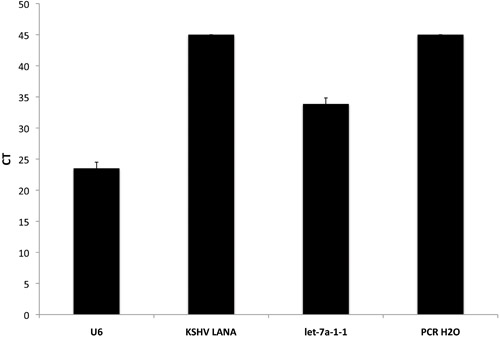
그림 2. qPCR 기반 사전 microRNA 배열의 내부 컨트롤 포함. 3 가지 유전자와 노 템플릿 컨트롤에 대한 원시 중부 표준시 값 표시됩니다. PCR H2O 우리 대조군 역할을하는 동안 U6는 내부 긍정적인 컨트롤로 사용됩니다. 하자 - 7A - 1 - 1 KSHV LANA 사용 세포주 또는 조직의 바이러스 상태에 따라 중 긍정적이거나 부정적인 컨트롤로 사용할 수 있지만 일반적으로 많은 다른 세포 유형에 표현 microRNA입니다.
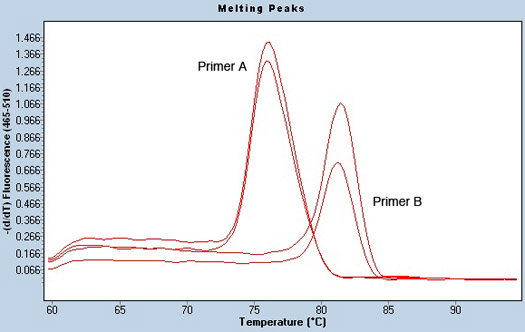
그림 3. 미리 microRNA의 primers 및 샘플 오염의 부족 피크 분석 녹는. 용융 곡선은 LightCycler 소프트웨어의 TM 전화 분석을 사용하여 얻은되었습니다. 중복에서 실행 동일한 시료에 대해 두 개의 다른 primers는 (프리머 A와 B) 표시됩니다. 각 프라이머에 대한 용해 온도가 구분됩니다. 그러나, 예제 샘플 오염의 부족을 보여주는 단 하나의 피크가 있습니다.
Access restricted. Please log in or start a trial to view this content.
토론
QPCR는 매우 민감한 연구에 샘플 사이의 유전자 발현 수준을 비교하는 데 사용할 수있는 분석뿐만 아니라 바이러스의 경우에는 양성을 결정하는 것입니다. qPCR 배열을 사용하는 장점은 짧은 기간의 각 샘플에 대해 많은 primers를 (우리의 경우, 186 프라이머 쌍)를 실행하는 능력입니다. 같은 Tecan 자유 에보 (Evo), 최대 더욱 줄일 수있는 실험을 설정하는 데 필요한 시간의 양을, 그리고 로봇의 정확성과...
Access restricted. Please log in or start a trial to view this content.
공개
감사의 말
이 작품은 NIH 보조금 DE018304, R01DE018281에 의해 지원되었다. KT는 T32 GM07092 - 34으로하고 그래드 이니셔티브에 클럽메드를 통해 하워드 휴스 의학 연구소 (HHMI)에서 채플 힐에서 노스캐롤라이나 대학에 부여에 의해 지원됩니다. PC는 T32 CA009156에 의해 지원됩니다.
Access restricted. Please log in or start a trial to view this content.
자료
| Name | Company | Catalog Number | Comments |
| Freedom Evo 150 | Tecan Group Ltd. | 30017587 | |
| Matrix Electronic Multichannel Pipette | Thermo Fisher Scientific, Inc. | 2001-MTX | (or any comparable Thermo Scientific multichannel that can pipette the volumes described above) |
| Matrix Integrity Filter Tips, Sterile | Thermo Fisher Scientific, Inc. | 7435 | (or comparable tip for multichannel used) |
| Lightcycler 480 SYBR Green 1 Master | Roche Group | 04 707 516 001 | |
| Lightcycler 480 Instrument II | Roche Group | 05015243001 | 384-well version |
| Lightcycler 480 Multiwell Plate 384, white | Roche Group | 04729749001 | |
| LightCycler 480 Sealing Foil | Roche Group | 04729757001 | |
| LightCycler 480 Multiple Plate Analysis Software | Roche Group | 05075122001 | |
| Eliminase Decontaminant | Decon Laboratories | 04-355-31 |
참고문헌
- Bustin, A. A. The MIQE Guidelines: Minimum Information for Publication of Quantitative Real-Time PCR Experiments. Clinical Chem. 55 (4), 611-622 (2009).
- Frégeau, C. J. Automated Processing of Forensic Casework Samples using Robotic Workstations Equipped with Nondisposable Tips - Contamination Prevention. J. Forensic Sci. 53 (3), 53-533 (2008).
- O'hara, A. J. Pre-micro RNA Signatures Delineate Stages of Endothelial Cell Transformation in Kaposi Sarcoma. PLoS Pathog. 5 (4), e1000389-e1000389 (2009).
Access restricted. Please log in or start a trial to view this content.
재인쇄 및 허가
JoVE'article의 텍스트 или 그림을 다시 사용하시려면 허가 살펴보기
허가 살펴보기This article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. 판권 소유