Zum Anzeigen dieser Inhalte ist ein JoVE-Abonnement erforderlich. Melden Sie sich an oder starten Sie Ihre kostenlose Testversion.
Method Article
Profiling von Pre-micro RNAs und microRNAs mittels quantitativer real-time PCR (qPCR) Arrays
In diesem Artikel
Zusammenfassung
Wir zeigen den Aufbau und die Analyse der Vor-microRNA 96-well-Arrays für die qPCR mit einem Roboter als auch von Hand mit einem Thermo Scientific Matrix Mehrkanalpipette.
Zusammenfassung
Quantitative real-time PCR (qPCR) ist als eine genaue und wertvolle Hilfe in der Profilierung der Genexpression Ebenen entstanden. Einer der vielen Vorteile ist eine untere Nachweisgrenze im Vergleich zu anderen Methoden der Genexpressions-Profiling bei der Verwendung kleiner Mengen von Input für jeden Test. Automatisierte qPCR Setup hat auf diesem Gebiet, indem sie für höhere Reproduzierbarkeit verbessert. Dank der günstigen und schnellen Aufbau ermöglicht Hochdurchsatz-Experimenten, so dass die Profilierung von vielen verschiedenen Genen gleichzeitig in jedem Experiment. Diese Methode zusammen mit internen Platte steuert reduziert auch experimentelle Variablen gemeinsam mit anderen Techniken. Wir haben vor kurzem entwickelte eine qPCR-Assay für die Profilierung von pre-microRNAs (pre-miRNAs) mit einem Satz von 186 Primerpaare. MicroRNAs haben als eine neue Klasse von kleinen, nicht-kodierende RNAs mit der Fähigkeit, viele mRNA-Ziele in der post-transkriptioneller Ebene regulieren entstanden. Diese kleinen RNAs werden zunächst durch RNA-Polymerase II als primäres miRNA (pri-miRNA) Transkript, die dann in die Vorläufer miRNA (pre-miRNA) gespalten wird transkribiert. Pre-miRNAs sind in das Zytoplasma, wo Dicer spaltet die Haarnadelschleife zu reifen miRNAs Ertrag exportiert. Erhöht in miRNA Ebenen können sowohl die Vorstufe und reifen miRNA Ebenen und Profilierung der beiden Formen beobachtet werden kann sinnvoll sein. Es gibt mehrere kommerziell erhältliche Assays für die reife miRNAs, aber ihre hohen Kosten können die Forscher von diesem Profiling-Technik abzuschrecken. Hier diskutieren wir eine kostengünstige, zuverlässige und SYBR-basierte qPCR-Methode des Profiling pre-miRNAs. Änderungen in pre-miRNA Ebenen spiegeln oft reife miRNA Änderungen und kann ein nützlicher Indikator für reife miRNA-Ausdruck sein. Allerdings kann die gleichzeitige Profilierung sowohl pre-miRNAs und reifen miRNAs optimal sein, da sie nicht-redundanten Informationen können dazu beitragen und geben Einblick in microRNA Verarbeitung. Darüber hinaus kann die hier beschriebene Technik erweitert, um die Profilierung der anderen Bibliothek Sets für spezifische Signalwege oder Krankheitserreger umfassen.
Protokoll
Der qPCR pre-miRNA Profiling Arrays können so eingerichtet werden, wie voll ausgestattet mit einem Tecan Freedom EVO-Roboter (A) oder von Hand mit der Matrix elektronischen Mehrkanalpipette (B) automatisiert.
1) Vorbereiten des Master-Mix, Primer-Platten und Proben.
- Primer-Platten mit 186 Primerpaare in 96-well-Format (insgesamt 2 Platten) am 12.05 sollte bei -80 ° C gelagert werden Thaw Platten bei Raumtemperatur, vortexen, zentrifugieren kurz vor Gebrauch.
- Zur Vorbereitung der Master-Mix, auftauen SYBR Green 2x PCR Mix bei Raumtemperatur. Jede Reaktion verwendet 8ul Master-Mix mit 2UL Primer. Die Zusammensetzung des Mastermix pro Reaktion ist 4ul SYBR-Mix, 3UL PCR-Wasser und 10-20 ng Proben-DNA oder cDNA.
- Für die Einrichtung eines jeden 96-Well-Platte Primer werden vier Master-Mix Rohre benötigt werden. Die Proben können bis zu 4 Proben pro Platte (singlicate), 2 Proben pro Platte (doppelte) oder einer einzigen Probe in vierfacher Ausfertigung ausgeführt werden.
- Bereiten Master-Mix durch die Kombination von SYBR-, Wasser-und Probe in jedem der 4-2ml Eppendorf-Röhrchen. Jedes Rohr sollte genug Mastermix für ca. 100 Reaktionen, so dass überschüssige zum Pipettieren Abfall. Vortex mischen.
A. Aufbau der pre-miRNA-Assays unter Verwendung der Freedom Tecan Evo Roboter.
- Nach der Initialisierung des Roboters und das Laden des EVOware Programm, spülen dreimal mit 30mls je System von Luftblasen, die mit Pipettiergenauigkeit stören klar.
- Richten Sie die Roboter-Plattform, die folgendes beinhalten:
- Labeled 384-Well-Platte
- 96-well-Primer Platte (1)
- Master-Mixes
- Eine Wanne mit 2% Bleichmittel
- Ein gefüllter System Fluidbehälter
- Leere Abfallbehälter
- Begin automatisierten Roboter ausführen, indem Sie "Run" zweimal.
- Am Ende des Programms, zu entfernen 384-Well-Platte und Dichtung mit LightCycler 480 Dichtfolie und kurz zentrifugieren. Ort versiegelt 384-Well-Platte in Position 1 des Hotels.
- Gehe zu 2,2 Step & Repeat für Primer 2 mit neuen Master-Mixes und ein neues 384-Well-Platte.
- Ort versiegelt Platte in Position 2 des Hotels.
- Öffnen Sie ein neues EVOware Programm für Laden der Lightcycler vom Hotel, und wählen Sie "Run" zweimal.
- Freedom Evo wird automatisch geladen jede Platte in den LightCycler und führen Sie den folgenden SYBR-Green I / HRM PCR-Programm:
Pre (1 Zyklus):
50 ° für 5 Minuten an der Rampe von 4,8 ° / sec
95 ° für 5 Minuten an der Rampe von 4,8 ° / sec
Amp (45 Zyklen):
95 ° für 15 Sekunden an der Rampe von 4,8 ° / sec
62 ° für 30 Sekunden an der Rampe von 2,5 ° / sec
(Single Datenerfassung während dieses Schritts)
Schmelzkurve:
95 ° für 5 sec an der Rampe von 4,8 ° / sec
60 ° für 1 Minute bei Anstieg von 2,5 ° / sec
95 ° kontinuierlich an der Rampe Rate von .11 ° / sec
mit 5 Akquisitionen pro ° C
Cool:
50 ° für 30 Sekunden an der Rampe von 25 ° / sec
- Freedom Evo wird automatisch geladen jede Platte in den LightCycler und führen Sie den folgenden SYBR-Green I / HRM PCR-Programm:
B. Aufbau der pre-miRNA-Assays unter Verwendung der Matrix elektronischen Mehrkanalpipette:
- Legen Sie Inhalte des Master-Mix Röhre 1 in ein Reservoir.
- Stellen Sie die elektronische Pipette absaugen 16ul des Master-Mix mit 2-8ul verzichten Schritte von einem Spülschritt gefolgt.
- Für Master-Mix 1, verzichten 8ul in jeden anderen auch der 384-Well-Platte (Set auf 384-well Spitze Abstand), beginnend mit gut A1. (Dh Brunnen A1, C1, E1, etc.) Für das zweite 8ul verzichten, gehen Sie zu Spalte 3 (dh Brunnen A3, C3, E3, etc.), dann purge über einen Abfallbehälter und entsorgen Tipps.
- Folgen Sie diesem Muster für die restlichen 5 Zyklen, bis Sie auch A23 zu erreichen.
- Wiederholen Sie dies für Master-Mix 2, (beginnend mit gut A2, C2, E2, etc.); Master-Mix 3 (Anfang an gut B1, D1, F1, etc.); Master-Mix 4 (ab B2, D2, F2, usw. .).
- Für Aliquotierung der Primer Platte, setzen elektronische Pipette absaugen und 2UL verzichten 2UL mit einem Spülschritt folgenden. Dispense 2UL von Primern aus Spalte 1 der 96-Well-Platte Primer (AH) in den 384-Well-Platte, Brunnen A1, C1, E1, etc. Purge über einen Abfallbehälter und entsorgen Tipps. Wiederholen Sie für Brunnen A2, C2, E2, B1, D1, F1, und B2, D2, F2, etc.
- Wiederholen Sie für die Zeilen 2-12 der 96-Well-Primer, schwebte über alle 2 Spalten für jeden Primer Spalte bis zur vollständigen 384-Well-Platte wurden aliquotiert hat.
- Seal-Platten mit einem LightCycler Dichtfolie und kurz zentrifugieren.
- Legen Platten in eine LightCycler 480 und radeln die Platten nach der folgenden Einstellungen mit dem SYBR Green I / HRM Erkennung Format:
Pre (1 Zyklus):
50 ° für 5 Minuten an der Rampe von 4,8 ° / sec
95 ° für 5 Minuten an der Rampe von 4,8 ° / sec
Amp (45 Zyklen):
95 ° für 15 sec bei Anstiegsgeschwindigkeitvon 4,8 ° / sec
62 ° für 30 Sekunden an der Rampe von 2,5 ° / sec
(Single Datenerfassung während dieses Schritts)
Schmelzkurve:
95 ° für 5 sec an der Rampe von 4,8 ° / sec
60 ° für 1 Minute bei Anstieg von 2,5 ° / sec
95 ° kontinuierlich an der Rampe Rate von .11 ° / sec
mit 5 Akquisitionen pro ° C
Cool:
50 ° für 30 Sekunden an der Rampe von 25 ° / sec - Wiederholen Sie die Verwendung von Primer Platte 2, Aliquotierung in ein frisches 384-Well-Platte mit frisch zubereiteten Mastermixen.
Geheimnisse zum Erfolg:
Ein wichtiges Merkmal bei der Gestaltung PCR-Arrays ist, dass Alle 186 Primerpaare die gleiche Annealingtemperatur haben, so dass die Platte bei der gleichen optimalen PCR-Lauf-Programm-Einstellungen können ausgeführt werden.
Jede neue Array sollte mithilfe drei Kontrollen vor dem Ausführen von Proben. Diese Kontrollen sind:
1 laufen alle Primer mit Wasser oder .1 x TE ausführen, um auszuschließen, Primer Kontamination.
1 von wechselnden Wasser / TE und der positiven Kontrolle laufen, keine Verschleppung zu gewährleisten.
3 läuft mit der gleichen positiven Kontrolle, um die Reproduzierbarkeit zwischen den Läufen zu gewährleisten.
Master-Mix sollte frisch zubereitet werden, und die Platten sollten innerhalb von 24 Stunden für optimale Ergebnisse ausgeführt werden
Primer Platte Folie sollte langsam entfernt werden, um das Risiko von Kontaminationen zwischen den Wells zu reduzieren.
Bei Verwendung eines Roboters mit festen Spitzen, 2% Bleichmittel waschen notwendig, um die Spitzen zwischen den einzelnen Pipettierschritt, durch eine Wasserspülung gefolgt waschen. Dies verhindert und beseitigt Verschleppung, die sonst kontaminieren könnten Daten und führen zu eindeutigen Ergebnisse.
Nach einer Platte durch die Lightcycler ausgeführt wird, sollte es nicht wieder in die PCR Setup Raum geöffnet werden. Dies verhindert, dass PCR Kontamination.
Repräsentative Ergebnisse:
qPCR Ergebnisse sind in der Regel durch CT-Werte als von der Lightcycler Analyse-Software ermittelt vertreten. Ein erfolgreicher laufen in der Regel aus einer Reihe von CTs für Proben, in der Regel zwischen 20-35 für Proben, die positiv sind. Die Wasserproben in einem guten Lauf sind immer an einem CT von> 40 und jedes Muster oder spezielle Vertiefungen mit einer CT über 37 sind als negativ oder nicht erkannt. In der Regel werden die Proben wodurch ein CT von <10 auch unzuverlässig und werden von der weiteren Analyse ausgeschlossen. Es gibt mehrere Möglichkeiten, qPCR-Daten und die Einbindung der internen Kontrollen in unserem Test zu analysieren hilft bei der Kontrolle für die Variation der Probe-Eingang. Die Pre-miR-Array enthält eine U6-Grundierung, die oft als Referenz-Gen auf CT-Werte von verschiedenen Proben zu normalisieren wird. Dieser normierte Wert wird als Delta-CT-Wert (DCT) bezeichnet. Die Ergebnisse werden oft als das rohe CT, die DCT-Wert graphisch dargestellt oder kann weiter auf standardisierte Werte analysiert werden.
In Abbildung 1, die Analyse der volle Vor-microRNA Array von vier verschiedenen Proben aus einem Zeitverlauf Experiment ist in Heat Map-Format präsentiert. Die relative Expression jedes pre-miRNA wird gezeigt und drei große, unterschiedliche Cluster entstanden. Die Mehrheit der pre-Mirs analysiert unterzog kleine, unbedeutende Änderungen wie erwartet (Abbildung 1A). Allerdings waren ein kleiner Teil der Pre-microRNAs signifikant verringert (Abbildung 1B) oder dramatisch zugenommen (Abbildung 1C) in der gesamten Zeitverlauf zu experimentieren. Ebenen des Ausdrucks der einzelnen Vor-microRNA wurden die 0h Zeitpunkt (Probe 1) als Grundlage Niveau normalisiert. In einigen Fällen können Sie feststellen, dass einige der Cluster-pre-microRNAs auch Cluster zusammen in der Heatmap-Analyse (ex: let-7a, Abbildung 1B). Je nach Experiment, Cluster entstehen kann, dass abhängig sind Virusinfektion, Zelltyp spezifische Expression von pre-microRNAs oder Pre-Mirs, dass durch ein gemeinsames Molekül oder-Signalweg reguliert werden.
Die pre-miRNA-Test, dass wir auch entwickelt haben, enthält eine Reihe von bekannten viralen pre-microRNAs und Gene, die durch das Kaposi-Sarkom assoziierten Herpesvirus (KSHV) und Epstein-Barr-Virus (EBV) in Ergänzung zu den menschlichen Vor-Mirs kodiert. Diese können als positive oder negative Kontrollen verwendet werden, je nach Zelllinie und virale Status der Proben verwendet. In Abbildung 2 sind die durchschnittlichen CTs von einem KSHV-negative Probe in vierfacher Ausführung gezeigt. Wichtig ist, dass KSHV LANA nicht in diesem Beispiel durch die hochsensitive qPCR-Arrays detektiert. Allerdings U6 - unsere internen Kontroll-und let-7a - waren ein hoch exprimierten humanen Pre-microRNA, in nachweisbaren Mengen exprimiert. Schließlich, wie erwartet, hat die negative Kontrolle der PCR-grade Wasser nicht zu einem qPCR Produkt.
Ein weiterer Indikator für eine erfolgreiche qPCR laufen ist ein guter Schmelzkurvenanalyse, die Einblick in die mögliche Kontamination in den Proben geben kann. Um eine Schmelztemperatur wird die Platte langsam nach der PCR ist komplett beheizt. Da die DNA-Stränge zu trennen, wird SYBR green veröffentlicht und Fluoreszenz-Signal abnimmt. Die Temperatur, bei der diese drop auftritt, ist grafisch dargestellt, und ist als die Schmelztemperatur der Probe bekannt. Abhängig von der DNA-Sequenz, Proben bei verschiedenen Temperaturen voneinander und aus dem Wasser negative Kontrolle, die nicht über eine Schmelztemperatur, da keine DNA vorhanden ist, zu schmelzen. Die PCR-Produkte von jedem Primerpaar sollte bei ähnlichen Temperaturen schmelzen. Zum Beispiel zeigt Abbildung 3 a Schmelzkurvenanalyse für zwei verschiedene Primer mit der gleichen Probe in zweifacher Ausfertigung. Es ist klar, dass die Schmelztemperatur für die beiden Primerpaare ist aber die Probe auf die exakt gleiche Temperatur schmelzenden für jede Wiederholung. Anzeichen für eine schlechte oder potenziell kontaminierten Probe kann mehrere Schmelzpeaks, was auf die Anwesenheit von zwei verschiedenen Quellen der Eingabe.
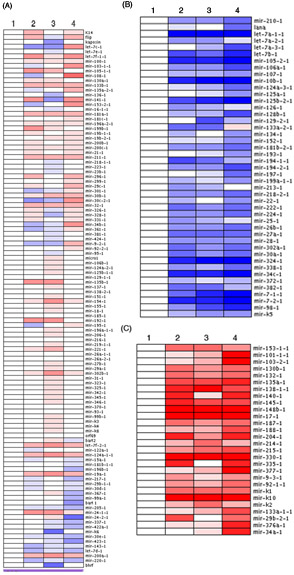
Abbildung 1. Pre-microRNA Signaturen entstehen durch Profilierung mit einem neuartigen qPCR-basierte Arrays. Die durchschnittliche deltaCTU6 wurde berechnet und standardisierte Werte wurden in ArrayMinerTM Software geladen, was zu 3 verschiedene Cluster als Heatmaps angezeigt. (A) Pre-microRNAs mit kleinen Veränderungen in der Expression gezeigt werden. Pre-microRNAs, deren Pegel deutlich zurückgegangen (B) oder erhöht (C) sind ebenfalls dargestellt. Blau entspricht unteren Ebenen des Ausdrucks, während rote Expression erhöht ist.
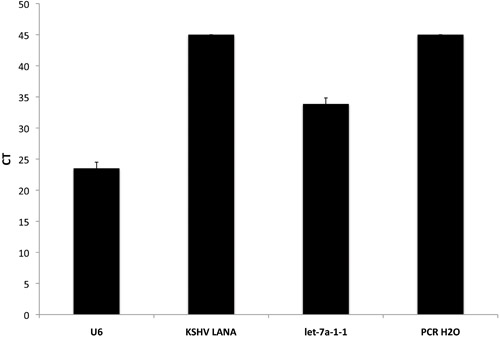
Abbildung 2. Einbeziehung der internen Kontrollen in der qPCR-basierte Pre-microRNA Array. Die rohen CT-Werte für 3 verschiedene Gene und keine Vorlage Kontrolle angezeigt. U6 wird als interne positive Kontrolle verwendet, während PCR H2O dient uns als negative Kontrolle. Let-7a-1-1 ist eine microRNA normalerweise in vielen verschiedenen Zelltypen exprimiert, während KSHV LANA entweder eine positive oder negative Kontrolle, auf die virale Status der Zelllinie oder Gewebe verwendet Basis verwendet werden können.
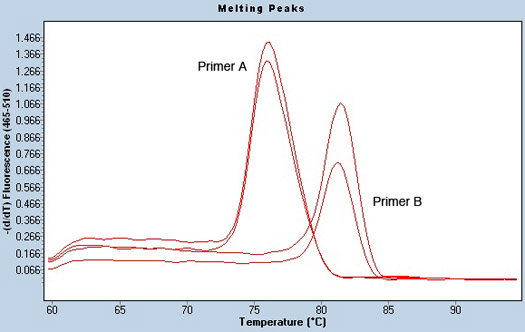
Abbildung 3. Schmelzpeak Analyse der Vor-microRNA Primer und der Mangel an Probenkontamination. Schmelzkurven wurden mit dem Tm Aufruf Analyse in der LightCycler-Software. Zwei verschiedene Primer (Primer A und B) für die gleiche Probe in zweifacher Ausführung gezeigt. Die Schmelztemperatur für jedes Primerpaar unterscheidet. Allerdings hat die Probe nur einen Peak, was die mangelnde Probenkontamination.
Access restricted. Please log in or start a trial to view this content.
Diskussion
QPCR ist ein hochempfindliches Verfahren, die zur Genexpression zwischen den Proben vergleichen in einer Studie werden können, sowie Positivität im Falle von Viren zu bestimmen. Der Vorteil der Verwendung von qPCR-Arrays ist die Fähigkeit, viele Primer (in unserem Fall, 186 Primerpaare) für jede Probe laufen in einem kurzen Zeitraum. Mit einem Pipettierroboter wie die Tecan Freedom EVO, der viel Zeit benötigt, um ein Experiment zu weiter reduziert werden kann, festgelegt, und die Genauigkeit und Konsistenz des Robo...
Access restricted. Please log in or start a trial to view this content.
Offenlegungen
Danksagungen
Diese Arbeit wurde vom NIH gewährt DE018304, R01DE018281 unterstützt. KT wird durch T32 GM07092-34 und durch einen Zuschuss an die University of North Carolina at Chapel Hill vom Howard Hughes Medical Institute (HHMI) durch die Med in Grad Initiative unterstützt. PC wird durch T32 CA009156 unterstützt.
Access restricted. Please log in or start a trial to view this content.
Materialien
| Name | Company | Catalog Number | Comments |
| Freedom Evo 150 | Tecan Group Ltd. | 30017587 | |
| Matrix Electronic Multichannel Pipette | Thermo Fisher Scientific, Inc. | 2001-MTX | (or any comparable Thermo Scientific multichannel that can pipette the volumes described above) |
| Matrix Integrity Filter Tips, Sterile | Thermo Fisher Scientific, Inc. | 7435 | (or comparable tip for multichannel used) |
| Lightcycler 480 SYBR Green 1 Master | Roche Group | 04 707 516 001 | |
| Lightcycler 480 Instrument II | Roche Group | 05015243001 | 384-well version |
| Lightcycler 480 Multiwell Plate 384, white | Roche Group | 04729749001 | |
| LightCycler 480 Sealing Foil | Roche Group | 04729757001 | |
| LightCycler 480 Multiple Plate Analysis Software | Roche Group | 05075122001 | |
| Eliminase Decontaminant | Decon Laboratories | 04-355-31 |
Referenzen
- Bustin, A. A. The MIQE Guidelines: Minimum Information for Publication of Quantitative Real-Time PCR Experiments. Clinical Chem. 55 (4), 611-622 (2009).
- Frégeau, C. J. Automated Processing of Forensic Casework Samples using Robotic Workstations Equipped with Nondisposable Tips - Contamination Prevention. J. Forensic Sci. 53 (3), 53-533 (2008).
- O'hara, A. J. Pre-micro RNA Signatures Delineate Stages of Endothelial Cell Transformation in Kaposi Sarcoma. PLoS Pathog. 5 (4), e1000389-e1000389 (2009).
Access restricted. Please log in or start a trial to view this content.
Nachdrucke und Genehmigungen
Genehmigung beantragen, um den Text oder die Abbildungen dieses JoVE-Artikels zu verwenden
Genehmigung beantragenWeitere Artikel entdecken
This article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Alle Rechte vorbehalten