É necessária uma assinatura da JoVE para visualizar este conteúdo. Faça login ou comece sua avaliação gratuita.
Method Article
Perfil dos pré-micro RNAs e microRNAs usando Quantitative PCR em tempo real (qPCR) Arrays
Neste Artigo
Resumo
Vamos demonstrar a configuração e análise de microRNA pré-96 também matrizes para QPCR usando um robô, bem como à mão com uma pipeta multicanal Thermo Scientific Matrix.
Resumo
Quantitativas PCR em tempo real (qPCR) surgiu como uma ferramenta precisa e valiosa em perfis níveis de expressão de genes. Uma de suas muitas vantagens é um limite inferior de detecção em comparação com outros métodos de perfil de expressão gênica durante o uso de quantidades menores de entrada para cada ensaio. QPCR configuração automatizada tem melhorado neste campo, permitindo maior reprodutibilidade. Sua configuração conveniente e rápida permite high-throughput experimentos, permitindo a caracterização de muitos genes diferentes simultaneamente em cada experimento. Este método, juntamente com os controles internos da placa também reduz variáveis experimentais comuns a outras técnicas. Recentemente, desenvolveu um ensaio de qPCR para o perfil de pré-microRNAs (pré-miRNAs), utilizando um conjunto de 186 pares de primers. MicroRNAs surgiram como uma nova classe de pequenos RNAs não-codificadores com a capacidade de regular as metas mRNA muitos ao nível pós-transcricional. Estes pequenos RNAs são transcritos primeiro pela RNA polimerase II como um miRNA primário (pri-miRNA) transcrição, que é então clivada para o miRNA precursor (pré-miRNA). Pré-miRNAs são exportados para o citoplasma, onde Dicer cliva o loop de hairpin para produzir miRNAs maduros. Aumento dos níveis de miRNA pode ser observado em ambas as precursor do miRNA maduro e níveis e perfis de ambas as formas podem ser úteis. Existem vários testes disponíveis comercialmente para miRNAs maduros, no entanto, seu alto custo pode dissuadir os pesquisadores a partir dessa técnica de perfil. Aqui, discutimos um custo-benefício, confiança, método qPCR SYBR baseada em perfis pré-miRNAs. Mudanças nos níveis pré-miRNA muitas vezes refletir as mudanças miRNA maduro e pode ser um indicador útil de expressão de miRNA maduro. No entanto, o perfil simultânea de ambos os pré-miRNAs e miRNAs maduros pode ser ótimo como eles podem contribuir com informações não redundantes e fornecer informações sobre processamento de microRNA. Além disso, a técnica descrita aqui pode ser expandido para englobar a caracterização de conjuntos de outra biblioteca para vias específicas ou patógenos.
Protocolo
A qPCR arrays pré-miRNA perfil pode ser configurado como totalmente automatizada com uma liberdade Tecan Evo robô (A) ou à mão com a pipeta multicanal Matrix eletrônico (B).
1) Prepare a mistura principal, placas primer e amostras.
- Primer placas contendo 186 pares de primers em formato de 96 poços (total de 2 placas) em 12:05 devem ser armazenadas a -80 ° C. Placas descongelar em temperatura ambiente, vortex e centrifugar brevemente antes de usar.
- Para preparar o mix master, descongelar SYBR Green 2x Mix PCR em temperatura ambiente. Cada reação usa master mix 8ul com primer 2ul. A composição do mix de mestre por reação é mix SYBR 4ul, 3ul água grau PCR e 10-20 ng de DNA da amostra ou cDNA.
- Para a configuração de cada placa cartilha de 96 poços, quatro tubos master mix serão necessários. Amostras pode ser executado quatro amostras por chapa (singlicate), 2 amostras por placa (duplicata) ou uma única amostra em quadruplicado.
- Prepare master mix, combinando água SYBR, e amostra em cada um dos tubos de 4 2ml Eppendorf. Cada tubo deve conter master mix suficiente para cerca de 100 reações, permitindo que o excesso de resíduos de pipetagem. Vortex para misturar.
A. Configuração de ensaio pré-miRNA usando o Freedom Tecan Evo robô.
- Após a inicialização do robô e carregamento do programa Evoware, lave três vezes com cada 30mls para limpar sistema de bolhas de ar que irá interferir com pipetagem precisão.
- Instalação da plataforma do robô para incluir o seguinte:
- Rotulados placa de 384 poços
- De 96 poços da placa primer (1)
- Mestre mistura
- A calha contendo lixívia 2%
- Um recipiente cheio de líquido do sistema
- Recipiente vazio
- Começar a executar robô automatizado, selecionando "Run" duas vezes.
- No final do programa, remova 384 prato e bem-selo com LightCycler folha de selagem 480 e centrifugar brevemente. Lugar selado placa de 384 poços na posição 1 do hotel.
- Vá para o passo 2.2 e repita para placa de primer 2 com mixes novo mestre e um novo prato de 384 poços.
- Coloque placa colada na posição 2 do hotel.
- Abra o programa Evoware novo para carregar o LightCycler do hotel e selecione "Run" duas vezes.
- Liberdade Evo carregará automaticamente cada placa no LightCycler e executar o seguinte SYBR verde-I / HRM programa de ciclos:
Pre (1 ciclo):
50 ° por 5 minutos a taxa de aumento de 4,8 ° / seg
95 ° por 5 minutos a taxa de aumento de 4,8 ° / seg
Amp (45 ciclos):
95 ° por 15 segundos a taxa de rampa de 4,8 ° / seg
62 ° por 30 segundos a uma taxa de rampa de 2,5 ° / seg
(Aquisição de dados único durante esta etapa)
Curva de fusão:
95 ° por 5 segundos a taxa de rampa de 4,8 ° / seg
60 ° para 1 minuto a taxa de aumento de 2,5 ° / seg
95 ° contínua a taxa de aumento de 0,11 ° / seg
com 5 aquisições por ° C
Cool:
50 ° por 30 segundos a uma taxa de rampa de 25 ° / seg
- Liberdade Evo carregará automaticamente cada placa no LightCycler e executar o seguinte SYBR verde-I / HRM programa de ciclos:
B. Instalação de ensaio pré-miRNA usando o Matrix pipeta multicanal electrónica:
- Conteúdo lugar de mestre tubo misture 1 em um reservatório.
- Definir a pipeta eletrônica para aspirar 16ul de mix master com 2-8ul dispensar passos seguidos por uma etapa de purga.
- Para mestre misture 1, dispensar 8ul em todos os outros bem da placa de 384 poços (conjunto de 384 bem-espaçamento ponta), começando bem A1. (Ie poços A1, C1, E1, etc) Para o segundo 8ul dispensar, mover para a coluna 3 (ie poços A3, C3, E3, etc), então purge ao longo de um recipiente de resíduos e dicas dispor.
- Siga este padrão para os restantes 5 ciclos até chegar bem A23.
- Repita o procedimento para mestre mix 2, (começando com A2 bem, C2, E2, etc); mestre mix 3 (início em B1 bem, D1, F1, etc); mestre mix 4 (início em B2, D2, F2, etc .).
- Para aliquotting de placa primer, conjunto pipeta eletrônica para aspirar e dispensar 2ul 2ul com um seguinte passo de purga. Dispense 2ul de primers da coluna 1 de 96 poços da placa primer (AH) na placa de 384 poços, poços A1, C1, E1, etc Purge ao longo de um recipiente de resíduos e dicas dispor. Repita o procedimento para poços A2, C2, E2, B1, D1, F1 e B2, D2, F2, etc
- Repita para as linhas 2-12 de primers de 96 poços, deslocando-se a cada 2 colunas para cada coluna primer, até placa de 384 poços completo foi aliquotado.
- Placas de vedação com uma folha de selagem LightCycler e centrifugar brevemente.
- Placas em um lugar LightCycler 480 e ciclo das placas de acordo com as seguintes configurações utilizando o SYBR Green I / HRM formato de detecção:
Pre (1 ciclo):
50 ° por 5 minutos a taxa de aumento de 4,8 ° / seg
95 ° por 5 minutos a taxa de aumento de 4,8 ° / seg
Amp (45 ciclos):
95 ° por 15 segundos a taxa de rampade 4,8 ° / seg
62 ° por 30 segundos a uma taxa de rampa de 2,5 ° / seg
(Aquisição de dados único durante esta etapa)
Curva de fusão:
95 ° por 5 segundos a taxa de rampa de 4,8 ° / seg
60 ° para 1 minuto a taxa de aumento de 2,5 ° / seg
95 ° contínua a taxa de aumento de 0,11 ° / seg
com 5 aquisições por ° C
Cool:
50 ° por 30 segundos a uma taxa de rampa de 25 ° / seg - Repita usando placa de primer 2, aliquotting em uma placa nova de 384 poços usando mistura mestre preparados na hora.
Segredos para o sucesso:
Uma característica importante ao projetar matrizes PCR é que todos os 186 pares de primers têm a mesma temperatura de recozimento, de modo que o prato pode ser executado no mesmo ideal PCR configurações do programa ser executado.
Cada nova matriz deve ser verificado utilizando três controles antes de amostras em execução. Esses controles são:
Uma execução de todos os primers executar com água ou 0,1 x TE para impedir a contaminação primer.
1 execução do alternando água / TE e controle positivo, para garantir que nenhum reporte.
3 é executado usando o mesmo controle positivo, para garantir a reprodutibilidade entre as execuções.
Mestre mistura deve ser preparada, e as placas devem ser executados dentro de 24 horas para melhores resultados
Folha de chapa de primer deve ser removido lentamente a reduzir o risco de contaminação entre poços.
Ao usar um robô com pontas fixas, lave bleach 2% é necessário lavar as pontas entre cada etapa de pipetagem, seguido por uma lavagem de água. Isso evita que e elimina transição, que poderiam contaminar os dados e levar a resultados inconclusivos.
Depois de uma placa é executado através do LightCycler, não deve ser aberta novamente na sala de configuração PCR. Isso ajuda a evitar a contaminação da PCR.
Resultados representativos:
qPCR resultados normalmente são representados por valores CT como determinado a partir do software de análise LightCycler. A temporada de sucesso geralmente consiste de uma série de CTs para amostras, geralmente entre 20-35 para as amostras que são positivas. As amostras de água em uma boa corrida estão sempre em um CT de> 40 e quaisquer amostras ou poços específico com um CT mais de 37 são considerados negativos ou não detectado. Como regra geral, as amostras produzindo um CT de <10 também são confiáveis e estão excluídos da análise posterior. Existem várias maneiras de analisar os dados qPCR ea inclusão de controles internos em nosso teste ajuda a controlar a variação de entrada de amostra. A matriz pré-miR inclui uma cartilha controle U6, que é frequentemente usado como um gene de referência para normalizar valores CT a partir de amostras diferentes. Este valor normalizado é referido como o delta CT valor (DCT). Resultados são representadas graficamente como o CT-primas, o valor DCT ou pode ainda ser analisada a valores padronizados.
Na Figura 1, a análise da matriz de pré microRNA-cheia de quatro amostras diferentes de um experimento timecourse é apresentado em formato de mapa de calor. A expressão relativa de cada pré-microRNA é mostrado e três grandes, grupos distintos surgiram. A maioria dos pré-Mirs analisados foram submetidos a mudanças pequenas e insignificantes como esperado (Figura 1A). No entanto, uma pequena parte da pré-microRNAs foram significativamente menores (Figura 1B) ou aumentaram dramaticamente (Figura 1C) durante todo o experimento timecourse. Níveis de expressão de cada pré-microRNA foram normalizados para o ponto no tempo 0h (amostra 1) como um nível basal. Em alguns casos, você pode notar que alguns dos pré-cluster microRNAs também se agrupam na análise heatmap (ex: deixe-7a Figura 1B). Dependendo do experimento, os clusters podem surgir que são dependentes de infecção viral, a expressão de células tipo específico de pré-microRNAs ou pré-Mirs que são regulados por uma molécula comum ou via de sinalização.
O ensaio de pré-microRNA que temos desenvolvido também inclui uma série de conhecidos viral pré-microRNAs e genes codificados pelo sarcoma de Kaposi Associated Herpesvirus (KSHV) e Epstein Barr (EBV), para além do humano pré-Mirs. Estes podem ser utilizados como controle positivo ou negativo, dependendo da linha de celular e status viral das amostras utilizadas. Na Figura 2, os TCs média de uma amostra KSHV-negativos executado em quatro vias são mostrados. Importante, KSHV LANA não foi detectado nesta amostra pela matriz qPCR altamente sensível. No entanto, U6 - nossos controles internos e deixe-7a - uma altamente expressa humana pré-microRNA, foram expressas em níveis detectáveis. Finalmente, como esperado, o controle negativo da PCR grau água não deu um produto qPCR.
Outro indicador de uma corrida qPCR de sucesso é uma boa análise da curva de fusão, que pode dar dicas sobre possível contaminação nas amostras. Para estabelecer uma temperatura de fusão, a placa é aquecida lentamente após a PCR é completa. Como as fitas de DNA separadas, SYBR green é liberada, e sinal de fluorescência diminui. A temperatura na qual este drop ocorre é gráfico, e é conhecida como a temperatura de fusão da amostra. Dependendo da seqüência de DNA, as amostras de derreter em temperaturas diferentes uns dos outros e do controle da água negativa, o que não deve ter uma temperatura de fusão já que nenhum DNA está presente. Os produtos de PCR de cada par de primer deve derreter em temperaturas semelhantes. Por exemplo, a Figura 3 mostra uma análise da curva de fusão por dois primers diferentes, utilizando a mesma amostra em duplicata. É claro que a temperatura de fusão é diferente para os dois pares de primers, mas a amostra está derretendo na temperatura exata mesma para cada repetição. Sinais de uma amostra ruim ou potencialmente contaminados podem incluir vários picos de fusão, sugerindo a presença de duas fontes diferentes de entrada.
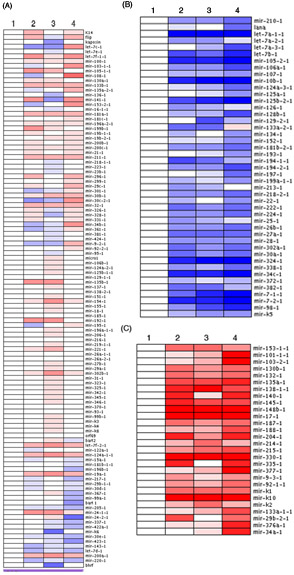
Figura 1. Pré-microRNA assinaturas emergir através de perfis usando um qPCR baseado romance matriz. O deltaCTU6 média foi calculada e os valores padronizados foram carregados no software ArrayMinerTM, gerando três grupos distintos exibido como heatmaps. (A) Pré-microRNAs com pequenas alterações na expressão são mostrados. Pré-microRNAs cujos níveis foram significativamente menores (B) ou aumentada (C) também são mostrados. Azul corresponde aos níveis mais baixos de expressão enquanto o vermelho representa o aumento dos níveis de expressão.
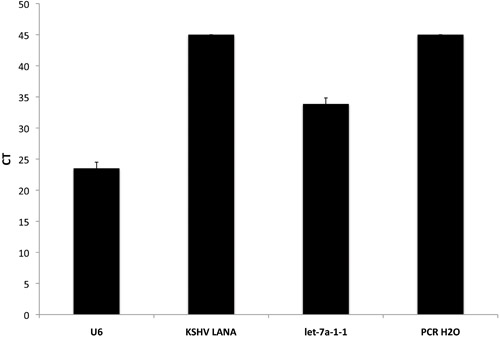
Figura 2. Inclusão de controles internos na qPCR baseado conjunto pré-microRNA. Os valores brutos CT por 3 genes diferentes e um modelo de controle não são mostradas. U6 é usado como um controle interno positivo, enquanto PCR H2O serve como nosso controle negativo. Deixe-7-1-1 é um microRNA normalmente expressa em muitos tipos diferentes de células, enquanto KSHV LANA pode ser usado como um controle positivo ou negativo, com base no status viral da linha de célula ou tecido usado.
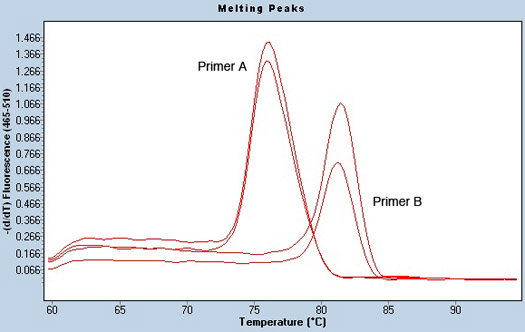
Figura 3. Melting análise do pico de microRNA pré-primers e falta de contaminação da amostra. Curvas de fusão foram obtidos utilizando a análise chamando Tm no software LightCycler. Dois primers diferentes (Primer A e B) para a mesma amostra executado em duplicado são mostrados. A temperatura de fusão para cada primer é distinta. No entanto, a amostra tem apenas um pico, demonstrando a falta de contaminação da amostra.
Access restricted. Please log in or start a trial to view this content.
Discussão
QPCR é um método altamente sensível que pode ser usado para comparar os níveis de expressão gênica entre as amostras em estudo, bem como para determinar a positividade no caso de vírus. A vantagem de usar arrays qPCR é a capacidade de executar muitos primers (no nosso caso, 186 pares de primers) para cada amostra em um curto período de tempo. Usando um robô de pipetagem, como o Freedom Tecan Evo, a quantidade de tempo necessário para definir um experimento up ainda pode ser reduzido, ea precisão ea consistê...
Access restricted. Please log in or start a trial to view this content.
Divulgações
Agradecimentos
Este trabalho foi financiado pelo NIH concede DE018304, R01DE018281. KT é apoiada por GM07092 T32-34 e por uma concessão para a University of North Carolina em Chapel Hill do Howard Hughes Medical Institute (HHMI) através do Med em Grad Initiative. PC é suportado pelo T32 CA009156.
Access restricted. Please log in or start a trial to view this content.
Materiais
| Name | Company | Catalog Number | Comments |
| Freedom Evo 150 | Tecan Group Ltd. | 30017587 | |
| Matrix Electronic Multichannel Pipette | Thermo Fisher Scientific, Inc. | 2001-MTX | (or any comparable Thermo Scientific multichannel that can pipette the volumes described above) |
| Matrix Integrity Filter Tips, Sterile | Thermo Fisher Scientific, Inc. | 7435 | (or comparable tip for multichannel used) |
| Lightcycler 480 SYBR Green 1 Master | Roche Group | 04 707 516 001 | |
| Lightcycler 480 Instrument II | Roche Group | 05015243001 | 384-well version |
| Lightcycler 480 Multiwell Plate 384, white | Roche Group | 04729749001 | |
| LightCycler 480 Sealing Foil | Roche Group | 04729757001 | |
| LightCycler 480 Multiple Plate Analysis Software | Roche Group | 05075122001 | |
| Eliminase Decontaminant | Decon Laboratories | 04-355-31 |
Referências
- Bustin, A. A. The MIQE Guidelines: Minimum Information for Publication of Quantitative Real-Time PCR Experiments. Clinical Chem. 55 (4), 611-622 (2009).
- Frégeau, C. J. Automated Processing of Forensic Casework Samples using Robotic Workstations Equipped with Nondisposable Tips - Contamination Prevention. J. Forensic Sci. 53 (3), 53-533 (2008).
- O'hara, A. J. Pre-micro RNA Signatures Delineate Stages of Endothelial Cell Transformation in Kaposi Sarcoma. PLoS Pathog. 5 (4), e1000389-e1000389 (2009).
Access restricted. Please log in or start a trial to view this content.
Reimpressões e Permissões
Solicitar permissão para reutilizar o texto ou figuras deste artigo JoVE
Solicitar PermissãoExplore Mais Artigos
This article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Todos os direitos reservados