È necessario avere un abbonamento a JoVE per visualizzare questo. Accedi o inizia la tua prova gratuita.
Method Article
Profilatura di pre-micro-RNA e microRNA con Quantitative Real-time PCR (qPCR) Array
* Questi autori hanno contribuito in egual misura
In questo articolo
Riepilogo
Dimostreremo la configurazione e l'analisi di pre-microRNA 96-array e per QPCR usando un robot così come a mano con una pipetta multicanale Thermo Scientific Matrix.
Abstract
Quantitativa real-time PCR (QPCR) è emerso come uno strumento preciso e prezioso nel profilo livelli di espressione genica. Uno dei suoi vantaggi è un limite di rilevazione più basso rispetto ad altri metodi di profili di espressione genica durante l'utilizzo di piccole quantità di ingresso per ciascun test. Automatizzate di configurazione qPCR ha migliorato questo settore, consentendo una maggiore riproducibilità. La sua configurazione comoda e rapida permette di high-throughput esperimenti, che consente la profilazione di molti geni diversi simultaneamente in ogni esperimento. Questo metodo insieme ai controlli piastra interna riduce anche variabili sperimentali comuni ad altre tecniche. Abbiamo recentemente sviluppato un test qPCR per la profilatura di pre-microRNA (pre-miRNA) utilizzando un set di 186 coppie di primer. I microRNA sono emersi come una nuova classe di piccoli RNA non codificanti, con la possibilità di regolare molti mRNA target a livello post-trascrizionale. Questi piccoli RNA vengono prima trascritti dalla RNA polimerasi II come un miRNA primario (pri-miRNA) trascrizione, che viene poi tagliata in miRNA precursore (pre-miRNA). Pre-miRNA vengono esportati nel citoplasma dove Dicer unirà il ciclo tornante cedere miRNA maturi. Aumenti dei livelli di miRNA possono osservare sia il precursore e livelli di miRNA maturo e profilatura di entrambe queste forme possono essere utili. Ci sono diversi test disponibili in commercio per il miRNA maturo, ma il loro costo elevato potrebbe scoraggiare i ricercatori questa tecnica di profilatura. Qui, si discute una soluzione conveniente, affidabile, SYBR metodo basato qPCR di profili pre-miRNA. Cambiamenti nei livelli di pre-miRNA spesso riflettere i cambiamenti miRNA maturo e può essere un utile indicatore di espressione miRNA maturi. Tuttavia, profilazione simultanea di entrambi i pre-miRNA e miRNA maturo può essere ottimale in quanto possono contribuire le informazioni ridondanti e permettono di percepire il trattamento di microRNA. Inoltre, la tecnica qui descritta può essere ampliata fino a comprendere la profilazione dei set di librerie per altri percorsi specifici o agenti patogeni.
Protocollo
Il pre-miRNA array profiling qPCR può essere impostato come completamente automatico con una libertà Tecan Evo robot (A) oppure a mano con la pipetta multicanale elettronica Matrix (B).
1) Preparare la master mix, piatti primer e campioni.
- Piastre di fondo contenente 186 coppie di primer in formato a 96 pozzetti (totale di 2 piastre) a 12:05 devono essere conservati a -80 ° C. Scongelare le piastre a temperatura ambiente, vortex e centrifugare brevemente prima dell'uso.
- Per preparare il mix master, disgelo fuori SYBR Green 2x Mix PCR a temperatura ambiente. Ogni reazione usa mix 8ul maestro con primer 2ul. La composizione del master mix per ogni reazione viene mix SYBR 4UL, 3UL acqua grado PCR e 10-20 ng campione di DNA o cDNA.
- Per la configurazione di ogni piatto fondo da 96 pozzetti, quattro tubi master mix sarà necessario. I campioni possono essere eseguiti 4 campioni per piastra (singlicate), 2 campioni per piastra (duplicato) o un singolo campione in quattro esemplari.
- Preparare master mix unendo SYBR, acqua e campione in ciascuno di 4-2 ml provette Eppendorf. Ogni tubo deve contenere mix abbastanza principale per circa 100 reazioni, permettendo in eccesso per pipettaggio rifiuti. Vortex per mescolare.
A. Configurazione di pre-miRNA test utilizzando il Freedom Tecan Evo robot.
- A seguito di inizializzazione del robot e il caricamento del programma Evoware, lavare tre volte con ogni 30mls per cancellare sistema di bolle d'aria che interferiscono con la precisione di pipettaggio.
- Installazione della piattaforma robot sono i seguenti:
- Etichettati 384 pozzetti
- 96 pozzetti fondo (1)
- Master Mix
- Una attraverso contenente il 2% di candeggina
- Un sistema di contenitori pieni di liquidi
- Rifiuti contenitore vuoto
- Iniziare a conduzione robot automatizzati selezionando la voce "Esegui" due volte.
- Alla fine del programma, rimuovere 384 pozzetti e sigillare con LightCycler 480 foglio di tenuta e centrifugare brevemente. Luogo sigillato piastra da 384 pozzetti in posizione 1 dell 'hotel.
- Passare al punto 2.2 e ripetere per piastra di fondo 2 con mescola nuovo padrone e una nuova piastra da 384 pozzetti.
- Inserire la piastra di tenuta in posizione 2 dell 'hotel.
- Aperto il nuovo programma per Evoware Caricamento del LightCycler dall'hotel e selezionare "Esegui" due volte.
- Freedom Evo caricherà automaticamente ogni piatto nel LightCycler ed eseguire il seguente SYBR-verde I / HRM programma dei cicli:
Pre (1 ciclo):
50 ° per 5 minuti a velocità di rampa di 4,8 ° / sec
95 ° per 5 minuti a velocità di rampa di 4,8 ° / sec
Amp (45 cicli):
95 ° per 15 secondi a velocità di rampa di 4,8 ° / sec
62 ° per 30 sec a velocità di rampa di 2,5 ° / sec
(Single di acquisizione dati in questa fase)
Curva di fusione:
95 ° per 5 secondi a velocità di rampa di 4,8 ° / sec
60 ° per 1 minuto a velocità di rampa di 2,5 ° / sec
95 ° continuo a velocità di rampa di 0,11 ° / sec
con 5 acquisizioni per ° C
Cool:
50 ° per 30 secondi a velocità di rampa di 25 ° / sec
- Freedom Evo caricherà automaticamente ogni piatto nel LightCycler ed eseguire il seguente SYBR-verde I / HRM programma dei cicli:
B. Configurazione di pre-miRNA test utilizzando la pipetta multicanale elettronica Matrix:
- Il contenuto del tubo posto master mix 1 in un serbatoio.
- Impostare la pipetta elettronica per aspirare 16ul di master mix con 2-8ul dispensare passi seguita da una fase di spurgo.
- Per master mix 1, dispensare 8ul in ogni altro bene del 384 pozzetti (impostato a 384 pozzetti spaziatura punta), a partire dal pozzetto A1. (Cioè pozzi A1, C1, E1, ecc) Per il secondo 8ul dispensare, spostarsi in colonna 3 (ossia pozzi A3, C3, E3, ecc) e poi eliminare su un contenitore dei rifiuti e suggerimenti smaltire.
- Seguite questo schema per i restanti 5 cicli fino a raggiungere ben A23.
- Ripetere per master mix 2, (a cominciare da ben A2, C2, E2, ecc); Master Mix 3 (a partire B1 bene, D1, F1, ecc); Master Mix 4 (inizio in B2, D2, F2, ecc .).
- Per aliquotting di piatto fondo, impostare pipetta elettronica per aspirare e dispensare 2ul 2ul con un seguito passo di spurgo. Dispensare 2ul di primer da colonna 1 a 96 pozzetti fondo (AH) nel 384-pozzetti, pozzi A1, C1, E1, ecc Purge su un contenitore dei rifiuti e suggerimenti smaltire. Ripetere l'operazione per pozzi A2, C2, E2, B1, D1, F1, e B2, D2, F2, ecc
- Ripetere per le righe 2-12 di primer a 96 pozzetti, passando sopra ogni 2 colonne per ogni colonna fondo, fino a completa a 384 pozzetti è stato aliquotted.
- Piastre di tenuta con un foglio di tenuta LightCycler e centrifugare brevemente.
- Piastre in un luogo LightCycler 480 e del ciclo di piatti secondo le seguenti impostazioni utilizzando il SYBR Green I / formato di rilevamento gestione delle risorse umane:
Pre (1 ciclo):
50 ° per 5 minuti a velocità di rampa di 4,8 ° / sec
95 ° per 5 minuti a velocità di rampa di 4,8 ° / sec
Amp (45 cicli):
95 ° per 15 sec a velocità di rampadi 4,8 ° / sec
62 ° per 30 sec a velocità di rampa di 2,5 ° / sec
(Single di acquisizione dati in questa fase)
Curva di fusione:
95 ° per 5 secondi a velocità di rampa di 4,8 ° / sec
60 ° per 1 minuto a velocità di rampa di 2,5 ° / sec
95 ° continuo a velocità di rampa di 0,11 ° / sec
con 5 acquisizioni per ° C
Cool:
50 ° per 30 secondi a velocità di rampa di 25 ° / sec - Ripetere con fondo piatto 2, aliquotting in un piatto fresco da 384 pozzetti utilizzando miscele maestro preparati al momento.
Segreti del successo:
Una caratteristica importante quando si progetta array PCR è che tutte le 186 coppie di primer hanno la stessa temperatura di ricottura, in modo che il piatto può essere eseguito con le stesse impostazioni ottimale del programma eseguire PCR.
Ogni nuovo array devono essere controllati utilizzando tre controlli prima della corsa dei campioni. Questi controlli sono:
1 pista di tutti i primer correre con acqua o 0,1 x TE per escludere la contaminazione primer.
1 pista di alternare acqua / TE e di controllo positivo, per assicurarsi che non riporto.
3 piste con lo stesso controllo positivo, per garantire la riproducibilità tra le esecuzioni.
Mix master deve essere preparata, e piatti deve essere eseguito entro 24 ore per ottenere risultati ottimali
Piastra di alluminio primer deve essere rimosso lentamente per ridurre il rischio di contaminazione tra pozzi.
Quando si utilizza un robot con le punte fisse, lavare candeggina al 2% è necessario lavare le punte tra ogni semina, seguita da un filo d'acqua. Questo previene ed elimina riporto, che potrebbero contaminare i dati e portare a risultati inconcludenti.
Dopo che un piatto è gestito attraverso il LightCycler, non dovrebbe essere riaperto nella stanza di impostazione della PCR. Questo aiuta a prevenire la contaminazione della PCR.
Rappresentante dei risultati:
qPCR risultati sono in genere rappresentati da valori di CT, come determinato dal software di analisi LightCycler. Un grande successo di solito consiste in una gamma di TA per i campioni, in genere tra 20-35 per i campioni che sono positivi. I campioni di acqua in una buona corsa sono sempre a un TC> 40 e di qualunque campione o pozzi specifiche con un CT oltre 37 sono considerati negativi o non rilevato. Come regola generale, i campioni ottenendo un CT di <10 sono inaffidabili e sono esclusi da ulteriori analisi. Ci sono diversi modi per analizzare i dati qPCR e l'inserimento dei controlli interni nei nostri test aiuta a controllare per la variazione di ingresso del campione. La pre-miR serie comprende un manuale di controllo U6, che è spesso utilizzato come gene di riferimento per normalizzare i valori di CT da campioni diversi. Questo valore normalizzato viene indicato come il delta CT valore (DCT). I risultati sono spesso rappresentate come le prime CT, il valore dCT o possono essere ulteriormente analizzati per valori standardizzati.
Nella figura 1, l'analisi della piena pre-microRNA array di quattro campioni diversi da un esperimento timecourse è presentato in formato carta di calore. L'espressione relativa di ogni pre-microRNA è mostrato e tre grandi, gruppi distinti emerse. La maggior parte dei pre-MIRS analizzato subito piccole variazioni insignificanti come previsto (Figura 1A). Tuttavia, una piccola porzione di pre-microRNA sono risultati significativamente diminuiti (Figura 1B) o aumentato notevolmente (Figura 1C) per tutto l'esperimento timecourse. I livelli di espressione di ogni pre-microRNA sono stati normalizzati al timepoint 0h (campione 1) come un livello base. In alcuni casi, è possibile notare che alcune delle cluster pre-microRNA anche raggruppare insieme nell'analisi heatmap (es: let-7, Figura 1B). A seconda della sperimentazione, i cluster possono emergere che dipendono da un'infezione virale, tipo di espressione di cellule specifiche di pre-microRNA o pre-MIRS che sono regolati da una molecola comune o via di segnalazione.
Il test di pre-microRNA che abbiamo sviluppato include anche una serie di note virale pre-microRNA e geni codificati da Sarcoma di Kaposi associato Herpesvirus (KSHV) e Epstein Barr Virus (EBV) in aggiunta alle pre-umano MIRS. Questi possono essere utilizzati come controlli positivi o negativi, a seconda della linea cellulare e lo stato virale dei campioni utilizzati. Nella figura 2, il TA media da un KSHV negativo campione eseguito in quadruplicato sono mostrati. È importante sottolineare che KSHV LANA non è stato rilevato in questo campione l'array qPCR altamente sensibile. Tuttavia, U6 - il nostro controllo interno e di let-7 bis - altamente espresso umano pre-microRNA, sono stati espressi a livelli rilevabili. Infine, come previsto, il controllo negativo della PCR acqua di grado non ha dato un prodotto qPCR.
Un altro indicatore di una corsa qPCR di successo è una buona analisi della curva di fusione, che può dare informazioni in potenziale contaminazione nei campioni. Per stabilire una temperatura di fusione, la piastra è riscaldata lentamente dopo la PCR è completa. Come i filamenti di DNA separati, SYBR green viene rilasciato, e il segnale di fluorescenza diminuisce. La temperatura alla quale questo drop verifica viene rappresentata graficamente, ed è conosciuta come la temperatura di fusione del campione. In base on la sequenza del DNA, i campioni fondono a temperature diverse le une dalle altre e dal controllo delle acque negativi, che non dovrebbe avere una temperatura di fusione in quanto non DNA è presente. I prodotti di PCR di ciascuna coppia di primer dovrebbe sciogliersi a temperature simili. Per esempio, la Figura 3 mostra l'analisi della curva di fusione per due primers diversi utilizzando lo stesso campione in duplicato. E 'chiaro che la temperatura di fusione è diversa per le due coppie di primer, ma il campione si sta sciogliendo ad una temperatura esattamente lo stesso per ogni replica. Segni di un campione di cattivo o potenzialmente contaminati possono includere diversi picchi di fusione, il che suggerisce la presenza di due diverse fonti di input.
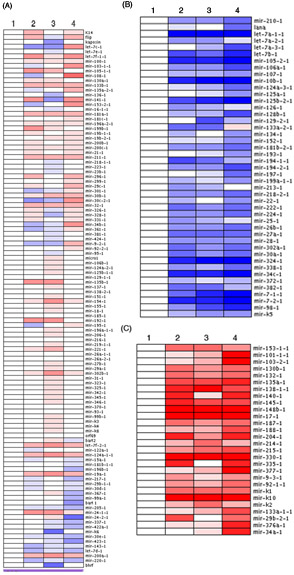
Figura 1. Pre-microRNA firme emergono attraverso il profiling usando un romanzo qPCR a base di array. La media è stata calcolata deltaCTU6 e valori standardizzati sono stati caricati nel software ArrayMinerTM, ottenendo 3 gruppi distinti visualizzato come heatmaps. (A) pre-microRNA con piccoli cambiamenti di espressione sono mostrati. Pre-microRNA i cui livelli sono risultati significativamente diminuiti (B) o aumentata (C) sono inoltre riportati. Blu corrisponde ad abbassare i livelli di espressione, mentre il rosso rappresenta un aumento dei livelli di espressione.
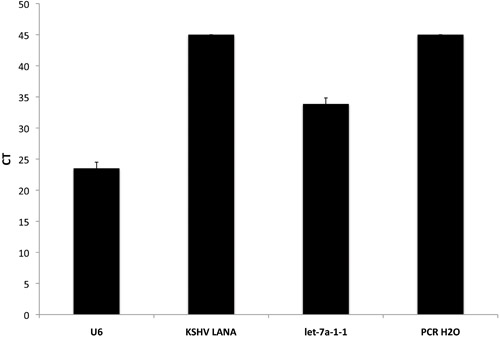
Figura 2. Inclusione dei controlli interni nella qPCR-based pre-microRNA array. I valori grezzi CT per 3 geni diversi e un modello di controllo non vengono visualizzati. U6 è usato come un controllo interno positivo mentre PCR H2O serve come il nostro controllo negativo. Let-7a-1-1 è un microRNA normalmente espressa in molti tipi cellulari diversi, mentre KSHV LANA può essere utilizzato sia come controllo positivo o negativo, in base allo stato virale della linea di cellule o tessuti utilizzati.
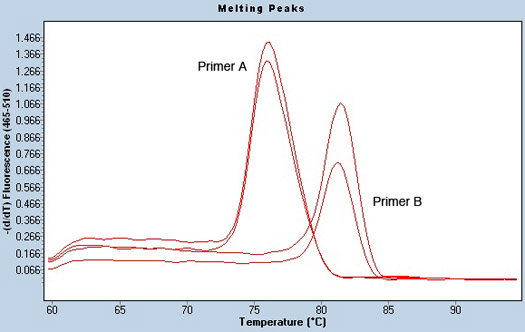
Figura 3. Picco di fusione analisi di pre-microRNA primer e la mancanza di contaminazione dei campioni. Le curve di fusione sono stati ottenuti utilizzando l'analisi chiamando Tm nel software LightCycler. Due primer diversi (Primer A e B) per lo stesso campione in doppio sono mostrati. La temperatura di fusione per ogni primer è distinto. Tuttavia, il campione ha un solo picco, dimostrando la mancanza di contaminazione dei campioni.
Access restricted. Please log in or start a trial to view this content.
Discussione
QPCR è un saggio altamente sensibile che può essere utilizzato per confrontare i livelli di espressione genica tra i campioni in studio, e di determinare positività in caso di virus. Il vantaggio di utilizzare array qPCR è la possibilità di eseguire molti primer (nel nostro caso, 186 coppie di primer) per ogni campione in un breve periodo di tempo. Utilizzando un robot pipettamento come la libertà Tecan Evo, la quantità di tempo necessario per impostare un esperimento up può essere ulteriormente ridotto, e l'...
Access restricted. Please log in or start a trial to view this content.
Divulgazioni
Riconoscimenti
Questo lavoro è stato supportato da sovvenzioni NIH DE018304, R01DE018281. KT è supportato da GM07092 T32-34 e da un finanziamento alla University of North Carolina a Chapel Hill da Howard Hughes Medical Institute (HHMI) attraverso il Med in Grad iniziativa. PC è supportato da T32 CA009156.
Access restricted. Please log in or start a trial to view this content.
Materiali
| Name | Company | Catalog Number | Comments |
| Freedom Evo 150 | Tecan Group Ltd. | 30017587 | |
| Matrix Electronic Multichannel Pipette | Thermo Fisher Scientific, Inc. | 2001-MTX | (or any comparable Thermo Scientific multichannel that can pipette the volumes described above) |
| Matrix Integrity Filter Tips, Sterile | Thermo Fisher Scientific, Inc. | 7435 | (or comparable tip for multichannel used) |
| Lightcycler 480 SYBR Green 1 Master | Roche Group | 04 707 516 001 | |
| Lightcycler 480 Instrument II | Roche Group | 05015243001 | 384-well version |
| Lightcycler 480 Multiwell Plate 384, white | Roche Group | 04729749001 | |
| LightCycler 480 Sealing Foil | Roche Group | 04729757001 | |
| LightCycler 480 Multiple Plate Analysis Software | Roche Group | 05075122001 | |
| Eliminase Decontaminant | Decon Laboratories | 04-355-31 |
Riferimenti
- Bustin, A. A. The MIQE Guidelines: Minimum Information for Publication of Quantitative Real-Time PCR Experiments. Clinical Chem. 55 (4), 611-622 (2009).
- Frégeau, C. J. Automated Processing of Forensic Casework Samples using Robotic Workstations Equipped with Nondisposable Tips - Contamination Prevention. J. Forensic Sci. 53 (3), 53-533 (2008).
- O'hara, A. J. Pre-micro RNA Signatures Delineate Stages of Endothelial Cell Transformation in Kaposi Sarcoma. PLoS Pathog. 5 (4), e1000389-e1000389 (2009).
Access restricted. Please log in or start a trial to view this content.
Ristampe e Autorizzazioni
Richiedi autorizzazione per utilizzare il testo o le figure di questo articolo JoVE
Richiedi AutorizzazioneEsplora altri articoli
This article has been published
Video Coming Soon