このコンテンツを視聴するには、JoVE 購読が必要です。 サインイン又は無料トライアルを申し込む。
Method Article
siRNA導入のための細胞型に特異的な抗HIV gp120のアプタマーの開発
要約
ナノモル親和性HIV - 1BA - Lのgp120に対するいくつかの2' -フルオロRNAアプタマーは、RNAのライブラリーからで隔離されています in vitroで SELEX手順。新しいデュアル阻害機能抗gp120のアプタマー - siRNAのキメラが作成され、全身の抗HIV治療のためにかなりの期待を示しています。
要約
HIVによる感染の世界的流行は、抗レトロウイルス薬の新しいクラスのための緊急の必要性を作成しています。相補的RNA転写産物の発現を阻害する低分子干渉(SI)RNAの強力な能力は、HIVを含む疾患の様々な治療薬の新しいクラスとして悪用されています。多くのこれまでの報告では、新たなRNAiベースanti-HIV/AIDS治療戦略は、かなりの期待を持っていることが示されているが、成功した治療への応用およびsiRNAの臨床翻訳の鍵の障害は、効率的な配信です。特に、RNAiベースの治療薬の安全性と有効性を検討し、それは特定の細胞集団または組織に標的細胞内のsiRNA導入のアプローチを開発することが極めて望ましい。 HIV - 1のgp120タンパク質、HIV - 1の表面上の糖タンパク質のエンベロープは、CD4細胞へのウイルスの侵入に重要な役割を果たしている。 HIV - 1のエントリをトリガし、細胞融合を開始することをgp120とCD4の相互作用は、薬物発見のための臨床的に関連する抗ウイルス戦略として検証されています。
ここで、我々はまず、2' - F改変抗HIV gp120のRNAアプタマーの選択と識別を議論する。従来のニトロセルロースフィルターSELEX法を用いて、ナノモル親和性を持ついくつかの新しいアプタマーは、50のランダムなNT RNAライブラリーから単離された。が正常に高い親和性で結合した種を得るためには、選択のストリンジェンシーは、慎重に条件を調整することによって制御される。選択されたアプタマーは、特異的に結合することができ、急速にHIV - 1エンベロープタンパク質を発現する細胞に取り込まれる。さらに、単独でアプタマーはHIV - 1感染を中和することができる。最高のアプタマー- 1に基づき、我々はまた、アプタマーやsiRNAの両方の部分が強力な抗HIV活性を持つ新規な二重の阻害機能抗gp120のアプタマー - siRNAのキメラを作成してください。さらに、我々はHIV - 1感染細胞へのsiRNAの細胞型特異的送達のためのgp120アプタマー - siRNAのキメラを利用する。キメラこの二重の機能は、抑制するHIV - 1感染の様々な核酸治療薬を(アプタマーとsiRNA)を組み合わせた高活性抗レトロウイルス療法(HAART)を失敗した患者のためのアプタマー - siRNAのキメラの魅力的な治療候補者を作るための大きな可能性を示しています。
プロトコル
1。 RNAライブラリの調製
- 出発DNAライブラリーは、ランダム配列の50ヌクレオチドを含んでおり、統合されたDNA技術(コーラル、アイオワ州)により合成した。 N 50 - - CAG ACG ACT CGC CCG - 3'(81 NT)AGG ACG ATG CGG一本鎖DNAオリゴのライブラリの順序は、5' - GGGです。ランダムな領域は、in vitro転写とRT - PCRのための3'タグのためのT7プロモーターを含む一定の地域、に挟まれている。 " - TAA TAC GAC TCA CTA TAG GGA GGA CGA TGC GG - 3'(32量体)と5'- TCG GGC GAG TCG TCT G - 3'(16 merの)、それぞれ5'および3'定数の配列は5です。 -20℃でアリコートで、水に濡らしたりしてストック溶液を作る
- PCRは、2mMのMgCl 2との各dNTP200μMのと一緒に、3μMの5' -および3' -プライマーの各々を用いて一本鎖DNAオリゴランダムライブラリ(0.4μM)を増幅する。オリジナルのDNAライブラリーの豊富さを維持するために、10サイクルのPCRを制限する。 PCR反応(10反応、反応あたり100μL)した後、QIAクイックゲル精製キット(QIAGEN)を用いて増幅された二本鎖DNAプールを回復する。
- 製造元の指示に従ってDuraScriptionキット(シャーロット、マディソン、WI)を用いてRNAのライブラリに、その結果dsDNAを変換します。転写反応混合物に、リボヌクレアーゼ耐性のRNAを生産する2' - F - CTPと2' - F - UTPとCTPとUTPを交換してください。一般的に、DNAテンプレートを精製1μgの、2μL10 ×バッファー、2μLのdATP、2μLdGTPを、2μL2' - F - dCTPを、2μL2' - F - dUTPを、2μLDTTを含有する反応の20μLを準備し、2μLのT7 RNA室温でポリメラーゼとは、37℃で6時間のためのCを(各dNTPが50 mMの。)
- その後、8%ポリアクリルアミド/ 7 M尿素ゲルによる鋳型DNAを除去し、精製するのDNase I(20μLのT7転写反応あたり1.5μL)との反応を消化。 UV分光光度法により精製したRNAライブラリを定量化する。
2アプタマーのin vitroでの発生で
- 選択する前に、選択バッファおよびリフォールディングバッファーを準備します。 100mMのHepes緩衝液(pH7.4)を含むHEPES緩衝液を準備します。室温で保管してpH値を調整するためにNaOHを使用して。 5mMのMgCl 2を ;の5mMのCaCl 2、13.5 mMのKClを50mMのHEPES、pH7.4、中、750 mMのNaClを含むRNAのリフォールディングバッファー(5xHBS)を準備する。低塩RNA結合バッファー(10mMのHEPES pH7.4の、50mMのNaCl、1mMのCaCl 2、1mMのMgCl 2、2.7 mMのKCl、10mMのDTT、0.01%BSAおよび高塩RNA結合バッファー(10準備mMのHEPES pH7.4の、150mMのNaCl、1mMのCaCl 2、1mMのMgCl 2、2.7 mMのKCl、10mMのDTT、0.01%BSAが)。-20℃で、これらのバッファを保存します
- 1-4を説明し、主としてSELEXを実行します。 1xHBS緩衝液(10mM HEPES pH7.4の、150mMのNaCl、1mMのCaCl 2、1mMのMgCl 2、2.7 mMの塩化カリウム)ですべての選考、リフォールディングRNAプール、3分してから、徐々に冷却するための95〜熱° Cの前に〜37℃ 37℃インキュベーションを続ける℃で10分間。
- 一般的に、ターゲットHIV - 1 バルのgp120蛋白質とのインキュベーション前に、30分間ニトロセルロースフィルター(HAWPフィルター、孔径0.45μm)へのRNAのプールをリフォールディングニトロセルロースフィルター、プレ吸着と非特異的結合を最小限にするために。
- SELEXラウンド1〜4の30分間、低塩濃度のRNA結合バッファー中の標的タンパク質との事前クリアRNAプールをインキュベートする。 SELEXの第4ラウンドの後、高塩濃度のRNA結合バッファーを使用してください。 SELEX進歩に伴い、gp120のタンパク質の量を削減し、アプタマー選択のストリンジェンシーを増加させるために、競合他社の酵母tRNAを増加させる。
- 選択の最初のサイクルでは、に200μLの低プレクリアランダムRNAプール(40μgの、1.5 nmolの、9x10 14分子)とHIV - 1 バルのgp120タンパク質を(0.23ナノモル、RNA /タンパク質比が6.5 / 1)インキュベート30分間室温で回転プラットフォーム上で塩RNA結合バッファー。
- プリ接液ニトロセルロースフィルターを通して反応を渡すと、1 mLの結合バッファーで洗浄する。
- 95℃で200μLの溶出緩衝液(7M尿素および5mM EDTA)°マイクロコンYM - 30カラムでフェノール/クロロホルム抽出し、濃縮し5分、のためのCとフィルタから結合したRNAを溶出する。
- ThermoScript RT - PCRシステム(Invitrogen)を用いて回収したRNAプールを転写とPCRの15サイクル増幅逆。
- QIAクイックゲル精製キットを用いて増幅した二本鎖DNAのプールを浄化し、選択の次のラウンドのために上記のように転記する。
3。フィルター結合アッセイによって監視SELEXの進捗状況
- フィルター結合アッセイによりアプタマーのSELEX進行状況を監視します。 T4ポリヌクレオチドキナーゼおよびγ- 32P - ATPとして5' -三リン酸を開始し、ラベルを削除するには、CIPとRNAプールを扱います。
- CIPの熱10pmolのは、95℃RNAライブラリを扱う℃で5分間、次に氷上で冷やしますのためのC。続いて、2を追加PNKバッファ、T4ポリヌクレオチドキナーゼの1μL、20μLのγ- 32 P - ATPおよび水の1μLμLの。
- 37℃で30分間、次に20 ^ムーを追加する、水のLとG - 50カラムで反応を浄化。最後に、250nmの最終濃度でラベルされたRNAの40μLを得る。
- アッセイの前に、1xHBS緩衝液(10mM HEPES pH7.4の、150mMのNaCl、1mMのCaCl 2、1mMのMgCl 2、2.7 mMの塩化カリウム)、95〜熱でリフォールディングされたRNAプール℃で3分間、その後に徐々に冷却するための37℃ 37℃インキュベーションを続ける℃で10分間。
- ここで例として結合反応液100μLを行う。末端標識gp120のタンパク質(100 nM)および30分間の高塩RNA結合バッファーで非特異的な競合他社のtRNA(100 nm)の10倍モル過剰のRNAプールを(10nM)をインキュベートする。
- プリウェットニトロセルロースフィルターによる結合反応液50μLを区切ります。
- 2 mLの結合バッファーでフィルターを洗浄し、多目的シンチレーションカウンター(ベックマンコールター)を介してフィルター上に保持された放射能をカウント。入力制御として、同時に結合反応の残りの50μLを数える。結合親和性としてインプットRNAにフィルター上に保持されたRNAのパーセントを計算する。
4。クローニング、シークエンシングとアラインメント
- それ以上の濃縮は、追加の選択ラウンド次のようにも観察されていない場合は、11ラウンド後は、その後のRNAプールの最大結合は潜在的に達している。
- ThermoScript RT - PCRシステム(Invitrogen社)と続いて、PCRにより得られたcDNAを増幅して使用して高濃縮アプタマーのプール(12 番目のRNAプール)を転写する逆。 QIAクイックゲル精製キット(QIAGEN)を用いてPCR産物を精製する。ゲル精製TAクローニングベクターをpCR 2.1(Invitrogen)にDNA産物をクローニングします。合計で、170個のクローンを接種し、さらに個々の配列を取得するためにDNA配列決定によって、それらを識別する。
- 個々のアプタマーの配列のアライメントに基づいて、6つの異なるグループに個々のクローンを分類する。各グループから1つの代表的なシーケンス(A - 1、-5、-9、-12、- 28とB - 68)ので、そのグループ内での相対的な量のさらなる特徴付けのために選んだ。
5。 in vitro転写によるアプタマーとキメラRNAの世代
- 直接PCRが2mMのMgCl 2との各dNTP200μMのと一緒に、2μMの5' -および3' -プライマーの各々を用いて二本鎖DNAテンプレートを生成し、QIAクイックゲル精製キットを用いて得られたPCR産物を回復する。
- DuraScriptionキット(シャーロット、マディソン、WI)を使用してPCR生成されたDNAテンプレートからキメラセンス鎖を転写する。転写反応混合物に、RNaseの分解に耐性であるRNAを生産する2' - F - CTPと2' - F - UTPで正規のCTPとUTPを交換してください。
- 通常は、μLDNAテンプレートの1μgの、2 mLの10xbuffer、2μLのdATP、2μLdGTPを、2μL2' - F - dCTPを、2μL2' - F - dUTPを、2μLDTTと2を含む反応の20μLをインキュベートT7 RNAは、37ポリメラーゼ℃で6時間インキュベートし、続いてフェノール抽出とエタノール沈殿に続くバイオスピン30列(Bio - Rad社)でそれを浄化する。
- インターフェロン応答を回避するために、さらに開始5' -三リン酸を除去するCIPによって転写されたRNAを扱う。 ℃で60分間37℃で転写産物の3μgの、バッファ3の6μLとCIPの0.25μLを含有する反応の合計60μLをインキュベートする。フェノール/クロロホルム強要とエタノール沈殿の後、再懸濁し、RNAペレットを水に。
- キメラを準備するには、バッファ、熱をリフォールディングに適切なアンチセンスRNAのみでセンス鎖のRNAを保有するキメラを組み合わせて95 ° C〜37クール後、3分および℃でゆっくりと。 37℃インキュベーションを続ける℃で10分間。フィニアルの1xHBSバッファのリフォールディングステップを実行します。たとえば、次のように10μMキメラセンス鎖の混合10μL、25μLシステムに10μMのアンチセンス鎖と5μLリフォールディングバッファー(5xHBS)の10μL。
6。ゲルシフトアッセイにより、解離定数の決定
- エンドP 32標識の代表、各グループとキメラのセンス鎖からアプタマー、上述のようにリフォールディング1xHBSバッファー中のRNA。
- 40%アクリルアミド/ビスソリューション、19.375 mLの水、10%過硫酸アンモニウム(APS)溶液を150μL、およびTEMED 30μLの3.125 mLで、10xTBEバッファー2.5 mLを混合することにより、5%のゲル25mlを準備します。ゲルは、約30分で重合してください。慎重にコームを削除し、実行しているバッファ(1xTBE)でウェルを洗浄するために針を備えた、30 mLの注射器を使用してください。
- ゲルユニットの組み立てを完了し、電源に接続してください。ゲルは4℃180 V℃で1時間のために事前に実行することができます。
- シリアルに、所望の濃度まで結合緩衝液でHIV - 1Balのgp120タンパク質を希釈する。最終的なRgp120のeaction濃度は0、1、5、10、20、40、80、160、320、640 nmです。 5' - Pの一定量のRNA(10nM)を30分間室温で回転プラットフォーム上で結合緩衝液中のgp120タンパク質濃度の上昇(反応のμL合計20)と32 -末端標識をインキュベートする。
- インキュベーション後、5%非変性ポリアクリルアミドゲルに5μLネイティブローディング緩衝液と負荷との結合反応の20μLを混合する。 1mMのEDTA、0.1%ブロモフェノールブルー、0.1%キシレンシアノールFF、0.1%オレンジG、40%グリセロール、10mMトリス- HCl、pH7.5を含むネイティブローディングバッファーを(4x)の準備。 -20℃でアリコートの店
- 以下の電気泳動(4で180 V℃で2時間、二次色素がゲルの中央に実行されるまでは)、蛍光画像の画面にゲルを公開し、台風スキャナを用いて放射能を定量化する。
- グラフパッドプリズムと非線形曲線の回帰を用いて解離定数を算出する。
7。フローサイトメトリーによる細胞表面結合研究
- サイレンサーsiRNAのラベリングキット(Ambion社)を用いて蛍光アプタマーとのキメラを生成する。 、5 mLの10 ×標識バッファー、15μLRNA(5μg)を、7.5μlの標識色素22.5μLのヌクレアーゼフリー水:次の順序で試薬を加える。 37合計50μLのラベリング反応℃で1時間インキュベートする。
- インキュベーション後、5.0μL(0.1容量)の5M NaClおよび125μL(2.5巻)冷100%エタノールを、追加して、よく混ぜます。 60分間-20℃でインキュベートする。 20分間4℃で最高速度で遠心。 175μLを70%EtOHで上清と洗浄ペレットを削除します。その後、空気乾燥した暗所でペレットとは、ヌクレアーゼフリー水15μLで標識RNAを中断。
- 260nmで、蛍光色素の吸光度の最大でラベルされたRNAの吸光度を測定します。が提供する計算機によると色素の比率とRNAの濃度:ベース計算http://www.ambion.com/techlib/append/base_dye.htmlを 。
- 上記のようなバッファをリフォールディングにおけるミックスCy3標識キメラのセンス鎖とアンチセンス鎖とリフォールディング。
- エイズの研究およびリファレンス試薬プログラム5、6を介しgp160とCHO - EEのコントロール細胞を発現するCHO - WTを取得する。 GMEM - S培地(400μMメチオニンスルホキシ(MSX)とグルタミン欠損最小必須培地)(ギブコ、Invitrogen)中で細胞を成長させる。 37℃加湿5%CO 2インキュベーター内で培養、細胞℃に
- 予熱洗浄バッファーでCHO - WT gp160またはCHO - EEのコントロール細胞を洗浄し、トリプシン処理し、プレートから切り離します。 500μLの結合緩衝液で2回細胞を洗浄した後、37℃で結合バッファーでインキュベートで細胞ペレットをresupend℃で30分間。細胞をペレット化し、その後は400 nMのCy3標識実験的なRNAを含む温めた結合バッファー50μLで再懸濁します。
- 37℃インキュベーション後℃で40分間、予熱の結合緩衝液500μLで細胞を3回洗浄し、最後に結合バッファー350μLで再懸濁し、37に温めた℃、直ちにフローサイトメトリーによって分析する。
8。ライブセルの共焦点顕微鏡による内在と細胞内局在の検討
- アッセイの一日前に、24時間で約70%コンフルエントにできるように2mLのGMEM - S培地に0.3x10 6に播種して35 mmのプレートにCHO - WT gp160とCHO - EEのコントロール細胞を成長させる
- 実験の日に、あらかじめ温めておいたPBS 1mLで細胞を洗浄。と37℃で30分間予め温めておいた完全増殖培地1mLでインキュベート℃に
- 前述のようにCy3標識アプタマー - siRNAのキメラを準備します。上記のようなアプタマースティック- siRNAの共役を形成するために、5' - Cy3標識センス鎖を含むsiRNAを棒でリフォールディングアプタマースティックをインキュベートする。
- アッセイの前に、4℃でアリコートで水とストア内のヘキスト33342(生きている細胞の核染色、Molecular Probes社、Invitrogen社、カリフォルニア州)0.15 mg / mLの溶液を調製
- 37℃で15分間が0.15 mg / mLのヘキスト33342での処理により細胞を染色すぐに、二回1.0mLの新鮮な培地で染料を洗い流し、新鮮な培地を加温2mLを交換してください。
- Cy3標識で37 5%CO2インキュベーター顕微鏡で生きた細胞の共焦点顕微鏡のためのメディアとインキュベートに100nMの最終濃度でアプタマー - siRNAのキメラ℃を追加
- 画像の40倍の倍率で水浸下で2光子共焦点顕微鏡システムを倒立ツァイスLSM 510メタを使用して15分毎に収集する。
9 in vitroで HIV - 1チャレンジとp24抗原アッセイで
- ATCCからCCRF - CEM細胞を購入する。 10%ウシ胎児血清(FBS、HyClone)、L -グルタミンと1xPen -連鎖球菌(ギブコ、インビトロジェン社)を補充したRPMI - 1640(Cellgro、Mediatech社)で育つ。 37℃加湿5%CO 2インキュベーターで培養細胞℃の
- 末梢血のmononucleを取得するホープ国立医療センター(診療所職員)の都市からの健康なドナーからのARサンプル。
- フィコール - ハイパックソリューション(Histopaque - 1077、Sigma)を介しての遠心分離により全血からPBMCを分離する。ダイナビーズCD8(Invitrogen社、CA)を使用してPBMCからCD8細胞を(T-cytotoxic/suppressor細胞)使い果たす。
- T細胞活性媒体(BioE、セントポール、ミネソタ州)で細胞を成長させる。 37℃加湿5%CO 2インキュベーターで培養細胞℃の
- エイズの研究およびリファレンス試薬のプログラムからのHIV - 1 IIIB及びNL4 - 3ウイルスとHIV - 1バルウイルスを入手してください。 -80℃でアリコートでウイルス、店舗℃の伝播後の
- HIVウイルス(IIIB、NL4 - 3またはバル)(MOI 0.001または0.005)とCCRF - CEM細胞またはヒトPBMCに感染。感染後の24時間後、静かに無料のウイルスを削除するには、PBSで3回細胞を洗浄。文化へ37 5%CO 2インキュベーター顕微鏡で感染した細胞℃で4日間を継続する。
- 前治療をRNAに、静かに無料のウイルスを削除するには、PBSで3回感染細胞を洗う。とインキュベート2 × 10 4感染細胞と3 × 4非感染細胞は、37 ° C(、三重アッセイウェルあたり100μL)で96ウェルプレートで400 nmでの最終濃度を実験的なRNAをリフォールディング。
- 異なる時間(3日、5日、7日と9 D)とp24アッセイまで-20℃で保存で培養上清を(1ウェルあたり10μL)を収集。
- HIV - 1 p24抗原ELISAキットを用いてp24抗原の解析を行います。
10。定量的RT - PCRアッセイでsiRNAの機能の検出
- HIVウイルス(IIIB、NL4 - 3またはバル)とCCRF - CEM細胞またはヒト末梢血単核細胞に感染し、上記のような実験的なRNAを(波長400nm)で扱います。
- 治療の7日後、細胞をペレット化し、STAT - 60でトータルRNAを分離する。ゲノムDNAを除去し、全RNA2μgを用いてcDNAを生成するためにDNase Iで全RNAを扱う。
- 、1.5 mLの10 × DNase処理バッファ、4μLRNA(2μg)を、0.5μLRNain阻害剤および1.0μLRNaseフリーのDNase Iのインキュベーション合計15μL反応37℃1℃8 mLのヌクレアーゼフリー水:以下の試薬を混合80時間、熱は℃で10分間、直後のDNase Iを失活氷上で反応を冷やして。
- 65℃、2μLランダムプライマー(μL/ 50 ng)をし、1μLのdNTP(10mM)を、上記反応混合物に、そして熱を追加℃で5分間。すぐに、氷上で反応を冷やします。
- 以下の試薬を追加します。5mLの5xFirstストランドバッファー、2.5 mLの0.1 M DTT、0.5 mLのRNain阻害剤および1.0 mLのMMLV - RTを。 25合わせて27 mLの反応℃で10分間および37℃℃で1時間インキュベートする。反応後、70℃の熱混合℃で15分間逆転写酵素を不活性化し、氷の上で寒さをのためのC。 cDNAは定量RT - PCR解析の準備ができています。
- 定量的RT - PCRは400nm(三重アッセイ)の最終濃度で2 × iQのSyberGreenマスターミックスと特異的プライマーセットを用いて標的遺伝子の発現を分析する。定量PCRのデータの正規化のための内部コントロールとしてGAPDHの発現を使用してください。
11。代表的な結果:
1。 BALのgp120 HIV - 1に対する新しいRNAアプタマーが単離され、特徴付けされています。
実験の節で説明したように、5'および3'末端に固定されたプライマー領域に挟まれた最初のDNAオリゴヌクレオチドライブラリを含む50 NTランダムな領域は、RNAプールに増幅し、転写される。この初期のライブラリは、さまざまな3次元構造の広大な配列にフォールド15 10までの多様なシーケンス(1 nmol)を、構成されています。最初のライブラリの高い複雑さと多様性は、ターゲットへの良好な結合親和性を持つアクティブな構造の存在を保証するかもしれない。
選択的にR5株のHIV - 1 BALのgp120エンベロープタンパク質7を結合する2' -フルオロ修飾RNAアプタマーを選択するためにin vitroで SELEXの手順( 図1) で採用。 図1に示すように、ニトロセルロースベースの選択の戦略は、非結合のRNA分子から特異的標的結合RNAを分離するために実行されます。タンパク質はニトロセルロースに付着するので、唯一のRNA /タンパク質複合体または凝集体が膜上に保持することができ、無料のRNAは洗い流されている。変性条件下で、結合したRNAを回収し、逆cDNAに転写し、二本鎖DNAに、と続いて次の選択サ イクルのための新しいRNAのプールを作成するための転写をインビトロで増幅されています。選択のストリンジェンシーは、標的タンパク質の量を削減し、競合他社のtRNAの量を増やすことによって増加する。 RNAプール、各選択のラウンドで使用されている蛋白質と競合tRNAの量を表1に示されています。
フィルター結合アッセイによってそれぞれのSELEXサイクル後の選択の進行状況を監視します。 RNAの%はFILに保持として結合親和性を評価するトータルRNAのプールでTER。出発RNAのプール(1 - RNA)は、膜上に保持された入力分子RNAの0.1%を示しています。しかし、9つの選択のラウンド後に第九RNAライブラリ(9 - RNA)がバインドされている入力RNAの9.72パーセントを持っています。追加の選択ラウンドが実施されたが、それ以上の濃縮は、RNAのプールの最大結合が( 図2A)に達したことを示唆し、観察ではありません。フィルター結合アッセイと同様に、ゲルシフトアッセイはまた、解離定数を決定するための最も一般的な戦略の1つです。この手順は簡単で便利です。 図2Bに示すように、ゲルシフトアッセイは、さらにRNAプールの結合活性を確認する。これらの結果は、標的タンパク質に対して高い結合特異性を有するいくつかの配位子が連続してこれらのRNAのプールに富むことを示している..
クローンとシーケンス高濃縮アプタマープール(12 - RNA)。個々のクローン化されたアプタマーの配列のアライメントによると、6種類のグループを表2に示すように分類されます。 (/ G)TTGAGGGACC(/ G):クローンの約40%(グループIおよびIIアプタマー)が保存された配列が含まれています。ため、そのグループ内での相対的豊富さのさらなる特徴付けのため:私達は各グループから1つの代表的なシーケンス(1、-5、-9、-12、- 28とB - 68など)を選択します。ネイティブゲル移動度シフトアッセイを通して、これらの代表的なアプタマーの解離定数(K d)は ( 図3A)計算されます。例えば、A - 1、アプタマーの最高は、52 nMの( 図3B)の見かけのKd値を持っています。図3Aに示すように、これらの選択されたアプタマーは、選択的に標的とHIV - 1 バルのgp120ではなく、HIVのgp120 CMのタンパク質と結合することができる。
2。抗gp120のアプタマーは、特異的に結合し、HIV gp160を発現する細胞によって内在化されており、細胞培養におけるHIV - 1感染を阻害する。
CHO - gp160安定HIVエンベロープ糖タンパク質gp160を発現する細胞は、選択される抗gp120のアプタマーの結合および内在化のためにテストするために使用されています。彼らはエンベロープ処理に必要なギャグでエンコードされたプロテアーゼが不足しているため、これらの細胞がgp120とgp41にgp160を処理しません。対照として、我々はgp160を発現しない親のCHO - EEの細胞株を使用。フローサイトメトリー分析( 図4A)は、Cy3標識アプタマーは特にCHO - gp160細胞に結合することが明らかになったが、対照CHO - EE細胞。さらに、リアルタイムの生きた細胞のZ軸共焦点顕微鏡は、Cy3標識アプタマーが選択的にCHO - gp160インキュベーションの2時間後の細胞( 図4Bおよび4C)が、CHO - EEのコントロール細胞(内内在化されていることを示し図4D)。図4Cはまた、アプタマーは、多分受容体を介したエンドサイトーシスを介して細胞に入るのgp120アプタマーを示唆して細胞質内に集約することを示しています。
HIV - 1チャレンジアッセイでは、HIV - 1感染- PBMC細胞はアプタマーと扱われます。アプタマーとは別の日の後の治療では、メディアのアリコートは、ウイルスのp24抗原レベル( 図4E)についてアッセイする。結果は、抗- gp120のアプタマーは、(1)ナノモル濃度でHIV - 1 p24を産生を阻害することを示している。
3。キメラアプタマー - siRNAの抗gp120の細胞型特異的siRNAデリバリーシステムとしての有効性を設計し、評価される
図5Aに示すように、アプタマーおよびsiRNAのセンス鎖のセグメントは、ヌクレアーゼ耐性2' -フルオロUTPおよび2' -フルオロCTPを含んでおり、in vitroでのバクテリオファージの転写により対応する二本鎖DNAテンプレートから合成される。分子の柔軟性を高めるために、2つのヌクレオチドリンカー(UU)はアプタマーとダイサー基板の部分の間に挿入されます。キメラを含むsiRNAを調製するために、in vitro転写されたキメラアプタマーセンス鎖のポリマーで修飾されていないアンチセンス鎖のRNAのモル濃度とアニールされています。ゲルシフトアッセイ( 図5B)とフローサイトメトリー( 図5C)からこれらのデータは、キメラが単独でアプタマーとほぼ同じ結合親和性を維持することを示している。リアルタイム共焦点顕微鏡( 図5D)から経時画像はA - 1チャンネルキメラが正常細胞の細胞質中に内在化することができますCy3標識ことを示している。予想通り、キメラのない取り込みは、CHO - EEコントロール細胞( 図5E)で観察されない。
同様に、RNAのウイルスの可能性は、HIV - 1チャレンジアッセイによって評価されます。 HIV p24抗原分析の結果(図6Aおよび6B)アプタマーとキメラの両方がp24を産生を抑制するが、最強の阻害はキメラCH - 1の治療で観察されていることを見る。
siRNAのコンポーネントは、APTAと一緒に機能していることを確認するMERは、感染細胞のキメラChのインターナリ- 1に続く、我々はまた、定量的RT - PCRの発現アッセイにより、TAT /回転遺伝子発現の阻害の相対的レベルを評価する。単独でアプタマーはTAT /回転の遺伝子発現( 図6Cおよび6D)に影響しなかったしながら我々は、キメラに感染した細胞の治療は、TAT /回転遺伝子のサイレンシング誘導することが可能であることがわかります。これらの結果は、アプタマー納入したsiRNAは、RNAiを引き起こすことをさらにサポートを提供しています。
| 選択に使用される蛋白質、RNAプールとtRNAの量 | |||||
| SELEXラウンド | ターゲット/ RNAの比 | gp120のタンパク質 | RNAプール | 競合他社のtRNA | 選択バッファ |
| 1 | 1/6.5 | 229.8 pmolの | 1.5 nmolの(40.1μg)を | 0 | 低塩SELEXバッファ |
| 2 | 1/6.5 | 114.9 pmolの | 0.75 nmolの(20.1μg)を | 0.25 nmolの(6.6μg)を | |
| 3 | 1月8日 | 76.6 pmolの | 0.625 nmolの(16.7μg)を | 0.25 nmolの(6.6μg)を | |
| 4 | 1月8日 | 76.6 pmolの | 0.625 nmolの(16.7μg)を | 0.25 nmolの(6.6μg)を | |
| 5 | 1月8日 | 38.3 pmolの | 0.306 nmolの(8.18μg)を | 0.5 nmolの(13.2μg)を | 高塩分SELEXバッファ |
| 6 | 1月8日 | 38.3 pmolの | 0.306 nmolの(8.18μg)を | 0.5 nmolの(13.2μg)を | |
| 7 | 1月10日 | 26.8 pmolの | 0.268 nmolの(7.16μg)を | 0.5 nmolの(13.2μg)を | |
| 8 | 1月10日 | 26.8 pmolの | pmolの0.268 nmolの(7.16μg)を | 1 nmolの(26.4μg)を | |
| 9 | 1月10日 | 15.3 pmolの | 0.153 nmolの(4.09μg)を | 1 nmolの(26.4μg)を | |
| 10 | 1月10日 | 15.3 pmolの | 0.153 nmolの(4.09μg)を | 1.5 nmolの(39.6μg)を | |
| 11 | 1/12.5 | 7.66 pmolの | 0.096 nmolの(2.56μg)を | 2ナノモル(52.8μg)を | |
| 12 | 1/12.5 | 7.66 pmolの | 0.096 nmolの(2.56μg)を | 2ナノモル(52.8μg)を | |
表1。選定条件。タンパク質の量は、それぞれの選択と選択バッファに使用されるRNAプールとtRNAが示されている。
| グループ | RNA | ランダムシーケンス | 周波数(140クローン) |
| グループI | A - 1 | AATTGAGGGACCA CGCGCTGCTTGTTGTGATAAGCAG TTTGT CG GATGG | 33(23.6%) |
| B - 7 | AATTGAGGGACCA ACGCGAGGATGTGGATAGTGTGTA TTTGC GT GATGG | 3 | |
| - 32 | AATTGAGGGACCG TTGGTAAAAGCCGGA AATTG AGCT TTTAC GGC GATGG | 5 | |
| B - 55 | AATTGAGGTACCGCG TTATTAGGAACA AATTG GAATTCTAAACGC GATGG | 2 | |
| A - 24 | AATAGAGGGACC CAGATATAGGCTACACGGATGATGGTGTATCTG GATGG | 1 | |
| B - 19 | AATAGAGGAACCG TTTCAGAAGACTACAGGTTAGTCCAATGAAGC GACGG | 1 | |
| B - 31 | AATAGAGGGACCG TGGACAATAATTTATGGTCA TTTATTGGCAC GATGG | 1 | |
| グループII | A - 12 | AGTAGAGGAACCA AGCAATGGATGAATGCAAAAGTGTAAATGCTT GATGG | 10(7.1%) |
| グループIII | A - 9 | TGAGTTTGGGTAAATTTCCGGTTTCGGTTTACTCACGAAAGATCGGTCGG | 15(10.7%) |
| グループIV | - 28 | TAAAGGAGGGAAGGATGAGACCGCACGAAAAATATCAGCATACG TTTGTG | 10(7.1%) |
| グループV | A - 5 | GAAACTAGTTTGAATAATGGTGTAGAGGAGGGTCAATAGTTTCG TTGGTG | 9(6.4%) |
| グループVI | B - 68 | ACATAGTAATGACACGGAGGATGGAGAAAAAACAGCCATCTCTTGACGGT | 2 |
| その他 | 孤児シーケンス | 48 |
表2。アライメントとRNAアプタマーの同定。選択の12 番目のラウンドに続いて、選択したRNAプールをクローニングし塩基配列を決定した。全140クローンの整列した後、抗- gp120のアプタマーの6つのグループが同定された。アプタマーコア領域(5' - 3')の唯一のランダムシーケンスが示されている。複数の周波数で発生する分離株が指定されています。
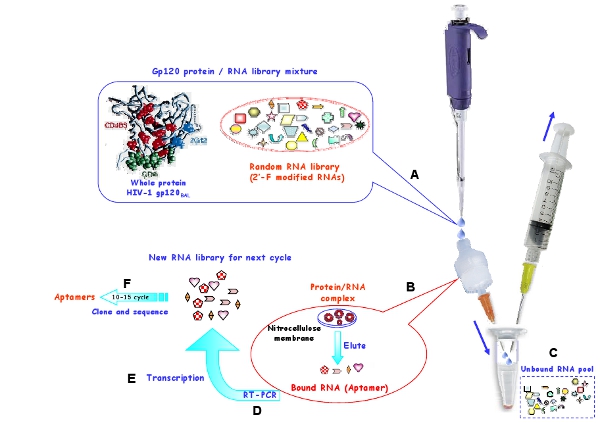
図1:HIV - 1バルのgp120タンパク質に対するRNAアプタマーを生成するために、ニトロセルロース膜を用いたin vitroの選択手順での模式図。 (A)開始のRNAプールと標的タンパク質が複合体を形成するためにインキュベートした。 (B)結合したRNA分子は、膜上に保持し、変性条件下で膜から溶出した。 (C)未結合のRNAを離れて洗浄した。 (D)選択されたRNAを逆転写とPCRにより増幅した。 (E)関連するDNAは、その後、次の選択サ イクルのための新しいRNAプールに転写した。 (F)10-15選択ラウンド後、選択されたアプタマーをクローニングしてシーケンスした。
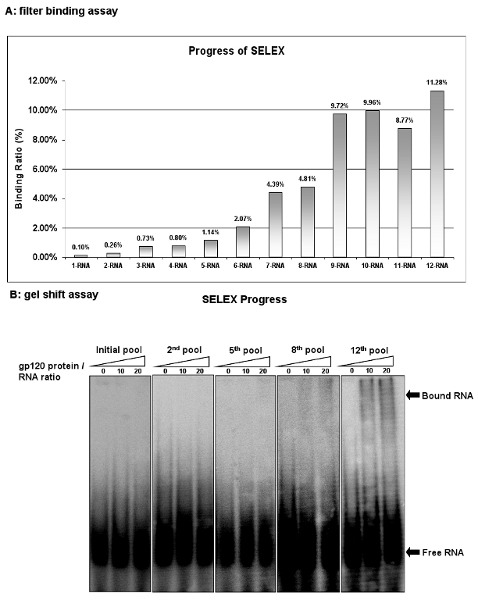
図2:HIV - 1 gp120のアプタマーの選択の進捗状況。 (A)各サイクルでのRNAプールの結合活性は、競合のtRNAをフィルター結合アッセイにより分析した。結合活性は、RNAがタンパク質/複雑なRNAでフィルター上に保持された入力の割合として算出した。 (B)各サイクルでのRNAプールの結合活性はゲルシフトアッセイにより分析した。 第 12 回 RNAのプールは、最も高い結合活性を示した。
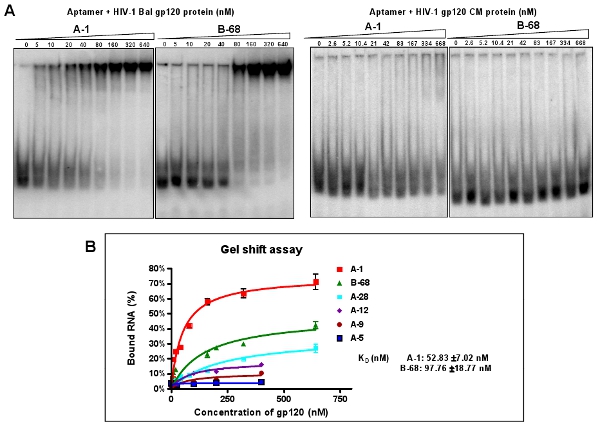
図3: バル gp120とHIV - 1に対して選択された個々のアプタマーの結合活性をアッセイ。 (A)5'末端P 32標識された個々のアプタマーは、標的のgp120タンパク質または非特異的なCMのタンパク質量の増加とともにインキュベートした。結合反応混合物は、ゲル移動度シフトアッセイにより分析した。アプタマー- 1とB - 68は、標的タンパク質との最高の結合親和性を示したが、タンパク質CMではない。データは4つの平均的な複製を表しています。ゲルシフトアッセイから(B)結合曲線。
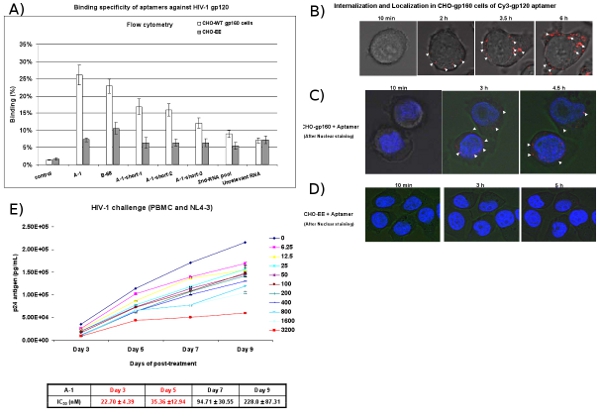
図4:細胞型特異的結合およびアプタマーの取り込みの研究。 Cy3標識RNAの(A)細胞表面の結合をフローサイトメトリーによって評価した。 Cy3標識RNAをCHO - gp160細胞とCHO - EEのコントロール細胞への結合について試験した。選択されたアプタマーは、細胞型特異的結合親和性を示した。第2回RNAプールと無関係のRNAをネガティブコントロールとして使用した。データは3つの平均的な複製を表しています。 (B)内部化の分析。 CHO - gp160細胞を35 mmのプレート中で増殖し、リアルタイムの生きた細胞の共焦点顕微鏡分析のための培地で1 Cy3標識の100 nMの濃度でインキュベートした。画像は15分に採取した。 40X倍率を使用して間隔。 (C、D)局在解析。 CHO - gp160細胞とCHO - EEのコントロール細胞を35 mmプレートで増殖させた。 A - 1 Cy3標識の100 nMのとのインキュベーション前に、細胞はヘキスト33342(生きている細胞の核染色)で染色し、リアルタイム共焦点顕微鏡を用いて分析した。 (E)選択される抗gp120のアプタマーは、以前にHIV - 1 NL4 - 3ウイルスに感染したヒトPBMC中のHIV - 1複製を阻害する。異なる濃度と時間ポアントが発表された。 IC50値が表示されました。データは、P24の3回の測定の平均値を表しています。
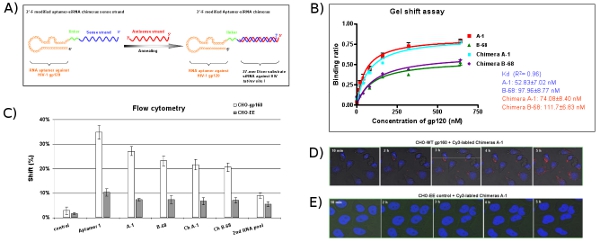
図5:アプタマー- siRNAのキメラ配信システムの設計と評価。 (A)回路図アプタマー- siRNAのキメラRNA:抗gp120のアプタマーの地域がgp120に結合し、siRNAは、HIV - 1 TAT / revの共通のエクソンをターゲットにしている責任がある。 2' -フルオロ改変アプタマー - siRNAのセンス一本鎖のキメラ分子を完了するために相補的なsiRNAのアンチセンス鎖のアニーリングに続く、共同転写した。アプタマーとsiRNAの間のリンカー(UU)が緑色で表示されます。 (B)と同等Kd値だけでなく、親のアプタマーを持っているアプタマー- siRNAのキメラRNAは特にHIV バルのgp120タンパク質を結合する。データは3つの平均的な複製を表しています。 (C)アプタマーの細胞型特異的結合の研究。 Cy3標識RNAをCHO - gp160細胞とCHO - EEのコントロール細胞への結合について試験した。セルシュルCy3標識RNAの顔のバインディングは、フローサイトメトリーによって評価した。選択されたアプタマーは、細胞型特異的結合親和性を示した。第2回RNAプールと無関係のRNAが陰性対照として使用した。データは2つの複製の平均を表しています。 (D、E)内部化と細胞内局在解析。 CHO - gp160細胞を35 mmプレートで増殖させ、ヘキスト33342(生きている細胞の核染色)で染色した。その後、細胞は、前述のようにリアルタイムの生細胞共焦点顕微鏡分析用のキメラCy3標識の100 nMの濃度で培地中でインキュベートした。
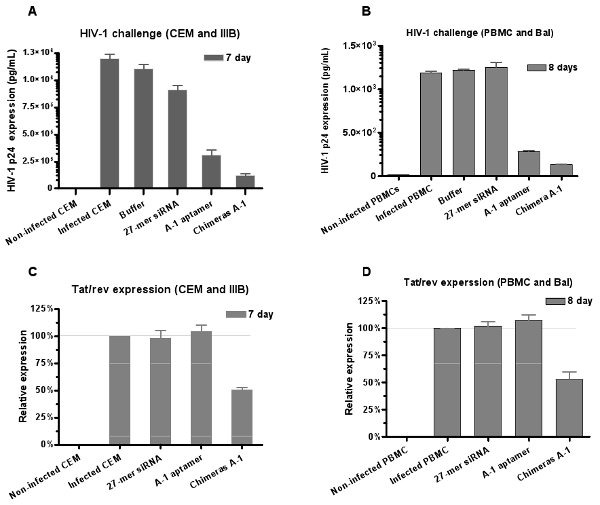
図6:アプタマー- siRNAのキメラにより媒介されるHIV - 1感染のデュアル阻害。抗gp120のアプタマーおよびアプタマー、siRNAのキメラの両方は、それぞれ、(A)CEM細胞(IIIB株)でHIV - 1感染を中和し、(B)は、ヒトの末梢血単核細胞(BAL株)文化。データは、P24の3回の測定の平均値を表しています。キメラは、siRNAがPBMC中のアプタマーダウンレギュレートTAT /回転遺伝子発現によって配信(C、D)ことを示すだけではアプタマーよりも強い阻害を示した。データは3つの平均的な複製を表しています。
ディスカッション
アプタマーは、in vitroで標的分子に8〜タイト結合、それによって高度に特異的な提供、具体的かつ安定的な三次元形状を想定して核酸を進化させています。低ナノモル結合親和性とそのターゲットへのアプタマーの絶妙な特異性がin vivoイメージング、および治療9 で 、それらの診断のための多目的なツールとして利用できます。標的siRNA導入のための...
開示事項
謝辞
我々は、有用な議論をブリッタヘーン、桂花日、ハリスSoiferとリサシェーラーに感謝。 CHO - EEとCHO - gp160細胞:この作品は、NIHのエイズ研究とレファレンス試薬プログラム、エイズの部、NIAID、NIHによって得られた健康AI29329と、以下の試薬をJJRに授与HL07470の国立研究所からの補助金によって支えられてライン、pNL4 - 3リュックベクトル、HIV - 1 DAIDSからBALのgp120、NIAID。
資料
| Name | Company | Catalog Number | Comments |
| 試薬の名前 | 会社 | カタログ番号 | コメント(省略可能) |
|---|---|---|---|
| MF -ミリポアメンブレンフィルタ | ミリポア | HAWP01300 | 孔径0.45μmの |
| Swinnexフィルターホルダー | ミリポア | SX0001300 | 13mmの直径 |
| QIAquickゲル抽出キット | QIAGEN | 28706 | DNAの精製 |
| マイクロコンYM - 30カラム | ミリポア | 42410 | RNAの濃度 |
| バイオスピン30カラム | Bio - Rad社 | 732-6250 | RNA精製 |
| TaqのPCRのDNAポリメラーゼ | シグマアルドリッチ | D1806 | |
| ThermoScript RT - PCRシステム | インビトロジェン | 11146-024 | |
| T7転写キットをDuraScribe | シャーロット | DS010925 | |
| PCR用のdNTP | ロッシュ | 1 581 295 | |
| リボ核酸、大腸菌からの転送 | シグマアルドリッチ | R1753 | tRNAの競合 |
| HIV - 1 Baの- Lのgp120タンパク質 | エイズの研究およびリファレンス試薬プログラム | 4961 | 標的タンパク質 |
| サイレンサーsiRNAの標識キット - Cy3標識 | アンビオン | 1632 | |
| 酸5月1日クロロホルム/フェノール溶液(pH 4.5) | アンビオン | AM9720 | |
| クロロホルム/イソプロパノール1分の24のソリューション | シグマ | C0549 | |
| ウシ腸ホスファターゼ(CIP) | ニューイングランドBioLab | M0290L | |
| T4ポリヌクレオチドキナーゼ | ニューイングランドBioLab | M0201L | |
| グリコーゲン | ロッシュ | 10 901 393 001 | RNA沈殿 |
| γ- 32 P - ATP | MPバイオメディカル | 013502002 | Radiactivity |
| 40パーセントAccuGel夜07時01分 | ナショナル診断 | EC - 850 | |
| 10xTBE | ナショナル診断 | EC - 860 | |
| N、N、N、N' -テトラメチルエチレンジアミン(TMEMD) | シグマアルドリッチ | T9281 | |
| 過硫酸アンモニウム(APS) | シグマアルドリッチ | A3678 | |
| L - methioineスルホキシ | シグマアルドリッチ | M5379 - 250ミリグラム | |
| RPMIメディア1640 | インビトロジェン | 11835-030 | |
| 重炭酸ナトリウム溶液、7.5%w / vの | インビトロジェン | 25080-094 | |
| 最小必須培地(MEM)(10) | インビトロジェン | 11430-030 | |
| MEM非必須アミノ酸(100) | インビトロジェン | 11140-050 | |
| をpCR 2.1とTAクローニングキット | インビトロジェン | K2040 - 01 |
参考文献
- Tuerk, C., Gold, L. Systematic evolution of ligands by exponential enrichment: RNA ligands to bacteriophage T4 DNA polymerase. Science. 249, 505-510 (1990).
- Ellington, A. D., Szostak, J. W. In vitro selection of RNA molecules that bind specific ligands. Nature. 346, 818-822 (1990).
- Robertson, D. L., Joyce, G. F. Selection in vitro of an RNA enzyme that specifically cleaves single-stranded DNA. Nature. 344, 467-468 (1990).
- Fitzwater, T., Polisky, B. A SELEX primer. Methods Enzymol. 267, 275-301 (1996).
- Weiss, C. D., White, J. M. Characterization of stable Chinese hamster ovary cells expressing wild-type, secreted, and glycosylphosphatidylinositol-anchored human immunodeficiency virus type 1 envelope glycoprotein. J Virol. 67, 7060-706 (1993).
- Vodicka, M. A. Indicator cell lines for detection of primary strains of human and simian immunodeficiency viruses. Virology. 233, 193-198> (1997).
- Zhou, J. Selection, characterization and application of new RNA HIV gp 120 aptamers for facile delivery of Dicer substrate siRNAs into HIV infected cells. Nucleic Acids Res. , (2009).
- Mayer, G. The chemical biology of aptamers. Angew Chem Int Ed Engl. 48, 2672-2689 (2009).
- Famulok, M., Hartig, J. S., Mayer, G. Functional aptamers and aptazymes in biotechnology, diagnostics, and therapy. Chem Rev. 107, 3715-3743 (2007).
- Chu, T. C., Twu, K. Y., Ellington, A. D., Levy, M. Aptamer mediated siRNA delivery. Nucleic Acids Res. 34, e73-e73 (2006).
- McNamara, J. O., 2nd, . Cell type-specific delivery of siRNAs with aptamer-siRNA chimeras. Nat Biotechnol. 24, 1005-1015 (2006).
- Dassie, J. P. Systemic administration of optimized aptamer-siRNA chimeras promotes regression of PSMA-expressing tumors. Nat Biotechnol. 27, 839-849 (2009).
- Zhou, J., Rossi, J. J. The therapeutic potential of cell-internalizing aptamers. Curr Top Med Chem. 9, 1144-1157 (2009).
- Zhou, J., Li, H., Li, S., Zaia, J., Rossi, J. J. Novel dual inhibitory function aptamer-siRNA delivery system for HIV-1 therapy. Mol Ther. 16, 1481-1489 (2008).
転載および許可
このJoVE論文のテキスト又は図を再利用するための許可を申請します
許可を申請This article has been published
Video Coming Soon
Copyright © 2023 MyJoVE Corporation. All rights reserved