Method Article
Preparazione del cloroplasto Sub-compartimenti da Arabidopsis per l'analisi di localizzazione della proteina di Immunoblotting o proteomica
In questo articolo
Riepilogo
Qui, descriviamo un metodo per purificare i cloroplasti intatti da foglie di Arabidopsis e loro tre sub-scomparti principali (busta, lo stroma e tilacoidi), utilizzando una combinazione di centrifugazione differenziale, gradienti di Percoll continui, e pendenze del saccarosio discontinuo. Il metodo è utile per subplastidial e localizzazione subcellulare delle proteine mediante immunoblotting e proteomica.
Abstract
Cloroplasti sono componenti importanti delle cellule vegetali. Tali plastidi compiono molte funzioni cruciali, come assimilazione di carbonio, zolfo e azoto, come pure la sintesi dei metaboliti essenziali. Questi organelli consistono dei seguenti tre sub-compartimenti chiavi. La busta, caratterizzata da due membrane, circonda l'organello e controlla la comunicazione del plastidio con altri compartimenti cellulari. Lo stroma è la fase solubile del cloroplasto e il sito principale dove l'anidride carbonica viene convertita in carboidrati. La membrana tilacoide è la rete di membrana interna che consiste della grana (piatto compresso ZSC) e lamelle (meno strutture dense), dove la fotosintesi oxygenic ha luogo. Il presente protocollo descrive passo per passo le procedure necessarie per la purificazione, utilizzando centrifugazione differenziale e gradienti di Percoll, dei cloroplasti intatti da Arabidopsise loro frazionamento, con pendenze del saccarosio, in tre Sub-compartimenti (cioè, busta, lo stroma e tilacoidi). Questo protocollo fornisce inoltre le istruzioni su come valutare la purezza di queste frazioni utilizzando marcatori associati ai vari sub-compartimenti cloroplasto. Il metodo qui descritto è prezioso per subplastidial la localizzazione delle proteine mediante immunoblotting, ma anche per subcellulare e subplastidial proteomica e altri studi.
Introduzione
Cloroplasti sono componenti importanti delle cellule vegetali. Essi derivano da un antenato cianobatterica che ha subito un'endosimbiosi e finalmente si è evoluto come un organulo durante evolution1,2. Tali organelli contengono tre scomparti principali (Figura 1). Il sistema di busta è composto di un interno e un esterno membrane che circondano l'organello. Questo sistema di doppia membrana contiene vari enzimi coinvolti nel metabolismo dei lipidi e pigmenti ed è per lo più dedicato al controllo della comunicazione tra plastidi e cytosol. Esso contiene vari sistemi di trasporto che consentono l'importazione delle proteine codificate e lo scambio di ioni e metaboliti tra il citosol e il cloroplasto regolando le funzioni metaboliche essenziali della pianta cella3,4 . Lo stroma, la fase solubile del cloroplasto, contiene gli enzimi del ciclo di Calvin (CO2 assimilazione), la sintesi di vari metaboliti tra cui aminoacidi e vitamine ed i macchinari di trascrizione e traduzione del plastidio. La membrana tilacoide è una rete di membrana interna ampiamente organizzato dove avviene la fase luminosa della fotosintesi. In tal modo, i cloroplasti sono il luogo dove vie metaboliche essenziali si verificano5.
Al fine di decifrare i nuovi meccanismi di regolazione che controllano la dinamica del cloroplasto e fisiologia, definendo la localizzazione sub-plastidiali delle proteine cloroplasto è così critico per supportare studi mirati con l'obiettivo di comprendere meglio le funzioni di proteine organismi di modello6. Per poter accedere alla localizzazione di vera subplastidial di queste proteine, è quindi essenziale per cominciare dalle frazioni di subplastidial altamente puro (busta membrane, stroma e tilacoidi). In questo contesto, l'obiettivo del presente protocollo è per purificare cloroplasti intatti da foglie di Arabidopsis mediante centrifugazione differenziale e pendenze continue di Percoll e frazionare li utilizzando pendenze del saccarosio discontinuo, in tre Sub-compartimenti (cioè, busta, lo stroma e tilacoidi). Il metodo qui descritto fornisce inoltre le istruzioni per valutare la purezza delle frazioni sub-organellari purificate mediante marcatori associati ai vari sub-compartimenti cloroplasto. Questo protocollo è prezioso per la localizzazione di subplastidial delle proteine mediante immunoblotting e per un'ulteriore analisi delle frazioni purificate mediante spettrometria di massa (MS)-basato su studi di proteomica.
Protocollo
1. preparazione del buffer, le soluzioni di riserva e gradienti
- Preparare le seguenti soluzioni stock che possono essere conservate fino a 6 mesi a 4 ° C.
- Preparare 1 L di Tampone tricina (1 M, pH 8,4) e Tampone tricina (1 M, pH 7,6). Regolare il pH con l'aggiunta di KOH pellet.
- Preparare 1 L di acido etilendiamminotetraacetico (EDTA, 0.5 M, pH 8) e 3-(N-morpholino) propano acido solfonico (MOPS) buffer (1 M, pH 7,8). Regolare il pH con l'aggiunta di NaOH pellet.
- Preparare 50 mL di MgCl2 (1m).
- Preparare 50 mL di proteasi inibitori soluzioni: phenylmethylsulfonyl fluoruro (PMSF istituito in isopropanolo, 100 mM), idrato di benzamidine cloridrato (100 mM) e ε-ammino acido caproico (50 mM).
Nota: Mentre PMSF e ammino acido caproico sono stabili in soluzione per mesi a 4 ° C, benzamidine soluzione deve essere conservata a-20 ° C.
- Preparare le seguenti soluzioni il giorno prima dell'esperimento e memorizzare tutte le soluzioni a 4 ° C.
- Preparare 4 L di macinazione medio pH 8,4 contenente Tricine-KOH (20 mM, pH 8,4), sorbitolo (0,4 M), EDTA (10 mM, pH 8) e NaHCO3 (10 mM). Regolare il pH con l'aggiunta di NaOH pellet. Aggiungere albumina di siero bovino (BSA) a 0,1% (p/v) appena prima dell'uso e mescolare bene.
- Preparare 500 mL di lavaggio pH medio (2x) 7,6 contenente Tricine-KOH (20 mM, pH 7.6), sorbitolo (0,8 M), MgCl2 (5 mM) ed EDTA (2,5 mM). Regolare il pH con l'aggiunta di NaOH pellet. Diluire la soluzione di questo tipo dopo la preparazione della soluzione di gradienti di Percoll ottenere lavaggio medio (1x).
- Preparare 200 mL di soluzione di gradienti di Percoll per purificazione del cloroplasto mescolando Percoll con lavaggio medio (2x) ad un volume uguale per ottenere una soluzione finale al 50% (v/v) Percoll / 0,4 M sorbitolo.
- Preparare 50 mL delle soluzioni di saccarosio per il frazionamento del cloroplasto mescolando MOPS (10 mM, pH 7,8), MgCl2 (4 mM) e le concentrazioni differenti di saccarosio (0,3 M, 0.6 M e 0,93 M).
- Preparare le seguenti sfumature e buffer prima di iniziare l'esperimento.
- Preparare sei tubi di gradienti di Percoll (ognuna contenente 30 mL di un 50% di Percoll / 0,4 M sorbitolo) mediante centrifugazione a 38.700 x g per 55 min a 4 ° C. Tenere il freno per evitare il mescolamento dei gradienti. Dopo centrifugazione, conservare le provette contenenti i gradienti preformati in una stanza fredda fino all'utilizzo.
- Preparare quattro tubi di pendenze del saccarosio, con ogni sfumatura formate da tre strati di saccarosio seguenti: 3 mL di 0,93 M, 2,5 mL di 0,6 M e 2 mL di saccarosio di 0,3 M. Sovrapposizione con attenzione ogni strato, utilizzando una pompa peristaltica con 0,93 M partendo dal basso e finitura con 0,3 M nella parte superiore.
- Preparare 50 mL di medium ipotonico per lisi cloroplasto contenente MOPS (10 mM, pH 7,8), MgCl2 (4 mM), PMSF (1 mM, istituito in isopropanolo), idrato di benzamidine cloridrato (1 mM) e ε-ammino acido caproico (0,5 mM). Memorizzare nel buffer il ghiaccio fino all'utilizzo.
- Preparare 50 mL di membrana lavaggio tampone MOPS (10 mM, pH 7,8), PMSF (1 mM), idrato di benzamidine cloridrato (1 mM) e ε-ammino acido caproico (0,5 mM). Memorizzare nel buffer il ghiaccio fino all'utilizzo.
2. crescita e la raccolta delle foglie di Arabidopsis
- Per la crescita di piante di Arabidopsis , preparare 4 vaschette di plastica grande (per una superficie totale di 0,5 a 1 m2) di piante di Arabidopsis di semina di 30 mg di semi in ogni vaschetta. Coltivare le piante di Arabidopsis per 5 settimane al ciclo di luce 12 h a 23 ° C (giorno) / 18 ° C (notte) con un'intensità di luce di 150 μM m-2 s-1.
- Incubare piante in una stanza buia e fredda (4 ° C) durante la notte prima dell'esperimento (per ridurre la quantità di granuli di amido nei cloroplasti).
- Pre-pesare un becher da 1 litro e poi metterlo sul ghiaccio prima di iniziare la raccolta di materiale fogliare.
- Raccogliere foglie di Arabidopsis evitando di suolo (compost). Ripesare il becher e registrare il peso del tessuto.
Nota: 400-500 g di materiale fogliare sono attesi da quattro padelle. - Omogeneizzare le foglie in una stanza fredda con 2 L di tampone di macinazione (aggiungere BSA prima dell'uso) tre volte / 2 s ogni volta, in un frullatore ad alta velocità.
- Filtrare l'omogeneizzato in una cella frigorifera utilizzando 4 strati di mussola e uno strato di nylon blutex. Premere delicatamente le foglie di omogenato dentro il blutex mussola/nylon per estrarre tutto il liquido.
- Recuperare il tessuto restante nella Coppa del frullatore per una seconda estrazione. Ripetere i passaggi da 2.5 e 2.6 utilizzando 2 L di macinatura media e nuovi 4-5 strati di mussola (in una stanza fredda).
3. purificazione dei cloroplasti grezzi mediante centrifugazione differenziale
- Allo stesso modo distribuire l'estratto grezzo cella in sei bottiglie da 500 mL e mettete le bottiglie sul ghiaccio prima centrifugazione. Centrifugare per 2 min, non appena la velocità massima (2.070 x g) è raggiunto (massima accelerazione e freno, 4 ° C).
- Delicatamente e scartare il surnatante.
- Aspirare il supernatante rimanente utilizzando una pompa di acqua e mantenere il pellet contenente cloroplasti grezzi concentrati sul ghiaccio.
- Risospendere delicatamente il pellet con l'aggiunta di un volume minimo di lavaggio medio (1x) (volume finale delle sospensioni combinato cloroplasto = 36 mL) utilizzando un pennello o una spatola di plastica curva. Utilizzare una pipetta da 10 mL per aggiungere 3 mL di media in ogni bottiglia di lavaggio.
Nota: Non utilizzare dispensare con punte molto fine per evitare la rottura dei cloroplasti. In alternativa, tagliare la punta di una pipetta con una lama di rasoio per generare un foro più grande blu. - Raccogliere i sedimento cloroplasti in uno tubo utilizzando una pipetta da 10 mL. Mescolare delicatamente capovolgendo la provetta per ottenere una sospensione omogenea prima del caricamento su gradienti di Percoll.
4. purificazione dei cloroplasti intatti su gradiente di Percoll continuo
- Caricare lentamente 6 mL della sospensione cloroplasto in cima a ciascuno dei sei gradienti di Percoll utilizzando una pipetta da 10 mL per evitare la rottura dei cloroplasti.
- Centrifugare le pendenze per 10 min a 4 ° C, utilizzando un rotore basculante, 13.300 x g.
Nota: L'accelerazione deve essere lento, e il freno deve essere scollegato (freno disinserito o decelerazione lenta) per evitare il mescolamento dei gradienti di Percoll. - Aspirare la fase superiore che contiene rotti cloroplasti e mitocondri intatti utilizzando una pompa di acqua e quindi recuperare intatti cloroplasti presenti nella parte inferiore (la banda larga di colore verde scuro) di fase con una pipetta da 10 mL. Fare attenzione a non aspirare i nuclei e detriti cellulari (che si trova nella parte inferiore del tubo) con cloroplasti intatti (Figura 2A).
- Diluire la sospensione del cloroplasto intatto con tampone di lavaggio (1x) 3-4 volte. Centrifugare per 2 min, non appena la velocità massima (2.070 x g, 4 ° C) è raggiunto (massima accelerazione e freno).
- Attentamente e scartare il surnatante.
- Completamente aspirare il supernatante rimanente con una pompa dell'acqua e mantenere la pallina di cloroplasti intatti concentrati sul ghiaccio.
- Prima di lisi del cloroplasto, tenere un'aliquota della frazione cloroplasto intatto di circa 1 mL di lavaggio medio (1x) per ulteriori analisi mediante elettroforesi del gel di poliacrilammide del sodio dodecil solfato (SDS-PAGE) e western blotting. Mantenere una piccola aliquota di questi cloroplasti intatti per la determinazione di concentrazione nella proteina. Conservare la frazione cloroplasto intatto in azoto liquido per ulteriori esperimenti.
5. lisi dei cloroplasti intatti utilizzando un Buffer ipotonico e purificazione dei sub-compartimenti cloroplasto su pendenze del saccarosio discontinuo
- Lisare i cloroplasti intatti purificati da risospendere il pellet in medium ipotonico contenente inibitori di proteasi (il volume finale non deve superare i 12 mL).
Nota: Da questo passaggio, l'uso di dispensare con estremità sottili (punte di blu) è possibile poiché l'integrità dei cloroplasti è no più essenziale (pipettaggio cloroplasti su e giù finchè pellet non è interamente risospese). Arabidopsis cloroplasti sono molto fragili (se paragonati a cloroplasti di pisello, per esempio) e loro Lisi sono quasi immediata dopo incubazione in medium ipotonico. - Caricare lentamente 3 mL di cloroplasti lisati sulla cima di ogni pendenze del saccarosio preformati utilizzando una pompa peristaltica.
- Ultracentrifuga i gradienti per 1 h a (70.000 x g, 4 ° C). Equilibrio di coppie di tubi mediante tampone ipotonico medio prima della centrifugazione.
- Recuperare con attenzione le proteine solubili stromale pipettando la fase superiore della sfumatura (3 mL da ogni sfumatura) (Figura 2B). Prendere un'aliquota per la determinazione della concentrazione di proteine7. Memorizzare lo stroma in azoto liquido per ulteriori esperimenti.
- Aspirare la fase superiore restante di ogni sfumatura fino alla fascia gialla con una pompa di acqua.
- Recuperare la fascia gialla (la busta) con una pipetta (circa 1 mL da ogni sfumatura). Piscina le buste in una provetta.
- Rimuovere la fase rimanente di ogni sfumatura fino al pellet thylakoid utilizzando una pompa dell'acqua.
6. lavaggio e concentrazione di tilacoide e sistemi di Membrane di busta
- Risospendere il pellet di thylakoid (verde pellet) in un volume minimo (2 mL) di membrana lavaggio buffer (1x) (con inibitori della proteasi).
- Diluire le sospensioni busta e tilacoidale 3-4 volte nel mezzo di lavaggio membrana (regolazione del volume di 10 mL) e ultracentrifuga per 1 h a (110.000 x g, 4 ° C). Bilanciare le coppie di tubi mediante membrana lavaggio tampone prima della centrifugazione.
- Aspirare accuratamente i surnatanti utilizzando una pompa dell'acqua.
- Aggiungere circa 100 µ l di tampone (con inibitori della proteasi) al pellet di busta di lavaggio della membrana. Prendere un'aliquota per la determinazione della concentrazione di proteine7. Conservare la preparazione di membrana purificata busta in azoto liquido.
- Tilacoidi risospendere in 3 mL di membrana lavaggio tampone (con inibitori della proteasi). Prendere un'aliquota per la determinazione della concentrazione di proteine7. Conservare frazione della membrana tilacoide in azoto liquido.
Risultati
Fasi successive della procedura conseguente cloroplasto purificata e loro sub-compartimenti sono riassunti nella Figura 2. Il gradiente di Percoll (Figura 2A) permette di distinguere i cloroplasti intatti da rotto cloroplasti e mitocondri (parte superiore della sfumatura) o nuclei e detriti cellulari (fondo della sfumatura). Dopo la rottura degli organelli Percoll-purificato grazie ad un shock osmotico, separate le frazioni risultanti su un gradiente di saccarosio (Figura 2B). Lo stroma (parte solubile del cloroplasto) sta galleggiando sulla superficie del gradiente di saccarosio. Le vescicole di membrana leggera busta vengono recuperate come una discreta banda gialla nell'interfaccia di saccarosio 0,6/0.93 M. Le vescicole di membrane tilacoidali più pesanti sono concentrate nella parte inferiore del tubo. Dopo il recupero, lavaggio e concentrazione delle frazioni due membrana, proteine sono quantificate e la composizione di tutte e quattro le frazioni è analizzata su un SDS-PAGE (Figura 2). Le corsie sono caricate su una base uguale proteina (20 µ g di ogni frazione purificata). Sapendo che questi contengono solo 1% di proteine dell'involucro e il 50% delle proteine dallo stroma o dai tilacoidi, questo tende a sovrastimare la contaminazione incrociata delle preparazioni purificate busta con altri sub-compartimenti cloroplasto. Tuttavia, questo metodo permette di rilevare piccole quantità di proteine cross-contaminazione la frazione di busta. Marcatori da ogni vano (cioè, proteine abbondanti) sono molto utili per valutare la contaminazione trasversale delle frazioni. Infatti, le frazioni di membrana tilacoide e busta dovranno contenere quantità molto basse della grande unità secondaria di RuBisCO (RBCL), la proteina più abbondante dallo stroma (50 kDa). Cloroplasti rotti si possono facilmente distinguere dai cloroplasti intatto a causa della perdita di questa proteina stromal8. La luce raccolta proteine complesse (LHCP) sono componenti di abbondanti thylakoid 25-kDa che dovrebbero a malapena (meno del 3%) contaminare busta membrane9. Infine, il translocator trioso fosfato (TPT) è una proteina di 30-kDa che è solo visibile nella frazione purificata busta a causa del suo forte arricchimento (vale a dire, da 50 a 100 x) nella frazione busta rispetto al cloroplasto intero estratti. Utilizzando il metodo descritto qui, Sub-compartimenti cloroplasto sono generalmente mal contaminazione incrociata come confermato mediante analisi western-blot (Figura 2D) basandosi su anticorpi diretti contro marcatori noti di tutti i tre sub-compartimenti: il solubili chetoacido reductoisomerase (KARI) dallo stroma, la busta del cloroplasto rame ATPasi (HMA1) e la luce raccolta proteine complesse (LHCP) dalle membrane tilacoidali. Contaminazione incrociata dei tre sub-compartimenti può essere quantificata utilizzando immunoblotting e la massa di analisi di spettrometria9. Mentre lo stroma è solitamente non contaminato da busta o thylakoid frazioni, le frazioni purificate busta contengono 3% delle proteine thylakoid e fino al 10% di proteine dallo stroma. Proteine dallo stroma scarsamente contaminano le membrane tilacoidali (meno dell'1%) ma tilacoidi contengono fino al 3% di proteine di membrana di busta. Più che avere un ruolo cruciale nell'identificare la posizione di genuino subplastidial delle proteine del cloroplasto, il presente metodo così limita anche conclusioni erronee sulla localizzazione di subplastidial di proteine derivanti da cross contaminazioni.
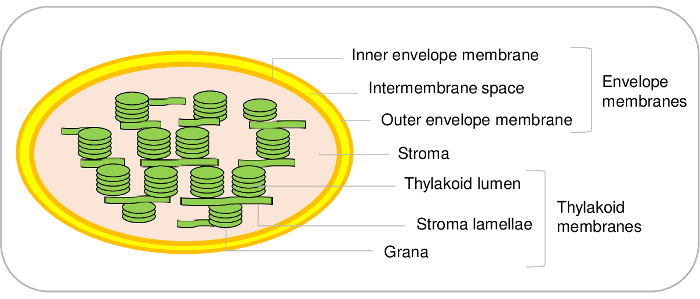
Figura 1: schema rappresentativo dei sub-compartimenti cloroplasto. Clicca qui per visualizzare una versione più grande di questa figura.
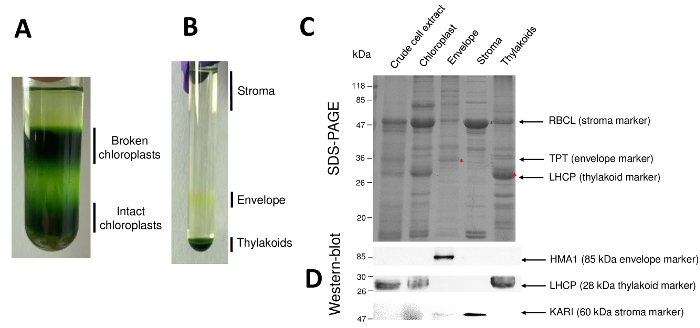
Figura 2: purificazione dei cloroplasti intatti e loro tre principali sub-scomparti utilizzando gradienti di Percoll e saccarosio. R. gradiente di Percoll consentendo la separazione dei cloroplasti rotti e intatti. B. gradiente di saccarosio che permette la separazione delle frazioni di stroma, busta e tilacoidale. C. rappresentante SDS-PAGE delle proteine da cloroplasti intatti e loro tre sub-scomparti principali che permette di visualizzare marcatori abbondanti da ogni sub-vano. Ogni corsia contiene 10 µ g di proteine. Marcatori di peso molecolare: RBCL, grande unità secondaria di RuBisCO (indicatore per lo stroma); TPT, translocator/trioso-fosfato (indicatore per la busta); LHCP, luce raccolta proteine complesse (indicatore per il thylakoid). D. gli esperimenti di Western-blot consentendo di rilevare gli indicatori specifici (utilizzando anticorpi specifici) da ogni sub-vano: il cloroplasto busta rame Atpasi HMA110, la luce raccolta proteine complesse LHCP dal thylakoid membrane11e il chetoacido reductoisomerase KARI dal stroma9. Clicca qui per visualizzare una versione più grande di questa figura.
Discussione
Il presente articolo mira al dettaglio il protocollo di passo dopo passo utilizzato per purificare i cloroplasti (e loro sub-compartimenti) da Arabidopsis thaliana. Poiché la disponibilità della sua sequenza completa del genoma quasi due decenni fa e di grandi raccolte di mutanti di inserimento resi disponibile alla comunità, Arabidopsis ora è ampiamente accettata come una pianta di modello. Tuttavia, mentre questa pianta è stato perfettamente adattata per approcci genetici, pianta gli scienziati dovuti adattare strumenti biochimici e fisiologici per questo modello emergente. Protocolli che permette di purificare fotosinteticamente attivi cloroplasti da foglie di affermati modelli biochimici come spinaci12 o pisello13 così ha dovuto essere adattato. Il primo metodo che descrive la purificazione dei cloroplasti di Arabidopsis è stato pubblicato nel 199814, poco prima del rilascio della sequenza del genoma di Arabidopsis . Diversi anni più tardi, semplici metodi per isolare i cloroplasti di Arabidopsis compatibili con studi volti ad analizzare in vitro importazione delle proteine in organelli purificati sono stati fatti disponibili15,16. Tuttavia, questi metodi non permetteva di combinare l'elevato grado di purezza e la conservazione di attività fotosintetica di cloroplasti purificati. Più recentemente i17, un metodo rapido è stata stabilita, che si basa sull'uso di gradienti di Percoll e permette di conservare quasi il 90% del tasso di fotosintesi misurato nelle foglie partenza di Arabidopsis.
Il protocollo descritto qui permette di purificare Arabidopsis cloroplasti a un eccellente livello di purezza. Infatti, immunologico rilevamento di contaminanti da altri compartimenti cellulari hanno dimostrato che gli organelli purificati sono privi di mitocondriale e della membrana plasmatica marcatori9,10. Questo protocollo è stato anche efficiente per purificare i cloroplasti da diversi ecotipi Arabidopsis 18, come Columbia (Col) o Wassilewskija (WS), vale a dire, gli ecotipi che sono stati utilizzati per genoma o espresso sequence tags (ESTs) sequenziamento progetti ma anche di generare mutanti di inserimento del T-DNA in Arabidopsis. In altre parole, quando gli studi di proteomica devono essere eseguite, il presente protocollo è compatibile con questi due ecotipi di riferimento da Arabidopsis. Infine, la resa dei cloroplasti utilizzando il presente protocollo è simile a quello ottenuto quando a partire dalle foglie di spinaci o piselli (cioè, 3%, come misurato dal contenuto clorofilla nel cloroplasto Percoll-purificato rispetto al totale quantità di clorofilla presente in partenza lascia). In termini di proteine, il rendimento è vicino a 50 mg di proteine cloroplasto, quando gli organelli sono purificati da 500 g di foglie di Arabidopsis 5-settimana-vecchio.
Raggiungere una buona resa (e l'integrità del cloroplasto), uno dovrebbe tuttavia prestare particolare attenzione a diversi passi quando si utilizza il presente protocollo. Il cloroplasto in Arabidopsis è una struttura estremamente fragile (questo non è il caso per i cloroplasti di pisello, per esempio). Specifica attenzione è così necessaria al fine di evitare la rottura su larga scala degli organelli durante purificazione. Il numero e le dimensioni dei granuli di amido presenti nei cloroplasti sono fondamentali per la preparazione dei cloroplasti intatti. Infatti, cloroplasti contenenti grano grande amido generalmente verrà interrotti durante le fasi di centrifugazione differenziale iniziale con l'obiettivo di concentrare il cloroplasto greggio frazioni12. Pertanto, le piante dovrebbero essere tenute durante la notte in una stanza buia e fredda (4 ° C) prima dell'esperimento, per ridurre la quantità di amido.
Nuovi utenti del presente protocollo potrebbero essere tentati di iniziare da grandi quantità di materiale fogliare (enorme rosette da vecchie piante di Arabidopsis con foglie più grandi) cercando di migliorare il recupero dei cloroplasti purificati. Tuttavia, nelle nostre mani, a partire da foglie giovani (5-week-old) è il miglior compromesso per combinare la resa, la purezza e l'integrità degli organelli purificati. Infatti, troppo vecchie foglie sono altamente arricchiti in composti fenolici che sono stati indicati per avere un impatto negativo su cloroplasto integrità19.
Infine, la fase di estrazione iniziale (rettifica del tessuto) è un altro passo fondamentale. Il processo di miscelazione deve essere limitato a pochi secondi. Come detto sopra, nuovi utenti potrebbero essere tentati di utilizzate la fusione dei più a lungo, così mi aspettavo di migliorare fortemente la resa degli organelli purificati. Tuttavia, se la fusione più efficacemente rilascia più materiale da foglie, sembra che la proporzione dei cloroplasti rotti aumenta rapidamente nell'estratto grezzo delle cellule. A causa di questo alto rapporto di rotto a cloroplasti intatti nel mezzo, ulteriori fasi di purificazione (separazione su gradienti di Percoll) sono fortemente influenzati e il rendimento della purificazione è inaspettatamente inferiore.
Disponibilità di protocolli specifici per purificare gli organelli hanno permesso una serie di alta velocità di trasmissione basato su proteomics esperimenti per essere condotti su campioni del cloroplasto. Questi dati sono stati resi disponibili in diverse banche dati pubbliche6, fornendo così ai biologi nel campo un'accurata localizzazione subcellulare (e subplastidial) per molte proteine del cloroplasto. Questo era particolarmente vero per le proteine dell'involucro in cui identità e posizione rimasta in gran parte sconosciuto prima di queste analisi, poiché le membrane busta rappresentano una componente minore cloroplasto (1-2% delle proteine cloroplasto) giocando un ruolo chiave nel cloroplasto metabolismo e biogenesi5,20. Mediante il protocollo descritto qui, abbiamo recentemente analizzato la composizione dei tre comparti principali cloroplasto da Arabidopsis (cioè, lo stroma, i tilacoidi e il sistema a membrana busta)9. Basato su un approccio di proteomica semi-quantitativa (contando spettrale), siamo stati in grado di valutare il partizionamento di centinaia di proteine in questi tre scomparti del cloroplasto.
Mentre il presente protocollo permette di purificare i tre scomparti principali del cloroplasto da Arabidopsis, è anche possibile distinguere ulteriori vani secondari nel cloroplasto. Infatti, il sistema a membrana busta è costituito l'interno e le membrane di busta esterna (Figura 1). Tuttavia, al meglio della nostra conoscenza, un metodo per purificare le membrane busta interna ed esterna da cloroplasti di Arabidopsis rimane essere stabilita. Membrane di busta interna ed esterna possono essere purificate da cloroplasti di22 21 o pisello di spinaci. La limitazione principale di Arabidopsis risultati per lo più dagli importi limitanti del materiale base. A partire da 500 g di foglie di Arabidopsis (che già richiede 1 m2 superficie in una camera di crescita) permette di purificare solo 100 µ g di proteine dell'involucro. D'altra parte, è facile iniziare con 5-10 kg di spinaci foglie dal mercato, per purificare grandi quantità di cloroplasti8 e terminare con una resa di 3-10 mg di proteine dell'involucro da questo materiale.
Lo stesso vale per i sub-compartimenti thylakoid. Infatti, i tilacoidi sono fatti di luce vescicole di membrana (lamelle) e strutture dense (grana) (Figura 1). Protocolli specifici sono disponibili per distinguere questi due compartimenti in Arabidopsis23,24. Ancora una volta, sulla base di un'analisi di proteomica quantitativa abbiamo recentemente inventariati le proteine presenti in questi due sub-compartimenti24. Questi approcci, insieme a un'indagine approfondita della letteratura, ha permesso la convalida o proporre ipotesi, la posizione di subplastidial di centinaia di proteine thylakoid. Tuttavia, è importante notare che microdomini di membrana aggiuntive sono presenti presso i margini ricurvi dei tilacoidi. Questi sub-compartimenti lipoproteina, o plastoglobuli, sono permanentemente accoppiati a membrane tilacoidali e contengono un insieme specifico di proteine25. Utilizzando il presente protocollo, così non è possibile distinguere queste proteine specifiche da altri componenti di tilacoide.
Alcuni componenti originali di busta, lo stroma o thylakoid (ben noti) mancano ancora dagli elenchi delle proteine rilevate. Insieme con analisi mirata di biochimiche ed immunologiche, il miglioramento continuo delle MS sensibilità sarà di grande aiuto per rivedere il contenuto del cloroplasto verso un repertorio completo della composizione dei suoi vari sub compartimenti.
Divulgazioni
Gli autori non hanno nulla a rivelare.
Riconoscimenti
Questo lavoro è stato supportato da una borsa di dottorato congiunta a IB dalla biologia vegetale INRA e divisione di allevamento e dal Labex GRAL (ANR-10-LABX-49-01). Vorremmo anche ringraziare il progetto ANR ANR-15-IDEX-02, Dr. Olivier Vallon (IBPC Parigi) per gli anticorpi contro LHCP e Dr. Renaud Dumas (LPCV, Grenoble) per gli anticorpi contro KARI.
Materiali
| Name | Company | Catalog Number | Comments |
| Percoll | GE Healthcare | 17089101 | |
| Tricine | Roth | 6977.2 | |
| Sorbitol | Roth | 6213.1 | |
| Ethylenediaminetetraacetic acid (EDTA) | Promega | H5032 | |
| NaHCO3 | Roth | 8551.1 | |
| Bovine serum albumin (BSA) | Roth | 8076.5 | |
| MgCl2 | Roth | 2189.1 | |
| Phenylmethylsulfonyl fluoride (PMSF) | Sigma | P7626 | |
| Benzamidine | Sigma | B6506 | |
| ε-amino caproic acid | Fluka | 21530 | |
| 3-(N-morpholino) propane sulfonic acid (MOPS) | Roth | 6979.3 | |
| Sucrose | Roth | 9286.2 | |
| Acrylamide stock: 30% (w/v) acrylamide, 0.8% (w/v) bisacrylamide | Roth | 3029.1 | |
| Tris | Fisher | BP152-5 | |
| Sodium dodecyl sulfate (SDS) | Roth | 1057.1 | |
| Tetramethylethylenediamine (TEMED) | Sigma | T-8133 | |
| Ammonium persulfate (APS) | Roth | 9592.1 | |
| Glycerol | Roth | 3783.1 | |
| Bromophenol blue | USB | US12370 | |
| Glycin | Roth | 3908.3 | |
| Gel staining medium | Clini-sciences | GEN-QC-STAIN | |
| Ethanol | CARLO ERBA | 528151 | |
| NaCl | Euromedex | 1112-A | |
| Triton X-100 | Promega | H5141 | |
| Fat-free milk powder | Régilait | ||
| HCl | Fisher | H/1150/PB15 | |
| KOH pellets | Sigma | 1.05012 | |
| NaOH pellets | CARLO ERBA | 480507 | |
| Anti-HMA1 antibody | Seigneurin-Berny et al, 2006 | Used at a 1:1000 dilution | |
| Anti-KARI antibody | Ferro et al, 2010 | Used at a 1:1000 dilution | |
| Anti-LHCP antibody | Vallon et al, 1991 | Used at a 1:25,000 dilution | |
| P-coumaric acid | Sigma | C-9008 | |
| Luminol (3-aminophalhydrazin) | Fluka | 9253 | |
| Dimethyl sulfoxide (DMSO). | Sigma | D5879 | |
| Large (30 cm × 45 cm) plastic cases | Puteaux | 162135 | |
| A. thaliana seeds | Around 30 mg of seeds for a whole case | ||
| Compost "Floragard" | Puteaux | 16311770 | |
| Growth rooms | 12-h light cycle, set at 23°C (day) / 18°C (night) with a light intensity of 150 μmol/m2/s. | ||
| Muslin or cheesecloth | Raffin | 70116 | 80-cm-large |
| Nylon blutex 50 μm aperture | Tripette et Renaud, Sailly Saillisel | 50 μm aperture | |
| Motor-driven blender, three speeds, 1 gallon (4 L) capacity | Waring Blender | ||
| Fixed-angle rotors JLA-10.500 (6 × 500-mL plastic bottles) | Beckman Coulter | ||
| Beckman JA-20 rotor | Beckman Coulter | ||
| JA-20 (6 × 50 mL polypropylene tubes) | Sorvall instruments | ||
| Swinging-bucket rotor JS-13.1 (6 × 50 mL polycarbonate tubes) | Beckman Coulter | ||
| SW 41 Ti rotor (6 × 13.2 mL ultraclear tubes) | Beckman Coulter | ||
| SW 41 Ti rotor tubes (13.2 mL ultraclear tubes) | Beckman Coulter | ||
| Ultracentrifuge (Beckman L7) | Beckman Coulter | ||
| Centrifuge (Beckman JSE-06D18) | Beckman Coulter | ||
| Microcentrifuge | Eppendorf 5415D or equivalent | ||
| Water pump connected to a Pasteur pipette via a plastic tube. | |||
| Nitrocellulose membranes | BA85, Schleicher and Schuell | ||
| Filter paper | 3MM, Whatman, Maidstone | ||
| Liquid nitrogen | |||
| Peristaltic pump | Gilson | ||
| Gel electrophoresis apparatus with the various accessories needed for protein separation by electrophoresis (combs, plates and casting apparatus). | Bio-Rad Protean 3 or equivalent | ||
| System for protein transfer to nitrocellulose membranes | Bio-Rad Protean 3 or equivalent |
Riferimenti
- Zimorski, V., Ku, C., Martin, W. F., Gould, S. B. Endosymbiotic theory for organelle origins. Current Opinion in Microbiology. 22, 38-48 (2014).
- Gould, S. B., Waller, R. F., McFadden, G. I. Plastid evolution. Annual Review of Plant Biology. 59, 491-517 (2008).
- Linka, N., Weber, A. P. Intracellular metabolite transporters in plants. Molecular Plant. 3 (1), 21-53 (2010).
- Block, M. A., Douce, R., Joyard, J., Rolland, N. Chloroplast envelope membranes: a dynamic interface between plastids and the cytosol. Photosynthesis Research. 92 (2), 225-244 (2007).
- Rolland, N., et al. The biosynthetic capacities of the plastids and integration between cytoplasmic and chloroplast processes. Annual Review of Genetics. 46, 233-264 (2012).
- Agrawal, G. K., et al. Plant organelle proteomics: collaborating for optimal cell function. Mass Spectrometry Reviews. 30 (5), 772-853 (2011).
- Chua, N. -. H. [40] Electrophoretic analysis of chloroplast proteins. Methods in Enzymology. 69, 434-446 (1980).
- Seigneurin-Berny, D., Salvi, D., Joyard, J., Rolland, N. Purification of intact chloroplasts from Arabidopsis and spinach leaves by isopycnic centrifugation. Current Protocols in Cell Biology. , (2008).
- Ferro, M., et al. AT_CHLORO, a comprehensive chloroplast proteome database with subplastidial localization and curated information on envelope proteins. Molecular & Cell Proteomics. 9 (6), 1063-1084 (2010).
- Seigneurin-Berny, D., et al. HMA1, a new Cu-ATPase of the chloroplast envelope, is essential for growth under adverse light conditions. Journal of Biological Chemistry. 281 (5), 2882-2892 (2006).
- Vallon, O., et al. Lateral redistribution of cytochrome b6/f complexes along thylakoid membranes upon state transitions. Proceedings of the National Academy of Sciences of the United States of America. 88 (18), 8262-8266 (1991).
- Douce, R. J. J., Edelman, M., Hallick, R. B., Chua, N. -. H. Purification of the chloroplast. Methods in Chloroplast Molecular Biology. , 239-256 (1982).
- Cerovic, Z. G., Plesnicar, M. An improved procedure for the isolation of intact chloroplasts of high photosynthetic capacity. Biochemical Journal. 223 (2), 543-545 (1984).
- Kunst, L. Preparation of physiologically active chloroplasts from Arabidopsis. Methods in Molecular Biology. 82, 43-48 (1998).
- Aronsson, H., Jarvis, P. A simple method for isolating import-competent Arabidopsis chloroplasts. FEBS Letters. 529 (2-3), 215-220 (2002).
- Ling, Q., Jarvis, P. Analysis of Protein Import into Chloroplasts Isolated from Stressed Plants. Journal of Visualized Experiments. (117), (2016).
- Seigneurin-Berny, D., Salvi, D., Dorne, A. J., Joyard, J., Rolland, N. Percoll-purified and photosynthetically active chloroplasts from Arabidopsis thaliana leaves. Plant Physiology and Biochemistry. 46 (11), 951-955 (2008).
- Salvi, D., Rolland, N., Joyard, J., Ferro, M. Purification and proteomic analysis of chloroplasts and their sub-organellar compartments. Methods in Molecular Biology. 432, 19-36 (2008).
- Walker, D. . The use of the oxygen electrode and fluorescence probes in simple measurements of photosynthesis. , (1990).
- Jarvis, P., Lopez-Juez, E. Biogenesis and homeostasis of chloroplasts and other plastids. Nature Reviews Molecular Cell Biology. 14 (12), 787-802 (2013).
- Block, M. A., Dorne, A. J., Joyard, J., Douce, R. Preparation and characterization of membrane fractions enriched in outer and inner envelope membranes from spinach chloroplasts. II. Biochemical characterization. Journal of Biological Chemistry. 258 (21), 13281-13286 (1983).
- Soll, J. Phosphoproteins and protein-kinase activity in isolated envelopes of pea (Pisum sativum L.) chloroplasts. Planta. 166 (3), 394-400 (1985).
- Moyet, L., Salvi, D., Tomizioli, M., Seigneurin-Berny, D., Rolland, N. Preparation of Membrane Fractions (Envelope, Thylakoids, Grana, and Stroma Lamellae) from Arabidopsis Chloroplasts for Quantitative Proteomic Investigations and Other Studies. Methods in Molecular Biology. 1696, 117-136 (2018).
- Tomizioli, M., et al. Deciphering thylakoid sub-compartments using a mass spectrometry-based approach. Molecular & Cellular Proteomics. 13 (8), 2147-2167 (2014).
- Spicher, L., Kessler, F. Unexpected roles of plastoglobules (plastid lipid droplets) in vitamin K1 and E metabolism. Current Opinion in Plant Biology. 25, 123-129 (2015).
Ristampe e Autorizzazioni
Richiedi autorizzazione per utilizzare il testo o le figure di questo articolo JoVE
Richiedi AutorizzazioneThis article has been published
Video Coming Soon