È necessario avere un abbonamento a JoVE per visualizzare questo. Accedi o inizia la tua prova gratuita.
Method Article
Fotometria multifibra per registrare l'attività neurale in animali liberamente
In questo articolo
Riepilogo
Questo protocollo descrive in dettaglio come implementare ed eseguire registrazioni di fotometria multifibra, come correggere gli artefatti indipendenti dal calcio e considerazioni importanti per l'imaging di fotometria a doppio colore.
Abstract
Registrare l'attività di un gruppo di neuroni in un animale liberamente mobile è un'impresa impegnativa. Inoltre, poiché il cervello viene sezionato in sottogruppi funzionali sempre più piccoli, diventa fondamentale registrare da proiezioni e/o sottopopolazioni geneticamente definite di neuroni. La fotometria in fibra è un approccio accessibile e potente in grado di superare queste sfide. Combinando metodologie ottiche e genetiche, l'attività neurale può essere misurata in strutture cerebrali profonde esprimendo indicatori di calcio codificati geneticamente, che traducono l'attività neurale in un segnale ottico che può essere facilmente misurato. Il protocollo attuale descrive in dettaglio i componenti di un sistema di fotometria multifibra, come accedere alle strutture cerebrali profonde per fornire e raccogliere la luce, un metodo per tenere conto degli artefatti di movimento e come elaborare e analizzare i segnali fluorescenti. Il protocollo descrive in dettaglio le considerazioni sperimentali quando si esegue l'imaging a singolo e doppio colore, da fibre ottiche impiantate singole o multiple.
Introduzione
La capacità di correlare le risposte neurali con aspetti specifici del comportamento di un animale è fondamentale per comprendere il ruolo che un particolare gruppo di neuroni svolge nel dirigere o rispondere a un'azione o uno stimolo. Data la complessità del comportamento animale, con la miriade di stati interni e stimoli esterni che possono influenzare anche le azioni più semplici, la registrazione di un segnale con risoluzione a prova singola dota i ricercatori degli strumenti necessari per superare questi Limitazioni.
La fotometria in fibra è diventata la tecnica di scelta per molti ricercatori nel campo delle neuroscienze dei sistemi a causa della sua relativa semplicità rispetto ad altre tecniche di registrazione in vivo, il suo elevato rapporto segnale-rumore e la capacità di registrare in una varietà di paradigmi comportamentali1,2,3,4,5,6,7,8. A differenza dei metodi elettrofisiologici tradizionali, la fotometria è l'approccio ottico più comunemente usato in combinazione con indicatori di calcio codificati geneticamente (GECI, la serie GCaMP)9. I CCIi cambiano la loro capacità di fluorescenza in base al fatto che siano legati o meno al calcio. Poiché la concentrazione interna di calcio nei neuroni è molto strettamente regolata e i canali di calcio legati alla tensione si aprono quando un neurone attiva un potenziale d'azione, aumenta la concentrazione interna di calcio, che si traduce in aumenti transitori capacità di un GECI di fluorescenza, può essere un buon proxy per la cottura neuronale9.
Con la fotometria in fibra, la luce di eccitazione viene diretta lungo una sottile fibra ottica multimodale nel cervello e un segnale di emissione viene raccolto attraverso la stessa fibra. Poiché queste fibre ottiche sono leggere e pieghevoli, un animale può muoversi in gran parte senza ostacoli, rendendo questa tecnica compatibile con una vasta gamma di test comportamentali e condizioni. Alcune condizioni, come i movimenti rapidi o la flessione del cavo patch in fibra ottica oltre il raggio in cui può mantenere la riflessione interna totale, possono introdurre artefatti del segnale. Per disambiguare il segnale dal rumore, possiamo sfruttare una proprietà di GCaMP nota come "punto isosbestico". In breve, con GCaMP, mentre la lunghezza d'onda della luce di eccitazione viene spostata a sinistra, la sua emissione nello stato legato al calcio diminuisce e l'emissione nello stato non legato al calcio aumenta marginalmente. Il punto in cui l'intensità relativa di queste due emissioni è uguale è definita punto isosbestico. Quando GCaMP è eccitato a questo punto, la sua emissione non è influenzata da cambiamenti nelle concentrazioni interne di calcio, e la varianza nel segnale è più spesso dovuta all'attenuazione del segnale da sovraccarico del cavo patch in fibra ottica o movimento del tessuto neurale rispetto alla fibra impiantata.
L'elettrofisiologia a unità singola è ancora il gold standard per le registrazioni in vivo liberamente in movimento grazie alla sua risoluzione a cella singola e a picco singolo. Tuttavia, può essere difficile individuare l'identità molecolare delle cellule registrate e l'analisi post-hoc può essere abbastanza laboriosa. Mentre la fotometria in fibra non ha la risoluzione a cella singola, permette ai ricercatori di porre domande impossibili da affrontare con tecniche tradizionali. Combinando strategie virali con animali transgenici, l'espressione delle GeCI può essere diretta a tipi neuronali geneticamente definiti per registrare l'attività neurale definita dalla popolazione o dalla proiezione, che può essere eseguita monitorando il segnale di calcio direttamente all'assone terminali10,11. Inoltre, impiantando più cannule in fibra ottica, è possibile monitorare contemporaneamente l'attività neurale da diverse regioni e percorsi del cervello nello stesso animale12,13.
In questo manoscritto, descriviamo una tecnica per la fotometria singola e multifibra, come correggere artefatti indipendenti dal calcio e dettagliamo come eseguire registrazioni mono e doppio colore. Forniamo anche esempi dei tipi di domande che consente di porre e dei loro livelli crescenti di complessità (vedere Figura 1). La configurazione della fotometria in fibra per le registrazioni multifibra descritte in questo protocollo può essere costruita utilizzando un elenco di materiali trovati allhttps://sites.google.com/view/multifp/hardware (Figura 2).
È essenziale che il sistema sia equipaggiato sia per lunghezze d'onda di eccitazione di 410 e 470 nm per l'emissione di fluorescenza indipendente dal calcio e dal calcio da GCaMP6 o le sue varianti. Per le configurazioni personalizzate o se non c'è nessun software disponibile per eseguire il sistema, il programma open source gratuito Bonsai (http://www.open-ephys.org/bonsai/) può essere utilizzato. In alternativa, la fotometria in fibra può essere eseguita attraverso MATLAB (ad esempio, https://github.com/deisseroth-lab/multifiber)12 o un altro linguaggio di programmazione14. Il software e l'hardware del sistema dovrebbero consentire la manipolazione di entrambi i LED da 410 nm e 470 nm e della telecamera, l'estrazione di immagini(Figura 2)e il calcolo dell'intensità media fluorescente nelle regioni di interesse (ROI) disegnate intorno alle fibre le immagini. L'output deve essere una tabella dei valori di intensità media registrati con i LED da 470 nm e 410 nm di ogni fibra nel cavo del cerotto. Quando si eseguono esperimenti multifibra, 400 m di fibre in bundle possono limitare il movimento dei topi. In questi casi, si consiglia di utilizzare cavi di patch da 200 m, che offrono maggiore flessibilità. Può anche essere possibile utilizzare cavi fittizi più piccoli durante l'allenamento dei topi.
È fondamentale essere in grado di estrarre i punti temporali per gli eventi di interesse durante l'acquisizione della fotometria in fibra. Se il sistema non fornisce facilmente un sistema integrato per integrare i TTL per eventi specifici, una strategia alternativa consiste nell'assegnare un timestamp ai singoli punti temporali registrati per allinearsi a orari ed eventi specifici durante l'esperimento. Il timestamp può essere fatto utilizzando l'orologio del computer.
Protocollo
Tutti gli esperimenti sono stati fatti in conformità con i Comitati istituzionali per la cura e l'uso degli animali dell'Università della California, San Diego, e la Canadian Guide for the Care and Use of Laboratory Animals e sono stati approvati dall'Université Laval Animal Protection comitato.
1. Allineamento del percorso ottico tra la telecamera CMOS (semiconduttore di ossido di metallo complementare) e il cavo patch individuale o ramificato
- Allenta tutte le viti sul traduttore a 5 assi (11, Figura 2B).
- Vite nel cavo patch (12, Figura 2B) all'adattatore [SMA (sotto-inminiatura A) o FC (connettore in fibra ottica)] che è apposto al traduttore a 5 assi.
- Accendere la luce di eccitazione 470 nm (1, Figura 2B) a bassa potenza (100 W) e posizionare la punta del patchcord che punta a un vetrino di plastica autofluorescente. Questo non ha alcuna influenza sulle registrazioni future, ma è esclusivamente per visualizzare il processo di allineamento.
- Registrare dalla fotocamera CMOS (13, Figura 2B) in modalità live. Aumentare il guadagno o regolare la tabella di ricerca (LUT) fino a quando l'immagine non è completamente nera. Il punto è quello di essere in grado di vedere un'immagine al punto focale dell'obiettivo (10, Figura 2B).
- Far avanzare il traduttore a 5 assi verso l'obiettivo, assicurando che la luce da 470 nm sia centrata sulla fibra all'estremità SMA o FC del cavo patch, fino a quando un'immagine non può essere risolta sulla fotocamera.
- Regolare gli assi X e Y fino a quando l'immagine non viene centrata e ben risolta.
- Visualizzate la luce emessa dall'estremità ferrule del cavo patch. Dovrebbe apparire come un cerchio isotropico. Se si utilizza un cavo di patch di ramificazione, la quantità di luce emessa alle estremità ferrule di ogni cavo di patch dovrebbe essere simile. Se il cerchio non è isotropico o la luce emessa non è uguale, regolare il traduttore a 5 assi nell'asse X-Y.
2. Impostazione di ROI intorno alle fibre per la misurazione dell'intensità media fluorescente
- Accendere tutte le luci di eccitazione per visualizzare meglio le fibre. Regolare il guadagno della fotocamera in modo che nessun pixel sia saturo e sia presente un'immagine chiara delle fibre.
- Registrare dal vivo o scattare un'immagine preliminare.
- Disegnare ROI intorno alle fibre e tenerli per la misurazione dei valori di intensità media durante le registrazioni (Figura 2A).
- Per registrazioni multiple in fibra, verificare l'indipendenza nei segnali.
- Record dal vivo da tutte le fibre.
- Puntare una fibra verso una fonte di luce e toccare con un dito. Le fluttuazioni molto grandi dovrebbero verificarsi esclusivamente in quel canale (perdita accettabile 1:1000).
- Se i segnali non sono indipendenti, ridisegnare i ROI più conservativi e ripetere il test di indipendenza.
- Per etichettare e tenere traccia di quale ROI corrisponde a quale fibra, nastro colorato o smalto può essere applicato alla fine delle fibre. Scatta una foto prima dell'inizio di qualsiasi esperimento come promemoria secondario.
3. Impostazione dell'arena di registrazione
- Appendere il cavo di patch sopra l'arena utilizzando supporti, morsetti o supporti.
- Assicurarsi che l'animale possa muoversi liberamente in tutta l'arena, disinibito dalla lunghezza della fibra.
- Se viene utilizzata una scatola operante o un campo aperto, assicurarsi che il cavo di patch sarà in grado di raggiungere l'animale con una flessione minima. Se questo richiede un colpo naso, assicurarsi che ci sia abbastanza spazio in testa per evitare la flessione della fibra. Evitare qualsiasi flessione o torsione eccessiva del cavo patch.
4. Registrazioni in vivo
NOTA: La procedura di impianto di cannula in fibra ottica per esperimenti di fotometria in fibra è identica alla procedura di optogenetica come descritto in Sparta et al15. Si consiglia di utilizzare cemento dentale (vedi Tabella dei materiali), che fornisce robusto ancoraggio della testata all'osso del cranio. Il cemento dentale sarà particolarmente utile nei casi in cui non è possibile utilizzare viti di ancoraggio.
- Ispezionare visivamente l'estremità distale delle fibre del cavo patch ad occhio e con un microscopio a minifibra. Se la superficie delle fibre è graffiata, rifinire le fibre utilizzando la lucidatura della fibra / pellicola di lamiera con grinta fine (1 m e 0,3 m).
- Pulire le estremità distese del cavo patch con 70% di etanolo e un applicatore di punta di cotone.
- Pulire le cannule in fibra ottica utilizzando il 70% di etanolo e un applicatore a punta di cotone.
- Collegare l'estremità ferrule del cavo patch alla fibra impiantata utilizzando un manicotto diviso in ceramica coperto con un tubo di strizzacervelli nero. Durante la connessione, assicurarsi che la manica sia stretta, altrimenti utilizzare una nuova manica.
NOTA: Ci sarà una grande quantità di perdita di segnale se c'è spazio tra il cordone del cerotto e l'impianto, e le registrazioni non funzioneranno. - Lasciare che l'animale si riprenda per alcuni minuti prima dell'inizio dei test comportamentali.
- Avviare la registrazione del segnale ottico ed eseguire l'esperimento.
- Durante la registrazione, tieni d'occhio la traccia dal vivo per garantire registrazioni di qualità. Si prevede che il segnale diminuisca rapidamente in funzione del tempo nei primi 2 min di registrazione. Questo effetto è causato dal decadimento LED mediato dal calore, per cui l'aumento di calore aumenta la resistenza dell'elemento ottico.
- Se si verifica un salto nel segnale che supera la cinetica on/off di GCaMP, questo è spesso un'indicazione che la manica non è abbastanza stretta e lo spazio tra il cavo patch e l'impianto sta cambiando. In questo caso, interrompere l'esperimento e ricollegare l'animale utilizzando una nuova manica.
5. Analisi dei dati di fotometria in fibra
NOTA: Questo è un metodo per l'analisi dei dati che funziona bene per la maggior parte delle registrazioni. Tuttavia, è possibile implementare approcci alternativi. Codice di esempio per l'analisi dei dati è disponibile qui: https://github.com/katemartian/Photometry_data_processing.
- Estrarre i valori di intensità di fluorescenza registrati da 470 nm (Int470) e 410 nm(Int410) LED, corrispondenti a ogni singola fibra.
- Ammorbidire ogni segnale utilizzando un algoritmo della media mobile (Figura 3A).
- Eseguire la correzione della linea di base di ogni segnale (Figura 3A e 3B) utilizzando l'algoritmo adattivo appesantito reppesantito penalmente Penalmente Penalized Least Squares (airPLS) (https://github.com/zmzhang/airPLS) per rimuovere la pendenza e la bassa frequenza fluttuazioni dei segnali.
- Standardizzare ogni segnale utilizzando il valore medio e la deviazione standard (Figura 3C):
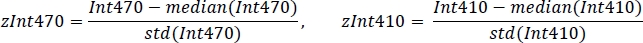
- Utilizzando la regressione lineare robusta non negativa, adattare i segnali daInt410 a z(Figura3D) alla funzione di regressione:
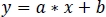
- Utilizzare i parametri della regressione lineare (a, b) per trovare nuovi valori di zInt410 montato su zInt470 (fitInt410, Figura 3D,E):
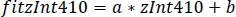
- Utilizzare i parametri della regressione lineare (a, b) per trovare nuovi valori di zInt410 montato su zInt470 (fitInt410, Figura 3D,E):
- Calcolare il valore dF/F normalizzato (z dF/F) (Figura 3F):
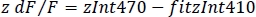
6. Registrazioni simultanee a doppio colore
- Aggiungere al sistema di fotometria un LED di 560 nm per eccitare il sensore di calcio fluorescente rosso e specchi e filtri dicromatici appropriati (vedi Kim et al., 2016 per una descrizione dettagliata)12.
- Aggiungere una barra di divisione dell'immagine tra l'obiettivo e la fotocamera CMOS per separare le lunghezze d'onda di emissione verde e rossa (vedere Figura 5). Lo splitter di immagini formerà due immagini speculari sul sensore della fotocamera, corrispondenti ai segnali rossi e verdi (ad esempio, un cavo patch con 3 rami creerà un'immagine con 6 fibre).
- Disegna ROI intorno a tutte le fibre in entrambi i colori come descritto sopra. Assicurarsi di identificare chiaramente ogni ROI con la fibra e il canale corrispondenti (verde e rosso) (Figura 4A).
- Attivare l'eccitazione simultanea con 470 nm e 560 nm LED e alternarli con 410 nm LED (Figura 5A).
7. Doppia analisi dei dati a colori
- Seguire i passaggi nella Sezione 5 per trovare fitInt410 per il segnale Int470 e calcolare z dF/F.
- Poiché il punto isosortico per i GERI con spostamento rosso è generalmente sconosciuto, il segnale registrato con LED di 410 nm nel canale verde può essere utilizzato per la correzione del movimento su entrambi i canali. Seguire i passaggi nella Sezione 5 per trovare fitInt410 per il segnale Int560 e calcolare z dF/F.
Risultati
Correlazioni neurali delle risposte comportamentali possono variare a seconda di una varietà di fattori. In questo esempio, abbiamo usato la fotometria in fibra in vivo per misurare l'attività dei terminali assoni dall'area ipotalamica laterale (LHA) che terminano nell'habenula laterale (LHb). I topi selvatici sono stati iniettati con un virus adeno-associato (AAV) codifica GCaMP6s (AAV-hSyn-GCaMP6s) nella LHA e una fibra ottica è stata impiantata con la punta immediatamente sopra la LHb (Figura 4...
Discussione
La fotometria in fibra è un approccio accessibile che consente ai ricercatori di registrare la dinamica del calcio sfuso da popolazioni neuronali definite in animali liberamente in movimento. Questo metodo può essere combinato con una vasta gamma di test comportamentali, tra cui attività "movimento pesante" come test di nuoto forzato2, paura-condizionamento18, interazioni sociali1,4, e altri7
Divulgazioni
Sage Aronson è il CEO e fondatore di Neurophotometrics Ltd., che vende sistemi di fotometria multifibra.
Riconoscimenti
Questo lavoro è stato sostenuto da una sovvenzione del Natural Sciences and Engineering Research Council del Canada (NSERC: RGPIN-2017-06131) a C.P. C. P. è un chercheur-Boursier FRSQ. Ringraziamo anche il Plateforme d'Outils Moléculaires (https://www.neurophotonics.ca/fr/pom) per la produzione dei vettori virali utilizzati in questo studio.
Materiali
| Name | Company | Catalog Number | Comments |
| 1/4"-20 Stainless Steel Cap Screw, 1" Long | Thorlabs | SH25S100 | |
| 1/4"-20 Stainless Steel Cap Screw, 1/2" Long | Thorlabs | SH25S050 | |
| 1/4"-20 Stainless Steel Cap Screw, 3/8" Long | Thorlabs | SH25S038 | |
| 1000 µm, 0.50 NA, SMA-SMA Fiber Patch Cable | Thorlabs | M59L01 | |
| 12.7 mm Optical Post | Thorlabs | TR30/M | |
| 12.7 mm Pedestal Post Holder | Thorlabs | PH20EM | |
| 15 V, 2.4 A Power Supply Unit with 3.5 mm Jack Connector for T-Cube | Thorlabs | KPS101 | |
| 20x objective | Thorlabs | RMS20X | #10 in Figure 2, #11 in Figure 5 |
| 30 mm Cage Cube with Dichroic Filter Mount | Thorlabs | CM1-DCH/M | #8-9 in Figure 2, #8-10 in Figure 5 |
| 405 nm LED | Doric Lenses | CLED_405 | #2 in Figure 2 |
| 410 nm bandpass filter | Thorlabs | FB410-10 | #5 in Figure 2; #7 in Figure 5 |
| 465 nm. LED | Doric Lenses | CLED_465 | #1 in Figure 2 |
| 470 nm bandpass filter | Thorlabs | FB470-10 | #4 in Figure 2; #6 in Figure 5 |
| 560 nm bandpass filter | Semrock | FF01-560/14-25 | #5 in Figure 5 |
| 560 nm LED | Doric Lenses | CLED_560 | #1 in Figure 3 |
| 5-axis kinematic Mount | Thorlabs | K5X1 | #11 in Figure 2, #12 in Figure 5 |
| Achromatic Doublet | Thorlabs | AC254-035-A-ML | #7 in Figure 2 |
| Adaptor for 405 collimator | Thorlabs | AD11F | #3 in Figure 2; #4 in Figure 5 |
| Adaptor for ajustable collimator | Thorlabs | AD127-F | #3 in Figure 2; #4 in Figure 5 |
| Aluminum Breadboard | Thorlabs | MB1824 | |
| Clamping Fork | Thorlabs | CF125 | |
| Cube connector | Thorlabs | CM1-CC | |
| Dual 493/574 dichroic | Semrock | FF493/574-Di01-25x36 | #10 in Figure 5 |
| Emission filter for GCaMP6 | Semrock | FF01-535/22-25 | #6 in Figure 2 |
| Enclosure with Black Hardboard Panels | Thorlabs | XE25C9 | |
| Externally SM1-Threaded End Cap for Machining | Thorlabs | SM1CP2M | |
| Fast-change SM1 Lens Tube Filter Holder | Thorlabs | SM1QP | #4-6 in Figure 2, #5-7 in Figure 5 |
| Fixed Collimator for 405 nm light | Thorlabs | F671SMA-405 | #3 in Figure 2; #4 in Figure 5 |
| Fixed collimator for 470 and 560 nm light | Thorlabs | F240SMA-532 | #3 in Figure 2; #4 in Figure 5 |
| Green emission filter | Semrock | FF01-520/35-25 | In light beam splitter |
| High-Resolution USB 3.0 CMOS Camera | Thorlabs | DCC3260M | #13 in Figure 2, #15 in Figure 5 |
| Light beam splitter | Neurophotometrics | SPLIT | #14 in Figure 5 |
| Longpass Dichroic Mirror, 425 nm Cutoff | Thorlabs | DMLP425R | #8 in Figure 2, #9 in Figure 5 |
| Longpass Dichroic Mirror, 495 nm Cutoff | Semrock | FF495-Di03 | #9 in Figure 2, #8 in Figure 5 |
| Metabond dental cement | C&B | ||
| M8 - M8 cable | Doric Lenses | Cable_M8-M8 | |
| Optic fiber cannulas | Doric Lenses | Need to specify that these will be used to photometry experiments requiring low autofluorescence | |
| Optic fiber Patchcords | Doric Lenses | Need to specify that these will be used to photometry experiments requiring low autofluorescence | |
| Red emission filter | Semrock | FF01-600/37-25 | In light beam splitter |
| T7 LabJack | LabJack | ||
| T-cube LED Driver | Thorlabs | LEDD1B | |
| USB 3.0 I/O Cable, Hirose 25, for DCC3240 | Thorlabs | CAB-DCU-T3 |
Riferimenti
- Gunaydin, L. A., et al. Natural Neural Projection Dynamics Underlying Social Behavior. Cell. 157 (7), 1535-1551 (2014).
- Proulx, C. D., et al. A neural pathway controlling motivation to exert effort. Proceedings of the National Academy of Sciences of the United States of America. 115 (22), 5792-5797 (2018).
- Muir, J., et al. In Vivo Fiber Photometry Reveals Signature of Future Stress Susceptibility in Nucleus Accumbens. Neuropsychopharmacology. 43 (2), 255-263 (2017).
- Wang, D., et al. Learning shapes the aversion and reward responses of lateral habenula neurons. eLife. 6, (2017).
- de Jong, J. W., et al. A Neural Circuit Mechanism for Encoding Aversive Stimuli in the Mesolimbic Dopamine System. Neuron. 101 (1), 133-151 (2018).
- Lerner, T. N., et al. Intact-Brain Analyses Reveal Distinct Information Carried by SNc Dopamine Subcircuits. Cell. 162 (3), 635-647 (2015).
- Calipari, E. S., et al. In vivo imaging identifies temporal signature of D1 and D2 medium spiny neurons in cocaine reward. Proceedings of the National Academy of Sciences of the United States of America. 113 (10), 2726-2731 (2016).
- González, A. J., et al. Inhibitory Interplay between Orexin Neurons and Eating. Current Biology. 26 (18), 2486-2491 (2016).
- Chen, T. -. W., et al. Ultrasensitive fluorescent proteins for imaging neuronal activity. Nature. 499 (7458), 295-300 (2013).
- Barker, D. J., et al. Lateral Preoptic Control of the Lateral Habenula through Convergent Glutamate and GABA Transmission. Cell Reports. 21 (7), 1757-1769 (2017).
- Siciliano, C. A., Tye, K. M. Leveraging calcium imaging to illuminate circuit dysfunction in addiction. Alcohol. 74, 47-63 (2018).
- Kim, C. K., et al. Simultaneous fast measurement of circuit dynamics at multiple sites across the mammalian brain. Nature Methods. 13 (4), 325-328 (2016).
- Sych, Y., Chernysheva, M., Sumanovski, L. T., Helmchen, F. High-density multi-fiber photometry for studying large-scale brain circuit dynamics. Nature Methods. 16 (6), 553-560 (2019).
- Akam, T., Walton, M. E. pyPhotometry: Open source Python based hardware and software for fiber photometry data acquisition. Scientific Reports. 9 (1), 3521 (2019).
- Sparta, D. R., et al. Construction of implantable optical fibers for long-term optogenetic manipulation of neural circuits. Nature Protocol. 7 (1), 12-23 (2011).
- Stamatakis, A. M., et al. Lateral Hypothalamic Area Glutamatergic Neurons and Their Projections to the Lateral Habenula Regulate Feeding and Reward. The Journal of Neuroscience. 36 (2), 302-311 (2016).
- Tervo, G. D., et al. A Designer AAV Variant Permits Efficient Retrograde Access to Projection Neurons. Neuron. 92 (2), 372-382 (2016).
- Yu, K., da Silva, P., Albeanu, D. F., Li, B. Central Amygdala Somatostatin Neurons Gate Passive and Active Defensive Behaviors. The Journal of Neuroscience. 36 (24), 6488-6496 (2016).
- Falkner, A. L., Grosenick, L., Davidson, T. J., Deisseroth, K., Lin, D. Hypothalamic control of male aggression-seeking behavior. Nature Neuroscience. 19 (4), 596-604 (2016).
- Ren, J., et al. Anatomically Defined and Functionally Distinct Dorsal Raphe Serotonin Sub-systems. Cell. 175 (2), 472-487 (2018).
- Barnett, L. M., Hughes, T. E., Drobizhev, M. Deciphering the molecular mechanism responsible for GCaMP6m’s Ca2+-dependent change in fluorescence. PLOS ONE. 12 (2), 0170934 (2017).
- Sun, F., et al. A Genetically Encoded Fluorescent Sensor Enables Rapid and Specific Detection of Dopamine in Flies, Fish, and Mice. Cell. 174 (2), 481-496 (2018).
- Patriarchi, T., et al. Ultrafast neuronal imaging of dopamine dynamics with designed genetically encoded sensors. Science. 360 (6396), (2018).
- Feng, J., et al. A Genetically Encoded Fluorescent Sensor for Rapid and Specific In Detection of Norepinephrine. Neuron. 102 (4), 745-761 (2019).
- Akerboom, J., et al. Genetically encoded calcium indicators for multi-color neural activity imaging and combination with optogenetics. Frontiers in Molecular Neuroscience. 6, 1-29 (2013).
- Dana, H., et al. Sensitive red protein calcium indicators for imaging neural activity. eLife. 5, (2016).
- Wang, H., Jing, M., Li, Y. Lighting up the brain: genetically encoded fluorescent sensors for imaging neurotransmitters and neuromodulators. Current Opinion in Neurobiology. 50, 171-178 (2018).
- Lu, L., et al. Wireless optoelectronic photometers for monitoring neuronal dynamics in the deep brain. Proceedings of the National Academy of Sciences. 115 (7), 1374-1383 (2018).
- Jennings, J. H., et al. Visualizing Hypothalamic Network Dynamics for Appetitive and Consummatory Behaviors. Cell. 160 (3), 516-527 (2014).
Ristampe e Autorizzazioni
Richiedi autorizzazione per utilizzare il testo o le figure di questo articolo JoVE
Richiedi AutorizzazioneThis article has been published
Video Coming Soon