Method Article
Aislamiento de núcleos de riñón de ratón adulto para secuenciación de ARN de un solo núcleo
* Estos autores han contribuido por igual
En este artículo
Resumen
Aquí, presentamos un protocolo para aislar núcleos de alta calidad de riñones de ratones congelados que mejoran la representación de los tipos de células renales medulares y evitan los artefactos de expresión génica de la disociación enzimática del tejido.
Resumen
Los riñones regulan diversos procesos biológicos como el agua, el electrolito y la homeostasis ácido-base. Las funciones fisiológicas del riñón son ejecutadas por múltiples tipos de células dispuestas en una arquitectura compleja a través del eje corticomedular del órgano. Los avances recientes en transcriptómica unicelular han acelerado la comprensión de la expresión génica específica del tipo de célula en la fisiología renal y la enfermedad. Sin embargo, los protocolos de disociación tisular basados en enzimas, que se utilizan con frecuencia para la secuenciación de ARN unicelular (scRNA-seq), requieren principalmente tejido fresco (no archivado), introducen respuestas de estrés transcripcional y favorecen la selección de abundantes tipos de células de la corteza renal, lo que resulta en una representación insuficiente de las células de la médula.
Aquí, presentamos un protocolo que evita estos problemas. El protocolo se basa en el aislamiento de núcleos a 4 °C del tejido renal congelado. Los núcleos se aíslan de una pieza central del riñón del ratón compuesta por la corteza, la médula externa y la médula interna. Esto reduce la sobrerrepresentación de las células corticales típicas de las muestras de riñón entero en beneficio de las células medulares, de modo que los datos representarán todo el eje corticomedular con suficiente abundancia. El protocolo es simple, rápido y adaptable y proporciona un paso hacia la estandarización de la transcriptómica de núcleos únicos en la investigación renal.
Introducción
Los riñones muestran una arquitectura tisular altamente compleja. Consisten en segmentos funcional y anatómicamente distintos a lo largo de un eje corticomedular y median funciones biológicas, como la regulación del volumen de líquido extracelular, el equilibrio electrolítico o la homeostasis ácido-base1.
Los avances en transcriptómica unicelular han permitido la caracterización en profundidad de tejidos complejos y han acelerado la comprensión de la expresión génica específica de segmentos y tipos celulares en la fisiología renal, el desarrollo y la enfermedad 2,3,4.
Sin embargo, los protocolos de disociación basados en enzimas que se utilizan con frecuencia para scRNA-seq muestran varios inconvenientes y restricciones. Dependiendo del protocolo, generan respuestas de estrés transcripcional y sesgo de disociación tisular hacia tipos de células corticales más fáciles de disociar 5,6. Aunque los protocolos que utilizan proteasas activas en frío para riñones embrionarios son capaces de mitigar las alteraciones transcripcionales relacionadas con el estrés, no logran superar el sesgo de disociación hacia las células corticales y podrían no ser fácilmente adaptables a diferentes tipos de tejidos renales enfermos7. Además, los enfoques unicelulares no son fácilmente compatibles con muestras de tejido congelado, lo que limita su aplicación principalmente a tejido fresco no archivado, lo que hace que la recolección de tejido sea un factor restrictivo6.
La secuenciación de ARN de un solo núcleo (snRNA-seq) puede eludir estas limitaciones 8,9. Aquí, presentamos un protocolo para el aislamiento de núcleos de una rebanada central de tejido renal de ratón adulto congelado (Figura 1)10. Nuestro protocolo es simple y proporciona un enfoque estandarizado para obtener bibliotecas de secuenciación de ARN con una representación equilibrada de diversos tipos de células renales para modelos experimentales que no implican cambios tisulares regionales fuertes. En este último caso, nuestro protocolo también se puede realizar con riñones enteros.
Protocolo
Todos los experimentos con animales se llevaron a cabo de acuerdo con la Ley de Bienestar Animal (TierSchG) y el Reglamento de Animales Experimentales de Bienestar Animal (TierSchVersV) y fueron autorizados por las autoridades locales y los Oficiales de Bienestar Animal de nuestra institución (MDC).
1. Preparación del tejido
- Prepare una placa de 6 pocillos que contenga 2 ml de 1x solución salina tamponada con fosfato (PBS) por pocillo para cada riñón que se obtendrá. Prepare una placa de 6 pocillos que contenga 2 ml de solución de estabilización de ARN por pocillo y riñón. Pre-enfríe ambas placas en hielo.
- Eutanasia a un ratón macho C57BL/6 de 3 a 6 meses de edad. Coloque el ratón en una bandeja de disección, fije las extremidades y esterilice el abdomen con etanol al 70%.
- Abra el abdomen hasta la caja torácica con fórceps y tijeras. Levante el intestino y otros órganos hacia un lado y retire los riñones cortando cuidadosamente el uréter, la arteria renal y la vena con una tijera. Lave el riñón en el PBS 1x helado previamente preparado y retire la fascia renal y cualquier grasa restante del riñón hasta que se elimine todo el tejido blanco (Figura 2A).
- Coloque el riñón en una placa de disección en frío y use un bisturí afilado o una cuchilla de afeitar para obtener una rebanada media de 1-2 mm. Asegúrese de que la pieza de tejido contenga todo el eje corticomedular (Figura 2B). Use tijeras y pinzas de microdisección para recortar cuidadosamente la corteza desde los lados de la pieza central. Dentro de la pieza de tejido disecado, los tres segmentos de la corteza, la médula externa y la médula interna deben ser claramente visibles (Figura 2C).
NOTA: La rebanada no debe exceder un grosor de 2 mm o un peso de 20 mg para garantizar cantidades tampón suficientes para una lisis tisular efectiva y para minimizar el ARN de fondo ambiental en las bibliotecas de ADNc. El ARN ambiental desperdicia la capacidad de secuencia, ya que no está asociado con núcleos individuales. - Transfiera la pieza renal a la solución de estabilización de ARN previamente preparada e incube durante 24 h a 4 °C para evitar la degradación del ARN. Después de 24 h, retirar la solución de estabilización de ARN y conservar el tejido a -80 °C hasta su uso posterior. Retire con cuidado el exceso de solución con papel de seda.
2. Aislamiento de núcleos
- Pasos de limpieza y preparación
- Limpie las mesas de trabajo y pipetas con etanol al 70% y solución de descontaminación de RNasa.
- Limpie un tubo de trituradora de tejido de 2 ml de fondo redondo y haga coincidir el mortero A y B con la solución de descontaminación de RNasa, seguido de etanol al 70% y agua libre de RNasa (1 juego de tubos y mortero por muestra). Deja que se seque completamente.
- Preenfriar la centrífuga a 4 °C.
- Etiquete y preenfríe tres tubos de recolección de 15 ml, un tubo de recolección de 1.5 ml, un tubo de recolección de 5 ml de clasificación celular activada por fluorescencia (FACS) y un tubo de molino seco para cada muestra en hielo.
- Preparación del tampón
- Calentar la solución madre del complejo ribonucleósido-vanadilo a 65 °C hasta que se reconstituya a una solución transparente verde-negra de acuerdo con las instrucciones del fabricante. 11
- Preparar 1x PBS que contenga 4% de albúmina sérica bovina (BSA) como se describe en la Tabla 1A. Además, prepare 1x PBS con 0.04% BSA (Tabla 1B). Filtrar ambas soluciones con un filtro de jeringa de membrana de acetato de celulosa (SFCA) sin surfactante de 0,2 μm y mantener en hielo hasta su uso posterior.
- Preparar el tampón de lisis de núcleos 1 (NLB1, Tabla 1C). Añadir 4 ml de tampón de lisis EZ para el tampón de lisis de núcleos 2 (NLB2, Tabla 1D) y 2 ml de BSA/PBS al 0,04 % para el tampón de suspensión de núcleos (NSB, Tabla 1E) a tubos de 15 ml. Agregue la solución inhibidora de RNasa a NLB2 y NSB directamente antes de usarla como se indica a continuación en el protocolo. Mantener en hielo hasta su uso posterior.
- Prepare el tampón de lisis EZ con sacarosa al 10% (Tampón de gradiente de sacarosa, Tabla 1F). Mezcle bien y filtre el tampón en un tubo fresco de 15 ml utilizando un filtro de jeringa de membrana SFCA de 0,2 μm. Mantener en hielo hasta su uso posterior.
- Homogeneización tisular y lisis celular
NOTA: Para minimizar la degradación del ARN, todos los pasos se llevan a cabo en hielo. El tubo del molinillo, la placa de Petri y todos los tampones deben enfriarse previamente. Todos los pasos de resuspensión se realizan pipeteando cuidadosamente la suspensión de los núcleos. No haga vortex la muestra para evitar las fuerzas de cizallamiento y el daño a los núcleos.- Tome la pieza de riñón congelada y transfiérala a una placa de Petri de poliestireno de 60 mm sobre hielo que contenga 1 ml de NLB1.
- Picar bien el tejido con una cuchilla de afeitar o un bisturí (Figura 3A).
- Corte la punta de una punta de pipeta de 1 ml y transfiera el tejido picado y el tampón al tubo del molinillo. Asegúrese de transferir todas las piezas de tejido. Lave la placa de Petri 5-10 veces con el tampón, si es necesario.
- Homogeneice la suspensión en hielo moviendo lentamente el mortero A, 25 veces hacia arriba y hacia abajo en el tubo del molinillo. Evite las burbujas de aire causadas por movimientos rápidos (Figura 3B).
- Pasar el homogeneizado a través de un filtro de 100 μm en un tubo de recolección de 15 ml preenfriado y lavar el filtro con otro 1 ml de NLB1.
- Lave el tubo del molinillo con tampón de lisis de núcleos EZ frío y deseche el tampón.
- Transfiera el homogeneizado de nuevo al tubo del molinillo y homogeneice la suspensión en hielo moviendo lentamente el mortero B, 15 veces hacia arriba y hacia abajo en el tubo del molinillo. Evite las burbujas de aire causadas por movimientos rápidos (Figura 3C).
- Transfiera el homogeneizado a un tubo de recolección preenfriado de 15 ml. Lave el tubo del molinillo con otros 2 ml de NLB1 y asegúrese de transferir todos los fragmentos de tejido al tubo de recolección. Incubar el homogeneizado (volumen total de 4 mL) durante 5 min en hielo para lisar las células.
- Purificación de núcleos
- Pasar el homogeneizado a través de un filtro de 40 μm a un tubo de recolección de 15 ml preenfriado. Girar el tubo de recogida durante 5 min a 500 x g a 4 °C en una centrífuga con un rotor de cuchara oscilante. Mientras tanto, agregue una solución de inhibidor de la RNasa a NLB2 (Tabla 1D).
- Retire el sobrenadante sin alterar la bolita. Resuspender cuidadosamente el pellet en 4 ml de NLB2.
- Coloque cuidadosamente la suspensión con un cojín de 1 ml de tampón de gradiente de sacarosa. Centrífuga a 500 x g durante 5 min a 4 °C en una centrífuga con rotor de cangilón oscilante. Mientras tanto, agregue una solución inhibidora de la RNasa a NSB (Tabla 1E).
- Después de la centrifugación, retire suavemente el tubo de recolección de la centrífuga y tenga cuidado de no molestar las dos capas cuando manipule el tubo de recolección. Los restos celulares son visibles entre las dos capas. Retire el sobrenadante con cuidado comenzando con los residuos. Retire el sobrenadante restante sin alterar el pellet del núcleo y vuelva a suspender cuidadosamente el pellet en 1 ml de NSB.
NOTA: El volumen de resuspensión depende de la cantidad de tejido utilizado para el aislamiento y del tamaño del pellet ganado después del último paso de centrifugación. Es posible que sea necesario adaptar el volumen al número esperado de núcleos. - Pasar el homogeneizado a través de un filtro de 20 μm en el tubo de recolección FACS de 5 ml preenfriado.
3. Clasificación de núcleos
- Añadir 20 μL de 4′,6-diamidino-2-fenilindol (DAPI) por ml de NSB a una concentración final de 2 μM al homogeneizado en el tubo de recogida del FACS y mezclar cuidadosamente. Incubar durante 5 minutos en hielo.
- Preparar el tubo colector de 1,5 ml preenfriado con 20 μL de BSA al 4% /1x PBS y añadir 0,5 μL de solución inhibidora de la RNasa a una concentración final de 1 U/μL. Proceder inmediatamente a la clasificación.
- Ordene los núcleos utilizando un clasificador de células.
- Mezclar brevemente la suspensión de núcleos antes de insertar el tubo de recogida FACS en el clasificador.
- Establezca una primera puerta P1 basada en la dispersión directa (FSC) y la dispersión lateral (SSC) para excluir escombros y agregados (Figura 4A).
- Para excluir núcleos vacíos o dañados y múltiplos, establezca una puerta posterior basada en el área DAPI frente a la altura DAPI (DAPI-A vs DAPI-H) (Figura 4B).
- Clasificar núcleos individuales en el tubo colector de 1,5 ml que contiene BSA al 4% /1x PBS con 1 solución U/μL de inhibidor de la RNasa preparada en 3.2.
4. Control de calidad
- Mida la concentración final de núcleos bajo un microscopio de fluorescencia o en una cámara de conteo automatizada en al menos dos recuentos independientes y evalúe la calidad de la suspensión (Figura 5).
NOTA: Las concentraciones óptimas están entre 700 - 1,200 núcleos / μL. Las concentraciones celulares más bajas, como 700 núcleos / μL, pueden ser preferibles ya que las bibliotecas de ADNc resultantes contenían menos ARN de fondo ambiental (transcripciones no asociadas con núcleos individuales). - Calcular el volumen requerido de suspensión de núcleos para la recuperación deseada de núcleos individuales secuenciados. Para evitar la agregación de núcleos y la degradación del ARN, proceda inmediatamente a la preparación de la biblioteca.
Resultados
Para determinar el rendimiento de nuestro protocolo, utilizamos el 10x Genomics Chromium Single Cell 3' Gene Expression Kit v3.1 para la preparación de la biblioteca y analizamos los datos de snRNA-seq con el paquete Seurat12,13.
La Figura 6 muestra los resultados de una biblioteca representativa de snRNA-seq. Para evaluar la calidad de nuestros núcleos, trazamos el número de genes contra el número de transcripciones (definidas por identificadores moleculares únicos (UMI)) coloreadas por la fracción de lecturas mitocondriales (Figura 6A). Los núcleos de buena calidad generalmente muestran un mayor número de lecturas, correlacionando UMI y números de genes, y bajas fracciones de lectura mitocondrial.
Para el análisis posterior, se excluyeron los núcleos con menos de 500 o más de 4000 genes contados, o más del 5% del ARN mitocondrial (n = 828). Solo se incluyeron genes expresados en un mínimo de tres núcleos. Detectamos alrededor de 20.000 genes en total en los 6.000 núcleos restantes con 1.600 genes medianos y 2.800 UMI medianas por núcleo (Figura 6B).
La agrupación se basó en genes altamente variables. Se identificaron un total de 18 conglomerados. Las identidades celulares se anotaron en función de genes marcadores conocidos (no se muestran). Los subgrupos de un tipo de célula se resumieron en un grupo que resultó en un total de 11 tipos de células distintas: podocitos, túbulo proximal (PT), miembro delgado (tL), miembro ascendente grueso (TAL), túbulo contorneado distal (DCT), túbulo de conexión (CNT), conducto colector principal y células intercaladas (CD-PC, A-IC, B-IC), epitelio medular profundo de la pelvis (DMEP) y endotelio. Los patrones de expresión génica de marcadores enriquecidos con conglomerados se visualizaron en un diagrama de puntos (Figura 6C) y grupos de tipo celular en una gráfica de incrustación de vecinos estocásticos distribuidos en t (t-SNE) (Figura 6D).
Para evaluar las distribuciones del tipo celular en nuestra muestra, se calculó el porcentaje de cada tipo de célula (Figura 6E) y se utilizó para determinar la relación de TP a TAL. El TP se encuentra principalmente en la corteza renal y con frecuencia está sobrerrepresentado en los conjuntos de datos de células individuales del riñón, ya que las células del PT son fáciles de disociar y muy abundantes en muestras de riñón completo. El TAL, por otro lado, se extiende a través de toda la médula externa14. Por lo tanto, la proporción de fracciones PT y TAL representa una buena medida para el enriquecimiento de los tipos de células medulares en un conjunto de datos de células individuales de riñón. En general, la relación PT/TAL en conjuntos de datos de riñón entero unicelular varió de 8 (datos no publicados de tejido renal completo tratado con proteasa fría) a 45 para tejido disociado enzimáticamente10,14,15. En el conjunto de datos snRNA-seq presentado aquí pudimos alcanzar una relación PT / TAL de 2. Este resultado ilustra que la eliminación del exceso de corteza durante la disección de tejidos combinada con snRNA-seq da como resultado una representación del tipo de célula renal sorprendentemente mejorada.
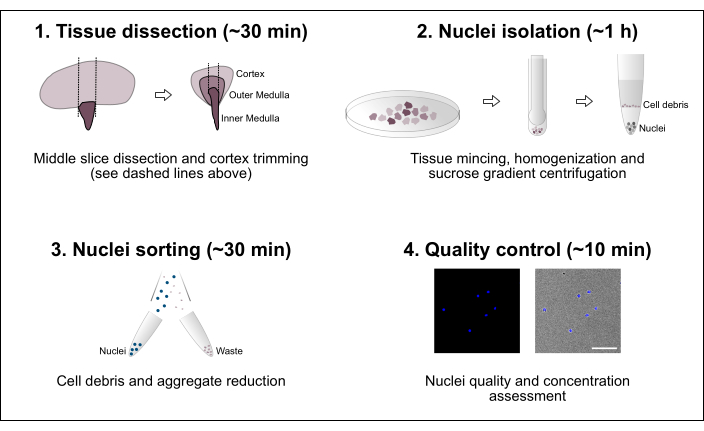
Figura 1: Descripción general esquemática del flujo de trabajo. El protocolo consta de cuatro pasos principales que incluyen la disección de tejidos seguida de aislamiento de núcleos, clasificación de núcleos y una evaluación final de pureza y concentración. Barra de escala = 100 μm. Haga clic aquí para ver una versión más grande de esta figura.
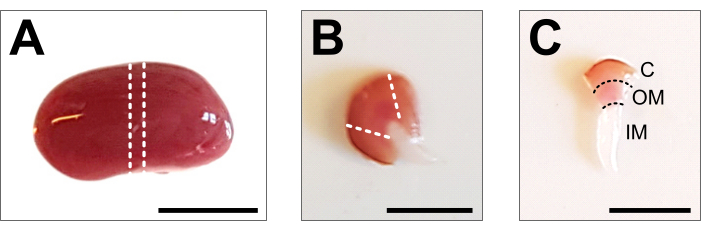
Figura 2: Disección renal y preparación tisular . (A) Imagen representativa de todo el riñón disecado. Las líneas punteadas indican los cortes necesarios para obtener un corte medio de 1-2 mm con una representación de todos los tipos de células renales. (B) Imagen representativa del corte medio obtenido. Las líneas punteadas indican los cortes para el recorte de la corteza desde el lado. (C) Imagen representativa de la pieza central del riñón con corteza recortada. La corteza (C), la médula externa (OM) y la médula interna (IM) son claramente visibles. Barra de escala = 500 μm. Haga clic aquí para ver una versión más grande de esta figura.

Figura 3: Homogeneización tisular y purificación de núcleos . (A) Imagen representativa que muestra tejido renal suficientemente picado. Barra de escala = 500 μm. (B) Homogeneizar después de la primera etapa de homogeneización (25 golpes con mortero A, tubo de molinillo de 2 ml). (C) Homogeneizar después de la segunda etapa de homogeneización (15 golpes con mortero B, tubo de molino de 2 ml). Haga clic aquí para ver una versión más grande de esta figura.
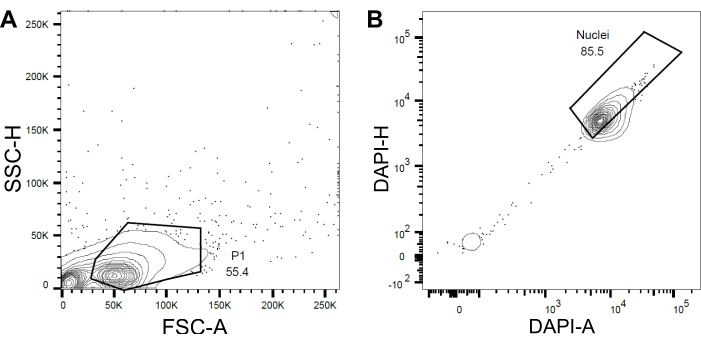
Figura 4: Estrategia de compuerta para la clasificación de núcleos. (A ) Se estableció una primera puerta P1 basada en la dispersión hacia adelante (FSC) frente a la dispersión lateral (SSC) para excluir escombros y agregados. (B ) Una puerta posterior basada en DAPI-Area (DAPI-A) vs DAPI-Height (DAPI-H) excluyó núcleos vacíos o dañados y múltiplos. Haga clic aquí para ver una versión más grande de esta figura.
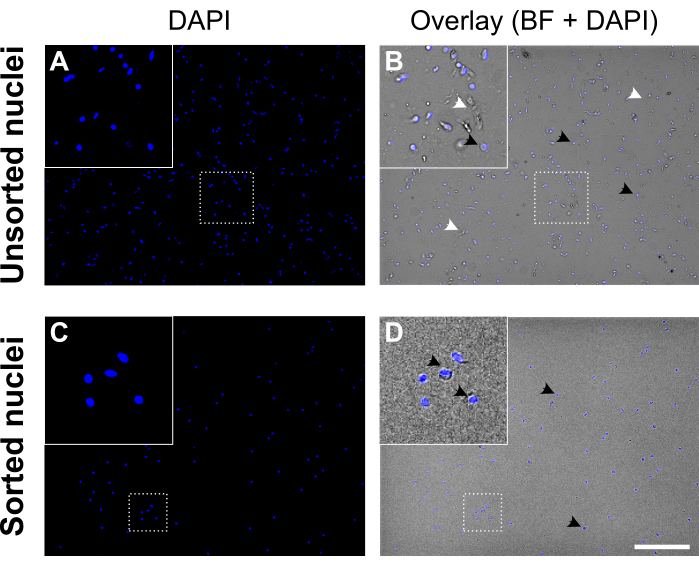
Figura 5: Suspensión de núcleos antes y después de la clasificación de los núcleos. (A, C) Núcleos teñidos con DAPI (azul). (B, D) Superposición de DAPI y canal brightfield (BF). Antes de clasificar (panel superior), la suspensión de núcleos contiene restos celulares y agregados (etiquetados con puntas de flecha blancas). Después de la clasificación (panel inferior) la suspensión de núcleos aparece mucho más limpia. Ejemplos de núcleos teñidos con DAPI están etiquetados con puntas de flecha negras. Los núcleos de buena calidad aparecen redondos y lisos con una membrana intacta y están bien separados, mientras que los núcleos de mala calidad aparecen arrugados y muestran pérdida de la membrana nuclear. Barra de escala = 250 μm. Haga clic aquí para ver una versión más grande de esta figura.
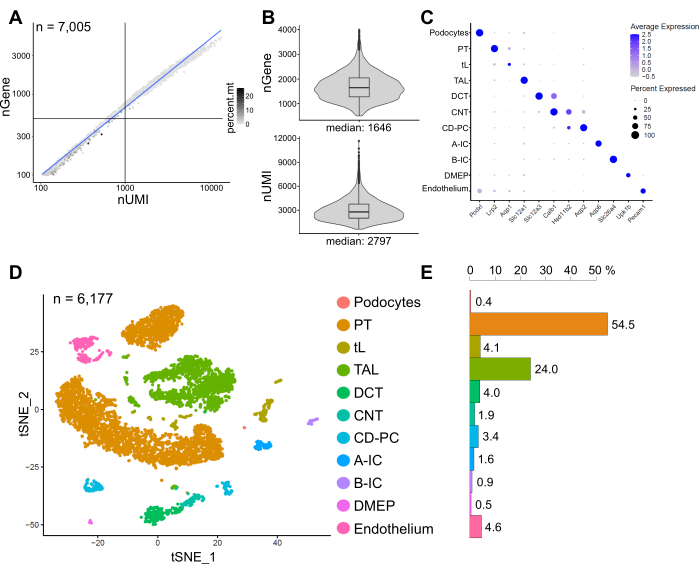
Figura 6: Control de calidad y análisis de un conjunto de datos representativo de snRNA-seq. (A) Número de genes (nGene) trazados contra el número de identificadores moleculares únicos (nUMI) coloreados por la fracción de lecturas mitocondriales (percent.mt). Los núcleos de baja calidad corresponden al cuadrante inferior izquierdo de la gráfica (n = 828) y fueron excluidos del análisis posterior. (B) Distribución y mediana de nGene y nUMI detectados por núcleo en el conjunto de datos snRNA-seq, que representan 6.177 núcleos (> 500 genes). Las bibliotecas se secuenciaron a una profundidad media de ~ 8.200 lecturas mapeadas por núcleo. (C) Diagrama de puntos que muestra los patrones de expresión génica de marcadores enriquecidos con racimo (eje x) para tipos de células individuales (eje y). El tamaño del punto corresponde a la proporción de células que expresan el gen indicado. El color corresponde a la expresión promedio. (D) Gráfica de incrustación de vecinos estocásticos distribuidos en T (t-SNE) de tipos de células identificadas. (E) Distribución del tipo de célula en el conjunto de datos snRNA-seq. TP: túbulo proximal; tL: miembro delgado; TAL: miembro ascendente grueso, DCT, túbulo contorneado distal; CNT: túbulo de conexión; CD-PC: células principales del conducto colector; A-IC: células intercaladas tipo A; B-IC: células intercaladas tipo B; DMEP: epitelio medular profundo de la pelvis. Haga clic aquí para ver una versión más grande de esta figura.
| Reactivo | Concentración final | Volumen (ml) | ||
| A) 4 % BSA/PBS | ||||
| Solución salina tamponada con fosfato (PBS) con 10% de albúmina bovina | 4% | 2 | ||
| PBS (solución salina tamponada con fosfato) 1X sin calcio ni magnesio | - | 3 | ||
| (B) 0,04 % BSA/PBS | ||||
| 4 % BSA / PBS | 0.04 % | 0.5 | ||
| PBS (solución salina tamponada con fosfato) 1X sin calcio ni magnesio | - | 49.5 | ||
| c) Tampón de lisis de núcleos 1 (NLB1) | ||||
| Tampón de lisis EZ nuclear | - | 4 | ||
| Inhibidor de la RNasa RiboLock (40 U/μL) | 1 U/μL | 0.1 | ||
| Complejo ribonucleósido-vanadilo (200 mM) | 10 mM | 0.2 | ||
| d) Tampón de lisis de núcleos 2 (NLB2) | ||||
| Tampón de lisis EZ nuclear | - | 4 | ||
| Inhibidor de la RNasa RiboLock (40 U/μL) | 1 U/μL | 0.1 | ||
| e) Tampón de suspensión de núcleos (NSB) | ||||
| 0,04 % BSA / PBS | - | 2 | ||
| Inhibidor de la RNasa RiboLock (40 U/μL) | 1 U/μL | 0.05 | ||
| f) Tampón de gradiente de sacarosa (sacarosa al 10 %) | ||||
| Peso 1 g de sacarosa | ||||
| Disolver en 6 ml de tampón de lisis EZ nuclear | ||||
| Llene hasta 10 ml con tampón de lisis EZ nuclear | ||||
| Filtrar a través de un filtro de jeringa de 0,2 μm en un tubo nuevo | ||||
Tabla 1: Recetas de solución: (A) Preparación de BSA/1x PBS al 4%. Filtrar con un filtro de jeringa de membrana SFCA de 0,2 μm y mantener en hielo hasta su uso. (B) Preparación de 0.04% BSA/1 x PBS. Filtrar con un filtro de jeringa de membrana SFCA de 0,2 μm y mantener en hielo hasta su uso. c) Preparación del tampón de lisis de núcleos 1 (NLB1). Los volúmenes indicados se proporcionan por muestra. Mantener en hielo hasta su uso. d) Preparación del tampón de lisis 2 de los núcleos (NLB2). Los volúmenes indicados se proporcionan por muestra. Agregue RiboLock RNase Inhibitor a NLB2 directamente antes de usarlo como se indica en el protocolo. Mantener en hielo hasta su uso. e) Preparación del tampón de suspensión de núcleos (NSB). Los volúmenes indicados se proporcionan por muestra. Agregue RiboLock RNase Inhibitor a NSB directamente antes de usarlo como se indica en el protocolo. Mantener en hielo hasta su uso. f) Preparación de tampón de gradiente de sacarosa. Filtrar con un filtro de jeringa de membrana SFCA de 0,2 μm y mantener en hielo hasta su uso.
Discusión
La transcriptómica unicelular avanza en la comprensión de la expresión génica específica del tipo de célula en la fisiología renal y la enfermedad. Aquí, proporcionamos un método simple y reproducible para aislar núcleos únicos de alta calidad de tejido renal de ratón congelado para snRNA-seq de una manera estandarizada.
Para snRNA-seq, es fundamental utilizar núcleos de alta calidad como entrada para la generación de bibliotecas y evitar la degradación del ARN durante el procesamiento del tejido. Por lo tanto, la incubación de piezas de tejido en solución de estabilización de ARN inmediatamente después de la disección es esencial para proteger y estabilizar el ARN celular y permite almacenar muestras a - 80 °C indefinidamente. Al aplicar este protocolo a tejido congelado sin tratamiento de solución de estabilización de ARN, como material de archivo, se requiere una ejecución de prueba y se debe evaluar la calidad del ARN, ya que observamos una pérdida significativa de integridad de ARN en tejido congelado a presión sin incubación previa en solución de estabilización de ARN.
En general, el manejo adecuado de la muestra es crucial para maximizar la recuperación de núcleos únicos intactos. Todos los pasos de resuspensión deben llevarse a cabo pipeteando cuidadosamente para evitar el esfuerzo cortante y el daño físico. Los tampones para la resuspensión final de núcleos y la clasificación de núcleos deben contener BSA para evitar la pérdida y agregación de núcleos.
Los volúmenes tampón en este protocolo están optimizados para muestras de tejido muy pequeñas (~ 15 mg). Es fundamental garantizar una lisis celular completa y un lavado suficiente para generar suspensiones de alta calidad. Los bloques de tejido más grandes o las muestras de riñón completo darán como resultado concentraciones excesivas de núcleos que conducen a la aglutinación y agregación, alta abundancia de ARN ambiental y mala calidad general de la suspensión. Si se procesan muestras más grandes u otros tejidos, se recomienda encarecidamente realizar pruebas para determinar los volúmenes de tampón óptimos para niveles mínimos de ARN ambiental. La calidad y las concentraciones de núcleos y ARN deben examinarse cuidadosamente, ya que la sobrecarga da como resultado un rendimiento general deficiente.
Además, grandes cantidades de restos celulares, causando altos niveles de ARN ambiental no asociados con núcleos individuales, influyen negativamente en los resultados de la secuenciación. La clarificación de la suspensión de los núcleos por centrifugación a través de un cojín de sacarosa mitiga este problema hasta cierto punto, pero también puede conducir a un sesgo en la representación del tipo celular al contraseleccionar contra núcleos densos y pequeños presentes, por ejemplo, en las células inmunes16. Si esto es motivo de preocupación, se debe omitir el gradiente de sacarosa. Por el contrario, encontramos que la citometría de flujo basada en la tinción DAPI fue crítica para reducir la cantidad de desechos celulares con el fin de producir una suspensión de núcleos únicos de alta calidad.
El aislamiento de núcleos individuales tiene ventajas considerables en comparación con los enfoques unicelulares8. Es compatible con el tejido correctamente congelado, haciendo más flexible la recolección de tejido, y evita la necesidad de disociación tisular basada en enzimas, que puede introducir respuestas de estrés transcripcional 6,17. Además, supera el sesgo de disociación que favorece la selección de tipos celulares fácilmente disociables de la corteza renal, lo que puede conducir a una subrepresentación de los tipos de células medulares en algunos enfoques basados en enzimas 5,6,10.
El uso de una pieza renal central en lugar de tejido renal completo ahorra aún más recursos y corrige la sobrerrepresentación de abundantes tipos de células como se describió anteriormente10. Sin embargo, dependiendo del modelo de ratón o fenotipo investigado, puede ser beneficioso utilizar muestras de riñón enteras en lugar de una sola rebanada media. Las muestras de riñón entero pueden ser más representativas de las proporciones celulares verdaderas, o los cambios que ocurren en todo el riñón, mientras que un corte medio recortado resultó ventajoso para los fenotipos medulares o cuando el material de la muestra era limitado. Esta decisión, por lo tanto, es muy específica para el usuario y debe considerarse cuidadosamente.
Divulgaciones
Los autores no tienen nada que revelar.
Agradecimientos
Agradecemos a la Plataforma de Genómica Científica del Centro Max Delbrück de Medicina Molecular de la Asociación Helmholtz, Berlín, por su apoyo técnico.
JL y KMSO fueron apoyados por el Grupo de Formación en Investigación GRK 2318 de la Fundación Alemana de Investigación (DFG) y por la Unidad de Investigación FOR 2841. KMSO fue apoyado por Collaborative Research Grant 1365. AB fue apoyado por la financiación del Premio Gottfried Wilhelm Leibniz de la DFG otorgado a NR.
Materiales
| Name | Company | Catalog Number | Comments |
| Cell sorter | - | - | For fluorescence-activated cell sorting (FACS); e.g. BD FACSAria Cell Sorter. |
| Centrifuge 5810 R | Eppendorf | 5811000015 | |
| Countess Cell Counting Chamber Slides | Invitrogen | C10228 | |
| Countess II FL Automated Cell Counter | Invitrogen | AMQAF1000 | Needs to contain DAPI light cube to count nuclei. Alternatively, nuclei can be counted manually under fluorescence microscope. |
| 4′,6-Diamidino-2-phenyl-indol-dihydrochlorid (DAPI) | Biotrend | 40043/b | Stock solution prepared with a concentration of 100 µM. Used for nuclei staining in a final concentration of 2 µM. |
| D(+)-Sucrose ≥99.9%, ultrapure DNAse-, RNAse-free | VWR | 0335-500G | |
| DNA LoBind Microcentrifuge Tubes (1.5 mL) | Eppendorf | 22431021 | |
| Ethanol, 70 % | - | - | |
| FACS tubes | pluriSelect | 43-10100-46 | |
| KIMBLE Dounce tissue grinder set 2 mL complete | Sigma-Aldrich | D8938-1SET | |
| Minisart Syringe Filters 0.2 µm | Sartorius | 16534-GUK | |
| Nuclease-free Water | Invitrogen | AM9937 | |
| Nuclei EZ Prep Nuclei Isolation Kit | Sigma-Aldrich | NUC-101 | Nuclei EZ Lysis Buffer (Product No. N3408) needed for buffer preparation. |
| PBS (Phosphate-Buffered Saline) 1X without calcium or magnesium | Corning | 21-040-CV | |
| Petri dishes, polystyrene 60 mm | Sigma-Aldrich | P5481 | |
| Phosphate-Buffered Saline (PBS) with 10% Bovine Albumin | Sigma-Aldrich | SRE0036 | |
| pluriStrainer Mini 100 µm | pluriSelect | 43-10100-46 | |
| pluriStrainer Mini 20 µm | pluriSelect | 43-10020-40 | |
| pluriStrainer Mini 40 µm | pluriSelect | 43-10040-40 | |
| Polystyrene Centrifuge Tube (15 mL) | Falcon | 352099 | |
| Razor blades | - | - | |
| RiboLock RNase Inhibitor (40 U/µL) | Thermo Fisher | EO0384 | |
| Ribonucleoside-vanadyl complex | New England Biolabs | S1402S | Follow manufacturer's instructions (https://international.neb.com/products/s1402-ribonucleoside-vanadyl-complex#Product%20Information). Upon use the 200 mM stock solution is reconstituted to a green-black clear solution by incubating at 65 °C. |
| RNAlater Stabilization Solution | Invitrogen | AM7020 | |
| RNase AWAY | Fisher Scientific | 11952385 |
Referencias
- Thomas, R. S. Kidney modeling and systems physiology. Wiley Interdisciplinary Reviews: Systems Biology and Medicine. 1 (2), 172-190 (2009).
- Potter, S. S. Single-cell RNA sequencing for the study of development, physiology and disease. Nature Reviews Nephrology. 14 (8), 479-492 (2018).
- Park, J., Liu, C. L., Kim, J., Susztak, K. Understanding the kidney one cell at a time. Kidney International. 96 (4), 862-870 (2019).
- Clark, A. R., Greka, A. The power of one: advances in single-cell genomics in the kidney. Nature Reviews Nephrology. 16 (2), 73-74 (2020).
- Lake, B. B., et al. A single-nucleus RNA-sequencing pipeline to decipher the molecular anatomy and pathophysiology of human kidneys. Nature Communications. 10 (1), 2832 (2019).
- Wu, H., Kirita, Y., Donnelly, E. L., Humphreys, B. D. Advantages of single-nucleus over single-cell RNA sequencing of adult kidney: Rare cell types and novel cell states revealed in fibrosis. Journal of the American Society of Nephrology. 30 (1), 23-32 (2019).
- Adam, M., Potter, A. S., Potter, S. S. Psychrophilic proteases dramatically reduce single-cell RNA-seq artifacts: a molecular atlas of kidney development. Development. 144 (19), 3625-3632 (2017).
- Habib, N., et al. Massively parallel single-nucleus RNA-seq with DroNc-seq. Nature Methods. 14 (10), 955-958 (2017).
- Muto, Y., et al. Single cell transcriptional and chromatin accessibility profiling redefine cellular heterogeneity in the adult human kidney. Nature Communications. 12 (1), 2190 (2021).
- Hinze, C., et al. Kidney single-cell transcriptomes predict spatial corticomedullary gene expression and tissue osmolality gradients. Journal of the American Society of Nephrology. 32 (2), 291 (2021).
- Berger, S. L. Isolation of cytoplasmic RNA: ribonucleoside-vanadyl complexes. Methods in Enzymology. 152, 227-234 (1987).
- Butler, A., Hoffman, P., Smibert, P., Papalexi, E., Satija, R. Integrating single-cell transcriptomic data across different conditions, technologies, and species. Nature Biotechnology. 36 (5), 411-420 (2018).
- Stuart, T., et al. Comprehensive Integration of single-cell data. Cell. 177 (7), 1888-1902 (2019).
- Park, J., et al. Single-cell transcriptomics of the mouse kidney reveals potential cellular targets of kidney disease. Science. 360 (6390), 758-763 (2018).
- Kirita, Y., Wu, H., Uchimura, K., Wilson, P. C., Humphreys, B. D. Cell profiling of mouse acute kidney injury reveals conserved cellular responses to injury. Proceedings of the National Academy of Sciences of the United States of America. 117 (27), 15874-15883 (2020).
- Schneeberger, S., et al. The neuroinflammatory interleukin-12 signaling pathway drives Alzheimer's disease-like pathology by perturbing oligodendrocyte survival and neuronal homeostasis. bioRxiv. , 441313 (2021).
- Nguyen, Q. H., Pervolarakis, N., Nee, K., Kessenbrock, K. experimental considerations for single-cell RNA sequencing approaches. Frontiers in Cell and Developmental Biology. 6, 108 (2018).
Reimpresiones y Permisos
Solicitar permiso para reutilizar el texto o las figuras de este JoVE artículos
Solicitar permisoThis article has been published
Video Coming Soon
ACERCA DE JoVE
Copyright © 2025 MyJoVE Corporation. Todos los derechos reservados