Zum Anzeigen dieser Inhalte ist ein JoVE-Abonnement erforderlich. Melden Sie sich an oder starten Sie Ihre kostenlose Testversion.
Method Article
Beurteilung der Knochenbruchheilung mittels Mikro-Computertomographie
* Diese Autoren haben gleichermaßen beigetragen
In diesem Artikel
Zusammenfassung
Die Mikro-Computertomographie (μCT) ist ein zerstörungsfreies bildgebendes Verfahren, das in präklinischen Studien zur Beurteilung der Knochenstruktur beiträgt, jedoch gibt es keinen Konsens über μCT-Verfahren zur Analyse der knochenheilenden Kallus. Diese Studie bietet ein schrittweises μCT-Protokoll, das die Überwachung der Frakturheilung ermöglicht.
Zusammenfassung
Die Mikro-Computertomographie (μCT) ist die gebräuchlichste bildgebende Modalität, um die dreidimensionale (3D) Morphologie von Knochen und neu gebildetem Knochen während der Frakturheilung in translationalen wissenschaftlichen Untersuchungen zu charakterisieren. Untersuchungen zur Heilung von Röhrenknochenbrüchen bei Nagetieren beinhalten in der Regel eine sekundäre Heilung und die Bildung eines mineralisierten Kallus. Die Form des gebildeten Kallus und die Dichte des neu gebildeten Knochens können je nach Zeitpunkt und Behandlung erheblich variieren. Während Standardmethoden zur Quantifizierung von Parametern intakter kortikaler und trabekulärer Knochen weit verbreitet und in kommerziell erhältliche Software eingebettet sind, gibt es keinen Konsens über Verfahren zur Analyse der heilenden Kallus. Das Ziel dieser Arbeit ist es, ein standardisiertes Protokoll zu beschreiben, das den Knochenvolumenanteil und die Kallusmineraldichte im heilenden Kallus quantifiziert. Das Protokoll beschreibt verschiedene Parameter, die bei der Bildgebung und Analyse berücksichtigt werden sollten, einschließlich der Probenausrichtung während der Bildgebung, der Größe des interessierenden Volumens und der Anzahl der Schichten, die konturiert werden, um die Hornhaut zu definieren.
Einleitung
Die Mikro-Computertomographie (μCT)-Bildgebung ist in der präklinischen Knochenforschung weit verbreitet und liefert nicht-invasive, hochauflösende Bilder zur Beurteilung der Mikrostruktur von Knochen 1,2,3,4,5. Bei der μCT handelt es sich um eine große Anzahl von Röntgenbildern, die von einer rotierenden Probe oder unter Verwendung einer rotierenden Röntgenquelle und eines Detektors aufgenommen werden. Algorithmen werden verwendet, um volumetrische 3D-Daten in Form eines Stapels von Bildschichten zu rekonstruieren. Die klinische CT ist der Goldstandard für die 3D-Bildgebung menschlicher Knochen, und die μCT ist eine häufig verwendete Technik zur Bewertung der Knochenheilungseffizienz bei Versuchstieren 1,2,3,4,6,7. Mineralisierter Knochen hat einen ausgezeichneten Röntgenkontrast, während Weichteile einen relativ schlechten Kontrast aufweisen, es sei denn, es wird ein Kontrastmittel verwendet. Bei der Beurteilung der Frakturheilung erzeugt die μCT Bilder, die detaillierte Informationen über die 3D-Struktur und Dichte des mineralisierten Kalluses liefern. In-vivo-μCT-Scans können auch zur longitudinalen und zeitlichen Beurteilung der Frakturheilung verwendet werden.
Die Quantifizierung von intaktem kortikalem und trabekulärem Knochen mittels μCT ist im Allgemeinen gut etabliert und standardisiert8. Obwohl präklinische Studien eine Vielzahl von Quantifizierungsmethoden verwenden, um die Frakturheilung zu analysieren 9,10,11, wurde ein detailliertes Protokoll der μCT-Bildanalyse zur Kallusquantifizierung noch nicht veröffentlicht. Daher ist es das Ziel dieser Studie, ein detailliertes Schritt-für-Schritt-Protokoll für die μCT-Bildgebung und -Analyse von Knochenheilungskallus bereitzustellen.
Protokoll
Das folgende Protokoll wurde entwickelt, um den langknochenheilenden Kallus, der von euthanasierten Mäusen gewonnen wurde, zu charakterisieren. Die meisten Schritte können jedoch auf Ratten angewendet und auch für In-vivo-Scans von Knochenbrüchen verwendet werden. Das Protokoll beschreibt ein bestimmtes μCT-System und eine bestimmte Bildverarbeitungs-, Analyse- und Visualisierungssoftware (siehe Materialtabelle), die Methodik ist jedoch allgemein auf andere Scanner und Software anwendbar. Das Protokoll wurde vom Institutional Animal Care and Use Committee des Pennsylvania State University College of Medicine genehmigt. Bei den in dieser Studie verwendeten Mäusen handelte es sich um 16 Wochen alte, männliche C57BL/6J-Mäuse (Durchschnittsgewicht 31,45 ± 3,2 g).
1. Gewebeentnahme und -konservierung
HINWEIS: Verwenden Sie ein geeignetes Frakturmodell für Mäuse. Für diese Studie wurde das Modell der offenen Tibiafraktur in der Mitte der Diaphysäre gemäß dem in12,13 beschriebenen Standardprotokoll verwendet.
- Am Ende des Frakturmodellexperiments wird die Maus euthanasiert, indem eine intraperitoneale Injektion von Ketamin oder Xylazin (500 mg/kg bzw. 50 mg/kg) verabreicht wird.
- Entnehmen Sie den gebrochenen Knochen mit einer Schere vom mittleren Oberschenkelknochen bis zum Tibiotalargelenk, ohne die Frakturstelle zu stören. Entfernen Sie die Muskeln, die den Knochen umgeben, und lassen Sie nur das Weichgewebe übrig, das in direktem Kontakt mit dem Knochen steht, um die Bruchstelle während der nachfolgenden Verarbeitungsschritte zu stützen. Entfernen Sie den Markstift mit einer geraden hämostatischen Mikromückenzange.
- Konservieren Sie die Proben in Formalin bei 4 °C oder in Kochsalzlösung bei -20 °C. Die Wahl des Konservierungsträgers hängt von den beabsichtigten Anwendungen ab, die der μCT nachgelagert sind. In dieser Studie wurden die Proben in Kochsalzlösung bei -20 °C konserviert.
2. μCT-Abtastung
- Probenvorbereitung
- Für das gleichzeitige Scannen mehrerer Proben legen Sie bis zu sechs Proben in eine speziell entwickelte, 3D-gedruckte Scanvorrichtung (Abbildung 1 A, B) oder ähnliches. Gleichzeitiges Scannen reduziert Scanzeit und -kosten. Die in dieser Studie verwendete kundenspezifische Vorrichtung enthält sechs Schlitze zur Aufnahme der langen Knochenproben und ein Mittelloch für ein Hydroxylapatit (HA)-Phantom (Abbildung 1A,B; Materialtabelle).
HINWEIS: Das HA-Phantom dient als Standard in Schritt 4.2 (siehe unten), um μCT-Einheiten (typischerweise Hounsfield) in HA-Dichte (mgHA/ccm) umzurechnen. - Platzieren Sie die vorbereitete Vorrichtung in einer Spritze oder einem konischen Röhrchen, das dem Durchmesser des Sichtfeldes entspricht (FOV; Abbildung 1C). In dieser Studie wurde eine 20-mm-Spritze verwendet, um das 21,5-mm-Sichtfeld anzupassen.
- Um zu verhindern, dass die Proben während des Scanvorgangs austrocknen, füllen Sie die Spritze oder das kanonische Röhrchen mit dem in Schritt 1.3 verwendeten Konservierungsmittel (in dieser Studie wurde Kochsalzlösung verwendet).
- Für das gleichzeitige Scannen mehrerer Proben legen Sie bis zu sechs Proben in eine speziell entwickelte, 3D-gedruckte Scanvorrichtung (Abbildung 1 A, B) oder ähnliches. Gleichzeitiges Scannen reduziert Scanzeit und -kosten. Die in dieser Studie verwendete kundenspezifische Vorrichtung enthält sechs Schlitze zur Aufnahme der langen Knochenproben und ein Mittelloch für ein Hydroxylapatit (HA)-Phantom (Abbildung 1A,B; Materialtabelle).
- Abtastung
- Vergewissern Sie sich vor dem Scannen, dass das μCT-Gerät wie folgt kalibriert ist: Platzieren Sie ein HA-Phantom auf der Mittellinie des μCT-Sichtfelds, scannen Sie das Phantom und messen Sie die Dichte von HA. Stellen Sie sicher, dass die gemessene Dichte mit der vom Hersteller angegebenen Dichte übereinstimmt.
- Richten Sie die Mittellinie der Probenvorrichtung an der ungefähren Mittellinie des μCT-Sichtfelds aus. Dadurch wird sichergestellt, dass sich die Proben innerhalb des Sichtfelds befinden und ihre Längsachsen ungefähr mit der axialen Richtung der resultierenden Bilder übereinstimmen.
HINWEIS: Diese standardisierte Ausrichtung kann später dazu beitragen, das Analyseverfahren weniger anfällig für Schwankungen zu machen, z. B. in der Menge des Gewebes, die innerhalb des interessierenden Volumens berücksichtigt wird. - Stellen Sie die Scanparameter des μCT-Systems ein (Materialtabelle). Die in dieser Studie verwendeten Parameter sind 10,5 μm (isotrope Voxelgröße), 55 kVp (Energie/Intensität), 145 μA (Strom) und 300 ms (Integrationszeit). Bestimmen Sie die Voxelgröße anhand der ungefähren Dicke der Trabekel der Maus (20-60 μm)8. Untersuchen Sie den Scan visuell in verschiedenen Ansichten, um sicherzustellen, dass er das gesamte Volumen aller Hornhautproben abdeckt.

Abbildung 1: Aufbau der kundenspezifischen Abtastvorrichtung. (A) Bilder der Abtastvorrichtung (oben) mit den sechs Probenschlitzen und des HA-Phantoms (unten). (B) Bilder, die die Langknochenprobe (oben) und das HA-Phantom (unten) zeigen, die in den dafür vorgesehenen Schlitzen platziert sind. (C) Bilder, die die Scanvorrichtung in einer 20-mm-Spritze zeigen. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.
3. Bildsegmentierung
HINWEIS: RAW-Bilder werden automatisch in Bildsequenzdaten rekonstruiert.
- Bildkonvertierung: Konvertieren Sie die rekonstruierten Bildsequenzdaten mit Hilfe einer Bildverarbeitungssoftware in DICOM-Bildsequenzen (siehe Materialtabelle). Importieren Sie DICOM-Bildsequenzen zur Bildverarbeitung, Analyse und Visualisierung in die Software (siehe Materialtabelle) (Abbildung 2A).
- Bildzuschneiden: Schneiden Sie jedes Bild stapelweise zu und stellen Sie sicher, dass das gesamte Sample im zugeschnittenen Volumen enthalten ist (Abbildung 2B). Speichern Sie das zugeschnittene Bild wie folgt: Klicken Sie oben links auf dem Bildschirm auf die Registerkarte Datei , wählen Sie Projekt speichern und wählen Sie dann Projektgröße minimieren aus den Optionen, die auf dem Bildschirm angezeigt werden. Die Datei wird im kommerziellen Softwareformat gespeichert.
- Bildrauschunterdrückung: Verwenden Sie eine Filtermethode, um den Rauschpegel zu reduzieren und Unschärfen wie folgt zu vermeiden.
- Klicken Sie auf die Registerkarte Datei und wählen Sie das Bild aus, das mit Open Data verarbeitet werden soll. Das geöffnete Bild wird im Projektansichtsfenster in der oberen linken Ecke des Bildschirms angezeigt.
- Klicken Sie mit der rechten Maustaste, um Bildverarbeitung und dann Filter-Sandbox auszuwählen. Klicken Sie auf Erstellen.
- Gehen Sie im Eigenschaftenfenster (in der linken unteren Ecke des Bildschirms) wie folgt vor: Wählen Sie Daten als Vorschautyp; Wählen Sie den Filtertyp aus dem Dropdown-Menü neben Filter aus. Wählen Sie 3D für die Interpretation; Wählen Sie Separierbar aus dem Dropdown-Menü neben Kernel-Typ; Geben Sie die Werte, die für die Standardabweichung und den Korngrößenfaktor verwendet werden sollen, in das verfügbare leere Feld daneben ein. Wählen Sie im Dropdown-Menü neben Ausgabe die Option Wie Eingabe aus. Klicken Sie auf Übernehmen.
HINWEIS: Die Wahl des Filtertyps (verfügbare Optionen sind bilateral, Box, Gauß, Median, rekursiv exponentiell, abgegrenzt, anisotrope Diffusion, nicht-lokale Mittelwerte, unscharfe Maskierung und FFT-Filter) und Parameter hängt vom Rauschpegel und der Voxelgröße der gescannten Bilder ab. Für den Gauß-Filter sind 3 x 3 x 3 und 5 x 5 x 5 häufig verwendete Werte für den Kerngrößenfaktor, und 0,5-2,0 wird üblicherweise für die Standardabweichung8 verwendet. In dieser Studie wurde ein Gauß-Filter angewendet, und 5 x 5 x 5 und 0,8 wurden für den Korngrößenfaktor bzw. die Standardabweichung verwendet.
- Neuausrichtung des Bildes
HINWEIS: Dies ist ein optionaler Schritt. Wenn während des Scanvorgangs eine Fehlausrichtung von Röhrenknochenproben relativ zu den Koordinatenachsen des Bildgebungssystems auftritt, kann eine digitale Ausrichtungsmethode angewendet werden, um die Fehlausrichtung zu korrigieren (Abbildung 2C).- Erstellen Sie wie folgt ein gerendertes 3D-Bild des Beispiels. Wählen Sie im Fenster der Projektansicht das gefilterte, zugeschnittene Bild (erstellt in Schritt 3.3) aus. Klicken Sie mit der rechten Maustaste, um Anzeige und dann Volumen-Rendering aus dem Dropdown-Menü auszuwählen, und klicken Sie dann auf Erstellen. Überprüfen Sie das 3D-gerenderte Bild visuell in der sagittalen und frontalen Ebene.
- Drehen Sie das gerenderte Volumen manuell, um eine gute Ausrichtung in der Längsachse zu erhalten. Wenden Sie die Transformation wie folgt auf die gedrehten Bilder an: Klicken Sie im Eigenschaftenfenster auf den Transformationseditor, gehen Sie dann zu Transformationseditor-Manipulator und wählen Sie Transformator aus dem Dropdown-Menü. Nun kann die Probe gedreht und neu ausgerichtet werden. Sobald der Neuausrichtungsprozess abgeschlossen ist, klicken Sie erneut auf den Transformationseditor , um das Bild zu sperren.
- Berechnen Sie das gefilterte Bild (das in Schritt 3.3 erstellt wurde) erneut, um wie folgt neue Bildschichten in der Querebene (axial) zu erstellen: Wählen Sie im Fenster Projektansicht das Bild aus Schritt 3.4.2 aus. Klicken Sie mit der rechten Maustaste, um Geometrietransformation und dann Transformiertes Bild neu berechnen aus dem Dropdown-Menü auszuwählen, und klicken Sie auf Erstellen. Wechseln Sie im Eigenschaftenfenster zu Daten , und führen Sie die folgenden Schritte aus: Wählen Sie für die Interpolation Standard aus dem Dropdown-Menü aus. Wählen Sie für mode die Option Extended; Wählen Sie für preserve (Beibehalten) die Option Voxel-Größe. Geben Sie für den Auffüllungswert Null in das leere Feld Verfügbares Feld ein. Klicken Sie dann auf Übernehmen.
- Definition des interessierenden Volumens (VOI)
- Gehen Sie die Querbildschnitte durch und identifizieren Sie die Mittelebene des Frakturkalus. Definieren Sie die VOI basierend auf den proximalen und distalen Enden des Kallus. In Fällen, in denen die Hornhautenden schwer zu definieren sind, definieren Sie die VOI auf der Grundlage eines standardisierten Abstands von der Kallusmittelebene (Abbildung 2D).
HINWEIS: Während der Heilungsphasen, die dem Knochenumbau vorausgehen, ist es einfach, die Ränder des mineralisierten Kallus zu definieren, da sich die trabekuläre Struktur des neu gebildeten gewebten Knochens von der kortikalen Struktur des ursprünglichen Knochens unterscheidet. Wenn jedoch die Umbauphase folgt, nimmt der neu gebildete Knochen allmählich die kortikale Struktur an; Daher wird es immer schwieriger, die Ränder der Hornhaut zu definieren.
- Gehen Sie die Querbildschnitte durch und identifizieren Sie die Mittelebene des Frakturkalus. Definieren Sie die VOI basierend auf den proximalen und distalen Enden des Kallus. In Fällen, in denen die Hornhautenden schwer zu definieren sind, definieren Sie die VOI auf der Grundlage eines standardisierten Abstands von der Kallusmittelebene (Abbildung 2D).

Abbildung 2: Bildsegmentierung . (A) Ein Bild, das sechs Proben innerhalb eines Scans zeigt. (B) Bildzuschnitt zur Isolierung einzelner Proben. (C) Digitale Ausrichtung zur Korrektur einer falsch ausgerichteten Längsachse (gelbe gestrichelte Linie). (D) Definition der VOI- und Kallusmittelebene. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.
4. Bildanalyse
- Segmentierung des Kallus und des kortikalen Knochens
- Konturieren Sie die äußere Begrenzung der Hornhaut halbautomatisch mit dem Segmentierungs-Lasso-Werkzeug mit den Optionen "Automatisch nachzeichnen" und "Kanten nachzeichnen" (Abbildung 3A) wie folgt:
- Klicken Sie nach dem erneuten Zusammensetzen der transformierten Bilder (Schritt 3.4.3) auf die Registerkarte Segmentierung in der zweiten Registerkartenzeile am oberen Rand des Bildschirms. Wählen Sie im Fenster des Segmentierungseditors ein transformiertes Bild (erstellt in Schritt 3.4.3) aus dem Dropdown-Menü neben Bild aus.
- Doppelklicken Sie im Fenster MATERIALIEN auf Hinzufügen. Auf diese Weise werden zwei Registerkarten mit den Namen Material3 und Material4 angezeigt. Klicken Sie mit der rechten Maustaste, um Material3 in Hornhaut und Material4 in kortikalen Knochen umzubenennen.
- Klicken Sie im Fenster AUSWAHL auf das Lasso-Symbol. Wählen Sie aus den angezeigten Optionen Freihand für den 2D-Modus, Innen für den 3D-Modus und sowohl Automatisch nachzeichnen als auch Kanten nachzeichnen für Optionen aus. Verwenden Sie das Lasso, um die interessierenden Bereiche zu markieren.
- Wiederholen Sie diesen Konturierungsschritt mit Schichten, die über den VOI abgetastet werden (Abbildung 3B). Die konturierten Scheiben können beabstandet werden (z. B. durch 20 Scheiben getrennt).
HINWEIS: In Bereichen mit komplexen Kallusstrukturen kann der Benutzer den Abstand zwischen den konturierten Schichten verringern, um mehr Fragmente zu erfassen (Abbildung 3A,B). - Interpolieren Sie über die konturierten Hornhautkonturen, um eine vollständige Hornhautbeschriftung (Abbildung 3C, D) wie folgt zu erstellen: Wählen Sie im Fenster MATERIALIEN die Callus-Datei (erstellt in Schritt 4.1.1.2) aus, klicken Sie oben auf dem Bildschirm auf die Registerkarte Auswahl und wählen Sie im Dropdown-Menü die Option Interpolieren. Klicken Sie im Fenster AUSWAHL auf das Pluszeichen.
- Öffnen Sie die Datei Cortical Bone, die Sie in Schritt 4.1.1.2 erstellt haben. Der kortikale Knochen einschließlich der Markhöhle wird segmentiert, wie in den Schritten 4.1.1 und 4.1.2 für den Kallus beschrieben. (Abbildung 4A,B). Interpolieren Sie den konturierten periostalen Kortex, um eine kortikale Knochenmarkierung zu erstellen, wie sie in Schritt 4.1.3 für den Kallus beschrieben wurde (Abbildung 4C,D).
- Berechnen Sie das konturierte Volumen und den mittleren Grauwert der Hornhaut wie folgt: Klicken Sie auf die Registerkarte Segmentierung in der oberen Zeile des Bildschirms und wählen Sie Materialstatistik aus dem Dropdown-Menü. Dadurch wird eine Tabelle generiert, die alle berechneten Werte enthält. Die Werte des kortikalen Knochens und des Kallus (nach Subtraktion des kortikalen Knochens) werden separat angegeben. Nachdem die Tabelle generiert wurde, klicken Sie auf In Workspace exportieren , um die Daten zu speichern.
- Konturieren Sie die äußere Begrenzung der Hornhaut halbautomatisch mit dem Segmentierungs-Lasso-Werkzeug mit den Optionen "Automatisch nachzeichnen" und "Kanten nachzeichnen" (Abbildung 3A) wie folgt:
- Umrechnung von Graustufeneinheiten in Knochenmineraldichte
- Schneiden Sie das 3D-Bild des 4,5-mm-HA-Phantoms (Abbildung 2B) aus dem gesamten Bild zu, und klicken Sie auf Segmentierung. Das Harz des HA-Phantoms enthält fünf kleine HA-Zylinder (Abbildung 1A). Definieren Sie für den HA-Zylinder mit der höchsten Dichte die erste und die letzte Schicht durch visuelle Inspektion.
- Zeichnen Sie zwei Kreise an der ersten und der letzten Schicht (ohne Kanten) mit dem Pinselwerkzeug (Abbildung 5A) wie folgt: Klicken Sie im Fenster MATERIALIEN viermal auf Hinzufügen . Klicken Sie mit der rechten Maustaste, um material3, material4, material5 und material6 in phantom1, phantom2, phantom3 bzw. phantom4 umzubenennen. Wählen Sie Phantom1 aus, klicken Sie auf das Pinselsymbol im Fenster AUSWAHL und verwenden Sie den Schieberegler, um die Pinselgröße (kreisförmiges Nachzeichnen) basierend auf der Größe des Phantoms anzupassen (die Größe des Kreises sollte kleiner sein als die des Phantoms).
- Wenden Sie die Interpolation zwischen den beiden Kreisen an, um ein Volumen für jeden HA-Zylinder (Abbildung 5B) wie folgt zu erstellen: Wählen Sie im Fenster MATERIALIEN die Option Phantom1 aus, klicken Sie auf die Registerkarte Auswahl in der oberen Zeile des Bildschirms und wählen Sie Interpolieren aus dem Dropdown-Menü. Klicken Sie im Fenster AUSWAHL auf das Pluszeichen.
- Wiederholen Sie den Segmentierungsprozess mit drei der verbleibenden HA-Zylinder, beginnend mit der zweithöchsten HA-Dichte und endend mit der zweitniedrigsten HA-Dichte (Abbildung 5B). Der Zylinder mit der geringsten HA-Dichte kann ausgeschlossen werden, da er oft schwer zu segmentieren ist.
- Verwenden Sie die generierten 3D-Beschriftungen, um die mittleren Grauwerte der vier analysierten HA-Zylinder zu berechnen. Tragen Sie mit Hilfe einer Tabelle (siehe Materialtabelle) oder ähnlichem die mittleren Grauwerte und die entsprechenden Knochenmineraldichtewerte (BMD) auf, die vom Phantomhersteller bereitgestellt werden. Generieren Sie eine Korrelationsgleichung zwischen BMD und den Grauwerten mithilfe der linearen Regression.
- Segmentierung des mineralisierten Kalluses und Berechnung der BMD
- Basierend auf der in Schritt 4.2.5 generierten Korrelationsgleichung und dem gewählten Schwellenwert, der zwischen mineralisiertem und unmineralisiertem Kallus unterscheidet, bestimmen Sie den entsprechenden Grauwertschwellenwert. Dementsprechend wird der Kallusbereich mit Grauwerten, die über dem Schwellenwert liegen, als mineralisierter Kallus und der Rest als nicht mineralisiert gekennzeichnet (Abbildung 6A,B). In dieser Studie wurden 250 mgHA/ccm als Schwellenwert für mineralisierte Kallhaut14,15 verwendet.
- Berechnen Sie das gesamte Kallus- und mineralisierte Kallusvolumen. Berechnen Sie anhand dieser Werte den Knochenvolumenanteil (mineralisiertes Kallusvolumen, normiert auf das Gesamtkallusvolumen = BV/TV). Verwenden Sie den gemessenen mittleren Grauwert für die gesamte Kallus, um die BMD der Hornhaut mit Hilfe der in Abschnitt 4.2.5 generierten Korrelationsgleichung zu berechnen.
HINWEIS: Basierend auf dem Ziel der Studie und der für die Analyse verwendeten Software können andere Parameter wie SMI (Strukturmodellindex), Trabekeldicke und Anisotropiegrad berechnet werden.

Abbildung 3: Segmentierung der äußeren Kallusgrenze. (A) Eine Kontur der äußeren Begrenzung der Hornhaut (rote Linie). (B) Konturen an Schichten, die über die VOI abgetastet wurden (rote Schichten). (C) Eine durch Interpolation erzeugte 3D-Kallusbeschriftung (rotes Volumen). (D) Ein Querschnitt der in C dargestellten Kallusmarkierung (einschließlich kortikaler Knochen). Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.

Abbildung 4: Segmentierung des kortikalen Knochens. (A) Eine Kontur der periostalen Oberfläche des Kortex (grüne Linie). (B) Konturen an Schichten, die über die VOI abgetastet wurden (grüne Schichten). (C) Eine 3D-Markierung des kortikalen Knochens (mit Markhöhle; grün) und des Kallus (rot), die aus interpolierten Markierungen des periostalen Kortex und des Kallus erstellt wurde. (D) Ein Querschnitt des Kallus (rot) und des kortikalen Knochens (mit der Markhöhle; grün). Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.

Abbildung 5: Umrechnung von Graustufeneinheiten in BMD. (A) Konturen des HA-Zylinders an der ersten und der letzten Schicht (rote Kreise). (B) 3D-interpolierte HA-Zylinder (links) und Querschnitte (rechts). Braun: höchste HA-Dichte; blau: zweithöchste HA-Dichte; violett: dritthöchste HA-Dichte; grün: vierthöchste HA-Dichte. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.
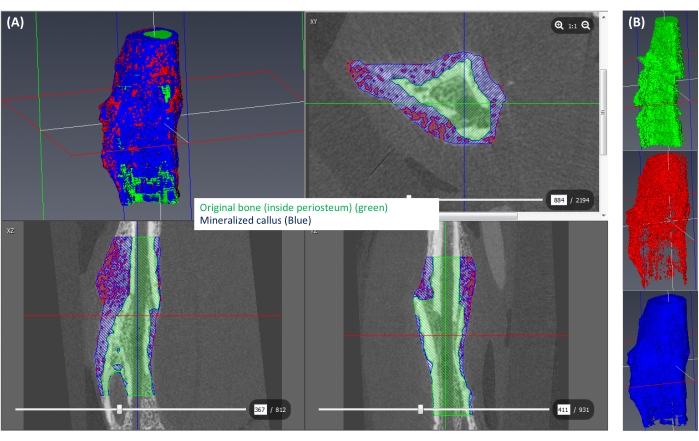
Abbildung 6: Segmentierung des mineralisierten Kallus. (A) Der mineralisierte Kallus (≥250 mgHA/ccm) ist blau dargestellt, der Rest des Kallus (<250 mgHA/ccm) ist rot und der dem ursprünglichen Knochen entsprechende Raum ist grün dargestellt. (B) Eine 3D-Ansicht jedes isolierten Etiketts. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.
Ergebnisse
Um die Knochenbildung während der Frakturheilung zu überwachen, wurde bei adulten, männlichen C75BL/6J-Mäusen eine mitteldiaphysäre offene Tibiafraktur induziert. Die Fraktur wurde mit einem Marknagel stabilisiert, einem etablierten Modell der Sekundärheilung13. Das Kallusgewebe wurde an den Tagen 14, 21 und 28 nach der Frakturentnommen 12. Diese Zeitpunkte repräsentieren verschiedene Phasen der Heilung. Die endochondrale Knochenbildung während der sekundären Knoch...
Diskussion
Der Zweck dieser Studie ist es, ein detailliertes Protokoll für die μCT-Analyse mit dem Ziel einer genauen Quantifizierung der 3D-mineralisierten Kallusstruktur zu beschreiben, die bei Knochen- und Frakturheilungsstudien oft von grundlegender Bedeutung ist. Das Protokoll nutzt eine universelle, hochmoderne 3D-Bildanalyse-Softwareplattform, die die Bildvisualisierung, Segmentierung/Beschriftung und Messungen von einfach bis komplex erleichtert.
Die zeitaufwändigste Aufgabe im Protokoll ist d...
Offenlegungen
Die Autoren haben keine Interessenkonflikte offenzulegen.
Danksagungen
Diese Arbeit wurde von den National Institutes of Health (NIH) R01 DK121327 zu R.A.E und R01 AR071968 zu F.K.
Materialien
| Name | Company | Catalog Number | Comments |
| 10% neutral buffered formalin | Fisher chemical | SF100-20 | Used for bone tissue fixation |
| Avizo | Thermo Scientific | Image processing and analysis software | |
| Hydroxyapatite phantom | Micro-CT HA D4.5, QRM | QRM-70128 | |
| Image Processing Language | Scanco | Used to convert raw images to DICOM images | |
| Micro-Mosquito Straight Hemostatic Forceps | Medline | Used to remove the intramedullary pin | |
| Microsoft Excel | Microsoft | Spreadsheet software | |
| Scanco mCT system (vivaCT 40) | Scanco | Used for µCT imaging |
Referenzen
- Rüegsegger, P., Koller, B., Müller, R. A microtomographic system for the nondestructive evaluation of bone architecture. Calcified Tissue International. 58 (1), 24-29 (1996).
- Müller, R., et al. Morphometric analysis of human bone biopsies: a quantitative structural comparison of histological sections and micro-computed tomography. Bone. 23 (1), 59-66 (1998).
- Waarsing, J. H., et al. Detecting and tracking local changes in the tibiae of individual rats: a novel method to analyse longitudinal in vivo micro-CT data. Bone. 34 (1), 163-169 (2004).
- Boyd, S. K., Davison, P., Müller, R., Gasser, J. A. Monitoring individual morphological changes over time in ovariectomized rats by in vivo micro-computed tomography. Bone. 39 (4), 854-862 (2006).
- Christiansen, B. A. Effect of micro-computed tomography voxel size and segmentation method on trabecular bone microstructure measures in mice. Bone Reports. 5, 136-140 (2016).
- Holdsworth, D. W., Thornton, M. M. Micro-CT in small animal and specimen imaging. Trends in Biotechnology. 20 (8), 34-39 (2002).
- Schambach, S. J., Bag, S., Schilling, L., Groden, C., Brockmann, M. A. Application of micro-CT in small animal imaging. Methods. 50 (1), 2-13 (2010).
- Bouxsein, M. L., et al. Guidelines for assessment of bone microstructure in rodents using micro-computed tomography. Journal of Bone and Mineral Research. 25 (7), 1468-1486 (2010).
- Morgan, E. F., et al. Micro-computed tomography assessment of fracture healing: Relationships among callus structure, composition, and mechanical function. Bone. 44 (2), 335-344 (2009).
- O'Neill, K. R., et al. Micro-computed tomography assessment of the progression of fracture healing in mice. Bone. 50 (6), 1357-1367 (2012).
- Bissinger, O., et al. Fully automated segmentation of callus by micro-CT compared to biomechanics. Journal of Orthopaedic Surgery and Research. 12 (1), 108 (2017).
- Brown, M. L., et al. Delayed fracture healing and increased callus adiposity in a C57BL/6J murine model of obesity-associated type 2 diabetes mellitus. PLOS One. 9 (6), 99656 (2014).
- Khajuria, D. K., et al. Aberrant structure of fibrillar collagen and elevated levels of advanced glycation end products typify delayed fracture healing in the diet-induced obesity mouse model. Bone. 137, 115436 (2020).
- Sigurdsen, U., Reikeras, O., Hoiseth, A., Utvag, S. E. Correlations between strength and quantitative computed tomography measurement of callus mineralization in experimental tibial fractures. Clinical Biomechanics. 26 (1), 95-100 (2011).
- Duvall, C. L., Taylor, W. R., Weiss, D., Wojtowicz, A. M., Guldberg, R. E. Impaired angiogenesis, early callus formation, and late stage remodeling in fracture healing of osteopontin-deficient mice. Journal of Bone and Mineral Research. 22 (2), 286-297 (2007).
- Gerstenfeld, L. C., et al. Comparison of effects of the bisphosphonate alendronate versus the RANKL inhibitor denosumab on murine fracture healing. Journal of Bone and Mineral Research. 24 (2), 196-208 (2009).
- Alentado, V. J., et al. Validation of the modified radiographic union score for tibia fractures (mRUST) in murine femoral fractures. Frontiers in Endocrinology. 13, 911058 (2022).
- Yu, K. E., et al. Enhancement of impaired MRSA-infected fracture healing by combinatorial antibiotics and modulation of sustained inflammation. Journal of Bone and Mineral Research. 37 (1), 1352-1365 (2022).
- Nyman, J. S., et al. Quantitative measures of femoral fracture repair in rats derived by micro-computed tomography. Journal of Biomechanics. 42 (7), 891-897 (2009).
- Fiset, S., et al. Experimental validation of the radiographic union score for tibial fractures (RUST) using micro-computed tomography scanning and biomechanical testing in an in-vivo rat model. The Journal of Bone and Joint Surgery. 100 (21), 1871-1878 (2018).
- Shefelbine, S. J., et al. Prediction of fracture callus mechanical properties using micro-CT images and voxel-based finite element analysis. Bone. 36 (3), 480-488 (2005).
- Liu, Y., et al. Glucocorticoid-induced delayed fracture healing and impaired bone biomechanical properties in mice. Clinical Interventions in Aging. 13, 1465-1474 (2018).
- Watson, P. J., Fitton, L. C., Meloro, C., Fagan, M. J., Gröning, F. Mechanical adaptation of trabecular bone morphology in the mammalian mandible. Scientific Reports. 8 (1), 7277 (2018).
- Nie, C., Wang, Z., Liu, X. The effect of depression on fracture healing and osteoblast differentiation in rats. Neuropsychiatric Disease and Treatment. 14, 1705-1713 (2018).
Nachdrucke und Genehmigungen
Genehmigung beantragen, um den Text oder die Abbildungen dieses JoVE-Artikels zu verwenden
Genehmigung beantragenThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Alle Rechte vorbehalten