Для просмотра этого контента требуется подписка на Jove Войдите в систему или начните бесплатную пробную версию.
Method Article
Высокоскоростной магнитный пинцет для наномеханических измерений на чувствительных к силе элементах
* Эти авторы внесли равный вклад
В этой статье
Резюме
Здесь мы описываем высокоскоростной магнитный пинцет, который выполняет наномеханические измерения чувствительных к силе биомолекул с максимальной частотой 1,2 кГц. Мы представляем его применение к шпилькам ДНК и комплексам SNARE в качестве модельных систем, но он также будет применим к другим молекулам, участвующим в механобиологических событиях.
Аннотация
Одномолекулярные магнитные пинцеты (МТ) служили мощными инструментами для принудительного опроса биомолекул, таких как нуклеиновые кислоты и белки, и поэтому готовы быть полезными в области механобиологии. Поскольку метод обычно основан на отслеживании магнитных шариков на основе изображений, ограничение скорости при записи и анализе изображений, а также тепловые колебания шариков долгое время препятствовали его применению при наблюдении небольших и быстрых структурных изменений в молекулах-мишенях. В этой статье подробно описаны методы построения и эксплуатации установки МТ высокого разрешения, которая может разрешать наноразмерную, миллисекундную динамику биомолекул и их комплексов. В качестве прикладных примеров демонстрируются эксперименты с шпильками ДНК и комплексами SNARE (механизм слияния мембран), в которых основное внимание уделяется тому, как их переходные состояния и переходы могут быть обнаружены в присутствии сил пиконетонного масштаба. Мы ожидаем, что высокоскоростные МТ будут по-прежнему обеспечивать высокоточные наномеханические измерения молекул, которые чувствуют, передают и генерируют силы в клетках, и тем самым углубят наше понимание механобиологии на молекулярном уровне.
Введение
Клетки активно ощущают и реагируют на механические раздражители. При этом многие биомолекулы проявляют силовые свойства, которые обеспечивают динамические структурные изменения. Хорошо оцененные примеры включают механочувствительные ионные каналы и цитоскелетные элементы, которые обеспечивают клетки ключевой механической информацией из окружающей среды.
Кроме того, молекулы, обладающие уникальной силовой природой, также можно считать механочувствительными в более широком смысле. Например, локальное образование и плавление дуплексов нуклеиновых кислот, а также структур более высокого порядка, таких как G-квадруплексы, играют решающую роль в репликации, транскрипции, рекомбинации и, в последнее время, редактировании генома. Более того, некоторые нейронные белки, участвующие в синаптических коммуникациях, выполняют свои функции, генерируя физические силы, которые превышают уровни типичных межмолекулярных взаимодействий. Независимо от того, на каком примере вы проводите исследование, изучение наномеханики вовлеченных биомолекул с высокой пространственно-временной точностью окажется очень полезным для выявления молекулярных механизмов связанных с ними механобиологических процессов 1,2,3.
Методы одномолекулярной силовой спектроскопии послужили мощными инструментами для изучения механических свойств биомолекул 2,4,5,6. Они могут отслеживать структурные изменения в нуклеиновых кислотах и белках одновременно с приложением силы, тем самым исследуя силозависимые свойства. Двумя хорошо известными установками являются оптический пинцет и магнитный пинцет (МТ), в которых используются шарики микронного размера для манипулирования молекулами 5,6,7,8. В этих платформах полистирол (для оптического пинцета) или магнитные шарики (для МТ) привязаны к молекулам-мишеням (например, нуклеиновым кислотам и белкам) через молекулярные «ручки», обычно состоящие из коротких фрагментов двухцепочечной ДНК (дцДНК). Затем шарики перемещаются, чтобы приложить силу, и визуализируются, чтобы отслеживать их местоположение, которые сообщают о структурных изменениях в молекулах-мишенях. Оптический и магнитный пинцет в значительной степени взаимозаменяемы в своих применениях, но существуют важные различия в их подходах к управлению силой. Оптический пинцет по своей сути представляет собой инструменты с позиционным зажимом, которые захватывают шарики в нужном положении, из-за чего приложенная сила колеблется, когда целевая конструкция претерпевает изменения формы; Увеличение растяжения, например, от раскладывания, ослабляет трос и снижает натяжение, и наоборот. Хотя активная обратная связь может быть реализована для управления силой в оптических пинцетах, MT, напротив, естественным образом работают как устройство силового зажима, используя преимущества стабильных магнитных сил в дальней зоне постоянными магнитами, которые также могут выдерживать возмущения окружающей среды.
Несмотря на свою долгую историю и простую конструкцию, МТ отстают от оптических пинцетов в их приложениях для высокоточных измерений, в основном из-за технических проблем с быстрым отслеживанием шариков. Однако в последнее время несколько групп совместно провели многогранное совершенствование как аппаратного, так и программного обеспечения для приборов машинного перевода 2,9,10,11,12,13,14,15,16,17,18,19 . В этой работе мы представляем пример такой установки, работающей на частоте 1,2 кГц, и описываем, как использовать ее для выполнения наномеханических измерений на чувствительных к силе биомолекулах. В качестве модельных систем мы используем шпильки ДНК и нейрональные комплексы SNARE и исследуем их быстрые структурные изменения в режиме пиконетона. Шпильки ДНК демонстрируют простые переходы с двумя состояниями в четко определенном диапазоне сил20,21 и, следовательно, служат игрушечными моделями для проверки работоспособности установки пинцета. Поскольку белки SNARE собираются в чувствительный к силе комплекс, который управляет слиянием мембран22, они также были тщательно изучены с помощью одномолекулярной силовой спектроскопии 14,23,24,25. Представлены стандартные подходы к анализу данных и извлечению полезной информации по термодинамике и кинетике. Мы надеемся, что эта статья может способствовать внедрению высокоточных МТ в механобиологические исследования и мотивировать читателей исследовать свои собственные чувствительные к силе системы, представляющие интерес.
протокол
Все материалы и оборудование, описанные в этом протоколе, перечислены в Таблице материалов. Программное обеспечение LabVIEW для работы с высокоскоростной установкой машинного перевода, описанной ниже, а также скрипты MATLAB для анализа демонстрационных данных размещены на GitHub (https://github.com/ShonLab/Magnetic-Tweezers) и находятся в открытом доступе.
1. Конструкция аппарата
ПРИМЕЧАНИЕ: Общий принцип построения высокоскоростного МТ аналогичен стандартным, обычным системам МТ, за исключением использования высокоскоростной комплементарной камеры на основе металл-оксидного полупроводника (КМОП) и мощного когерентного источника света (рис. 1). Обратитесь к другим источникам для получения более подробной информации о стандартных приборах МТ 5,26,27.
- Установите инвертированный микроскоп на антивибрационный оптический стол. Установите высокоскоростную CMOS-камеру и фреймграббер.
- Постройте сцену перевода для манипулирования магнитами в 3D. Установите моторизованный линейный столик (ход >20 мм) вертикально поверх ручного столика XY.
ПРИМЕЧАНИЕ: Вертикальное движение управляет усилием, тогда как ступень XY предназначена для ручного выравнивания магнитов по оптической оси для первоначальной сборки установки. - Установите роторный шаговый двигатель и систему ремней и шкивов для вращающихся магнитов.
ПРИМЕЧАНИЕ: Ремень передает вращательное движение между валом двигателя и магнитами, находящимися на расстоянии нескольких сантиметров друг от друга. Вращение магнитов является внутренним для поступательной манипуляции. - Установите магниты. Используйте акриловый держатель (заказанный у компании-производителя; см. дополнительный рисунок S1), который может плотно разместить два одинаковых магнита параллельно, с четко определенным зазором 1 мм между магнитами (рис. 1B). Чтобы использовать максимальное усилие, достижимое с данной парой магнитов, отрегулируйте вертикальное положение подвижного столика так, чтобы нижняя поверхность магнитов совпадала с плоскостью образца при перемещении в самое нижнее положение.
ПРИМЕЧАНИЕ: Обратитесь к Lipfert et al. для получения дополнительной информации о конструкции держателя и конфигурациимагнитов 28. Высота и ориентация магнитов контролируются программным обеспечением LabVIEW в сочетании со сбором данных. - При просмотре с помощью объектива с малым увеличением выровняйте магниты по центру поля зрения. Убедитесь, что вращение магнитов не вызывает большого смещения центра пары магнитов.
ПРИМЕЧАНИЕ: Если средняя точка между магнитами вращается вокруг оси вращения, вполне вероятно, что магниты смещены от центра из-за несовершенного держателя. Небольшой уровень смещения по отношению к размеру зазора допустим, так как вращение магнита предназначено только для проверки тросов и приложения крутящих моментов в определенных приложениях. - Установите суперлюминесцентный диод (SLD) для подсветки бусин. Пропустите луч через зазор 1 мм между двумя магнитами. Убедитесь, что луч правильно коллимирован, чтобы поместиться в зазоре, и освещение не затеняется магнитами.
- Установите пьезосканер на наконечник и установите объектив с 100-кратным погружением в масло (числовая апертура [NA]: 1,45) для отслеживания борта. Чтобы избежать потенциальных артефактов в результатах отслеживания, убедитесь, что освещение поддерживается равномерно при перемещении магнитов. Наконец, отрегулируйте уровень освещенности до максимальной яркости без насыщения пикселей.
ПРИМЕЧАНИЕ: Для сравнения различных источников света для высокоскоростного слежения за шариками см. Dulin et al.29.
2. Калибровка магнитной силы
- Используя полимеразную цепную реакцию (ПЦР; см. Таблицу 1), подготовьте 5 фрагментов дцДНК kbp (с использованием праймера B, праймера Z_5k и λ-ДНК), которые помечены биотином на одном конце (для поверхностного прикрепления) и азидом на другом конце (для прикрепления шариков).
- Следуя разделу 6, подготовьте проточную ячейку с молекулами 5 kbp.
- Следуя разделу 7, определите хорошую конструкцию бортового троса, проверив ее удлинение и вращение. В частности, обязательно выбирайте валик с минимальной траекторией вращения (т.е. с радиусом <200 нм), чтобы минимизировать смещение высоты валика из-за нецентрированного крепления30,31. Как только хороший трос будет идентифицирован, начните отслеживание борта, ссылаясь на раздел 9.
- Если установка новая, охарактеризуйте ее шум и стабильность для надежных измерений с высоким разрешением. Поместите магнит на расстоянии ~ 3 мм от поверхности проточной ячейки (чтобы применить >10 пН и подавить броуновское движение шарика), отследите z-положение шарика на частоте 1,2 кГц и вычислите отклонение Аллана (AD) от z-координатного временного ряда32,33 (рис. 2C). Убедитесь, что значения AD в несколько нанометров достижимы в высокоскоростном режиме (<0,1 с) и что дифференциальное отслеживание (положение магнитного валика относительно эталонного валика) снижает AD в более длительном временном масштабе.
ПРИМЕЧАНИЕ: Обычно мы получаем AD <3 нм при максимальной частоте (разрешение 1,2 кГц или 0,83 мс), а AD продолжает уменьшаться, по крайней мере, до 10 с, что подразумевает минимальный дрейф. Другие сообщили об аналогичных значениях на аналогичных установках 9,10,11,12,34. - С магнитами в положении покоя (F ~ 0 pN) запишите координаты x и y привязанного шарика на частоте 1,2 кГц. Запишите положение в течение достаточно длительного периода (т. е. достаточно большего, чем характерное время релаксации флуктуации35), чтобы броуновское движение было достаточно выборизировано.
ПРИМЕЧАНИЕ: Здесь направление x соответствует направлению магнитного поля, тогда как движение в y представляет собой поперечное движение, перпендикулярное полю. - Переместите магниты ближе к проточной ячейке и повторяйте измерения положения шарика, пока магниты не коснутся верхней части проточной ячейки. Двигайтесь большими шагами (например, 1-2 мм), когда магниты находятся на расстоянии более 7 мм от плоскости образца (поскольку приложенная сила медленно увеличивается в дальнем поле магнитов), но постепенно уменьшайте размер шага (например, 0,1-0,5 мм) по мере приближения для более точной калибровки при более высоких уровнях силы (рис. 2B).
- Рассчитайте силу в каждом положении магнита, d, используя любой из двух альтернативных методов (предоставляется сценарий MATLAB «калибровка силы.m», включающий оба метода; см. Дополнительный файл 1).
- Измерьте дисперсию y-координат
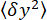 бусины (рис. 2D) и среднее z-положение
бусины (рис. 2D) и среднее z-положение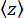 бусины относительно самого низкого положения (рис. 2B, внизу). Затем используйте уравнение (1)7,27,36 для оценки силы (с фиксированным радиусом валика R = 1,400 нм и тепловой энергией k RT = 4.11 пН∙нм):
бусины относительно самого низкого положения (рис. 2B, внизу). Затем используйте уравнение (1)7,27,36 для оценки силы (с фиксированным радиусом валика R = 1,400 нм и тепловой энергией k RT = 4.11 пН∙нм):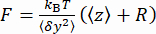 (1)
(1) - В качестве альтернативы можно рассчитать спектральную плотность мощности (PSD) y-координат Sy (рис. 2E). Определите приложенную силу F, подгонив двойную лоренцеву модель37 к измеренному Sy с помощью уравнения (2).
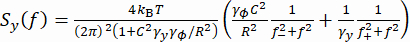 (2)
(2)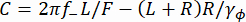 Здесь , R — радиус валика, γ yи γφ — коэффициенты поступательного и вращательного сопротивления соответственно (оцениваемые по уравнению Стокса-Эйнштейна), k RT — тепловая энергия, f+ и f- две характеристические частоты, полученные с помощью уравнения (3).
Здесь , R — радиус валика, γ yи γφ — коэффициенты поступательного и вращательного сопротивления соответственно (оцениваемые по уравнению Стокса-Эйнштейна), k RT — тепловая энергия, f+ и f- две характеристические частоты, полученные с помощью уравнения (3).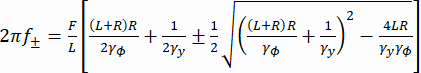 (3)
(3)
ПРИМЕЧАНИЕ: Поскольку удлинитель троса L является функцией силы, которая следует хорошо зарекомендовавшей себя модели червячной цепи (WLC), приведенные выше выражения оставляют F в качестве единственного подходящего параметра (для простоты мы устанавливаем R равным 1,400 нм, потому что он распределяется по всем уровням силы, и точное значение не оказывает заметного влияния на результаты). При необходимости размытие при движении и сглаживание при получении изображения с помощью камеры должны рассматриваться как38,39, но этот эффект незначителен в наших высокоскоростных измерениях выше 1 кГц с привязями 5 кбит/с.
- Измерьте дисперсию y-координат
- Повторите шаги 2.4-2.7 для еще нескольких конструкций. Исследуйте от трех до пяти различных шариков, чтобы усреднить изменчивость силы между магнитными шариками.
ПРИМЕЧАНИЕ: Изменение силы между используемыми магнитными шариками следует учитывать для определения правильного количества конструкций для усреднения. Эта изменчивость невелика, но может привести к погрешности измеряемой силы более чем на 1 пН даже для коммерческих продуктов31. Для большинства применений, где абсолютное определение задействованных сил не имеет решающего значения, обычно достаточно усреднения результатов калибровки трех-пяти шариков. Альтернативный подход к учету этого изменения заключается в измерении силы с помощью отдельных тросов в начале эксперимента, что может занять много времени. Другой вариант состоит в том, чтобы встроить шпильки, которые распаковываются на известных уровнях силы в каждую конструкцию31. - Построите график измеренной силы в зависимости от расстояния до магнита и подгоните двойную экспоненциальную функцию к данным (рис. 2F), используя уравнение (4).
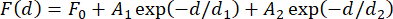 (4)
(4)
Здесь F0 (базовая линия), A 1 и A 2 (амплитуды) и d 1 и d2 (константы распада) являются подходящими параметрами. Убедитесь, что значения силы из двух методов, а также результирующие двойные экспоненциальные подгонки в значительной степени совпадают (рис. 2F, G).
ПРИМЕЧАНИЕ: Чтобы убедиться, что калибровка силы выполнена правильно, проверьте соотношение силы и удлинения зондируемых конструкций, построив график зависимости удлинения от измеренной силы. - Чтобы скорректировать смещение высоты валика zв результате зависящего от силы наклона магнитных шариков30,31, оцените z от бокового смещения xoff, учитывая геометрию смещенногоот центра троса с радиусом валика, используя уравнение (5), и примените значения к измеренным значениям расширения. Этот шаг реализован в скрипте MATLAB "force calibration.m" (строки 252-254).
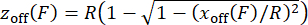 (5)
(5)
ПРИМЕЧАНИЕ: Несмотря на то, что эта поправка вносит небольшие изменения в удлинение, особенно для шариков с малым радиусом вращения (<200 нм), это смещение часто критически влияет на упругий отклик, как видно из перехода от рисунка 2H к рисунку 2I30,31. - Проверьте длину персистенции Lp , подгонив расширяемую модель WLC к данным с помощью уравнения (6).
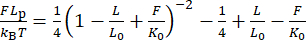 (6)
(6)
Здесь L0 — длина контура (1,7 мкм для 5 кбит/с), а K0 — модуль энтальпического растяжения.
ПРИМЕЧАНИЕ: Несмотря на то, что L p дцДНК хорошо принят как 40-50 нм в типичном буфере, таком как фосфатно-буферный физиологический раствор (PBS), формула WLC, применяемая к коротким молекулам (<5 kbp), систематически недооценивает L p, поскольку L0 уменьшаетсяна 31,40. Это связано с тем, что классическая модель WLC предполагает полимер, длина цепи которого достаточно больше, чем длина его стойкости. Здесь мы получили Lp = 40 ± 3 нм для конструкции 5 kbp (рис. 2H), а поправка на расширение в дальнейшем дала однородный K0 от 1,100 ± 200 пN (рис. 2I). Применение конечной моделиWLC 31,40, а также поправки на негауссованность в распределениирасширения 41 несколько увеличит Lp. - После проверки калибровки силы примените полученные параметры подгонки двухэкспоненциальной модели к предоставленному программному обеспечению LabVIEW (дополнительный файл 2) и подождите, пока программное обеспечение вычислит текущую силу в режиме реального времени по показаниям двигателя (т. е. положению магнита). Поскольку аналитическое выражение для обратной функции d(F) недоступно, подготовьте справочную таблицу d по сравнению с F с шагом 0,1 пН путем численной оценки целевых уровней силы d. Сохраните эту таблицу в программном обеспечении, чтобы управлять управлением усилием.
3. Синтез ДНК шпилек
ПРИМЕЧАНИЕ: Конструкции шпилек ДНК для экспериментов МТ получают путем ПЦР-амплификации области 510.н. в λ-ДНК с двумя пользовательскими праймерами, один из которых содержит структуру шпильки на своем 5'-конце (рис. 3A). Таким образом, мотив шпильки помещается на один конец продукта ПЦР.
- Подготовьте грунтовки.
- Прямой праймер: Праймер B_hp, который мечен 5'-биотином для прикрепления поверхности стекла и связывается с λ-ДНК. Этот праймер содержит мотив шпильки со стержнем 8.н. и петлей 6 нт, 5 футов до области λ-связывания.
- Обратный праймер: Праймер Z_hp, помеченный 5'-азидом для магнитного крепления шариков и связывающийся с λ-ДНК на расстоянии 1 кбп от прямого праймера.
- Настройте и запустите ПЦР с λ-ДНК (матрицей), nTaq-полимеразой и стандартными условиями ПЦР (см. Таблицу 1). Очистите продукт с помощью коммерческого набора для очистки.
- Измерьте концентрацию ДНК путем поглощения ультрафиолета на длине волны 260 нм (A260) и выполните электрофорез в агарозном геле (2% гель) (см. Таблицу 2), чтобы проверить размер продукта. Типичный выход составляет ~ 35 мкл раствора ~ 600 нМ.
4. Получение белков SNARE
ПРИМЕЧАНИЕ: Нейрональные комплексы SNARE собираются путем объединения трех очищенных белков крысы, экспрессируемых из кишечной палочки: VAMP2 / синаптобревин-2, синтаксин-1A и SNAP-25 (рис. 3B). Чтобы облегчить их сборку, синтаксин и SNAP-25 экспрессируются совместно с фрагментом VAMP2 (без N-концевой области; называемой «ΔN-VAMP2») в структуру, называемую «ΔN-комплексом», а затем смешиваются с полноразмерным VAMP2 после присоединения ДНК с образованием полных комплексов.
- Подготовьте плазмиды, содержащие кДНК, для экспрессии белков SNARE (последовательности ДНК для всех плазмид приведены в таблице материалов).
- Подготовьте 6× VAMP2 с тегом His, у которого отсутствует трансмембранный домен (2-97; L32C/I97C для дисульфидных связей) клонирован в вектор pET28a.
- Подготовьте синтаксин-1А, в котором отсутствует Habc и трансмембранный домен (замены 191-267, I202C/I266C для дисульфидных связей), клонированный вместе с 6×His-меченым ΔN-VAMP2 (49-96) в вектор pETDuet-1.
- Подготовьте полноразмерную изоформу b SNAP-25 (2-206, все от C до A), клонированную в вектор pET28a. Это будет использовано для подготовки комплексов ΔN.
- Подготовьте полноразмерную изоформу b SNAP-25 с 6×тегом HIS (1-206, все от C до A), клонированную в вектор pET28a, для прямого добавления в буфер анализа MT для повторной сборки комплексов SNARE после развертывания.
- Подготовьте две пробирки с клетками Rosetta (DE3) E. coli . Трансформируйте одну группу плазмидами VAMP2 (из шага 4.1.1), одну с синтаксином-1A/ΔN-VAMP2 и немечеными плазмидами SNAP-25 (из этапов 4.1.2 и 4.1.3) для экспрессии ΔN-комплекса, а другую с помощью плазмид SNAP-25 с тегом His (из шага 4.1.4).
- Перенесите трансформированные клетки в бульон Лурия-Бертани (LB) с соответствующими антибиотиками (здесь канамицин и хлорамфеникол для VAMP2 и SNAP-25 с тегом His; канамицин, хлорамфеникол и ампициллин для ΔN-комплекса). Выращивают их при 37 °C в встряхивающем инкубаторе (220 об/мин) до тех пор, пока оптическая плотность (OD) бульона не достигнет 0,7-0,9.
- Добавьте 1 мМ изопропил β-d-1-тиогалактопиранозид (IPTG) для индукции экспрессии белка и инкубируйте клетки в течение 3-4 ч при 37 ° C в встряхивающем инкубаторе (220 об/мин).
- Гранулируют клетки центрифугированием культуры при 4 500 × г в течение 15 мин при 4 ° C.
- Подготовьте буферы для очистки белка (см. табл. 2).
- Суспендировать клеточные гранулы, экспрессирующие SNARE, в 40 мл ледяного буфера лизиса и лизировать клетки ультразвуком на льду (амплитуда 15%, 5 с включена и 5 с выключена, всего 30 минут).
- Центрифугируют лизат при 15 000 × г в течение 30 мин при 4 °C для удаления нерастворимых материалов.
- Пропустите надосадочную жидкость через гравитационную колонну, заполненную 1 мл смолы Ni-NTA. Промойте смолу промывочным буфером A, затем промывочным буфером B и разбавьте белки 10 мл элюирующего буфера.
- Удалите трис (2-карбоксиэтил) фосфин (TCEP) и имидазол из элюента с помощью обессоливающей колонны (следуйте инструкциям производителя). Разбавьте образец PBS.
- Сконцентрируйте белки с помощью центробежных фильтров (отсечка 10 кДа) до ~ 70 мкМ, сохраняя при этом белки в PBS (обычно с получением 2 мл). Измерьте концентрацию белка либо с помощью ультрафиолетового (УФ) поглощения на длине волны 280 нм (A280), либо с помощью анализа Брэдфорда.
- Приготовьте небольшие аликвоты, заморозьте в жидком азоте и храните при температуре -80 °C до использования.
ПРИМЕЧАНИЕ: Полные комплексы SNARE будут собраны после конъюгации ΔN-комплекса на ручке ДНК (см. ниже).
5. Прикрепление дескрипторов ДНК
ПРИМЕЧАНИЕ: Две ручки дцДНК 510.н., содержащие первичные аминогруппы на одном конце, сначала подготавливают с помощью ПЦР, а затем аминогруппы превращают в малеимидные группы с помощью бифункционального сшивающего агента SM (PEG) 2. Затем две ручки ковалентно связываются с комплексами SNARE через их цистеиновые группы для сайт-специфической конъюгации (рис. 3B).
- Подготовьте грунтовки.
- Подготовьте прямые праймеры: праймер B (для амплификации ручки B), который мечен 5'-биотином для прикрепления к поверхности стекла и связывается с λ-ДНК; Праймер Z (для усиления рукоятки Z), помеченный 5'-азидом для крепления магнитных шариков и имеющий ту же последовательность, что и праймер B.
- Подготовьте обратный праймер: праймер N (общий для ручки B и ручки Z), который мечен 5'-амином для конъюгации белка и связывается с λ-ДНК на расстоянии 510.н. от прямого праймера.
- Настройте и запустите два набора ПЦР-реакций (18 пробирок по 200 мкл для каждой ручки) с λ-ДНК (матрицей), nTaq-полимеразой и стандартными условиями ПЦР (см. Таблицу 1). Очистите продукт с помощью набора для очистки ПЦР и промойте каждую ручку 45 мкл сверхчистой воды. Используйте минимальный объем воды, чтобы получить высокую концентрацию ручек для эффективной реакции на последующих этапах.
- Измерьте концентрацию ДНК с помощью A260. Типичный выход составляет ~ 650 мкл раствора ~ 2 мкМ для каждой ручки. Держите небольшие образцы отдельно друг от друга для последующей проверки при электрофорезе в агарозном геле.
- Реагируйте на каждую рукоятку (1 мкМ в PBS) с 5 мМ SM(PEG)2. Инкубировать при комнатной температуре при аккуратном вращении. Через 1 ч используйте набор для очистки ДНК для удаления непрореагировавшего SM(PEG)2. Облейте каждую ручку 250 мкл PBS для получения ~ 2 мкМ растворов.
- Смешайте растворы Handle B и ΔN-комплекса в молярном соотношении 1:16 (например, 1 мкМ Handle B и 16 мкМ ΔN-комплекса) в PBS и инкубируйте в течение 2 ч при комнатной температуре с перемешиванием. Держите отдельно небольшой образец для электрофореза в агарозном геле.
- Добавьте раствор VAMP2 в 2,5-кратном молярном избытке над ΔN-комплексом, используемым на предыдущем этапе. Выдерживают смесь еще 1 ч при комнатной температуре при перемешивании. На этом этапе собираются полные комплексы SNARE.
- Удалите свободные белки путем буферного обмена со свежим PBS и центробежным фильтром (отсечка 100 кДа): центрифуга при 14 000 × г в течение 5 мин при 4 °C, повторить не менее 6 раз и запустить в течение 15 мин для последнего отжима. Измерьте увеличение соотношения A260/A280, чтобы контролировать удаление свободных белков. Держите отдельно небольшой образец для электрофореза в агарозном геле.
- Добавьте ручку Z в раствор в 15-кратном молярном избытке над ручкой B. Поддерживайте концентрацию ручки Z по крайней мере выше 1 мкМ, чтобы облегчить реакцию. Инкубируйте смесь в течение ночи при температуре 4 °C при перемешивании.
- Проверьте промежуточные продукты (ручка B и ее белковые конъюгаты) и конечный продукт (комплекс SNARE с двумя ручками) с помощью электрофореза в агарозном геле (рис. 3B, вставка) (см. Таблицу 2).
ПРИМЕЧАНИЕ: Если белки успешно прикреплены к ручке B, будет обнаружен сдвиг подвижности. В частности, образование полных комплексов SNARE на ручках ДНК может быть подтверждено их устойчивостью к додецилсульфату натрия (SDS), в отличие от ΔN-комплексов, которые разбираются в SDS и оставляют только синтаксин, связанный с ДНК (сравните b и c на рисунке 3B). - Приготовьте небольшие аликвоты, заморозьте в жидком азоте и храните при температуре -80 °C до использования.
ПРИМЕЧАНИЕ: Несмотря на то, что окончательный раствор содержит непрореагировавшие ручки, во время сборки образца в проточной ячейке будет выбрана только искомая конструкция, дважды меченная биотином и азидом.
6. Изготовление проточных ячеек
ПРИМЕЧАНИЕ: Проточные ячейки для измерений МТ изготовлены из двух стеклянных покровных стекол, соединенных между собой двусторонним скотчем (рис. 3C). Одно покровное стекло покрыто смесью ПЭГ и биотинилированного полиэтиленгликоля (ПЭГ), чтобы избежать неспецифического связывания и обеспечить специфическую привязку молекул-мишеней через связь биотин-нейтравидин (рис. 3D). Затем растворы материалов для экспериментов МТ последовательно вливаются в проточную ячейку с помощью шприцевого насоса (рис. 3C, D).
- Подготовьте два стеклянных покровных стекла, по одному для верхней (24 мм × 50 мм, толщина No 1,5) и нижней (24 мм × 60 мм, толщина No 1,5) поверхности. Очистите покровные стекла ультразвуком в 1 м КОН в течение 30 минут. После обработки ультразвуком промойте покровные стекла дистиллированной водой и держите в воде до следующего шага.
- ПЭГилатировать нижнее покровное стекло в соответствии с опубликованными протоколами42,43. Используйте N- [3- (триметоксисилил) пропил] этилендиамин для силанизации и смесь биотина-ПЭГ-SVA и mPEG-SVA 1:100 (ww) в 100 мМ бикарбонатном буфере. Держите ПЭГилированные покровные стекла сухими при температуре -20 °C и храните их в течение нескольких недель.
- В день экспериментов выньте ПЭГилированные покровные стекла и высушите их феном с помощью азотного пистолета. Визуально осмотрите их на наличие грязи, чтобы убедиться, что они чистые.
- Чтобы сделать каналы для образцов, подготовьте полоски двустороннего скотча шириной ~2 мм и положите четыре полоски на нижнее покровное стекло (ПЭГилированная поверхность вверх), параллельные друг другу и отделенные друг от друга ~5 мм (рис. 3C).
ПРИМЕЧАНИЕ: Таким образом, в одной проточной ячейке можно создать три канала для образцов шириной 5 мм. - Поместите верхнее защитное стекло в центр нижнего покровного стекла, оставив ~ 5 мм пространства на коротких краях для входных и выходных отверстий каналов. Аккуратно прижмите пинцетом заднюю часть верхнего покровного стекла, чтобы надежно закрыть каналы.
- Чтобы сделать впускной резервуар, обрежьте край наконечника пипетки объемом 200 мкл. Вырежьте ~ 10 мм из более широкого отверстия, чтобы обеспечить удержание ~ 200 мкл раствора. Сделайте три из них для трех каналов потока. Чтобы настроить выпускные отверстия, подготовьте три иглы для шприца, которые подходят к трубке для шприцевого насоса.
- Используя 5-минутную эпоксидную смолу, приклейте резервуары и игольчатые ступицы к проточной ячейке. Убедитесь, что сформировано полное уплотнение, чтобы избежать утечки, и что каналы не заблокированы излишками клея. Дайте ему высохнуть не менее 30 минут.
7. Сборка бортовых тросовых конструкций
ПРИМЕЧАНИЕ: Растворы материалов для экспериментов МТ, в том числе для конструкций бортовых тросов, последовательно вводятся в проточные ячейки с помощью шприцевого насоса (рис. 3C, D).
- Подготовьте магнитные бусины. Возьмите 5 мг шариков M270-эпоксидной смолы из исходного раствора (~ 3,3 × 10 шариков8 в 167,5 мкл диметилформамида) и замените растворитель фосфатным буфером (см. Таблицу 2) путем магнитной сепарации шариков.
- Подготовьте шарики при ~ 1,1 × 109 шариках мл-1 в фосфатном буфере с 1 М сульфатом аммония и вступите в реакцию с 2 мМ дибензоциклооктина (DBCO)-NH2. Выдерживают смесь в течение 3 ч на вращающемся миксере при комнатной температуре. После реакции промойте шарики 3 раза свежим фосфатным буфером, чтобы удалить непрореагировавшие молекулы.
ПРИМЕЧАНИЕ: Вымытые шарики можно хранить без дополнительного вращения при температуре 4 °C в течение нескольких недель перед использованием. - Подсоедините иглу на выходе проточного канала к шприцевому насосу с помощью полиэтиленовой трубки. Уравновесьте каналы с помощью PBS.
- Последовательно вводите в канал путем всасывания с помощью насоса следующие растворы: NeutrAvidin, целевые конструкции (шпильки ДНК или комплексы SNARE с ручками ДНК), эталонные полистирольные шарики и магнитные шарики с покрытием DBCO. Перед использованием тщательно перемешайте растворы шариков, чтобы диспергировать потенциальные агрегаты шариков.
- Смойте несвязанные шарики, приложив 0,1 пН силы.
ПРИМЕЧАНИЕ: Приложение небольшой силы, направленной вверх, облегчает удаление несвязанных шариков и помогает избежать разрыва специально связанных конструкций бортового троса. - Для экспериментов с комплексами SNARE включите 1,5 мкМ SNAP-25 в конечный буфер.
ПРИМЕЧАНИЕ: Свободные молекулы SNAP-25 могут повторно связывать комплексы SNARE после развертывания и позволяют проводить повторные измерения на одном комплексе.
8. Идентификация целевых конструкций
- На поверхности канала проточной ячейки найдите магнитные шарики, которые привязаны к одиночным молекулам целевой конструкции. Убедитесь, что рядом находится эталонная бусина.
- Поверните бусину-кандидата и убедитесь, что она свободно вращается. Если шарик привязан к нескольким молекулам, он проявляет ограниченное движение.
- Вращайте бусину на несколько полных оборотов и узнайте радиус вращения (эта функция реализована в предусмотренном программном обеспечении). Желательно выбирать бусину с небольшим радиусом вращения.
ПРИМЕЧАНИЕ: Этот радиус показывает, насколько валик смещен от центра оси троса, что случайным образом определяется при сборке борт-трос30,31. Во всех экспериментах минимальное смещение бусины по центру смягчает многие артефакты, связанные с большим отношением радиуса валика к удлинению троса, которое мы используем. - Увеличьте усилие от 0 до 5 пН, чтобы определить хорошие бусины с одной привязью. Обратите внимание на большое изменение дифракционной картины бусины, возникающее в результате растяжения троса со скоростью 1 кбит/с (или эквивалентных двух ручек с частотой 510.н.). Если дифракционная картина существенно не изменится, уменьшите силу до нуля и найдите другой валик-кандидат.
ПРИМЕЧАНИЕ: Подъем бусины ~ 300 нм можно легко заметить по необработанным изображениям без фактического запуска процесса отслеживания.
9. Отслеживание борта для измерений удлинения
ПРИМЕЧАНИЕ: Отслеживание бусин осуществляется путем анализа изображений бусин в режиме реального времени в программном обеспечении LabVIEW, поставляемом с этой статьей. Метод слежения и его варианты использовались в большинстве традиционных систем МТ и объяснялись в предыдущей литературе 2,5,7,26. Измеряя положение магнитного шарика относительно неподвижного эталонного валика (т. е. дифференциальное отслеживание), измерения положения становятся чрезвычайно устойчивыми к внешнему возмущению.
- После того, как подходящая магнитная бусина будет расположена вместе с эталонной бусиной, нажмите кнопку «Калибровка », чтобы начать подготовку к отслеживанию бусин.
- Нажмите на бусины на изображении, чтобы определить расположение бусин. Затем изображения будут обрезаны до областей интереса (ROI) (например, 150 x 150 пикселей для бусины 3 мкм) вокруг бусин, а затем дополнительно проанализированы для извлечения точных координат бусин.
- Дождитесь завершения вращения магнита. Этот процесс записывает x- и y-координаты валика (путем вычисления 2D-перекрестной корреляции44 или с использованием радиальной симметрии45 изображений валика с сопоставимой производительностью) при вращении магнитов для документирования смещенного от центра прикреплениявалика 31.
- Для слежения в направлении z дождитесь, пока программа сгенерирует справочную таблицу дифракционных изображений бусин на разных расстояниях от фокальной плоскости. Это осуществляется путем ступенчатого перемещения линзы объектива с помощью пьезосканера с равноудаленными шагами и записи усредненных по флуктуациям изображений шариков в каждом положении. Затем z-координаты бусин в реальных экспериментах определяются путем сравнения изображений бусин в реальном времени с таблицей поиска с интерполяцией7.
- Когда генерация справочных таблиц будет завершена, включите слежение и автофокусировку (нажмите кнопки Track ? и AF? Buttons) и нажмите кнопку «Получить », чтобы начать запись положения бусин.
ПРИМЕЧАНИЕ: Автофокусировка не является обязательной, но рекомендуется для коррекции дрейфа ступени в z во время съемки.
10. Схемы принудительного применения
- Эксперименты с наращиванием силы: Чтобы проверить соотношение силы и удлинения конструкции, примените увеличение силы вверх и вниз с постоянной скоростью нагрузки (± 1,0 пН с-1) (рис. 4A). Например, примените три раунда цикла 0-20-0 pN, чтобы проверить общую длину конструкции и кривую разгибания усилия ручек.
- Указав параметры троса в программном обеспечении, наложите кривую силового удлинения WLC поверх измеренных данных и определите, привязан ли целевой шарик подлинной конструкцией образца с правильными ручками ДНК. В качестве отправной точки используйте известную длину контура (например, ~340 нм для дцДНК 1 кбит/с) и длину персистенции WLC (30-45 нм для короткой дцДНК31). При необходимости примените метод коррекции расширения, описанный в шаге 2.11.
- Если конструкция проверена, подробно изучите реакцию силы-расширения, чтобы найти дополнительное расширение, возникающее в результате молекул-мишеней-шпилек или комплексов SNARE.
- Эксперименты с постоянной силой: Постепенно изменяйте приложенную силу дискретными шагами, чтобы исследовать силовую чувствительность молекул-мишеней (рис. 4B).
ПРИМЕЧАНИЕ: МТ позволяют проводить простые и эффективные эксперименты с постоянной силой, потому что приложенная сила поддерживается постоянной, когда магниты удерживаются неподвижно.- Для шпилек ДНК приложите усилие 4-8 пН с шагом 0,2-0,5 пН и измерьте положение шарика в течение ~ 10 с на каждом уровне силы.
- Для комплексов SNARE приложите усилие 14-16 пН с шагом 0,1-0,2 пН и измерьте положение валика в течение ~10 с на каждом уровне силы.
- Эксперименты с силовыми прыжками: Наблюдайте за переходными событиями комплексов SNARE.
ПРИМЕЧАНИЕ: Эксперименты с силовыми скачками, как и эксперименты с постоянной силой, включают изменения уровней силы. Однако силовые скачки используют более резкие изменения приложенной силы, что позволяет отслеживать события, вызванные силой в исследуемых молекулах, такие как внезапный разрыв белковых комплексов. Например, поскольку комплексы SNARE проявляют структурный гистерезис при циклическом движениисилы 23, информативно проводить эксперименты с силовыми скачками и измерять задержку перехода (рис. 4C).- Распаковки: Отслаивание молекулы VAMP2 от интактного тройного комплекса SNARE, оставляя бинарный комплекс синтаксина-1A и SNAP-25.
- Повторное застежка: Застежка-молния распакованной молекулы VAMP2 для регенерации интактного комплекса SNARE.
- Разворачивается: Полная разборка комплекса SNARE сопровождается полной диссоциацией SNAP-25. Только молекулы VAMP2 и синтаксина остаются в конструкции после развертывания.
- Складывание: Регенерация комплекса SNARE при связывании свободной молекулы SNAP-25 с буфера.
- При 2 pN индуцируют сборку интактного комплекса SNARE, ожидая (~ 30 с) ассоциации свободной молекулы SNAP25. Внезапное уменьшение протяженности наблюдается при образовании комплекса SNARE.
- Чтобы наблюдать за событиями распаковки, подождите несколько секунд при 10-12 пН, а затем резко переходите к 14-15 пН с максимально возможной скоростью двигателя. В зависимости от целевой силы комплекс SNARE будет демонстрировать либо обратимый переход между частично распакованными промежуточными продуктами (как в экспериментах с постоянной силой), либо скачок ~ 25 нм в более высокое, разархивированное состояние после случайного времени ожидания (или задержки).
- Чтобы наблюдать за событиями повторного застеживания, уменьшите усилие до 10-12 пН сразу после того, как будет наблюдаться расстегивание. Опять же, комплекс SNARE демонстрирует стохастический переход в нижнее состояние с застежкой-молнией после некоторой случайной задержки. Если разворачивание произошло после распаковки, комплекс не сможет застегнуться повторно, так как молекула SNAP-25 будет отсутствовать.
- Чтобы наблюдать за разворачивающимися событиями, подождите более длительный период после того, как будет наблюдаться распаковка, чтобы обнаружить дальнейшее увеличение расширения (~ 2 нм).
11. Анализ данных
ПРИМЕЧАНИЕ: Типы анализа, который можно проводить с данными МТ, зависят от целевой системы. Тем не менее, существуют общие подходы к извлечению полезной информации из соответствующих экспериментов, описанных на рисунке 4. Все анализы выполняются с помощью MATLAB (R2021a) с использованием пользовательских кодов, предоставленных в этой статье. Эти коды генерируют графики, используя те же данные, что и в этой статье. Обратите внимание, что в то время как необработанные данные отслеживания 100 Гц были взяты непосредственно для анализа, данные отслеживания 1,2 кГц обычно подвергались медианной фильтрации (с пятиточечным скользящим окном) перед анализом для уменьшения шума (за исключением анализа шума).
- Эксперименты с наращиванием силы: Проанализируйте соотношение силы и растяжения (например, упругость полимеров) и переведите силу для извлечения информации о наномеханических свойствах.
- Эксперименты с постоянной силой: Проанализируйте популяции состояний и время пребывания (или скорость перехода) в зависимости от силы для извлечения структурных (например, областей, участвующих в переходе), термодинамических (например, разность свободных энергий) и кинетических (например, энергетический барьер) параметров конформационных изменений.
- Эксперименты с силовыми прыжками: Проанализируйте кинетику разрыва (например, белок-белковые взаимодействия и связывание рецептор-лиганд) или время жизни переходных промежуточных продуктов (например, разворачивание биомолекул) для извлечения стабильности молекул-мишеней и их состояний.
- В качестве репрезентативных приложений проанализируйте данные образцов для шпилек ДНК и комплексов SNARE:
- Переходы шпильки ДНК с двумя состояниями: сила расстегивания, расстояние открытия, силовая зависимость сдвига популяции, а также измерения назначения состояний и скорости перехода с помощью скрытой марковской модели (HMM) (предоставлены коды MATLAB).
- Конформационные изменения комплексов SNARE: сила распаковки, силовая зависимость промежуточных состояний и латентности распаковки, гистерезис при повторном застежке и поведение разворачивания/сворачивания.
ПРИМЕЧАНИЕ: Модели расширения силы для ручек ДНК, шпилек ДНК и конформаций комплекса SNARE приведены в предыдущих ссылках14,31.
Результаты
Калибровка по усилию
Результаты двух методов измерения силы (дисперсия бокового смещения шариков и анализ спектра мощности) отличались на 0-2 пН (рис. 2G). Согласно результатам, приведенным на рисунке 2F, мы можем надежно достигать до 30 пН с помощь?...
Обсуждение
В этой работе мы представили установку одномолекулярной силовой спектроскопии, которая может наблюдать структурные изменения биомолекул с высокой пространственно-временной точностью. Используемая высокоскоростная CMOS-камера снимает 1 200 кадров с-1 с разрешением 1 280 x 1 024, что позв...
Раскрытие информации
У авторов нет конфликтов интересов, о которых можно было бы заявить.
Благодарности
Эта работа была поддержана грантом Национального исследовательского фонда Кореи (NRF), финансируемым правительством Кореи (MSIT) (NRF-2022R1C1C1012176, NRF-2021R1A4A1031754 и NRF-2021R1A6A1A10042944). S.-H.R. была поддержана грантом NRF (2021R1C1C2009717).
Материалы
| Name | Company | Catalog Number | Comments |
| Materials for construct synthesis | |||
| Agarose gel electrophoresis system | Advance | Mupid-2plus | |
| DNA ladder | Bioneer | D-1037 | |
| nTaq polymerase | Enzynomics | P050A | |
| PCR purification kit | LaboPass | CMR0112 | |
| PEGylated SMCC crosslinker / SM(PEG)2 | ThermoFisher Scientific | 22102 | For SNARE–DNA coupling |
| Primer B | Bioneer | 5'-Biotin/TCGCCACCATCATTTCCA-3' | For 5-kbp force calibration construct and DNA handles |
| Primer B_hp | IDT | 5'-Biotin/TTTTTTTTTTGTTCTCTATTT TTTTAGAGAAC /AP site/ /AP site/ TCGCCACCATCATTTCCA-3' | For hairpin construct |
| Primer N | Bioneer | 5'-C6Amine/CATGTGGGTGACGCGAAA-3' | For DNA handles |
| Primer Z | Bioneer | 5'-Azide/TCGCCACCATCATTTCCA-3' | For DNA handles |
| Primer Z_5k | Bioneer | 5'-Azide/TTAGAGAGTATGGGTATATGACA TCG-3' | For 5-kbp force calibration construct |
| Primer Z_hp | Bioneer | 5'-Azide/GTGGCAGCATGACACC-3' | For hairpin construct |
| SYBR Safe DNA Gel Stain | ThermoFisher Scientific | S33102 | |
| λ-DNA | Bioneer | D-2510 | Template strand for PCR |
| DNA sequences for SNARE proteins | |||
| 6×His-tagged SNAP-25b (2-206; capitalized) in pET28a | homemade | tggcgaatgggacgcgccctgtagcggcgca ttaagcgcggcgggtgtggtggttacgcgca gcgtgaccgctacacttgccagcgccctagc gcccgctcctttcgctttcttcccttccttt ctcgccacgttcgccggctttccccgtcaag ctctaaatcgggggctccctttagggttccg atttagtgctttacggcacctcgaccccaaa aaacttgattagggtgatggttcacgtagtg ggccatcgccctgatagacggtttttcgccc tttgacgttggagtccacgttctttaatagt ggactcttgttccaaactggaacaacactca accctatctcggtctattcttttgatttata agggattttgccgatttcggcctattggtta aaaaatgagctgatttaacaaaaatttaacg cgaattttaacaaaatattaacgtttacaat ttcaggtggcacttttcggggaaatgtgcgc ggaacccctatttgtttatttttctaaatac attcaaatatgtatccgctcatgaattaatt cttagaaaaactcatcgagcatcaaatgaaa ctgcaatttattcatatcaggattatcaata ccatatttttgaaaaagccgtttctgtaatg aaggagaaaactcaccgaggcagttccatag gatggcaagatcctggtatcggtctgcgatt ccgactcgtccaacatcaatacaacctatta atttcccctcgtcaaaaataaggttatcaag tgagaaatcaccatgagtgacgactgaatcc ggtgagaatggcaaaagtttatgcatttctt tccagacttgttcaacaggccagccattacg ctcgtcatcaaaatcactcgcatcaaccaaa ccgttattcattcgtgattgcgcctgagcga gacgaaatacgcgatcgctgttaaaaggaca attacaaacaggaatcgaatgcaaccggcgc aggaacactgccagcgcatcaacaatatttt cacctgaatcaggatattcttctaatacctg gaatgctgttttcccggggatcgcagtggtg agtaaccatgcatcatcaggagtacggataa aatgcttgatggtcggaagaggcataaattc cgtcagccagtttagtctgaccatctcatct gtaacatcattggcaacgctacctttgccat gtttcagaaacaactctggcgcatcgggctt cccatacaatcgatagattgtcgcacctgat tgcccgacattatcgcgagcccatttatacc catataaatcagcatccatgttggaatttaa tcgcggcctagagcaagacgtttcccgttga atatggctcataacaccccttgtattactgt ttatgtaagcagacagttttattgttcatga ccaaaatcccttaacgtgagttttcgttcca ctgagcgtcagaccccgtagaaaagatcaaa ggatcttcttgagatcctttttttctgcgcg taatctgctgcttgcaaacaaaaaaaccacc gctaccagcggtggtttgtttgccggatcaa gagctaccaactctttttccgaaggtaactg gcttcagcagagcgcagataccaaatactgt ccttctagtgtagccgtagttaggccaccac ttcaagaactctgtagcaccgcctacatacc tcgctctgctaatcctgttaccagtggctgc tgccagtggcgataagtcgtgtcttaccggg ttggactcaagacgatagttaccggataagg cgcagcggtcgggctgaacggggggttcgtg cacacagcccagcttggagcgaacgacctac accgaactgagatacctacagcgtgagctat gagaaagcgccacgcttcccgaagggagaaa ggcggacaggtatccggtaagcggcagggtc ggaacaggagagcgcacgagggagcttcca gggggaaacgcctggtatctttatagtcctgt cgggtttcgccacctctgacttgagcgtcga tttttgtgatgctcgtcaggggggcggagcc tatggaaaaacgccagcaacgcggccttttt acggttcctggccttttgctggccttttgct cacatgttctttcctgcgttatcccctgatt ctgtggataaccgtattaccgcctttgagtg agctgataccgctcgccgcagccgaacgacc gagcgcagcgagtcagtgagcgaggaagcgg aagagcgcctgatgcggtattttctccttac gcatctgtgcggtatttcacaccgcatatat ggtgcactctcagtacaatctgctctgatgc cgcatagttaagccagtatacactccgctat cgctacgtgactgggtcatggctgcgccccg acacccgccaacacccgctgacgcgccctga cgggcttgtctgctcccggcatccgcttaca gacaagctgtgaccgtctccgggagctgcat gtgtcagaggttttcaccgtcatcaccgaaa cgcgcgaggcagctgcggtaaagctcatcag cgtggtcgtgaagcgattcacagatgtctgc ctgttcatccgcgtccagctcgttgagtttc tccagaagcgttaatgtctggcttctgataa agcgggccatgttaagggcggttttttcctg tttggtcactgatgcctccgtgtaaggggga tttctgttcatgggggtaatgataccgatga aacgagagaggatgctcacgatacgggttac tgatgatgaacatgcccggttactggaacgt tgtgagggtaaacaactggcggtatggatgc ggcgggaccagagaaaaatcactcagggtc aatgccagcgcttcgttaatacagatgtaggt gttccacagggtagccagcagcatcctgcga tgcagatccggaacataatggtgcagggcgc tgacttccgcgtttccagactttacgaaaca cggaaaccgaagaccattcatgttgttgctc aggtcgcagacgttttgcagcagcagtcgct tcacgttcgctcgcgtatcggtgattcattc tgctaaccagtaaggcaaccccgccagccta gccgggtcctcaacgacaggagcacgatcat gcgcacccgtggggccgccatgccggcgata atggcctgcttctcgccgaaacgtttggtgg cgggaccagtgacgaaggcttgagcgagggc gtgcaagattccgaataccgcaagcgacagg ccgatcatcgtcgcgctccagcgaaagcggt cctcgccgaaaatgacccagagcgctgccgg cacctgtcctacgagttgcatgataaagaag acagtcataagtgcggcgacgatagtcatgc cccgcgcccaccggaaggagctgactgggtt gaaggctctcaagggcatcggtcgagatccc ggtgcctaatgagtgagctaacttacattaa ttgcgttgcgctcactgcccgctttccagtc gggaaacctgtcgtgccagctgcattaatga atcggccaacgcgcggggagaggcggtttgc gtattgggcgccagggtggtttttcttttca ccagtgagacgggcaacagctgattgccctt caccgcctggccctgagagagttgcagcaag cggtccacgctggtttgccccagcaggcgaa aatcctgtttgatggtggttaacggcgggat ataacatgagctgtcttcggtatcgtcgtat cccactaccgagatatccgcaccaacgcgca gcccggactcggtaatggcgcgcattgcgcc cagcgccatctgatcgttggcaaccagcatc gcagtgggaacgatgccctcattcagcattt gcatggtttgttgaaaaccggacatggcact ccagtcgccttcccgttccgctatcggctga atttgattgcgagtgagatatttatgccagc cagccagacgcagacgcgccgagacagaa cttaatgggcccgctaacagcgcgatttgctgg tgacccaatgcgaccagatgctccacgccca gtcgcgtaccgtcttcatgggagaaaataat actgttgatgggtgtctggtcagagacatca agaaataacgccggaacattagtgcaggcag cttccacagcaatggcatcctggtcatccag cggatagttaatgatcagcccactgacgcgt tgcgcgagaagattgtgcaccgccgctttac aggcttcgacgccgcttcgttctaccatcga caccaccacgctggcacccagttgatcggcg cgagatttaatcgccgcgacaatttgcgacg gcgcgtgcagggccagactggaggtggcaac gccaatcagcaacgactgtttgcccgccagt tgttgtgccacgcggttgggaatgtaattca gctccgccatcgccgcttccactttttcccg cgttttcgcagaaacgtggctggcctggttc accacgcgggaaacggtctgataagagacac cggcatactctgcgacatcgtataacgttac tggtttcacattcaccaccctgaattgactc tcttccgggcgctatcatgccataccgcgaa aggttttgcgccattcgatggtgtccgggat ctcgacgctctcccttatgcgactcctgcat taggaagcagcccagtagtaggttgaggccg ttgagcaccgccgccgcaaggaatggtgcat gcaaggagatggcgcccaacagtcccccggc cacggggcctgccaccatacccacgccgaaa caagcgctcatgagcccgaagtggcgagccc gatcttccccatcggtgatgtcggcgatata ggcgccagcaaccgcacctgtggcgccggtg atgccggccacgatgcgtccggcgtagagga tcgagatctcgatcccgcgaaattaatacga ctcactataggggaattgtgagcggataaca attcccctctagaaataattttgtttaactt taagaaggagatataccATGGGCAGC AGCCATCATCATCATCATCACA GCAGCGGCCTGGTGCCGCGC GGCAGCCATACTAGCGGAGAT ATCGCCGAGGACGCAGACAT GCGCAATGAGCTGGAGGAGA TGCAGAGGAGGGCTGACCAG CTGGCTGATGAGTCCCTGGA AAGCACCCGTCGCATGCTGC AGCTGGTTGAAGAGAGTAAA GATGCTGGCATCAGGACTTT GGTTATGTTGGATGAGCAAG GCGAACAACTGGAACGCATT GAGGAAGGGATGGACCAAAT CAATAAGGACATGAAAGAAG CAGAAAAGAATTTGACGGAC CTAGGAAAATTCGCCGGCCT TGCCGTGGCCCCCGCCAAC AAGCTTAAATCCAGTGATGC TTACAAAAAAGCCTGGGGC AATAATCAGGATGGAGTAGT GGCCAGCCAGCCTGCCCG TGTGGTGGATGAACGGGAG CAGATGGCCATCAGTGGTG GCTTCATCCGCAGGGTAAC AAATGATGCCCGGGAAAAT GAGATGGATGAGAACCTG GAGCAGGTGAGCGGCATC ATCGGAAACCTCCGCCAC ATGGCTCTAGACATGGGCA ATGAGATTGACACCCAGA ATCGCCAGATCGACAGGA TCATGGAGAAGGCTGATT CCAACAAAACCAGAATTG ATGAAGCCAACCAACGTG CAACAAAGATGCTGGGAA GTGGTTAAggatccgaattcgag ctccgtcgacaagcttgcggccgcactc gagcaccaccaccaccaccactgagat ccggctgctaacaaagcccgaaagga agctgagttggctgctgccaccgctgag caataactagcataaccccttggggcct ctaaacgggtcttgaggggttttttgctga aaggaggaactatatccggat | |
| 6×His-tagged VAMP2 (2-97, L32C/I97C; capitalized) in pET28a | homemade | tggcgaatgggacgcgccctgtagcggcgca ttaagcgcggcgggtgtggtggttacgcgca gcgtgaccgctacacttgccagcgccctagc gcccgctcctttcgctttcttcccttccttt ctcgccacgttcgccggctttccccgtcaag ctctaaatcgggggctccctttagggttccg atttagtgctttacggcacctcgaccccaaa aaacttgattagggtgatggttcacgtagtg ggccatcgccctgatagacggtttttcgccc tttgacgttggagtccacgttctttaatagt ggactcttgttccaaactggaacaacactca accctatctcggtctattcttttgatttata agggattttgccgatttcggcctattggtta aaaaatgagctgatttaacaaaaatttaacg cgaattttaacaaaatattaacgtttacaat ttcaggtggcacttttcggggaaatgtgcgc ggaacccctatttgtttatttttctaaatac attcaaatatgtatccgctcatgaattaatt cttagaaaaactcatcgagcatcaaatgaaa ctgcaatttattcatatcaggattatcaata ccatatttttgaaaaagccgtttctgtaatg aaggagaaaactcaccgaggcagttccatag gatggcaagatcctggtatcggtctgcgatt ccgactcgtccaacatcaatacaacctatta atttcccctcgtcaaaaataaggttatcaag tgagaaatcaccatgagtgacgactgaatcc ggtgagaatggcaaaagtttatgcatttctt tccagacttgttcaacaggccagccattacg ctcgtcatcaaaatcactcgcatcaaccaaa ccgttattcattcgtgattgcgcctgagcga gacgaaatacgcgatcgctgttaaaaggaca attacaaacaggaatcgaatgcaaccggcgc aggaacactgccagcgcatcaacaatatttt cacctgaatcaggatattcttctaatacctg gaatgctgttttcccggggatcgcagtggtg agtaaccatgcatcatcaggagtacggataa aatgcttgatggtcggaagaggcataaattc cgtcagccagtttagtctgaccatctcatct gtaacatcattggcaacgctacctttgccat gtttcagaaacaactctggcgcatcgggctt cccatacaatcgatagattgtcgcacctgat tgcccgacattatcgcgagcccatttatacc catataaatcagcatccatgttggaatttaa tcgcggcctagagcaagacgtttcccgttga atatggctcataacaccccttgtattactgt ttatgtaagcagacagttttattgttcatga ccaaaatcccttaacgtgagttttcgttcca ctgagcgtcagaccccgtagaaaagatcaaa ggatcttcttgagatcctttttttctgcgcg taatctgctgcttgcaaacaaaaaaaccacc gctaccagcggtggtttgtttgccggatcaa gagctaccaactctttttccgaaggtaactg gcttcagcagagcgcagataccaaatactgt ccttctagtgtagccgtagttaggccaccac ttcaagaactctgtagcaccgcctacatacc tcgctctgctaatcctgttaccagtggctgc tgccagtggcgataagtcgtgtcttaccggg ttggactcaagacgatagttaccggataagg cgcagcggtcgggctgaacggggggttcgtg cacacagcccagcttggagcgaacgacctac accgaactgagatacctacagcgtgagctatg agaaagcgccacgcttcccgaagggagaaa ggcggacaggtatccggtaagcggcagggtc ggaacaggagagcgcacgagggagcttcca gggggaaacgcctggtatctttatagtcctgt cgggtttcgccacctctgacttgagcgtcga tttttgtgatgctcgtcaggggggcggagcc tatggaaaaacgccagcaacgcggccttttt acggttcctggccttttgctggccttttgct cacatgttctttcctgcgttatcccctgatt ctgtggataaccgtattaccgcctttgagtg agctgataccgctcgccgcagccgaacgacc gagcgcagcgagtcagtgagcgaggaagc ggaagagcgcctgatgcggtattttctccttac gcatctgtgcggtatttcacaccgcatatat ggtgcactctcagtacaatctgctctgatgc cgcatagttaagccagtatacactccgctat cgctacgtgactgggtcatggctgcgccccg acacccgccaacacccgctgacgcgccctga cgggcttgtctgctcccggcatccgcttaca gacaagctgtgaccgtctccgggagctgcat gtgtcagaggttttcaccgtcatcaccgaaa cgcgcgaggcagctgcggtaaagctcatcag cgtggtcgtgaagcgattcacagatgtctgc ctgttcatccgcgtccagctcgttgagtttc tccagaagcgttaatgtctggcttctgataa agcgggccatgttaagggcggttttttcctg tttggtcactgatgcctccgtgtaaggggga tttctgttcatgggggtaatgataccgatga aacgagagaggatgctcacgatacgggttac tgatgatgaacatgcccggttactggaacgt tgtgagggtaaacaactggcggtatggatgc ggcgggaccagagaaaaatcactcagggtc aatgccagcgcttcgttaatacagatgtaggt gttccacagggtagccagcagcatcctgcga tgcagatccggaacataatggtgcagggcgc tgacttccgcgtttccagactttacgaaaca cggaaaccgaagaccattcatgttgttgctc aggtcgcagacgttttgcagcagcagtcgct tcacgttcgctcgcgtatcggtgattcattc tgctaaccagtaaggcaaccccgccagccta gccgggtcctcaacgacaggagcacgatcat gcgcacccgtggggccgccatgccggcgata atggcctgcttctcgccgaaacgtttggtgg cgggaccagtgacgaaggcttgagcgagggc gtgcaagattccgaataccgcaagcgacagg ccgatcatcgtcgcgctccagcgaaagcggt cctcgccgaaaatgacccagagcgctgccgg cacctgtcctacgagttgcatgataaagaag acagtcataagtgcggcgacgatagtcatgc cccgcgcccaccggaaggagctgactgggtt gaaggctctcaagggcatcggtcgagatccc ggtgcctaatgagtgagctaacttacattaa ttgcgttgcgctcactgcccgctttccagtc gggaaacctgtcgtgccagctgcattaatga atcggccaacgcgcggggagaggcggtttgc gtattgggcgccagggtggtttttcttttca ccagtgagacgggcaacagctgattgccctt caccgcctggccctgagagagttgcagcaag cggtccacgctggtttgccccagcaggcgaa aatcctgtttgatggtggttaacggcgggat ataacatgagctgtcttcggtatcgtcgtat cccactaccgagatatccgcaccaacgcgca gcccggactcggtaatggcgcgcattgcgcc cagcgccatctgatcgttggcaaccagcatc gcagtgggaacgatgccctcattcagcattt gcatggtttgttgaaaaccggacatggcact ccagtcgccttcccgttccgctatcggctga atttgattgcgagtgagatatttatgccagc cagccagacgcagacgcgccgagacagaa cttaatgggcccgctaacagcgcgatttgctgg tgacccaatgcgaccagatgctccacgccca gtcgcgtaccgtcttcatgggagaaaataat actgttgatgggtgtctggtcagagacatca agaaataacgccggaacattagtgcaggcag cttccacagcaatggcatcctggtcatccag cggatagttaatgatcagcccactgacgcgt tgcgcgagaagattgtgcaccgccgctttac aggcttcgacgccgcttcgttctaccatcga caccaccacgctggcacccagttgatcggcg cgagatttaatcgccgcgacaatttgcgacg gcgcgtgcagggccagactggaggtggcaac gccaatcagcaacgactgtttgcccgccagt tgttgtgccacgcggttgggaatgtaattca gctccgccatcgccgcttccactttttcccg cgttttcgcagaaacgtggctggcctggttc accacgcgggaaacggtctgataagagacac cggcatactctgcgacatcgtataacgttac tggtttcacattcaccaccctgaattgactc tcttccgggcgctatcatgccataccgcgaa aggttttgcgccattcgatggtgtccgggat ctcgacgctctcccttatgcgactcctgcat taggaagcagcccagtagtaggttgaggccg ttgagcaccgccgccgcaaggaatggtgcat gcaaggagatggcgcccaacagtcccccggc cacggggcctgccaccatacccacgccgaaa caagcgctcatgagcccgaagtggcgagccc gatcttccccatcggtgatgtcggcgatata ggcgccagcaaccgcacctgtggcgccggtg atgccggccacgatgcgtccggcgtagagga tcgagatctcgatcccgcgaaattaatacga ctcactataggggaattgtgagcggataaca attcccctctagaaataattttgtttaactt taagaaggagatataccATGGGCAGC AGCCATCATCATCATCATCAC AGCAGCGGCCTGGTGCCGC GCGGCAGCCATATGGCAGAT CTCTCGGCTACCGCTGCCAC CGTCCCGCCTGCCGCCCCG GCCGGCGAGGGTGGCCCCC CTGCACCTCCTCCAAATCTTA CCAGTAACAGGAGATGCCAG CAGACCCAGGCCCAGGTGG ATGAGGTGGTGGACATCATG AGGGTGAATGTGGACAAGGT CCTGGAGCGAGACCAGAAG CTATCGGAACTGGATGATCG CGCAGATGCCCTCCAGGCA GGGGCCTCCCAGTTTGAAA CAAGTGCAGCCAAGCTCAA GCGCAAATACTGGTGGAAA AACCTCAAGATGATGTGCTA Aggatccgaattcgagctccgtcg acaagcttgcggccgcactcgagcaccacca ccaccaccactgagatccggctgctaacaaa gcccgaaaggaagctgagttggctgctgcca ccgctgagcaataactagcataaccccttgg ggcctctaaacgggtcttgaggggttttttg ctgaaaggaggaactatatccggat | |
| 6×His-tagged ΔN-VAMP2 (49–96; capitalized) and Syntaxin-1A (191–267, I202C/I266C; capitalized) in pETDuet-1 | homemade | ggggaattgtgagcggataacaattcccctc tagaaataattttgtttaactttaagaagga gatataccATGGGCAGCAGCCATCA TCATCATCATCACAGCAGCGG CCTGGAAGTTCTGTTCCAGGG GCCCGGTAATGTGGACAAGGT CCTGGAGCGAGACCAGAAGCT ATCGGAACTGGATGATCGCGC AGATGCCCTCCAGGCAGGGGC CTCCCAGTTTGAAACAAGTGC AGCCAAGCTCAAGCGCAAATAC TGGTGGAAAAACCTCAAGATGAT GTAAgcggccgcataatgcttaagtcgaaca gaaagtaatcgtattgtacacggccgcataa tcgaaattaatacgactcactataggggaat tgtgagcggataacaattccccatcttagta tattagttaagtataagaaggagatatacat ATGGCCCTCAGTGAGATCGAGA CCAGGCACAGTGAGTGCATC AAGTTGGAGAACAGCATCCG GGAGCTACACGATATGTTCAT GGACATGGCCATGCTGGTGG AGAGCCAGGGGGAGATGATT GACAGGATCGAGTACAATGTG GAACACGCTGTGGACTACGTG GAGAGGGCCGTGTCTGACACC AAGAAGGCCGTCAAGTACCAG AGCAAGGCACGCAGGAAGAA GTGCATGATCTAActcgagtc tggtaaagaaaccgctgctgcgaaatttgaa cgccagcacatggactcgtctactagcgcag cttaattaacctaggctgctgccaccgctga gcaataactagcataaccccttggggcctct aaacgggtcttgaggggttttttgctgaaag gaggaactatatccggattggcgaatgggac gcgccctgtagcggcgcattaagcgcggcgg gtgtggtggttacgcgcagcgtgaccgctac acttgccagcgccctagcgcccgctcctttc gctttcttcccttcctttctcgccacgttcg ccggctttccccgtcaagctctaaatcgggg gctccctttagggttccgatttagtgcttta cggcacctcgaccccaaaaaacttgattagg gtgatggttcacgtagtgggccatcgccctg atagacggtttttcgccctttgacgttggag tccacgttctttaatagtggactcttgttcc aaactggaacaacactcaaccctatctcggt ctattcttttgatttataagggattttgccg atttcggcctattggttaaaaaatgagctga tttaacaaaaatttaacgcgaattttaacaa aatattaacgtttacaatttctggcggcacg atggcatgagattatcaaaaaggatcttcac ctagatccttttaaattaaaaatgaagtttt aaatcaatctaaagtatatatgagtaaactt ggtctgacagttaccaatgcttaatcagtga ggcacctatctcagcgatctgtctatttcgt tcatccatagttgcctgactccccgtcgtgt agataactacgatacgggagggcttaccatc tggccccagtgctgcaatgataccgcgagac ccacgctcaccggctccagatttatcagcaa taaaccagccagccggaagggccgagcgca gaagtggtcctgcaactttatccgcctccatc cagtctattaattgttgccgggaagctagag taagtagttcgccagttaatagtttgcgcaa cgttgttgccattgctacaggcatcgtggtg tcacgctcgtcgtttggtatggcttcattca gctccggttcccaacgatcaaggcgagttac atgatcccccatgttgtgcaaaaaagcggtt agctccttcggtcctccgatcgttgtcagaa gtaagttggccgcagtgttatcactcatggt tatggcagcactgcataattctcttactgtc atgccatccgtaagatgcttttctgtgactg gtgagtactcaaccaagtcattctgagaata gtgtatgcggcgaccgagttgctcttgcccg gcgtcaatacgggataataccgcgccacata gcagaactttaaaagtgctcatcattggaaa acgttcttcggggcgaaaactctcaaggatc ttaccgctgttgagatccagttcgatgtaac ccactcgtgcacccaactgatcttcagcatc ttttactttcaccagcgtttctgggtgagcaaa aacaggaaggcaaaatgccgcaaaaaagg gaataagggcgacacggaaatgttgaatact catactcttcctttttcaatcatgattgaag catttatcagggttattgtctcatgagcgga tacatatttgaatgtatttagaaaaataaac aaataggtcatgaccaaaatcccttaacgtg agttttcgttccactgagcgtcagaccccgt agaaaagatcaaaggatcttcttgagatcct ttttttctgcgcgtaatctgctgcttgcaaa caaaaaaaccaccgctaccagcggtggtttg tttgccggatcaagagctaccaactcttttt ccgaaggtaactggcttcagcagagcgcaga taccaaatactgtccttctagtgtagccgta gttaggccaccacttcaagaactctgtagca ccgcctacatacctcgctctgctaatcctgt taccagtggctgctgccagtggcgataagtc gtgtcttaccgggttggactcaagacgatag ttaccggataaggcgcagcggtcgggctgaa cggggggttcgtgcacacagcccagcttgga gcgaacgacctacaccgaactgagataccta cagcgtgagctatgagaaagcgccacgcttccc gaagggagaaaggcggacaggtatccggta agcggcagggtcggaacaggagagcgcac gagggagcttccagggggaaacgcctggtatc tttatagtcctgtcgggtttcgccacctctg acttgagcgtcgatttttgtgatgctcgtca ggggggcggagcctatggaaaaacgccagc aacgcggcctttttacggttcctggccttttg ctggccttttgctcacatgttctttcctgcg ttatcccctgattctgtggataaccgtatta ccgcctttgagtgagctgataccgctcgccgc agccgaacgaccgagcgcagcgagtcagtg agcgaggaagcggaagagcgcctgatgcgg tattttctccttacgcatctgtgcggtatttc acaccgcatatatggtgcactctcagtacaa tctgctctgatgccgcatagttaagccagta tacactccgctatcgctacgtgactgggtca tggctgcgccccgacacccgccaacacccgc tgacgcgccctgacgggcttgtctgctcccg gcatccgcttacagacaagctgtgaccgtct ccgggagctgcatgtgtcagaggttttcacc gtcatcaccgaaacgcgcgaggcagctgcgg taaagctcatcagcgtggtcgtgaagcgatt cacagatgtctgcctgttcatccgcgtccag ctcgttgagtttctccagaagcgttaatgtc tggcttctgataaagcgggccatgttaaggg cggttttttcctgtttggtcactgatgcctc cgtgtaagggggatttctgttcatgggggta atgataccgatgaaacgagagaggatgctca cgatacgggttactgatgatgaacatgcccg gttactggaacgttgtgagggtaaacaactg gcggtatggatgcggcgggaccagagaaaaa tcactcagggtcaatgccagcgcttcgttaa tacagatgtaggtgttccacagggtagccag cagcatcctgcgatgcagatccggaacataa tggtgcagggcgctgacttccgcgtttccag actttacgaaacacggaaaccgaagaccatt catgttgttgctcaggtcgcagacgttttgc agcagcagtcgcttcacgttcgctcgcgtat cggtgattcattctgctaaccagtaaggcaa ccccgccagcctagccgggtcctcaacgaca ggagcacgatcatgctagtcatgccccgcgc ccaccggaaggagctgactgggttgaaggct ctcaagggcatcggtcgagatcccggtgcct aatgagtgagctaacttacattaattgcgtt gcgctcactgcccgctttccagtcgggaaac ctgtcgtgccagctgcattaatgaatcggcc aacgcgcggggagaggcggtttgcgtattgg gcgccagggtggtttttcttttcaccagtga gacgggcaacagctgattgcccttcaccgcc tggccctgagagagttgcagcaagcggtcca cgctggtttgccccagcaggcgaaaatcctg tttgatggtggttaacggcgggatataacat gagctgtcttcggtatcgtcgtatcccacta ccgagatgtccgcaccaacgcgcagcccgga ctcggtaatggcgcgcattgcgcccagcgcc atctgatcgttggcaaccagcatcgcagtgg gaacgatgccctcattcagcatttgcatggt ttgttgaaaaccggacatggcactccagtcg ccttcccgttccgctatcggctgaatttgat tgcgagtgagatatttatgccagccagccag acgcagacgcgccgagacagaacttaatggg cccgctaacagcgcgatttgctggtgaccca atgcgaccagatgctccacgcccagtcgcgt accgtcttcatgggagaaaataatactgttg atgggtgtctggtcagagacatcaagaaata acgccggaacattagtgcaggcagcttccac agcaatggcatcctggtcatccagcggatag ttaatgatcagcccactgacgcgttgcgcga gaagattgtgcaccgccgctttacaggcttc gacgccgcttcgttctaccatcgacaccacc acgctggcacccagttgatcggcgcgagatt taatcgccgcgacaatttgcgacggcgcgtg cagggccagactggaggtggcaacgccaatc agcaacgactgtttgcccgccagttgttgtg ccacgcggttgggaatgtaattcagctccgc catcgccgcttccactttttcccgcgttttc gcagaaacgtggctggcctggttcaccacgc gggaaacggtctgataagagacaccggcata ctctgcgacatcgtataacgttactggtttc acattcaccaccctgaattgactctcttccg ggcgctatcatgccataccgcgaaaggtttt gcgccattcgatggtgtccgggatctcgacg ctctcccttatgcgactcctgcattaggaag cagcccagtagtaggttgaggccgttgagca ccgccgccgcaaggaatggtgcatgcaagga gatggcgcccaacagtcccccggccacgggg cctgccaccatacccacgccgaaacaagcgc tcatgagcccgaagtggcgagcccgatcttc cccatcggtgatgtcggcgatataggcgcca gcaaccgcacctgtggcgccggtgatgccgg ccacgatgcgtccggcgtagaggatcgagat cgatctcgatcccgcgaaattaatacgactc actata | |
| SNAP-25b (1–206, all C to A; capitalized) in pET28a | homemade | tggcgaatgggacgcgccctgtagcggcgca ttaagcgcggcgggtgtggtggttacgcgca gcgtgaccgctacacttgccagcgccctagc gcccgctcctttcgctttcttcccttccttt ctcgccacgttcgccggctttccccgtcaag ctctaaatcgggggctccctttagggttccg atttagtgctttacggcacctcgaccccaaa aaacttgattagggtgatggttcacgtagtg ggccatcgccctgatagacggtttttcgccc tttgacgttggagtccacgttctttaatagt ggactcttgttccaaactggaacaacactca accctatctcggtctattcttttgatttata agggattttgccgatttcggcctattggtta aaaaatgagctgatttaacaaaaatttaacg cgaattttaacaaaatattaacgtttacaat ttcaggtggcacttttcggggaaatgtgcgc ggaacccctatttgtttatttttctaaatac attcaaatatgtatccgctcatgaattaatt cttagaaaaactcatcgagcatcaaatgaaa ctgcaatttattcatatcaggattatcaata ccatatttttgaaaaagccgtttctgtaatg aaggagaaaactcaccgaggcagttccatag gatggcaagatcctggtatcggtctgcgatt ccgactcgtccaacatcaatacaacctatta atttcccctcgtcaaaaataaggttatcaag tgagaaatcaccatgagtgacgactgaatcc ggtgagaatggcaaaagtttatgcatttctt tccagacttgttcaacaggccagccattacg ctcgtcatcaaaatcactcgcatcaaccaaa ccgttattcattcgtgattgcgcctgagcga gacgaaatacgcgatcgctgttaaaaggaca attacaaacaggaatcgaatgcaaccggcgc aggaacactgccagcgcatcaacaatatttt cacctgaatcaggatattcttctaatacctg gaatgctgttttcccggggatcgcagtggtg agtaaccatgcatcatcaggagtacggataa aatgcttgatggtcggaagaggcataaattc cgtcagccagtttagtctgaccatctcatct gtaacatcattggcaacgctacctttgccat gtttcagaaacaactctggcgcatcgggctt cccatacaatcgatagattgtcgcacctgat tgcccgacattatcgcgagcccatttatacc catataaatcagcatccatgttggaatttaa tcgcggcctagagcaagacgtttcccgttga atatggctcataacaccccttgtattactgt ttatgtaagcagacagttttattgttcatga ccaaaatcccttaacgtgagttttcgttcca ctgagcgtcagaccccgtagaaaagatcaaa ggatcttcttgagatcctttttttctgcgcg taatctgctgcttgcaaacaaaaaaaccacc gctaccagcggtggtttgtttgccggatcaa gagctaccaactctttttccgaaggtaactg gcttcagcagagcgcagataccaaatactgt ccttctagtgtagccgtagttaggccaccac ttcaagaactctgtagcaccgcctacatacc tcgctctgctaatcctgttaccagtggctgc tgccagtggcgataagtcgtgtcttaccggg ttggactcaagacgatagttaccggataagg cgcagcggtcgggctgaacggggggttcgtg cacacagcccagcttggagcgaacgacctac accgaactgagatacctacagcgtgagctatg agaaagcgccacgcttcccgaagggagaaa ggcggacaggtatccggtaagcggcagggtc ggaacaggagagcgcacgagggagcttcc agggggaaacgcctggtatctttatagtcctgt cgggtttcgccacctctgacttgagcgtcga tttttgtgatgctcgtcaggggggcggagcc tatggaaaaacgccagcaacgcggccttttt acggttcctggccttttgctggccttttgct cacatgttctttcctgcgttatcccctgatt ctgtggataaccgtattaccgcctttgagtg agctgataccgctcgccgcagccgaacgacc gagcgcagcgagtcagtgagcgaggaagc ggaagagcgcctgatgcggtattttctccttac gcatctgtgcggtatttcacaccgcatatat ggtgcactctcagtacaatctgctctgatgc cgcatagttaagccagtatacactccgctat cgctacgtgactgggtcatggctgcgccccg acacccgccaacacccgctgacgcgccctga cgggcttgtctgctcccggcatccgcttaca gacaagctgtgaccgtctccgggagctgcat gtgtcagaggttttcaccgtcatcaccgaaa cgcgcgaggcagctgcggtaaagctcatcag cgtggtcgtgaagcgattcacagatgtctgc ctgttcatccgcgtccagctcgttgagtttc tccagaagcgttaatgtctggcttctgataa agcgggccatgttaagggcggttttttcctg tttggtcactgatgcctccgtgtaaggggga tttctgttcatgggggtaatgataccgatga aacgagagaggatgctcacgatacgggttac tgatgatgaacatgcccggttactggaacgt tgtgagggtaaacaactggcggtatggatgc ggcgggaccagagaaaaatcactcagggtc aatgccagcgcttcgttaatacagatgtaggt gttccacagggtagccagcagcatcctgcga tgcagatccggaacataatggtgcagggcgc tgacttccgcgtttccagactttacgaaaca cggaaaccgaagaccattcatgttgttgctc aggtcgcagacgttttgcagcagcagtcgct tcacgttcgctcgcgtatcggtgattcattc tgctaaccagtaaggcaaccccgccagccta gccgggtcctcaacgacaggagcacgatcat gcgcacccgtggggccgccatgccggcgata atggcctgcttctcgccgaaacgtttggtgg cgggaccagtgacgaaggcttgagcgagggc gtgcaagattccgaataccgcaagcgacagg ccgatcatcgtcgcgctccagcgaaagcggt cctcgccgaaaatgacccagagcgctgccgg cacctgtcctacgagttgcatgataaagaag acagtcataagtgcggcgacgatagtcatgc cccgcgcccaccggaaggagctgactgggtt gaaggctctcaagggcatcggtcgagatccc ggtgcctaatgagtgagctaacttacattaa ttgcgttgcgctcactgcccgctttccagtc gggaaacctgtcgtgccagctgcattaatga atcggccaacgcgcggggagaggcggtttgc gtattgggcgccagggtggtttttcttttca ccagtgagacgggcaacagctgattgccctt caccgcctggccctgagagagttgcagcaag cggtccacgctggtttgccccagcaggcgaa aatcctgtttgatggtggttaacggcgggat ataacatgagctgtcttcggtatcgtcgtat cccactaccgagatatccgcaccaacgcgca gcccggactcggtaatggcgcgcattgcgcc cagcgccatctgatcgttggcaaccagcatc gcagtgggaacgatgccctcattcagcattt gcatggtttgttgaaaaccggacatggcact ccagtcgccttcccgttccgctatcggctga atttgattgcgagtgagatatttatgccagc cagccagacgcagacgcgccgagacagaa cttaatgggcccgctaacagcgcgatttgctgg tgacccaatgcgaccagatgctccacgccca gtcgcgtaccgtcttcatgggagaaaataat actgttgatgggtgtctggtcagagacatca agaaataacgccggaacattagtgcaggcag cttccacagcaatggcatcctggtcatccag cggatagttaatgatcagcccactgacgcgt tgcgcgagaagattgtgcaccgccgctttac aggcttcgacgccgcttcgttctaccatcga caccaccacgctggcacccagttgatcggcg cgagatttaatcgccgcgacaatttgcgacg gcgcgtgcagggccagactggaggtggcaac gccaatcagcaacgactgtttgcccgccagt tgttgtgccacgcggttgggaatgtaattca gctccgccatcgccgcttccactttttcccg cgttttcgcagaaacgtggctggcctggttc accacgcgggaaacggtctgataagagacac cggcatactctgcgacatcgtataacgttac tggtttcacattcaccaccctgaattgactc tcttccgggcgctatcatgccataccgcgaa aggttttgcgccattcgatggtgtccgggat ctcgacgctctcccttatgcgactcctgcat taggaagcagcccagtagtaggttgaggccg ttgagcaccgccgccgcaaggaatggtgcat gcaaggagatggcgcccaacagtcccccggc cacggggcctgccaccatacccacgccgaaa caagcgctcatgagcccgaagtggcgagccc gatcttccccatcggtgatgtcggcgatata ggcgccagcaaccgcacctgtggcgccggtg atgccggccacgatgcgtccggcgtagagga tcgagatctcgatcccgcgaaattaatacga ctcactataggggaattgtgagcggataaca attcccctctagaaataattttgtttaactt taagaaggagatataccATGGCCGA GGACGCAGACATGCGCAATG AGCTGGAGGAGATGCAGAGG AGGGCTGACCAGCTGGCTGA TGAGTCCCTGGAAAGCACCC GTCGCATGCTGCAGCTGGTT GAAGAGAGTAAAGATGCTGG CATCAGGACTTTGGTTATGTT GGATGAGCAAGGCGAACAAC TGGAACGCATTGAGGAAGGG ATGGACCAAATCAATAAGGAC ATGAAAGAAGCAGAAAAGAAT TTGACGGACCTAGGAAAATTC GCCGGCCTTGCCGTGGCCCC CGCCAACAAGCTTAAATCCAG TGATGCTTACAAAAAAGCCTG GGGCAATAATCAGGATGGAGT AGTGGCCAGCCAGCCTGCCC GTGTGGTGGATGAACGGGAG CAGATGGCCATCAGTGGTGGC TTCATCCGCAGGGTAACAAAT GATGCCCGGGAAAATGAGATG GATGAGAACCTGGAGCAGGT GAGCGGCATCATCGGAAACCT CCGCCACATGGCTCTAGACAT GGGCAATGAGATTGACACCCA GAATCGCCAGATCGACAGGAT CATGGAGAAGGCTGATTCCAA CAAAACCAGAATTGATGAAGC CAACCAACGTGCAACAAAGAT GCTGGGAAGTGGTTAA ctcgagcaccaccaccaccaccactgag atccggctgctaacaaagcccgaaagga agctgagttggctgctgccaccgctgagc aataactagcataaccccttggggcctc taaacgggtcttgaggggttttttgctgaa aggaggaactatatccggat | |
| Materials for protein purificaiton | |||
| 2-Mercaptoethanol | SIGMA | M3148-25ML | |
| Agar | LPS Solution | AGA500 | |
| Ampicillin, Sodium salt | PLS | AC1043-005-00 | |
| Chloramphenicol | PLS | CR1023-050-00 | |
| Competent cells (E. coli) | Novagen | 70956 | Rosetta(DE3)pLysS |
| Glycerol | SIGMA | G5516-500ML | |
| HEPES | SIGMA | H4034-100G | |
| Hydrochloric acid / HCl | SIGMA | 320331-500ML | |
| Imidazole | SIGMA | I2399-100G | |
| Isopropyl β-D-1-thiogalactopyranoside / IPTG | SIGMA | 10724815001 | |
| Kanamycin Sulfate | PLS | KC1001-005-02 | |
| Luria-Bertani (LB) Broth | LPS Solution | LB-05 | |
| Ni-NTA resin | Qiagen | 30210 | |
| PD MiniTrap G-25 (desalting column) | Cytiva | GE28-9180-07 | For instructions, see: https://www.cytivalifesciences.com/en/us/shop/chromatography/prepacked-columns/desalting-and-buffer-exchange/pd-minitrap-desalting-columns-with-sephadex-g-25-resin-p-06174 |
| Phenylmethylsulfonyl fluoride / PMSF | ThermoFisher Scientific | 36978 | |
| Plasmids for SNARE proteins | cloned in house | N/A | Available upon request |
| Protease inhibitor cocktail | genDEPOT | P3100 | |
| Sodium chloride | SIGMA | S5886-500G | |
| Sodium phosphate dibasic / Na2HPO4 | SIGMA | S7907-100G | |
| Sodium phosphate monobasic / NaH2PO4 | SIGMA | S3139-250G | |
| Tris(2-carboxyethyl)phosphine / TCEP | SIGMA | C4706-2G | |
| Trizma base | SIGMA | T1503-250G | |
| Materials for sample assembly | |||
| Biotin-PEG-SVA | LAYSAN BIO | BIO-PEG-SVA-5K-100MG & MPEG-SVA-5K-1g | For PEGylation |
| Dibenzocyclooctyne-amine / DBCO-NH2 | SIGMA | 761540-10MG | For bead coating |
| Double-sided tape | 3M | 136 | For flow cell assembly |
| Epoxy glue | DEVCON | S-208 | For flow cell assembly |
| Glass coverslip for bottom surface | VWR | 48393-251 | Rectangular, 60×24 mm, #1.5 |
| Glass coverslip for top surface | VWR | 48393-241 | Rectangular, 50×24 mm, #1.5 |
| Magnetic bead | ThermoFisher Scientific | 14301 | Dynabeads M-270 Epoxy, 2.8 μm |
| mPEG-SVA | LAYSAN BIO | mPEG-SVA 1g | For PEGylation |
| N,N-Dimethylformamide / DMF | SIGMA | D4551-250ML | For bead coating |
| N-[3-(trimethoxysilyl)propyl]ethylenediamine | SIGMA | 104884-100ML | For PEGylation |
| Neutravidin | ThermoFisher Scientific | 31000 | For sample tethering |
| Phosphate buffered saline / PBS, pH 7.2 | PLS | PR2007-100-00 | |
| Plastic syringe | Norm-ject | A5 | 5 ml, luer tip |
| Polyethylene Tubing | SCI | BB31695-PE/4 | PE-60 |
| Reference bead | SPHEROTECH | SVP-30-5 | Streptavidin-coated Polystyrene Particles; 3.0-3.4 µm |
| Syringe needle | Kovax | 21G-1 1/4'' | 21 G |
| Syringe pump | KD SCIENTIFIC | 788210 | |
| Equipment for magnetic tweezer instrument | |||
| 1-axis motorized microtranslation stage | PI | M-126.PD1 | For vertical positioning of magnets |
| 2-axis manual translation stage | ST1 | LEE400 | For alignment of magnets to the optical axis |
| Acrylic holder for magnets | DaiKwang Precision | custum order | Drawing available upon request |
| Frame grabber | Active Silicon | AS-FBD-4XCXP6-2PE8 | |
| High-speed CMOS camera | Mikrotron | EoSens 3CXP | |
| Inverted microscope | Olympus | IX73P2F-1-2 | |
| Neodymium magnets | LG magnet | ND 10x10x12t | Dimension: 10 mm × 10 mm × 12 mm; two needed |
| Objective lens | Olympus | UPLXAPO100XO | Oil-immersion, NA 1.45 |
| Objective lens nanopositioner | Mad City Labs | Nano-F100S | |
| Rotation stepper motor | AUTONICS | A3K-S545W | For rotating magnets |
| Superluminescent diode | QPHOTONICS | QSDM-680-2 | 680 nm |
| Software | |||
| LabVIEW | National Instruments | v20.0f1 | |
| MATLAB | MathWorks | v2021a |
Ссылки
- Le, S., Liu, R., Lim, C. T., Yan, J. Uncovering mechanosensing mechanisms at the single protein level using magnetic tweezers. Methods. 94, 13-18 (2016).
- Choi, H. -. K., Kim, H. G., Shon, M. J., Yoon, T. -. Y. High-resolution single-molecule magnetic tweezers. Annual Review of Biochemistry. 91 (1), 33-59 (2022).
- Yang, T., Park, C., Rah, S. -. H., Shon, M. J. Nano-precision tweezers for mechanosensitive proteins and beyond. Molecules and Cells. 45 (1), 16-25 (2022).
- Neuman, K. C., Nagy, A. Single-molecule force spectroscopy: optical tweezers, magnetic tweezers and atomic force microscopy. Nature Methods. 5 (6), 491-505 (2008).
- De Vlaminck, I., Dekker, C. Recent advances in magnetic tweezers. Annual Review of Biophysics. 41 (1), 453-472 (2012).
- Bustamante, C. J., Chemla, Y. R., Liu, S., Wang, M. D. Optical tweezers in single-molecule biophysics. Nature Reviews Methods Primers. 1, 25 (2021).
- Gosse, C., Croquette, V. Magnetic tweezers: micromanipulation and force measurement at the molecular level. Biophysical Journal. 82 (6), 3314-3329 (2002).
- Smith, S. B., Finzi, L., Bustamante, C. Direct mechanical measurements of the elasticity of single DNA molecules by using magnetic beads. Science. 258 (5085), 1122-1126 (1992).
- Lansdorp, B. M., Tabrizi, S. J., Dittmore, A., Saleh, O. A. A high-speed magnetic tweezer beyond 10,000 frames per second. Review of Scientific Instruments. 84 (4), 044301 (2013).
- Cnossen, J. P., Dulin, D., Dekker, N. H. An optimized software framework for real-time, high-throughput tracking of spherical beads. Review of Scientific Instruments. 85 (10), 103712 (2014).
- Dulin, D., et al. High spatiotemporal-resolution magnetic tweezers: calibration and applications for DNA dynamics. Biophysical Journal. 109 (10), 2113-2125 (2015).
- Huhle, A., et al. Camera-based three-dimensional real-time particle tracking at kHz rates and Ångström accuracy. Nature Communications. 6 (1), 5885 (2015).
- Popa, I., et al. A HaloTag anchored ruler for week-long studies of protein dynamics. Journal of the American Chemical Society. 138 (33), 10546-10553 (2016).
- Shon, M. J., Kim, H., Yoon, T. -. Y. Focused clamping of a single neuronal SNARE complex by complexin under high mechanical tension. Nature Communications. 9 (1), 3639 (2018).
- Tapia-Rojo, R., Eckels, E. C., Fernández, J. M. Ephemeral states in protein folding under force captured with a magnetic tweezers design. Proceedings of the National Academy of Sciences. 116 (16), 7873-7878 (2019).
- Löf, A., et al. Multiplexed protein force spectroscopy reveals equilibrium protein folding dynamics and the low-force response of von Willebrand factor. Proceedings of the National Academy of Sciences. 116 (38), 18798-18807 (2019).
- Tapia-Rojo, R., Alonso-Caballero, A., Fernandez, J. M. Direct observation of a coil-to-helix contraction triggered by vinculin binding to talin. Science Advances. 6 (21), (2020).
- Rieu, M., et al. Parallel, linear, and subnanometric 3D tracking of microparticles with Stereo Darkfield Interferometry. Science Advances. 7 (6), (2021).
- Rieu, M., Valle-Orero, J., Ducos, B., Allemand, J. -. F., Croquette, V. Single-molecule kinetic locking allows fluorescence-free quantification of protein/nucleic-acid binding. Communications Biology. 4 (1), 1083 (2021).
- Woodside, M. T., et al. Nanomechanical measurements of the sequence-dependent folding landscapes of single nucleic acid hairpins. Proceedings of the National Academy of Sciences. 103 (16), 6190-6195 (2006).
- Camunas-Soler, J., Ribezzi-Crivellari, M., Ritort, F. Elastic properties of nucleic acids by single-molecule force spectroscopy. Annual Review of Biophysics. 45 (1), 65-84 (2016).
- Südhof, T. C., Rothman, J. E. Membrane fusion: grappling with SNARE and SM proteins. Science. 323 (5913), 474-477 (2009).
- Gao, Y., et al. Single reconstituted neuronal SNARE complexes zipper in three distinct stages. Science. 337 (6100), 1340-1343 (2012).
- Zorman, S., et al. Common intermediates and kinetics, but different energetics, in the assembly of SNARE proteins. eLife. 3, e03348 (2014).
- Zhang, Y., Hughson, F. M. Chaperoning SNARE folding and assembly. Annual Review of Biochemistry. 90 (1), 581-603 (2021).
- Vilfan, I. D., Lipfert, J., Koster, D. A., Lemay, S. G., Dekker, N. H. Magnetic tweezers for single-molecule experiments. Handbook of Single-Molecule Biophysics. , 371-395 (2009).
- You, H., Le, S., Chen, H., Qin, L., Yan, J. Single-molecule manipulation of G-quadruplexes by magnetic tweezers. Journal of Visualized Experiments. (127), e56328 (2017).
- Lipfert, J., Hao, X., Dekker, N. H. Quantitative modeling and optimization of magnetic tweezers. Biophysical Journal. 96 (12), 5040-5049 (2009).
- Dulin, D., Barland, S., Hachair, X., Pedaci, F. Efficient illumination for microsecond tracking microscopy. PLoS One. 9 (9), e107335 (2014).
- Klaue, D., Seidel, R. Torsional stiffness of single superparamagnetic microspheres in an external magnetic field. Physical Review Letters. 102 (2), 028302 (2009).
- Shon, M. J., Rah, S. -. H., Yoon, T. -. Y. Submicrometer elasticity of double-stranded DNA revealed by precision force-extension measurements with magnetic tweezers. Science Advances. 5 (6), 1697 (2019).
- Czerwinski, F., Richardson, A. C., Oddershede, L. B. Quantifying noise in optical tweezers by Allan variance. Optics Express. 17 (15), 13255-13269 (2009).
- Lansdorp, B. M., Saleh, O. A. Power spectrum and Allan variance methods for calibrating single-molecule video-tracking instruments. Review of Scientific Instruments. 83 (2), 025115 (2012).
- Ostrofet, E., Papini, F. S., Dulin, D. High spatiotemporal resolution data from a custom magnetic tweezers instrument. Data in Brief. 30, 105397 (2020).
- Yu, Z., et al. A force calibration standard for magnetic tweezers. Review of Scientific Instruments. 85 (12), 123114 (2014).
- Strick, T. R., Allemand, J. -. F., Bensimon, D., Bensimon, A., Croquette, V. The elasticity of a single supercoiled DNA molecule. Science. 271 (5257), 1835-1837 (1996).
- Daldrop, P., Brutzer, H., Huhle, A., Kauert, D. J., Seidel, R. Extending the range for force calibration in magnetic tweezers. Biophysical Journal. 108 (10), 2550-2561 (2015).
- te Velthuis, A. J. W., Kerssemakers, J. W. J., Lipfert, J., Dekker, N. H. Quantitative guidelines for force calibration through spectral analysis of magnetic tweezers data. Biophysical Journal. 99 (4), 1292-1302 (2010).
- Ostrofet, E., Papini, F. S., Dulin, D. Correction-free force calibration for magnetic tweezers experiments. Scientific Reports. 8 (1), 15920 (2018).
- Seol, Y., Li, J., Nelson, P. C., Perkins, T. T., Betterton, M. D. Elasticity of short DNA molecules: theory and experiment for contour lengths of 0.6-7 µm. Biophysical Journal. 93 (12), 4360-4373 (2007).
- Burnham, D. R., Vlaminck, I. D., Henighan, T., Dekker, C. Skewed Brownian fluctuations in single-molecule magnetic tweezers. PLoS One. 9 (9), 108271 (2014).
- Paul, T., Myong, S. Protocol for generation and regeneration of PEG-passivated slides for single-molecule measurements. STAR Protocols. 3 (1), 101152 (2022).
- Lee, H. -. W., et al. Profiling of protein-protein interactions via single-molecule techniques predicts the dependence of cancers on growth-factor receptors. Nature Biomedical Engineering. 2 (4), 239-253 (2018).
- Cheezum, M. K., Walker, W. F., Guilford, W. H. Quantitative comparison of algorithms for tracking single fluorescent particles. Biophysical Journal. 81 (4), 2378-2388 (2001).
- Parthasarathy, R. Rapid, accurate particle tracking by calculation of radial symmetry centers. Nature Methods. 9 (7), 724-726 (2012).
- Woodside, M. T., Block, S. M. Reconstructing folding energy landscapes by single-molecule force spectroscopy. Annual Review of Biophysics. 43 (1), 19-39 (2014).
- Evans, E., Ritchie, K. Dynamic strength of molecular adhesion bonds. Biophysical Journal. 72 (4), 1541-1555 (1997).
- Zhang, Y. Energetics, kinetics, and pathway of SNARE folding and assembly revealed by optical tweezers. Protein Science. 26 (7), 1252-1265 (2017).
- Chen, H., et al. Improved high-force magnetic tweezers for stretching and refolding of proteins and short DNA. Biophysical Journal. 100 (2), 517-523 (2011).
- Cho, S., et al. Tension exerted on cells by magnetic nanoparticles regulates differentiation of human mesenchymal stem cells. Biomaterials Advances. 139, 213028 (2022).
- Shon, M. J., Cohen, A. E. Nano-mechanical measurements of protein-DNA interactions with a silicon nitride pulley. Nucleic Acids Research. 44 (1), 7 (2016).
- Cheng, Y. Single-particle cryo-EM-How did it get here and where will it go. Science. 361 (6405), 876-880 (2018).
- Jumper, J., et al. Highly accurate protein structure prediction with AlphaFold. Nature. 596 (7873), 583-589 (2021).
- Neupane, K., et al. Direct observation of transition paths during the folding of proteins and nucleic acids. Science. 352 (6282), 239-242 (2016).
- Choi, H. -. K., et al. Watching helical membrane proteins fold reveals a common N-to-C-terminal folding pathway. Science. 366 (6469), 1150-1156 (2019).
- Kim, C., et al. Extreme parsimony in ATP consumption by 20S complexes in the global disassembly of single SNARE complexes. Nature Communications. 12 (1), 3206 (2021).
Перепечатки и разрешения
Запросить разрешение на использование текста или рисунков этого JoVE статьи
Запросить разрешениеThis article has been published
Video Coming Soon
Авторские права © 2025 MyJoVE Corporation. Все права защищены