Method Article
Organizatörü yakalama Merhaba-C: Yüksek çözünürlüklü, genom çapında organizatörü etkileşimlerin profil oluşturma
* Bu yazarlar eşit katkıda bulunmuştur
Bu Makalede
Özet
DNA arttırıcılar gibi düzenleyici elemanlarının Gen ifadesinin fiziksel olarak hedef gen rehberleri, sık sık büyük genomik mesafeleri kapsayan uzun menzilli kromozom etkileşimleri aracılığıyla irtibata geçerek kontrol. Organizatörü yakalama Hi-C (PCHi-C) hedef genleri için potansiyel yasal sıralarının atama etkinleştirme rehberleri ve distal bölgeler arasındaki önemli etkileşimler tanımlar.
Özet
Üç boyutlu genom organizasyonu onun işlevine bağlıdır. Örneğin, sık sık önemli (bazı durumlarda yüzlerce kilobases) genomik mesafeler köprü ve yan yol fiziksel temas yoluyla onların hedef genlerin spatio-temporal ifade transkripsiyon arttırıcılar gibi düzenleyici elemanlarının kontrol Yakındaki genler. İnsan genomu büyük çoğunluğu var bilinmeyen bir tahmini bir milyon arttırıcılar limanlar gen hedefler. Hedef genleri distal düzenleyici bölgeleri atama böylece gen ifadesi denetim anlamanız çok önemlidir. Bir tek denemede tüm rehberleri için organizatörü yakalama Hi-C (PCHi-C) distal organizatörü etkileşim bölgeleri (PIRs), genom çapında algılamasını etkinleştirmek için geliştirilmiştir. PCHi-C, son derece karmaşık Hi-C kitaplıklar organizatörü serileri biotinylated RNA yemler tamamlayıcı bütün organizatörü içeren kısıtlama parçaları sonuna kadar binlerce çözüm karma seçim yoluyla için özel olarak zenginleştirilmiş. Sonra açılan organizatörü dizileri ve arttırıcılar ve diğer potansiyel düzenleyici elemanlarının gibi sık etkileşim eşleri için amaçtır. Yüksek üretilen iş eşleştirilmiş uç sıralama sonra kısıtlama parça düzeyinde önemli PIRs tanımlamak için her kısıtlama organizatörü bakmaksızın parçası bir istatistiksel test uygulanır. PCHi-C uzun menzilli organizatörü etkileşimlerin bir atlas onlarca insan ve fare hücre tipleri oluşturmak için kullandık. Bu organizatörü interactome haritalar memeli gen ifadesi denetim daha büyük bir anlayış için hedef genleri sözde düzenleyici bölgeleri atama ve Tercihli kayma organizatörü organizatörü etkileşim ağları açığa katkıda bulunmuştur. Bu bilgileri ayrıca insan genetik hastalık ve potansiyel hastalık genler, tanımlaması kodlamayan hastalık ilişkili bağlayarak anlamak için yüksek alaka vardır sıra türevleri veya kontrol dizilerini için onların hedef genlerin yakınında.
Giriş
Kanıt biriken üç boyutlu genom organizasyonu nükleer süreçler, gen harekete geçirmek1,2,3, baskı4 de dahil olmak üzere bir dizi önemli bir işlevsel rol oynar öneriyor ,5,6,7,8, rekombinasyon9,10, DNA onarım11, DNA çoğaltma12,13, ve hücresel yaşlanma14. Uzak arttırıcılar uygun spatio-temporal gen ifadesi denetim için gerekli olan15,16,17, düzenleyen Rehberleri kayma yakınında bulunur. Distal arttırıcılar hedef Gen transkripsiyonu18,19,20,21,22ve 'kromatin döngü zorla' için gerekli olan artırıcı silme işlemlerini göster bir artırıcı ve onun hedef organizatörü Hbb odağı içinde arasında mühendislik hayvan zinciri transkripsiyon harekete geçirmek23götürmek için yeterli olduğunu gösterir. Ayrıca, genler ektopik arttırıcılar kontrol altında getirmek genom düzenlemeler uygunsuz gen harekete geçirmek ve hastalık24,25,26neden olabilir. Birlikte, bu örnekler organizatörü artırıcı etkileşimleri gen kontrolü için gerekli olan ve uygun gen ekspresyonu emin olmak için sıkı düzenleme gerektirir göstermektedir. İnsan ve fare genleri her tahmin edilmektedir yaklaşık bir milyon arttırıcılar liman için. Bu arttırıcılar büyük çoğunluğu için hedef genlerin bilinmemektedir ve 'kuralları' organizatör ve arttırıcılar arasında kötü anlaşılır. Onların hedef genlerin transkripsiyon arttırıcılar atama böylece memeli gen ifadesi denetim deşifre içinde büyük bir meydan okuma olarak kalır.
Üç boyutlu genom mimari anlayışımız 3 C27 (kromozom conformation yakalama) giriş ve onun türevleri28,29,30,31 tarafından devrim . Bu tekniklerin, Hi-C (yüksek işlem hacmi kromozom conformation yakalama) en güçlü bir hücre nüfus içinde kromozom etkileşimler tüm topluluğu tanımlamak için tasarlanmıştır. Merhaba C kitaplıklar, genellikle üretilen milyonlarca hücre, son derece karmaşık bir tahmini 1011 bağımsız ligasyonu ürünlerinde insan genomu32~ 4 kb parçaları arasında. Bir sonucu, bireysel kısıtlama arasındaki etkileşimler güvenilir ve tekrarlanabilir tanımlaması (içerenler gibi bir organizatörü veya artırıcı) dan parçaları gibi Hi-C veri Hi-C kitaplıklar için Ultra Derin sıralama tabi olan sürece mümkün değildir, Hangi Hi-C kitaplıklar rutin olarak hazırlama laboratuarlar için ekonomik açıdan mantıklı bir çözüm değil. Bu eksiklik aşmak için organizatörü yakalama Hi-özellikle ligasyonu urun organizatörü içeren Hi-C kitaplıklar zenginleştirmek için C geliştirdi. Biz iki nedenden dolayı Rehberleri üzerinde duruldu. İlk olarak, organizatörü artırıcı kişiler uygun gen ifade düzeyleri (bkz. Yukarıdaki referanslar) çok sayıda çalışma için çok önemli olduğu gösterilmiştir ve organizatör hücre tipleri arasında büyük ölçüde sabit olduğundan, ikinci olarak, aynı yakalama yem sistemi sorgulamak için kullanılabilir birden çok hücre tipleri ve koşullar karşısında yasal devresi. Çözüm Hi-C kitaplıklar hibridizasyon on binlerce biotinylated RNA 120mers Hi-C ligasyonu ürünleri organizatörü içeren ve manyetik boncuklar streptavidin kaplı üzerinde sonraki yakalama için tamamlayıcı ile bizim yaklaşım kullanır. PCHi-C kütüphaneleri ile bu sonuçlar çok Organizatör önemli ölçüde yüksek frekanslarda bakmaksızın parçaları tanımlaması sadece odaklanan özgün Hi-C Kütüphanesi karşılaştırıldığında karmaşıklığı azaltmak.
Biz PCHi-C bir insan ve fare hücre tipleri daha iyi gen ifadesi denetim tarafından açığa çıkarmak uzun menzilli distal organizatörü etkileşen bölgeleri sözde düzenleyici fonksiyonu ile anlamak için katkıda bulunmak gibi rasgele kullanılmış çekirdeği üç boyutlu uzayda organizatörü organizatörü kişiler. Çalışmalar çok sayıda hücre türleri33,34,35,36,37,38arasında, yüz binlerce organizatörü artırıcı kişiler eşledikten 39, fare embriyonik kök hücre7kayma genom Polycomb baskıcı karmaşık aracılı kuruluşunda tanımlanan, büyük ölçekli organizatörü interactomes hücresel başkalaşım37sırasında, uyumlanma gösterdi 38 , 39ve bağlantılı kodlamayan hastalık ilişkili türevleri gen Rehberleri35sıra.
PCHi-C organizatör ile etkileşim DNA dizileri genom çapında topluluğu eşleştirmek için ideal bir yöntemdir. İlgili yaklaşımlar, gibi yakalama Hi-C ( konuyabakın) sürekli genomik bölgeler yüksek çözünürlüklü etkileşim profilleri seçilen genomik bölgeler için elde etmek için seçtiğiniz yöntemi vardır. PCHi-C ve yakalama Hi-C bir deneysel (tek fark olduğunu yakalama sistemi seçimi) açısından, son derece benzer böylece tavsiye ve yönergeleri biz sağlamak için her iki yaklaşımın tabidir. Burada, PCHi-c ayrıntılı bir açıklama mevcut Biz mantığı ve PCHi-C deney tasarımı anahat bir adım adım PCHi-C Kütüphanesi nesil protokol sağlar ve nasıl PCHi-C kitaplıklar kalitesini yüksek kaliteli veri vermeye protokolünde çeşitli adımlar takip edilebilir göstermektedir.
Protokol
1. Formaldehit fiksasyon
-
Hücre hazırlık: en az 2 x 107 hücre başına deney ile başlar.
- Kültüründe yetiştirilen hücreler için kültür orta hücrelerde resuspend. Ex vivo hücreler için 1 x Dulbecco'nın modifiye kartal Orta (% 10 (vol/vol) fetal Sığır serum ile (FBS) takıma DMEM), resuspend.
- Yapisan hücrelerin kültür orta kaldırın ve 30.625 mL % 10 (vol/vol) FBS, oda sıcaklığında (RT; 20-25 ° C) ile taze orta ekleyin.
- Süspansiyon hücrelerin, toplamak ve santrifüj kapasitesi 400 x g ve 20 ° C 3 dk. Kaldır süpernatant için hücreleri ve hücre Pelet 30.625 ml % 10 (vol/vol) FBS RT. yönü ile orta yeniden askıya alma
- Sağlam dokular için tripsin (% 0.05 ila %2.5 son konsantrasyonu, hücre türüne bağlı olarak) veya tek hücre süspansiyon elde etmek için homojenizasyon dounce kullanın. Bu ek adımdan sonra hücre süspansiyon hücreler gibi davran.
-
% 16 metanol-Alerjik paraformaldehyde (açık ampül sadece önceden kullanmak için) 4.375 mL %2 (vol/vol) son bir konsantrasyon için ekleyin. RT nazik bir rocker üzerinde karıştırma ile 10 min için düzeltme.
Dikkat: Paraformaldehyde tehlikeli bir kimyasaldır. Uygun sağlık ve Güvenlik Yönetmeliği izleyin. - Reaksiyon 5 mL taze hazırlanmış 1 M buz gibi glisin de ekleyerek gidermek. 5 min için nazik RT sallanan ile karıştırın ve zaman zaman ters çevirme ile 15 dakika buz üzerinde kuluçkaya.
-
Yıkama ve sabit hücreleri toplamak.
- Yapışık hücreleri için süpernatant kaldırmak, buz gibi 1 PBS pH 7,4 x 10 mL plaka duvara eklemek ve çıkarmak o. Buz gibi 1 PBS pH 7,4 x 1 mL ekleyin, bir hücre kazıyıcı ve transfer 50 mL tüp içine kullanarak hücreleri toplamak. İki kez mümkün olduğunca çok sayıda hücreleri toplamak için yineleyin. Buz gibi PBS 50 mL son hacim için mantıklı.
- Süspansiyon hücrelerin, santrifüj hücreleri 760 x g ve 5 min için 4 ° C süpernatant kaldırmak ve hücre Pelet 50 mL buz gibi PBS pH 7.4 yeniden askıya alma.
- Hücreleri 400 x g ve 4 ° C'de 10 dakika santrifüj kapasitesi ve dikkatli bir şekilde süpernatant çıkarın. Hücre Pelet sıvı azot içinde donmuş ve daha sonra birkaç ay için-80 ° C'de depolanan ek olabilir.
2. hücre lizis
- Yeniden askıya hücre Pelet buz gibi lizis taze hazırlanan tampon 50 ml (10 mM Tris-HCl pH 8, %0,2 (vol/vol) Igepal CA-630, 10 mM NaCl ve bir tablet proteaz inhibitörü kokteyl) ve karıştırın. 30 dk için buz üzerinde kuluçkaya, zaman zaman ters çevirme ile karıştırın. Çekirdeklerin 760 x g ve 5 min için 4 ° C, santrifüj kapasitesi ve süpernatant kaldırın.
3. hinDIII sindirim
- Hücre çekirdeği 1,25 x kısıtlama arabelleği 2 ile yıkayın. Hücre Pelet buz gibi 1,25 x kısıtlama arabellek 2 1 ml ve transfer 1,5 mL tüp içine yeniden askıya. 760 x g ve 5 min için 4 ° C, çekirdek spin ve süpernatant kaldırın.
- Hücre pelet 1,25 x kısıtlama arabellek 2 1790 µL içinde yeniden askıya alma. 5 aliquots, her 5-10 milyon 1,25 kısıtlama arabellek 2 x 358 µL hücreleri içeren olun.
- 11 µL % 10 (wt/vol) SDS sallamak başı aliquot bir thermomixer olarak 37 ° C'de 30 dk dk (devir/dakika) başına 950 devrimler ekleyin. Hücre kümeleri görünüyorsa, pipetting tarafından kabarcıklar kaçınarak ayırmak.
- %10 75 µL eklemek Triton X-100 (vol/vol) sallamak 950 devir/dakika ve 15 dakika içinde bir thermomixer 37 ° C başı aliquot. Hücre kümeleri görünüyorsa, pipetting tarafından kabarcıklar kaçınarak ayırmak.
-
12 µL 100 U/µL HinDIII 100, (Toplam 1.200 adet) aliquot başına ekleyin ve 37 ° C gece (O/N) bir thermomixer 950 rpm'de sallayarak süre kuluçkaya.
- Sindirim denetimi için enzim (sindirilmemiş denetimi) eklemeden önce örneği (her aliquot 5 µL) yeni bir tüp 25 µL aktarmak ve enzim (sindirilir denetimi) ekledikten sonra aynı işlemi tekrarlayın. Hi-C kütüphanesi olarak aynı şekilde her iki tüpler kuluçkaya.
- Ertesi sabah, aliquot 100 U/µL HinDIII (toplamda 500 adet) 5 µL ekleyin ve bir thermomixer 950 rpm'de sallayarak süre için 2 h 37 ° C'de kuluçkaya.
-
Sindirim denetimi: (bkz: 3.5.1) sindirilir ve sindirilmemiş kontrollerin crosslink ters (adım 6), fenol: kloroform çıkarma ve DNA yağış (7. adım) gerçekleştirin.
- Bir çift bir HinDIII site span astar tasarım. Aynı bölgede bir HinDIII site span yok astar başka bir çifti Tasarla. Kantitatif PCR (Q-PCR) astar tasarım Primer3 kullanarak (http://bioinfo.ut.ee/primer3-0.4.0/) ve aşağıdaki parametreleri:
Astar boyutu: en iyi 20 (dk.: 18, azami: 27); Astar Tm: En iyi 60 (dk.: 57, azami: 63); Astar CG % içeriği: dk.: 20, azami: 80; Amplicon boyutu: RT-PCR ~ 100 bp (geleneksel PCR ~ 300 bp); Kütüphane mispriming: insan (insan astar) ya da kemirgen ve basit (fare astar). - 4 ortalama Cts (eşik döngüsü) elde etmek için Q-PCR gerçekleştirmek: Ct [D; H] bir HinDIII sitesi [H]; span astar çifti ile sindirilir örnek [D] aldı, CT [D;-] olarak sindirilir örnekten [D] bir HinDIII sitesi [-]; span yok astar çifti ile elde edilen CT [U; H sindirilmemiş örnek [U] bir HinDIII site span astar çifti ile elde edilen], CT [U;-] olarak sindirilmemiş örnekten [U] bir HinDIII sitesi [-] span yok astar çifti ile elde edilen. Sindirim yüzdesini hesaplamak: % sindirim 100-100/2 =(Ct[D,H]-Ct[D,-]) - (Ct[U,H]-Ct[U,-]).
- Bir çift bir HinDIII site span astar tasarım. Aynı bölgede bir HinDIII site span yok astar başka bir çifti Tasarla. Kantitatif PCR (Q-PCR) astar tasarım Primer3 kullanarak (http://bioinfo.ut.ee/primer3-0.4.0/) ve aşağıdaki parametreleri:
4. Biotinylation kısıtlama parçası çıkıntılar
- Biotinylation ana karışım hazırlamak: 30,6 µL (Moleküler Biyoloji grade) kısıtlama tampon 2, H2O 10.2 µL x 10, 10 mM dCTP 7.65 µL 10 mM dGTP 7.65 µL 10 mM dTTP 7.65 µL 191.25 µL 0.4 mM biotin-14-dATP ve 5000 U/mL DNA polimeraz 51 µL ben büyük () Klenow) parçası.
- Biotinylation ana Mix aliquot, mix başına 60 µL ekleyin ve 1s 700 rpm (thermomixer) 5 sallayarak için 37 ° C'de kuluçkaya s, 30 s. 1 saat sonra aliquots buza koyun.
5. içinde-çekirdek ligasyonu
- Tüp ligasyonu ana karışım hazırlamak: 10 T4 DNA ligaz arabellek, 10 mg/ml sığır Serum albümin 51 µL x 510 µL (100 x BSA), su (Moleküler Biyoloji grade) 1754.4 µL ve 1 U/µL T4 DNA ligaz 127.5 µL ( Tablo malzemelerigörmek).
- 479 µL aliquot mix başına ligasyonu ana karışımı ekleyin ve 4 h 5 için 700 RPM sallayarak için 16 ° C'de kuluçkaya s her 2 dakikada bir thermomixer içinde.
- 30 dk RT. az kuluçkaya
6. Crosslink ters
- Tüm aliquots (yüksek hızlı Santrifüjü için uygun) 50 mL santrifüj tüp içinde birleştirir.
- 10 mg/ml RNase A, mix, 62.5 µL ekleyin ve 37 ° C'de 30 dk için kuluçkaya
- 10 mg/ml İndinavir K, mix, 300 µL ekleyin ve 37 ° C'de 30 dk için kuluçkaya
- Reaksiyon O/N (ya da en az 4 h) 65 ° C'de kuluçkaya Ertesi sabah, 10 mg/ml İndinavir K, mix, 300 µL ekleyin ve 65 ° C'de 1 h için kuluçkaya
7. DNA arıtma
- 4337.5 µL TLE arabellek (10 mM Tris-HCl pH 8.0; 0,1 mM EDTA pH 8.0) ve karışımı ekleyin.
- 1 birim (10 mL) fenol faz 8.0, 10 s ve Santrifüj için girdap RT ve 20.000 x g 3 dk. Transfer 9 mL üst (sulu) faz için yeni bir 50 mL tüp ekleyin.
Dikkat: Fenol tehlikeli bir kimyasaldır. Uygun sağlık ve Güvenlik Yönetmeliği izleyin. - TLE arabelleğe kalan sulu faz 2 mL ekleyin, girdap 10 s ve RT ve 20.000 x g 3 dakika süreyle santrifüj 2.5 mL sulu faz yeni tüp içine aktarmak için kimden adım 7.2 son hacim yapma, 11,5 mL. Daha düşük (organik) faz içeren tüp atmak.
- 1 hacmi (11,5 mL) fenol: kloroform: izoamil alkol (25:24:1), 10 s ve Santrifüj için girdap RT ve 20.000 x g 3 dk. 11 Transfer mL üst (sulu) faz için yeni bir 50 mL tüp ekleyin. 7.3 arasındaki adımları yineleyin. Toplam Numune hacmi şimdilik 13.5 mL.
- 3 M sodyum asetat pH 5.2 ve 33,75 mL buz gibi % 100 etanol, mix, 1,35 mL ekleyin ve-80 ° c 45 dk için kuluçkaya veya bir alternatif olarak-20 ° C'de gece
- 4 ° C ve 20.000 x g 10 dk santrifüj kapasitesi, süpernatant kaldırmak, pelet 1 mL % 70 (vol/vol) etanol taze hazırlanmış yeniden askıya alma ve yeni bir tüp aktarın.
- 4 ° C'de ve benchtop santrifüj 3 min için tam hızda santrifüj kapasitesi, sonra süpernatant kaldırın.
- Pelet 1 mL buz soğuk % 70 (vol/vol) etanol yeniden askıya alma ve 7,7 arasındaki adımları yineleyin. 10 dk 37 ° C'de Pelet Makinası ve TLE arabellek 650 µL içinde yeniden askıya alma. DNA verim çift iplikçikli DNA ölçmek için bir floresan tabanlı tahlil kullanarak belirleyin.
Not: Protokol burada donma ve örnek-80 ° c birkaç ay veya daha kısa bir süre için-20 ° C'de depolamak ek tarafından duraklatılmış.
8. kalite kontrol
- Monitör Kütüphane bütünlüğü ve ligasyonu DNA Elektroforez tarafından. Çalıştır 200 ng, bir % 0,8 özel/1 x TBE jel İnternet üzerinde. DNA bir grup üzerinde 10 kb olarak çalıştırmalısınız.
- Bilinen hücre tipi sabit kısa - ve uzun - menzil etkileşimler tarafından geleneksel PCR algılamak. Kullanım 100 ng şablonunun PCR reaksiyon DNA. PCR astar tasarım yakın ve kısıtlama siteleri (bkz: 3.7.1) Yukarıdaki talimatları uygulayarak doğru. Kalite kontrol fare ve insan Hi-C kitaplıklar için astar serilerini Tablo 1' de listelenmiştir.
-
Doldurma ve ligasyonu kontrolü: amplicons üzerinden içeren jel şeritler kesip kontrol 8.2, jel-DNA özü ve DNA 4 bireysel PCR reaksiyonları ile aynı astar bileşimleri için şablon olarak kullanmak.
- PCR arıtma seti kullanarak amplicons arındırmak ve DNA toplama ölçmek.
- Her amplicon 15 µL son bir hacim içinde dört sindirim reaksiyonları (HinDIII [a], NheI [b], HinDIII + NheI [c] ve hiçbir enzim [d]) hazırlamak: 500 amplicon ng, 10 kısıtlama arabellek 2.1, 10 mg/ml sığır Serum albümin 0.15 µL x 1,5 µL (100 x BSA) ve 0.1 µL (10 adet) enzim (HinDIII [a], NheI [b], HinDIII + NheI [c] veya su [d]).
- 37 ° C'de 1 h için sindirmek, sonra sindirim reaksiyonları %1,5 (wt/vol) özel/1 x TBE jel üzerinde çalıştırın.
9. DNA fragmantasyonu
- Örnek yeni bir tüp 50,5 µg aktarmak ve TLE arabellek 655 µL. Split örnek son bir birime ( Tablo malzemelerigörmek) 5 sonication tüpleri her şişe için Kütüphane (10 µg) 130 µL ekleyerek ekleyebilirsiniz. Kesme ~ 400 boyutuna bir ultra-sonicator kan basıncı ( Tablo malzemelerigörmek) aşağıdaki parametreleri kullanarak: görev faktörü: % 10; tepe olay gücü (w): 140; devir-de patlama: 200; Süresi: 55 s.
- Bir taze 2 mL tüp sonicated örnek toplamak.
10. çift taraflı SPRI-boncuk boyutu seçimi
- Mix SPRI (katı faz ters çevrilebilir immobilizasyon) boncuk çözüm de ters çevirme tarafından yeni bir tüp boncuk çözüm 1.85 mL transfer ve RT için 15 dakikadır getir.
- Su (Moleküler Biyoloji grade) 350 µL örneğe Ekle (son Hacim 1 mL).
- SPRI boncuk çözüm 600 µL örneğe Ekle (toplam hacmi 1.6 mL; DNA SPRI çözüm oranı: 0,6-1), RT, 5 min için kuluçkaya ve spin benchtop santrifüj örnek toplamak 2-3 sn için örnek.
- Kapağı açın, 5 dk, transfer yeni bir tüp açık süpernatant için manyetik ayırma stand örnek yerleştirin ve boncuk atmak.
- SPRI boncuk ikinci boyutu seçimi adım için konsantre: SPRI Transfer 930 µL yeni bir tüp boncuk, yer manyetik ayırma stand 5 min ve atma için net süpernatant. Boncuk SPRI boncuk çözüm 310 µL içinde yeniden askıya alma.
- Konsantre SPRI boncuk (adım 10,5) 300 µL örneğe Ekle (Toplam birim 1,9 mL; oranı SPRI DNA çözümdür şimdi 0,9-1), RT 5 min için kuluçkaya ve spin kapağı için 2-3 s. dikkatle açık bir benchtop santrifüj içinde örnek , 5 min için manyetik ayırma stand tüpü yerleştirin ve süpernatant atmak.
- Örnek tüp manyetik ayırma stand 1 mL taze hazırlanmış % 70 etanol (vol/vol) eklemek, 30 s ve atma süpernatant için kuluçkaya. İki kez tekrarlayın.
- Kuru boncuk bir thermomixer (tüp kapak açık) için en fazla 5 dk. eklemek 300 µL TLE arabelleğe örnek, mix, 37 ° C'de ve oda sıcaklığında 10 dakika için kuluçkaya.
- Örnek bir benchtop santrifüj için 2-3 sn içinde spin, kapağı açın ve tüp manyetik ayırma stand 5 dk. Transfer yer süpernatant yeni bir tüp temizleyin ve boncuk atmak.
11. biotin/Streptavidin Pull-down ligasyonu ürünleri
- Arabellekleri hazırlamak: 1 TB arabellek x (5mM Tris-HCl pH 8.0; %0,05 0,5 mM EDTA; 1 M NaCl ara 20); 2 x NTB arabellek (10 mM Tris-HCl pH 8.0; 1 mM EDTA; 2 M NaCl); 1 x NTB arabellek (5 mM Tris-HCl pH 8.0; 0,5 mM EDTA; 1 M NaCl).
- Manyetik streptavidin birleştiğinde boncuk ( Tablo malzemelerigörmek) 200 µL yeni bir tüp içine ekleyin, 1 dk. için manyetik ayırma stand yerleştirin ve süpernatant kaldırın.
-
Boncuk 1 x TB arabelleği 500 µL ile iki kez yıkayın.
- Her yıkama adımında biotin aşağı açılır, sonunda tamir ve Temizleme biotin DNA uçları olmayan bakmaksızın, dATP takip ve bağdaştırıcı ligasyonu adımları için boncuk karşılık gelen arabellekte yeniden askıya alma, RT ve 3 dk 15 dönüş/dak, döndürmek, benchtop santrifüj tüpü spin 2-3 s, 3 dk için manyetik ayırma stand tüpü yerleştirin ve süpernatant kaldırmak.
- Boncuk 2 x NTB arabellek 300 µL içinde yeniden askıya alma. Boncuk ve örnek (600 µL toplam hacim) mix ve 3 rpm'de dönen bir tekerlek üzerinde 15 dakika RT kuluçkaya.
- Boncuk manyetik ayırma stand 3 min için geri kazanmak ve açık süpernatant kaldırın. Boncuk iki kez 500 µL 1 x NTB arabellek içinde'ilk ve o zaman 1 x ligasyonu arabelleği 200 µL yıkayın. Boncuk 10 x ligasyonu arabellek 50 µL içinde yeniden askıya alma.
12. son onarım ve Temizleme biotin sigara bakmaksızın DNA ucunda
- Örnek (Toplam 50 µL) 2.5 mM dNTP mix (12.5 µL her dNTP 10 mm), 18.1 µL olan 3.000 U/mL T4 DNA polimeraz, 10.000 U/mL T4 PNK, 18.1 µL 50 µL ile birleştirmek 5000 U/mL DNA polimeraz 3.7 µL ben büyük (Klenow) parçalara ayırması ve H2o 360.1 µL
- Mix ve 1s 5 sallayarak için 20 ° C'de kuluçkaya s 700 RPM her 2 dakikada bir thermomixer içinde.
- Boncuk manyetik ayırma standında geri, açık süpernatant kaldırmak ve boncuk 1 TB arabellek x 500 µL iki kez yıkayın.
- 1 500 µL bir makinaya ardından 1 x NTB arabelleği 500 µL boncuk yıkayın x TLE.
- Boncuk manyetik ayırma standında geri, açık süpernatant kaldırmak ve boncuk 1 x TLE arabellek 415 µL içinde yeniden askıya alma.
13. dATP takip
- Örnek (415 µL) 10 kısıtlama tampon 2, 5 µL 10 mM dATP ve 5 U/µL Klenow exo-eksi 30 µL x 50 µL ile birleştirir.
- Mix ve 30 dk, 5 sallayarak için 37 ° C'de kuluçkaya s 700 RPM her 2 dakikada bir thermomixer içinde.
- Boncuk manyetik ayırma standında geri, açık süpernatant kaldırmak ve boncuk 1 TB arabellek x 500 µL iki kez yıkayın.
- Boncuk 1 x NTB arabellek 500 µL içinde yıkayın.
14. adaptör ligasyonu
- 1 tüp ligasyonu tepki arabellek x 200 µL boncuk yıkayın ( Tablo malzemelerigörmek).
- Boncuk 1 x ligasyonu tepki arabellek 200 µL içinde yeniden askıya alma. DNA ligaz 4 µL ekleyin ( Tablo malzemelerigörmek) ve 15 µM 16 µL önceden komplementer PE bağdaştırıcıları (ön tavlama PE bağdaştırıcıları tarafından eşit miktarda PE bağdaştırıcısı 1 ve 2 PE bağdaştırıcısı (hem 30 µM) karıştırma ve RT, bir kaç dakikalığına kuluçka). RT 15dk için kuluçkaya.
- Boncuk manyetik ayırma standında geri, açık süpernatant kaldırmak ve boncuk 1 TB arabellek x 500 µL iki kez yıkayın.
- Boncuk 1 x NTB arabellek 500 µL içinde yıkayın. 1 kısıtlama tampon 2, x 100 µL sonra yıkama boncuk boncuk 1 kısıtlama arabellek 2 x 50 µL yeniden askıya ve yeni bir tüp içine aktarın.
15. Merhaba-C Kütüphanesi amplifikasyon
- PCR ana karışım hazırlamak: 5 x Phusion arabelleği; 100 µL 6 µL 25 µM PE PCR astar 1.0; 6 µL 25 µM PE PCR astar 2.0; 14 µL dNTP Mix (10 mM her); Phusion polimeraz 6 µL; H2O. 318 µL
- Mix PCR ana karışımı (toplamda 500 µL), boncuk ile 50 µL 10 aliquots içinde bölmek ve aşağıdaki koşullar kullanarak PCR tarafından yükseltmek:
30'lu 98 ° c
7 devir: 10 s 98 ° c; 30 s 65 ° c; 30 s 72 ° c
7 dk. 72 ° c - PCR reaksiyonları yeni bir tüp içine toplamak, boncuk (500 µL) manyetik ayırma stand ve transfer süpernatant yeni bir tüpün içine geri kazanmak.
-
SPRI boncuk kullanarak Kütüphane DNA arındırmak.
- Mix SPRI boncuk, boncuk yeni bir tüp 460 µL transfer ve RT için getirmek için 15 dk. eklemek 450 µL PCR reaksiyonları (son hacim 950 µL) için SPRI boncuk RT, 5 min için kuluçkaya ve örnek bir benchtop santrifüj için örnek toplamak 2-3 sn içinde spin.
- Kapağı açın, manyetik ayırma stand 5 min için örnek yerleştirin ve süpernatant kaldırın.
- Boncuk manyetik ayırma kürsüye tutmak, 1 mL % 70 etanol (vol/vol) eklemek tüp boncuk açık alan üzerinde örnek, 30 s ve atma süpernatant için bırakın.
- 15.4.3 iki kez daha tekrarlayın.
- Boncuk bir thermomixer (tüp kapak açık) için en fazla 5 dk içinde 37 ° C'de kuru.
- Örnek için mix, 51 µL TLE arabelleği ekleyin ve bir thermomixer 950 rpm'de sallayarak 37 ° C'de 10 dakika için kuluçkaya.
- Örnek bir benchtop santrifüj için 2-3 sn içinde spin, kapağı açın ve tüp manyetik ayırma stand 5 dk. Transfer yer süpernatant yeni bir tüp temizleyin ve boncuk atmak.
- Hi-C Kütüphanesi konsantrasyonu ölçmek. PCR güçlendirme 7 tur sonra düzenli olarak 500-1500 ng Hi-C Kitaplığı edinin.
16. melez çözüm yakalama
Not: Engelleyici ve tampon (SHS1-4) çözümleri aşağıda olan SureSelect ( Tablo malzemelerigörmek) kit.
- Hi-C Kitaplığı yeni bir tüp içine 1 µg transfer 500 ng ve örnek bir vakum yoğunlaştırıcı buharlaşır (bkz: Malzemeler tablo; 45 ° C; vakum basıncı: düzey 30.0, rampa 5) kadar kuru.
- Buharlaştırılmış Hi-C Kütüphanesi 3.6 µL (Moleküler Biyoloji grade) H2O, kütük parçası 1, pencere engelleyicisi 2 2.5 µL ve özel pencere engelleyicisi 0.6 µL 2.5 µL ekleyerek yeniden askıya alma.
- Örnek yeni bir PCR tüp şerit bir kuyunun içine aktarmak, PCR kap şerit ile kapatın ve buza koyun. Etiket "D" (için Hi-C DNA) olarak.
- Hibridizasyon arabellek hazırlamak: 12.5 µL SHS1 arabelleği; SHS2 arabellek 0.5 µL; 5 µL SHS3 arabelleği; 6.5 µL SHS4 arabelleği.
- 5 dakika içinde bir thermomixer için 65 ° C'de kuluçkaya. Yeni bir PCR tüp şerit bir kuyunun içine aktarmak, PCR kap şerit ile kapatın ve RT. etiketini "H" (için hibridizasyon arabellek) bırakın.
- Yeni bir PCR tüp şerit bir kuyunun içine 100 ng/µL biotinylated 5 µL RNA problar (mağaza-80 ° C ve tezcan kullanmadan önce buzda); karıştırın. SRNase b (RNase inhibitörü) 0.5 µL ve 1,5 µL (Moleküler Biyoloji grade) H2O.
- PCR tüp şerit bir PCR kap şerit ve buz üzerinde yer ile kapatın. Etiket "R" (RNA) olarak.
- PCR makine aşağıdaki parametreleri kullanarak ayarlayın:
5 min 95 ° c; 25 h 65 ° c; ısıtmalı kapak; 29 µL PCR reaksiyon birim.
Not: PCR makinesi örnek buharlaşma önlemek için çalışırken mümkün olduğunca çabuk tüm işlemler sırasında devam edin. - "D" PCR tüp şerit PCR makinesinde yerleştirin, PCR makine kapağını kapatın ve PCR reaksiyon başlar. PCR program 65 ° C ulaştığında, PCR makine kapağını açın ve "H" PCR tüp şerit PCR makinesinde yerleştirin. PCR makine kapağını kapatın ve kuluçkaya 3 dk. açık PCR makinesi kapak, "R" PCR tüp PCR makinede şerit ve PCR makinesi yakın bir yer.
- 2 dakika sonra PCR makinesi kapağı ve tüm PCR tüp şeritler açın. 13 µL iyi "h" iyi "R", o zaman tüm cilt içine iyi "d" iyi "R" aktarın. Yukarı ve aşağı tepki karıştırmak için 3 kez damlalıklı yakın PCR tüp şerit, kaldır "H" ve "D" PCR şeritler tüp ve PCR makine kapağını kapatın. Tepki için 24 h 65 ° C'de kuluçkaya.
17. yalıtım organizatörü parçası içeren ligasyonu ürünleri
Not: Aşağıdaki adımları SureSelect adaptör kiti ve Kütüphane ile (bkz. Tablo Materals) yapılması tavsiye edilir.
- Yıkama arabelleği örnek 65 ° C'de 2 1.5 mL önceden önceden ısıtmak.
- Manyetik boncuklar streptavidin birleştiğinde ( Tablo malzemelerigörmek) 60 µL yeni bir tüp içine ekleyin, 1 dk. için manyetik ayırma stand üzerine yerleştirin ve süpernatant kaldırın.
- Boncuk üç kez 1 bağlayıcı arabelleğe x 200 µL ile yıkayın.
Not: çekim sonrası yalıtım organizatörü içeren ligasyonu ürünlerin sırasında her yıkama adım için boncuk karşılık gelen arabellekte yeniden askıya alma, 3 dk RT ve dönen bir tekerlek üzerinde 15 dönüş/dak için döndürmek, usulca toplamak 2-3 s benchtop santrifüj tüpü spin örnek, yer manyetik ayırma tüpü standı 3 dk ve Kaldır süpernatant için. - Boncuk 1 bağlayıcı arabelleğe x 200 µL yeniden askıya. PCR makinesi ve PCR tüp şerit (PCR programı hala çalışır durumdayken) açmak ve hibridizasyon tepki manyetik boncuklar ile tüp içine aktarın. RT 30 dk 3 rpm'de dönen bir tekerlek üzerinde kuluçkaya.
- Boncuk manyetik ayırma standında geri kazanmak ve açık süpernatant kaldırın. Boncuk yıkama tampon 1, mix, 500 µL yeniden askıya alma ve bir thermomixer 950 rpm'de sallayarak süre 20 ° C'de 15 dakika kuluçkaya.
- Boncuk manyetik ayırma stand 3 min için geri kazanmak ve açık süpernatant kaldırın. Boncuk yıkama arabellek 2 500 µL yeniden askıya alma, mix ve 65 ° C'de 10 dakika içinde bir thermomixer 950 rpm'de sallayarak süre kuluçkaya. 17,5 iki kez daha tekrarlayın.
- Boncuk manyetik ayırma standında geri, açık süpernatant kaldırmak ve boncuk 1 kısıtlama arabellek 2 x 200 µL yeniden askıya. Boncuk manyetik ayırma stand açmak, süpernatant kaldırmak ve boncuk 1 kısıtlama arabellek 2 x 30 µL içine yeniden askıya alma.
18. PCHi-C Kütüphanesi amplifikasyon
- PCR ana karışım hazırlamak: 5 x PCR arabelleği (Phusion arabellek) 60 µL, 25 µM PE PCR astar 1.0, 25 µM PE PCR astar 2.0, dNTP mix (10 mM her), Phusion polimeraz 3.6 µL ve H2o 190.8 µL 8.4 µL 3.6 µL 3.6 µL
- (Toplam 300 µL), boncuk ile mix PCR ana karışımı bölün ve 50 µL PCR yükseltmek-aşağıdaki koşullar kullanarak 6 aliquots:
30 s 98 ° c
4 döngüleri: 10 98 ° c, 30 s s 65 ° c, 30 s 72 ° c
7 dk. 72 ° c - Tüm PCR reaksiyonları yeni tüp içinde toplamak, mıknatıs üstündeki geri kazanmak ve süpernatant nakletmek (300 µL; PCHi-C kütüphanesi içerir) içinde yeni bir tüp.
- SPRI boncuk, küçük 15,4 açıklanan adımları kullanarak PCHi-C Kütüphanesi arındırmak.
- PCHi-C Kütüphanesi konsantrasyonu ölçmek.
Sonuçlar
Organizatörü yakalama Hi-C fare7,34,36,39 ve insan33,35,37,38 Hi-C kitaplıklar için zenginleştirmek için kullanılan organizatörü etkileşimler. (HiCap adlı) benzer bir iletişim kuralı tarafından Sandberg grubu40tarif edilmiştir. Şekil 1A şematik iş akışı organizatörü yakalama Hi-c için gösterir. Burada açıklanan iletişim kuralında, Hi-C kitaplıklar-çekirdek ligasyonu41sahte ligasyonu ürünleri42önemli ölçüde azaltılmış birkaç sonuç, kullanarak oluşturulur. PCHi-c, son derece karmaşık fare veya insan Hi-C kitaplıklar için çözüm hibridizasyon tabi olan ve 39,021 biotinylated RNA'ların 22,225 fare organizatörü içeren HindIII kısıtlama parçaları tamamlayıcı veya 37,608 biotinylated RNA'ları kullanarak yakalamak 22,076 insan organizatörü içeren HindIII kısıtlama parçaları, sırasıyla hedefleme. Kısıtlama parçaları içeren organizatörü biri veya her iki uçta da bireysel biotinylated RNA'ların (Şekil 1B) tarafından hedeflenebilir. Bulduğumuz her iki yakalama bireysel Rehberleri (Şekil 1 c; çiğ sıra okuma) neredeyse iki kat, geliştirilmiş kapsama beklendiği gibi sona erer. Böylece, ne zaman (biz tavsiye biotinylated RNA'ların her iki ucunda da bir kısıtlama parçası için tamamlayıcı yakalanması için kullanmak içinYani, tekrarlayan olmayan bölgelerde), mümkün.
PCHi-C Kitaplığı kalite Kütüphane hazırlık sırasında erken bir aşamada değerlendirmek için biz iki denetim DNA ligasyonu ve arıtma, yukarıda açıklanan31olarak sonra gerçekleştirmek. İlk tüp ligasyonu ürünler olduğu gibi 3 C27yükseltmek için belirli astar çiftleri kullanmaktır. Astar çiftleri (Tablo 1) hücre tipi sabit uzun menzilli ligasyonu ürünleri yükseltmek için kullanıyoruz, böyle Myc gen ve onun bilinen arttırıcılar yaklaşık 2 Mb uzakta (Şekil 2A) bulunan veya Hist1 odağı (genler arasında tarafından 1.5 Mb ayrılmış olarak) ve doğrusal yakın ('kısa menzilli denetimi') bulunan iki bölgeler arasında.
İkinci kalite kontrol Klenow-aracılı doldurma sırasında biotin birleşmesiyle kısıtlama sitesi çıkıntılar biotin-dATP ile verimliliğini belirlemek için yapılır. Başarılı Klenow doldurma ve ortadan kaybolması sonraki künt uçlu ligasyonu sonuçlarında arasında yeni bir NheI tanıma sitesi (Şekil 2B oluşumu DNA molekülleri HindIII durumunda ve bir tüp ligasyonu ürünün orijinal kısıtlama sitesinin ). HindIII NheI sindirilmiş ligasyonu ürün için biotin birleşme verimlilik doğrudan bir okuma oranıdır. Yüksek kaliteli kitaplıkları yakınındaki tamamlamak NheI sindirim ligasyonu ürünleri (Şekil 2B) var ise bir kalitesiz Hi-C Kütüphanesi HindIII sindirim, yüksek düzeyde gösterir.
Sonra (biotin streptavidin Hi-C ligasyonu ürün boyutu seçili, bağdaştırıcı ligasyonu ve yakalama öncesi PCR aşağı çekin sonraYani, ) Hi-C Kitaplığı hazırlık, Hi-C Kütüphanesi bütünlüğü ve boyut dağılımı (Şekil Bioanalyzer tarafından değerlendirilir 2 C). aynı denetimi PCHi-C Kitaplığı hazırlık (Yani, sonra hibridizasyon yakalama ligasyonu ürünleri organizatörü içeren ve çekim sonrası PCR) sonunda yapılır. Hi-C ve PCHi-C Bioanalyzer profilleri karşılaştırmasını gösterir beklendiği gibi Hi-C kitaplıklarıdır karşılık gelen PCHi-C kitaplıklar çok daha yoğun, ama belgili tanımlık kütüphaneci boyutu dağılımı son derece benzerdir yakalama adım gösteren PCHi-C (Şekil 2C, D) bir boyutu önyargı tanıtmak değildir.
Eşleştirilmiş uç sıralama sonra PCHi-C okuma eşleştirilir, kalite kontrol ve HiCUP boru hattı43kullanarak filtre. Yüksek kaliteli PCHi-C kitaplıkları içerir % 70-90 'geçerli çiftleri arasında' (Yani, eşleştirilmiş uç sıra okuma doğrusal genomik haritada; komşu değil iki kısıtlama parçaları arasında Şekil 3A, B). -Çekirdek ligasyonu protokol41,42kullanarak, trans yüzdesi (Yani, eşleştirilmiş uç sıra farklı kromozom üzerinde bulunan iki kısıtlama parçaları arasında okur) çiftleri genellikle düşüktür okumak, 5 ile % 25 arasında kromozom bölgelerin ve iyi Kütüphane kalite gösteren varlığını yansıtan. Doğrudan karşılaştırma 'geçerli çiftleri' Hi-C kitaplıklar ve onların karşılık gelen PCHi-C kitaplıklar35arasında yüzdesi tüm durumlarda geçerli çiftleri yüzdesini gösterir daha yüksek PCHi-C kitaplıklarda (3B rakam). Bu PCHi-C (Şekil 3 c) olarak geçerli olmayan 'aynı iç parçası' okuma yüzdesi bir azalma eşlik ediyor. Bu, beklenen yakalama adım sadece organizatörü içeren ligasyonu ürünler için zenginleştirir, ama aynı zamanda parçaları kısıtlama parçası amaçları için kısıtlama üzerinde yakalama oligos konumu nedeniyle (bkz. Şekil 1B) gibi.
Filtreleme HiCUP sonra yakalama verimliliği belirleriz. PCHi-C kitaplıkları geçerli sıralama okuma HiCUP süzme sonra üç tür içerir:
1.) Organizatörü: genom okur (Yani, yakalanan organizatörü parçası ve genom herhangi bir yerinde bir sigara organizatörü HindIII kısıtlama parçası arasında okuma)
2.) Organizatörü: organizatörü okur (iki yakalanan organizatörü parçaları arasında okuma)
3.) genom: genom okur (arka plan Hi-C ligasyonu ürünleri nerede ligasyonu ürün ortakları hiçbiri haritalar için yakalanan organizatör). Bunlar önce aşağı akım analizleri atılır.
Yüksek kaliteli PCHi-C kitaplıkları (3D şekil) % 65-90 arasında yakalama verimliliği (Kategori 1 ve 2 yukarıdaki toplamı) sahiptir. PCHi-C sonuçlanan Hi-C kitaplıklar için doğrudan bir karşılaştırma gösterir bir ~ 15-fold zenginleştirme organizatörü içeren ligasyonu ürünleri için (3D şekil), bazı durumlarda 17-fold. Bu varsayımsal maksimum yakın olduğunu (19.6-fold) PCHi-C, genom kısıtlama parçaları yüzdesi bağlıdır için zenginleştirme kaplı yakalama sistemi tarafından. Yakalama sistemleri daha az kısıtlama parçaları44,45,46hedefleme tasarlayarak büyük zenginlik elde edilebilir.
Hücre türü ve soy-özgüllük33,34,35, hücresel başkalaşım37,38,39 sırasında belirgin değişikliklerle organizatörü interactomes analizini gösterir . Rakamlar 4 ve 5 lineage özgüllük ve farklılaşma dinamiği belirli Rehberleri örnekler. Örneğin, ALAD yapısal tüm hücrelerde ifade edilir ancak onun ifade upregulated erythroblasts47olduğunu. ALAD düzenleyici tüm hematopoetik hücreler içinde birkaç distal parçaları kurar ve özellikle de erythroblasts (Şekil 4) ek etkileşimleri yürütmektedir. B hücreleri, T hücreleri, ama hücre tipi belirli etkileşimler de dahil olmak üzere monosit, nötrofil ve megakaryocytes () içinde miyeloid soyundan hücrelerdeki etkileşimleri onlarca çok az etkileşimlerde hiçbir istatistiksel olarak anlamlı etkileşimler Il-8 gösterir Şekil 5). Bu örnekler, PCHi-C hücre tipi belirli interactomes çözmeye ve düzenleyici potansiyeli ile organizatörü etkileşim bölgeleri belirlemek için nasıl kullanılabileceğini göstermektedir.
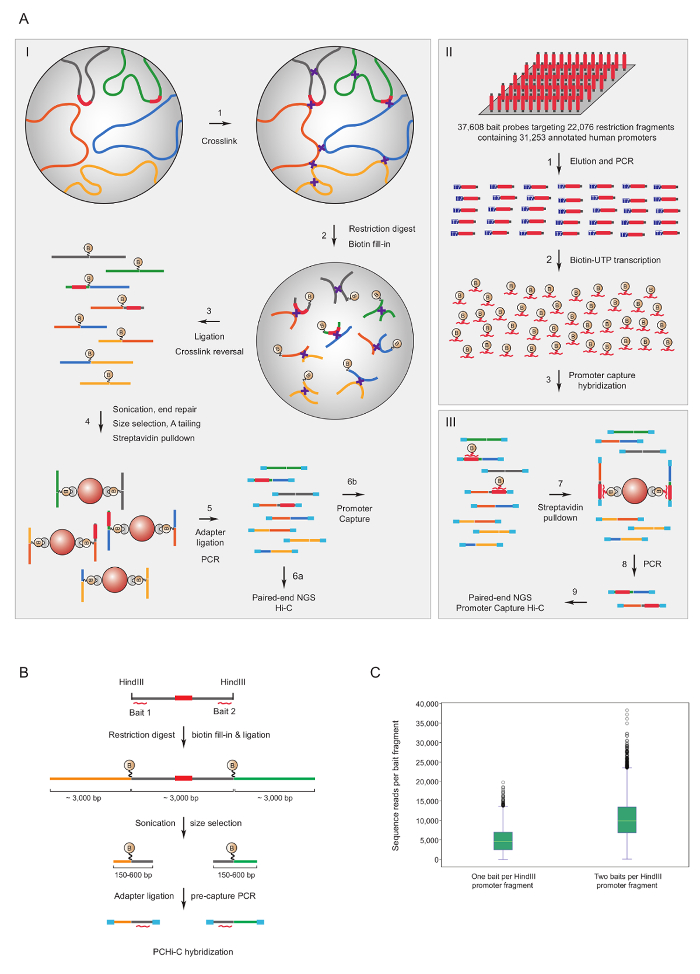
Resim 1 : Organizatörü yakalama Hi-C mantığı ve yakalama yem tasarım. PCHi-c (A) şematik iş akışı -Çekirdek ligasyonu Hi-C41,42 (I) biotinylated RNA yemler (II) ile çözüm hibridizasyon izledi (burada tasvir) insan kısıtlama parçaları hedefleme veya fare gen Rehberleri (III). (B) tasarım PCHi-c için yem. Biotinylated RNA yakalama yemler (kırmızı eğri çizgiler) Organizatörü içeren kısıtlama parçaları (gri; Eğer kısıtlama bulundukları organizatörü dizileri (kırmızı) kendilerini sadece RNA yakalama yemler tarafından hedeflenir Not biter karşı tasarlanmıştır parça biter). Sıra tamamlayıcılık hibridizasyon RNA yem ve DNA hedef arasındaki aracılığıyla izole ve sonraki organizatörü içeren kısıtlama parçaları (gri) ve onların etkileşen kısıtlama parçaları (sarı ve yeşil) oluşan ligasyonu ürünleri biotin streptavidin açılan, A. (C) karşılaştırma PCHi-C yakalama verimliliği organizatörü içeren kısıtlama parçaları için gösterildiği gibi iki RNA yem yakalama problar (şematik b bakınız) bir RNA yem yakalama sonda vs tarafından hedef. Bu rakam daha büyük bir versiyonunu görüntülemek için buraya tıklayınız.
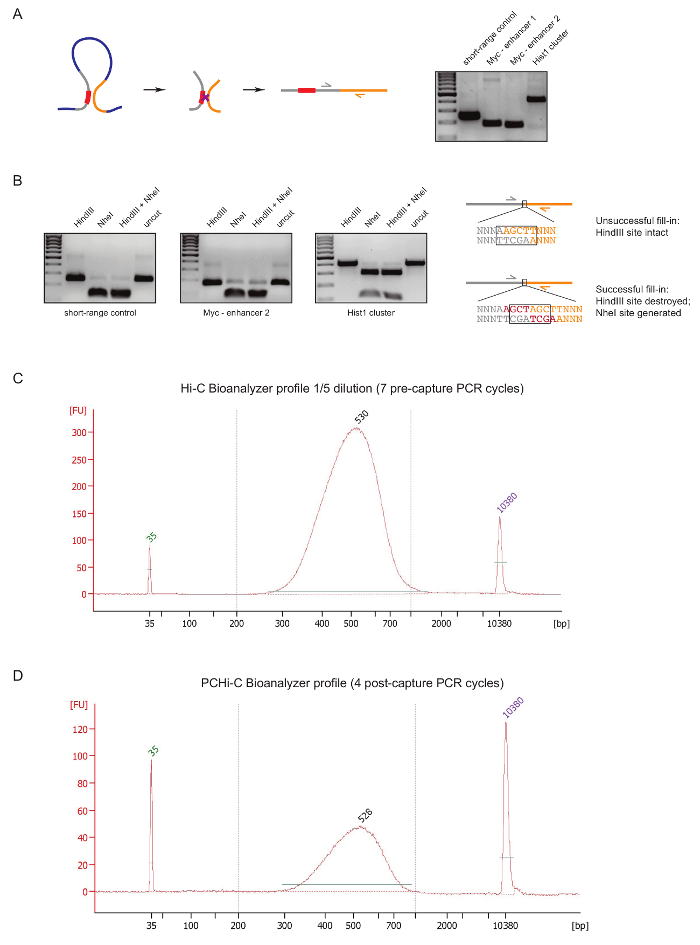
Resim 2 : PCHi-C öncesi sıralama kalite denetimleri. (A) sol, organizatörü ve PIR, bir organizatörü içeren kısıtlama parçası (gri; organizatörü sıra kırmızı) ve bir PIR kısıtlama parçası (sarı) oluşan bir Hi-C ligasyonu üründe sonuçlanan arasında kayma yan yana şematik. Doğru DNA (solda şematik tasvir) belirli astar çiftleri kullanılarak güçlendirilmiş Hi-C ligasyonu ürünlere örnekler gösterilen Elektroforez jel. (B) yaptı, HindIII, NheI ve HindIII/NheI kısıtlama temsilcisi örnekleri digests Hi-C ligasyonu ürünleri (A gösterilen PCR ürünleri). Doğru DNA'ın şematik sıra başarısız takip Hi-C ligasyonu sonra (üst) veya başarılı (alt) dNTP Klenow doldurularak kısıtlama kavşaklar ve sonraki ligasyonu. (C) temsilcisi Hi-C Kitaplığı bioanalyzer profil (1/5 seyreltme). (D) temsilcisi PCHi-C Kitaplığı bioanalyzer profil (seyreltme). Bu rakam daha büyük bir versiyonunu görüntülemek için buraya tıklayınız.
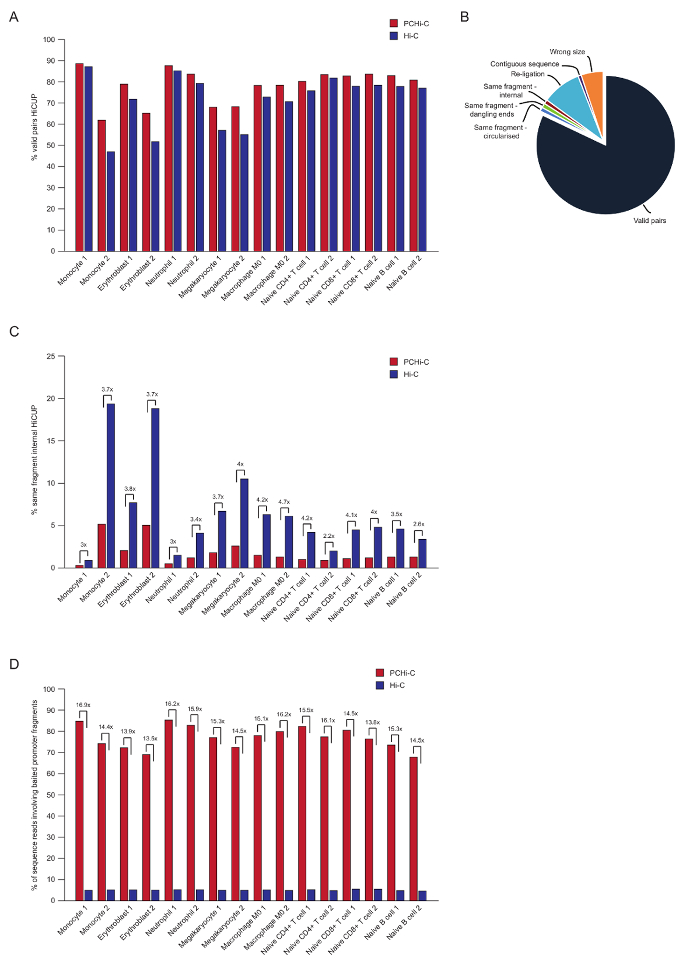
Şekil 3 : PCHi-C sonrası sıralama kalite denetimleri. (A) yüzde geçerli sıralama karşılaştırılması HiCUP43 Hi-C kitaplıklar (Javierre et al.verilerden, 201635) karşılık gelen PCHi-C vs içinde işleme sonra çiftleri okuyun. (B) temsilcisi HiCUP PCHi-C sonuç geçerli gösterilen çiftleri ve aşağı akım analizleri (Javierre et al.verilerden, 201635) önce atılır diğer sıralı Kategoriler okuyun. (C) HiCUP sonra PCHi-C vs Hi-C kitaplıklar (Javierre et al.verilerden, 201635) karşılık gelen işleme yüzdesi 'aynı parça iç' karşılaştırılması okur. (D) karşılaştırma yüzde sırası içeren baited organizatörü parçaları (yakalama verimliliği) PCHi-C vs Hi-C kitaplıklar (Javierre ve ark., 201635verileri) karşılık gelen okur. Bu rakam daha büyük bir versiyonunu görüntülemek için buraya tıklayınız.
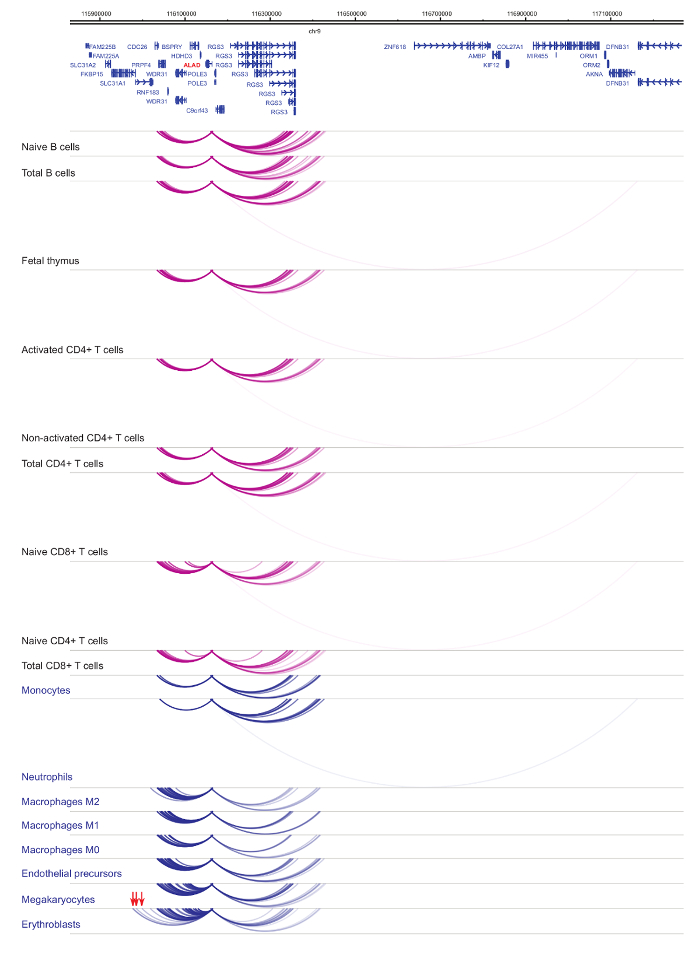
Şekil 4: ALAD PCHi-C profili insan hematopoetik hücrelerin. Organizatörü etkileşimleri myeloid hücre tiplerinin mavi kemer gösterilir ve organizatörü etkileşimleri lenfoid hücre tiplerinin mor kemer gösterilir. Erythroblast özel etkileşimleri (Javierre et al.verilerden, 201635) kırmızı oklarla belirtilmiştir. Bu rakam daha büyük bir versiyonunu görüntülemek için buraya tıklayınız.
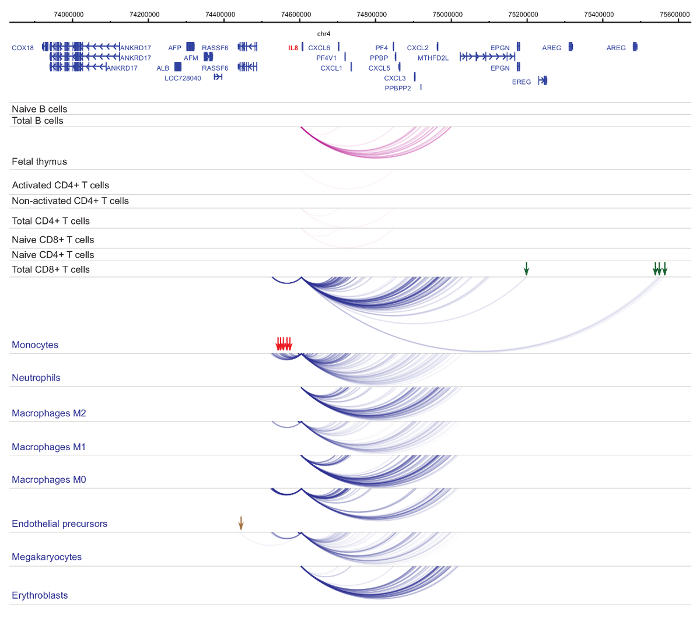
Şekil 5 : IL8 PCHi-C profili insan hematopoetik hücrelerin. Organizatörü etkileşimleri myeloid hücre tiplerinin mavi kemer gösterilir ve organizatörü etkileşimleri lenfoid hücre tiplerinin mor kemer gösterilir. Monosit özel etkileşimleri yeşil oklarla belirtilmiştir, nötrofil özel etkileşimleri kırmızı oklarla belirtilmiştir ve megakaryocyte özgü etkileşim kahverengi bir okla (Javierre et al.verilerden, 201635) belirtilir. Bu rakam daha büyük bir versiyonunu görüntülemek için buraya tıklayınız.
| İnsan | ||||||||
| Adı | Sıra | Kromozom | Strand | Başlangıç GRCh38/hg38 | Son GRCh38/hg38 | 3 C etkileşimleri ve biotin birleşme sınamak için astar bileşimleri | ||
| HS AHF64 Dekker | GCATGCATTAGCCTCTGCTGTTCTCTGAAATC | 11 | + | 116803960 | 116803991 | hs AHF66 Dekker ile birlikte kullanın | ||
| HS AHF66 Dekker | CTGTCCAAGTACATTCCTGTTCACAAACCC | 11 | + | 116810219 | 116810248 | hs AHF64 Dekker ile birlikte kullanın | ||
| HS MYC odağı | GGAGAACCGGTAATGGCAAA | 8 | - | 127733814 | 127733833 | hs MYC +1820 ile birlikte veya hs MYC-538 kullanmak | ||
| HS MYC +1820 | AAAATGCCCATTTCCTTCTCC | 8 | + | 129554527 | 129554547 | hs MYC odağı ile birlikte kullanın | ||
| HS MYC-538 | TGCCTGATGGATAGTGCTTTC | 8 | - | 127195696 | 127195716 | hs MYC odağı ile birlikte kullanın | ||
| HS HIST1 F | AAGCAGGAAAAGGCATAGCA | 6 | + | 26207174 | 26207193 | hs HIST1 R ile birlikte kullanın | ||
| HS HIST1 R | TCTTGGGTTGTGGGACTTTC | 6 | + | 27771575 | 27771594 | hs HIST1 F ile birlikte kullanın | ||
| Fare | ||||||||
| Sıra | Kromozom | Strand | Başlangıç GRCm38/mm10 | Son GRCm38/mm10 | 3 C etkileşimleri ve biotin birleşme sınamak için astar bileşimleri | |||
| TCATGAGTTCCCCACATCTTTG | 8 | + | 84841090 | 84841111 | mm Calr2 ile birlikte kullanın | |||
| CTGTGGGCACCAGATGTGTAAAT | 8 | + | 84848519 | 84848541 | mm Calr1 ile birlikte kullanın | |||
| TATCAAGGGTGCCCGTCACCTTCAGC | 6 | + | 125163098 | 125163123 | Gapdh4 Dekker ile birlikte kullanın | |||
| GGGCTTTTATAGCACGGTTATAAAGT | 6 | + | 125163774 | 125163799 | Gapdh3 Dekker ile birlikte kullanın | |||
| GGAGGAGGGAAAAGGAGTGATT | 6 | + | 52212829 | 52212850 | mm Hoxa13 ile birlikte kullanın | |||
| CAGGCATTATTTGCTGAGAACG | 6 | - | 52253490 | 52253511 | mm Hoxa7 ile birlikte kullanın | |||
| GGGTAATGGTGTCACTAACTGG | 13 | + | 23571284 | 23571305 | mm Hist1h3e veya mm Hist1h4i ile birlikte kullanın | |||
| GGGTTTGATGAGTTGGTGAAG | 13 | + | 23566541 | 23566561 | mm Hist1h2ae ile birlikte kullanın | |||
| TTGGGCCAAAGCCTATATGA | 13 | + | 22043085 | 22043104 | mm Hist1h2ae ile birlikte kullanın | |||
Tablo 1: Kalite kontrol insan için astar serileri ve fare Hi-C kitaplıklar.
Tartışmalar
Modüler tasarım organizatörü yakalama Hi-C
Organizatörü yakalama Hi-C özellikle Rehberleri ilgili etkileşimleri için Hi-C kitaplıklar zenginleştirmek için tasarlanmıştır. Bu etkileşimler yalnızca bir alt kümesini ligasyonu ürünleri bir Hi-C Kitaplığı'nda oluşturmaktadır.
Yakalama Hi-C yakalama sistemini değiştirerek herhangi bir genomik bölge ve bölgeler ilgi için Hi-C kitaplıklar zenginleştirmek için kolayca değiştirilebilir. Yakalama bölgeleri-ebilmek var olmak sürekli genomik kesimleri44,45,46,48, olmuştur arttırıcılar tespit PCHi-C (' ters yakalama Hi-C'35) veya DNaz ben aşırı duyarlı siteleri49 . Yakalama sisteminin boyutunu bağlı olarak deneysel kapsam ayarlanabilir. Örneğin, Dryden ve ark. meme kanseri44ile ilişkili üç gen çöller içinde 519 yem parçaları hedefleyin. Yakalama sistemi tarafından Martin ve ark. Her iki sürekli genomik segmentleri hedefleyen ('Bölge yakalama': Toplam 211 genomik bölgeleri; 2,131 kısıtlama parçaları) ve seçili Rehberleri (3,857 gen organizatör)45.
SureSelect kitaplıklarıdır farklı boyut aralıkları kullanılabilir: 1 kb ile 499 kb (5.190 – 4,806), 2.9 MB (5.190 – 4,816), 500 kb ve 3 Mb-5.9 Mb (5.190 – 4,831). Her tek tek yakalama biotin-RNA 120 nükleotit uzun olduğu için bu sistemleri yakalamak en fazla 4,158 karşılamak, 24,166 ve 49,166 tek tek yakalama probları, anılan sıraya göre. Bu 2.079, 12,083 ve 24,583 hedeflenen kısıtlama parçaları, sırasıyla karşılık gelen (kısıtlama parçaları için numaraları alt sınırlar iki tek tek yakalama problar her kısıtlama için dizayn edilebilir varsayımını temel olduğunu unutmayın parça — bu durum her kısıtlama için olmayacaktır nedeniyle tekrarlayan dizileri gerçekte (Ayrıca bkz: Şekil 1B, C) parçası, daha yüksek kullanılabilir yakalama probları sabit bir sayı için targetable kısıtlama parçalarının ortaya çıkmıştır ).
Burada açıklanan protokol Restriksiyon enzimi ile uzun menzilli etkileşimleri ortaya çıkarmak için 6 bp tanıma site kullanımı temel alır. Restriksiyon enzimi ile 4 bp tanıma site daha proksimal etkileşimleri büyük çözünürlük için de olası40,49kullanmaktır.
PCHi-C sınırlamaları
Bir doğal tüm kromozom conformation yakalama deneyleri onların çözünürlük kitaplığın oluşturulmasında kullanılan Restriksiyon enzimi tarafından belirlenir kısıtlamasıdır. Aynı kısıtlama parçası üzerinde bulunan DNA öğeleri arasındaki etkileşimleri 'C-type' deneyleri için görünmez olur. Ayrıca, PCHi-C, bazı durumlarda birden fazla transkripsiyon başlangıç site aynı organizatörü içeren kısıtlama parçası üzerinde bulunan ve PIRs bazı durumlarda her iki aktif ve baskıcı histon işaretleri hangi kesin olarak belirlemek zor yasal yapma liman öğeleri etkileşimleri, arabuluculuk ve organizatörü etkileşimlerin düzenleyici çıktı tahmin etmek. Enzimleri ile 4 bp tanıma siteleri kullanarak bu sorunu azaltır ama büyük ölçüde artan Hi-C Kitaplığı karmaşıklık pahasına gelir (4 bp tanıma sitesi enzimleri ile oluşturulan Hi-C kitaplıklarıdır Hi-C den en az 100 kat daha karmaşık Kitaplıklar) 6 bp tanıma sitesi enzimleri ile oluşturulan ve sonraki nesil sıralama için ilgili maliyetler.
Geçerli PCHi-C Protokolü malzeme, nadir hücre tipleri organizatörü etkileşimlerin analizi iyileştirmelerden başlangıç olarak hücreleri milyonlarca gerekir başka bir kısıtlamadır. Organizatörü kişiler'hücre popülasyonlarının 10.000 100.000 hücreleri (örneğin hücreleri hematopoetik kök hücre veya erken embriyonik gelişim sırasında) ile sorgulama etkinleştirmek için PCHi-C değiştirilmiş bir sürümü bu nedenle yakalama değerli bir ek olurdu Merhaba C araç.
Son olarak, üzerinde formaldehit tespit itimat tüm yöntemleri gibi PCHi-C yalnızca, Windows fiksasyon zaman noktasında 'dondurulmuş' etkileşimleri kaydeder. Böylece, kinetik ve organizatörü etkileşimleri dinamiklerini incelemek için Süper çözünürlük canlı cep mikroskobu gibi yöntemleri gerekli PCHi-c ile birlikte
Yöntemleri kayma kromozom organizasyon yüksek çözünürlükte incelemek için
Kromozom etkileşim kütüphanelerin büyük karmaşıklık istatistiksel anlamlılık ile iki belirli kısıtlama parçaları arasında etkileşim ürünlerin güvenilir kimlik yasaklar. Bu sorunu aşmak için sıra yakalama Hi-C33,34,40,44 veya belirli etkileşimler için 3 C50,51 kitaplıkları zenginleştirmek için kullanılmıştır. Hi-C kitaplıklar 3 C kitaplıklar için zenginleştirme adım kullanmanın en büyük avantajı Hi-C, hakiki ligasyonu ürünler için bir zenginleştirme adım 3 C kapsamaktadir. Sonuç olarak, geçerli okuma yüzdesi PCHi-C kütüphanelerde yaklaşık HiCUP süzme sonra yakalama-C'de kütüphaneler50civarında bulunan geçerli % 5-8, okur daha 10 kat yüksektir. Sahlen ve ark. doğrudan yakalama-C HiCap, kullandığı PCHi-C gibi Hi-C kitaplıklar için 3 C kitaplıklar kullanır yakalama-C aksine yakalama zenginleştirme ile karşılaştırıldığında. Bizim bulgular ile tutarlı, buldular yakalama-C kütüphaneler çoğunlukla un ve birleştirilmiş parçaları40/ oluşmaktadır. Ayrıca, yakalama-C kitaplıklar40daha yüksek bir karmaşıklık HiCap kütüphaneleri vardı.
Yakalama-C, yeni nesil yakalama-C52 (NG yakalama-C) adı verilen bir türevi olarak orijinal kullanılan problar örtüşen yerine PCHi-C33,34, daha önce kurulan kısıtlama parçası sonunda, başına bir oligo kullanır Yakalama-C Protokolü50. Bu mütevazi, yakalama-C'ye göre geçerli okuma yüzdesi artar ama NG yakalama-C istihdam yakalama zenginleştirme iki sıralı Tur ve PCR sayısının nispeten yüksek devir (20-24 döngüleri, toplam 11 döngüleri için PCHi-C için genellikle karşılaştırıldığında), hangi kaçınılmaz olarak daha yüksek sayıda sıra çoğaltmaları ve alt Kütüphane karmaşıklık sonuçlarında. PCHi-C en iyileştirme sırasında deneme deneylerde bulduk (değil çoğaltılamaz benzersizyani ) yüzdesini çiftleri sadece oldu yaklaşık %15 19 PCR döngüleri kullanıldığında okumak (yakalama 13 devir öncesi + 6 devir sonrası yakalama; verileri gösterilmez), ancak en iyi duruma getirme PCR döngüleri, daha küçük bir numarayla genellikle benzersiz okuma çiftleri % 75-90'ı verir. Böylece, PCR devir sayısını önemli ölçüde azaltarak bilgilendirici sıralı veri miktarını artırır.
Son yöntemi ChIP kromozom etkileşimleri faiz (HiChIP53) belirli bir protein tarafından aracılı odaklanmak için Hi-C ile birleştirir. Benzer mantığı üzerinde dayanır, çim54, karşılaştırıldığında HiChIP veri bilgilendirici sıra okuma,53çağrı güven derecesi yüksek etkileşim için izin daha yüksek bir sayıda içerir. Doğrudan karşılık gelen HiChIP karşılaştırmak çok ilginç olacak ve yakalama Hi-C veri kümeleri bir kez (bir antikor cohesin birim Smc1a karşı kullanarak HiChIP53 ile yakalama Hi-C tüm Smc1a için kısıtlama bağlı örnek için kullanılabilir parçaları) yan yana. Bu iki yaklaşım arasında bir doğal fark yakalama Hi-C kromatin immunoprecipitation dayanmaz ve bu nedenle protein doluluk bakılmaksızın kromozom etkileşimleri sorguya yapabiliyor olmasıdır. Bu gibi PRC1 fare ESC kayma genom mimari7anahtar bir düzenleyici tanımlamak için kullanılan belirli faktörü bağlayıcı olup 3D genom organizasyonu karşılaştırmasını sağlar.
PCHi-C ve GWAS
Genom çapında dernek çalışmaları (GWAS) % 95'den büyük hastalığı ile ilişkili olduğunu ortaya sıra türevleri büyük mesafelerde kez protein kodlayıcı genlerin55için genom kodlamayan bölgelerinde bulunur. GWAS türevleri çoğu zaman bulundu yakın DNaz ben aşırı duyarlı siteleri, hangi potansiyel yasal faaliyet sıralarıyla özelliğidir. PCHi-C ve yakalama Hi-C GWAS risk loci meme kanseri44, kolorektal kanser48ve otoimmün hastalığı35,45,46karıştığı Rehberleri bağlamak için yaygın olarak kullanılmaktadır. PCHi-C çalışma üzerinde 17 farklı insan hematopoietik hücre sıra türevleri trombosit ve kırmızı kan hücresi belirli özellikleri ile ilişkili ağırlıklı olarak bulundu, ancak türleri SNPs otoimmün hastalığı ile ilişkili lenfoid hücrelerde PIRs içinde zenginleştirilmiş bulundu makrofajlar ve erythroblasts, sırasıyla35,56. Böylece, doku belirli organizatörü PCHi-C tarafından ele geçen interactomes hastalık ilişkili kodlamayan işlevini anlamak için yardımcı olabilir türevleri sıra ve yeni potansiyel hastalık genleri terapötik müdahale için tanımlar.
Organizatörü etkileşim bölgeleri özellikleri
Kanıt bağlantı organizatörü interactomes gen ifade denetlemek için birkaç satırlık. İlk olarak, birçok PCHi-C araştırma (yüksek) ifade genlerin rehberleri ile etkileşim genomik bölgeler H3K27 asetilasyon ve p300 bağlama33,34 gibi artırıcı aktivitesiyle ilişkilendirilmiş işaretleri zenginleştirilmiş göstermiştir , 37. gen ifade düzey ve etkileşen arttırıcılar, artan gen ekspresyonu arttırıcılar sonucu artırıcı etkileri34,35düzeyleri düşündüren sayısı arasında pozitif bir korelasyon bulduk. İkinci olarak, doğal olarak meydana gelen Ifade eQTLs35tarafından etkilenen aynı genlere bağlı PIRs yılında niceliksel Özellik loci (eQTLs) zenginleştirilmiş ifade. Üçüncü olarak, gezi57 ' PCHi-C veri entegre ederek, Cairns vd. PIRs fare ESCs içinde eşleme gezi muhabir gen gen ekspresyonu reporter genler tümleştirme sitelerde daha organizatörü etkileşim bölgelerde daha güçlü muhabir göstermek bulundu PIRs transkripsiyon düzenleyici faaliyet sahip gösteren 58. Birlikte, bu bulgular PCHi-C tarafından çeşitli fare ve insan hücre türleri ele geçen organizatörü interactomes gen ifadesi denetim için anahtar yasal modülleri dahil öneririm.
Bu arttırıcılar yalnızca küçük bir bölümü (~ % 20) temsil eden tüm PIRs ortaya PCHi-C33tarafından,34, fazlalaştı. Diğer PIRs doğrudan transkripsiyon düzenleyici fonksiyonları yerine yapısal veya topolojik rolleri olabilir. Ancak, kanıt da PCHi-C klasik artırıcı işaretleri liman değil DNA öğeleri düzenleyici fonksiyonu ile ortaya çıkarmak. Bir insan lenfoid hücre kültürünü BRD7 organizatörü muhabir gen deneyleri33artırıcı faaliyete sahip gösterilmiştir bir bölge artırıcı işaretleri yoksun etkileşimli olarak bulundu. Benzer özelliklere sahip düzenleyici elemanlarının Şu anda takdir daha bol olabilir. Örneğin, gen ekspresyonu kontrol ancak artırıcı yoksun olan düzenleyici DNA öğeleri tanımlanan işaretsiz düzenleyici elemanlarının (UREs) için bir CRISPR tabanlı ekran59işaretler.
Diğer durumlarda, PIRs liman transkripsiyon baskı ile ilişkili kromatin işaretleri gösterilmiştir. PIRs ve PRC1 içinde fare ESCs bağlı etkileşen Rehberleri bastırılmış genleri taşıyan geniş bir kayma ağ baskıcı H3K27me37işaretlemek meşgul edildi. İnsan lymphoblastoid hücrelerde, uzak bir öğe BCL6 organizatörü ile etkileşim BCL6 transkripsiyon onun yerel bağlamda bastırmak için çalışabilir düşündüren transgene muhabir gen ifade33, baskı altında.
Doluluk insan ESCs ve NECs37 kromatin yalıtkan protein CTCF PIRs yeni bir sınıfı temsil edebilir için zenginleştirilmiş PIRs. Toplu olarak, bu sonuçlar PIRs gen düzenleyici faaliyetleri henüz işlevsel olarak karakterize edilebilir topluluğu liman öneririz.
Açıklamalar
Yazarlar ifşa gerek yok.
Teşekkürler
Valeriya Malysheva el yazması ve Şekil 1 ilgili uzman yardım eleştirel okuma için teşekkür ediyoruz. Bu eser tıbbi araştırma Konseyi, İngiltere (Bay/L007150/1) ve İngiltere'de Biyoteknoloji ve Biyolojik Bilimler Araştırma Konseyi, UK (1/J004480/BB) tarafından desteklenmiştir.
Malzemeler
| Name | Company | Catalog Number | Comments |
| 16% (vol/vol) paraformaldehyde solution | Agar Scientific | R1026 | |
| Dulbecco's Modified Eagle Medium (DMEM) 1x | Life Technologies | 41965-039 | |
| Fetal bovine serum (FBS) sterile filtered | Sigma | F9665 | |
| Low-retention filter tips | Starlab | S1180-3810, S1180-1810, S1180-8810 and S1182-1830 | |
| 10x PBS pH 7.4 | Life Technologies | 70011-036 | |
| Molecular biology grade water | Sigma-Aldrich | W4502 | |
| 1 M Tris-HCl pH 8.0 | Life Technologies | 15568-025 | |
| IGEPAL CA-630 | Sigma-Aldrich | I8896 | |
| 5 M NaCl | Life Technologies | 24740-011 | |
| Protease inhibitor cocktail (EDTA-free) | Roche Diagnostics | 11873580001 | |
| Restriction buffer 2 (10x NEBuffer 2) | New England Biolabs | B7002 | |
| DNA LoBind tube, 1.5 mL | Eppendorf | 0030 108.051 | |
| DNA LoBind tube, 2 mL | Eppendorf | 30108078 | |
| 20% (wt/vol) SDS | Bio-Rad Laboratories | 161-0418 | |
| 20% (vol/vol) Triton X-100 | Sigma-Aldrich | T8787 | |
| HindIII, 100 U/uL | New England Biolabs | R0104 | |
| 10 mM dCTP | Life Technologies | 18253-013 | |
| 10 mM dGTP | Life Technologies | 18254-011 | |
| 10 mM dTTP | Life Technologies | 18255-018 | |
| 0.4 mM Biotin-14-dATP | Life Technologies | 19524-016 | |
| DNA polymerase I large (Klenow) fragment 5000 units/mL | New England Biolabs | M0210 | |
| 10x T4 DNA ligase reaction buffer | New England Biolabs | B0202 | |
| 100x 10mg/ml Bovine Serum Albumin | New England Biolabs | B9001 | |
| T4 DNA ligase, 1 U/μL | Invitrogen | 15224-025 | |
| RNase A | Roche | 10109142001 | |
| Proteinase K, recombinant, PCR grade | Roche | 3115836001 | |
| 20 000×g 50 ml centrifuge tube | VWR | 525-0156 | |
| 0.5 M EDTA pH 8.0 | Life Technologies | 15575-020 | |
| Phenol pH 8.0 | Sigma | P4557 | |
| Phenol: Chloroform: Isoamyl Alcohol 25:24:1 | Sigma | P3803 | |
| Sodium acetate pH 5.2 | Sigma | S7899 | |
| Quant-iT PicoGreen | Invitrogen | P7589 | |
| QIAquick Gel Extraction Kit | Qiagen | 28704 | |
| QIAquick PCR Purification Kit | Qiagen | 28104 | |
| Restriction buffer 2.1 (10x NEBuffer 2.1) | New England Biolabs | B7202 | |
| NheI, 100U/uL | New England Biolabs | R0131 | |
| Micro TUBE AFA Fiber Pre-slit snap cap 6x16mm vials | Covaris | 520045 | For sonication |
| SPRI beads (Agencourt AMPure XP) | Beckman Coulter | A63881 | |
| Dynabeads MyOne Streptavidin C1 beads | Invitrogen | 65001 | |
| Tween 20 | Sigma | P9416 | |
| 10 mM dATP | Life Technologies | 18252-015 | |
| T4 DNA polymerase 3000 units/mL | New England Biolabs | M0203 | |
| T4 PNK 10000 units/mL | New England Biolabs | M0201 | |
| Klenow exo minus 5000 units/mL | New England Biolabs | M0212 | |
| Quick ligation reaction buffer | New England Biolabs | B6058 | |
| NEB DNA Quick ligase | New England Biolabs | M2200 | |
| PE adapter 1.0 (5'-P-GATCGGAAGAGCGGTTCAGC AGGAATGCCGAG-3') | Illumina | ||
| PE adapter 2.0 (5'-ACACTCTTTCCCTACACGACGCT CTTCCGATCT-3') | Illumina | ||
| NEB Phusion PCR kit | New England Biolabs | M0530 | |
| PE PCR primer 1.0 (5'-AATGATACGGCGACCACCGA GATCTACACTCTTTCCCTAC ACGACGCTCTTCCGATCT-3') | Illumina | ||
| PE PCR primer 2.0 (5'-CAAGCAGAAGACGGCATACGA GATCGGTCTCGGCATTCCT GCTGAACCGCTCTTCCGATCT-3') | Illumina | ||
| PCR strips | Agilent Technologies | 410022 and 401425 | |
| SureSelect SSEL TE Reagent ILM PE full adaptor kit | Agilent Technologies | 931108 | |
| SureSelect custom 3-5.9 Mb library | Agilent Technologies | 5190-4831 | custom design mouse or human PCHi-C system |
| Dynabeads MyOne Streptavidin T1 beads | Invitrogen | 65601 | |
| E220 high-performance focused ultra-sonicator | Corvaris | E220 |
Referanslar
- Osborne, C. S., et al. Active genes dynamically colocalize to shared sites of ongoing transcription. Nature Genetics. 36, 1065-1071 (2004).
- Schoenfelder, S., et al. Preferential associations between co-regulated genes reveal a transcriptional interactome in erythroid cells. Nature Genetics. 42, 53-61 (2010).
- de Wit, E., et al. The pluripotent genome in three dimensions is shaped around pluripotency factors. Nature. 501, 227-231 (2013).
- Bantignies, F., et al. Polycomb-dependent regulatory contacts between distant Hox loci in Drosophila. Cell. 144, 214-226 (2011).
- Engreitz, J. M., et al. The Xist lncRNA exploits three-dimensional genome architecture to spread across the X chromosome. Science. 341, 1237973(2013).
- Denholtz, M., et al. Long-range chromatin contacts in embryonic stem cells reveal a role for pluripotency factors and polycomb proteins in genome organization. Cell Stem Cell. 13, 602-616 (2013).
- Schoenfelder, S., et al. Polycomb repressive complex PRC1 spatially constrains the mouse embryonic stem cell genome. Nature Genetics. 47, 1179-1186 (2015).
- Kundu, S., et al. Polycomb Repressive Complex 1 generates discrete compacted domains that change during differentiation. Molecular Cell. 65, 432-446 (2017).
- Skok, J. A., Gisler, R., Novatchkova, M., Farmer, D., de Laat, W., Busslinger, M. Reversible contraction by looping of the Tcra and Tcrb loci in rearranging thymocytes. Nature Immunology. 8, 378-387 (2007).
- Zhang, Y., et al. Spatial organization of the mouse genome and its role in recurrent chromosomal translocations. Cell. 148, 908-921 (2012).
- Aymard, F., et al. Genome-wide mapping of long-range contacts unveils clustering of DNA double-strand breaks at damaged active genes. Nature Structural & Molecular Biology. 24, 353-361 (2017).
- Ryba, T., et al. Evolutionarily conserved replication timing profiles predict long-range chromatin interactions and distinguish closely related cell types. Genome Research. 20, 761-770 (2010).
- Pope, B. D., et al. Topologically associating domains are stable units of replication-timing regulation. Nature. 515, 402-405 (2014).
- Chandra, T., et al. Global reorganization of the nuclear landscape in senescent cells. Cell Reports. 10, 471-483 (2015).
- Carter, D., Chakalova, L., Osborne, C. S., Dai, Y. F., Fraser, P. Long-range chromatin regulatory interactions in vivo. Nature Genetics. 32, 623-626 (2002).
- Tolhuis, B., Palstra, R. J., Splinter, E., Grosveld, F., de Laat, W. Looping and interaction between hypersensitive sites in the active beta-globin locus. Molecular Cell. 10, 1453-1465 (2002).
- Amano, T., Sagai, T., Tanabe, H., Mizushina, Y., Nakazawa, H., Shiroishi, T. Chromosomal dynamics at the Shh locus: limb bud-specific differential regulation of competence and active transcription. Developmental Cell. 16, 47-57 (2009).
- Zuniga, A., et al. Mouse limb deformity mutations disrupt a global control region within the large regulatory landscape required for Gremlin expression. Genes & Development. 18, 1553-1564 (2004).
- Sagai, T., Hosoya, M., Mizushina, Y., Tamura, M., Shiroishi, T. Elimination of a long-range cis-regulatory module causes complete loss of limb-specific Shh expression and truncation of the mouse limb. Development. 132, 797-803 (2005).
- D'Haene, B., et al. Disease-causing 7.4 kb cis-regulatory deletion disrupting conserved non-coding sequences and their interaction with the FOXL2 promotor: implications for mutation screening. PLoS Genet. 5, e1000522(2009).
- Sur, I. K., et al. Mice lacking a Myc enhancer that includes human SNP rs6983267 are resistant to intestinal tumors. Science. 338, 1360-1363 (2012).
- Herranz, D., et al. A NOTCH1-driven MYC enhancer promotes T cell development, transformation and acute lymphoblastic leukemia. Nature Medicine. 20, 1130-1137 (2014).
- Deng, W., et al. Controlling long-range genomic interactions at a native locus by targeted tethering of a looping factor. Cell. 149, 1233-1244 (2012).
- Groschel, S., et al. A single oncogenic enhancer rearrangement causes concomitant EVI1 and GATA2 deregulation in leukemia. Cell. 157, 369-381 (2014).
- Lupianez, D. G., et al. Disruptions of topological chromatin domains cause pathogenic rewiring of gene-enhancer interactions. Cell. 161, 1012-1025 (2015).
- Franke, M., et al. Formation of new chromatin domains determines pathogenicity of genomic duplications. Nature. 538, 265-269 (2016).
- Dekker, J., Rippe, K., Dekker, M., Kleckner, N. Capturing chromosome conformation. Science. 295, 1306-1311 (2002).
- Simonis, M., et al. Nuclear organization of active and inactive chromatin domains uncovered by chromosome conformation capture-on-chip (4C). Nature Genetics. 38, 1348-1354 (2006).
- Zhao, Z., et al. Circular chromosome conformation capture (4C) uncovers extensive networks of epigenetically regulated intra- and interchromosomal interactions. Nature Genetics. 38, 1341-1347 (2006).
- Dostie, J., et al. Chromosome Conformation Capture Carbon Copy (5C): A massively parallel solution for mapping interactions between genomic elements. Genome Research. 16, 1299-1309 (2006).
- Lieberman-Aiden, E., et al. Comprehensive mapping of long-range interactions reveals folding principles of the human genome. Science. 326, 289-293 (2009).
- Belton, J. M., McCord, R. P., Gibcus, J. H., Naumova, N., Zhan, Y., Dekker, J. Hi-C: a comprehensive technique to capture the conformation of genomes. Methods. 58, 268-276 (2012).
- Mifsud, B., et al. Mapping long-range promoter contacts in human cells with high-resolution capture Hi-C. Nature Genetics. 47, 598-606 (2015).
- Schoenfelder, S., et al. The pluripotent regulatory circuitry connecting promoters to their long-range interacting elements. Genome Res. 25, 582-597 (2015).
- Javierre, B. M., et al. Lineage-specific genome architecture links enhancers and non-coding disease variants to target gene promoters. Cell. 167, 1369-1384 (2016).
- Wilson, N. K., et al. Integrated genome-scale analysis of the transcriptional regulatory landscape in a blood stem/progenitor cell model. Blood. 127, e12-e23 (2016).
- Freire-Pritchett, P., et al. Global reorganisation of cis-regulatory units upon lineage commitment of human embryonic stem cells. Elife. 6, (2017).
- Rubin, A. J., et al. Lineage-specific dynamic and pre-established enhancer-promoter contacts cooperate in terminal differentiation. Nature Genetics. 49, 1522-1528 (2017).
- Siersbaek, R., et al. Dynamic rewiring of promoter-anchored chromatin loops during adipocyte differentiation. Molecular Cell. 66, 420-435 (2017).
- Sahlen, P., et al. Genome-wide mapping of promoter-anchored interactions with close to single-enhancer resolution. Genome Biology. 16, 156(2015).
- Nagano, T., et al. Single-cell Hi-C reveals cell-to-cell variability in chromosome structure. Nature. 502, 59-64 (2013).
- Nagano, T., Varnai, C., Schoenfelder, S., Javierre, B. M., Wingett, S. W., Fraser, P. Comparison of Hi-C results using in-solution versus in-nucleus ligation. Genome Biology. 16, 175(2015).
- Wingett, S., et al. HiCUP: pipeline for mapping and processing Hi-C data. F1000 Res. 4, 1310(2015).
- Dryden, N. H., et al. Unbiased analysis of potential targets of breast cancer susceptibility loci by Capture Hi-C. Genome Research. 24, 1854-1868 (2014).
- Martin, P., et al. Capture Hi-C reveals novel candidate genes and complex long-range interactions with related autoimmune risk loci. Nature Communications. 6, 10069(2015).
- McGovern, A., et al. Capture Hi-C identifies a novel causal gene, IL20RA, in the pan-autoimmune genetic susceptibility region 6q23. Genome Biol.ogy. 17, 212(2016).
- Hodge, D., et al. A global role for EKLF in definitive and primitive erythropoiesis. Blood. 107, 3359-3370 (2006).
- Jager, R., et al. Capture Hi-C identifies the chromatin interactome of colorectal cancer risk loci. Nature Communications. 6, 6178(2015).
- Joshi, O., et al. Dynamic reorganization of extremely long-range promoter-promoter Interactions between two states of pluripotency. Cell Stem Cell. 17, 748-757 (2015).
- Hughes, J. R., et al. Analysis of hundreds of cis-regulatory landscapes at high resolution in a single, high-throughput experiment. Nature Genetics. 46, 205-212 (2014).
- Kolovos, P., et al. Targeted Chromatin Capture (T2C): A novel high-resolution high-throughput method to detect genomic interactions and regulatory elements. Epigenetics Chromatin. 7, 10(2014).
- Davies, J. O., et al. Multiplexed analysis of chromosome conformation at vastly improved sensitivity. Nature Methods. 13, 74-80 (2016).
- Mumbach, M. R., et al. HiChIP: efficient and sensitive analysis of protein-directed genome architecture. Nature Methods. 13, 919-922 (2016).
- Fullwood, M. J., et al. An oestrogen-receptor-alpha-bound human chromatin interactome. Nature. 462, 58-64 (2009).
- Maurano, M. T., et al. Systematic localization of common disease-associated variation in regulatory DNA. Science. 337, 1190-1195 (2012).
- Petersen, R., et al. Platelet function is modified by common sequence variation in megakaryocyte super enhancers. Nat. Commun. 8, 16058(2017).
- Akhtar, W., et al. Chromatin position effects assayed by thousands of reporters integrated in parallel. Cell. 154, 914-927 (2013).
- Cairns, J., et al. CHiCAGO: Robust detection of DNA looping interactions in Capture Hi-C data. Genome Biology. 17, 127(2016).
- Rajagopal, N., et al. High-throughput mapping of regulatory DNA. Nature Biotechnology. 34, 167-174 (2016).
Yeniden Basımlar ve İzinler
Bu JoVE makalesinin metnini veya resimlerini yeniden kullanma izni talebi
Izin talebiThis article has been published
Video Coming Soon
JoVE Hakkında
Telif Hakkı © 2020 MyJove Corporation. Tüm hakları saklıdır