Method Article
Промоутер захват Привет C: С высоким разрешением, геном общесистемной Профилирование взаимодействия промоутер
* Эти авторы внесли равный вклад
В этой статье
Резюме
ДНК нормативных элементов, таких как усилители, контроль экспрессии генов связавшись физически целевой генным стимуляторам, часто через большой дальности хромосомных взаимодействия, охватывающих большие расстояния геномной. Промоутер захватить Hi-C (PCHi-C) идентифицирует значимых взаимодействий между промоутеров и дистальной регионами, позволяя назначение потенциал регулирования последовательностей для их целевых генов.
Аннотация
Трёхмерный Организации генома связано с ее функции. Например нормативных элементов, таких как транскрипционный анализ усилители управляют пространственно временных выражение их целевых генов через физический контакт, часто преодоление значительно (в некоторых случаях сотни kilobases) геномных расстояния и обход близлежащих генов. Геном человека затаивает примерно один миллион усилители, подавляющее большинство из которых имеют неизвестные цели ген. Присвоение их генов-мишеней дистальной регулирования регионах важно таким образом понять управления выражения гена. Мы разработали промоутер захватить Hi-C (PCHi-C), чтобы включить обнаружение генома общесистемной дистальной промоутер взаимодействующих областей (Пирс), для всех промоутеров в одном эксперименте. В PCHi-C весьма сложные Hi-C библиотек специально обогащены промоутер последовательностей подборку в решение для совместной работы с тысячами биотинилированным РНК приманки дополняет концы всех промоутер содержащих ограничение фрагментов. Цель состоит в том, затем потяните вниз промоутер последовательностей и их частое взаимодействие партнеров таких усилителей и других потенциальных регуляторных элементов. После высокой пропускной способности в паре конец последовательности, статистический тест применяется для каждого фрагмента промоутер лигируют ограничений для выявления значительных Пирс на уровне фрагмент ограничение. Мы использовали PCHi-C для создания атласа на большие расстояния промоутер взаимодействий в десятки человека и типы клеток мыши. Эти карты interactome промоутер способствовали более глубокому пониманию млекопитающих ген выражение управления присвоения предполагаемого регулирования регионов их генов-мишеней и выявление преференциальных пространственных промоутер промоутер взаимодействия сетей. Эта информация также имеет большое значение для понимания генетических болезней человека и выявление потенциальных болезней генов, связывая некодирующих болезни связанные последовательности вариантов или поблизости управляющие последовательности для их целевых генов.
Введение
Накопление доказательств свидетельствует о том, что трехмерной Организации генома играет важную функциональную роль в диапазоне ядерных процессов, в том числе генов активации1,2,3, репрессии4 ,5,6,7,8, рекомбинация9,10, ДНК ремонт11, репликации ДНК12,13, и 14клеточного старения. Далекие усилители находятся в пространственной близости к промоутеров, что они регулируют15,16,17, которая необходима для надлежащего пространственно временных ген выражение элемента управления. Усилитель удаления показывают, что дистальной усилители важны для целевого гена транскрипции18,19,20,21,22и «вынуждены хроматина цикла» показывает что инженерных привязывая между enhancer и его целевой промоутер в локусе ГБД является достаточным для привода transcriptional активации23. Кроме того перестановки генома, которые приносят гены под контролем внематочная усилители может привести к неуместным генов активации и болезни24,25,26. Вместе эти примеры иллюстрируют, что промоутер усилитель взаимодействия необходимы для управления гена и требуют жесткой регулирования для обеспечения соответствующего гена выражение. Человека и мыши геномов каждого оцениваются в гавани около одного миллиона усилители. Для подавляющего большинства этих усилителей генов-мишеней неизвестны, и плохо понимаются «правила применения вооруженной силы» между промоутеров и усилители. Таким образом назначение transcriptional усилители для их целевых генов остается серьезной проблемой в расшифровке млекопитающих ген выражение элемента управления.
Наше понимание архитектуры трехмерной генома революционизировал введение 3C27 (хромосома конформации захвата) и его варианты28,,2930,31 . Самый мощный из этих методов, Hi-C (высокая пропускная способность хромосома конформации захват) предназначена для идентификации весь ансамбль хромосомных взаимодействий в популяции клеток. Привет-C библиотек, обычно создается из миллионов клеток, являются весьма сложными с приблизительно 1011 независимых лигирование продуктов между фрагментами ~ 4 kb в геноме человека32. Как следствие, надежных и воспроизводимых определение взаимодействий между отдельными ограничение фрагменты (например, содержащие промоутер или усилитель) от Hi-C данных невозможно, если библиотеки Hi-C подвергаются Ультра глубокое последовательности, которая не является экономически жизнеспособным решением для лабораторий, регулярно готовит Hi-C библиотек. Чтобы обойти этот недостаток, мы разработали промоутер захватить Hi-C специально обогатить промоутер содержащие продукты лигирование от Hi-C библиотек. Мы сосредоточились на промоутеров по двум причинам. Во-первых промоутер усилитель контактов было показано, чтобы иметь решающее значение для надлежащего ген выражение уровней в многочисленных исследованиях (см. ссылки выше), и во-вторых, как промоутеров во многом инвариантны между типами клеток, одной и той же системы приманки захвата может использоваться для допросов регулирования цепь на нескольких типов клеток и условий. Наш подход опирается на решение гибридизации Hi-C библиотек с десятками тысяч биотинилированным РНК 120mers дополняет промоутер содержащих продуктов перешнуровки Hi-C и последующего захвата на стрептавидина покрытием магнитные бусы. Это приводит к PCHi-C библиотек с значительно снижена сложность по сравнению с исходной библиотеке Hi-C, сосредоточив внимание только на идентификации фрагментов, которые лигируют промоутеров на значительно высоких частотах.
Мы использовали PCHi-C в типы клеток мыши для содействия более глубокому пониманию управления выражение гена, раскрытие дальнего дистальной промоутер взаимодействующих регионах с предполагаемым функции регулирования и ряд человека, а также не случайные Промоутер промоутер контактов в трехмерном пространстве ядра. Эти исследования сопоставлены сотни тысяч промоутер усилитель контактов через многочисленные ячейки типы33,34,35,36,,3738, 39, определены Организации Polycomb репрессивных комплекс-опосредованной пространственных генома мыши эмбриональные стволовые клетки7, продемонстрировали масштабный переоснащая промоутер interactomes в клеточной дифференциации37, 38 , 39и связанных некодирующих болезни связанные последовательности вариантов гена промоутеров35.
PCHi-C является метод идеально подходит для карты генома общесистемной ансамбль последовательностей ДНК, взаимодействующих с промоутерами. Соответствующих подходов, таких как захват Привет-C непрерывного геномной регионов (см. обсуждение) являются методом выбора для получения высокого разрешения взаимодействия профили для выбранных регионах геномной. PCHi-C и захватить Hi-C чрезвычайно схожи с экспериментальной точки зрения (единственное различие является выбор системы захвата), так что рекомендации и руководящие принципы, мы предоставляем применимы для обоих подходов. Здесь мы представляем детальное описание PCHi-C. Мы изложить обоснование и дизайн PCHi-C эксперимента, предоставляют пошаговые протокол поколения библиотека PCHi-C и иллюстрируют, как качество библиотек PCHi-C может контролироваться на различных этапах в протоколе для получения качественных данных.
протокол
1. формальдегид фиксации
-
Сотовый подготовка: Начните с минимум 2 x 107 ячеек на эксперимент.
- Для ячеек, выросли в культуре Ресуспензируйте клетки в культурной среде. Для ex vivo клеток Ресуспензируйте в 1 x Дульбекко изменение орел среднего (DMEM), дополнены 10% (vol/vol) плода бычьим сывороточным (ФБС).
- Для адэрентных клеток удалите питательной среды и добавьте 30.625 мл свежей среды с 10% (vol/vol) FBS при комнатной температуре (RT; 20-25 ° C).
- Для подвески клеток собирать и центрифуги клетки на 400 x g и 20 ° C для 3 мин удалить супернатант и вновь приостановить Пелле клеток в 30.625 мл среды с 10% (vol/vol) FBS в рт.
- Для твердых тканей используйте трипсина (0,05% до 2,5% концентрации выпускных экзаменов, в зависимости от типа клеток) или dounce, гомогенизации получить одну ячейку подвеска. После этого дополнительного шага лечить клетки как подвеска клетки.
-
Добавления 4.375 мл метанола бесплатно параформальдегида 16% (открытые ампуле только до использования) до конечной концентрации 2% (vol/vol). Исправление для 10 мин на RT с нежным смешивания на рокер.
Предупреждение: Параформальдегида является опасных химических веществ. Соблюдайте соответствующие правила здоровья и безопасности. - Утолите реакции, добавив 5 мл свежеприготовленные 1 M ледяной глицина. Смешайте 5 мин с плавное покачивание на RT и затем инкубировать на льду на 15 мин с периодически переворачивать.
-
Вымойте и собирать фиксированных ячеек.
- Для адэрентных клеток удалить супернатант, добавить 10 мл ледяной 1 x PBS рН 7,4 на стену плиты и удалите его. Добавьте 1 mL ледяной 1 x PBS рН 7,4, собирать клеток с использованием клеток скребок и передачи в 50 мл трубки. Повторите дважды, чтобы собрать как многие клетки как можно скорее. Добавьте ледяной PBS до окончательный объем 50 мл.
- Для подвески клетки клетки центрифуги на 760 x g и 4 ° C за 5 мин, удалить супернатант и вновь приостановить Пелле клеток в 50 мл ледяной PBS рН 7,4.
- Центрифуга клетки на 400 x g и 4 ° C на 10 минут и тщательно удалить супернатант. Пелле клетки может быть оснастки заморожены в жидком азоте и впоследствии хранятся при температуре-80 ° C в течение нескольких месяцев.
2. Lysis клетки
- Вновь приостановить Пелле клеток в 50 мл-свежеприготовленные ледяной литического буфера (10 мм трис-HCl рН 8, 0,2% (vol/vol) Igepal CA-630, 10 мм NaCl и одна таблетка ингибитор протеазы коктейль) и перемешать. Инкубировать на льду для 30 минут, изредка смешивать, инвертирование. Центрифуга ядер на 760 x g и 4 ° C на 5 мин и удалить супернатант.
3. ХинdIII пищеварение
- Вымойте клеточных ядер с ограничением буфера 1,25 x 2. Вновь приостановите Пелле клеток в 1 мл ледяной 1,25 x ограничение буфера 2 и передачи в 1,5 мл трубку. Спина ядер на 760 x g и 4 ° C на 5 мин и удалить супернатант.
- Вновь приостановите Пелле клеток в 1790 мкл ограничение буфера 1,25 x 2. Сделайте 5 аликвоты, каждый из которых содержит 5-10 миллионов клеток в 358 мкл 1,25 x ограничение буфера 2.
- Мкл 11 10% (wt/vol) SDS Алиготе и трясти на 950 оборотов в минуту (об/мин) для 30 минут при 37 ° C в thermomixer. Если появляются скопления клеток, разъединять, дозирование, избегая пузыри.
- 75 мкл 10% Тритон X-100 (vol/vol) за Алиготе и сотрясения при 37 ° C в течение 15 мин в thermomixer 950 об/мин. Если появляются скопления клеток, разъединять, дозирование, избегая пузыри.
-
Мкл 12 100 U/мкл ХинdIII 100 (1200 единиц в общей сложности) за Алиготе и инкубировать при 37 ° C ночь (O/N) при встряхивании в 950 об/мин в thermomixer.
- Для управления пищеварение передача 25 мкл пример (5 мкл от каждого Алиготе) в новой трубки перед добавлением энзима (непереваренных управления) и повторить ту же процедуру после добавления фермента (переваривается управления). Инкубируйте обеих трубок, так же, как библиотека Hi-C.
- На следующее утро 5 мкл 100 U/мкл ХинdIII (500 единиц в общей сложности) за Алиготе и инкубировать при 37 ° C втечение 2 ч при встряхивании в 950 об/мин в thermomixer.
-
Пищеварение управления: для перевариваются и непереваренных элементов управления (см. 3.5.1), выполняют crosslink разворота (шаг 6), извлечение фенола: хлороформ и ДНК осадков (шаг 7).
- Дизайн пары праймеров, которые охватывают ХинdIII сайта. В том же регионе дизайн другой пары праймеров, которые не охватывают ХинdIII сайта. Дизайн праймеров для количественного PCR (Q-ПЦР) с помощью Primer3 (http://bioinfo.ut.ee/primer3-0.4.0/) и следующие параметры:
Грунтовка размер: оптимальный 20 (мин: 18, Макс.: 27); Праймер Tm: Оптимальный 60 (мин: 57, Макс.: 63); Грунтовка CG % содержание: мин.: 20, Макс.: 80; Ампликон размер: RT-PCR ~ 100 bp (для обычных ПЦР ~ 300 bp); Mispriming библиотека: человека (человека грунтовки) или грызун и простой (мыши грунты). - Выполнять Q-ПЦР для получения 4 среднее Cts (порог цикл): Ct [D; H], полученные из переваренной образца [D] с парой праймеров, которые охватывают ХинdIII сайта [H]; CT [D;-], полученные из переваренной образца [D] с парой праймеров, которые не охватывают ХинdIII сайта [-]; CT [U; H], полученные из непереваренной образца [U] с парой праймеров, которые охватывают ХинdIII сайта; CT [U;-], полученные из непереваренной образца [U] с парой праймеров, которые не охватывают ХинdIII сайта [-]. Рассчитать процент пищеварения как: пищеварение % = 100-100/2(Ct[D,H]-Ct[D,-]) - (Ct[U,H]-Ct[U,-]).
- Дизайн пары праймеров, которые охватывают ХинdIII сайта. В том же регионе дизайн другой пары праймеров, которые не охватывают ХинdIII сайта. Дизайн праймеров для количественного PCR (Q-ПЦР) с помощью Primer3 (http://bioinfo.ut.ee/primer3-0.4.0/) и следующие параметры:
4. Biotinylation ограничение фрагмент свесов
- Подготовка Мастер микс biotinylation: 30,6 мкл 10 x ограничение буфера 2, 10,2 мкл H2O (молекулярной биологии класса), 7,65 мкл 10 мм дЦТФ, 7,65 мкл 10 мм dGTP, 7,65 мкл 10 мм dTTP, 191.25 мкл 0,4 мм биотин-14-dATP и 51 мкл 5000 ед/мл ДНК полимеразы я большой ( Фрагменты) фрагмент.
- 60 мкл biotinylation мастер смесь аликвота, микс и инкубировать при 37 ° C за 1 ч, тряски на 700 об/мин (thermomixer) для 5 s, каждый 30 s. После 1 h место аликвоты на льду.
5. в ядре лигирование
- Подготовить лигирование мастер смесь: 510 мкл 10 x T4 ДНК лигаза буфера, 51 мкл 10 мг/мл бычьим сывороточным альбумином (БСА 100 x), 1754.4 мкл воды (молекулярной биологии класса) и 127,5 мкл 1 U/мкл T4 ДНК лигаза (см. Таблицу материалы).
- 479 мкл лигирование мастер смеси на аликвота смеси и Инкубируйте на 16 ° C в течение 4 ч, тряски на 700 об/мин за 5 s каждые 2 мин в thermomixer.
- Инкубируйте 30 мин на RT.
6. Crosslink разворота
- Объединить все аликвоты в 50 мл пластиковых пробирок (подходит для высокоскоростной центрифуги).
- 62,5 мкл 10 мг/мл РНКазы A, смесь и Инкубируйте 30 мин при температуре 37 ° C.
- Добавить 300 мкл 10 mg/ml протеиназы K, микс и Инкубируйте 30 мин при температуре 37 ° C.
- Инкубируйте реакции O/N (или по крайней мере 4 h) на 65 ° C. На следующее утро 300 мкл 10 mg/ml протеиназы K, микс и инкубировать 1 час при температуре 65 ° C.
7. ДНК очистки
- 4337.5 мкл буфера TLE (10 мм трис-HCl рН 8,0; 0,1 мм ЭДТА рН 8.0) и перемешать.
- Добавьте 1 тома (10 mL) фенол рН 8,0, вихревые для 10 s и центрифуги на RT и 20000 x g для 3 мин передачи 9 мл верхняя (водный) фазы в новый Тюбик 50 мл.
Предупреждение: Фенол является опасных химических веществ. Соблюдайте соответствующие правила здоровья и безопасности. - Добавить 2 мл TLE буфера для оставшихся водной фазе, вихревые для передачи 10 s и центрифуги на RT и 20000 x g на 3 мин 2,5 мл водной фазы в новую трубу от шаг 7.2, делая окончательный объем 11,5 мл. Выбросите тубы этапа Нижняя (органических).
- Добавьте 1 тома (11,5 мл) фенол: хлороформ: изоамиловый спирт (25:24:1), вихревой 10 s и центрифуги на RT и 20000 x g для 3 мин передачи 11 мл верхняя (водный) фазы в новый Тюбик 50 мл. Повторите шаг 7.3. Объем общей выборки теперь будет 13,5 мл.
- Добавьте 1.35 мл 3 M натрия ацетата рН 5.2 и 33,75 мл ледяной 100% этанола, микс и Инкубируйте на-80 ° C для 45 мин или альтернативно на ночь при-20 ° C.
- Центрифуга на 4 ° C и 20000 x g за 10 мин, удалить супернатант, вновь приостановить Пелле в 1 мл-свежеприготовленные 70% этанола (vol/vol) и передать новой трубки.
- Центрифуга на 4 ° C и на полной скорости на 3 мин в настольная центрифуга, а затем удалить супернатант.
- Вновь приостановить Пелле в 1 мл, лед холодной 70% этанола (vol/vol) и повторите шаг 7,7. Сухие гранулы при 37 ° C 10 мин и вновь приостановить в 650 мкл буфера TLE. Определите доходность ДНК с помощью анализа на основе флуоресценции для количественного определения двуцепочечной ДНК.
Примечание: Протокол может быть приостановлена здесь, оснастки, замораживания и хранения образцов в-80 ° C в течение нескольких месяцев или в-20 ° C на более короткий период времени.
8. контроль качества
- Мониторинг целостности библиотека и перевязка методом электрофореза ДНК. Запуск 200 нг библиотеки на геле агарозы/1 x TBE 0,8%. ДНК должен работать как группа свыше 10 КБ.
- Обнаружить известный тип ячейки инвариантные краткосрочного и долгосрочного взаимодействия обычных ПЦР. Использование 100 ng шаблона ДНК в реакции PCR. Дизайн праймеры PCR, близко и к ограничения сайтов после инструкции выше (см. 3.7.1). В таблице 1перечислены последовательностей праймера для контроля качества человека Hi-C библиотек и мыши.
-
Контроль заполнения и перевязка: вырезать полосы гель, содержащий ампликонов от 8.2, гель экстракт ДНК и используйте ДНК как шаблон для 4 индивидуальных реакций PCR с идентичными грунтовка комбинаций.
- Очистить ампликонами, используя комплект для очистки ПЦР и количественного определения концентрации ДНК.
- Подготовить четыре пищеварение реакции (ХинdIII [], NheI [b], ХинdIII + NheI [c] и не фермент [d]) для каждого ампликон в окончательном объеме 15 мкл: 500 нг ампликонами, 1.5 мкл 10 x ограничение буфера 2.1, 0,15 мкл 10 мг/мл бычьим сывороточным альбумином (БСА 100 x) и 0.1 мкл (10 единиц) фермента (ХинdIII [], NheI [b], ХинdIII + NheI [c] или воды [d]).
- Дайджест за 1 ч при 37 ° C, а затем запустить пищеварение реакции на геле агарозы/1 x TBE 1,5% (wt/vol).
9. ДНК фрагментации
- Передача 50,5 мкг образца в новой трубки и добавить буфер TLE окончательный объем 655 мкл. Сплит образца в 5 sonication ампул (см. Таблицу материалы) путем добавления 130 мкл библиотеки (10 мкг) для каждого флакона. Сдвиг размер ~ 400 bp в ультра sonicator (см. Таблицу материалы) с использованием следующих параметров: коэффициент: 10%; пик инцидента мощность (Вт): 140; циклов за взрыв: 200; время: 55 s.
- Сбор sonicated образца в свежий 2-мл пробирку.
10. двухсторонняя Спри шарик выбор размера
- Mix Спри (твердой фазы реверсивные иммобилизации) шарик раствор хорошо, инвертирование, передать новой трубки 1,85 мл раствора из бисера и довести до RT на 15 мин.
- 350 мкл воды (молекулярной биологии класса) в образце (конечный объем 1 мл).
- 600 мкл раствора Спри шарик образца (общий объем 1,6 мл; соотношение Спри решения ДНК: 0,6-1), Проинкубируйте 5 мин на RT и спина образца в настольная центрифуга для 2-3 s для сбора образцов.
- Откройте крышку, поместите образец на магнитной сепарации постоять 5 мин, передачи ясно супернатант в новой трубки и отбросить бусины.
- Концентрат Спри Бусины для второй этап выбора размера: передачи 930 мкл Спри бисер в новой трубки, на магнитной сепарации постоять 5 мин и отменить ясно супернатант. Вновь приостановите бисер в 310 мкл раствора Спри шарик.
- 300 мкл концентрированного Спри бусины (шаг 10.5) образца (общий объем 1,9 мл; соотношение Спри решение ДНК в настоящее время 0,9-1), Инкубируйте на RT на 5 мин, и спина образца в настольная центрифуга для 2-3 s. тщательно открыть крышку , поместите трубки на магнитной сепарации постоять 5 мин и удалить супернатант.
- Добавить 1 мл свежеприготовленные 70% этанола (vol/vol) образец трубки на стенде магнитной сепарации, Инкубируйте 30 s и удалить супернатант. Повторите дважды.
- Сухие шарики при 37 ° C в thermomixer (открытой крышкой трубы) для не более чем 5 минут добавить 300 мкл TLE буфера выборки, смешать и проинкубируйте втечение 10 мин при комнатной температуре.
- Спиновые образца в настольная центрифуга для 2-3 s, откройте крышку и место трубку на магнитной сепарации постоять 5 мин передачи очистить супернатант в новой трубки и отбросить бусины.
11. биотина/стрептавидина выпадающее лигирование продукции
- Подготовить буферов: 1 x ТБ буфера (ЭДТА 0,5 мм, 5 мм трис-HCl рН 8,0; 0,05%; 1 M NaCl анимации 20); 2 x НТБ буфера (10 мм трис-HCl рН 8,0; 1 мм ЭДТА 2 М NaCl); 1 x NTB буфер (5 мм трис-HCl рН 8,0; 0,5 мм ЭДТА; 1 M NaCl).
- 200 мкл магнитных сочетании стрептавидина бусины (см. Таблицу материалы) в новой трубки, поместите его на стенде магнитной сепарации за 1 мин и удалить супернатант.
-
Вымойте бусины дважды с 500 мкл буфера 1 x ТБ.
- Для каждого мыть шаг во время биотина выпадающее, конец ремонт и удаление биотина в-лигируют заканчивается ДНК, dATP хвостохранилища и адаптер лигирование шаги, вновь приостановить бисер в соответствующего буфера, поворот на RT и 15 об/мин за 3 мин, спина трубку в настольная центрифуга для 2-3 s Поместите трубку на стенде магнитной сепарации на 3 мин и удалить супернатант.
- Вновь приостановите бусины в 300 мкл буфера 2 x NTB. Смешать бусины и образец (600 мкл общего объема) и Инкубируйте на RT 15 мин на вращающееся колесо на 3 об/мин.
- Вернуть бусы на стенде магнитной сепарации на 3 мин и удалить супернатант ясно. Вымойте бусины дважды в 500 мкл буфера 1 x NTB первой, а затем в 200 мкл 1 x лигирование буфера. Вновь приостановите бисер в 50 мкл 10 x лигирование буфера.
12. конец ремонт и удаление биотина на концах-лигируют ДНК
- Объединить образец (50 мкл в общей сложности) с 50 мкл 2,5 мм dNTP смеси (12,5 мкл 10 мм каждого dNTP), 18,1 µL из 3000 ед/мл T4 ДНК-полимеразы, 18,1 мкл 10 000 ед/мл T4 ПНК, 3.7 мкл 5000 ед/мл ДНК полимеразы фрагмент я большой (фрагменты) и 360.1 мкл H2O.
- Смешать и Инкубируйте на 20 ° C для 1 h, пожимая 5 s на 700 об/мин каждые 2 мин в thermomixer.
- Вернуть бусы на стенде магнитной сепарации, удалить супернатант ясно и мыть бусины дважды в 500 мкл 1 x ТБ буфера.
- Вымойте бусины в 500 мкл 1 x NTB буфера, следуют один мыть в 500 мкл 1 x TLE.
- Вернуть бусы на стенде магнитной сепарации, удалить супернатант ясно и вновь приостановить бусины в 415 мкл буфера 1 x TLE.
13. dATP хвостохранилища
- Пример (415 мкл) в сочетании с 50 мкл 10 x ограничение буфера 2, 5 мкл dATP 10 мм и 30 мкл 5 U/мкл фрагменты exo минус.
- Смешать и инкубировать при 37 ° C в течение 30 мин, пожимая 5 s на 700 об/мин каждые 2 мин в thermomixer.
- Вернуть бусы на стенде магнитной сепарации, удалить супернатант ясно и мыть бусины дважды в 500 мкл 1 x ТБ буфера.
- Вымойте бусины в 500 мкл буфера 1 x NTB.
14. адаптер лигирование
- Вымойте бусины в 200 мкл 1 x буфер реакции перешнуровки (см. Таблицу материалы).
- Вновь приостановите бусины в 200 мкл буфера реакции перешнуровки 1 x. 4 мкл ДНК лигаза (см. Таблицу материалы) и 16 мкл 15 мкм предварительно отожженная PE адаптеры (предварительно отжига адаптеры PE путем смешивания равных объемах PE адаптер 1 и PE адаптер 2 (как на 30 мкм) и инкубировать в течение нескольких минут на RT). Инкубируйте на RT на 15 мин.
- Вернуть бусы на стенде магнитной сепарации, удалить супернатант ясно и мыть бусины дважды в 500 мкл 1 x ТБ буфера.
- Вымойте бусины в 500 мкл буфера 1 x NTB. Затем, стирать бусины в 100 мкл 1 x ограничение буфера 2, вновь приостановить бусины в 50 мкл 1 x ограничение буфера 2 и передачи в новой трубки.
15. Привет C библиотека усилители
- Подготовка мастер смесь ПЦР: 100 мкл буфера 5 x Phusion; 6 мкл 25 мкм праймера PE PCR 1.0; 6 мкл 25 мкм праймера PE PCR 2.0; 14 мкл dNTP смеси (10 мм); 6 мкл Phusion полимеразы; 318 мкл H2O.
- Мастер смесь смесь ПЦР с бисером (500 мкл в общей сложности), разделить на аликвоты 10 50 мкл и усилить методом ПЦР с использованием следующих условий:
30 лет на 98 ° C
7 циклов: 10 s на 98 ° C; 30 сек при 65 ° C; 30 сек при 72 ° C
7 мин при 72 ° C - Собирать ПЦР-реакции в новой трубки, вернуть бусы на магнитной сепарации стенд и передачу супернатанта (500 мкл) в новую трубу.
-
Очищайте библиотеки ДНК с помощью Спри бусины.
- Спри микс бисер, передача 460 мкл бисера в новой трубки и принести RT для 15 минут добавить 450 мкл Спри бусы для ПЦР-реакции (окончательный объем 950 мкл), Проинкубируйте 5 мин на RT и спина образца в настольная центрифуга для 2-3 s для сбора образцов.
- Откройте крышку, поместите образец на магнитной сепарации постоять 5 мин и удалить супернатант.
- Сохраняя бисер на стенде магнитной сепарации, добавьте 1 мл 70% этанола (vol/vol) образец трубки над площадью ясно из бисера, оставьте на 30 сек и удалить супернатант.
- Повторите шаг 15.4.3 вдвое больше.
- Сухие шарики при 37 ° C в thermomixer (открытой крышкой трубы) не более 5 мин.
- Добавление 51 мкл буфера TLE, микс и инкубировать в течение 10 минут при 37 ° C, тряски на 950 об/мин в thermomixer.
- Спиновые образца в настольная центрифуга для 2-3 s, откройте крышку и место трубку на магнитной сепарации постоять 5 мин передачи очистить супернатант в новой трубки и отбросить бусины.
- Количественно определить концентрацию Hi-C-библиотеки. После 7 раундов амплификации PCR мы регулярно получать 500 – 1500 нг Hi-C библиотеки.
16. гибрид в решение захват
Примечание: Блокатор и буфера (SHS1-4) ниже решений являются от SureSelect комплекта (см. Таблицу материалы).
- Передачи 500 нг 1 мкг Hi-C библиотеки в новой трубки и выпарить образца на вакууме концентратор (см. Таблицу материалов; 45 ° C; вакуумного давления: уровень 30,0, рамп 5) до полного высыхания.
- Вновь приостановите выварочная Hi-C библиотека, добавив 3.6 мкл H2O (молекулярной биологии класса), 2,5 мкл блокатор 1, 2.5 мкл блокатор 2 и 0,6 мкл пользовательских окон.
- Передача образцов в колодец новой полосы трубки PCR, закройте с полосой Кап ПЦР и место на льду. Ярлык как «D» (для ДНК-C).
- Подготовьте гибридизации буфера: 12,5 мкл буфера SHS1; 0.5 мкл буфера SHS2; 5 мкл буфера SHS3; 6,5 мкл буфера SHS4.
- Инкубируйте на 65 ° C для 5 минут в thermomixer. Передача в колодец новой полосы трубки PCR, закройте с полосой Кап ПЦР и держать на RT. Label как «H» (для гибридизации буфера).
- В колодец новой полосы трубки PCR Смешайте 5 мкл 100 нг/мкл биотинилированным зонды RNA (магазин-80 ° C и оттепели на льду непосредственно перед использованием); 0.5 мкл SRNase B (АБС битор РНКазы) и 1,5 мкл H2O (молекулярной биологии класса).
- Закройте трубку газа ПЦР с ПЦР колпачок газа и место на льду. Ярлык как «R» (для РНК).
- Настройка машины ПЦР, используя следующие параметры:
5 мин при 95 ° C; 25 мин при температуре 65 ° C; крышка с подогревом; 29 мкл том реакции PCR.
Примечание: Действовать как можно быстрее во время всех процедур во время ПЦР машины для того, чтобы избежать испарения образца. - Место в «D» PCR трубки газа в машину ПЦР, закройте крышку машины ПЦР и начать реакции PCR. Когда программа ПЦР достигает 65 ° C, открыть крышку машины ПЦР и «H» PCR трубки газа в машину ПЦР. Закройте крышку машины ПЦР и Инкубируйте 3 мин открыть крышку машины ПЦР, место «R» ПЦР-пробирку полосы на ПЦР машины и закрыть машину ПЦР.
- Через 2 мин Откройте крышку машины ПЦР и все ПЦР трубки полоски. Перевести 13 мкл хорошо «H» в хорошо «R», то все тома хорошо «D» в хорошо «R». Накапайте вверх и вниз 3 раза смешать реакции, закройте трубку газа PCR, удаление «H» и «D» ПЦР трубки полоски и закройте крышку машины ПЦР. Инкубируйте реакции при 65 ° C для 24 h.
17. изоляция промоутер фрагмент-содержащих продуктов перешнуровки
Примечание: Следующие шаги рекомендуется делать с SureSelect комплект адаптера и библиотеки (см. Таблицу Materals).
- Предварительно теплой 1,5 мл буфера 2 мыть за образец при 65 ° C заранее.
- 60 мкл стрептавидина в сочетании магнитных шариков (см. Таблицу материалы) в новой трубки, место на стенде магнитной сепарации за 1 мин и удалить супернатант.
- Вымойте бусины три раза с 200 мкл 1 x привязки буфера.
Примечание: Для каждого шага мыть во время изоляции после захвата промоутер содержащих продуктов перешнуровки вновь приостановить бусины в соответствующего буфера, поворот на 3 мин в RT и 15 rpm на вращающееся колесо, мягко спина трубку в настольная центрифуга для 2-3 s для сбора образца, место трубку на магнитной сепарации постоять 3 мин и удалить супернатант. - Вновь приостановите бусины в 200 мкл 1 x привязки буфера. Открыть машину ПЦР и ПЦР-пробирку полосы (в то время как все еще запущена программа ПЦР) и передачи гибридизации реакции в трубку с магнитной бусины. Инкубируйте на RT 30 мин на вращающееся колесо на 3 об/мин.
- Вернуть бусы на стенде магнитной сепарации и удалить супернатант ясно. Вновь приостановить бусины в 500 мкл буфера мытья 1, перемешивают и Инкубируйте 15 мин при 20 ° C при встряхивании в 950 об/мин в thermomixer.
- Вернуть бусы на стенде магнитной сепарации на 3 мин и удалить супернатант ясно. Вновь приостановить бусины в 500 мкл буфера мытья 2, смешать и инкубировать 10 мин при температуре 65 ° C при встряхивании в 950 об/мин в thermomixer. Повторите шаг 17,5 вдвое больше.
- Вернуть бусы на стенде магнитной сепарации, удалить супернатант ясно и повторно приостановить бисером промаркированные 1 x ограничение буфера 2. Вернуть бусы на стенде магнитной сепарации, удалить супернатант и вновь приостановить бусины в 30 мкл 1 x ограничение буфера 2.
18. PCHi-C библиотека усилители
- Подготовка мастер смесь ПЦР: 60 мкл буфера 5 x PCR (Phusion буфер), 3.6 мкл 25 мкм праймера PE PCR 1.0, 3.6 мкл 25 мкм PE PCR праймер 2.0, 8.4 мкл dNTP смеси (по 10 мм), 3.6 мкл Phusion полимеразы и 190,8 мкл H2O.
- Мастер смесь смесь ПЦР с бисером (300 мкл в общей сложности), разделить на 6 аликвоты 50 мкл, и ПЦР усилить с помощью следующих условий:
30 s на 98 ° C
4 циклов: 10 s на 98 ° C, 30 s при 65 ° C, 30 сек при 72 ° C
7 мин при 72 ° C - Собрать все ПЦР-реакции в новой трубки, вернуть бусы на магните и передачи супернатант (300 мкл; содержит библиотеки PCHi-C) в к новой пробке.
- Очищайте PCHi-C библиотеки с помощью Спри бусы, следующие шаги, описанные выше до 15,4.
- Количественного определения концентрации PCHi-C-библиотеки.
Результаты
Промоутер захватить Hi-C был использован для обогащения мыши7,34,,3639 и35,человека33,37,38 Hi-C библиотек Промоутер взаимодействий. Аналогичный протокол (названный HiCap) был описан Sandberg группы40. Рисунок 1A показана схема рабочего процесса для промоутера захватить Hi-C. В протоколе, описанные здесь Привет-C библиотек создаются с помощью в ядро перевязка41, которая приводит к значительно снизить количество ложных лигирование продукции42. Для PCHi-C весьма сложные мыши или человека Hi-C библиотек подвергаются-решение гибридизации и захватить с помощью 39,021 РНК биотинилированным дополняет 22,225 мыши промоутер содержащих HindIII фрагментов, или 37,608 биотинилированным РНК на 22,076 человека промоутер содержащих HindIII фрагментов, соответственно. Промоутер, содержащие фрагменты ограничения могут быть направлены на одной или обеих концах отдельных биотинилированным РНК (рис. 1B). Мы обнаружили, что захват обоих заканчивается улучшение охвата отдельных промоутеров (Рисунок 1 c; сырой последовательности прочтений) почти в два раза, как ожидалось. Таким образом, всякий раз, когда возможно (т.е. в регионах, не повторяющихся), мы советуем использовать биотинилированным RNAs дополняет обоих концах ограничения для захвата.
Чтобы оценить качество библиотека PCHi-C на раннем этапе в ходе подготовки библиотеки, мы выполняем два элемента управления после перевязки ДНК и очистки, как описано выше31. Первый заключается в использовании конкретных грунтовка пар для того чтобы усилить продуктов перешнуровки как 3 C27. Мы используем грунтовка пар (Таблица 1) для того чтобы усилить тип ячейки инвариантные дальнего лигирование продуктов, например между Гене Myc и его известных усилители расположен около 2 МБ прочь (рисунок 2A) или между генов (Локус) Hist1 разделенных 1,5 МБ) и между двумя регионами, расположен в непосредственной близости линейной («малой дальности управления»).
Второй контроль качества проводится для определения эффективности включения биотина во время заполнить фрагменты опосредованной ограничение сайта свесы с биотин dATP. Успешно заполнить фрагменты и результаты последующего перешнуровка Туп конца в исчезновении оригинального сайта ограничение между молекулами ДНК лигирование продукта и в случае HindIII в формировании нового сайта NheI признание (Рисунок 2Б ). Отношение HindIII продукт усваивается лигирование NheI является прямым Индикация эффективности включения биотин. Низкое качество Hi-C библиотека покажет высокий уровень HindIII пищеварение, тогда как высокое качество библиотек имеют почти полное NheI пищеварение продуктов перешнуровки (рис. 2B).
После Hi-C библиотека подготовки (т.е., после того, как биотин стрептавидина тянуть вниз размер отдельных продуктов перешнуровки Hi-C, адаптер перевязки и до захвата ПЦР) целостность и размер распространение библиотеки Hi-C оценивается по Bioanalyzer (Рисунок 2 C). тот же элемент управления осуществляется в конце PCHi-C библиотека подготовки (т.е., после захвата гибридизации промоутер содержащих продуктов перешнуровки и после захвата ПЦР). Сравнение профилей Hi-C и PCHi C Bioanalyzer показывает, как и ожидалось, Привет-C библиотеки являются гораздо более концентрированной, чем соответствующие библиотеки PCHi-C, но размер распределение библиотек очень похож, указывая, что захват шаг в PCHi-C не ввести размер смещения (рис. 2 c, D).
После паре конец последовательности, читает PCHi-C сопоставляются, качество контролируется и фильтруются с помощью конвейера HiCUP43. Высокое качество PCHi-C библиотеки содержат между 70-90% «действительный пар» (то есть, в паре конец последовательности читает между двумя ограничение фрагментов, которые не соседствуют на линейной геномной карте; Рисунок 3А, Б). С помощью перевязки в ядро протокол41,42, процент транс читать пары (то есть, в паре конец последовательности читает между двумя фрагментами ограничения, которые расположены на разных хромосомах) обычно низкий, между 5 и 25% что отражает существование хромосома территорий и с указанием качества хорошей библиотеки. Прямое сравнение доли пар «действительно» между Hi-C библиотек и их соответствующие библиотеки PCHi-C35, показывает, что во всех случаях процент допустимых пар находится выше PCHi-C библиотек (рисунок 3B). Это сопровождается снижением доли недействительный «же фрагмент внутреннего» читает в PCHi-C (рис. 3 c). Это ожидается, как шаг записи не только для промоутер содержащих продуктов перешнуровки обогащает, но также для ограничения фрагмент концы, из-за позиции oligos захвата на ограничение фрагменты (см. Рисунок 1B).
После фильтрации HiCUP мы определить эффективность захвата. PCHi-C библиотеки содержат три типа недопустимая последовательность читает после фильтрации HiCUP:
1.) промоутер: читает генома (то есть, читает между фрагмент захваченных промоутер и фрагмент ограничение не промоутер HindIII нигде в геноме)
2.) промоутер: промоутер читает (читает между двух захваченных промоутер фрагменты)
3.) генома: читает генома (фон Hi-C лигирование продуктов, где ни один из партнеров продукт перешнуровки сопоставляется захваченных промоутер). Эти отбрасываются до течению анализы.
Высокое качество PCHi-C библиотек имеют эффективность захвата (сумма категорий 1 и 2 выше) между 65 – 90% (Рисунок 3D). Прямое сравнение Hi-C библиотек показывает, что PCHi-C приводит к ~ 15 раз обогащения промоутер-перевязка продуктов, содержащих (Рисунок 3D), в некоторых случаях 17-fold. Это близко к гипотетической максимум (19.6-fold) обогащения для PCHi-C, который зависит от доли фрагментов генома, охватываемых системой захвата. Большего обогащения может достигаться путем разработки систем захвата ориентации меньше ограничение фрагментов44,45,46.
Анализ interactomes промоутер демонстрирует ячейки типа и линии специфика33,34,35, с выраженными изменениями в клеточной дифференциации37,38,39 . Рисунки 4 и 5 показаны примеры линии специфичность и дифференциации динамики на конкретных промоутеров. К примеру ALAD выражается конститутивно во всех клетках, но его выражение upregulated в erythroblasts47. Промоутер ALAD контакты несколько дистальной фрагменты во всех гемопоэтических клеток и участвует в дополнительные взаимодействия конкретно в erythroblasts (рис. 4). ИЛ-8 показывает без статистически значимых взаимодействий в клетки B, очень немногие взаимодействия в Т-клеток, но десятки взаимодействий в клетки миелоидного линии, включая тип ячейки специфических взаимодействий в megakaryocytes, моноцитов и нейтрофилов ( Рисунок 5). Эти примеры демонстрируют, как PCHi-C может использоваться для разгадать типа клеток конкретных interactomes и идентифицировать промоутер взаимодействующих регионах с нормативно-правовой потенциал.
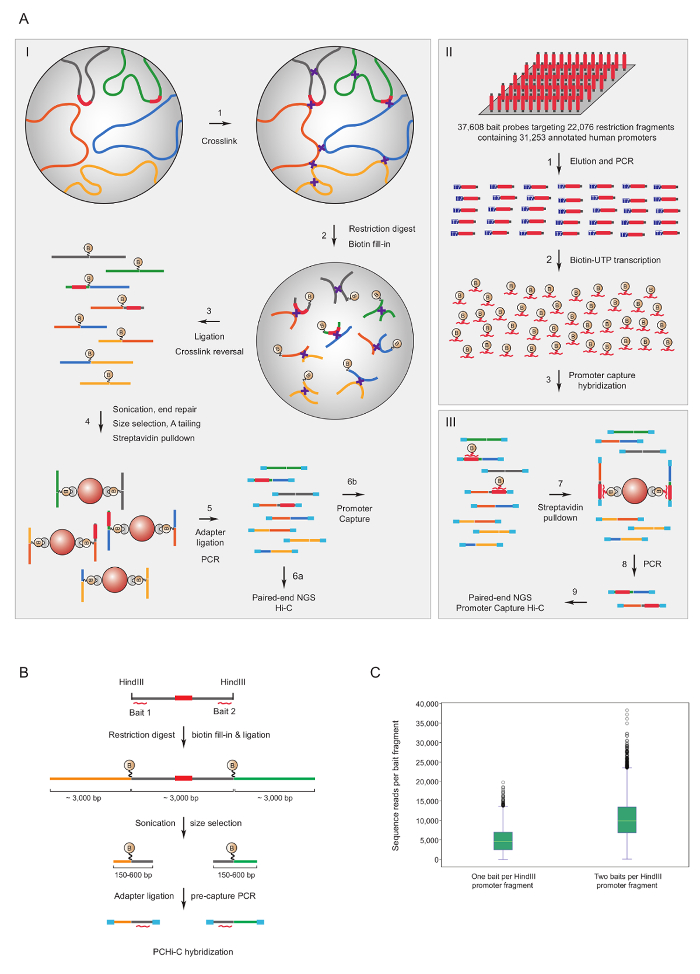
Рисунок 1 : Промоутер захватить Hi-C обоснование и захвата приманки дизайн. (A) схема рабочего процесса PCHi-C. В ядро лигирование Hi-C41,42 (I) следуют-решение гибридизации с приманками биотинилированным РНК (II) ориентация фрагментов всех человека (изображенный здесь) или мыши ген промоутеров (III). (B) приманки дизайн для PCHi-C. Против концы промоутер содержащих ограничение фрагментов (серый; Обратите внимание, что промоутер последовательности, сами (красный) только являются мишенью приманки захвата РНК, если они расположены на ограничение предназначены биотинилированным РНК захвата приманки (красные кривые линии) фрагмент заканчивается). Перешнуровка продуктов, состоящих из промоутер содержащих ограничение фрагментов (серый) и их взаимодействия фрагментов (желтый и зеленый) изолированные путем гибридизации последовательности взаимодополняемости между приманки РНК и ДНК целевой и последующего биотин стрептавидина пулдаун, как показано в A. (C) сравнение PCHi-C улавливания промоутер содержащих ограничение фрагментов мишенью одну приманку РНК захвата зонд против двух РНК приманки захвата зонды (см. схема в B). Пожалуйста, нажмите здесь, чтобы посмотреть большую версию этой фигуры.
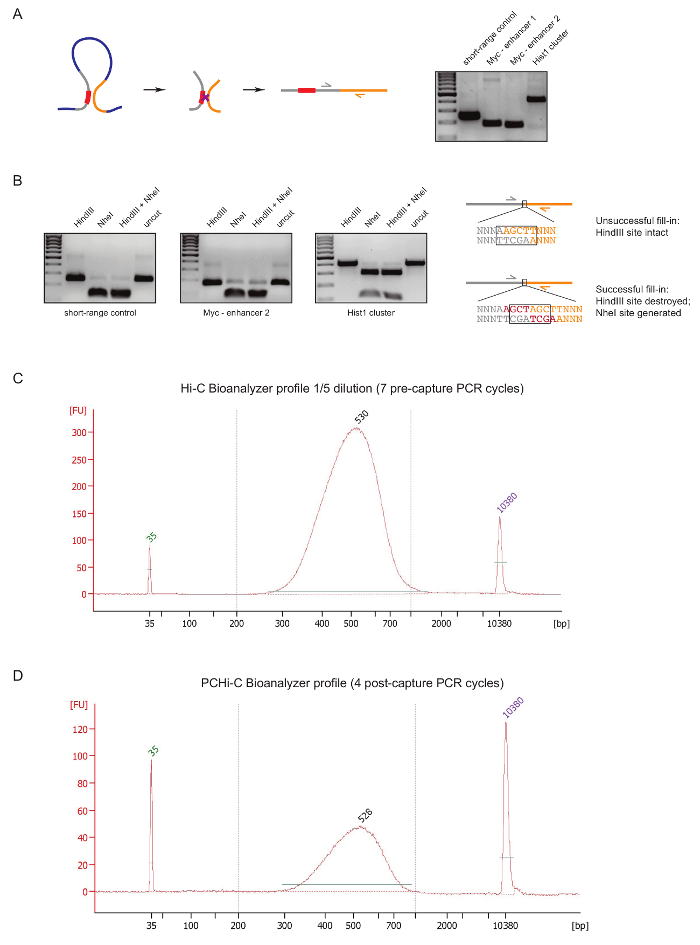
Рисунок 2 : PCHi-C контроль качества до последовательности. (A) слева, схема пространственного сопоставление между промоутер и Пир, что приводит к Hi-C лигирование продукт, состоящий из промоутер содержащих ограничение фрагмент (серый; промоутер последовательности в красном) и фрагмент ограничение ПИР (желтый). Право, ДНК гель электрофорез показаны примеры продуктов перешнуровки Hi-C усиливается с помощью конкретных грунтовка пар (как показано на схеме слева). (B) слева, показательные примеры HindIII, NheI и HindIII/NheI ограничения краткие сборники Hi-C перевязки (продукты PCR показано на A). Справа, схема ДНК последовательность после перевязки Hi-C после неудачной (вверху) или заполнить фрагменты dNTP успешных (внизу) ограничение развязок и последующей перевязкой. (C) представитель Hi-C библиотека bioanalyzer профиль (разбавления 1/5). (D) представитель PCHi-C библиотека bioanalyzer профиль (без разбавления). Пожалуйста, нажмите здесь, чтобы посмотреть большую версию этой фигуры.
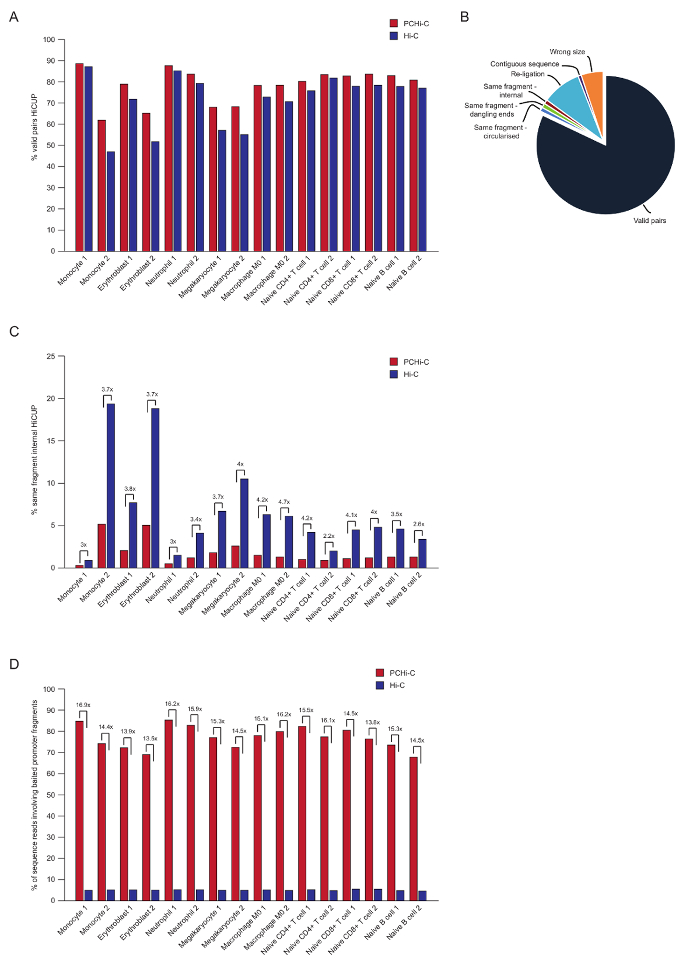
Рисунок 3 : PCHi-C после виртуализации контроля качества. (A) сравнение процент допустимые последовательности читать пар после HiCUP43 обработки в PCHi-C против соответствующих библиотек Hi-C (данные из Хавьерре et al., 2016-35). (B) представитель HiCUP PCHi-C результат показаны допустимые читать пар и другие категории последовательности, которые отбрасываются до течению анализы (данные из Хавьерре et al., 2016-35). (C) сравнение процентных «же фрагмент внутреннего» читает после HiCUP обработки в PCHi-C против соответствующих библиотек Hi-C (данные из Хавьерре et al., 2016-35). (D) сравнения последовательности процент читает с участием наживкой промоутер фрагменты (эффективности) в PCHi-C против соответствующих библиотек Hi-C (данные из Хавьерре et al., 2016-35). Пожалуйста, нажмите здесь, чтобы посмотреть большую версию этой фигуры.
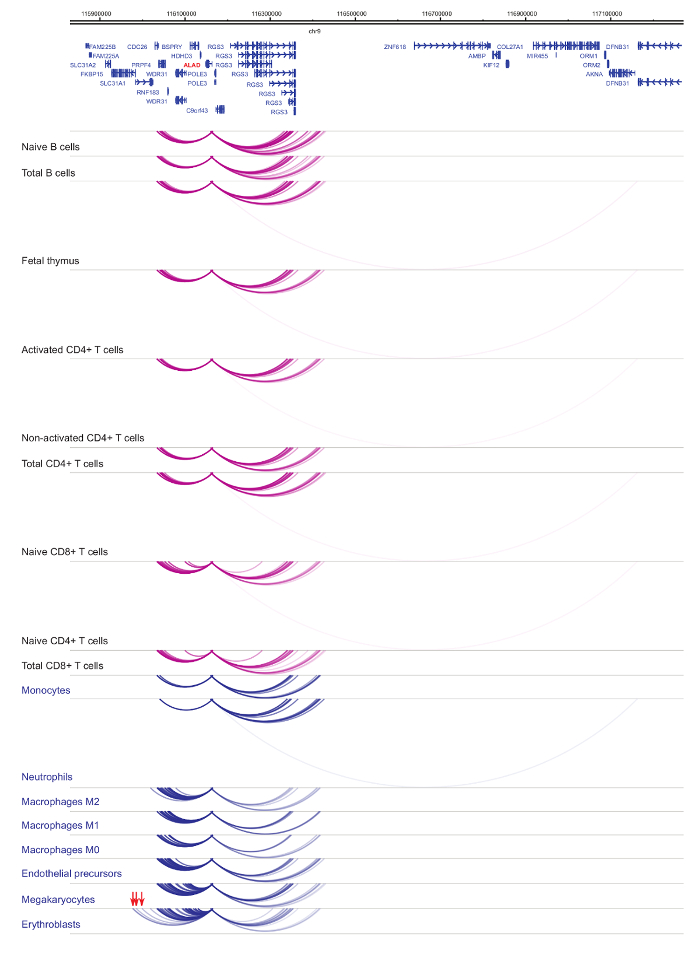
Рисунок 4: ALAD PCHi-C профиль гемопоэтических клеток человека. Промоутер взаимодействия типов миелоидных клеток показаны как синие арки, а промоутер взаимодействия типов лимфоидных клеток являются как фиолетовый арки. Erythroblast специфических взаимодействий обозначаются красными стрелками (данные из Хавьерре et al., 2016-35). Пожалуйста, нажмите здесь, чтобы посмотреть большую версию этой фигуры.
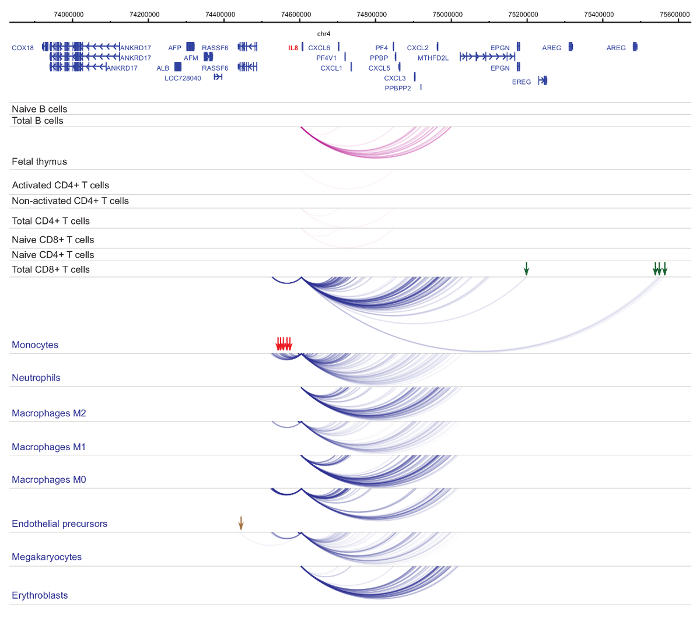
Рисунок 5 : ИЛ 8 PCHi-C профиль гемопоэтических клеток человека. Промоутер взаимодействия типов миелоидных клеток показаны как синие арки, а промоутер взаимодействия типов лимфоидных клеток являются как фиолетовый арки. Моноцит специфических взаимодействий обозначаются зелеными стрелками, нейтрофилов специфических взаимодействий обозначаются красными стрелками и мегакариоцитов конкретного взаимодействия обозначается коричневый стрелка (данные из Хавьерре et al., 2016-35). Пожалуйста, нажмите здесь, чтобы посмотреть большую версию этой фигуры.
| Человека | ||||||||
| Имя | Последовательность | Хромосома | Стрэнд | Начало GRCh38/hg38 | Конец GRCh38/hg38 | Грунтовка комбинации, чтобы проверить взаимодействие 3C и биотин инкорпорации | ||
| HS AHF64 Деккер | GCATGCATTAGCCTCTGCTGTTCTCTGAAATC | 11 | + | 116803960 | 116803991 | использование в сочетании с hs AHF66 Деккер | ||
| HS AHF66 Деккер | CTGTCCAAGTACATTCCTGTTCACAAACCC | 11 | + | 116810219 | 116810248 | использование в сочетании с hs AHF64 Деккер | ||
| HS MYC Локус | GGAGAACCGGTAATGGCAAA | 8 | - | 127733814 | 127733833 | использование в сочетании с hs MYC +1820 или hs MYC-538 | ||
| HS MYC +1820 | AAAATGCCCATTTCCTTCTCC | 8 | + | 129554527 | 129554547 | использование в сочетании с hs MYC Локус | ||
| HS MYC-538 | TGCCTGATGGATAGTGCTTTC | 8 | - | 127195696 | 127195716 | использование в сочетании с hs MYC Локус | ||
| HS HIST1 F | AAGCAGGAAAAGGCATAGCA | 6 | + | 26207174 | 26207193 | использование в сочетании с hs HIST1 R | ||
| HS HIST1 R | TCTTGGGTTGTGGGACTTTC | 6 | + | 27771575 | 27771594 | использование в сочетании с hs HIST1 F | ||
| Мышь | ||||||||
| Последовательность | Хромосома | Стрэнд | Начало GRCm38/мм10 | Конец GRCm38/мм10 | Грунтовка комбинации, чтобы проверить взаимодействие 3C и биотин инкорпорации | |||
| TCATGAGTTCCCCACATCTTTG | 8 | + | 84841090 | 84841111 | использование в сочетании с мм Calr2 | |||
| CTGTGGGCACCAGATGTGTAAAT | 8 | + | 84848519 | 84848541 | использование в сочетании с мм Calr1 | |||
| TATCAAGGGTGCCCGTCACCTTCAGC | 6 | + | 125163098 | 125163123 | использование в сочетании с Gapdh4 Деккер | |||
| GGGCTTTTATAGCACGGTTATAAAGT | 6 | + | 125163774 | 125163799 | использование в сочетании с Gapdh3 Деккер | |||
| GGAGGAGGGAAAAGGAGTGATT | 6 | + | 52212829 | 52212850 | использование в сочетании с мм Hoxa13 | |||
| CAGGCATTATTTGCTGAGAACG | 6 | - | 52253490 | 52253511 | использование в сочетании с мм Hoxa7 | |||
| GGGTAATGGTGTCACTAACTGG | 13 | + | 23571284 | 23571305 | использование в сочетании с Hist1h3e или мм Hist1h4i | |||
| GGGTTTGATGAGTTGGTGAAG | 13 | + | 23566541 | 23566561 | использование в сочетании с мм Hist1h2ae | |||
| TTGGGCCAAAGCCTATATGA | 13 | + | 22043085 | 22043104 | использование в сочетании с мм Hist1h2ae | |||
Таблица 1: Грунтовка последовательности для контроля качества человека и мыши Hi-C библиотек.
Обсуждение
Модульная конструкция промоутер захватить Hi-C
Промоутер захватить Hi-C предназначен для специально обогатить Hi-C библиотеки для взаимодействия с участием промоутеров. Эти взаимодействия составляют только подмножество лигирование продуктов, присутствующих в библиотеке Hi-C.
Захват Привет-C можно легко изменить для обогащения Hi-C библиотек для любой геномной региона или регионов интерес, изменив систему захвата. Захват регионов может быть непрерывной геномной сегментов44,45,46,48, усилители, которые были определены в PCHi-C (35'обратный захват Hi-C'), или DNase сверхчувствительным сайты49 . Размер захвата системы может корректироваться в зависимости от области экспериментальной. Например, Драйден и др. целевые 519 приманки фрагментов в трех пустынь генов, связанных с раком молочной железы44. Система захвата Мартин и др. цели обеих непрерывной геномной сегментов («Региона захватить»: 211 геномной регионов в общей сложности; 2 131 фрагментов) и промоутеров (3857 генным стимуляторам)45.
SureSelect библиотеки доступны в разные типоразмеры: 1 КБ до 499 kb (5 190 – 4,806), 500 КБ до 2,9 МБ (5 190 – 4,816) и 3 МБ до 5,9 МБ (5 190 – 4,831). Как каждый индивидуальный захват биотин РНК длиной 120 нуклеотидов, они захватить систем вместить максимум 4,158, 24,166 и 49,166 отдельных захватить датчиков, соответственно. Это соответствует 2,079, 12,083 и 24,583 целевых фрагментов, соответственно (Обратите внимание, что номера для фрагментов нижней границы, основывается на предположении, что два отдельных захвата зонды могут быть разработаны для каждого ограничения фрагмент — в действительности из-за повторяющихся последовательностей это не будет случай для каждого ограничения фрагмент (см. также Рисунок 1B, C), приводит в большее количество фрагментов ориентации ограничение для постоянного числа доступных захвата зонды ).
Протокол, описанные здесь основана на использовании энзима ограничения с узлом признание 6 bp раскрыть на большие расстояния взаимодействия. С помощью энзима ограничения с 4 сайта признание bp для большей резолюции более проксимальном взаимодействий является также возможно40,49.
Ограничения PCHi-C
Одно ограничение, свойственное все хромосомы конформации захвата анализов является, что их резолюции определяется энзима ограничения, используемый для создания библиотеки. Взаимодействия, которые происходят между ДНК элементов, расположенных на один и тот же фрагмент ограничения являются невидимыми для «C-тип» анализов. Кроме того в PCHi-C, в некоторых случаях более чем один сайт начало транскрипции может быть расположен на тот же промоутер содержащих ограничение фрагмент, и Пирс в некоторых случаях гавани оба активных и репрессивных гистона знаки, что делает его трудно определить который регулирования элементы посредником взаимодействий и предсказать регулирования выходной промоутер взаимодействий. С помощью энзимов ограничения с 4 bp признание сайтов устраняет эту проблему, но происходит за счет значительно возросшей сложности библиотека Hi-C (Hi-C библиотек, созданных с энзимами ограничения распознавания сайта 4 bp по крайней мере в 100 раз более сложной, чем Hi-C библиотеки, созданные с энзимами ограничения распознавания сайта 6 bp) и связанные с этим расходы для следующего поколения последовательности.
Еще одним ограничением является, что текущий протокол PCHi-C требует миллионы клеток как исходного материала, исключающего анализ взаимодействий промоутер в типах редких клеток. Модифицированная версия PCHi-C с тем чтобы допрос промоутер контактов в клеточных популяций с 10 000 до 100 000 клеток (например, клетки во время раннего эмбрионального развития или гемопоэтических стволовых клеток) поэтому бы ценным дополнением к захвату Привет-C панели инструментов.
Наконец как все методы, которые полагаются на формальдегид фиксации, PCHi-C только записи взаимодействий, которые являются «заморожены» в момент времени фиксации. Таким образом для изучения кинетики и динамика промоутер взаимодействий, требуются методы, такие как супер резолюции микроскопия живой клетки вместе с PCHi-C.
Методы для того чтобы рассечь пространственных хромосома Организации с высоким разрешением
Огромные сложности хромосомных взаимодействия библиотек запрещает надежной идентификации продуктов взаимодействия между двух конкретных фрагментов с статистической значимости. Чтобы обойти эту проблему, захват последовательности был использован для обогащения Hi-C-33,-34,-40,-44 или 3 C50,51 библиотек для специфических взаимодействий. Основным преимуществом использования Hi-C библиотеки свыше 3C для обогащения шаг является Hi-C, в отличие от 3C, включает обогащение шаг для подлинного лигирование продуктов. Как следствие процент действительно читает в PCHi-C библиотек примерно в 10 раз выше, чем в захват-C библиотек50, в которой содержится около 5-8% действительный читает после фильтрации HiCUP. Sahlen et al. непосредственно сравнивать захвата-C HiCap, который, как PCHi-C использует библиотеки Hi-C для захвата обогащения, в отличие от захвата-C, которая использует 3 C библиотек. В соответствии с нашими выводами, они обнаружили, что захват-C библиотек главным образом состоят из ООН перевязаны фрагменты40. Кроме того HiCap библиотек был выше сложность, чем захват-C библиотек40.
Вариант захвата-C, под названием следующего поколения захвата-C52 (нг захвата-C) использует один oligo на конец фрагмента ограничения, ранее созданного в PCHi-C33,34, вместо дублирования датчики, используемые в оригинале Захват-C протокол50. Это увеличивает процент действительно читает по сравнению с захвата-C скромно, но нг захвата-C использует два последовательных раундов захвата обогащения, и относительно большое количество PCR циклов (20-24 циклов в общей сложности, по сравнению с 11 циклов обычно для PCHi-C), которая неизбежно приводит к росту числа дубликатов последовательности и Нижняя библиотека сложности. В пробные эксперименты во время оптимизации PCHi-C, мы обнаружили, что процент уникальных (т.е., не дублируются) читать пар был только около 15% когда мы использовали 19 циклов PCR (13 циклов предварительного захвата + 6 циклов после захвата; данные не показаны), однако Оптимизация на меньшее количество циклов PCR, обычно дает 75 – 90% уникальных чтения пар. Таким образом уменьшение числа циклов PCR значительно увеличивает количество информативный последовательности данных.
Бронирование метод сочетает в себе чип с Hi-C, чтобы сосредоточиться на хромосомные взаимодействий при посредничестве специфического протеина интереса (53HiChIP). По сравнению с Цзя-PET54, которая основана на аналогичные обоснования, HiChIP данные содержат большее количество информативный последовательности читает, позволяя больше доверия взаимодействия вызова53. Это будет очень интересно непосредственно сравнить соответствующие HiChIP и наборы данных захватить Hi-C когда они становятся доступными (к примеру, HiChIP, используя антитело против когезинов группы Smc1a53 с захвата Hi-C для всех Smc1a связано ограничение фрагменты) бок о бок. Одно неотъемлемое различие между этими двумя подходами, что захватить Hi-C не полагаться на иммунопреципитации chromatin и поэтому способен опрашивания хромосомных взаимодействия независимо от белка размещение. Это позволяет сравнение 3D генома Организации в наличие или отсутствие привязки конкретных факторов, как была использована для выявления PRC1 как ключевым регулятором мыши ESC пространственных генома архитектура7.
PCHi-C и GWAS
Геном всей ассоциации исследований (GWAS) показали, что более чем 95% болезней связанные последовательности вариантов расположены в регионах некодирующих генома, часто на большие расстояния, чтобы белок кодирование генов55. GWAS варианты являются часто найдены в непосредственной близости от DNase я сверхчувствительным сайтов, который является отличительной чертой последовательностей с потенциальными регулирующей деятельности. PCHi-C и захватить Hi-C широко использовались для связи промоутеров GWAS риска локусов причастны рака груди44,48колоректального рака и аутоиммунных заболеваний35,45,46. PCHi-C исследование на 17 различных гемопоэтических клеток человека типы нашел SNPs, связанные с аутоиммунным заболеванием обогатились в Пирс в лимфоидных клеток, тогда как последовательность варианты, связанные с тромбоцитов и эритроцитов специфические черты преимущественно были найдены в макрофаги и erythroblasts, соответственно35,56. Таким образом конкретные промоутер ткани тип interactomes обнаружили в PCHi-C может помочь понять функцию некодирующих болезни связанные последовательности вариантов и выявлять новых потенциальных болезней генов для терапевтической интервенции.
Особенности взаимодействия промоутер регионов
Несколько линий доказательств связать промоутер interactomes управления выражения гена. Во-первых несколько PCHi-C исследования показали, что геномной регионов, взаимодействующих с промоутерами (высоко) выразил генов обогащаются в метках, связанные с деятельностью enhancer, таких как H3K27 ацетилирования и p300 привязки33,34 , 37. Мы нашли положительная корреляция между уровнем выражение гена и количество взаимодействующих усилители, предполагая, что эффект суммирования результата усилители в выражении увеличение гена уровнях34,35. Во-вторых естественным выражение в Пирс, которые подключены к же генов, экспрессия которых зависит от eQTLs35обогащены локусов количественных признаков (eQTLs). В-третьих путем интеграции путешествие57 и данных PCHi-C, Кэрнс et al. обнаружили, что путешествие репортер генов сопоставление Пирс в мыши ЭСК Показать сильнее репортер экспрессии генов, чем репортер генов в интеграции сайтов в регионах, не взаимодействующих промоутер 58, указав, что Пирс обладают транскрипционный анализ нормативной деятельности. Вместе эти результаты показывают, что промоутер interactomes обнаружили PCHi-C в различных мыши и типы клеток человека включают в себя ключевые нормативные модули для управления выражения гена.
Стоит отметить, что усилители представляют лишь малую долю (~ 20%) из всех Пирс, обнаружили PCHi-C33,34. Другие пиры могут иметь структурные или топологических ролей, вместо того, чтобы прямые транскрипционный анализ регулирующих функций. Однако есть также доказательства того, что PCHi-C может раскрыть элементы ДНК с функции регулирования, которые не гавани классической усилитель знаки. В линии лимфоидных клеток человека промоутер BRD7 было обнаружено взаимодействовать с регионом лишенный знаков Улушитель, что было показано, обладают усилитель активность Репортер ген анализов33. Нормативные элементы с аналогичными характеристиками могут быть более обильны, чем в настоящее время высоко. Например основанный ТРИФОСФАТЫ экран для регулирования ДНК элементы выявленных немаркированных регуляторных элементов (Урес) которые контролируют экспрессию генов, но лишены усилитель марки59.
В других случаях было показано PIRs гавани хроматина знаков, связанные с транскрипционный анализ репрессий. Пирс и взаимодействующих промоутеров, обязательность PRC1 в мыши ЭСК занимались обширные пространственные сети репрессированных генов, учитывая что репрессивные Марк H3K27me37. В человеческой лимфобластоидных клетках элемент далекой, взаимодействующих с BCL6 промоутер репрессированных трансген Репортер ген выражение33, предполагая, что он может функционировать для подавления BCL6 транскрипции в своем собственном контексте.
Пирс, обогащенного для размещения белка хроматина изолятор CTCF в человеческих ЭСК и NECs37 может представлять еще один класс Пирс. Вместе эти результаты показывают, что Пирс гавани коллекцию гена регулирования деятельности еще функционально охарактеризовать.
Раскрытие информации
Авторы не имеют ничего сообщать.
Благодарности
Мы благодарим Валерия Малышева для критических чтении рукописи и квалифицированную помощь с рис. Эта работа была поддержана Советом медицинских исследований, Великобритания (MR/L007150/1) и Великобритании биотехнологии и биологических наук исследовательский совет, Великобритания (BB/J004480/1).
Материалы
| Name | Company | Catalog Number | Comments |
| 16% (vol/vol) paraformaldehyde solution | Agar Scientific | R1026 | |
| Dulbecco's Modified Eagle Medium (DMEM) 1x | Life Technologies | 41965-039 | |
| Fetal bovine serum (FBS) sterile filtered | Sigma | F9665 | |
| Low-retention filter tips | Starlab | S1180-3810, S1180-1810, S1180-8810 and S1182-1830 | |
| 10x PBS pH 7.4 | Life Technologies | 70011-036 | |
| Molecular biology grade water | Sigma-Aldrich | W4502 | |
| 1 M Tris-HCl pH 8.0 | Life Technologies | 15568-025 | |
| IGEPAL CA-630 | Sigma-Aldrich | I8896 | |
| 5 M NaCl | Life Technologies | 24740-011 | |
| Protease inhibitor cocktail (EDTA-free) | Roche Diagnostics | 11873580001 | |
| Restriction buffer 2 (10x NEBuffer 2) | New England Biolabs | B7002 | |
| DNA LoBind tube, 1.5 mL | Eppendorf | 0030 108.051 | |
| DNA LoBind tube, 2 mL | Eppendorf | 30108078 | |
| 20% (wt/vol) SDS | Bio-Rad Laboratories | 161-0418 | |
| 20% (vol/vol) Triton X-100 | Sigma-Aldrich | T8787 | |
| HindIII, 100 U/uL | New England Biolabs | R0104 | |
| 10 mM dCTP | Life Technologies | 18253-013 | |
| 10 mM dGTP | Life Technologies | 18254-011 | |
| 10 mM dTTP | Life Technologies | 18255-018 | |
| 0.4 mM Biotin-14-dATP | Life Technologies | 19524-016 | |
| DNA polymerase I large (Klenow) fragment 5000 units/mL | New England Biolabs | M0210 | |
| 10x T4 DNA ligase reaction buffer | New England Biolabs | B0202 | |
| 100x 10mg/ml Bovine Serum Albumin | New England Biolabs | B9001 | |
| T4 DNA ligase, 1 U/μL | Invitrogen | 15224-025 | |
| RNase A | Roche | 10109142001 | |
| Proteinase K, recombinant, PCR grade | Roche | 3115836001 | |
| 20 000×g 50 ml centrifuge tube | VWR | 525-0156 | |
| 0.5 M EDTA pH 8.0 | Life Technologies | 15575-020 | |
| Phenol pH 8.0 | Sigma | P4557 | |
| Phenol: Chloroform: Isoamyl Alcohol 25:24:1 | Sigma | P3803 | |
| Sodium acetate pH 5.2 | Sigma | S7899 | |
| Quant-iT PicoGreen | Invitrogen | P7589 | |
| QIAquick Gel Extraction Kit | Qiagen | 28704 | |
| QIAquick PCR Purification Kit | Qiagen | 28104 | |
| Restriction buffer 2.1 (10x NEBuffer 2.1) | New England Biolabs | B7202 | |
| NheI, 100U/uL | New England Biolabs | R0131 | |
| Micro TUBE AFA Fiber Pre-slit snap cap 6x16mm vials | Covaris | 520045 | For sonication |
| SPRI beads (Agencourt AMPure XP) | Beckman Coulter | A63881 | |
| Dynabeads MyOne Streptavidin C1 beads | Invitrogen | 65001 | |
| Tween 20 | Sigma | P9416 | |
| 10 mM dATP | Life Technologies | 18252-015 | |
| T4 DNA polymerase 3000 units/mL | New England Biolabs | M0203 | |
| T4 PNK 10000 units/mL | New England Biolabs | M0201 | |
| Klenow exo minus 5000 units/mL | New England Biolabs | M0212 | |
| Quick ligation reaction buffer | New England Biolabs | B6058 | |
| NEB DNA Quick ligase | New England Biolabs | M2200 | |
| PE adapter 1.0 (5'-P-GATCGGAAGAGCGGTTCAGC AGGAATGCCGAG-3') | Illumina | ||
| PE adapter 2.0 (5'-ACACTCTTTCCCTACACGACGCT CTTCCGATCT-3') | Illumina | ||
| NEB Phusion PCR kit | New England Biolabs | M0530 | |
| PE PCR primer 1.0 (5'-AATGATACGGCGACCACCGA GATCTACACTCTTTCCCTAC ACGACGCTCTTCCGATCT-3') | Illumina | ||
| PE PCR primer 2.0 (5'-CAAGCAGAAGACGGCATACGA GATCGGTCTCGGCATTCCT GCTGAACCGCTCTTCCGATCT-3') | Illumina | ||
| PCR strips | Agilent Technologies | 410022 and 401425 | |
| SureSelect SSEL TE Reagent ILM PE full adaptor kit | Agilent Technologies | 931108 | |
| SureSelect custom 3-5.9 Mb library | Agilent Technologies | 5190-4831 | custom design mouse or human PCHi-C system |
| Dynabeads MyOne Streptavidin T1 beads | Invitrogen | 65601 | |
| E220 high-performance focused ultra-sonicator | Corvaris | E220 |
Ссылки
- Osborne, C. S., et al. Active genes dynamically colocalize to shared sites of ongoing transcription. Nature Genetics. 36, 1065-1071 (2004).
- Schoenfelder, S., et al. Preferential associations between co-regulated genes reveal a transcriptional interactome in erythroid cells. Nature Genetics. 42, 53-61 (2010).
- de Wit, E., et al. The pluripotent genome in three dimensions is shaped around pluripotency factors. Nature. 501, 227-231 (2013).
- Bantignies, F., et al. Polycomb-dependent regulatory contacts between distant Hox loci in Drosophila. Cell. 144, 214-226 (2011).
- Engreitz, J. M., et al. The Xist lncRNA exploits three-dimensional genome architecture to spread across the X chromosome. Science. 341, 1237973(2013).
- Denholtz, M., et al. Long-range chromatin contacts in embryonic stem cells reveal a role for pluripotency factors and polycomb proteins in genome organization. Cell Stem Cell. 13, 602-616 (2013).
- Schoenfelder, S., et al. Polycomb repressive complex PRC1 spatially constrains the mouse embryonic stem cell genome. Nature Genetics. 47, 1179-1186 (2015).
- Kundu, S., et al. Polycomb Repressive Complex 1 generates discrete compacted domains that change during differentiation. Molecular Cell. 65, 432-446 (2017).
- Skok, J. A., Gisler, R., Novatchkova, M., Farmer, D., de Laat, W., Busslinger, M. Reversible contraction by looping of the Tcra and Tcrb loci in rearranging thymocytes. Nature Immunology. 8, 378-387 (2007).
- Zhang, Y., et al. Spatial organization of the mouse genome and its role in recurrent chromosomal translocations. Cell. 148, 908-921 (2012).
- Aymard, F., et al. Genome-wide mapping of long-range contacts unveils clustering of DNA double-strand breaks at damaged active genes. Nature Structural & Molecular Biology. 24, 353-361 (2017).
- Ryba, T., et al. Evolutionarily conserved replication timing profiles predict long-range chromatin interactions and distinguish closely related cell types. Genome Research. 20, 761-770 (2010).
- Pope, B. D., et al. Topologically associating domains are stable units of replication-timing regulation. Nature. 515, 402-405 (2014).
- Chandra, T., et al. Global reorganization of the nuclear landscape in senescent cells. Cell Reports. 10, 471-483 (2015).
- Carter, D., Chakalova, L., Osborne, C. S., Dai, Y. F., Fraser, P. Long-range chromatin regulatory interactions in vivo. Nature Genetics. 32, 623-626 (2002).
- Tolhuis, B., Palstra, R. J., Splinter, E., Grosveld, F., de Laat, W. Looping and interaction between hypersensitive sites in the active beta-globin locus. Molecular Cell. 10, 1453-1465 (2002).
- Amano, T., Sagai, T., Tanabe, H., Mizushina, Y., Nakazawa, H., Shiroishi, T. Chromosomal dynamics at the Shh locus: limb bud-specific differential regulation of competence and active transcription. Developmental Cell. 16, 47-57 (2009).
- Zuniga, A., et al. Mouse limb deformity mutations disrupt a global control region within the large regulatory landscape required for Gremlin expression. Genes & Development. 18, 1553-1564 (2004).
- Sagai, T., Hosoya, M., Mizushina, Y., Tamura, M., Shiroishi, T. Elimination of a long-range cis-regulatory module causes complete loss of limb-specific Shh expression and truncation of the mouse limb. Development. 132, 797-803 (2005).
- D'Haene, B., et al. Disease-causing 7.4 kb cis-regulatory deletion disrupting conserved non-coding sequences and their interaction with the FOXL2 promotor: implications for mutation screening. PLoS Genet. 5, e1000522(2009).
- Sur, I. K., et al. Mice lacking a Myc enhancer that includes human SNP rs6983267 are resistant to intestinal tumors. Science. 338, 1360-1363 (2012).
- Herranz, D., et al. A NOTCH1-driven MYC enhancer promotes T cell development, transformation and acute lymphoblastic leukemia. Nature Medicine. 20, 1130-1137 (2014).
- Deng, W., et al. Controlling long-range genomic interactions at a native locus by targeted tethering of a looping factor. Cell. 149, 1233-1244 (2012).
- Groschel, S., et al. A single oncogenic enhancer rearrangement causes concomitant EVI1 and GATA2 deregulation in leukemia. Cell. 157, 369-381 (2014).
- Lupianez, D. G., et al. Disruptions of topological chromatin domains cause pathogenic rewiring of gene-enhancer interactions. Cell. 161, 1012-1025 (2015).
- Franke, M., et al. Formation of new chromatin domains determines pathogenicity of genomic duplications. Nature. 538, 265-269 (2016).
- Dekker, J., Rippe, K., Dekker, M., Kleckner, N. Capturing chromosome conformation. Science. 295, 1306-1311 (2002).
- Simonis, M., et al. Nuclear organization of active and inactive chromatin domains uncovered by chromosome conformation capture-on-chip (4C). Nature Genetics. 38, 1348-1354 (2006).
- Zhao, Z., et al. Circular chromosome conformation capture (4C) uncovers extensive networks of epigenetically regulated intra- and interchromosomal interactions. Nature Genetics. 38, 1341-1347 (2006).
- Dostie, J., et al. Chromosome Conformation Capture Carbon Copy (5C): A massively parallel solution for mapping interactions between genomic elements. Genome Research. 16, 1299-1309 (2006).
- Lieberman-Aiden, E., et al. Comprehensive mapping of long-range interactions reveals folding principles of the human genome. Science. 326, 289-293 (2009).
- Belton, J. M., McCord, R. P., Gibcus, J. H., Naumova, N., Zhan, Y., Dekker, J. Hi-C: a comprehensive technique to capture the conformation of genomes. Methods. 58, 268-276 (2012).
- Mifsud, B., et al. Mapping long-range promoter contacts in human cells with high-resolution capture Hi-C. Nature Genetics. 47, 598-606 (2015).
- Schoenfelder, S., et al. The pluripotent regulatory circuitry connecting promoters to their long-range interacting elements. Genome Res. 25, 582-597 (2015).
- Javierre, B. M., et al. Lineage-specific genome architecture links enhancers and non-coding disease variants to target gene promoters. Cell. 167, 1369-1384 (2016).
- Wilson, N. K., et al. Integrated genome-scale analysis of the transcriptional regulatory landscape in a blood stem/progenitor cell model. Blood. 127, e12-e23 (2016).
- Freire-Pritchett, P., et al. Global reorganisation of cis-regulatory units upon lineage commitment of human embryonic stem cells. Elife. 6, (2017).
- Rubin, A. J., et al. Lineage-specific dynamic and pre-established enhancer-promoter contacts cooperate in terminal differentiation. Nature Genetics. 49, 1522-1528 (2017).
- Siersbaek, R., et al. Dynamic rewiring of promoter-anchored chromatin loops during adipocyte differentiation. Molecular Cell. 66, 420-435 (2017).
- Sahlen, P., et al. Genome-wide mapping of promoter-anchored interactions with close to single-enhancer resolution. Genome Biology. 16, 156(2015).
- Nagano, T., et al. Single-cell Hi-C reveals cell-to-cell variability in chromosome structure. Nature. 502, 59-64 (2013).
- Nagano, T., Varnai, C., Schoenfelder, S., Javierre, B. M., Wingett, S. W., Fraser, P. Comparison of Hi-C results using in-solution versus in-nucleus ligation. Genome Biology. 16, 175(2015).
- Wingett, S., et al. HiCUP: pipeline for mapping and processing Hi-C data. F1000 Res. 4, 1310(2015).
- Dryden, N. H., et al. Unbiased analysis of potential targets of breast cancer susceptibility loci by Capture Hi-C. Genome Research. 24, 1854-1868 (2014).
- Martin, P., et al. Capture Hi-C reveals novel candidate genes and complex long-range interactions with related autoimmune risk loci. Nature Communications. 6, 10069(2015).
- McGovern, A., et al. Capture Hi-C identifies a novel causal gene, IL20RA, in the pan-autoimmune genetic susceptibility region 6q23. Genome Biol.ogy. 17, 212(2016).
- Hodge, D., et al. A global role for EKLF in definitive and primitive erythropoiesis. Blood. 107, 3359-3370 (2006).
- Jager, R., et al. Capture Hi-C identifies the chromatin interactome of colorectal cancer risk loci. Nature Communications. 6, 6178(2015).
- Joshi, O., et al. Dynamic reorganization of extremely long-range promoter-promoter Interactions between two states of pluripotency. Cell Stem Cell. 17, 748-757 (2015).
- Hughes, J. R., et al. Analysis of hundreds of cis-regulatory landscapes at high resolution in a single, high-throughput experiment. Nature Genetics. 46, 205-212 (2014).
- Kolovos, P., et al. Targeted Chromatin Capture (T2C): A novel high-resolution high-throughput method to detect genomic interactions and regulatory elements. Epigenetics Chromatin. 7, 10(2014).
- Davies, J. O., et al. Multiplexed analysis of chromosome conformation at vastly improved sensitivity. Nature Methods. 13, 74-80 (2016).
- Mumbach, M. R., et al. HiChIP: efficient and sensitive analysis of protein-directed genome architecture. Nature Methods. 13, 919-922 (2016).
- Fullwood, M. J., et al. An oestrogen-receptor-alpha-bound human chromatin interactome. Nature. 462, 58-64 (2009).
- Maurano, M. T., et al. Systematic localization of common disease-associated variation in regulatory DNA. Science. 337, 1190-1195 (2012).
- Petersen, R., et al. Platelet function is modified by common sequence variation in megakaryocyte super enhancers. Nat. Commun. 8, 16058(2017).
- Akhtar, W., et al. Chromatin position effects assayed by thousands of reporters integrated in parallel. Cell. 154, 914-927 (2013).
- Cairns, J., et al. CHiCAGO: Robust detection of DNA looping interactions in Capture Hi-C data. Genome Biology. 17, 127(2016).
- Rajagopal, N., et al. High-throughput mapping of regulatory DNA. Nature Biotechnology. 34, 167-174 (2016).
Перепечатки и разрешения
Запросить разрешение на использование текста или рисунков этого JoVE статьи
Запросить разрешениеThis article has been published
Video Coming Soon
Авторские права © 2025 MyJoVE Corporation. Все права защищены