A subscription to JoVE is required to view this content. Sign in or start your free trial.
Method Article
إنتاج الجينات الحذف في الإشريكيّة القولونية بتوصيل P1 مع أشرطة الكاسيت المهربة مقاومة المضادات الحيوية
In This Article
Summary
وهنا يقدم بروتوكول لاستخدام الموجودة مسبقاً بنيات الحذف كاسيت مقاومة المضادات الحيوية كأساس لتحقيق طفرات الحذف في سلالات كولاي الأخرى. يمكن تعبئة هذه الطفرات الحذف وإدراجها في محور المقابلة من سلالة المستلم باستخدام توصيل عاثية P1.
Abstract
نهج أول دراسة مهمة الجينات غير معروف في البكتيريا خلق المغلوب هذا الجين. هنا، يمكننا وصف بروتوكول قوية وسريعة لنقل طفرات الجينات الحذف من سلالة الإشريكيّة القولونية واحد إلى آخر باستخدام توصيل المعمم مع عاثية P1. يتطلب هذا الأسلوب أن الطفرة تكون انتقائية (مثلاً، استناداً إلى اضطرابات الجينات باستخدام غرز كاسيت المضادات الحيوية). مثل هذه الأشرطة المضادات الحيوية يمكن تعبئتها من سلالة مانحة وأدخلت سلالة المستفيدة من تهم بسرعة وتوليد متحولة حذف جينات بسهولة. يمكن تصميم كاسيت المضادات الحيوية تشمل فلبس التعرف على المواقع التي تسمح بختان الإناث الكاسيت ريكومبيناسي الخاصة بالموقع لإنتاج المغلوب نظيفة مع تسلسل ندبة ~ 100-قاعدة-زوج-منذ فترة طويلة في الجينوم. إظهار البروتوكول بانقطاع الجين تاما الترميز عاملاً الجمعية المعنية في نشوء حيوي أوتوترانسبورتير، واختبار تأثير هذا المغلوب على نشوء حيوي ووظيفة هما أوتوترانسبورتير تريميريك أدهيسينس. على الرغم من حذف الجينات بتوصيل P1 قصوره، بسهولة وسرعة تنفيذه جعلها بديلاً جذاباً لأساليب أخرى لحذف الجينات.
Introduction
نهج أول مشترك لدراسة وظيفة الجين هو أداء الطفرات المغلوب ومراعاة النمط الظاهري الناتج عن ذلك. وهذا يسمى أيضا علم الوراثة العكسي. بكتيريا كولاي كانت العمود الفقري للبيولوجيا الجزيئية للسنوات السبعين الأخيرة، أو حتى، بسبب السهولة في استزراع وبه الانقياد للتلاعب بالجينات1. وقد وضعت عدة أساليب لإنتاج الجينات الحذف في كولاي، بما في ذلك علامة تبادل الطفرات2،3 ، وفي الآونة الأخيرة، ريكومبينيرينج باستخدام λ الأحمر أو ET راك نظم4،5 , 6.
في نظام مستخدمة على نطاق واسع، وتحل محلها متواليات ترميز الجينات الفردية كاسيت مقاومة للمضادات الحيوية التي يمكن فيما بعد أن اقتطعت من5،كروموسوم7. يتم استبدال تسلسل الترميز، على سبيل المثال بكاسيت مقاومة كاناميسين (أساسه)، الذي يحيط به فلبس (حزب العمل الفيجي) الاعتراف بالمواقع المستهدفة (معاهدة سجل الأفلام) على كلا جانبي. يتم التعرف على مواقع FRT قبل recombinase حزب العمل الفيجي، الذي يتوسط جزئ خاصة بالموقع بين المواقع FRT مما يؤدي إلى حذف الكاسيت كان. وبهذه الطريقة، يمكن تحقيق حذف كامل لتسلسل الجين المعطى للترميز، تاركين وراءهم تسلسل ندبة الحد أدنى فقط من حوالي 100 قاعدة أزواج (bp) (الشكل 1).
فقط على مدى عقد من الزمان، وقد وضعت ما يسمى مجموعة كيو. هذه هي مكتبة بكتيرية استناداً إلى أحد مختبرات قياسية السلالة K12 كولاي ، حيث تم حذفها تقريبا جميع الجينات غير الضرورية على حدة بواسطة λ جزئ الأحمر7،8. وقد استنساخ ضمن هذه المجموعة كل تسلسل ترميز واحد استبداله كاسيت مقاومة كان المهربة. جمع كيو ثبت أن تكون أداة مفيدة للعديد من التطبيقات9. واحد مثل هذا الطلب هو إنتاج طفرات الحذف في سلالات كولاي الأخرى. يمكن تعبئة الكاسيت كان من استنساخ حذف معين قبل عموما ترانسدوسينج باكتيريوفاجيس، مثل P110،،من1112،،من1314. ثم يمكن استخدام أسهم بالعاثية أعدت من سلالة من هذا القبيل لتصيب مستلم سلالة كولاي من الفائدة، حيث تردد منخفض ولكن موثوق بها كان المنطقة المحتوية على كاسيت يمكن إدراجها في جينوم المتلقي من جزئ مثلى (الشكل 2). يمكن تحديد ترانسدوكتانتس للنمو عن طريق الوسائط التي تحتوي على أساسه. وفي أعقاب ذلك، إذا كان المطلوب هو إزالة كاسيت المقاومة للمضادات الحيوية، يمكن توفيره recombinase حزب العمل الفيجي إلى سلالة ترانسدوكتانت في ترانس. بعد علاج بلازميد المحتوية على حزب العمل الفيجي، الذي يحمل علامة مقاومة الأمبيسلّين (أمبير)، يتم فحص الحيوانات المستنسخة أساسه ومراعية لامبير ل، والختان الصحيح لتسلسل البرية من نوع الترميز وكاسيت كان يتم التحقق من قبل مستعمرة [بكر].
هنا، يقدم بروتوكول مفصلاً، تصف كل خطوة من الخطوات في إنتاج المغلوب سلالة كولاي استناداً إلى الاستراتيجية المبينة أعلاه. على سبيل مثال، يظهر حذف من الجينات تاما . بترميز تاما بروتين بيتا لبرميل غشاء خارجي الذي هو جزء من النقل والجمعية الوحدة النمطية (TAM)، الذي تشترك في نشوء حيوي لبعض البروتينات أوتوترانسبورتير وبيلي15،،من1617. ثم استخدم هذه السلالة المغلوب لدراسة تأثير الحذف تاما على نشوء حيوي هما أوتوترانسبورتير تريميريك أدهيسينس (TAAs) وأدهيسين واليرسينيا يادا كولاي الغلوبولين المناعي (Ig)-ملزمة "ﻷعمالهم عيبد" 18،19.
Protocol
1-سلالات والبلازميدات
-
السلالات البكتيرية
- استخدام سلالات BW25113 كولاي 5، JW4179 (BW25113 tamA::kan)7، BL21(DE3)20و BL21ΔABCF21. انظر الجدول للمواد للحصول على مزيد من المعلومات.
-
باكتيريوفاجيس
- استخدام بالعاثية P1فير لتوصيل عامة. تخزين في بالعاثية أسهم سائلة مع بضع قطرات من كلوروفورم (راجع الخطوة 2، 2). للحصول على مزيد من المعلومات، راجع الجدول للمواد.
-
البلازميدات
- استخدام البلازميدات التالية في هذا البروتوكول: pCP2022ويادا pIBA223pEibD1024. والبلازميدات التحكم، استخدم باسك-IBA2 و pET22b (انظر الجدول للمواد).
-
ظروف النمو
- تنتشر البكتيريا في ليسوجيني مرق (رطل) متوسطة25 مع قوي يهز (180-200 لفة في الدقيقة) في 37 درجة مئوية أو 30 درجة مئوية في حالة BL21ΔABCF والسلالات التي تحتوي على pCP20.
- أداء بلازميد علاج في 43 درجة مئوية.
- لوسيلة متينة، وتكملة رطل مع أجار 1% (w/v).
- لاجار العلوي، الملحق رطل مع 0.7% أجار و 10 مم كاكل2 والاوتوكلاف المتوسطة. تستخدم شركة نفط الجنوب متوسطة للانتعاش بعد انهانسر26.
- استخدام التركيزات التالية للمضادات الحيوية: 100 ميكروغرام/مل لامبير و 25 ميكروغرام/مل لشخص
2-إعداد بالعاثية ليستي
-
الإصابة بسلالة المانحين
- تنمو سلالة المانحة JW4197 في 5 مل متوسطة رطل تستكمل مع 10 مم كاكل2 وكان اختيارياً (25 ميكروغرام/مل) كثافة بصرية في 600 نانومتر (OD600) ~ 1.0. قياس القيمة600 OD باستخدام جهاز المطياف الضوئي.
- جعل سلسلة تمييع المخزون بالعاثية P1 الموجودة في الأجل المتوسط ليبره: أوصى بتخفيف ما بين 10-3 إلى 10-7.
- ميكس 200 ميكروليتر من تعليق البكتيرية و 100 ميكروليتر من إضعاف بالعاثية معين في أنبوب الطرد المركزي 15 مل أو ما يعادلها. إعداد العديد من الأنابيب كتخفيف بالعاثية. احتضان هذه الأنابيب لمدة 20 دقيقة عند 37 درجة مئوية دون المصافحة.
- إضافة ~ 3 مل أجار أعلى المنصهر (~ 50 درجة مئوية) وتستكمل مع 10 ملم كاكل2 للأنابيب وخلط المحتويات جيدا قبل فورتيكسينج الأنابيب قريبا، وصب الخلائط على لوحات رطل بريوارميد جعل الطبقات حتى.
- احتضان لوحات بين عشية وضحاها في 37 درجة مئوية.
-
إعداد ليستي
- وفي اليوم التالي اختر لوحة مع نمو لويحات بالعاثية شبه المتلاقية. على لوحة شبه المتلاقية، حوالي نصف مساحة اللوحة من الواضح (الشكل 3).
- كشط الطبقة الأعلى أجار من صفيحة باستخدام حلقة تطعيم أو أداة مماثلة واجار أعلى في أنبوب الطرد مركزي. أضف 1 – 2 مل رطل وقطره من كلوروفورم ودوامه الأنبوب بشدة ل ~ 1 دقيقة إضافة كلوروفورم في غطاء دخان.
- الطرد المركزي في أنبوب لمدة 15 دقيقة في 4000 x ز أو أسرع بيليه أجار والخلايا البكتيرية.
- نقل المادة طافية إلى أنبوب ميكروسينتريفوجي جديدة، تجنب تحمل أكثر من أي حطام من بيليه. 2 إضافة قطرات كلوروفورم وتخزين في 4-10 درجة مئوية. إضافة كلوروفورم في غطاء دخان. لا تجمد بالعاثية ليستي كهذا سيؤدي إلى انخفاض كبير في عدد الجسيمات المعدية.
-
تحديد عيار ليستي
- ينمو BW25113 في رطل وتستكمل مع 10 ملم كاكل2 في 37 درجة مئوية حتى تصل إلى الثقافة التطوير التنظيمي600 ~ 1.0.
- إعداد سلسلة تمييع بالعاثية في رطل (مثلاً، 10-6–10-9).
ملاحظة: كن حذراً إلى بيبيت العينة من الجزء العلوي من ليساتي لتجنب نقل كلوروفورم لتخفيف. - ميكس 200 ميكروليتر من تعليق البكتيرية و 100 ميكروليتر من إضعاف بالعاثية معين في أنبوب الطرد المركزي 15 مل أو ما يعادلها. إعداد العديد من الأنابيب كتخفيف بالعاثية. احتضان هذه الأنابيب لمدة 20 دقيقة عند 37 درجة مئوية دون المصافحة.
- إضافة ~ 3 مل أجار أعلى المنصهر (~ 50 درجة مئوية) وتستكمل مع 10 ملم كاكل2 للأنابيب وخلط المحتويات جيدا قبل فورتيكسينج الأنابيب قريبا، وصب الخليط على لوحات رطل بريوارميد جعل الطبقات حتى.
- احتضان لوحات بين عشية وضحاها في 37 درجة مئوية.
- وفي اليوم التالي إحصاء عدد اللوحات (مناطق واضحة في حصيرة البكتيرية) لكل لوحة وحساب عيار من ليستي باستخدام الصيغة التالية:

على سبيل المثال: في لوحات مع تخفيف 10-7و 10-8و 10-9، هناك 231، 27، ولويحات 2، على التوالي. كما تم استخدام 100 ميكروليتر من كل تمييع للعدوى، وأن يتم التعبير عنها عيار كتشكيل اللوحة وحدات (بفو)/مل، عامل الطلاء هو 10. يتم إدخال هذه الأرقام في الصيغة: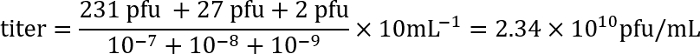
وهكذا، عيار حوالي 2 × 1010 بالعاثية المعدية الجسيمات/مل.
3-توصيل P1
-
إعداد الخلايا المتلقية
- تنمو سلالة المتلقي BL21ΔABCF في رطل استكملها بكثافة بصرية في 600 نانومتر (OD600) ~ 1.0. استخدام جهاز المطياف الضوئي قياس قيمة600 OD.
- حساب حجم بالعاثية ليستي اللازمة لتحقيق تعدد العدوى (وزارة الداخلية) في القيمة 0.5. لحساب وزارة الداخلية، تقدير عدد البكتيريا استناداً إلى OD600 للثقافة. افترض أن قيمة600 OD 1.0 يناظر ~ 109 زيمبابوي/مل. حساب حجم المطلوبة استناداً إلى عيار معروف.
على سبيل المثال (استناداً إلى عيار حسابها في المثال بعد الخطوة 2.3.6):
- إضافة كاكل2 إلى الثقافة المتلقية سلالة إلى 10 ملم، ومزيج. أخذ 1 مل الثقافة لتوصيل.
-
توصيل المنفذ
- إضافة حجم المناسبة بالعاثية ليساتي إلى 1 مل ثقافة المتلقية (بما في ذلك كاكل2 في 10 ملم) المحسوبة في الخطوة 3.1.2 وتخلط بلطف.
ملاحظة: احرص على "الماصة؛" العينة من الجزء العلوي من لتجنب نقل كلوروفورم إلى المزيج. - بشكل ثابت في احتضان هذا المزيج لمدة 20 دقيقة عند 37 درجة مئوية.
- إيقاف العدوى عن طريق إضافة سترات الصوديوم، الأس الهيدروجيني 5.5، إلى 100 مم.
- الطرد المركزي البكتيريا (5,000 س ز 2 دقيقة) وإزالة المادة طافية، ثم ريسوسبيند عليها في 1 مل رطل جديدة تستكمل مع سترات الصوديوم 100 مم، الرقم الهيدروجيني 5.5.
- تغسل الخلايا أكثر مرتين كما في الخطوة 3.2.4 لضمان إزالة فاجيس الحرة والكالسيوم.
- ريسوسبيند البكتيريا في 1 مل رطل جديدة تستكمل مع سترات الصوديوم 100 مم، الرقم الهيدروجيني 5.5. احتضان هذه البكتيريا في 30 درجة مئوية ح 1 مع الهز (> 100 لفة في الدقيقة).
- تجمع البكتيريا بالطرد المركزي (5,000 س ز 2 دقيقة) وريسوسبيند لهم في ~ 100 ميكروليتر من رطل مع سترات الصوديوم 100 مم، الرقم الهيدروجيني 5.5.
- انتشار البكتيريا على صفيحة رطل وتستكمل مع كان في 25 ميكروغرام/مل وسترات الصوديوم 10 مم، الرقم الهيدروجيني 5.5، وتنمو البكتيريا في 30 درجة مئوية حتى تظهر المستعمرات (~ 24 h).
- إضافة حجم المناسبة بالعاثية ليساتي إلى 1 مل ثقافة المتلقية (بما في ذلك كاكل2 في 10 ملم) المحسوبة في الخطوة 3.1.2 وتخلط بلطف.
-
تحديد ترانسدوكتانتس
- مرة واحدة قد نمت المستعمرات على لوحة التحديد، ريستريك لهم على أساسه + رطل للمستعمرات واحدة وينمو بمعدل 30 درجة مئوية حتى تظهر المستعمرات واحدة.
4-نسق الكاسيت كان
-
التحول مع جزئ بلازميد
- جعل اليكتروكومبيتينت سلالة مقاومة لكان BL21ΔABCF.
- تنمو السلالة من مستعمرة واحدة في الطازجة رطل (5 مل) وتستكمل مع كان (25 ميكروغرام/مل) في 30 درجة مئوية حتى يصبح OD600 ~0.5 – 0.7.
- من هنا، القيام بالخطوات التالية عند 4 درجة مئوية أو على الجليد. الطرد المركزي البكتيريا (5,000 س ز لمدة 10 دقائق) وإزالة المادة طافية.
- أغسل بيليه الخلية مع الماء المقطر المثلج مرتين بتكرار الخطوة السابقة الطرد المركزي، وأخيراً، ريسوسبيند بيليه الخلية في 100 ميليلتر من المثلج والغليسيرول 10%.
- أن تهدئة 1 مم انهانسر الترعة على الجليد.
- إضافة pg 1 من بلازميد الحمض النووي (pCP20) إلى تعليق خلية ومزيج التعليق بلطف وأنه نقل إلى ومبومو انهانسر تبريد.
- تعيين اليكتروبوراتور إلى 1.8 كيلو فولت واليكتروبوراتي الخلايا.
- إنقاذ الخلايا المحولة بإضافة 1 مل من شركة نفط الجنوب المتوسطة وتزايد عليها ح 1 في 30 درجة مئوية.
- لوحة 100 ميليلتر الخلايا LB + أمبير لوحات وتنمو الخلايا عند 30 درجة مئوية بين عشية وضحاها.
- جعل اليكتروكومبيتينت سلالة مقاومة لكان BL21ΔABCF.
-
تحريض جزئ
- لوحة المستعمرات اختيار واحد من رطل + أمبير وتطعيم رطل جديدة مع حذف كل المضادات الحيوية.
- تنمو الخلايا في درجة الحرارة غير المتساهلة (43 درجة مئوية) بين عشية وضحاها للحث على التعبير عن حزب العمل الفيجي ريكومبيناسي.
-
تحديد ريكومبينانتس
- جعل تخفيف المسلسل ولوحة 50 ميليلتر من5-10 106 إضعاف على ألواح غير انتقائية وأنها تنمو بين عشية وضحاها في 30 درجة مئوية.
5-تحقق حذف الجينات
-
التحقق من فقدان بلازميد وممارسو الناجحة
- الانتصارات المستعمرات واحدة من لوحات إعدادها في الخطوة 4.3.1 على أساسه + رطل + أمبير، رطل، ولوحات رطل دون المضادات الحيوية، بهذا الترتيب. للمساعدة في streaking، استخدم شبكة مستعمرة (انظر 1 ملف تكميلي). تنمو لوحات عند 30 درجة مئوية حتى تظهر المستعمرات (~ 24 h).
- اختيار 10 – 20 الحيوانات المستنسخة التي نمت على اللوحة غير انتقائية ولكن فشلت في النمو في وسائل الإعلام الانتقائي لمزيد من التحقق.
-
إضافية للتحقق من مستعمرة [بكر]
- أداء مستعمرة [بكر] مع الإشعال المرافقة تاما الترميز تسلسل (انظر الجدول للمواد).
- إعداد مزيج PCR رئيسي. مقدار المزيج المطلوبة يعتمد على العدد المستعمرات لفحصها (انظر المثال في الجدول 1). مزيج الكواشف على الجليد، إضافة بوليميريز آخر.
- مزيج مزيج الرئيسي بكر جيدا، ثم الاستغناء عن 20 ميليلتر في أنابيب قطاع بكر. اختيار كمية صغيرة من كل مستعمرة فحص استخدام تلميح ماصة معقمة وإضافته إلى أنبوب. تذكر أن تدرج سلالة المستلم الأصلي للمقارنة. بشكل اختياري، وتشمل أيضا سلالة المانحين.
- تشغيل رد فعل بكر (انظر الجدول 2 للبرنامج المستخدم).
- تعد من 1% [اغروس] هلام.
- قياس 0.5 غرام من [اغروس] هلام 50 مل، وإضافة 50 مل من المخزن المؤقت تاي (40 مم تريس وحمض الخليك 20 مم 1 مم يدتا، pH 8.0). حرارة الخليط في فرن ميكروويف حتى قد حلت جميع [اغروس].
- مرة واحدة قد حلت [اغروس]، بارد الحل إلى ما يقرب من 50 درجة مئوية، ثم قم بإضافة 5 ميكروليتر من تلطيخ صبغ الحمض النووي. مزيج الحل جيد وأنه صب في علبة جل في غرفة صب. إدراج صف واحد أو أكثر من الامشاط جيدا حيث لا توجد آبار كافية لجميع العينات، فضلا عن علامات الحجم الجزيئي. السماح للهلام لمدة 30 دقيقة.
- بمجرد اكتمال تشغيل PCR، إضافة 4 ميليلتر من 6 × صبغ تحميل الحمض النووي لكل عينة. ضع الجل في قاعة التفريد وإضافة تاي المخزن المؤقت حتى يتم تغطية الآبار.
- تطبيق 10 – 15 ميليلتر من كل بكر كرد فعل لبئر في الهلام. أيضا إضافة 5 ميليلتر علامة الحجم الجزيئي. ثم قم بتشغيل جل لمدة 30 دقيقة في 75 الخامس.
- بمجرد اكتمال التشغيل صورة جل تستخدم تصوير الضوء الأزرق.
6-غير ذلك من التقنيات
-
تعبير البروتين
ملاحظة: قد تم التعبير عن اختبار البروتينات (يادا وعيبد)23،24ووصف بالتفصيل في مكان آخر. هذا ملخص موجز للخطوات الرئيسية.- تحويل BL21ΔABCF Δث تاماايث والبلازميدات اللازمة (يادا pIBA2 و pEibD10 ووالبلازميدات التحكم المقابلة) وحدد ترانسفورمانتس رطل + أمبير.
- للتعبير البروتين، تطعيم 100 مل المتوسطة رطل + أمبير مع 1 مل ثقافة بين عشية وضحاها تحول البكتيريا وتنمو هذه في 30 درجة مئوية حتى سجل منتصف المرحلة، في الوقت الذي، والحث على إنتاج البروتين مع أما أنهيدروتيتراسيكليني (في 100 نانوغرام/مليلتر) أو الأيزوبروبيل ثيوجالاكتوسيدي (في 0.5 مم).
- بعد قياس التعكر الثقافات ح 2 الاستقراء عند 30 درجة مئوية، وجمع عدد من الخلايا المقابلة إلى 50 مل OD600 = 1.0 من سينتريفوجينج الثقافات لمدة 15 دقيقة في س 4,000 ز. أغسل بيليه x 1 مع 10 ملم حبيس، ودرجة الحموضة 7.4، ومن ثم أما تخزينها في-20 درجة مئوية أو العملية كذلك كما هو الحال في الخطوة 6، 2.
-
استخراج الغشاء الخارجي
ملاحظة: يتم استخراج الغشاء الخارجي كما هو موضح بالتفصيل قبل ليو et al. 27-فيما يلي موجز للخطوات الرئيسية.- ريسوسبيند بيليه الخلية في 1 مل 10 مم حبيس، درجة الحموضة 7.4، تستكمل مع ملم 10 MgCl2 والحركة2، lysozyme (0.1 مغ/مل)، وقليل من الدناز أنا.
- الخلايا (مثلاً، استخدام الخافق حبة).
- الطرد المركزي الخلايا قريبا (2 دقيقة في س 15,600 ز) إزالة الحطام خلية وتحريك المادة طافية على أنبوب جديد.
- الطرد المركزي الخلايا لمدة 30 دقيقة في س 16,000 ز، بعد ذلك، ريسوسبيند بيليه في ميكروليتر 400 من 1% N-لوريل ساركوسيني في 10 ملم حبيس، درجة الحموضة 7.4.
- احتضان الخلايا مع الإثارة لمدة 30 دقيقة في درجة حرارة الغرفة، بعد ذلك، الطرد المركزي لهم لمدة 30 دقيقة كما ذكر أعلاه.
- أغسل بيليه x 1 مع 200 ميكروليتر من 10 ملم حبيس، درجة الحموضة 7.4، شفافة وثم ريسوسبيند أنه في 30 ميكروليتر من 10 ملم حبيس وإضافة 10 ميكروليتر من 4 × الحزب الديمقراطي الصربي صفحة نموذج المخزن المؤقت.
-
فحوصات النشاط
ملاحظة: إجراء فحوصات الحزب الديمقراطي الصربي صفحة والنشاط يادا وعيبد ك وصف28. ويرد أدناه الخطوات الأساسية. -
الحزب الديمقراطي الصربي صفحة
- الحرارة العينات عند 50 درجة مئوية لمدة 5 دقائق قبل تحميلها على جل بولياكريلاميدي، لتجنب يشوه البروتينات.
- بعد الانفصال في الحزب الديمقراطي الصربي صفحة، نقل البروتينات إلى غشاء ثنائي الفلوريد (PVDF) الفينيليدن.
- بعد النقل، كتلة الغشاء مع مسحوق حليب منزوع الدسم 2% في برنامج تلفزيوني.
-
وصمة عار في أقصى غرب يادا-الكولاجين
- وبعد تجميد، إضافة نوع الكولاجين البقري المخفف أنا في المخزن المؤقت لحظر لتركيز من 10 ميكروغرام/مل واحتضان الغشاء ح 1.
- يغسل الغشاء 2 x مع برنامج تلفزيوني + 0.05% Tween20 (برنامج تلفزيوني-T).
- إضافة جسم الأولية (COL الكولاجين المضادة [مونوكلونل]-1) للغشاء، والمخفف 1:2,000 في المخزن المؤقت لحظر.
- بعد حضانة الغشاء ح 1، يغسل 2 x كما هو مذكور في الخطوة 6.3.2.2 ثم قم بإضافة جسم الثانوي [الماعز الماوس المضادة IgG-الفجل البيروكسيديز (HRP) المتقارن]، المضيفين المخفف في المخزن المؤقت لحظر.
- احتضان الغشاء ح 1، ثم غسله x 2 مع برنامج تلفزيوني-ت. إضافة تعزيز الركيزة تشيميلومينيسسينت للغشاء طبقاً لإرشادات الشركة المصنعة، والكشف عن الفرقة باستخدام كاميرا CCD.
-
مفتش عيبد مقايسة الربط
- وبعد تجميد، إضافة تضعف جسم ثانوي (الماعز المضادة أرنب HRP)، 1:2,000 في المخزن المؤقت لحظر.
- احتضان الغشاء ح 1، ثم غسله x 2 مع برنامج تلفزيوني. تنفيذ الكشف عن تشيميلومينيسسينت كما هو مذكور في الخطوة 6.3.2.5.
النتائج
جيل من تاما المغلوب من BL21ΔABCF:
وقد استخدمت سابقا الاستراتيجية المبينة أعلاه لإنتاج سلالة مشتقة من BL21(DE3)، سلالة مختبر قياسية المستخدمة لإنتاج البروتين، وهو الأمثل لإنتاج البروتين الغشاء الخارجي ودعا BL21ΔABCF21. ويفتقر إلى أرب...
Discussion
توصيل P1 طريقة سريعة وقوية وموثوق بها لتوليد الجينات الحذف في كولاي. وهذا يتجلى هنا ترانسدوسينج متحولة حذف تاما من سلالة مانحة كيو إلى مستلم المستمدة من BL21. المراحل الرئيسية في عملية توصيل يتم إنتاج ترانسدوسينج ليستي، توصيل نفسها، وختان كاسيت المقاومة أساسه، والتحقق من المغلوب ب...
Disclosures
الكتاب ليس لها علاقة بالكشف عن.
Acknowledgements
تم الحصول على سلالات مجموعة كيو من المشروع بيوريسورسي الوطني (رجال، اليابان): كولاي. ونحن نشكر "ديرك لينكه" (قسم العلوم البيولوجية، جامعة أوسلو) على دعمه المستمر. تم تمويل هذا العمل من قبل مجلس البحوث منحة النرويج الشباب الباحث 249793 (لجاك ليو جيم).
Materials
| Name | Company | Catalog Number | Comments |
| Strains | |||
| E. coli BW25113 | NIG | ME6092 | Wild-type strain of Keio collection |
| E. coli BL21(DE3) | Merck | 69450-3 | Expression strain |
| E. coli BL21DABCF | Addgene | 102270 | Derived from BL21(DE3) |
| E. coli JW4179 | NIG | JW4179-KC | tamA deletion mutant |
| P1 vir | NIG | HR16 | Generally transducing bacteriophage |
| Plasmids | |||
| pCP20 | CGSC | 14177 | conditionally replicating plasmid with FLP |
| pASK-IBA2 | IBA GmbH | 2-1301-000 | expression vector |
| pEibD10 | N/A | N/A | for production of EibD; plasmid available on request |
| pET22b+ | Merck | 69744-3 | expression vector |
| pIBA2-YadA | N/A | N/A | for production of YadA; plasmid available on request |
| Chemicals | |||
| Acetic acid | ThermoFisher | 33209 | |
| Agar | BD Bacto | 214010 | |
| Agarose | Lonza | 50004 | |
| Ampicillin | Applichem | A0839 | |
| Anhydrotetracycline | Abcam | ab145350 | |
| anti-collagen type I antibody COL-1 | Sigma | C2456 | |
| Bovine collagen type I | Sigma | C9791 | |
| Calcium chloride | Merck | 102382 | |
| Chloroform | Merck | 102445 | |
| Di-sodium hydrogen phosphate | VWR | 28029 | |
| DNA dye | Thermo | S33102 | |
| DNA molecular size marker | New England BioLabs | N3232S | |
| DNase I | Sigma | DN25 | |
| dNTP mix | New England Biolabs | N0447 | |
| ECL HRP substrate | Advansta | K-12045 | |
| EDTA | Applichem | A2937 | |
| Glycerol | VWR | 24388 | |
| goat anti-mouse IgG-HRP | Santa Cruz | sc-2005 | |
| goat anti-rabbit IgG-HRP | Agrisera | AS10668 | |
| HEPES | VWR | 30487 | |
| Isopropyl thiogalactoside | VWR | 43714 | |
| Kanamycin | Applichem | A1493 | |
| Lysozyme | Applichem | A4972 | |
| Magnesium chloride | VWR | 25108 | |
| Manganese chloride | Sigma | 221279 | |
| N-lauroyl sarcosine | Sigma | L9150 | |
| Skim milk powder | Sigma | 70166 | |
| Sodium chloride | VWR | 27808 | |
| tamA forward primer | Invitrogen | N/A | Sequence 5'-GAAAAAAGG ATATTCAGGAGAAAATGTG-3' |
| tamA reverse primer | Invitrogen | N/A | Sequence 5'-TCATAATT CTGGCCCCAGACC-3' |
| Taq DNA polymerase | New England Biolabs | M0267 | |
| Tri-sodium citrate | Merck | 106448 | |
| Tryptone | VWR | 84610 | |
| Tween20 | Sigma | P1379 | |
| Yeast extract | Merck | 103753 | |
| Equipment | |||
| Agarose gel electrophoresis chamber | Hoefer | SUB13 | |
| Bead beater | Thermo | FP120A-115 | |
| CCD camera | Kodak | 4000R | |
| Electroporation cuvettes | Bio-Rad | 165-2089 | |
| Electroporation unit | Bio-Rad | 1652100 | |
| Gel imager | Nippon Genetics | GP-03LED | |
| Incubating shaker | Infors HT | Minitron | |
| Incubator | VWR | 390-0482 | |
| Microcentrifuge | Eppendorf | 5415D | |
| Microwave oven | Samsung | CM1099A | |
| PCR machine | Biometra | Tpersonal | |
| PCR strips | Axygen | PCR-0208-CP-C | |
| pH meter | Hanna Instruments | HI2211-01 | |
| PVDF membrane | ThermoFisher | 88518 | |
| SDS-PAG electrophoresis chamber | ThermoFisher | A25977 | |
| Tabletop centrifuge | Beckman Coulter | B06322 | |
| Vortex mixer | Scientific Industries | SI-0236 | |
| Water bath | GFL | D3006 | |
| Wet transfer unit | Hoefer | TE22 |
References
- Blount, Z. D. The unexhausted potential of E. coli. eLife. 4, e05826 (2015).
- Hamilton, C. M., Aldea, M., Washburn, B. K., Babitzke, P., Kushner, S. R. New method for generating deletions and gene replacements in Escherichia coli. Journal of Bacteriology. 171 (9), 4617-4622 (1989).
- Link, A. J., Phillips, D., Church, G. M. Methods for generating precise deletions and insertions in the genome of wild-type Escherichia coli: application to open reading frame characterization. Journal of Bacteriology. 179 (20), 6228-6237 (1997).
- Sawitzke, J. A., Thomason, L. C., Costantino, N., Bubunenko, M., Datta, S., Court, D. L. Recombineering: in vivo genetic engineering in E. coli, S. enterica, and beyond. Methods in Enzymology. 421, 171-199 (2007).
- Datsenko, K. A., Wanner, B. L. One-step inactivation of chromosomal genes in Escherichia coli K-12 using PCR products. Proceedings of the National Academy of Sciences of the United States of America. 97 (12), 6640-6645 (2000).
- Muyrers, J. P., Zhang, Y., Stewart, A. F. Techniques: Recombinogenic engineering--new options for cloning and manipulating DNA. Trends in Biochemical Sciences. 26 (5), 325-331 (2001).
- Baba, T., et al. Construction of Escherichia coli K-12 in-frame, single-gene knockout mutants: the Keio collection. Molecular Systems Biology. 2, (2006).
- Yamamoto, N., et al. Update on the Keio collection of Escherichia coli single-gene deletion mutants. Molecular Systems Biology. 5, 335 (2009).
- Baba, T., Huan, H. -. C., Datsenko, K., Wanner, B. L., Mori, H. The applications of systematic in-frame, single-gene knockout mutant collection of Escherichia coli K-12. Methods in Molecular Biology. 416, 183-194 (2008).
- Chattopadhyay, M. K., Tabor, C. W., Tabor, H. Polyamines are not required for aerobic growth of Escherichia coli: preparation of a strain with deletions in all of the genes for polyamine biosynthesis. Journal of Bacteriology. 191 (17), 5549-5552 (2009).
- Xie, X., Wong, W. W., Tang, Y. Improving simvastatin bioconversion in Escherichia coli by deletion of bioH. Metabolic Engineering. 9 (4), 379-386 (2007).
- Maeda, T., Sanchez-Torres, V., Wood, T. K. Escherichia coli hydrogenase 3 is a reversible enzyme possessing hydrogen uptake and synthesis activities. Applied Microbiology and Biotechnology. 76 (5), 1035-1042 (2007).
- Meehan, B. M., Landeta, C., Boyd, D., Beckwith, J. The disulfide bond formation pathway is essential for anaerobic growth of Escherichia coli. Journal of Bacteriology. 199 (16), (2017).
- Niba, E. T. E., Naka, Y., Nagase, M., Mori, H., Kitakawa, M. A genome-wide approach to identify the genes involved in biofilm formation in E. coli. DNA Research. 14 (6), 237-246 (2008).
- Heinz, E., et al. Conserved features in the structure, mechanism, and biogenesis of the inverse autotransporter protein family. Genome Biology and Evolution. 8 (6), 1690-1705 (2016).
- Selkrig, J., et al. Discovery of an archetypal protein transport system in bacterial outer membranes. Nature Structural & Molecular Biology. 19 (5), 506-510 (2012).
- Stubenrauch, C., et al. Effective assembly of fimbriae in Escherichia coli depends on the translocation assembly module nanomachine. Nature Microbiology. 1 (7), 16064 (2016).
- Leo, J. C., Goldman, A. The immunoglobulin-binding Eib proteins from Escherichia coli are receptors for IgG Fc. Molecular Immunology. 46 (8-9), 1860-1866 (2009).
- Mühlenkamp, M., Oberhettinger, P., Leo, J. C., Linke, D., Schütz, M. S. Yersinia adhesin A (YadA) - Beauty & beast. International Journal of Medical Microbiology. 305 (2), 252-258 (2015).
- Studier, F. W., Moffatt, B. A. Use of bacteriophage T7 RNA polymerase to direct selective high-level expression of cloned genes. Journal of Molecular Biology. 189 (1), 113-130 (1986).
- Meuskens, I., Michalik, M., Chauhan, N., Linke, D., Leo, J. C. A new strain collection for improved expression of outer membrane proteins. Frontiers in Cellular and Infection Microbiology. 7, 464 (2017).
- Cherepanov, P. P., Wackernagel, W. Gene disruption in Escherichia coli: TcR and KmR cassettes with the option of Flp-catalyzed excision of the antibiotic-resistance determinant. Gene. 158 (1), 9-14 (1995).
- Grosskinsky, U., et al. A conserved glycine residue of trimeric autotransporter domains plays a key role in Yersinia adhesin A autotransport. Journal of Bacteriology. 189 (24), 9011-9019 (2007).
- Leo, J. C., et al. The structure of E. coli IgG-binding protein D suggests a general model for bending and binding in trimeric autotransporter adhesins. Structure. 19 (7), 1021-1030 (1993).
- Bertani, G. Studies on lysogenesis. I. The mode of phage liberation by lysogenic Escherichia coli. Journal of Bacteriology. 62 (3), 293-300 (1951).
- Hanahan, D. Studies on transformation of Escherichia coli with plasmids. Journal of Molecular Biology. 166 (4), 557-580 (1983).
- Leo, J. C., Oberhettinger, P., Linke, D. Assessing the outer membrane insertion and folding of multimeric transmembrane β-barrel proteins. Methods in Molecular Biology. , 157-167 (2015).
- Mikula, K. M., Leo, J. C., Łyskowski, A., Kedracka-Krok, S., Pirog, A., Goldman, A. The translocation domain in trimeric autotransporter adhesins is necessary and sufficient for trimerization and autotransportation. Journal of Bacteriology. 194 (4), 827-838 (2012).
- Thomason, L. C., Costantino, N., Court, D. L., Ausubel, F. M. E. coli genome manipulation by P1 transduction. Current Protocols in Molecular Biology. , (2007).
- Franklin, N. C. Mutation in gal U gene of E. coli blocks phage P1 infection. Virology. 38 (1), 189-191 (1969).
- Jeong, H., et al. Genome sequences of Escherichia coli B strains REL606 and BL21(DE3). Journal of Molecular Biology. 394 (4), 644-652 (2009).
- Greene, E. C. DNA Sequence Alignment during Homologous Recombination. Journal of Biological Chemistry. 291 (22), 11572-11580 (2016).
- Watt, V. M., Ingles, C. J., Urdea, M. S., Rutter, W. J. Homology requirements for recombination in Escherichia coli. Proceedings of the National Academy of Sciences of the United States of America. 82 (14), 4768-4772 (1985).
- Ho, T. D., Waldor, M. K. Enterohemorrhagic Escherichia coli O157:H7 gal mutants are sensitive to bacteriophage P1 and defective in intestinal colonization. Infection and Immunity. 75 (4), 1661-1666 (2006).
- Ikeda, H., Tomizawa, J. I. Transducing fragments in generalized transduction by phage P1. I. Molecular origin of the fragments. Journal of Molecular Biology. 14 (1), 85-109 (1965).
- Murooka, Y., Harada, T. Expansion of the host range of coliphage P1 and gene transfer from enteric bacteria to other Gam-negative bacteria. Applied and Environmental Microbiology. 38 (4), 754-757 (1979).
- Goldberg, R. B., Bender, R. A., Streicher, S. L. Direct selection for P1-sensitive mutants of enteric bacteria. Journal of Bacteriology. 118 (3), 810-814 (1974).
- O'Connor, K. A., Zusman, D. R. Coliphage P1-mediated transduction of cloned DNA from Escherichia coli to Myxococcus xanthus: use for complementation and recombinational analyses. Journal of Bacteriology. 155 (1), 317-329 (1983).
Reprints and Permissions
Request permission to reuse the text or figures of this JoVE article
Request PermissionExplore More Articles
This article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. All rights reserved