このコンテンツを視聴するには、JoVE 購読が必要です。 サインイン又は無料トライアルを申し込む。
Method Article
物品税課税対象の抗生物質耐性カセット P1 伝達による大腸菌の遺伝子の削除を生産
要約
ここで他の大腸菌の欠失変異株を作るための基盤として、既存の抗生物質抵抗カセット削除構造の使用のためのプロトコルを提案する.このような欠失変異体を動員し、P1 ファージ伝達を使用して受信者負担の対応する軌跡に挿入することができます。
要約
細菌の未知遺伝子の機能を勉強する最初のアプローチは、この遺伝子のノックアウトを作成することです。ここでは、バクテリオファージ P1 と一般化伝達を使用して別に 1 つのエシェリヒア属大腸菌の緊張から遺伝子欠失変異体を転送するための堅牢で高速なプロトコルについて述べる。このメソッドは、変異が選択可能な (例えば、抗生物質のカセット挿入を使用して遺伝子破壊に基づく) にする必要があります。このような抗生物質のカセット ドナーひずみから動員できる興味の受信者にすばやく導入し、遺伝子失変異を簡単に生成します。抗生物質のカセットは、ゲノムのシーケンスでのみ 〜 100 ベース ペア長い傷跡きれいなノックアウトを生成するサイト固有のリコンビナーゼによってカセットの切除ができる flippase の認識部位を含むように設計できます。プロトコルを示す細胞の生合成に関与するアセンブリ因子多摩遺伝子をノックアウトし、器官にこのノックアウトの効果と 2 つの三量体細胞アドヘシンの機能をテストします。P1 伝達によって遺伝子の欠失は、その制限があります、使いやすさとその実装の速度は、遺伝子欠失の他の方法に代わる。
概要
遺伝子の機能を検討する共通の最初のアプローチは、ノックアウト変異を実行し、結果の表現型を観察することです。これはまた逆遺伝学と呼ばれます。最後の 70 年間またはそう、その培養と遺伝子操作1こうして容易なため細菌エシェリヒア属大腸菌分子生物学の主力となっています。エシェリヒア属大腸菌、マーカー交換変異2,3と、最近では、λ 赤または Rac ET システム4,5を使用してジーンを含む遺伝子の削除を生成するいくつかの方法が開発されています。,6。
広く使われているシステムでは、個々 の遺伝子のコード配列が染色体5,7から後で摘出することができます抗生物質耐性カセットに置き換えられます。いずれかの側に flippase (FLP) 認識対象 (FRT) サイトが並ぶされているカナマイシン (菅) 抵抗カセットによって例えばコーディング シーケンスが置き換えられます。FRT サイトは菅カセットの削除につながる FRT サイト間の部位特異的組換えを媒介する FLP リコンビナーゼによって認識されます。この方法で、約 100 塩基対 (bp) (図 1) のシーケンスでのみ最小限の傷跡を残して、ある特定の遺伝子のコーディング シーケンスの完全な削除を実現できます。
わずか 10 年前、いわゆる慶應義塾コレクションが開発されました。ほぼすべての非本質的な遺伝子が λ 赤組換え7,8によって削除された個別に標準検査大腸菌K12 株に基づく細菌ライブラリです。このコレクションの各内のクローンは、物品税課税対象の菅抵抗カセットに置き換えられます 1 つのコーディング シーケンスを持っています。慶應コレクションは、多くのアプリケーション9便利なツールを証明しています。そのようなアプリケーションは、他の大腸菌の欠失変異株の生産です。特定削除クローンから菅カセットは、バクテリオファージ P110、11,12,13,14などを一般に伝達によって動員することができます。そのようなストレスから調製したファージ株式使用できます受信者、関心の大腸菌に感染する、低いが信頼性の高い周波数菅でカセットを含む地域に組み込むことが受信者のゲノムの相同組み換えによる(図 2)。菅含有培地で成長形質を選択できます。この後、抗生物質耐性カセットの除去が必要な場合、FLP リコンビナーゼはトランスで transductant 株に指定できます。抵抗、アンピシリン (Amp) のマーカーを運ぶ、FLP を含むプラスミドを硬化後、菅とアンプに敏感なクローンを上映するほか、正しい切除野生型のコーディング シーケンスと菅カセットはコロニー PCR によって確認されます。
ここでは、ノックアウト上記戦略に基づく大腸菌の菌株の生産のステップのそれぞれを記述する詳細なプロトコルが表示されます。例として、多摩遺伝子の削除が示されています。多摩は、アセンブリ モジュール (TAM)、特定の細胞蛋白質と線毛15,16,17の生合成に関与する輸送の一部である外膜 β バレル型タンパク質をエンコードします。このノックアウトのひずみを 2 つの三量体細胞アドヘシン (TAAs)、エルシニア双球菌矢田、エシェリヒア属大腸菌免疫グロブリン (Ig) の器官に多摩の削除の影響を調べる使用して-バインド TAA EibD18,19。
プロトコル
1 系統とプラスミド
-
細菌の緊張
- 大腸菌株 BW251135JW4179 (BW25113 tamA::kan)7、BL21(DE3)20、および BL21ΔABCF21を使用します。詳細については、材料の表を参照してください。
-
バクテリオファージ
- 一般的な伝達にバクテリオファージ P1virを使用します。クロロホルムを数滴の液体在庫としてバクテリオファージを格納 (手順 2.2 参照)。詳細については、材料の表を参照してください。
-
プラスミド
- このプロトコルでは、次のプラスミドを使用: pCP2022、pIBA2 矢田23、および24pEibD10。制御プラスミドとして pASK IBA2 と pET22b (材料の表を参照) を使用します。
-
成長の条件
- ホストゲノム スープ (LB) 中25 37 ° C または BL21ΔABCF と pCP20 を含む系統の場合 30 ° C で活発な動揺 (180-200 rpm) の中の細菌に伝達します。
- 43 ° C で硬化性プラスミドを実行します。
- 固体培地の寒天 1% (w/v) ポンドを補足します。
- 上の寒天の 0.7% 寒天と 10 mM CaCl2とオートクレーブ中で LB を補足します。エレクトロポレーション26後回復のため SOC 培地を使用します。
- 抗生物質のため下記の濃度を使用: アンプと菅の 25 μ g/mL の 100 μ g/mL
2 ファージ Lysate を準備します。
-
ドナー株の感染
- 5 mL の LB 培 10 mM CaCl2とし、必要に応じて菅でドナー株 JW4197 を成長 (25 μ g/mL) 600 の光学濃度に 〜 1.0 nm (外径600)。分光光度計を用いた OD600の値を測定します。
- LB 培地で既存の P1 バクテリオファージ株式の希釈系列を作る: 10-3 10-7~ 間の希釈を推奨します。
- 細菌懸濁液と与えられたファージ希釈 15 mL 遠心管またはそれと同等の 100 μ L のミックス 200 μ L。バクテリオファージ希釈として多くのチューブを用意します。振ることがなく 37 ° C で 20 分間チューブを孵化させなさい。
- チューブに 10 mM CaCl2を添加した溶の上の寒天 (~ 50 ° C) 〜 3 mL を加えてミックス内容に徹底的にボルテックス チューブ、まもなくも層を作る prewarmed LB プレートの上に混合物を注ぐ。
- 37 ° C で一晩版を孵化させなさい
-
ライセート作製
- 次の日は、バクテリオファージのプラクの半合流成長とプレートを選択します。半合流プレートの約半分の表面積プレートは明らか (図 3)。
- 接種ループまたは同様のツールを使用してこのようなプレートから上の寒天層をこすり、遠心管中上の寒天を配置します。ヒューム フードの LB の 1-2 mL とクロロホルムと渦 ~ 1 分追加、クロロホルムは積極的にチューブのドロップを追加します。
- 寒天と菌体ペレットへ 4,000 g以上で 15 分の管を遠心します。
- ペレットからの残骸の上に運ぶ回避する新鮮な遠心チューブに上清を移動します。クロロホルムの 2 滴を追加し、4-10 ° c. でライセートを格納ヒューム フードのクロロホルムを追加します。これは伝染性の粒子の数の大幅な削減になりますので、ライセートすぎるバクテリオファージは凍結しないでください。
-
溶解液の力価を決定します。
- 37 ° C で 10 mM CaCl2文化 〜 1.0 の外径 φ600まで添加した LB の BW25113 に成長します。
- LB (例えば、10-6– 10-9) の lysate のバクテリオファージの希釈系列を調製します。
注: 必ずピペット、サンプルを希釈するクロロホルムの転送を避けるために lysate の上から。 - 細菌懸濁液と与えられたファージ希釈 15 mL 遠心管またはそれと同等の 100 μ L のミックス 200 μ L。バクテリオファージ希釈として多くのチューブを用意します。振ることがなく 37 ° C で 20 分間チューブを孵化させなさい。
- チューブに 10 mM CaCl2を添加した溶の上の寒天 (~ 50 ° C) 〜 3 mL を加えてミックス内容に徹底的にボルテックス チューブ、まもなくも層を作る prewarmed LB プレートの上に混合物を注ぐ。
- 37 ° C で一晩版を孵化させなさい
- 次の日は、各プレートのプラーク (細菌のマットで明確な地域) の数をカウントし、次の数式を使用して、溶解液の力価を計算します。

例: 希釈 10-7, 10-810-9とプレートにあり 231、27、2 プラク、それぞれ。感染症の各希釈 100 μ L を用いてプラーク形成力価が表されるので、単位 (pfu)/mL、めっき因子は 10。これらの数値は、数式に入力されます。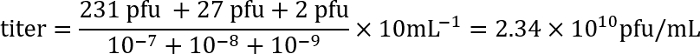
したがって、力価は約 2 × 1010伝染性のバクテリオファージの粒子/mL です。
3. P1 伝達
-
受信者のセルの準備
- 600 の光学濃度に添加した LB の受信者株 BL21ΔABCF を成長 〜 1.0 nm (外径600)。分光光度計を使用して、外径 φ600の値を測定します。
- ライセートのバクテリオファージの感染 (MOI) 値が 0.5 の多様性を達成するために必要なボリュームを計算します。慣性モーメントを計算するには、文化の OD600に基づいて細菌の数を見積もる。外径600値 1.0 が 10 〜9 cfu/mL に対応することを想定してください。ライセートの知られている力価に基づいて必要な容量を計算します。
(2.3.6 の手順の後の例で計算力価に基づく) の例:
- 10 mm 受信者ひずみ文化に CaCl2を加え、混ぜます。文化伝達のための 1 mL を取る。
-
伝達を行う
- 3.1.2 のステップで計算される (CaCl2 10 mm 含む) 受信者の文化の 1 mL に溶解バクテリオファージの適切なボリュームを追加、軽く混ぜます。
注: は、ミックスにクロロホルムを転送しないようにするライセートの上からサンプルをピペットように注意します。 - 37 ° C で 20 分のミックスを静的にインキュベートします。
- 100 mm pH 5.5、クエン酸ナトリウムを追加することによって感染を停止します。
- 細菌 (5,000 × gを 2 分間) を遠心し、上清を除去、100 mM クエン酸ナトリウム、pH 5.5 と補われる新鮮な LB の 1 mL でそれらを再懸濁します。
- 3.2.4 無料ファージとカルシウムの除去を確保するためのステップのようにさらに 2 回細胞を洗浄します。
- 100 mM クエン酸ナトリウム、pH 5.5 と補われる新鮮な LB の 1 mL 中の細菌を再懸濁します。30 ° C (> 100 rpm) の揺れで 1 h で細菌を孵化させなさい。
- 遠心分離 (5,000 × gを 2 分間) によって細菌を収集し、100 mM クエン酸ナトリウム、pH 5.5 の LB の ~ 100 μ L でそれらを再懸濁します。
- 25 μ g/mL と 10 mM クエン酸ナトリウム、pH 5.5、菅と補われる LB プレートに細菌を広げるし、植民地 (~ 24 h) が表示されるまでに、30 ° C でバクテリアします。
- 3.1.2 のステップで計算される (CaCl2 10 mm 含む) 受信者の文化の 1 mL に溶解バクテリオファージの適切なボリュームを追加、軽く混ぜます。
-
形質を選択します。
- 植民地選択板で育った、単一コロニーのためポンド + 菅にそれらを restreak、シングル コロニーが表示されるまで、30 ° C で成長します。
4. 常套菅カセット
-
組換えのプラスミッドの変形
- 菅耐性 BL21ΔABCF ひずみ electrocompetent を確認します。
- 菅首相は補われる新鮮な LB (5 mL) の単一コロニーから株を成長 (25 μ g/mL) 30 ° c まで OD600 ~0.5 – 0.7。
- ここから、4 ° c または氷の上、次の手順を実行します。細菌 (5,000 × gで 10 分間) を遠心し、上清を除去します。
- 2 回前の遠心分離のステップを繰り返すことによって冷たい蒸留水細胞ペレットを洗浄し、最後に、氷冷 10% グリセロールの 100 μ L で細胞ペレットを再懸濁します。
- 氷の上 1 mm エレクトロポレーション キュベットを冷やします。
- 細胞懸濁液にプラスミド (pCP20) の 1 pg を追加、穏やか、懸濁液に混合し、冷却エレクトロポレーション キュベットに転送。
- 本体を 1.8 に設定 kV および electroporate 細胞。
- SOC 培地 1 mL を追加し、30 ° C で 1 時間のそれらの成長によって変換されたセルを救出します。
- ポンド上のセルの 100 μ L をプレート + アンプ板し一晩 30 ° C で細胞を成長します。
- 菅耐性 BL21ΔABCF ひずみ electrocompetent を確認します。
-
再結合の誘導
- LB + アンプからシングル コロニーをピックアップ プレートし、すべての抗生物質を省略する新鮮な LB を接種します。
- FLP リコンビナーゼの発現を誘導するために一晩の非寛容な温度 (43 ° C) でセルを育てなさい。
-
Recombinants を選択します。
- シリアルの希薄を作ると非選択式ナンバー プレートに 105-106希釈の 50 μ L をプレートし、一晩 30 ° c. でそれを育てる
5. 遺伝子欠失の検証
-
成功した組換えプラスミドの損失の検証
- ストリーク プレート ステップ 4.3.1 LB + 菅、LB + アンプで準備からこの順序で、抗生物質がない LB プレート シングル コロニー。ストリー キングの支援するためコロニー グリッドを使用 (補足のファイル 1を参照してください)。植民地 (~ 24 h) が表示されるまで、30 ° C でプレートを成長します。
- 非選択的プレート上に成長したが、さらに検証のための選択培地で成長に失敗しました 10-20 クローンを選択します。
-
コロニー PCR による追加検証
- 並ぶ多摩コーディング配列のプライマーとコロニー PCR を実行 (材料の表を参照してください)。
- PCR のマスターの組合せを準備します。必要なミックスは、(表 1の例を参照してください) テストするコロニーの数によって異なります。氷の試薬を混ぜて、最後ポリメラーゼを追加します。
- PCR マスター ミックスを混和し、PCR ストリップのチューブに 20 μ L を分注します。少量の滅菌ピペット チップを使用して上映される各コロニーをピックアップし、それをチューブに追加します。比較のため、元の受信者ひずみを含めるようにしてください。必要に応じて、ドナーの負担があります。
- PCR の反作用を実行 (使用プログラムの表 2を参照)。
- 1% の agarose のゲルを準備します。
- 50 mL ゲルの agarose の 0.5 g を測定し、50 mL の TAE バッファー (40 mM トリス、20 mM 酢酸と 1 ミリメートルの EDTA、pH 8.0) を追加します。すべて agarose が溶けるまで電子レンジでの混合物を加熱します。
- Agarose が解散後約 50 ° C にソリューションを冷却し、DNA 染色色素の 5 μ L を追加します。ソリューションをよく混合し、鋳造の部屋でゲル トレイにそれを注ぐ。すべてのサンプルを分子量マーカーの十分な井戸があります、よく櫛の 1 つまたは複数の行を挿入します。30 分に設定するゲルを許可します。
- PCR の実行が完了すると、各サンプルに 4 μ L の DNA 読み込み染料 × 6 を追加します。ゲルの電気泳動室にし、井戸が覆われているまでに TAE バッファーを追加します。
- 10-15 μ L をゲルの井戸に各 PCR の反作用から適用されます。分子量マーカーの 5 μ L を追加もできます。その後、75 V で 30 分のためのゲルを実行します。
- 実行が完了すると、青の光の撮像素子を使用してゲルをイメージします。
6. その他のテクニック
-
蛋白質の表現
注: テスト蛋白質 (矢田・ EibD) の表情をしています23,24詳細他の場所で説明されています。これは、主な手順の概要です。- BL21ΔABCF Δ多摩 wi 番目に必要なプラスミド (pIBA2 矢田と pEibD10 と対応する制御プラスミド) を変換し、LB + アンプの形質転換体の選択。
- 蛋白質の表現のため一晩培養変形中の 1 mL の LB 培地 + アンプの 100 mL を接種してその時点での中間ログ段階まで 30 ° C で栽培、(100 ng/mL) でいずれかの anhydrotetracycline を持つ蛋白質の生産を誘発するか(0.5 mM) でイソプロピル thiogalactoside。
- 30 ° C で誘導の 2 h 文化の濁度を測定し、OD600で 50 mL に対応するセルの数を収集した後は、4,000 x gで 15 分間文化を遠心分離によって 1.0 を = します。餌を洗浄 1 x 10 mm HEPES、pH 7.4 では、し、いずれかは-20 ° C またはプロセス ステップ 6.2 のようにさらにそれでそれを保存します。
-
外膜抽出
メモ: 外膜抽出は実行されますレオらによって詳細に説明されているよう27. 主な手順を以下に記載します。- 10 mm HEPES、pH 7.4 では、1 mL の細胞ペレットを再懸濁します補足 10 mM MgCl2と MnCl2リゾチーム (0.1 mg/mL)、DNase のピンチと私。
- (例えばビード ・ ビーターを使用して) 細胞を溶解させます。
- 細胞の残骸を削除し、新鮮なチューブに上清を移動直後のセル (15,600 x gで 2 分) を遠心します。
- 16,000 x gで 30 分間細胞を遠心分離後、1 %n ラウロイル サルコシン 10 mM HEPES、pH 7.4 の 400 μ L でペレットを再懸濁します。
- その後、室温で 30 分間撹拌で細胞をインキュベート、上記として 30 分のそれらを遠心分離機します。
- 洗って、半透明ペレットを 10 mM HEPES、pH 7.4 では、200 μ l 1 x 10 mM HEPES の 30 μ L でそれを再懸濁します、4 x SDS-PAGE サンプルバッファーの 10 μ L を追加。
-
活性測定法
注: は、として記述されている28矢田と EibD の SDS-PAGE と活性アッセイを実行します。主な手順は以下のとおり。 -
SDS ページ
- 5 分の 50 ° c のサンプルを加熱すると、タンパク質の変性を避けるために、ポリアクリルアミドのゲルにロードする前に。
- SDS ページの分離、ポリフッ化ビニリデン (PVDF) 二フッ化膜に蛋白質を転送します。
- 転送後、PBS で 2% 脱脂ミルク パウダーと膜をブロックします。
-
矢田コラーゲン遠い西部のしみ方
- ブロック後、10 μ g/mL の濃度にブロック バッファーで希釈私牛由来のコラーゲンの種類を追加、1 h の膜を孵化させなさい。
- 2 膜を洗浄する PBS + 0.05% Tween20 x (PBS-T)。
- ブロック バッファー内膜に一次抗体 (モノクローナル抗コラーゲン COL 1)、希薄化後の 1:2,000 を追加します。
- 1 h の膜をインキュベート後ステップ 6.3.2.2 で述べたように 2 x を洗浄し、ブロック バッファーで希釈 1: 10,000、二次抗体 [ヤギ抗マウス IgG 西洋わさびペルオキシダーゼ (HRP) 共役] を追加します。
- 1 h の膜をインキュベートして洗って PBS T で 2 倍製造元の指示に従って膜強化化学発光基質を追加し、CCD カメラを使用してバンドを検出します。
-
EibD IgG 結合の試金
- 後に、ブロック (ヤギ抗うさぎ HRP)、二次抗体希釈バッファーの妨害に 1:2,000 を追加します。
- 1 h の膜をインキュベートして洗って PBS で 2 倍。前述のステップ 6.3.2.5 化学ルミネセンスの検出を実行します。
結果
BL21ΔABCF のノックアウトから多摩の生成:
上記の戦略は、BL21(DE3) で、外膜タンパク質の生産用に最適化され、BL21ΔABCF21と呼ばれるタンパク質の生産に使用される標準的な実験室ひずみのデリバティブ歪みを生成する以前使用されています。この株は、豊富な外膜蛋白質のために符号化する 4 つの遺伝?...
ディスカッション
P1 伝達は、エシェリヒア属大腸菌の遺伝子の削除を生成するための高速、堅牢で、信頼性の高い方法です。BL21 由来の受信者に慶應義塾ドナーひずみから多摩失変異を伝達でここでこれを示します。伝達プロセスの主要な段階は、ライセートの生産、伝達自体、菅抵抗カセットの切除および PCR によるノック アウトの検証です。合計では、プロセスは約 1 週間ほどかかります、?...
開示事項
著者が明らかに何もありません。
謝辞
慶應コレクション系統は、ナショナルバイオ リソース プロジェクト (遺伝研, 日本) から得られた:エシェリヒア属大腸菌。彼の継続サポートありがとうダーク リンケ (バイオ サイエンス学科、オスロ大学)。この作品は、ノルウェー若手研究者グラント 249793 (ジャック C. レオ) に研究評議会によって賄われていた。
資料
| Name | Company | Catalog Number | Comments |
| Strains | |||
| E. coli BW25113 | NIG | ME6092 | Wild-type strain of Keio collection |
| E. coli BL21(DE3) | Merck | 69450-3 | Expression strain |
| E. coli BL21DABCF | Addgene | 102270 | Derived from BL21(DE3) |
| E. coli JW4179 | NIG | JW4179-KC | tamA deletion mutant |
| P1 vir | NIG | HR16 | Generally transducing bacteriophage |
| Plasmids | |||
| pCP20 | CGSC | 14177 | conditionally replicating plasmid with FLP |
| pASK-IBA2 | IBA GmbH | 2-1301-000 | expression vector |
| pEibD10 | N/A | N/A | for production of EibD; plasmid available on request |
| pET22b+ | Merck | 69744-3 | expression vector |
| pIBA2-YadA | N/A | N/A | for production of YadA; plasmid available on request |
| Chemicals | |||
| Acetic acid | ThermoFisher | 33209 | |
| Agar | BD Bacto | 214010 | |
| Agarose | Lonza | 50004 | |
| Ampicillin | Applichem | A0839 | |
| Anhydrotetracycline | Abcam | ab145350 | |
| anti-collagen type I antibody COL-1 | Sigma | C2456 | |
| Bovine collagen type I | Sigma | C9791 | |
| Calcium chloride | Merck | 102382 | |
| Chloroform | Merck | 102445 | |
| Di-sodium hydrogen phosphate | VWR | 28029 | |
| DNA dye | Thermo | S33102 | |
| DNA molecular size marker | New England BioLabs | N3232S | |
| DNase I | Sigma | DN25 | |
| dNTP mix | New England Biolabs | N0447 | |
| ECL HRP substrate | Advansta | K-12045 | |
| EDTA | Applichem | A2937 | |
| Glycerol | VWR | 24388 | |
| goat anti-mouse IgG-HRP | Santa Cruz | sc-2005 | |
| goat anti-rabbit IgG-HRP | Agrisera | AS10668 | |
| HEPES | VWR | 30487 | |
| Isopropyl thiogalactoside | VWR | 43714 | |
| Kanamycin | Applichem | A1493 | |
| Lysozyme | Applichem | A4972 | |
| Magnesium chloride | VWR | 25108 | |
| Manganese chloride | Sigma | 221279 | |
| N-lauroyl sarcosine | Sigma | L9150 | |
| Skim milk powder | Sigma | 70166 | |
| Sodium chloride | VWR | 27808 | |
| tamA forward primer | Invitrogen | N/A | Sequence 5'-GAAAAAAGG ATATTCAGGAGAAAATGTG-3' |
| tamA reverse primer | Invitrogen | N/A | Sequence 5'-TCATAATT CTGGCCCCAGACC-3' |
| Taq DNA polymerase | New England Biolabs | M0267 | |
| Tri-sodium citrate | Merck | 106448 | |
| Tryptone | VWR | 84610 | |
| Tween20 | Sigma | P1379 | |
| Yeast extract | Merck | 103753 | |
| Equipment | |||
| Agarose gel electrophoresis chamber | Hoefer | SUB13 | |
| Bead beater | Thermo | FP120A-115 | |
| CCD camera | Kodak | 4000R | |
| Electroporation cuvettes | Bio-Rad | 165-2089 | |
| Electroporation unit | Bio-Rad | 1652100 | |
| Gel imager | Nippon Genetics | GP-03LED | |
| Incubating shaker | Infors HT | Minitron | |
| Incubator | VWR | 390-0482 | |
| Microcentrifuge | Eppendorf | 5415D | |
| Microwave oven | Samsung | CM1099A | |
| PCR machine | Biometra | Tpersonal | |
| PCR strips | Axygen | PCR-0208-CP-C | |
| pH meter | Hanna Instruments | HI2211-01 | |
| PVDF membrane | ThermoFisher | 88518 | |
| SDS-PAG electrophoresis chamber | ThermoFisher | A25977 | |
| Tabletop centrifuge | Beckman Coulter | B06322 | |
| Vortex mixer | Scientific Industries | SI-0236 | |
| Water bath | GFL | D3006 | |
| Wet transfer unit | Hoefer | TE22 |
参考文献
- Blount, Z. D. The unexhausted potential of E. coli. eLife. 4, e05826 (2015).
- Hamilton, C. M., Aldea, M., Washburn, B. K., Babitzke, P., Kushner, S. R. New method for generating deletions and gene replacements in Escherichia coli. Journal of Bacteriology. 171 (9), 4617-4622 (1989).
- Link, A. J., Phillips, D., Church, G. M. Methods for generating precise deletions and insertions in the genome of wild-type Escherichia coli: application to open reading frame characterization. Journal of Bacteriology. 179 (20), 6228-6237 (1997).
- Sawitzke, J. A., Thomason, L. C., Costantino, N., Bubunenko, M., Datta, S., Court, D. L. Recombineering: in vivo genetic engineering in E. coli, S. enterica, and beyond. Methods in Enzymology. 421, 171-199 (2007).
- Datsenko, K. A., Wanner, B. L. One-step inactivation of chromosomal genes in Escherichia coli K-12 using PCR products. Proceedings of the National Academy of Sciences of the United States of America. 97 (12), 6640-6645 (2000).
- Muyrers, J. P., Zhang, Y., Stewart, A. F. Techniques: Recombinogenic engineering--new options for cloning and manipulating DNA. Trends in Biochemical Sciences. 26 (5), 325-331 (2001).
- Baba, T., et al. Construction of Escherichia coli K-12 in-frame, single-gene knockout mutants: the Keio collection. Molecular Systems Biology. 2, (2006).
- Yamamoto, N., et al. Update on the Keio collection of Escherichia coli single-gene deletion mutants. Molecular Systems Biology. 5, 335 (2009).
- Baba, T., Huan, H. -. C., Datsenko, K., Wanner, B. L., Mori, H. The applications of systematic in-frame, single-gene knockout mutant collection of Escherichia coli K-12. Methods in Molecular Biology. 416, 183-194 (2008).
- Chattopadhyay, M. K., Tabor, C. W., Tabor, H. Polyamines are not required for aerobic growth of Escherichia coli: preparation of a strain with deletions in all of the genes for polyamine biosynthesis. Journal of Bacteriology. 191 (17), 5549-5552 (2009).
- Xie, X., Wong, W. W., Tang, Y. Improving simvastatin bioconversion in Escherichia coli by deletion of bioH. Metabolic Engineering. 9 (4), 379-386 (2007).
- Maeda, T., Sanchez-Torres, V., Wood, T. K. Escherichia coli hydrogenase 3 is a reversible enzyme possessing hydrogen uptake and synthesis activities. Applied Microbiology and Biotechnology. 76 (5), 1035-1042 (2007).
- Meehan, B. M., Landeta, C., Boyd, D., Beckwith, J. The disulfide bond formation pathway is essential for anaerobic growth of Escherichia coli. Journal of Bacteriology. 199 (16), (2017).
- Niba, E. T. E., Naka, Y., Nagase, M., Mori, H., Kitakawa, M. A genome-wide approach to identify the genes involved in biofilm formation in E. coli. DNA Research. 14 (6), 237-246 (2008).
- Heinz, E., et al. Conserved features in the structure, mechanism, and biogenesis of the inverse autotransporter protein family. Genome Biology and Evolution. 8 (6), 1690-1705 (2016).
- Selkrig, J., et al. Discovery of an archetypal protein transport system in bacterial outer membranes. Nature Structural & Molecular Biology. 19 (5), 506-510 (2012).
- Stubenrauch, C., et al. Effective assembly of fimbriae in Escherichia coli depends on the translocation assembly module nanomachine. Nature Microbiology. 1 (7), 16064 (2016).
- Leo, J. C., Goldman, A. The immunoglobulin-binding Eib proteins from Escherichia coli are receptors for IgG Fc. Molecular Immunology. 46 (8-9), 1860-1866 (2009).
- Mühlenkamp, M., Oberhettinger, P., Leo, J. C., Linke, D., Schütz, M. S. Yersinia adhesin A (YadA) - Beauty & beast. International Journal of Medical Microbiology. 305 (2), 252-258 (2015).
- Studier, F. W., Moffatt, B. A. Use of bacteriophage T7 RNA polymerase to direct selective high-level expression of cloned genes. Journal of Molecular Biology. 189 (1), 113-130 (1986).
- Meuskens, I., Michalik, M., Chauhan, N., Linke, D., Leo, J. C. A new strain collection for improved expression of outer membrane proteins. Frontiers in Cellular and Infection Microbiology. 7, 464 (2017).
- Cherepanov, P. P., Wackernagel, W. Gene disruption in Escherichia coli: TcR and KmR cassettes with the option of Flp-catalyzed excision of the antibiotic-resistance determinant. Gene. 158 (1), 9-14 (1995).
- Grosskinsky, U., et al. A conserved glycine residue of trimeric autotransporter domains plays a key role in Yersinia adhesin A autotransport. Journal of Bacteriology. 189 (24), 9011-9019 (2007).
- Leo, J. C., et al. The structure of E. coli IgG-binding protein D suggests a general model for bending and binding in trimeric autotransporter adhesins. Structure. 19 (7), 1021-1030 (1993).
- Bertani, G. Studies on lysogenesis. I. The mode of phage liberation by lysogenic Escherichia coli. Journal of Bacteriology. 62 (3), 293-300 (1951).
- Hanahan, D. Studies on transformation of Escherichia coli with plasmids. Journal of Molecular Biology. 166 (4), 557-580 (1983).
- Leo, J. C., Oberhettinger, P., Linke, D. Assessing the outer membrane insertion and folding of multimeric transmembrane β-barrel proteins. Methods in Molecular Biology. , 157-167 (2015).
- Mikula, K. M., Leo, J. C., Łyskowski, A., Kedracka-Krok, S., Pirog, A., Goldman, A. The translocation domain in trimeric autotransporter adhesins is necessary and sufficient for trimerization and autotransportation. Journal of Bacteriology. 194 (4), 827-838 (2012).
- Thomason, L. C., Costantino, N., Court, D. L., Ausubel, F. M. E. coli genome manipulation by P1 transduction. Current Protocols in Molecular Biology. , (2007).
- Franklin, N. C. Mutation in gal U gene of E. coli blocks phage P1 infection. Virology. 38 (1), 189-191 (1969).
- Jeong, H., et al. Genome sequences of Escherichia coli B strains REL606 and BL21(DE3). Journal of Molecular Biology. 394 (4), 644-652 (2009).
- Greene, E. C. DNA Sequence Alignment during Homologous Recombination. Journal of Biological Chemistry. 291 (22), 11572-11580 (2016).
- Watt, V. M., Ingles, C. J., Urdea, M. S., Rutter, W. J. Homology requirements for recombination in Escherichia coli. Proceedings of the National Academy of Sciences of the United States of America. 82 (14), 4768-4772 (1985).
- Ho, T. D., Waldor, M. K. Enterohemorrhagic Escherichia coli O157:H7 gal mutants are sensitive to bacteriophage P1 and defective in intestinal colonization. Infection and Immunity. 75 (4), 1661-1666 (2006).
- Ikeda, H., Tomizawa, J. I. Transducing fragments in generalized transduction by phage P1. I. Molecular origin of the fragments. Journal of Molecular Biology. 14 (1), 85-109 (1965).
- Murooka, Y., Harada, T. Expansion of the host range of coliphage P1 and gene transfer from enteric bacteria to other Gam-negative bacteria. Applied and Environmental Microbiology. 38 (4), 754-757 (1979).
- Goldberg, R. B., Bender, R. A., Streicher, S. L. Direct selection for P1-sensitive mutants of enteric bacteria. Journal of Bacteriology. 118 (3), 810-814 (1974).
- O'Connor, K. A., Zusman, D. R. Coliphage P1-mediated transduction of cloned DNA from Escherichia coli to Myxococcus xanthus: use for complementation and recombinational analyses. Journal of Bacteriology. 155 (1), 317-329 (1983).
転載および許可
このJoVE論文のテキスト又は図を再利用するための許可を申請します
許可を申請さらに記事を探す
This article has been published
Video Coming Soon
Copyright © 2023 MyJoVE Corporation. All rights reserved