Для просмотра этого контента требуется подписка на Jove Войдите в систему или начните бесплатную пробную версию.
Method Article
Производство гена удалений в Escherichia coli , P1 трансдукция с подакцизных антибиотикорезистентности кассеты
В этой статье
Резюме
Здесь мы представляем собой протокол для использования уже существующих антибиотикам сопротивление кассета удаления конструкции как основу для принятия удаления мутантов в других штаммов E. coli . Такие перегласовки удаления могут быть мобилизованы и вставлен в соответствующий Локус штамм получателей с помощью P1 бактериофага трансдукции.
Аннотация
Первый подход для изучения функции неизвестных генов бактерий является создание плей офф этого гена. Здесь мы описываем надежный и быстрый протокол для передачи мутации гена удаления из одного штамма кишечной палочки в другой с помощью обобщенного трансдукции бактериофага P1. Этот метод требует, чтобы мутация выбора (например, на основе гена сбоев с помощью антибиотиков кассеты вставок). Таких антибиотиков кассеты могут быть мобилизованы из штамма доноров и введена в получателей штамм интерес к быстро и легко создавать ген мутанта удаления. Антибиотик кассеты могут быть разработаны включить Флиппазы признание сайты, которые позволяют иссечение кассеты по участкам рекомбиназа производить чистую нокаут с только ~ 100-base пара длинный шрам последовательности генома. Мы продемонстрировать протокола путем стучать вне Джин Тама , кодирования фактор Ассамблеи, занимающихся биогенеза autotransporter и проверить эффект от этого плей офф на биогенеза и функции двух адгезины trimeric autotransporter. Хотя гена удаления трансдукции P1 имеет свои ограничения, легкость и скорость ее реализации сделать его привлекательной альтернативой для других методов удаления ген.
Введение
Первый общий подход для изучения функции гена — для выполнения нокаут-мутагенеза и наблюдать результате фенотип. Это также называется обратной генетики. Бактерия E. coli была рабочая лошадка молекулярной биологии за последние 70 лет или так, благодаря простоте его культивирования и его ответственностью генетические манипуляции1. Несколько методов были разработаны производить Джин удалений в E. coli, включая маркер обмен мутагенеза2,3 и, совсем недавно, recombineering, используя λ красный или Rac ET систем4,5 , 6.
В широко используется система кодирования последовательностей отдельных генов заменяются антибиотикорезистентности кассету, которая позже может быть подакцизным хромосома5,7. Кодирования последовательности заменяются, например кассета сопротивления канамицин (Кан), который окружен Флиппазы (FLP) признание целевой (ФРТ) сайты с обеих сторон. FRT сайты признаны рекомбиназа ФЛП, которая опосредует участкам рекомбинация между сайтами ФРТ, ведущих к удаления кассеты Кан. Таким образом может быть достигнуто полное удаление данного гена кодирующая последовательность, оставив только минимальный шрам последовательность приблизительно 100 пар оснований (ВР) (рис. 1).
Чуть более десяти лет назад так называемый Keio коллекция была разработана. Это бактериальный Библиотека, на базе стандартного лаборатории штамм E. coli K12, где почти все несущественные гены индивидуально были удалены λ красный рекомбинации7,8. Клоны в этой коллекции имеют один кодирующая последовательность, заменены подакцизных кассеты сопротивления Кан. Коллекции Keio оказалась полезным инструментом для многих приложений9. Одним из таких приложений является производство удаления мутантов в других штаммов E. coli . Кан кассету с клон данного исключения могут быть мобилизованы путем обычно преобразователя бактериофагов, таких как P110,11,12,,1314. Фаговые складе, подготовленных от такого напряжения может затем использоваться для заразить получателя штамм E. coli интерес, где с частотой низкий, но надежный Кан кассеты содержащих региона могут быть включены в геноме получателя, гомологичная рекомбинация (Рисунок 2). Transductants могут быть выбраны для роста на Кан содержащих носителе. После этого если удаление антибиотикорезистентности кассеты, ФЛП рекомбиназа могут поставляться штамм transductant в транс. После отверждения ФЛП содержащих плазмида, который носит маркер сопротивления ампициллин (Amp), Кан и Amp чувствительных клоны проверяются для, и правильный иссечение одичал тип кодирования последовательности и кассеты с Кан проверяются колонии PCR.
Здесь представлен подробный протокол, описывающих каждый из этапов в производстве нокаут-штамм E. coli , основанный на изложенной выше стратегии. В качестве примера показано удаление Джин Тама . Тама кодирует белок β-баррель внешней мембраны, который является частью транспорта и сборки модуля (TAM), который участвует в биогенеза некоторых белков autotransporter и пили15,16,17. Этот штамм нокаут-затем был использован для изучения эффекта удаления Тама биогенеза два trimeric autotransporter адгезинов (ТААС), Yersinia адгезина яда и иммуноглобулина E. coli (Ig)-привязка ТАА EibD 18,19.
протокол
1. штаммы и плазмиды
-
Штаммы бактерий
- Используйте E. coli штаммов BW251135, JW4179 (BW25113 tamA::kan)7, BL21(DE3)20и21BL21ΔABCF. Посмотреть Таблицу материалы для получения дополнительной информации.
-
Бактериофаги
- Использование фага P1Вир для общего трансдукции. Хранить Фаговые как жидкий запас с нескольких капель хлороформа (см. шаг 2.2). Для получения дополнительной информации см. Таблицу материалов.
-
Плазмиды
- Используйте следующие плазмид в настоящем протоколе: pCP2022, pIBA2-YadA23и24pEibD10. Как управления плазмид используйте pASK-IBA2 и pET22b (см. Таблицу материалы).
-
Условия роста
- Распространение бактерий в lysogeny бульон (LB) средний25 с энергичной встряхивания (180 – 200 об/мин) при 37 ° C или 30 ° C в случае BL21ΔABCF и штаммов, содержащие pCP20.
- Выполните плазмида отверждения на 43 ° C.
- Для средне-твердых дополнение LB с 1% агар (w/v).
- Для топ агар дополнение LB с 0,7% агар и 10 мм CaCl2 и автоклав среды. Используйте средство SOC для восстановления после электропорации26.
- Используйте следующие концентрации антибиотиков: 100 мкг/мл для Amp и 25 мкг/мл для Kan.
2. Подготовка Фаговые Lysate
-
Инфекция штамма доноров
- Расти штамм доноров JW4197 в 5 мл среды LB, дополнены с 10 мм CaCl2 и, при необходимости, с Кан (25 мкг/мл) для оптической плотности на 600 Нм (600OD) ~ 1.0. Измерьте значение600 ОД, с помощью спектрофотометра.
- Сделать серию разрежения имеющегося Фаговые P1 в среде LB: Рекомендуемые разведений между 10-3 до 10-7.
- Микс 200 мкл бактериальных подвеска и 100 мкл данного Фаговые разрежения в 15 мл пластиковых пробирок или эквивалент. Подготовьте столько трубы как Фаговые разведениях. Инкубируйте трубы для 20 минут при 37 ° C без встряхивания.
- Добавить ~ 3 мл расплавленного Топ агар (~ 50 ° C) с 10 мм CaCl2 к трубкам, перемешать содержимое путем vortexing трубы вскоре и залить смеси на подогретую фунтов пластин сделать даже слои.
- Инкубировать пластины на ночь при 37 ° C.
-
Лизатных подготовка
- Следующий день выберите тарелку с полу вырожденная роста Фаговые бляшки. На полу вырожденная тарелку, ясно примерно половину площади поверхности пластины (рис. 3).
- Скрип Топ агар слой от пластины, с помощью цикла прививок или аналогичный инструмент и место Топ агар в пластиковых пробирок. Добавить 1-2 мл фунтов и капля хлороформе и вихревой трубки энергично для ~ 1 мин добавить хлороформ в зонта.
- Центрифуга трубки для 15 мин 4000 x g или быстрее в гранулах в агар и бактериальной клетки.
- Переместите супернатант свежие microcentrifuge трубки, избегая над любой мусор из гранул. Добавьте 2 капли хлороформе и хранить lysate 4 – 10 ° c. Добавление хлороформ в зонта. Не заморозить Фаговые lysate, поскольку это приведет к значительному сокращению числа инфекционных частиц.
-
Определение lysate титр
- Растут BW25113 в LB, дополненные 10 мм CaCl2 при 37 ° C до культуры достигает ОД600 ~ 1.0.
- Подготовьте ряд разбавление Фаговые lysate в фунтов (например, 10-6– 10-9).
Примечание: Будьте осторожны накапайте образца от верхней части lysate во избежание передачи хлороформ в разведениях. - Микс 200 мкл бактериальных подвеска и 100 мкл данного Фаговые разрежения в 15 мл пластиковых пробирок или эквивалент. Подготовьте столько трубы как Фаговые разведениях. Инкубируйте трубы для 20 минут при 37 ° C без встряхивания.
- Добавить ~ 3 мл расплавленного Топ агар (~ 50 ° C) с 10 мм CaCl2 к трубкам, перемешать содержимое путем vortexing трубы вскоре и вылить смесь на подогретую LB пластины, чтобы сделать даже слои.
- Инкубировать пластины на ночь при 37 ° C.
- На следующий день подсчитать количество бляшек (ясно регионов в бактериальных мат) для каждой пластины и рассчитать титр lysate с использованием следующей формулы:

Пример: На плитах с разведениях 10-7, 10-8и 10-9, есть 231, 27 и 2 таблички, соответственно. 100 мкл каждого разведения был использован для инфекции, и поскольку титр выражается как бляшкообразующих единицах (ОРП) / мл, коэффициент покрытия составляет 10. Эти числа вводятся в формулу: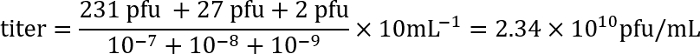
Таким образом титр составляет приблизительно 2 x 1010 инфекционных Фаговые частицы/мл.
3. P1 трансдукция
-
Подготовка получателей клетки
- Расти получателей штамм BL21ΔABCF в дополнение к оптической плотности в 600 фунтов Нм (600OD) ~ 1.0. Используйте спектрофотометр для измерения значение600 ОД.
- Рассчитайте объем Фаговые lysate необходимо достичь разносторонность инфекции (МВД) значение 0.5. Чтобы вычислить МВД, оцените количество бактерий, основанный на од600 культуры. Предположим, что значение600 ОД 1.0 соответствует9 ~ 10 кое/мл. Расчет необходимого объема на основе известных титр lysate.
Пример (основанный на титр, рассчитанные в примере после шага 2.3.6):
- Добавить2 CaCl получателя штамм культуры до 10 мм и перемешать. Возьмите 1 мл культуры для трансдукции.
-
Выполнение трансдукция
- Добавьте соответствующий объем Фаговые lysate 1 мл получателей культуры (включая CaCl2 10 мм), рассчитанные на шаге 3.1.2 и осторожно перемешать.
Примечание: Будьте внимательны Пипетка образец из верхней части lysate чтобы избежать переноса хлороформ в смеси. - Статически Инкубируйте смесь для 20 минут при 37 ° C.
- Остановите инфекцию, добавив цитрат натрия, рН 5,5, до 100 мм.
- Центрифуга бактерий (5000 x g на 2 мин) и удалить супернатант затем Ресуспензируйте их в 1 мл свежего LB дополнена натрия цитрат 100 мм, pH 5.5.
- Мыть дважды больше как шаг 3.2.4 для обеспечения удаления бесплатно бактериофаги и кальция клетки.
- Ресуспензируйте бактерий в 1 мл свежего LB дополнена натрия цитрат 100 мм, pH 5.5. Инкубируйте бактерий на 30 ° C в течение 1 ч при встряхивании (> 100 об/мин).
- Собирать бактерии центрифугированием (5000 x g на 2 мин) и Ресуспензируйте их в ~ 100 мкл фунтов с натрия цитрат 100 мм, pH 5.5.
- Распространение бактерий на плите LB дополнена Кан на 25 мкг/мл и 10 мм цитрат натрия, рН 5,5 и расти бактерий при 30 ° C до колонии появляются (~ 24 h).
- Добавьте соответствующий объем Фаговые lysate 1 мл получателей культуры (включая CaCl2 10 мм), рассчитанные на шаге 3.1.2 и осторожно перемешать.
-
Выбор transductants
- После колонии выросли на пластину отбора, restreak их на LB + Кан для одного колоний и растут при температуре 30 ° C до одной колонии появляются.
4. Вырезание Кан кассеты
-
Преобразование с рекомбинацией плазмиды
- Сделайте electrocompetent штамм Кан устойчивостью BL21ΔABCF.
- Расти штамм от одной колонии в свежих фунтов (5 мл), дополнены Кан (25 мкг/мл) при 30 ° C до тех пор, пока ОД600 ~0.5 – 0,7.
- С этого момента выполняют следующие шаги при температуре 4 ° C или на льду. Центрифуга для бактерий (5000 x g 10 мин) и удалить супернатант.
- Помыть лепешка ячейки с ледяной дистиллированной водой дважды, повторив предыдущий шаг центрифугирования и, наконец, Ресуспензируйте Пелле клеток в 100 мкл ледяной 10% глицерина.
- Охладиться 1 мм электропорации кюветы на льду.
- Добавить 1 pg плазмидной ДНК (pCP20) в суспензии клеток, осторожно перемешать подвеска и передать его охлаждением электропорации кювет.
- Установите electroporator 1,8 кв и electroporate клетки.
- Спасти трансформированных клеток, добавив 1 мл среды SOC и выращивать их в течение 1 ч при температуре 30 ° C.
- Тарелка µL 100 клеток на LB + Amp плиты и расти клеток при 30 ° C на ночь.
- Сделайте electrocompetent штамм Кан устойчивостью BL21ΔABCF.
-
Индукция рекомбинации
- Выбрать один колоний от LB + Amp пластины и прививать свежие LB, минуя все антибиотики.
- Растут на ночь, чтобы побудить выражение ФЛП рекомбиназа клетки на не разрешительной температуры (43 ° C).
-
Выбор рекомбинантов
- Сделать серийных разведений и пластины 50 мкл 105-106 разрежения на неселективной пластины и вырастить его на ночь на 30 ° C.
5. Проверка удаления гена
-
Проверка потери плазмиды и успешных рекомбинации
- Полоска одной колонии от плиты, подготовленную на этапе 4.3.1 на LB + Кан, LB + Amp и ФУНТ пластин без антибиотиков, в этом порядке. Для оказания помощи в полос, используйте сетку колонии (см. дополнительный файл 1). Растут плиты при 30 ° C до колонии появляются (~ 24 h).
- Выберите 10 – 20 клоны, которые выросли на неселективной пластины, но не растут на выборочной СМИ для дальнейшей проверки.
-
Дополнительная проверка колонии PCR
- Выполните колонии PCR с праймерами, окаймляющие Тама кодирования последовательности (см. Таблицу материалы).
- Подготовка мастер смесь ПЦР. Количество смеси необходимо зависит количество колоний для проверки (см. пример в таблице 1). Смешать реактивы на льду, добавить полимеразы последнего.
- Тщательно перемешать смесь ПЦР мастер, а затем отказаться от 20 мкл в пробирки ПЦР полосы. Выберите небольшое количество каждой колонии, чтобы проверяться с помощью стерильной пипеткой кончика и добавить его в трубку. Не забудьте включить исходный штамм получателей для сравнения. При необходимости также включают штамм доноров.
- Запуск реакции PCR (см. таблицу 2 для программы, используемой).
- Подготовка геля агарозы 1%.
- Для 50 мл геля измерения 0,5 г агарозы и добавьте 50 мл буфер TAE (40 мм трис, уксусная кислота 20 мм и 1 мм ЭДТА, рН 8,0). Нагрейте смесь в микроволновой печи до тех пор, пока все агарозы распущен.
- После того, как распалась агарозы, прохладном решение примерно 50 ° C, а затем добавьте 5 мкл ДНК, окрашивание красителя. Раствор тщательно перемешать и залить его в каппу в камере литья. Вставьте одну или несколько строк хорошо Комбс так, что есть достаточно скважин для всех образцов, а также размер молекулярных маркеров. Разрешить гель для 30 мин.
- После завершения выполнения ПЦР мкл 4 6 x ДНК загрузки краситель для каждого образца. Место в зале электрофореза геля и добавьте буфер TAE до лунки покрыты.
- Применяют 10 – 15 мкл из каждой реакции PCR в колодец в геле. Также 5 мкл молекулярного размера маркера. Затем запустите гель для 30 мин на 75 V.
- После завершения запуска изображение гель с помощью томографа голубой свет.
6. другие методы
-
Выражение протеина
Примечание: Выражение тест белков (болтовня и EibD) были подробно описаны в других местах2423,. Это краткий обзор основных шагов.- Преобразование BL21ΔABCF ΔТама with необходимые плазмид (pIBA2-YadA и pEibD10 и соответствующего управления плазмид) и выберите для трансформантов на LB + Amp.
- Для выражения протеина, прививать 100 мл с 1 мл раствора на ночь культуры бактерий, преобразованные LB средний + Amp и расти на 30 ° C до этапа середине журнала, в котором время, стимулировать производство белка с либо anhydrotetracycline (на 100 нг/мл) или Изопропиловый thiogalactoside (на 0,5 мм).
- После того, как 2 h индукции на 30 ° C, измерения мутности культур и собирать количество ячеек, соответствующих 50 мл на од600 = 1.0 центрифугированием культур для 15 мин на 4000 x g. Помыть лепешка 1 x 10 мм HEPES, рН 7,4, а затем либо храните его при 20 ° C или процесса он также как и шаг 6.2.
-
Экстракция внешней мембраны
Примечание: Наружной мембраны добыча осуществляется как описано в деталях Лео et al. 27. ниже кратко излагаются основные шаги.- Ресуспензируйте Пелле клеток в 1 мл 10 мм HEPES, рН 7,4, дополнены MgCl 10 мм2 и НКД2, лизоцим (0,1 мг/мл) и щепотку DNase I.
- Лизируйте клетки (например, с помощью било шарик).
- Центрифуга клетки вскоре (2 мин на 15600 x g) чтобы удалить ячейки мусор и переместить супернатант свежие трубки.
- Центрифуга клетки для 30 мин на 16000 x g, после чего, Ресуспензируйте гранулы в 400 мкл 1% N-лауроил sarcosine в 10 мм HEPES, рН 7,4.
- Клетки с агитации за 30 мин при комнатной температуре, после чего инкубировать, центрифуга их на 30 минут, как указано выше.
- Вымойте полупрозрачных гранул 1 x с 200 мкл 10 мм HEPES, рН 7,4, а затем Ресуспензируйте в 30 мкл 10 мм HEPES и добавить 10 мкл 4 x буфер образца SDS-PAGE.
-
Деятельность анализов
Примечание: Выполните SDS-PAGE и деятельность анализов для яда и EibD как описано28. Ниже кратко излагаются основные шаги. -
SDS-PAGE
- Тепло образцов на 50 ° C за 5 мин до их загрузки на геле полиакриламида, чтобы избежать денатурации белков.
- После разделения в SDS-PAGE передать белки винилидена фторид (PVDF) мембраны.
- После передачи блокировать мембраны с 2% сухого обезжиренного молока порошок в PBS.
-
YadA коллаген далеко Западная помарка
- После блокирования, добавить говядину коллаген типа, которую я разведен в блокирующем буфере до концентрации 10 мкг/мл и Проинкубируйте мембрану за 1 час.
- Промойте мембрану 2 x с PBS + 0,05% Tween20 (PBS-T).
- Добавьте основное антитело (моноклонального анти коллаген COL-1) мембраны, разбавленным стоматологов в блокирующем буфере.
- После инкубации мембран для 1 h, мыть 2 x, как указано в шаге 6.3.2.2, а затем добавить вторичные антитела [Коза анти мыши IgG хрен пероксидазы (ПХ) конъюгата], разбавленным мэм в блокирующем буфере.
- Проинкубируйте мембрану за 1 ч, затем промыть 2 x с PBS-T. Добавьте расширение Хемилюминесцентный субстрат мембрана в соответствии с инструкциями производителя и обнаружить группы с помощью CCD камеры.
-
EibD-IgG пробирного привязки
- После блокирования, добавьте вторичное антитело (коза анти кролик ПХ), разбавленный стоматологов в блокирующем буфере.
- Проинкубируйте мембрану за 1 ч, затем промыть 2 x с PBS. Хемилюминесцентный обнаружения как указано в шаге 6.3.2.5.
Результаты
Поколение Тама нокаут-о BL21ΔABCF:
Стратегия, изложенная выше ранее использовался для создания производных штамм BL21(DE3), Стандартный лабораторный штамм, используемых для производства белка, который оптимизирован для производства белка...
Обсуждение
Трансдукция P1 является быстрый, надежный и надежный метод для генерации гена удалений в E. coli. Здесь это подтверждается преобразователя Тама удаления мутант из штамма доноров Keio BL21-производные получателю. Основные этапы в процессе трансдукции являются производство преобразо?...
Раскрытие информации
Авторы не имеют ничего сообщать.
Благодарности
Кэйо коллекции штаммов были получены от национального проекта Биоресурс (Ниг, Япония): E. coli. Мы благодарим Дирк Линке (Департамент Biosciences, Университет Осло) за его неизменную поддержку. Эта работа финансировалась исследовательским советом Норвегии молодой исследователь Грант 249793 (Jack C. Leo).
Материалы
| Name | Company | Catalog Number | Comments |
| Strains | |||
| E. coli BW25113 | NIG | ME6092 | Wild-type strain of Keio collection |
| E. coli BL21(DE3) | Merck | 69450-3 | Expression strain |
| E. coli BL21DABCF | Addgene | 102270 | Derived from BL21(DE3) |
| E. coli JW4179 | NIG | JW4179-KC | tamA deletion mutant |
| P1 vir | NIG | HR16 | Generally transducing bacteriophage |
| Plasmids | |||
| pCP20 | CGSC | 14177 | conditionally replicating plasmid with FLP |
| pASK-IBA2 | IBA GmbH | 2-1301-000 | expression vector |
| pEibD10 | N/A | N/A | for production of EibD; plasmid available on request |
| pET22b+ | Merck | 69744-3 | expression vector |
| pIBA2-YadA | N/A | N/A | for production of YadA; plasmid available on request |
| Chemicals | |||
| Acetic acid | ThermoFisher | 33209 | |
| Agar | BD Bacto | 214010 | |
| Agarose | Lonza | 50004 | |
| Ampicillin | Applichem | A0839 | |
| Anhydrotetracycline | Abcam | ab145350 | |
| anti-collagen type I antibody COL-1 | Sigma | C2456 | |
| Bovine collagen type I | Sigma | C9791 | |
| Calcium chloride | Merck | 102382 | |
| Chloroform | Merck | 102445 | |
| Di-sodium hydrogen phosphate | VWR | 28029 | |
| DNA dye | Thermo | S33102 | |
| DNA molecular size marker | New England BioLabs | N3232S | |
| DNase I | Sigma | DN25 | |
| dNTP mix | New England Biolabs | N0447 | |
| ECL HRP substrate | Advansta | K-12045 | |
| EDTA | Applichem | A2937 | |
| Glycerol | VWR | 24388 | |
| goat anti-mouse IgG-HRP | Santa Cruz | sc-2005 | |
| goat anti-rabbit IgG-HRP | Agrisera | AS10668 | |
| HEPES | VWR | 30487 | |
| Isopropyl thiogalactoside | VWR | 43714 | |
| Kanamycin | Applichem | A1493 | |
| Lysozyme | Applichem | A4972 | |
| Magnesium chloride | VWR | 25108 | |
| Manganese chloride | Sigma | 221279 | |
| N-lauroyl sarcosine | Sigma | L9150 | |
| Skim milk powder | Sigma | 70166 | |
| Sodium chloride | VWR | 27808 | |
| tamA forward primer | Invitrogen | N/A | Sequence 5'-GAAAAAAGG ATATTCAGGAGAAAATGTG-3' |
| tamA reverse primer | Invitrogen | N/A | Sequence 5'-TCATAATT CTGGCCCCAGACC-3' |
| Taq DNA polymerase | New England Biolabs | M0267 | |
| Tri-sodium citrate | Merck | 106448 | |
| Tryptone | VWR | 84610 | |
| Tween20 | Sigma | P1379 | |
| Yeast extract | Merck | 103753 | |
| Equipment | |||
| Agarose gel electrophoresis chamber | Hoefer | SUB13 | |
| Bead beater | Thermo | FP120A-115 | |
| CCD camera | Kodak | 4000R | |
| Electroporation cuvettes | Bio-Rad | 165-2089 | |
| Electroporation unit | Bio-Rad | 1652100 | |
| Gel imager | Nippon Genetics | GP-03LED | |
| Incubating shaker | Infors HT | Minitron | |
| Incubator | VWR | 390-0482 | |
| Microcentrifuge | Eppendorf | 5415D | |
| Microwave oven | Samsung | CM1099A | |
| PCR machine | Biometra | Tpersonal | |
| PCR strips | Axygen | PCR-0208-CP-C | |
| pH meter | Hanna Instruments | HI2211-01 | |
| PVDF membrane | ThermoFisher | 88518 | |
| SDS-PAG electrophoresis chamber | ThermoFisher | A25977 | |
| Tabletop centrifuge | Beckman Coulter | B06322 | |
| Vortex mixer | Scientific Industries | SI-0236 | |
| Water bath | GFL | D3006 | |
| Wet transfer unit | Hoefer | TE22 |
Ссылки
- Blount, Z. D. The unexhausted potential of E. coli. eLife. 4, e05826 (2015).
- Hamilton, C. M., Aldea, M., Washburn, B. K., Babitzke, P., Kushner, S. R. New method for generating deletions and gene replacements in Escherichia coli. Journal of Bacteriology. 171 (9), 4617-4622 (1989).
- Link, A. J., Phillips, D., Church, G. M. Methods for generating precise deletions and insertions in the genome of wild-type Escherichia coli: application to open reading frame characterization. Journal of Bacteriology. 179 (20), 6228-6237 (1997).
- Sawitzke, J. A., Thomason, L. C., Costantino, N., Bubunenko, M., Datta, S., Court, D. L. Recombineering: in vivo genetic engineering in E. coli, S. enterica, and beyond. Methods in Enzymology. 421, 171-199 (2007).
- Datsenko, K. A., Wanner, B. L. One-step inactivation of chromosomal genes in Escherichia coli K-12 using PCR products. Proceedings of the National Academy of Sciences of the United States of America. 97 (12), 6640-6645 (2000).
- Muyrers, J. P., Zhang, Y., Stewart, A. F. Techniques: Recombinogenic engineering--new options for cloning and manipulating DNA. Trends in Biochemical Sciences. 26 (5), 325-331 (2001).
- Baba, T., et al. Construction of Escherichia coli K-12 in-frame, single-gene knockout mutants: the Keio collection. Molecular Systems Biology. 2, (2006).
- Yamamoto, N., et al. Update on the Keio collection of Escherichia coli single-gene deletion mutants. Molecular Systems Biology. 5, 335 (2009).
- Baba, T., Huan, H. -. C., Datsenko, K., Wanner, B. L., Mori, H. The applications of systematic in-frame, single-gene knockout mutant collection of Escherichia coli K-12. Methods in Molecular Biology. 416, 183-194 (2008).
- Chattopadhyay, M. K., Tabor, C. W., Tabor, H. Polyamines are not required for aerobic growth of Escherichia coli: preparation of a strain with deletions in all of the genes for polyamine biosynthesis. Journal of Bacteriology. 191 (17), 5549-5552 (2009).
- Xie, X., Wong, W. W., Tang, Y. Improving simvastatin bioconversion in Escherichia coli by deletion of bioH. Metabolic Engineering. 9 (4), 379-386 (2007).
- Maeda, T., Sanchez-Torres, V., Wood, T. K. Escherichia coli hydrogenase 3 is a reversible enzyme possessing hydrogen uptake and synthesis activities. Applied Microbiology and Biotechnology. 76 (5), 1035-1042 (2007).
- Meehan, B. M., Landeta, C., Boyd, D., Beckwith, J. The disulfide bond formation pathway is essential for anaerobic growth of Escherichia coli. Journal of Bacteriology. 199 (16), (2017).
- Niba, E. T. E., Naka, Y., Nagase, M., Mori, H., Kitakawa, M. A genome-wide approach to identify the genes involved in biofilm formation in E. coli. DNA Research. 14 (6), 237-246 (2008).
- Heinz, E., et al. Conserved features in the structure, mechanism, and biogenesis of the inverse autotransporter protein family. Genome Biology and Evolution. 8 (6), 1690-1705 (2016).
- Selkrig, J., et al. Discovery of an archetypal protein transport system in bacterial outer membranes. Nature Structural & Molecular Biology. 19 (5), 506-510 (2012).
- Stubenrauch, C., et al. Effective assembly of fimbriae in Escherichia coli depends on the translocation assembly module nanomachine. Nature Microbiology. 1 (7), 16064 (2016).
- Leo, J. C., Goldman, A. The immunoglobulin-binding Eib proteins from Escherichia coli are receptors for IgG Fc. Molecular Immunology. 46 (8-9), 1860-1866 (2009).
- Mühlenkamp, M., Oberhettinger, P., Leo, J. C., Linke, D., Schütz, M. S. Yersinia adhesin A (YadA) - Beauty & beast. International Journal of Medical Microbiology. 305 (2), 252-258 (2015).
- Studier, F. W., Moffatt, B. A. Use of bacteriophage T7 RNA polymerase to direct selective high-level expression of cloned genes. Journal of Molecular Biology. 189 (1), 113-130 (1986).
- Meuskens, I., Michalik, M., Chauhan, N., Linke, D., Leo, J. C. A new strain collection for improved expression of outer membrane proteins. Frontiers in Cellular and Infection Microbiology. 7, 464 (2017).
- Cherepanov, P. P., Wackernagel, W. Gene disruption in Escherichia coli: TcR and KmR cassettes with the option of Flp-catalyzed excision of the antibiotic-resistance determinant. Gene. 158 (1), 9-14 (1995).
- Grosskinsky, U., et al. A conserved glycine residue of trimeric autotransporter domains plays a key role in Yersinia adhesin A autotransport. Journal of Bacteriology. 189 (24), 9011-9019 (2007).
- Leo, J. C., et al. The structure of E. coli IgG-binding protein D suggests a general model for bending and binding in trimeric autotransporter adhesins. Structure. 19 (7), 1021-1030 (1993).
- Bertani, G. Studies on lysogenesis. I. The mode of phage liberation by lysogenic Escherichia coli. Journal of Bacteriology. 62 (3), 293-300 (1951).
- Hanahan, D. Studies on transformation of Escherichia coli with plasmids. Journal of Molecular Biology. 166 (4), 557-580 (1983).
- Leo, J. C., Oberhettinger, P., Linke, D. Assessing the outer membrane insertion and folding of multimeric transmembrane β-barrel proteins. Methods in Molecular Biology. , 157-167 (2015).
- Mikula, K. M., Leo, J. C., Łyskowski, A., Kedracka-Krok, S., Pirog, A., Goldman, A. The translocation domain in trimeric autotransporter adhesins is necessary and sufficient for trimerization and autotransportation. Journal of Bacteriology. 194 (4), 827-838 (2012).
- Thomason, L. C., Costantino, N., Court, D. L., Ausubel, F. M. E. coli genome manipulation by P1 transduction. Current Protocols in Molecular Biology. , (2007).
- Franklin, N. C. Mutation in gal U gene of E. coli blocks phage P1 infection. Virology. 38 (1), 189-191 (1969).
- Jeong, H., et al. Genome sequences of Escherichia coli B strains REL606 and BL21(DE3). Journal of Molecular Biology. 394 (4), 644-652 (2009).
- Greene, E. C. DNA Sequence Alignment during Homologous Recombination. Journal of Biological Chemistry. 291 (22), 11572-11580 (2016).
- Watt, V. M., Ingles, C. J., Urdea, M. S., Rutter, W. J. Homology requirements for recombination in Escherichia coli. Proceedings of the National Academy of Sciences of the United States of America. 82 (14), 4768-4772 (1985).
- Ho, T. D., Waldor, M. K. Enterohemorrhagic Escherichia coli O157:H7 gal mutants are sensitive to bacteriophage P1 and defective in intestinal colonization. Infection and Immunity. 75 (4), 1661-1666 (2006).
- Ikeda, H., Tomizawa, J. I. Transducing fragments in generalized transduction by phage P1. I. Molecular origin of the fragments. Journal of Molecular Biology. 14 (1), 85-109 (1965).
- Murooka, Y., Harada, T. Expansion of the host range of coliphage P1 and gene transfer from enteric bacteria to other Gam-negative bacteria. Applied and Environmental Microbiology. 38 (4), 754-757 (1979).
- Goldberg, R. B., Bender, R. A., Streicher, S. L. Direct selection for P1-sensitive mutants of enteric bacteria. Journal of Bacteriology. 118 (3), 810-814 (1974).
- O'Connor, K. A., Zusman, D. R. Coliphage P1-mediated transduction of cloned DNA from Escherichia coli to Myxococcus xanthus: use for complementation and recombinational analyses. Journal of Bacteriology. 155 (1), 317-329 (1983).
Перепечатки и разрешения
Запросить разрешение на использование текста или рисунков этого JoVE статьи
Запросить разрешениеСмотреть дополнительные статьи
This article has been published
Video Coming Soon
Авторские права © 2025 MyJoVE Corporation. Все права защищены