É necessária uma assinatura da JoVE para visualizar este conteúdo. Faça login ou comece sua avaliação gratuita.
Method Article
Produzindo a deleção do Gene na Escherichia coli por transdução P1 com fitas de resistência aos antibióticos sujeitos
Neste Artigo
Resumo
Aqui nós apresentamos um protocolo para o uso de construções de exclusão de resistência aos antibióticos-gaveta pré-existente, como uma base para a tomada de mutantes de exclusão em outras estirpes de Escherichia coli . Tais mutações de exclusão podem ser mobilizadas e inseridas o locus correspondente de uma estirpe de destinatário usando transdução de bacteriófago P1.
Resumo
Uma primeira abordagem para estudar a função de um gene desconhecido em bactérias é criar um knock-out deste gene. Aqui, descrevemos um protocolo robusto e rápido para transferência de mutações do gene exclusão de uma estirpe de Escherichia coli para outro usando transdução generalizada com o bacteriófago P1. Este método requer que a mutação seja selecionável (por exemplo, baseado em interrupções de gene usando inserções de gaveta antibiótico). Tais fitas antibióticas podem ser mobilizadas a partir de uma estirpe de doador e introduziram uma destinatário estirpe de interesse para rapidamente e facilmente geram um mutante de apagamento do gene. A gaveta antibiótica pode ser projetada para incluir sites de reconhecimento de flipase que permitem que a excisão da fita por um site-specific recombinase para produzir um nocaute limpo com apenas uma sequência de ~ 100-base-par-longa cicatriz no genoma. Demonstramos o protocolo ao nocautear o gene de tamA codifica um fator de montagem envolvido na biogênese de autotransporter e testar o efeito desse nocaute sobre a biogênese e a função de dois autotransporter trimeric adesinas. Embora a supressão do gene por transdução P1 tem suas limitações, a facilidade e rapidez de sua implementação seja uma alternativa atraente para outros métodos de apagamento do gene.
Introdução
Uma primeira abordagem comum para estudar a função de um gene é executar o mutagenesis de mata-mata e observar o fenótipo resultante. Isto também é denominado genética reversa. A bactéria e. coli tem sido o carro-chefe da biologia molecular nos últimos 70 anos, ou então, devido à facilidade do seu cultivo e sua acessibilidade para manipulação genética1. Vários métodos foram desenvolvidos para produzir exclusões de gene em e. coli, incluindo troca de marcador a mutagênese2,3 e, mais recentemente, recombineering usando o λ vermelho ou Rac ET sistemas4,5 , 6.
Em um sistema amplamente utilizado, codificação de sequências de genes individuais são substituídas por uma gaveta de resistência aos antibióticos que pode mais tarde ser extirpada do cromossoma5,7. As sequências de codificação são substituídas, por exemplo, uma gaveta de resistência de canamicina (Kan), que é ladeada por sites de destino (FRT) de reconhecimento de flipase (FLP) em ambos os lados. Os sites da FRT são reconhecidos pela recombinase FLP, que medeia a site-specific recombinação entre os sites FRT, levando à supressão da fita Kan. Desta forma, uma exclusão total de sequência de código de um determinado gene pode ser conseguida, deixando para trás apenas uma sequência de cicatriz mínima de aproximadamente 100 pares de bases (bp) (Figura 1).
Pouco mais de uma década atrás, foi desenvolvida a coleção chamada de Keio. Esta é uma biblioteca bacteriana com base em um padrão de laboratório cepa de Escherichia coli K12, onde quase todos os genes não essenciais foram individualmente excluídos por λ recombinação vermelho7,8. Os clones dentro de cada coleção tem uma sequência de código substituída com uma gaveta de resistência Kan sujeitas. A coleção de Keio provou para ser uma ferramenta útil para muitas aplicações9. Uma tal aplicação é a produção de mutantes de exclusão em outras estirpes de Escherichia coli . A gaveta de Kan de um clone de exclusão determinada pode ser mobilizada por geralmente transducing bacteriófagos, tais como P110,11,12,13,14. Um estoque de fago preparado a partir de uma tensão tão grande pode ser usado para infectar um destinatário Escherichia coli cepa de interesse, onde em uma frequência baixa, mas de confiança o Kan região contendo gaveta pode ser incorporada o genoma destinatário por recombinação homóloga (Figura 2). Transductants podem ser selecionados para o crescimento em meio de Kan-contendo. Em seguida, se a remoção da fita resistência aos antibióticos é desejada, a recombinase FLP pode ser fornecido para a estirpe transductant em trans. Após a cura o plasmídeo contendo FLP, que transporta um marcador de resistência a ampicilina (Amp), clones de Kan e Amp-sensíveis são selecionados para, e a correta excisão de sequência de codificação a selvagem-tipo e a gaveta de Kan são verificadas por PCR de colônia.
Aqui, é apresentado um protocolo detalhado, descrevendo cada uma das etapas na produção de uma estirpe de Escherichia coli , baseado na estratégia delineada acima de mata-mata. Como exemplo, uma deleção do gene da tamA é demonstrada. tamA codifica uma proteína de β-barril de membrana exterior que é uma parte do transporte e módulo de montagem (TAM), que está envolvida na biogênese de certas proteínas autotransporter e pili15,16,17. Esta estirpe de mata-mata foi usado para examinar o efeito da exclusão tamA sobre a biogênese de dois autotransporter trimeric adesinas (TAAs), a Yersinia adesina YadA e a Escherichia coli imunoglobulinas (Ig)-vinculação TAA EibD 18,19.
Protocolo
1. cepas e plasmídeos
-
Estirpes bacterianas
- Use a e. coli as estirpes BW251135, JW4179 (BW25113 tamA::kan)7, BL21(DE3)20e BL21ΔABCF21. Consulte Tabela de materiais para mais informações.
-
Bacteriófagos
- Use o fago P1vir para a transdução geral. Armazenar a Praga como um estoque líquido com algumas gotas de clorofórmio (consulte a etapa 2.2). Para obter mais informações, consulte Tabela de materiais.
-
Plasmídeos
- Use os seguintes plasmídeos neste protocolo: pCP2022, pIBA2-YadA23e pEibD1024. Como plasmídeos de controle, use pASK-IBA2 e pET22b (ver Tabela de materiais).
-
Condições de crescimento
- Propaga as bactérias em um lisogenia caldo (LB) médio25 com agitação vigorosa (180 – 200 rpm) a 37 ° C ou 30 ° C, no caso de tensões que contém pCP20 e BL21ΔABCF.
- Executar o plasmídeo curando a 43 ° C.
- Para um meio sólido, suplemento LB com ágar de 1% (p/v).
- Para ágar superior, suplemento LB com 0,7% agar e 10 mM CaCl2 e autoclave a médio. Use meio de SOC para a recuperação após electroporation26.
- Use as seguintes concentrações de antibióticos: 100 μg/mL para o Amp e 25 μg/mL para Kan.
2. preparar um Phage lisado
-
Infecção da estirpe dos doadores
- Crescer a tensão do doador JW4197 em 5 mL de LB suplementado com 10 mM CaCl2 e opcionalmente com Kan (25 µ g/mL) para uma densidade óptica em 600 nm (OD600) de ~ 1.0. Medir o valor de600 OD usando um espectrofotômetro.
- Fazer uma série de diluição de um stock de fago P1 existente no meio de LB: recomendado diluições são entre 10-3 a 10-7.
- Mix de 200 μL da suspensão bacteriana e 100 μL de uma diluição do fago determinado em um tubo de centrífuga de 15 mL ou equivalente. Prepare os tubos como muitos como diluições do fago. Incube os tubos por 20 min a 37 ° C, sem agitação.
- Adicionar ~ 3 mL de ágar superior fundido (~ 50 ° C) suplementado com 10 mM CaCl2 para os tubos, homogeneizar o conteúdo vortexing dos tubos em breve e despeje as misturas em placas LB escaldadas para fazer camadas mesmo.
- Incubar as placas durante a noite a 37 ° C.
-
Lisado preparação
- No dia seguinte escolha uma placa com um crescimento semi confluente de placas do fago. Em um prato semi confluente, aproximadamente metade da área de superfície da placa é evidente (Figura 3).
- Raspar a camada superior de ágar de tal uma placa usando um loop de inoculação ou uma ferramenta semelhante e coloque o ágar superior num tubo de centrífuga. Adicionar 1 a 2 mL de LB e uma gota de clorofórmio e vortex o tubo vigorosamente para ~ 1 min Add o clorofórmio em uma coifa.
- Centrifugar o tubo durante 15 minutos a 4.000 x g ou mais rápido para o agar e células bacterianas de Pelotas.
- Mova o sobrenadante para um tubo de microcentrifugadora fresco, evitando carregando ao longo de todos os restos da pelota. Adicione 2 gotas de clorofórmio e armazenar o lisado 4 e 10 ° c. Adicione clorofórmio em uma coifa. Não congele o fago lisado como isso irá resultar em uma redução significativa do número de partículas infecciosas.
-
Determinação do título de lisado
- Cresce BW25113 em LB suplementado com 10 mM CaCl2 a 37 ° C até a cultura atinge uma OD600 ~ 1.0.
- Prepare uma série de diluição do fago lisado em LB (por exemplo, 10-6– 10-9).
Nota: Tenha cuidado para pipetar amostra do topo do lisado para evitar a transferência de clorofórmio para as diluições. - Mix de 200 μL da suspensão bacteriana e 100 μL de uma diluição do fago determinado em um tubo de centrífuga de 15 mL ou equivalente. Prepare os tubos como muitos como diluições do fago. Incube os tubos por 20 min a 37 ° C, sem agitação.
- Adicionar ~ 3 mL de ágar superior fundido (~ 50 ° C) suplementado com 10 mM CaCl2 para os tubos, homogeneizar o conteúdo vortexing dos tubos em breve e despeje a mistura em placas LB escaldadas para fazer camadas mesmo.
- Incubar as placas durante a noite a 37 ° C.
- No dia seguinte contar o número de placas (regiões claras na esteira da bacteriana) por cada placa e calcular o título do lisado, usando a seguinte fórmula:

Exemplo: Em placas com as diluições 10-7, 10-8e 10-9, existem 231, 27 e 2 placas, respectivamente. Como 100 μL de cada diluição foi utilizado para a infecção, e porque o título é expresso como formadoras de placa unidades (pfu) / mL, o fator do chapeamento é 10. Estes números são inseridos na fórmula: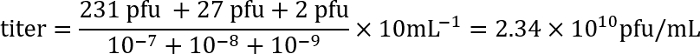
Assim, o título é aproximadamente 2 x 1010 do fago infecciosas partículas/mL.
3. P1 transdução
-
Preparando as células destinatários
- Crescer o destinatário estirpe BL21ΔABCF em LB suplementado com uma densidade óptica em 600 nm (OD600) de ~ 1.0. Use um espectrofotômetro para medir o valor de600 OD.
- Calcule o volume do fago lisado necessário para a realização de uma multiplicidade de valor de infecção (MOI) de 0,5. Para calcular o MOI, estime o número de bactérias baseada o OD600 da cultura. Suponha que um valor de600 OD de 1.0 corresponde ~ 109 UFC/mL. Calcule o volume necessário com base do título conhecido do lisado.
Exemplo (com base do título calculado no exemplo após etapa 2.3.6):
- Adicione o CaCl2 para a cultura de estirpe destinatário a 10 mM e misture. Tome 1 mL da cultura para a transdução.
-
Realizando a transdução
- Adicionar o volume apropriado do fago lisado a 1 mL de cultura destinatário (incluindo CaCl2 a 10 mM), conforme calculado no passo 3.1.2 e misture delicadamente.
Nota: Tenha cuidado para pipetar amostra do topo do lisado para evitar transferir clorofórmio à mistura. - Estaticamente, incubar a mistura por 20 min a 37 ° C.
- Pare a infecção pela adição de citrato de sódio, pH 5,5 a 100 mM.
- Centrifugar as bactérias (5.000 x g por 2 min) e remover o sobrenadante e, em seguida, resuspenda-los em 1 mL de fresco LB suplementado com citrato de sódio de 100 mM, pH 5,5.
- Lave as células mais duas vezes como na etapa 3.2.4 para garantir a remoção do fago livre e cálcio.
- Ressuspender as bactérias em 1 mL de fresco LB suplementado com citrato de sódio de 100 mM, pH 5,5. Incube a bactéria a 30 ° C, durante 1 h com agitação (> 100 rpm).
- Recolher as bactérias por centrifugação (5.000 x g por 2 min) e Resuspenda-los em ~ 100 μL de LB com citrato de sódio de 100 mM, pH 5,5.
- Espalhar as bactérias em uma placa LB suplementado com Kan a 25 µ g/mL e citrato de sódio de 10 mM, pH 5,5 e crescer a bactéria a 30 ° C até colônias aparecem (~ 24 h).
- Adicionar o volume apropriado do fago lisado a 1 mL de cultura destinatário (incluindo CaCl2 a 10 mM), conforme calculado no passo 3.1.2 e misture delicadamente.
-
Selecionando as transductants
- Uma vez que as colónias têm crescido na placa de seleção, restreak-los na LB + Kan para colônias única e crescer a 30 ° C até colônias única aparecem.
4. extirpando a gaveta de Kan
-
A transformação com plasmídeo de recombinação
- Fazer o electrocompetent de estirpe BL21ΔABCF Kan-resistente.
- Crescer a tensão de uma única colônia em fresco LB (5 mL), suplementado com Kan (25 µ g/mL) a 30 ° C até que o OD600 ~0.5-0,7.
- A partir daqui, execute as etapas a seguir a 4 ° C ou em gelo. Centrifugar as bactérias (5.000 x g durante 10 minutos) e remover o sobrenadante.
- Lave o centrifugado com água destilada gelada duas vezes, repetindo o passo anterior de centrifugação e, finalmente, resuspenda o pellet de células em 100 µ l de gelado 10% de glicerol.
- Arrefecer cubetas de eletroporação de 1 mm no gelo.
- Adicionar 1 pg de plasmídeo (pCP20) para a suspensão de eritrócitos, misture-os delicadamente a suspensão e transferi-lo para uma cubeta de eletroporação de refrigeração.
- Definir o electroporator para 1,8 kV e electroporate as células.
- Resgate de células transformadas, adicionando 1 mL de meio SOC e fazê-los crescer por 1h a 30 ° C.
- Placa de 100 µ l de células na LB + Amp placas e crescer as células a 30 ° C durante a noite.
- Fazer o electrocompetent de estirpe BL21ΔABCF Kan-resistente.
-
Indução da recombinação
- Palheta simples colônias do LB + Amp placa e inocular fresco LB, omitindo todos os antibióticos.
- Crescem as células à temperatura não-permissivo (43 ° C) durante a noite para induzir a expressão de recombinase FLP.
-
Seleção de recombinantes
- Fazer diluições em série e 50 µ l de uma diluição de6 105-10 em placas não-seletivo da placa e cultivá-lo durante a noite a 30 ° C.
5. verificação da exclusão Gene
-
Verificação da perda do plasmídeo e recombinação de sucesso
- Colônia de raia única dos pratos preparados na etapa 4.3.1 na LB Kan, LB + Amp e placas LB sem antibióticos, nesta ordem. Para ajudar as estrias, usar uma grade de Colônia (ver arquivo complementar 1). Crescem as placas a 30 ° C até colônias aparecem (~ 24 h).
- Pegue 10 – 20 clones que têm crescido na placa não-seletivo, mas não conseguiram crescer na mídia seletiva para posterior verificação.
-
Verificação suplementar por PCR de colônia
- Realizar uma colônia PCR com primers flanqueando o tamA sequência de código (consulte a Tabela de materiais).
- Prepare uma mistura PCR a mestre. A quantidade de mistura necessária depende do número de colónias a ser testado (Veja o exemplo na tabela 1). Misture os reagentes no gelo, adicionar o polymerase última.
- Homogeneizar a mistura de mestre de PCR e, em seguida, diluir 20 µ l em tubos de uma tira PCR. Pegue uma pequena quantidade de cada colônia para ser exibido usando uma ponta de pipeta estéril e adicioná-lo para um tubo. Lembre-se de incluir a estirpe de destinatário original para comparação. Opcionalmente, também inclua a estirpe do doador.
- Executar uma reação de PCR (ver tabela 2 para o programa utilizado).
- Prepare um gel de agarose a 1%.
- Para um gel 50 mL, medir 0,5 g de agarose e adicionar 50 mL de tampão TAE (40 mM Tris, ácido acético de 20 mM e 1 mM EDTA, pH 8.0). Aqueça a mistura em um forno de microondas até dissolver toda da agarose.
- Uma vez que a agarose dissolveu, arrefecer a solução para cerca de 50 ° C e adicione 5 μL de DNA coloração tintura. Misture a solução bem e despeje em uma bandeja de gel em uma câmara de fundição. Insira uma ou mais linhas de pentes bem assim há poços suficientes para todas as amostras, bem como marcadores de tamanho molecular. Permitir que o gel definir por 30 min.
- Uma vez concluída a execução PCR, adicione 4 µ l de 6 x tintura do carregamento de DNA de cada amostra. Colocar o gel na câmara de eletroforese e adicionar tampão TAE até os poços estão cobertos.
- Aplica 10 a 15 µ l de cada reação de PCR para um poço no gel. Também Adicione 5 µ l de um marcador de tamanho molecular. Em seguida, execute o gel por 30 min a 75 V.
- Uma vez concluída a execução, o gel usando um gerador de imagens de luz azul da imagem.
6. outras técnicas
-
Expressão da proteína
Nota: A expressão de proteínas de teste (YadA e EibD) tem sido descrito em detalhe em outro lugar,23,24. Este é um breve resumo das principais etapas.- Transformar BL21ΔABCF ΔtamA with os necessários plasmídeos (YadA-pIBA2 e pEibD10 e os correspondente plasmídeos de controle) e selecione para transformants na LB + Amp.
- Para a expressão da proteína, inocular 100 mL de meio LB + Amp 1ml da cultura de bactérias transformadas durante a noite e crescer estas a 30 ° C até a fase log de mid, momento em que, induzir a produção de proteína com qualquer anhydrotetracycline (a 100 ng/mL) ou thiogalactoside isopropílico (a 0,5 mM).
- Depois de 2h de indução a 30 ° C, medir a turbidez das culturas e coletar um número de células, correspondente a 50 mL no OD600 = 1.0 por centrifugação as culturas por 15 min a 4.000 x g. Lave o pellet 1x com 10 mM HEPES, pH 7,4 e em seguida ou armazená-lo a-20 ° C ou processo etapa 6.2 ainda mais.
-
Extração de membrana externa
Nota: Extração de membrana externa é realizada conforme descrito em detalhe por Leo et al . 27. as principais etapas estão resumidas a seguir.- Ressuspender o pellet de células em 1 mL de 10 mM HEPES, pH 7,4, suplementado com 10 mM MgCl2 e MnCl2, lisozima (0,1 mg/mL) e uma pitada de DNase eu.
- Lise as células (por exemplo, usando um batedor do grânulo).
- Centrifugar as células em breve (2 min a 15.600 x g) para remover os detritos celulares e mover o sobrenadante para um tubo de fresco.
- Centrifugar as células por 30 min a 16.000 x g, após o qual, resuspenda o pellet em 400 μL de 1% N-lauroyl sarcosina em 10 mM HEPES, pH 7,4.
- Incube as celulas com agitação por 30 min à temperatura ambiente, após o qual, centrifugue-los durante 30 min como acima.
- Lave o translúcido da pelota 1x com 200 μL de 10 mM HEPES, pH 7,4, resuspenda-lo em 30 μL de 10 mM HEPES e adiciona 10 μL de tampão de amostra SDS-PAGE de 4x.
-
Ensaios de atividade
Nota: Realizar ensaios de SDS-PAGE e atividade para YadA e EibD como descrito28. As principais etapas estão resumidas a seguir. -
SDS-PAGE
- Aquece as amostras a 50 ° C por 5 min antes de carregá-los em um gel de poliacrilamida, para evitar a desnaturação das proteínas.
- Após a separação em SDS-PAGE, transferi as proteínas para uma membrana (PVDF) de difluoreto de polivinilideno.
- Após a transferência, bloquear a membrana com pó de leite desnatado 2% em PBS.
-
Borrão do extremo-oeste YadA-colágeno
- Após o bloqueio, adicionar tipo de colágeno bovino que diluído em tampão de bloqueio a uma concentração de 10 μg/mL e incubar a membrana por 1h.
- Lavar a membrana 2 x com PBS + 0,05% Tween20 (PBS-T).
- Adicione o anticorpo primário (anticorpo monoclonal anticolágeno COL-1) para a membrana, 1:2,000 diluído em tampão de bloqueio.
- Após a incubação da membrana por 1h, lave 2x como mencionado na etapa 6.3.2.2 e em seguida, adicione o anticorpo secundário [conjugado de peroxidase (HRP) de rábano-IgG de anticabra rato], 1:10,000 diluído em tampão de bloqueio.
- Incubar a membrana para 1 h e, em seguida, lave-o 2 vezes com PBS-T. Adicionar um substrato quimioluminescente reforçado para a membrana de acordo com as instruções do fabricante e detectar a banda usando uma câmera CCD.
-
EibD-IgG ensaio
- Após o bloqueio, adicione um anticorpo secundário (de cabra anticoelho HRP), diluído 1:2,000 em tampão de bloqueio.
- Incubar a membrana para 1 h e, em seguida, lave-o 2 x com PBS. Execute a detecção quimioluminescente como mencionado na etapa 6.3.2.5.
Resultados
Geração de um tamA Knock-out de BL21ΔABCF:
A estratégia acima descrita anteriormente foi usada para produzir uma tensão derivada de BL21(DE3), uma estirpe de laboratório padrão usada para a produção de proteína, que é otimizada para a produção de proteínas de membrana exterior e chamada BL21ΔABCF21. Esta tensão não possui quatro genes que codificam para proteínas de membrana extern...
Discussão
Transdução de P1 é um método rápido, robusto e confiável para a geração de exclusões de gene em e. coli. Isso é demonstrado aqui pelo transducing um mutante de exclusão de tamA de uma estirpe de doador de Keio para um destinatário BL21-derivado. As grandes etapas do processo de transdução são a produção do transducing lisada, a transdução em si, a excisão da fita Kan resistência e a verificação do knock-out pelo PCR. No total, o processo leva cerca de 1 semana e requer sem método...
Divulgações
Os autores não têm nada para divulgar.
Agradecimentos
Cepas de coleção de Keio foram obtidas a partir do projeto nacional de BioResource (NIG, Japão): Escherichia coli. Agradecemos seu apoio contínuo Dirk Linke (departamento de Biociências, Universidade de Oslo). Este trabalho foi financiado pelo Conselho de pesquisa da Noruega jovem pesquisador grant 249793 (Jack C. Leo).
Materiais
| Name | Company | Catalog Number | Comments |
| Strains | |||
| E. coli BW25113 | NIG | ME6092 | Wild-type strain of Keio collection |
| E. coli BL21(DE3) | Merck | 69450-3 | Expression strain |
| E. coli BL21DABCF | Addgene | 102270 | Derived from BL21(DE3) |
| E. coli JW4179 | NIG | JW4179-KC | tamA deletion mutant |
| P1 vir | NIG | HR16 | Generally transducing bacteriophage |
| Plasmids | |||
| pCP20 | CGSC | 14177 | conditionally replicating plasmid with FLP |
| pASK-IBA2 | IBA GmbH | 2-1301-000 | expression vector |
| pEibD10 | N/A | N/A | for production of EibD; plasmid available on request |
| pET22b+ | Merck | 69744-3 | expression vector |
| pIBA2-YadA | N/A | N/A | for production of YadA; plasmid available on request |
| Chemicals | |||
| Acetic acid | ThermoFisher | 33209 | |
| Agar | BD Bacto | 214010 | |
| Agarose | Lonza | 50004 | |
| Ampicillin | Applichem | A0839 | |
| Anhydrotetracycline | Abcam | ab145350 | |
| anti-collagen type I antibody COL-1 | Sigma | C2456 | |
| Bovine collagen type I | Sigma | C9791 | |
| Calcium chloride | Merck | 102382 | |
| Chloroform | Merck | 102445 | |
| Di-sodium hydrogen phosphate | VWR | 28029 | |
| DNA dye | Thermo | S33102 | |
| DNA molecular size marker | New England BioLabs | N3232S | |
| DNase I | Sigma | DN25 | |
| dNTP mix | New England Biolabs | N0447 | |
| ECL HRP substrate | Advansta | K-12045 | |
| EDTA | Applichem | A2937 | |
| Glycerol | VWR | 24388 | |
| goat anti-mouse IgG-HRP | Santa Cruz | sc-2005 | |
| goat anti-rabbit IgG-HRP | Agrisera | AS10668 | |
| HEPES | VWR | 30487 | |
| Isopropyl thiogalactoside | VWR | 43714 | |
| Kanamycin | Applichem | A1493 | |
| Lysozyme | Applichem | A4972 | |
| Magnesium chloride | VWR | 25108 | |
| Manganese chloride | Sigma | 221279 | |
| N-lauroyl sarcosine | Sigma | L9150 | |
| Skim milk powder | Sigma | 70166 | |
| Sodium chloride | VWR | 27808 | |
| tamA forward primer | Invitrogen | N/A | Sequence 5'-GAAAAAAGG ATATTCAGGAGAAAATGTG-3' |
| tamA reverse primer | Invitrogen | N/A | Sequence 5'-TCATAATT CTGGCCCCAGACC-3' |
| Taq DNA polymerase | New England Biolabs | M0267 | |
| Tri-sodium citrate | Merck | 106448 | |
| Tryptone | VWR | 84610 | |
| Tween20 | Sigma | P1379 | |
| Yeast extract | Merck | 103753 | |
| Equipment | |||
| Agarose gel electrophoresis chamber | Hoefer | SUB13 | |
| Bead beater | Thermo | FP120A-115 | |
| CCD camera | Kodak | 4000R | |
| Electroporation cuvettes | Bio-Rad | 165-2089 | |
| Electroporation unit | Bio-Rad | 1652100 | |
| Gel imager | Nippon Genetics | GP-03LED | |
| Incubating shaker | Infors HT | Minitron | |
| Incubator | VWR | 390-0482 | |
| Microcentrifuge | Eppendorf | 5415D | |
| Microwave oven | Samsung | CM1099A | |
| PCR machine | Biometra | Tpersonal | |
| PCR strips | Axygen | PCR-0208-CP-C | |
| pH meter | Hanna Instruments | HI2211-01 | |
| PVDF membrane | ThermoFisher | 88518 | |
| SDS-PAG electrophoresis chamber | ThermoFisher | A25977 | |
| Tabletop centrifuge | Beckman Coulter | B06322 | |
| Vortex mixer | Scientific Industries | SI-0236 | |
| Water bath | GFL | D3006 | |
| Wet transfer unit | Hoefer | TE22 |
Referências
- Blount, Z. D. The unexhausted potential of E. coli. eLife. 4, e05826 (2015).
- Hamilton, C. M., Aldea, M., Washburn, B. K., Babitzke, P., Kushner, S. R. New method for generating deletions and gene replacements in Escherichia coli. Journal of Bacteriology. 171 (9), 4617-4622 (1989).
- Link, A. J., Phillips, D., Church, G. M. Methods for generating precise deletions and insertions in the genome of wild-type Escherichia coli: application to open reading frame characterization. Journal of Bacteriology. 179 (20), 6228-6237 (1997).
- Sawitzke, J. A., Thomason, L. C., Costantino, N., Bubunenko, M., Datta, S., Court, D. L. Recombineering: in vivo genetic engineering in E. coli, S. enterica, and beyond. Methods in Enzymology. 421, 171-199 (2007).
- Datsenko, K. A., Wanner, B. L. One-step inactivation of chromosomal genes in Escherichia coli K-12 using PCR products. Proceedings of the National Academy of Sciences of the United States of America. 97 (12), 6640-6645 (2000).
- Muyrers, J. P., Zhang, Y., Stewart, A. F. Techniques: Recombinogenic engineering--new options for cloning and manipulating DNA. Trends in Biochemical Sciences. 26 (5), 325-331 (2001).
- Baba, T., et al. Construction of Escherichia coli K-12 in-frame, single-gene knockout mutants: the Keio collection. Molecular Systems Biology. 2, (2006).
- Yamamoto, N., et al. Update on the Keio collection of Escherichia coli single-gene deletion mutants. Molecular Systems Biology. 5, 335 (2009).
- Baba, T., Huan, H. -. C., Datsenko, K., Wanner, B. L., Mori, H. The applications of systematic in-frame, single-gene knockout mutant collection of Escherichia coli K-12. Methods in Molecular Biology. 416, 183-194 (2008).
- Chattopadhyay, M. K., Tabor, C. W., Tabor, H. Polyamines are not required for aerobic growth of Escherichia coli: preparation of a strain with deletions in all of the genes for polyamine biosynthesis. Journal of Bacteriology. 191 (17), 5549-5552 (2009).
- Xie, X., Wong, W. W., Tang, Y. Improving simvastatin bioconversion in Escherichia coli by deletion of bioH. Metabolic Engineering. 9 (4), 379-386 (2007).
- Maeda, T., Sanchez-Torres, V., Wood, T. K. Escherichia coli hydrogenase 3 is a reversible enzyme possessing hydrogen uptake and synthesis activities. Applied Microbiology and Biotechnology. 76 (5), 1035-1042 (2007).
- Meehan, B. M., Landeta, C., Boyd, D., Beckwith, J. The disulfide bond formation pathway is essential for anaerobic growth of Escherichia coli. Journal of Bacteriology. 199 (16), (2017).
- Niba, E. T. E., Naka, Y., Nagase, M., Mori, H., Kitakawa, M. A genome-wide approach to identify the genes involved in biofilm formation in E. coli. DNA Research. 14 (6), 237-246 (2008).
- Heinz, E., et al. Conserved features in the structure, mechanism, and biogenesis of the inverse autotransporter protein family. Genome Biology and Evolution. 8 (6), 1690-1705 (2016).
- Selkrig, J., et al. Discovery of an archetypal protein transport system in bacterial outer membranes. Nature Structural & Molecular Biology. 19 (5), 506-510 (2012).
- Stubenrauch, C., et al. Effective assembly of fimbriae in Escherichia coli depends on the translocation assembly module nanomachine. Nature Microbiology. 1 (7), 16064 (2016).
- Leo, J. C., Goldman, A. The immunoglobulin-binding Eib proteins from Escherichia coli are receptors for IgG Fc. Molecular Immunology. 46 (8-9), 1860-1866 (2009).
- Mühlenkamp, M., Oberhettinger, P., Leo, J. C., Linke, D., Schütz, M. S. Yersinia adhesin A (YadA) - Beauty & beast. International Journal of Medical Microbiology. 305 (2), 252-258 (2015).
- Studier, F. W., Moffatt, B. A. Use of bacteriophage T7 RNA polymerase to direct selective high-level expression of cloned genes. Journal of Molecular Biology. 189 (1), 113-130 (1986).
- Meuskens, I., Michalik, M., Chauhan, N., Linke, D., Leo, J. C. A new strain collection for improved expression of outer membrane proteins. Frontiers in Cellular and Infection Microbiology. 7, 464 (2017).
- Cherepanov, P. P., Wackernagel, W. Gene disruption in Escherichia coli: TcR and KmR cassettes with the option of Flp-catalyzed excision of the antibiotic-resistance determinant. Gene. 158 (1), 9-14 (1995).
- Grosskinsky, U., et al. A conserved glycine residue of trimeric autotransporter domains plays a key role in Yersinia adhesin A autotransport. Journal of Bacteriology. 189 (24), 9011-9019 (2007).
- Leo, J. C., et al. The structure of E. coli IgG-binding protein D suggests a general model for bending and binding in trimeric autotransporter adhesins. Structure. 19 (7), 1021-1030 (1993).
- Bertani, G. Studies on lysogenesis. I. The mode of phage liberation by lysogenic Escherichia coli. Journal of Bacteriology. 62 (3), 293-300 (1951).
- Hanahan, D. Studies on transformation of Escherichia coli with plasmids. Journal of Molecular Biology. 166 (4), 557-580 (1983).
- Leo, J. C., Oberhettinger, P., Linke, D. Assessing the outer membrane insertion and folding of multimeric transmembrane β-barrel proteins. Methods in Molecular Biology. , 157-167 (2015).
- Mikula, K. M., Leo, J. C., Łyskowski, A., Kedracka-Krok, S., Pirog, A., Goldman, A. The translocation domain in trimeric autotransporter adhesins is necessary and sufficient for trimerization and autotransportation. Journal of Bacteriology. 194 (4), 827-838 (2012).
- Thomason, L. C., Costantino, N., Court, D. L., Ausubel, F. M. E. coli genome manipulation by P1 transduction. Current Protocols in Molecular Biology. , (2007).
- Franklin, N. C. Mutation in gal U gene of E. coli blocks phage P1 infection. Virology. 38 (1), 189-191 (1969).
- Jeong, H., et al. Genome sequences of Escherichia coli B strains REL606 and BL21(DE3). Journal of Molecular Biology. 394 (4), 644-652 (2009).
- Greene, E. C. DNA Sequence Alignment during Homologous Recombination. Journal of Biological Chemistry. 291 (22), 11572-11580 (2016).
- Watt, V. M., Ingles, C. J., Urdea, M. S., Rutter, W. J. Homology requirements for recombination in Escherichia coli. Proceedings of the National Academy of Sciences of the United States of America. 82 (14), 4768-4772 (1985).
- Ho, T. D., Waldor, M. K. Enterohemorrhagic Escherichia coli O157:H7 gal mutants are sensitive to bacteriophage P1 and defective in intestinal colonization. Infection and Immunity. 75 (4), 1661-1666 (2006).
- Ikeda, H., Tomizawa, J. I. Transducing fragments in generalized transduction by phage P1. I. Molecular origin of the fragments. Journal of Molecular Biology. 14 (1), 85-109 (1965).
- Murooka, Y., Harada, T. Expansion of the host range of coliphage P1 and gene transfer from enteric bacteria to other Gam-negative bacteria. Applied and Environmental Microbiology. 38 (4), 754-757 (1979).
- Goldberg, R. B., Bender, R. A., Streicher, S. L. Direct selection for P1-sensitive mutants of enteric bacteria. Journal of Bacteriology. 118 (3), 810-814 (1974).
- O'Connor, K. A., Zusman, D. R. Coliphage P1-mediated transduction of cloned DNA from Escherichia coli to Myxococcus xanthus: use for complementation and recombinational analyses. Journal of Bacteriology. 155 (1), 317-329 (1983).
Reimpressões e Permissões
Solicitar permissão para reutilizar o texto ou figuras deste artigo JoVE
Solicitar PermissãoThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Todos os direitos reservados