A subscription to JoVE is required to view this content. Sign in or start your free trial.
Method Article
הפקת ג'ין מחיקות ב- Escherichia coli מאת P1 התמרה חושית עם קלטות עמידות לאנטיביוטיקה ניתן לניתוח
In This Article
Summary
כאן אנו מציגים פרוטוקול עבור השימוש קיימות מגירות עמידות לאנטיביוטיקה מחיקה בונה כבסיס ליצירת מוטציות מחיקה זנים אחרים e. coli . מוטציות מחיקה כאלה יכולים להיות גייסה ונוסף מיקומה המקביל של זן נמען באמצעות התמרה חושית bacteriophage P1.
Abstract
בגישה הראשונה ללמוד את תפקוד ג'ין לא ידוע של חיידקים היא ליצור את מדהימה של הגן הזה. כאן, אנו מתארים את פרוטוקול חזקה ומהירה להעברת מחיקה מוטציות מהמאמץ Escherichia coli אחד למשנהו באמצעות bacteriophage P1 שימוש התמרה חושית מוכללת. שיטה זו דורשת כי המוטציה להיות ניתן לבחירה (למשל, בהתבסס על הפרעות גנטי באמצעות קלטת לאנטיביוטיקה הוספות). קלטות לאנטיביוטיקה כזה יכול להיות גייסה מכל זן התורם מוחדרים זן הנמען עניין במהירות, בקלות ליצור מוטציה מחיקה ג'ין. ניתן לעצב בקלטת לאנטיביוטיקה כדי לכלול flippase זיהוי אתרים המאפשרים הכריתה של הקלטת על ידי recombinase בייעודי לאתר כדי לייצר את מדהימה נקי עם רק צלקת ~ 100-בסיס-זוג-ארוך רצף הגנום. אנו מדגימים את הפרוטוקול על ידי לדפוק את הגן של טאמה קידוד גורם הרכבה מעורב autotransporter להן, לבחון את השפעת הזה מעלף על להן והתפקוד של שני trimeric autotransporter adhesins. למרות המחיקה הגן על ידי התמרה חושית P1 יש מגבלות משלו, קלות ומהירות של יישומה להגיע חלופה אטרקטיבית בשיטות אחרות של ג'ין מחיקה
Introduction
גישה ראשונה משותפת ללמוד את הפונקציה של הגן הוא ביצוע מעלף מוטגנזה מכוונת ולבחון את פנוטיפ וכתוצאה מכך. זה נקרא גם גנטיקה הפוכה. חיידק e. coli כבר סוס עבודה של ביולוגיה מולקולרית במשך 70 השנים האחרונות, או אז, בגלל הקלות של culturing שלה, שלה amenability מניפולציה גנטית1. פותחו מספר שיטות כדי לייצר ג'ין מחיקות ב e. coli, כולל סמן exchange מוטגנזה מכוונת2,3 , לאחרונה, recombineering באמצעות λ אדום או Rac ET מערכות4,5 , 6.
במערכת בשימוש נרחב, קידוד רצף של גנים יחידניים מוחלפים קלטת עמידות לאנטיביוטיקה זה אפשר להוציא אחר כך5,של כרומוזום7. רצפי קידוד מוחלפים, למשל קלטת ההתנגדות kanamycin (קאן), אשר מוקף flippase (FLP) זיהוי אתרי היעד (והשפלתם) משני הצדדים. האתרים והשפלתם מזוהים על-ידי recombinase FLP, אשר שמתווכת בייעודי לאתר רקומבינציה בין האתרים והשפלתם שמוביל המחיקה של קלטת קאן. בדרך זו, פעולת מחיקה מלאה של רצף קידוד של גן מסוים יכולה להיות מושגת, משאיר מאחור רק צלקת מינימלית רצף של בסיסי כ-100 זוגות (bp) (איור 1).
רק לפני כעשור, פותחה האוסף קאיו כביכול. זוהי ספרייה חיידקי בהתבסס על מעבדה סטנדרטיים זן K12 e. coli , שבו כמעט כל הגנים שאינם חיוניים נמחקו בנפרד על ידי λ רקומבינציה אדום7,8. בתוך אוסף זה כל שלשיבוטים רצף קידוד אחד הוחלפו קלטת התנגדות ניתן לניתוח קאן. האוסף קאיו הוכיחה להיות כלי שימושי עבור יישומים רבים9. יישום אחד כזה הוא הייצור של מחיקה מוטציות בזנים אחרים e. coli . יכול להיות גייסה בקלטת קאן שיבוט נתון מחיקה מאת בדרך כלל transducing bacteriophages, כגון P110,11,12,13,14. מניה phage שהוכנו בכזה מתח יכול לשמש לאחר מכן להדביק את הנמען e. coli זן של עניין, איפה בתדר נמוך אבל אמין קן אזור המכילים קלטת ניתן לשלב לתוך הגנום הנמען מאת רקומבינציה הומולוגית (איור 2). Transductants ניתן לבחור את הצמיחה על המדיום המכיל קאן. בעקבות זאת, אם הסרת הקלטת עמידות לאנטיביוטיקה היא הרצויה, recombinase FLP יכולות להיות מסופקות המתח transductant טרנס. לאחר ריפוי של פלסמיד המכיל FLP, אשר נושאת סמן ההתנגדות אמפיצילין (אמפר), שיבוטים קאן לבין מגבר רגיש מוקרנים במשך ולאחר הכריתה הנכון של הרצף פראי-סוג קידוד ובקלטת קאן אומתו המושבה PCR.
. הנה, פרוטוקול מפורט מוצג, המתארת את כל השלבים בהפקת זן e. coli בהתבסס על האסטרטגיה שפורטו לעיל את מדהימה. כדוגמה, הוא הפגין מחיקה של הגן בתמ א . תמ א מקודד חלבון β-חבית של הממברנה החיצונית היא חלק של הובלה, הרכבה מודול (TAM), אשר עוסקת להן autotransporter חלבונים מסוימים, pili15,16,17. זן זה מעלף שימש אז כדי לבחון את ההשפעה של המחיקה תמ א על להן של trimeric autotransporter שתי adhesins (תעש), adhesin Yersinia את ידה של אימונוגלובולין e. coli (Ig)-מחייב TAA EibD 18,19.
Protocol
1. זנים, פלסמידים
-
זני חיידקים
- השתמש e. coli זנים BW251135, JW4179 (BW25113 tamA::kan)7, BL21(DE3)20, BL21ΔABCF21. ראה טבלה של חומרים למידע נוסף.
-
Bacteriophages
- להשתמש את phage P1vir התמרה חושית כללי. לאחסן את phage כמו מניה נוזלי עם כמה טיפות של כלורופורם (ראה שלב 2.2). לקבלת מידע נוסף, ראה טבלה של חומרים.
-
פלסמידים
- להשתמש את פלסמידים הבאים בפרוטוקול זה: pCP2022, pIBA2-בלה23, ו pEibD1024. כמו שליטה פלסמידים, להשתמש pASK-IBA2 pET22b (ראה טבלה של חומרים).
-
תנאי הגידול
- הפצת חיידקים lysogeny מרק (LB) בינוני25 ברעידות נמרצת (180-200 סל ד) 37 ° C או 30 מעלות צלזיוס במקרה של BL21ΔABCF, זנים המכילים pCP20.
- לבצע פלסמיד ריפוי-43 מעלות צלזיוס.
- עבור מדיום מוצק, תוספת ליברות עם 1% אגר (w/v).
- עבור אגר העליון, תוספת ליברות עם-0.7% אגר ו- 10 מ"מ CaCl2 ו אוטוקלב המדיום. שימוש SOC בינוני ההתאוששות לאחר אלקטרופורציה26.
- השתמש הריכוזים הבאים על אנטיביוטיקה: μg/mL 100 אמפר ו 25 μg/mL עבור קאן.
2. הכנת Phage של Lysate
-
זיהום של המתח התורם
- לגדול המתח התורם JW4197 ב 5 מ ל LB בינוני בתוספת 10 מ מ CaCl2 , באופן אופציונלי עם קאן (25 µg/mL) צפיפות אופטית על 600 nm (OD600) של ~ 1.0. למדוד את הערך600 OD באמצעות ספקטרופוטומטרים.
- להפוך סדרת דילול מניות phage הקיים P1 המדיום LB: מומלץ דילולים הן בין 10-3 כדי 10-7.
- ΜL מיקס 200 של המתלה חיידקי, 100 μL לדילול phage נתון צינור צנטריפוגה 15 מ"ל או שווה ערך. להכין צינורות רבים כמו phage דילולים. דגירה הצינורות כעשרים דקות ב 37 ° C ללא רועד.
- הוסף ~ 3 מ"ל של אגר מותכת העליון (~ 50 ° C) בתוספת 10 מ"מ CaCl2 עד הצינורות, לערבב התוכן ביסודיות על ידי vortexing הצינורות זמן קצר, שופכים את תערובות על גבי לוחות LB prewarmed כדי להפוך אפילו שכבות.
- דגירה הלוחות בן לילה ב 37 º C.
-
הכנה lysate
- למחרת היום לבחור צלחת עם גידול confluent למחצה של phage הפלאק. על צלחת confluent למחצה, בערך חצי את פני השטח של הצלחת ברור (איור 3).
- לגרד את השכבה העליונה אגר מצלחת כזה באמצעות ללולאה חיסון או כלי דומה ולמקם את אגר העליון שפופרת צנטרפוגה. מוסיפים 1-2 מ ל LB טיפה של כלורופורם וב מערבולת הצינור נמרצות עבור מינימלית ~ 1 הוסף הכלורופורם בשכונה fume.
- Centrifuge את הצינור למשך 15 דקות ב 4000 x g או מהר יותר הצניפה אגר של תאים חיידקיים.
- להעביר את תגובת שיקוע צינור microcentrifuge טריים, הימנעות נושא מעל כל פסולת בגדר. להוסיף 2 טיפות של כלורופורם ולאחסן את lysate ב 4 – 10 ° C. הוסף כלורופורם בשכונה fume. אין להקפיא את phage lysate כמו זה יגרום לירידה משמעותית של מספר חלקיקים זיהומיות.
-
קביעת כייל את lysate
- לגדול BW25113 ב ק ג בתוספת 10 מ מ CaCl2 ב 37 ° C עד התרבות מגיע עם יתר600 של ~ 1.0.
- להכין סדרת דילול phage lysate ב ליברות (למשל, 10-6–10-9).
הערה: להיות זהירים כדי pipet הדגימה מהחלק העליון של lysate כדי למנוע העברת כלורופורם דילולים. - ΜL מיקס 200 של המתלה חיידקי, 100 μL לדילול phage נתון צינור צנטריפוגה 15 מ"ל או שווה ערך. להכין צינורות רבים כמו phage דילולים. דגירה הצינורות כעשרים דקות ב 37 ° C ללא רועד.
- הוסף ~ 3 מ"ל של אגר מותכת העליון (~ 50 ° C) בתוספת 10 מ"מ CaCl2 עד הצינורות, לערבב התוכן ביסודיות על ידי vortexing הצינורות זמן קצר, שופכים את התערובת על גבי לוחות LB prewarmed כדי להפוך אפילו שכבות.
- דגירה הלוחות בן לילה ב 37 º C.
- למחרת לספור את הפלאק (ברור אזורים המזרן חיידקי) עבור כל אחד מהלוחות ולחשב את כייל של lysate באמצעות הנוסחה הבאה:

דוגמה: על צלחות עם דילולים 10-7, 10-8ו 10-9, 231, 27, ויש 2 לוחות, בהתאמה. כפי μL 100 של כל דילול שימשו את הזיהום, ומכיוון כייל מבוטאת ויוצרים רובד יחידות (pfu) / mL, הגורם ציפוי הוא 10. מספרים אלו מוזנות לנוסחה: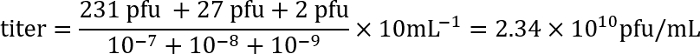
לכן, כייל הוא כ 2 x 1010 phage זיהומיות חלקיקים/mL.
3. התמרה חושית P1
-
הכנת התאים הנמען
- לגדול המתח הנמען BL21ΔABCF ב ק ג בתוספת צפיפות אופטית-600 nm (OD600) של ~ 1.0. להשתמש ספקטרופוטומטרים כדי למדוד את הערך600 OD.
- חישוב נפח phage lysate צורך להשיג את ריבוי של זיהום (MOI) ערך של 0.5. כדי לחשב את MOI, להעריך את מספר החיידקים בהתבסס על ה OD600 של התרבות. נניח כי ערך600 OD של 1.0 מקביל ~ 109 cfu/mL.... לחשב את אמצעי האחסון הדרושים בהתאם את כייל הידוע של lysate.
דוגמה (מבוסס על כייל החישוב בדוגמה לאחר שלב 2.3.6):
- מוסיפים CaCl2 לתרבות זן הנמען עד 10 מ"מ ומערבבים. קח 1 מ"ל של התרבות עבור התמרה חושית.
-
ביצוע התמרה חושית
- להוסיף נפח מתאים של phage lysate 1 מ"ל של תרבות הנמען (כולל CaCl2 -10 מ מ) כפי שמחושבת בשלב 3.1.2 ומערבבים בעדינות.
הערה: יש להיזהר pipette המדגם מהחלק העליון של lysate כדי למנוע העברה של כלורופורם לתערובת. - דגירה באופן סטטי את התערובת למשך 20 דקות ב 37 º C.
- לעצור את הזיהום על-ידי הוספת סודיום ציטרט, pH 5.5, 100 מ מ.
- Centrifuge החיידק (5,000 x g למשך 2 דקות), הסר את תגובת שיקוע, לאחר מכן resuspend אותם ב- 1 מ ל LB טרי בתוספת 100 מ מ סודיום ציטרט, pH 5.5.
- לשטוף את התאים עוד פעמיים, כמו שלב 3.2.4 כדי להבטיח הסרת phages חינם וסידן.
- Resuspend החיידק ב 1 מ"ל של LB טרי בתוספת 100 מ מ סודיום ציטרט, pH 5.5. דגירה החיידק ב 30 ° C עבור h 1 ברעידות (> 100 סל"ד).
- לאסוף את החיידקים על ידי צנטריפוגה (5,000 x g למשך 2 דקות), resuspend אותם בμl ~ 100 של ליברות עם 100 מ מ סודיום ציטרט, pH 5.5.
- התפשטות החיידקים בצלחת LB בתוספת קאן ב 25 µg/mL ו 10 מ מ סודיום ציטרט, pH 5.5, ולגדול החיידק ב 30 ° C עד מושבות יופיעו (~ 24 שעות).
- להוסיף נפח מתאים של phage lysate 1 מ"ל של תרבות הנמען (כולל CaCl2 -10 מ מ) כפי שמחושבת בשלב 3.1.2 ומערבבים בעדינות.
-
בחירה של transductants
- ברגע מושבות גדלו על הצלחת הבחירה, restreak אותם על ק ג + קן על מושבות יחיד ו לגדול ב 30 מעלות צלזיוס עד יחיד מושבות מופיעים.
4. excising את הקלטת קאן
-
הטרנספורמציה עם רקומבינציה פלסמיד
- להפוך את electrocompetent זן עמיד קאן BL21ΔABCF.
- תגדל את זן מושבה בודדת ב- LB טריים (5 מ"ל) בתוספת קאן (25 µg/mL) ב 30 ° C עד ה OD600 ~0.5 – 0.7.
- מכאן ואילך, לבצע את הפעולות הבאות ב 4 ° C או על קרח. Centrifuge החיידק (5,000 x g 10 דקות) ולהסיר את תגובת שיקוע.
- לשטוף את צניפה תא עם קרח מים מזוקקים פעמיים על-ידי חזרה על השלב הקודם צנטריפוגה ו, לבסוף, resuspend תא גלולה ב 100 µL של גליצרול 10% קר כקרח.
- לקרר 1 מ"מ אלקטרופורציה וואקום על קרח.
- להוסיף עמוד 1 של פלסמיד דנ א (pCP20) התליה תא, לערבב בעדינות את המתלים, וכן להעביר אותו cuvette אלקטרופורציה מקורר.
- הגדר את electroporator 1.8 kV, electroporate התאים.
- הצלה טרנספורמציה תאים על-ידי הוספת 1 מ"ל של מדיום SOC וגדל אותם עבור 1 h ב- 30 ° C.
- צלחת 100 µL של התאים ב- LB + מגבר צלחות ולגדול התאים ב 30 מעלות צלזיוס למשך הלילה.
- להפוך את electrocompetent זן עמיד קאן BL21ΔABCF.
-
אינדוקציה של רקומבינציה
- מושבות פיק יחיד מ LB + Amp לוח, לחסן LB טריים תוך השמטת כל אנטיביוטיקה.
- לגדל את התאים בטמפרטורה שאינו מתירניות (43 ° C) לילה לזירוז הביטוי של FLP recombinase.
-
בחירת ציטוקינים-חלבונים רקומביננטיים
- להפוך דילולים טורי, צלחת µL 50 לדילול6 5-10 10 על צלחות לא בררניים, לגדל את זה בן לילה ב- 30 ° C.
5. אימות של המחיקה ג'ין
-
אימות של אובדן פלסמיד, רקומבינציה מוצלח
- מושבות יחיד רצף של הצלחות מוכן בשלב 4.3.1 על ליברות + קאן, ליברות + מגבר ו LB צלחות ללא אנטיביוטיקה, בצו זה. כדי לסייע ומבטא, השתמש ברשת המושבה (ראה 1 קובץ משלים). לגדל את הצלחות ב 30 ° C עד מושבות יופיעו (~ 24 שעות).
- לבחור שיבוטים 10 – 20 גדל על הצלחת לא בררניים אך נכשל לגדול באמצעי התקשורת סלקטיבית לצורך אימות נוסף.
-
אימות נוסף מאת המושבה PCR
- לבצע מושבה PCR עם תחל איגוף תמ א קידוד רצף (ראה טבלה של חומרים).
- להכין תערובת PCR מאסטר. מידת שילוב הדרוש תלוי מספר מושבות להיבדק (עיין בדוגמה טבלה 1). לערבב את ריאגנטים על קרח, מוסיפים את פולימראז אחרונה.
- לערבב את תערובת הבסיס של PCR ביסודיות, אז לוותר על 20 µL לתוך הצינורות של רצועת ה-PCR. לאסוף כמות קטנה של כל מושבה שיוקרנו באמצעות טיפ פיפטה סטרילי והוסף אותו לרכבת התחתית. זכור לכלול המתח הנמענים המקוריים עבור השוואה. באופן אופציונלי, כוללים גם המתח התורם.
- להפעיל את תגובת ה-PCR (ראו טבלה 2 התוכנית המשמשת).
- הכנת ג'ל agarose 1%.
- עבור 50 מ ל ג'ל, למדוד 0.5 גר' agarose ולהוסיף 50 מ של מאגר טה (40 מ מ טריס, חומצה אצטית 20 מ מ ו- 1 מ מ EDTA, pH 8.0). מחממים את התערובת בתנור מיקרוגל עד agarose כל יש התפרקה.
- ברגע agarose פיזר, מגניב הפתרון עד כ 50 ° C, ולאחר מכן להוסיף 5 μL של ה-DNA מכתים לצבוע. מערבבים היטב את הפתרון ויוצקים אותו לתוך מגש ג'ל בתוך תא הליהוק. הוסף שורה אחת או יותר של מסרקים היטב כדי שלא יהיו בארות מספיק עבור כל דגימות, כמו גם סמני גודל מולקולרי. לאפשר את הג'ל להגדיר למשך 30 דקות.
- לאחר סיום המרוץ PCR, להוסיף 4 µL של 6 x DNA הטעינה לצבוע כל דגימה. מקם את הג'ל בבית הבליעה אלקטרופורזה ולהוסיף טה מאגר עד הבארות מכוסים.
- החל µL 10-15 מ כל התגובה PCR לבאר, הג'ל. גם להוסיף 5 µL של סמן גודל מולקולרי. לאחר מכן הפעל את הג'ל במשך 30 דקות ב-75 V.
- לאחר סיום המרוץ, תמונה הג'ל באמצעות imager אור כחול.
6. טכניקות אחרות
-
ביטוי חלבון
הערה: הביטוי של בדיקת חלבונים (בלה ו- EibD) היו מתוארות בפרוטרוט במקום אחר23,24. זהו סיכום קצר של השלבים העיקריים.- להפוך BL21ΔABCF Δתמ א wהמדומה של פלסמידים הכרחי (pIBA2-בלה, pEibD10, פלסמידים התואם את הבקרה) ו בחר עבור transformants ליברות + כח.
- עבור הביטוי חלבון, לחסן 100 מ ל LB בינוני + מגבר עם 1 מ"ל של תרבות לילה של חיידקים טרנספורמציה, גדלים אלה ב 30 ° C עד שלב אמצע יומן, ובו בזמן, לגרום לייצור חלבון או anhydrotetracycline (תמורת 100 ננוגרם למ"ל) או איזופרופיל thiogalactoside (ב- 0.5 מ מ).
- לאחר 2 h האינדוקציה ב 30 מעלות צלזיוס, למדוד את עכירות של התרבויות ולאסוף של מספר תאים המתאימים 50 מ ל- OD600 = 1.0 מאת צריך שתוציאו את התרבויות למשך 15 דקות ב x 4,000 גרם. לשטוף את גלולה 1 x עם 10 מ מ HEPES, pH 7.4 ולאחר מכן גם לאחסן אותו ב-20 ° C או תהליך זה יותר כמו שלב 6.2.
-
הממברנה החיצונית החילוץ
הערה: הממברנה החיצונית החילוץ מתבצע כפי שתואר בפירוט על ידי ליאו. et al. 27. השלבים העיקריים מסוכמים להלן.- Resuspend בגדר תא ב- 1 מ ל 10 מ מ HEPES, pH 7.4, בתוספת 10 מ מ MgCl2 , MnCl2, ליזוזים (0.1 mg/mL) ו קמצוץ של DNase אני.
- Lyse התאים (למשל, באמצעות מכה חרוז).
- Centrifuge התאים זמן קצר (2 דקות ב 15,600 x g) כדי להסיר שאריות תאים ולעבור את תגובת שיקוע צינור טריים.
- Centrifuge התאים למשך 30 דקות ב- 16,000 x g, לאחר מכן, resuspend בגדר ב 400 μL של 1% sarcosine N-lauroyl ב- 10 מ מ HEPES, pH 7.4.
- דגירה התאים עם עצבנות למשך 30 דקות בטמפרטורת החדר, לאחר מכן, centrifuge אותם למשך 30 דקות כמפורט לעיל.
- תשטוף שקוף גלולה 1 x עם 200 μL 10 מ מ HEPES, pH 7.4, ואז resuspend אותו בμl 30 10 מ מ HEPES, להוסיף 10 μL של 4 x מרחביות-דף לדוגמה מאגר.
-
פעילות מבחני
הערה: לבצע מבחני מרחביות-דף ופעילות בלה, EibD כפי שמתואר28. השלבים העיקריים מסוכמים להלן. -
עמוד למען חברה דמוקרטית
- מחממים את הדגימות ב 50 מעלות צלזיוס במשך 5 דקות לפני והעמיסו אותם ג'ל לזיהוי, כדי להימנע denaturing את החלבונים.
- לאחר ההפרדה בדף-מרחביות, להעביר את החלבונים קרום difluoride (PVDF) polyvinylidene.
- לאחר המעבר, לחסום את הקרום עם 2% אבקת חלב רפרפתי ב- PBS.
-
בלה-קולגן רחוק-מערב חשופה
- לאחר חסימת, להוסיף קולגן סוג שאני מדולל במאגר חסימה ריכוז μg/מ"ל, דגירה קרום לשעה.
- לשטוף את קרום 2 x עם PBS + 0.05% Tween20 (PBS-T).
- הוסף את נוגדן ראשוני (monoclonal אנטי-קולגן COL-1) למוח, 1:2,000 מדולל במאגר חסימה.
- לאחר המקננת הקרום עבור 1 h, לשטוף 2 x כפי שהוזכר בשלב 6.3.2.2 ולאחר מכן להוסיף את הנוגדן משנית [עז העכבר אנטי איג-חזרת peroxidase (HRP) המספר המשלים], 1:10,000 מדולל במאגר חסימה.
- דגירה הקרום עבור 1 h, ולאחר מכן לשטוף אותה 2 x עם PBS-טי להוסיף המצע chemiluminescent משופרת של הקרום על פי הוראות היצרן, לזהות את הלהקה בעזרת מצלמה CCD.
-
אג EibD איגוד וזמינותו
- לאחר חסימת, להוסיף נוגדנים משניים (עז נגד ארנב HRP), מדולל 1:2,000 במאגר חסימה.
- דגירה הקרום עבור 1 h, ולאחר מכן לשטוף אותה 2 x עם PBS. לבצע זיהוי chemiluminescent כפי שהוזכר בשלב 6.3.2.5.
תוצאות
מהדור תמ א הנוק-אאוט של BL21ΔABCF:
האסטרטגיה שפורטו לעיל בעבר שימש כדי לייצר זן נגזרות של BL21(DE3), זן מעבדה סטנדרטי המשמש לייצור חלבון, אשר אופטימיזציה עבור ייצור החלבון הממברנה החיצונית, שנקרא BL21ΔABCF21. זן זה חסר ארבעה גנים קידוד ?...
Discussion
התמרה חושית P1 הוא שיטה מהירה, חזקה ואמינה ליצירת ג'ין מחיקות ב e. coli. זה מומחש פה transducing מוטציה מחיקה תמ א של זן התורם קאיו לנמען נגזר BL21. השלבים העיקריים בתהליך התמרה חושית הן את הייצור של transducing lysate, את התמרה חושית עצמה, הכריתה של קלטת ההתנגדות קאן, ואימות של עקום החוצה על ידי ה-PCR. בסך...
Disclosures
המחברים אין לחשוף.
Acknowledgements
קאיו אוסף זנים התקבלו מפרוייקט BioResource הלאומית (כוש, יפן): e. coli. אנו מודים החיבור דירק (המחלקה של החיים, אוניברסיטת אוסלו) על תמיכתו המתמשכת. עבודה זו מומן על ידי המועצה למחקר של חוקר צעיר נורבגיה גרנט 249793 (כדי ג'ק ג לאו).
Materials
| Name | Company | Catalog Number | Comments |
| Strains | |||
| E. coli BW25113 | NIG | ME6092 | Wild-type strain of Keio collection |
| E. coli BL21(DE3) | Merck | 69450-3 | Expression strain |
| E. coli BL21DABCF | Addgene | 102270 | Derived from BL21(DE3) |
| E. coli JW4179 | NIG | JW4179-KC | tamA deletion mutant |
| P1 vir | NIG | HR16 | Generally transducing bacteriophage |
| Plasmids | |||
| pCP20 | CGSC | 14177 | conditionally replicating plasmid with FLP |
| pASK-IBA2 | IBA GmbH | 2-1301-000 | expression vector |
| pEibD10 | N/A | N/A | for production of EibD; plasmid available on request |
| pET22b+ | Merck | 69744-3 | expression vector |
| pIBA2-YadA | N/A | N/A | for production of YadA; plasmid available on request |
| Chemicals | |||
| Acetic acid | ThermoFisher | 33209 | |
| Agar | BD Bacto | 214010 | |
| Agarose | Lonza | 50004 | |
| Ampicillin | Applichem | A0839 | |
| Anhydrotetracycline | Abcam | ab145350 | |
| anti-collagen type I antibody COL-1 | Sigma | C2456 | |
| Bovine collagen type I | Sigma | C9791 | |
| Calcium chloride | Merck | 102382 | |
| Chloroform | Merck | 102445 | |
| Di-sodium hydrogen phosphate | VWR | 28029 | |
| DNA dye | Thermo | S33102 | |
| DNA molecular size marker | New England BioLabs | N3232S | |
| DNase I | Sigma | DN25 | |
| dNTP mix | New England Biolabs | N0447 | |
| ECL HRP substrate | Advansta | K-12045 | |
| EDTA | Applichem | A2937 | |
| Glycerol | VWR | 24388 | |
| goat anti-mouse IgG-HRP | Santa Cruz | sc-2005 | |
| goat anti-rabbit IgG-HRP | Agrisera | AS10668 | |
| HEPES | VWR | 30487 | |
| Isopropyl thiogalactoside | VWR | 43714 | |
| Kanamycin | Applichem | A1493 | |
| Lysozyme | Applichem | A4972 | |
| Magnesium chloride | VWR | 25108 | |
| Manganese chloride | Sigma | 221279 | |
| N-lauroyl sarcosine | Sigma | L9150 | |
| Skim milk powder | Sigma | 70166 | |
| Sodium chloride | VWR | 27808 | |
| tamA forward primer | Invitrogen | N/A | Sequence 5'-GAAAAAAGG ATATTCAGGAGAAAATGTG-3' |
| tamA reverse primer | Invitrogen | N/A | Sequence 5'-TCATAATT CTGGCCCCAGACC-3' |
| Taq DNA polymerase | New England Biolabs | M0267 | |
| Tri-sodium citrate | Merck | 106448 | |
| Tryptone | VWR | 84610 | |
| Tween20 | Sigma | P1379 | |
| Yeast extract | Merck | 103753 | |
| Equipment | |||
| Agarose gel electrophoresis chamber | Hoefer | SUB13 | |
| Bead beater | Thermo | FP120A-115 | |
| CCD camera | Kodak | 4000R | |
| Electroporation cuvettes | Bio-Rad | 165-2089 | |
| Electroporation unit | Bio-Rad | 1652100 | |
| Gel imager | Nippon Genetics | GP-03LED | |
| Incubating shaker | Infors HT | Minitron | |
| Incubator | VWR | 390-0482 | |
| Microcentrifuge | Eppendorf | 5415D | |
| Microwave oven | Samsung | CM1099A | |
| PCR machine | Biometra | Tpersonal | |
| PCR strips | Axygen | PCR-0208-CP-C | |
| pH meter | Hanna Instruments | HI2211-01 | |
| PVDF membrane | ThermoFisher | 88518 | |
| SDS-PAG electrophoresis chamber | ThermoFisher | A25977 | |
| Tabletop centrifuge | Beckman Coulter | B06322 | |
| Vortex mixer | Scientific Industries | SI-0236 | |
| Water bath | GFL | D3006 | |
| Wet transfer unit | Hoefer | TE22 |
References
- Blount, Z. D. The unexhausted potential of E. coli. eLife. 4, e05826 (2015).
- Hamilton, C. M., Aldea, M., Washburn, B. K., Babitzke, P., Kushner, S. R. New method for generating deletions and gene replacements in Escherichia coli. Journal of Bacteriology. 171 (9), 4617-4622 (1989).
- Link, A. J., Phillips, D., Church, G. M. Methods for generating precise deletions and insertions in the genome of wild-type Escherichia coli: application to open reading frame characterization. Journal of Bacteriology. 179 (20), 6228-6237 (1997).
- Sawitzke, J. A., Thomason, L. C., Costantino, N., Bubunenko, M., Datta, S., Court, D. L. Recombineering: in vivo genetic engineering in E. coli, S. enterica, and beyond. Methods in Enzymology. 421, 171-199 (2007).
- Datsenko, K. A., Wanner, B. L. One-step inactivation of chromosomal genes in Escherichia coli K-12 using PCR products. Proceedings of the National Academy of Sciences of the United States of America. 97 (12), 6640-6645 (2000).
- Muyrers, J. P., Zhang, Y., Stewart, A. F. Techniques: Recombinogenic engineering--new options for cloning and manipulating DNA. Trends in Biochemical Sciences. 26 (5), 325-331 (2001).
- Baba, T., et al. Construction of Escherichia coli K-12 in-frame, single-gene knockout mutants: the Keio collection. Molecular Systems Biology. 2, (2006).
- Yamamoto, N., et al. Update on the Keio collection of Escherichia coli single-gene deletion mutants. Molecular Systems Biology. 5, 335 (2009).
- Baba, T., Huan, H. -. C., Datsenko, K., Wanner, B. L., Mori, H. The applications of systematic in-frame, single-gene knockout mutant collection of Escherichia coli K-12. Methods in Molecular Biology. 416, 183-194 (2008).
- Chattopadhyay, M. K., Tabor, C. W., Tabor, H. Polyamines are not required for aerobic growth of Escherichia coli: preparation of a strain with deletions in all of the genes for polyamine biosynthesis. Journal of Bacteriology. 191 (17), 5549-5552 (2009).
- Xie, X., Wong, W. W., Tang, Y. Improving simvastatin bioconversion in Escherichia coli by deletion of bioH. Metabolic Engineering. 9 (4), 379-386 (2007).
- Maeda, T., Sanchez-Torres, V., Wood, T. K. Escherichia coli hydrogenase 3 is a reversible enzyme possessing hydrogen uptake and synthesis activities. Applied Microbiology and Biotechnology. 76 (5), 1035-1042 (2007).
- Meehan, B. M., Landeta, C., Boyd, D., Beckwith, J. The disulfide bond formation pathway is essential for anaerobic growth of Escherichia coli. Journal of Bacteriology. 199 (16), (2017).
- Niba, E. T. E., Naka, Y., Nagase, M., Mori, H., Kitakawa, M. A genome-wide approach to identify the genes involved in biofilm formation in E. coli. DNA Research. 14 (6), 237-246 (2008).
- Heinz, E., et al. Conserved features in the structure, mechanism, and biogenesis of the inverse autotransporter protein family. Genome Biology and Evolution. 8 (6), 1690-1705 (2016).
- Selkrig, J., et al. Discovery of an archetypal protein transport system in bacterial outer membranes. Nature Structural & Molecular Biology. 19 (5), 506-510 (2012).
- Stubenrauch, C., et al. Effective assembly of fimbriae in Escherichia coli depends on the translocation assembly module nanomachine. Nature Microbiology. 1 (7), 16064 (2016).
- Leo, J. C., Goldman, A. The immunoglobulin-binding Eib proteins from Escherichia coli are receptors for IgG Fc. Molecular Immunology. 46 (8-9), 1860-1866 (2009).
- Mühlenkamp, M., Oberhettinger, P., Leo, J. C., Linke, D., Schütz, M. S. Yersinia adhesin A (YadA) - Beauty & beast. International Journal of Medical Microbiology. 305 (2), 252-258 (2015).
- Studier, F. W., Moffatt, B. A. Use of bacteriophage T7 RNA polymerase to direct selective high-level expression of cloned genes. Journal of Molecular Biology. 189 (1), 113-130 (1986).
- Meuskens, I., Michalik, M., Chauhan, N., Linke, D., Leo, J. C. A new strain collection for improved expression of outer membrane proteins. Frontiers in Cellular and Infection Microbiology. 7, 464 (2017).
- Cherepanov, P. P., Wackernagel, W. Gene disruption in Escherichia coli: TcR and KmR cassettes with the option of Flp-catalyzed excision of the antibiotic-resistance determinant. Gene. 158 (1), 9-14 (1995).
- Grosskinsky, U., et al. A conserved glycine residue of trimeric autotransporter domains plays a key role in Yersinia adhesin A autotransport. Journal of Bacteriology. 189 (24), 9011-9019 (2007).
- Leo, J. C., et al. The structure of E. coli IgG-binding protein D suggests a general model for bending and binding in trimeric autotransporter adhesins. Structure. 19 (7), 1021-1030 (1993).
- Bertani, G. Studies on lysogenesis. I. The mode of phage liberation by lysogenic Escherichia coli. Journal of Bacteriology. 62 (3), 293-300 (1951).
- Hanahan, D. Studies on transformation of Escherichia coli with plasmids. Journal of Molecular Biology. 166 (4), 557-580 (1983).
- Leo, J. C., Oberhettinger, P., Linke, D. Assessing the outer membrane insertion and folding of multimeric transmembrane β-barrel proteins. Methods in Molecular Biology. , 157-167 (2015).
- Mikula, K. M., Leo, J. C., Łyskowski, A., Kedracka-Krok, S., Pirog, A., Goldman, A. The translocation domain in trimeric autotransporter adhesins is necessary and sufficient for trimerization and autotransportation. Journal of Bacteriology. 194 (4), 827-838 (2012).
- Thomason, L. C., Costantino, N., Court, D. L., Ausubel, F. M. E. coli genome manipulation by P1 transduction. Current Protocols in Molecular Biology. , (2007).
- Franklin, N. C. Mutation in gal U gene of E. coli blocks phage P1 infection. Virology. 38 (1), 189-191 (1969).
- Jeong, H., et al. Genome sequences of Escherichia coli B strains REL606 and BL21(DE3). Journal of Molecular Biology. 394 (4), 644-652 (2009).
- Greene, E. C. DNA Sequence Alignment during Homologous Recombination. Journal of Biological Chemistry. 291 (22), 11572-11580 (2016).
- Watt, V. M., Ingles, C. J., Urdea, M. S., Rutter, W. J. Homology requirements for recombination in Escherichia coli. Proceedings of the National Academy of Sciences of the United States of America. 82 (14), 4768-4772 (1985).
- Ho, T. D., Waldor, M. K. Enterohemorrhagic Escherichia coli O157:H7 gal mutants are sensitive to bacteriophage P1 and defective in intestinal colonization. Infection and Immunity. 75 (4), 1661-1666 (2006).
- Ikeda, H., Tomizawa, J. I. Transducing fragments in generalized transduction by phage P1. I. Molecular origin of the fragments. Journal of Molecular Biology. 14 (1), 85-109 (1965).
- Murooka, Y., Harada, T. Expansion of the host range of coliphage P1 and gene transfer from enteric bacteria to other Gam-negative bacteria. Applied and Environmental Microbiology. 38 (4), 754-757 (1979).
- Goldberg, R. B., Bender, R. A., Streicher, S. L. Direct selection for P1-sensitive mutants of enteric bacteria. Journal of Bacteriology. 118 (3), 810-814 (1974).
- O'Connor, K. A., Zusman, D. R. Coliphage P1-mediated transduction of cloned DNA from Escherichia coli to Myxococcus xanthus: use for complementation and recombinational analyses. Journal of Bacteriology. 155 (1), 317-329 (1983).
Reprints and Permissions
Request permission to reuse the text or figures of this JoVE article
Request PermissionExplore More Articles
This article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. All rights reserved