Un abonnement à JoVE est nécessaire pour voir ce contenu. Connectez-vous ou commencez votre essai gratuit.
Method Article
Produisant des destructions de gène chez Escherichia coli par Transduction P1 avec des Cassettes de la résistance aux antibiotiques soumis à accises
Dans cet article
Résumé
Nous présentons ici un protocole pour l’utilisation des préexistants de résistance aux antibiotiques-cassette suppression des constructions comme base pour l’établissement des mutants de délétion dans autres souches d’e. coli . Ces mutations de délétion peuvent être mobilisées et insérées dans le locus correspondant d’une souche à l’aide de transduction bactériophage P1.
Résumé
Une première approche pour étudier la fonction du gène inconnu chez les bactéries est de créer un knock-out de ce gène. Nous décrivons ici un protocole robuste et rapide pour transférer des mutations de délétion des gènes d’une souche d’Escherichia coli à l’autre à l’aide de transduction généralisée avec le bactériophage P1. Cette méthode nécessite que la mutation soit sélectionnable (p. ex., issu des perturbations de gène à l’aide d’insertions de cassette antibiotique). Ces cassettes antibiotiques peuvent être mobilisés à partir d’une souche de donateur et introduit dans une souche réceptrice d’intérêt à rapidement et facilement génèrent un mutant de délétion du gène. La cassette d’antibiotiques peut être conçue pour inclure des sites de reconnaissance de flippase qui permettent à l’excision de la cassette par une recombinase spécifique au site pour produire un knock-out propre avec seulement une séquence de ~ 100-base-paire-longue cicatrice dans le génome. Nous démontrer le protocole en assommant le tamA gène codant pour un facteur d’assemblage impliqué dans la biogenèse autotransporter et tester l’effet de cette knock out sur la biogenèse et fonction de deux adhésines autotransporter trimère. Bien que la délétion du gène par transduction P1 a ses limites, la facilité et la rapidité de sa mise en œuvre font une alternative intéressante aux autres méthodes de suppression de gène.
Introduction
Une première approche commune pour étudier la fonction d’un gène doit effectuer knock out mutagenèse et observer le phénotype résultant. C’est aussi nommé génétique inverse. La bactérie e. coli a été le cheval de bataille de la biologie moléculaire pour les 70 dernières années ou ainsi, en raison de la facilité de sa culture et sa susceptibilité de manipulation génétique1. Plusieurs méthodes ont été développées pour produire des destructions de gène chez e. coli, y compris le marqueur échange mutagénèse2,3 et, plus récemment, recombineering utilisant le λ rouge ou Rac ET systèmes4,5 , 6.
Dans un système largement utilisé, les séquences codantes de gènes individuels sont remplacées par une cassette de résistance aux antibiotiques qui peut plus tard être excisée du chromosome5,7. Les séquences codantes sont remplacés, par exemple par une cassette de résistance kanamycine (Kan), qui est flanquée de sites de cible (FRT) de reconnaissance flippase (FLP) de chaque côté. Les sites FRT sont reconnus par la recombinase FLP, qui intervient dans la recombinaison site-spécifique entre les sites FRT conduisant à la suppression de la cassette de Kan. De cette façon, une suppression complète de la séquence codante d’un gène donné peut être atteint, laissant derrière lui qu’une suite de cicatrice minime d’environ 100 paires de bases (PB) (Figure 1).
Juste plus de dix ans, la collection dite de Keio a été développée. Il s’agit d’une bibliothèque bactérienne basée sur un standard de laboratoire souche d’e. coli K12, où presque tous les gènes non essentiels ont été individuellement supprimés par λ recombinaison rouge7,8. Les clones au sein de cette collection chaque ont une séquence codante remplacée avec une cassette de résistance Kan soumis à accises. La collection de Keio s’est avéré pour être un outil utile pour nombreuses applications9. Une telle application est la production de mutants de délétion dans autres souches d’e. coli . La cassette Kan d’un clone de suppression donné peut être mobilisée par transduction généralement les bactériophages, telles que P110,11,12,13,14. Un stock de phage préparé à partir d’une telle souche peut alors servir à infecter un destinataire la souche e. coli d’intérêt, où à une fréquence faible mais fiable la Kan région cassette contenant peut être incorporée dans le génome du destinataire par recombinaison homologue (Figure 2). Transductants peuvent être sélectionnés pour la croissance sur le milieu contenant du Kan. Suite à cela, si le retrait de la cassette de résistance aux antibiotiques est souhaitée, la recombinase FLP peut être fournie à la souche transduites en trans. Après durcissement du plasmide contenant du FLP, qui transporte un marqueur de résistance ampicilline (Amp), clones Kan et Amp sensibles sont testées pour, et l’excision correcte de la séquence codante de type sauvage et la cassette Kan sont vérifiées par PCR sur colonie.
Ici, un protocole détaillé est présenté, décrivant chacune des étapes dans la production d’un knock-out souche e. coli , basé sur la stratégie décrite ci-dessus. À titre d’exemple, une délétion du gène tamA est démontrée. tamA , code pour une protéine β-Canon de la membrane externe qui est une partie du Transport et de montage Module (TAM), qui est impliqué dans la biogenèse de certaines protéines autotransporter et pili15,16,17. Cette souche de knock out a ensuite été utilisée pour étudier l’effet de la suppression de tamA sur la biogenèse des deux adhésines autotransporter trimère (AVA), l’adhésine Yersinia YadA et l’e. coli immunoglobuline (Ig)-liaison TAA EibD 18,,19.
Protocole
1. les souches et les plasmides
-
Souches bactériennes
- Utilisation de l' e. coli souches BW251135, JW4179 (BW25113 tamA::kan)7, BL21 (DE3)20et21de la BL21ΔABCF. Voir Table des matières pour plus d’informations.
-
Bactériophages
- Utilisez le phage P1vir pour la transduction générale. Stocker le phage comme un stock liquid avec quelques gouttes de chloroforme (Voir l’étape 2.2). Pour plus d’informations, consultez la Table des matières.
-
Plasmides
- Utilisez les plasmides suivants dans ce protocole : pCP2022, pIBA2-bla bla23et24de la pEibD10. Comme les plasmides de contrôle, utilisez pASK-IBA2 et pET22b (voir la Table des matières).
-
Conditions de croissance
- Propager les bactéries dans un bouillon (LB) de lysogénie, à moyenne25 avec agitation vigoureuse (180 / 200 tr/min) à 37 ° C ou 30 ° C dans le cas de souches contenant pCP20 et BL21ΔABCF.
- Effectuer le plasmide un durcissement à 43 ° C.
- Pour un milieu solide, Supplément LB avec 1 % d’agar (p/v).
- Pour albums agar, Supplément LB avec 0,7 % d’agar et 10 mM CaCl2 et stériliser le milieu. Utilisez SOC moyenne pour la récupération après électroporation26.
- Utilisez les concentrations suivantes pour les antibiotiques : 100 µg/mL pour les Amp et 25 µg/mL pour kan.
2. préparer un lysat de phages
-
Infection de la souche de donateurs
- Cultiver la souche donneur JW4197 dans 5 mL de LB additionné avec 10 mM CaCl2 et éventuellement avec Kan (25 µg/mL) à une densité optique à 600 nm (OD600) de ~ 1.0. Mesurer la valeur de600 OD à l’aide d’un spectrophotomètre.
- Faire une série de dilution d’un stock de phage P1 existant dans le milieu LB : recommandés par dilutions sont entre 10-3 à 10-7.
- Mix 200 μl de la suspension bactérienne et 100 μL d’une dilution de phage donnée dans un tube à centrifuger de 15 mL ou l’équivalent. Préparer les tubes autant que les dilutions de phages. Incuber les tubes pendant 20 min à 37 ° C sans agitation.
- Ajouter ~ 3 mL de gélose haut fondu (~ 50 ° C) avec 10 mM CaCl2 aux tubes, homogénéiser le contenu au vortex les tubes peu de temps et verser les mélanges dans des assiettes LB préchauffées à faire des couches même.
- Incuber les boîtes pendant une nuit à 37 ° C.
-
Préparation de lysate
- Le jour suivant, choisissez une plaque avec une croissance semi confluente de plaques phage. Sur une plaque semi confluente, environ la moitié la superficie de la plaque est claire (Figure 3).
- Gratter la couche de gélose haut de comme une plaque à l’aide d’une boucle de l’inoculation ou un outil similaire et placer l’agar en haut de la page dans un tube à centrifuger. Ajouter 1 à 2 mL de LB et une goutte de chloroforme et vortex le tube vigoureusement pendant environ 1 min. ajouter le chloroforme sous une hotte.
- Centrifuger le tube pendant 15 min à 4 000 x g ou plus rapide pour granuler la gélose et les cellules bactériennes.
- Déplacez le surnageant dans un tube de microcentrifuge fraîches, évitant le report de tous les débris de la pastille. Ajouter 2 gouttes de chloroforme et de stocker le lysat 4 – 10 ° c. Ajouter chloroforme sous une hotte. Ne pas congeler le phage lysat car cela se traduira par une réduction significative du nombre de particules infectieuses.
-
Déterminer le titre lysat
- Pousser BW25113 dans LB additionné de 10 mM CaCl2 à 37 ° C jusqu'à ce que la culture atteigne une OD600 ~ 1.0.
- Préparer une série de dilution du phage lysat en LB (p. ex., 10-6-10-9).
Remarque : Veillez à pipette l’échantillon du haut du lysat pour éviter de transférer le chloroforme pour les dilutions. - Mix 200 μl de la suspension bactérienne et 100 μL d’une dilution de phage donnée dans un tube à centrifuger de 15 mL ou l’équivalent. Préparer les tubes autant que les dilutions de phages. Incuber les tubes pendant 20 min à 37 ° C sans agitation.
- Ajouter ~ 3 mL de gélose haut fondu (~ 50 ° C) avec 10 mM CaCl2 aux tubes, homogénéiser le contenu au vortex les tubes peu et versez le mélange dans des assiettes LB préchauffées à faire des couches même.
- Incuber les boîtes pendant une nuit à 37 ° C.
- Le lendemain, compter le nombre de plaques (régions claires dans le tapis bactérien) pour chaque plaque et calculer le titre du lysat en utilisant la formule suivante :

Exemple : Sur les plaques avec la dilution 10-7, 10-8et 10-9, il y a 231, 27 et 2 plaques, respectivement. 100 μl de chaque dilution est utilisé pour l’infection, et que le titre est exprimé en formant unités (pfu) / mL, le facteur de placage est 10. Ces chiffres sont entrés dans la formule :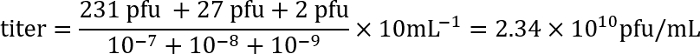
Ainsi, le titre est d’environ 2 x 1010 bactériophage infectieux particules/mL.
3. P1 Transduction
-
Préparer les cellules réceptrices
- Cultiver la souche réceptrice BL21ΔABCF dans LB complété à une densité optique à 600 nm (OD600) de ~ 1.0. Utilisez un spectrophotomètre pour mesurer la valeur de600 OD.
- Calculer le volume du phage lysat nécessaire à la réalisation d’une multiplicité de valeur d’infection (MOI) de 0.5. Pour calculer la MOI, estimer le nombre de bactéries basé sur l' OD600 de la culture. Supposons qu’une valeur de600 OD de 1,0 correspond à ~ 109 UFC/mL. Calculer le volume nécessaire basée sur le titre connu du lysate.
Exemple (basé sur le titre calculé dans l’exemple après l’étape 2.3.6) :
- Ajouter CaCl2 , à la culture de la souche réceptrice à 10 mM et mélanger. Prendre 1 mL de la culture pour la transduction.
-
Exécution de transduction
- Ajouter le volume approprié de lysat de phages à 1 mL de culture bénéficiaire (y compris CaCl2 à 10 mM) tel que calculé à l’étape 3.1.2 et mélanger doucement.
Remarque : Veillez à Pipeter l’échantillon du haut du lysat pour éviter de transférer le chloroforme au mélange. - Statiquement Incuber le mélange pendant 20 min à 37 ° C.
- Arrêtez l’infection en ajoutant le citrate de sodium, pH 5,5 à 100 mM.
- Centrifuger les bactéries (5 000 x g pendant 2 min) et retirez le surnageant, puis les remettre en suspension dans 1 mL de frais LB additionné de 100 mM du citrate de sodium, pH 5,5.
- Laver les cellules deux fois plus qu’à l’étape 3.2.4 pour assurer l’élimination des phages libres et en calcium.
- Remettre en suspension les bactéries dans 1 mL de frais LB additionné de 100 mM du citrate de sodium, pH 5,5. Incuber les bactéries à 30 ° C pendant 1 h avec agitation (> 100 tr/min).
- Récolter les germes par centrifugation (5 000 x g pendant 2 min) et les remettre en suspension dans ~ 100 μL de LB avec 100 mM du citrate de sodium, pH 5,5.
- Transmettre la bactérie sur une plaque LB additionné de Kan à 25 µg/mL et 10 mM du citrate de sodium, pH 5.5 et se développer les bactéries à 30 ° C jusqu'à ce que les colonies apparaissent (~ 24 h).
- Ajouter le volume approprié de lysat de phages à 1 mL de culture bénéficiaire (y compris CaCl2 à 10 mM) tel que calculé à l’étape 3.1.2 et mélanger doucement.
-
En sélectionnant les transductants
- Une fois que les colonies ont augmenté sur la plaque de sélection, leur restreak sur LB + Kan des colonies individuelles et poussent à 30 ° C jusqu'à ce que les colonies individuelles apparaissent.
4. excisant la cassette Kan
-
La transformation avec le plasmide de recombinaison
- Faire l’electrocompetent de souche BL21ΔABCF Kan-résistant.
- Cultiver la souche utilisée par une seule colonie en frais LB (5 mL) additionné de Kan (25 µg/mL) à 30 ° C jusqu'à ce que l' OD600 est ~0.5 – 0,7.
- Partir de là, réalisez les étapes suivantes à 4 ° C ou sur la glace. Centrifuger les bactéries (5 000 x g pendant 10 min) et éliminer le surnageant.
- Laver le culot cellulaire avec eau distillée glacée deux fois en répétant l’étape précédente de la centrifugation et, enfin, remettre en suspension le culot dans 100 µL de glacé 10 % de glycérol.
- Refroidir les cuves d’électroporation de 1 mm sur la glace.
- Ajoute la suspension cellulaire de 1 pg d’ADN (pCP20) plasmidique, mélanger la suspension doucement et transférer dans une cuvette d’électroporation refroidi.
- La valeur de l’électroporateur à 1,8 kV et electroporate les cellules.
- Sauver les cellules transformées en ajoutant 1 mL de milieu SOC et leur culture pendant 1 h à 30 ° C.
- Plaque de 100 µL de cellules sur LB + Amp plaques et cultiver les cellules à 30 ° C durant la nuit.
- Faire l’electrocompetent de souche BL21ΔABCF Kan-résistant.
-
Induction de la recombinaison
- Prélever des colonies unique du LB + Amp sur plaque et ensemencer les frais LB en omettant tous les antibiotiques.
- Cultiver les cellules à la température non permissif (43 ° C) pendant la nuit pour induire l’expression de la recombinase FLP.
-
Sélection des recombinants
- Faire des dilutions sériées et plaque 50 µL d’une dilution de6 105-10 sur plaques-bloquant sélectif et développez-le du jour au lendemain à 30 ° C.
5. vérification de la délétion de gène
-
Vérification de la perte de plasmides et de recombinaison réussie
- Strie unique colonies sur les plaques préparées à l’étape 4.3.1 sur LB, LB + Kan, Amp et LB sans antibiotiques, dans cet ordre. Pour vous aider dans les rayures, utiliser une grille de colonie (voir supplémentaire 1 fichier). Pousser les plaques à 30 ° C jusqu'à ce que les colonies apparaissent (~ 24 h).
- Sélectionner les clones de 10 – 20 qui ont cultivé sur la plaque non sélectif, mais n’ont pas poussé sur des milieux sélectifs pour vérification supplémentaire.
-
Vérification supplémentaire par PCR sur colonie
- Effectuer une colonie PCR avec des amorces flanquant le tamA séquence codante (voir Table des matières).
- Préparer un mélange maître de PCR. La quantité de mélange nécessaire dépend du nombre de colonies à tester (Voir l’exemple dans le tableau 1). Mélanger les réactifs sur la glace, ajouter en dernier la polymérase.
- Bien mélanger le mélange maître de PCR, puis diluer 20 µL dans des tubes d’une bande PCR. Prendre une petite quantité de chaque colonie à être projeté à l’aide d’un embout de la pipette stérile et l’ajouter à un tube. N’oubliez pas d’inclure la souche réceptrice originale à titre de comparaison. Éventuellement aussi inclure la souche de donateurs.
- Exécuter une réaction de PCR (voir le tableau 2 pour le programme utilisé).
- Préparer un gel d’agarose à 1 %.
- Pour un gel de 50 mL, mesurer 0,5 g d’agarose et ajouter 50 mL de tampon TAE (40 mM Tris, d’acide acétique 20 mM et 1 mM EDTA, pH 8,0). Faire chauffer le mélange dans un four à micro-ondes jusqu'à ce que tous les agarose a dissous.
- Une fois que l’agarose est dissout, refroidir la solution à environ 50 ° C et ensuite ajouter 5 μl d’ADN coloration teinture. Bien mélanger la solution et versez-la dans un bac de gel dans une chambre de coulée. Insérez une ou plusieurs rangées de peignes bien afin qu’il y a des puits suffisants pour tous les échantillons, ainsi que des marqueurs de taille moléculaire. Laisser le gel durant 30 min.
- Une fois que la série PCR est terminée, ajouter 4 µL de 6 x colorant de chargement de l’ADN à chaque échantillon. Placer le gel dans la chambre d’électrophorèse et ajouter le tampon TAE jusqu'à ce que les puits sont couverts.
- Appliquer 10-15 µL de chaque réaction de PCR dans un puits dans le gel. Également ajouter 5 µL de marqueur de poids moléculaire. Exécutez ensuite le gel pendant 30 min à 75 V.
- Une fois que la série est terminée, l’image du gel à l’aide d’un imageur de lumière bleue.
6. autres Techniques
-
Expression de la protéine
Remarque : L’expression des protéines de test (YadA et EibD) a été décrit en détail ailleurs23,24. Il s’agit d’un bref résumé des principales étapes.- Transformer BL21ΔABCF ΔtamA with les plasmides nécessaires (pIBA2-YadA et pEibD10 et les plasmides de contrôle correspondante), puis sélectionnez pour transformants sur LB + Amp.
- Pour l’expression de la protéine, ensemencer 100 mL de milieu LB + Amp avec 1 mL d’une culture d’une nuit ou plus de bactéries transformées et faire croître les à 30 ° C jusqu'à ce que phase logarithmique, date à laquelle, induire la production de protéine avec soit anhydrotétracycline (à 100 ng/mL) ou isopropylique thiogalactoside (à 0,5 mM).
- Après 2 h d’induction à 30 ° C, mesurer la turbidité des cultures et de recueillir un certain nombre de cases correspondant aux 50 mL à OD600 = 1.0 en centrifugeant les cultures à 4 000 x gpendant 15 minutes. Laver le culot 1 x 10 mm HEPES, pH 7,4 et ensuite soit conserver à-20 ° C ou processus il encore comme au point 6.2.
-
Extraction de la membrane externe
NOTE : Extraction de la membrane externe est effectuée comme décrit en détail par Leo et al. 27. les principales étapes sont résumées ci-dessous.- Remettre le culot dans 1 mL de 10 mM HEPES, pH 7,4, additionné de 10 mM MgCl2 et MnCl2, lysozyme (0,1 mg/mL) et une pincée de DNase I.
- Lyse des cellules (par exemple, à l’aide d’un batteur de perle).
- Centrifuger les cellules peu de temps (2 min à 15 600 x g) pour supprimer les débris cellulaires et déplacer le surnageant dans un tube frais.
- Centrifuger les cellules pendant 30 min à 16 000 x g, après quoi, resuspendre le culot dans 400 ml de 1 % N-lauroyl sarcosine dans 10 mM HEPES, pH 7,4.
- Incuber les cellules avec agitation pendant 30 min à température ambiante, après quoi, les Centrifuger pendant 30 min comme indiqué ci-dessus.
- Laver le translucide-pellet 1 x avec 200 μl de 10 mM HEPES, pH 7,4, puis il resuspendre dans 30 μL de 10 mM HEPES et ajoute 10 μL de 4 x tampon échantillon SDS-PAGE.
-
Tests d’activité
Remarque : Effectuez des essais SDS-PAGE et activité pour YadA et EibD comme décrit28. Les principales étapes sont résumées ci-dessous. -
SDS-PAGE
- Faire chauffer les échantillons à 50 ° C pendant 5 min avant de les charger sur un gel de polyacrylamide, pour éviter la dénaturation des protéines.
- Après la séparation en SDS-PAGE, transférer les protéines dans une membrane de polyvinylidène difluoride (PVDF).
- Après le transfert, bloquer la membrane avec la poudre de lait écrémé 2 % dans du PBS.
-
Tache d’extrême-ouest de YadA-collagène
- Après le blocage, ajout d’un type de collagène bovin que j’ai dilué dans un tampon bloquant à une concentration de 10 μg/mL et incuber la membrane pendant 1 h.
- Laver la membrane 2 x avec PBS + 0.05 % Tween20 (PBS-T).
- Ajoutez l’anticorps primaire (anticorps monoclonal anti-collagène COL-1) à la membrane, 1:2,000 dilué dans un tampon bloquant.
- Après incubation de la membrane pendant 1 h, laver 2 x comme indiqué dans l’étape 6.3.2.2, puis ajoutez l’anticorps secondaire [conjugué de peroxydase de raifort (HRP) goat anti-mouse IgG-raifort], dilué 1/10 000 dans un tampon bloquant.
- Incuber la membrane pendant 1 h, puis le laver 2 x avec PBS-T. Ajouter un substrat chimioluminescent renforcé à la membrane selon les instructions du fabricant et détecter la bande à l’aide d’une caméra CCD.
-
EibD-IgG essai de liaison
- Après le blocage, ajouter un anticorps secondaire (chèvre anti-lapin HRP), dilué 1:2,000 dans un tampon bloquant.
- Incuber la membrane pendant 1 h, puis le laver 2 x avec PBS. Effectuer la détection par chimiluminescence comme indiqué dans l’étape 6.3.2.5.
Résultats
Génération d’un tamA Knock out de BL21ΔABCF :
La stratégie décrite ci-dessus a déjà été utilisée pour produire une souche dérivée de BL21 (DE3), une souche de laboratoire standard utilisée pour la production de protéines, qui est optimisée pour la production de protéines de membrane externe et appelée BL21ΔABCF21. Cette souche n’a pas de quatre gènes codant pour des protéine...
Discussion
Transduction P1 est une méthode rapide, robuste et fiable pour générer des destructions de gène chez e. coli. Cela est démontré ici de transduction d’un mutant de délétion tamA d’une souche de donneur de Keio à un destinataire BL21 dérivés. Les grandes étapes dans le processus de transduction sont la production de la transduction lysate, la transduction lui-même, l’excision de la cassette de résistance Kan et la vérification de l’entrée défonçable par PCR. Au total, le processu...
Déclarations de divulgation
Les auteurs n’ont rien à divulguer.
Remerciements
Keio collection souches ont été obtenues par le projet National de BioResource (NIG, Japon) : e. coli. Nous remercions Dirk Linke (département de Biosciences, Université d’Oslo) pour son appui constant. Ce travail a été financé par le Conseil de recherche de bourse de jeune chercheur de Norvège 249793 (à Jack C. Leo).
matériels
| Name | Company | Catalog Number | Comments |
| Strains | |||
| E. coli BW25113 | NIG | ME6092 | Wild-type strain of Keio collection |
| E. coli BL21(DE3) | Merck | 69450-3 | Expression strain |
| E. coli BL21DABCF | Addgene | 102270 | Derived from BL21(DE3) |
| E. coli JW4179 | NIG | JW4179-KC | tamA deletion mutant |
| P1 vir | NIG | HR16 | Generally transducing bacteriophage |
| Plasmids | |||
| pCP20 | CGSC | 14177 | conditionally replicating plasmid with FLP |
| pASK-IBA2 | IBA GmbH | 2-1301-000 | expression vector |
| pEibD10 | N/A | N/A | for production of EibD; plasmid available on request |
| pET22b+ | Merck | 69744-3 | expression vector |
| pIBA2-YadA | N/A | N/A | for production of YadA; plasmid available on request |
| Chemicals | |||
| Acetic acid | ThermoFisher | 33209 | |
| Agar | BD Bacto | 214010 | |
| Agarose | Lonza | 50004 | |
| Ampicillin | Applichem | A0839 | |
| Anhydrotetracycline | Abcam | ab145350 | |
| anti-collagen type I antibody COL-1 | Sigma | C2456 | |
| Bovine collagen type I | Sigma | C9791 | |
| Calcium chloride | Merck | 102382 | |
| Chloroform | Merck | 102445 | |
| Di-sodium hydrogen phosphate | VWR | 28029 | |
| DNA dye | Thermo | S33102 | |
| DNA molecular size marker | New England BioLabs | N3232S | |
| DNase I | Sigma | DN25 | |
| dNTP mix | New England Biolabs | N0447 | |
| ECL HRP substrate | Advansta | K-12045 | |
| EDTA | Applichem | A2937 | |
| Glycerol | VWR | 24388 | |
| goat anti-mouse IgG-HRP | Santa Cruz | sc-2005 | |
| goat anti-rabbit IgG-HRP | Agrisera | AS10668 | |
| HEPES | VWR | 30487 | |
| Isopropyl thiogalactoside | VWR | 43714 | |
| Kanamycin | Applichem | A1493 | |
| Lysozyme | Applichem | A4972 | |
| Magnesium chloride | VWR | 25108 | |
| Manganese chloride | Sigma | 221279 | |
| N-lauroyl sarcosine | Sigma | L9150 | |
| Skim milk powder | Sigma | 70166 | |
| Sodium chloride | VWR | 27808 | |
| tamA forward primer | Invitrogen | N/A | Sequence 5'-GAAAAAAGG ATATTCAGGAGAAAATGTG-3' |
| tamA reverse primer | Invitrogen | N/A | Sequence 5'-TCATAATT CTGGCCCCAGACC-3' |
| Taq DNA polymerase | New England Biolabs | M0267 | |
| Tri-sodium citrate | Merck | 106448 | |
| Tryptone | VWR | 84610 | |
| Tween20 | Sigma | P1379 | |
| Yeast extract | Merck | 103753 | |
| Equipment | |||
| Agarose gel electrophoresis chamber | Hoefer | SUB13 | |
| Bead beater | Thermo | FP120A-115 | |
| CCD camera | Kodak | 4000R | |
| Electroporation cuvettes | Bio-Rad | 165-2089 | |
| Electroporation unit | Bio-Rad | 1652100 | |
| Gel imager | Nippon Genetics | GP-03LED | |
| Incubating shaker | Infors HT | Minitron | |
| Incubator | VWR | 390-0482 | |
| Microcentrifuge | Eppendorf | 5415D | |
| Microwave oven | Samsung | CM1099A | |
| PCR machine | Biometra | Tpersonal | |
| PCR strips | Axygen | PCR-0208-CP-C | |
| pH meter | Hanna Instruments | HI2211-01 | |
| PVDF membrane | ThermoFisher | 88518 | |
| SDS-PAG electrophoresis chamber | ThermoFisher | A25977 | |
| Tabletop centrifuge | Beckman Coulter | B06322 | |
| Vortex mixer | Scientific Industries | SI-0236 | |
| Water bath | GFL | D3006 | |
| Wet transfer unit | Hoefer | TE22 |
Références
- Blount, Z. D. The unexhausted potential of E. coli. eLife. 4, e05826 (2015).
- Hamilton, C. M., Aldea, M., Washburn, B. K., Babitzke, P., Kushner, S. R. New method for generating deletions and gene replacements in Escherichia coli. Journal of Bacteriology. 171 (9), 4617-4622 (1989).
- Link, A. J., Phillips, D., Church, G. M. Methods for generating precise deletions and insertions in the genome of wild-type Escherichia coli: application to open reading frame characterization. Journal of Bacteriology. 179 (20), 6228-6237 (1997).
- Sawitzke, J. A., Thomason, L. C., Costantino, N., Bubunenko, M., Datta, S., Court, D. L. Recombineering: in vivo genetic engineering in E. coli, S. enterica, and beyond. Methods in Enzymology. 421, 171-199 (2007).
- Datsenko, K. A., Wanner, B. L. One-step inactivation of chromosomal genes in Escherichia coli K-12 using PCR products. Proceedings of the National Academy of Sciences of the United States of America. 97 (12), 6640-6645 (2000).
- Muyrers, J. P., Zhang, Y., Stewart, A. F. Techniques: Recombinogenic engineering--new options for cloning and manipulating DNA. Trends in Biochemical Sciences. 26 (5), 325-331 (2001).
- Baba, T., et al. Construction of Escherichia coli K-12 in-frame, single-gene knockout mutants: the Keio collection. Molecular Systems Biology. 2, (2006).
- Yamamoto, N., et al. Update on the Keio collection of Escherichia coli single-gene deletion mutants. Molecular Systems Biology. 5, 335 (2009).
- Baba, T., Huan, H. -. C., Datsenko, K., Wanner, B. L., Mori, H. The applications of systematic in-frame, single-gene knockout mutant collection of Escherichia coli K-12. Methods in Molecular Biology. 416, 183-194 (2008).
- Chattopadhyay, M. K., Tabor, C. W., Tabor, H. Polyamines are not required for aerobic growth of Escherichia coli: preparation of a strain with deletions in all of the genes for polyamine biosynthesis. Journal of Bacteriology. 191 (17), 5549-5552 (2009).
- Xie, X., Wong, W. W., Tang, Y. Improving simvastatin bioconversion in Escherichia coli by deletion of bioH. Metabolic Engineering. 9 (4), 379-386 (2007).
- Maeda, T., Sanchez-Torres, V., Wood, T. K. Escherichia coli hydrogenase 3 is a reversible enzyme possessing hydrogen uptake and synthesis activities. Applied Microbiology and Biotechnology. 76 (5), 1035-1042 (2007).
- Meehan, B. M., Landeta, C., Boyd, D., Beckwith, J. The disulfide bond formation pathway is essential for anaerobic growth of Escherichia coli. Journal of Bacteriology. 199 (16), (2017).
- Niba, E. T. E., Naka, Y., Nagase, M., Mori, H., Kitakawa, M. A genome-wide approach to identify the genes involved in biofilm formation in E. coli. DNA Research. 14 (6), 237-246 (2008).
- Heinz, E., et al. Conserved features in the structure, mechanism, and biogenesis of the inverse autotransporter protein family. Genome Biology and Evolution. 8 (6), 1690-1705 (2016).
- Selkrig, J., et al. Discovery of an archetypal protein transport system in bacterial outer membranes. Nature Structural & Molecular Biology. 19 (5), 506-510 (2012).
- Stubenrauch, C., et al. Effective assembly of fimbriae in Escherichia coli depends on the translocation assembly module nanomachine. Nature Microbiology. 1 (7), 16064 (2016).
- Leo, J. C., Goldman, A. The immunoglobulin-binding Eib proteins from Escherichia coli are receptors for IgG Fc. Molecular Immunology. 46 (8-9), 1860-1866 (2009).
- Mühlenkamp, M., Oberhettinger, P., Leo, J. C., Linke, D., Schütz, M. S. Yersinia adhesin A (YadA) - Beauty & beast. International Journal of Medical Microbiology. 305 (2), 252-258 (2015).
- Studier, F. W., Moffatt, B. A. Use of bacteriophage T7 RNA polymerase to direct selective high-level expression of cloned genes. Journal of Molecular Biology. 189 (1), 113-130 (1986).
- Meuskens, I., Michalik, M., Chauhan, N., Linke, D., Leo, J. C. A new strain collection for improved expression of outer membrane proteins. Frontiers in Cellular and Infection Microbiology. 7, 464 (2017).
- Cherepanov, P. P., Wackernagel, W. Gene disruption in Escherichia coli: TcR and KmR cassettes with the option of Flp-catalyzed excision of the antibiotic-resistance determinant. Gene. 158 (1), 9-14 (1995).
- Grosskinsky, U., et al. A conserved glycine residue of trimeric autotransporter domains plays a key role in Yersinia adhesin A autotransport. Journal of Bacteriology. 189 (24), 9011-9019 (2007).
- Leo, J. C., et al. The structure of E. coli IgG-binding protein D suggests a general model for bending and binding in trimeric autotransporter adhesins. Structure. 19 (7), 1021-1030 (1993).
- Bertani, G. Studies on lysogenesis. I. The mode of phage liberation by lysogenic Escherichia coli. Journal of Bacteriology. 62 (3), 293-300 (1951).
- Hanahan, D. Studies on transformation of Escherichia coli with plasmids. Journal of Molecular Biology. 166 (4), 557-580 (1983).
- Leo, J. C., Oberhettinger, P., Linke, D. Assessing the outer membrane insertion and folding of multimeric transmembrane β-barrel proteins. Methods in Molecular Biology. , 157-167 (2015).
- Mikula, K. M., Leo, J. C., Łyskowski, A., Kedracka-Krok, S., Pirog, A., Goldman, A. The translocation domain in trimeric autotransporter adhesins is necessary and sufficient for trimerization and autotransportation. Journal of Bacteriology. 194 (4), 827-838 (2012).
- Thomason, L. C., Costantino, N., Court, D. L., Ausubel, F. M. E. coli genome manipulation by P1 transduction. Current Protocols in Molecular Biology. , (2007).
- Franklin, N. C. Mutation in gal U gene of E. coli blocks phage P1 infection. Virology. 38 (1), 189-191 (1969).
- Jeong, H., et al. Genome sequences of Escherichia coli B strains REL606 and BL21(DE3). Journal of Molecular Biology. 394 (4), 644-652 (2009).
- Greene, E. C. DNA Sequence Alignment during Homologous Recombination. Journal of Biological Chemistry. 291 (22), 11572-11580 (2016).
- Watt, V. M., Ingles, C. J., Urdea, M. S., Rutter, W. J. Homology requirements for recombination in Escherichia coli. Proceedings of the National Academy of Sciences of the United States of America. 82 (14), 4768-4772 (1985).
- Ho, T. D., Waldor, M. K. Enterohemorrhagic Escherichia coli O157:H7 gal mutants are sensitive to bacteriophage P1 and defective in intestinal colonization. Infection and Immunity. 75 (4), 1661-1666 (2006).
- Ikeda, H., Tomizawa, J. I. Transducing fragments in generalized transduction by phage P1. I. Molecular origin of the fragments. Journal of Molecular Biology. 14 (1), 85-109 (1965).
- Murooka, Y., Harada, T. Expansion of the host range of coliphage P1 and gene transfer from enteric bacteria to other Gam-negative bacteria. Applied and Environmental Microbiology. 38 (4), 754-757 (1979).
- Goldberg, R. B., Bender, R. A., Streicher, S. L. Direct selection for P1-sensitive mutants of enteric bacteria. Journal of Bacteriology. 118 (3), 810-814 (1974).
- O'Connor, K. A., Zusman, D. R. Coliphage P1-mediated transduction of cloned DNA from Escherichia coli to Myxococcus xanthus: use for complementation and recombinational analyses. Journal of Bacteriology. 155 (1), 317-329 (1983).
Réimpressions et Autorisations
Demande d’autorisation pour utiliser le texte ou les figures de cet article JoVE
Demande d’autorisationThis article has been published
Video Coming Soon