Se requiere una suscripción a JoVE para ver este contenido. Inicie sesión o comience su prueba gratuita.
Method Article
Producción de canceladuras del Gene en Escherichia coli por transducción de P1 con casetes de resistencia a los antibióticos
En este artículo
Resumen
Aquí presentamos un protocolo para el uso de preexistentes antibiótico resistencia-cassette borrado construcciones como base para la fabricación de mutantes de deleción en otras cepas de e. coli . Tales mutaciones de la canceladura pueden ser movilizados e inserta en el correspondiente locus de una cepa receptora mediante transducción de bacteriófago P1.
Resumen
Una primera aproximación al estudio de la función de un gen desconocido en bacterias es crear un knock-out de este gen. Aquí, describimos un protocolo robusto y rápido para la transferencia de mutaciones de la canceladura de genes de una cepa de Escherichia coli a otra mediante la transducción generalizada con el bacteriófago P1. Este método requiere que la mutación sea seleccionable (p. ej., basado en interrupciones del gen mediante inserciones de cassette antibiótico). Tales cintas antibióticas pueden movilizar de una cepa donante e introducen en una cepa receptora de interés rápidamente y generan fácilmente un mutante de canceladura del gene. El cassette de antibiótico puede diseñarse incluir flippase sitios de reconocimiento que permiten la supresión de la cinta por un recombinase específico para producir un knock-out limpiado con sólo una secuencia de ~ 100-base-par-larga cicatriz en el genoma. Demostrar el Protocolo por la anulación del factor conjunto implicado en la biogénesis autotransporter la codificación del gene de tamA y probar el efecto de esta eliminatoria en la biogénesis y la función de dos autotransporter trímero adhesinas. Aunque la canceladura del gene por transducción P1 tiene sus limitaciones, la facilidad y rapidez de su aplicación hacen una alternativa atractiva a otros métodos de canceladura del gene.
Introducción
Una primera aproximación común para estudiar la función de un gen es realizar mutagénesis de knock-out y observar el fenotipo resultante. Esto también se llama genética reversa. La bacteria e. coli ha sido el caballo de batalla de la biología molecular durante los últimos años 70 o así, debido a la facilidad de su cultivo y su receptividad a la manipulación genética1. Varios métodos han sido desarrollados para producir deleciones del gen en e. coli, incluyendo marcador cambio mutagénesis2,3 y, más recientemente, recombineering usando la λ rojo o ET Rac sistemas4,5 , 6.
En un sistema ampliamente utilizado, secuencias de codificación de genes individuales se sustituyen por una cinta de resistencia a los antibióticos que más adelante puede ser suprimida del cromosoma5,7. Las secuencias de codificación son sustituidas, por ejemplo por una cinta de resistencia kanamicina (Kan), que es flanqueada por los sitios de destino (FRT) flippase (FLP) reconocimiento a ambos lados. Los sitios FRT son reconocidos por el recombinase FLP, que media específica recombinación entre los sitios FRT conduce a la supresión de la cassette de Kan. De esta manera, se logra un borrado completo de la secuencia de codificación de un gen determinado, dejando sólo una secuencia mínima cicatriz de aproximadamente 100 pares de bases (PB) (figura 1).
A poco más de una década atrás, se desarrolló la llamada colección de Keio. Se trata de una biblioteca bacteriana basada en un estándar de laboratorio cepa K12 de e. coli , donde casi todos los genes no esenciales fueron borrados individualmente por λ recombinación rojo7,8. Los clones dentro de cada colección tienen una secuencia de codificación substituida con una cinta de resistencia los Kan. La colección de Keio ha demostrado para ser una herramienta útil para muchas aplicaciones9. Un tal uso es la producción de mutantes de deleción en otras cepas de e. coli . El cassette de Kan de un clon de eliminación dada puede ser movilizado por transducción generalmente de bacteriófagos, como P110,11,12,13,14. Un stock de fagos preparado a partir de tal tensión puede utilizarse para infectar a un recipiente e. coli cepa de interés, donde en una frecuencia baja pero confiable el Kan región que contiene el cassette puede ser incorporado en el genoma receptor por recombinación homóloga (Figura 2). Transductants pueden ser seleccionados para el crecimiento en el medio que contiene Kan. Después de esto, si se desea el retiro de la cinta de resistencia a los antibióticos, el recombinase FLP se puede suministrar a la cepa transductant en trans. Después de curar el plásmido que contiene el FLP, que lleva un marcador de resistencia a ampicilina (Amp), clones de Kan y sensible a Amp son evaluados para, y la supresión correcta de la secuencia de codificación de tipo salvaje y el cassette de Kan son verificados por PCR de Colonia.
Aquí, se presenta un protocolo detallado, describiendo cada uno de los pasos en la producción de un knock-out cepa de e. coli , basado en la estrategia que se ha señalado anteriormente. Por ejemplo, se demostró una canceladura del gene del tamA . tamA codifica una proteína de β-barril de membrana externa que es una parte del transporte y montaje módulo (TAM), que participa en la biogénesis de ciertas proteínas autotransporter y pili15,16,17. Esta cepa knock-out entonces fue utilizada para examinar el efecto de la supresión de tamA en la biogénesis de dos autotransporter trímero adhesinas (TAAs), la adhesina de Yersinia YadA y la inmunoglobulina (Ig) de la e. coli -vinculante EibD TAA 18,19.
Protocolo
1. las cepas y plásmidos
-
Cepas bacterianas
- Uso de la e. coli cepas BW251135, JW4179 (BW25113 tamA::kan)7, BL21(DE3)20y BL21ΔABCF21. Para más información ver la Tabla de materiales .
-
Bacteriófagos
- Utilice el fago P1vir para la transducción general. Almacenar el phage como un líquido con unas gotas de cloroformo (ver paso 2.2). Para obtener más información, vea Tabla de materiales.
-
Plásmidos
- Utilice el siguiente plásmidos en este protocolo: pCP2022, pIBA2-YadA23y pEibD1024. Plásmidos de control, utilizar IBA2 pASK y pET22b (véase Tabla de materiales).
-
Condiciones de crecimiento
- Propagar las bacterias en un medio25 de lisogenia caldo (LB) con vigoroso (180 – 200 rpm) a 37 ° C o 30 ° C en el caso de las cepas que contienen pCP20 y BL21ΔABCF.
- Realizar plásmido curado a 43 ° C.
- De un medio sólido, suplemento LB con agar al 1% (p/v).
- Para top agar, suplemento LB con 0,7% de agar y 10 mM de CaCl2 y autoclave el medio. Medio SOC de uso para la recuperación después de la electroporación26.
- Utilizar las siguientes concentraciones de antibióticos: 100 μg/mL de Amp y 25 μg/mL para el Kan.
2. preparación de un lisado de fagos
-
Infección de la cepa donante
- Crecer la cepa donante JW4197 en 5 mL de medio LB suplementada con 10 mM de CaCl2 y opcionalmente con Kan (25 μg/mL) a una densidad óptica a 600 nm (OD600) de ~ 1.0. Mide el valor de OD600 usando un espectrofotómetro.
- Hacer una serie de diluciones de un stock de fagos P1 existente en el medio LB: recomienda diluciones son entre 10-3 a 10-7.
- Mezcla 200 μL de la suspensión bacteriana y 100 μL de una dilución de phage dado en un tubo de centrífuga de 15 mL o equivalente. Preparar tantos tubos como diluciones de fago. Incubar los tubos durante 20 min a 37 ° C sin agitación.
- Añadir 3 mL de agar superior fundida (~ 50 ° C) suplementado con 10 mM de CaCl2 en los tubos, homogeneizar el contenido con un vórtex los tubos poco y vierta la mezcla en placas LB precalentadas para hacer capas incluso.
- Incubar las placas durante la noche a 37 ° C.
-
Preparación de lisado
- Al día siguiente elegir un plato con un semi-confluente crecimiento de placas de fagos. En un plato semi confluente, aproximadamente la mitad la superficie de la placa es evidente (figura 3).
- Raspar la capa de agar superior de dicha placa con un asa de inoculación o una herramienta similar y colocar el agar superior en un tubo de centrífuga. Añadir 1-2 mL de LB y una gota de cloroformo y agitar el tubo vigorosamente durante ~ 1 min agregar el cloroformo en una campana de humos.
- Centrifugar el tubo durante 15 min a 4.000 x g o más rápido para que sedimenten las células bacterianas y agar.
- Pasar el sobrenadante a un tubo de microcentrífuga fresco, evitando transportar cualquier residuo de la pastilla. Añadir 2 gotas de cloroformo y almacenar el lisado a 4 – 10 ° C. Añadir cloroformo en una campana de humos. No congele el phage lisado, esto resultará en una reducción significativa del número de partículas infecciosas.
-
Determinar el título del lisado
- Crecer BW25113 en LB suplementado con 10 mM de CaCl2 a 37 ° C hasta que el cultivo alcanza un OD600 de ~ 1.0.
- Preparar una serie de diluciones de los fagos lisado en LB (p. ej., 10-6-10-9).
Nota: Tenga cuidado al pipetear la muestra desde la parte superior del lisado para evitar transferencia de cloroformo a las diluciones. - Mezcla 200 μL de la suspensión bacteriana y 100 μL de una dilución de phage dado en un tubo de centrífuga de 15 mL o equivalente. Preparar tantos tubos como diluciones de fago. Incubar los tubos durante 20 min a 37 ° C sin agitación.
- Añadir 3 mL de agar superior fundida (~ 50 ° C) suplementado con 10 mM de CaCl2 en los tubos, homogeneizar el contenido con un vórtex los tubos poco y vierta la mezcla en placas LB precalentadas para hacer capas incluso.
- Incubar las placas durante la noche a 37 ° C.
- Al día siguiente cuenta el número de placas (regiones claras en la placa bacteriana) para cada placa y calcular el título del lisado mediante la fórmula siguiente:

Ejemplo: En las placas de las diluciones 10-710-8y 10-9, hay 231, 27 y 2 placas, respectivamente. 100 μL de cada dilución se utilizó para la infección, y porque el título se expresa como placa formando unidades (pfu) / mL, el factor de la galjanoplastia es 10. Estos números se introducen en la fórmula: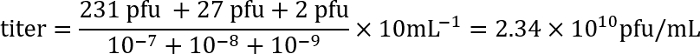
Así, el título es aproximadamente 2 x 1010 infecciosas fagos partículas/mL.
3. P1 transducción
-
Preparación de las células del receptoras
- Crecer la cepa receptora BL21ΔABCF en LB complementado a una densidad óptica a 600 nm (OD600) de ~ 1.0. Use un espectrofotómetro para medir el valor de OD600 .
- Calcular el volumen del lisado de fagos para alcanzar una multiplicidad de valor de infección (MOI) de 0,5. Para calcular el MOI, estimar el número de bacterias basado en OD600 de la cultura. Asume que un valor de600 OD de 1.0 corresponde a ~ 109 UFC/mL. Calcular el volumen requerido basado en el conocido título del lisado.
Ejemplo (basado en la concentración calculada en el ejemplo después paso 2.3.6):
- Añadir CaCl2 a la cultura receptora cepa a 10 mM y mezclar. Tomar 1 mL del cultivo para la transducción.
-
Realizar la transducción de señales
- Añadir el volumen adecuado de lisado de fagos a 1 mL de la cultura receptora (incluyendo CaCl2 a 10 mM) calculado en el paso 3.1.2 y mezclar suavemente.
Nota: Tenga cuidado al pipetear la muestra desde la parte superior del lisado para evitar transferencia de cloroformo a la mezcla. - Estáticamente, incubar la mezcla por 20 min a 37 ° C.
- Detener la infección mediante la adición de citrato de sodio, pH 5.5, a 100 mM.
- Centrifugue las bacterias (5.000 x g durante 2 min) Quite el sobrenadante y resuspender en 1 mL de LB fresco suplementado con citrato de sodio 100 mM, pH 5,5.
- Lavan las células dos veces más que en el paso 3.2.4 para la eliminación de calcio y phages gratis.
- Resuspender las bacterias en 1 mL de LB fresco suplementado con citrato de sodio 100 mM, pH 5,5. Incubar las bacterias a 30 ° C por 1 h con agitación (> 100 rpm).
- Recoger las bacterias por centrifugación (5.000 x g durante 2 min) y resuspender en ~ 100 μL de LB con citrato de sodio 100 mM, pH 5,5.
- Trasmitir las bacterias en una placa LB suplementado con Kan en 25 μg/mL y citrato de sodio 10 mM, pH 5,5 y crecer las bacterias a 30 ° C hasta que las colonias (~ 24 h).
- Añadir el volumen adecuado de lisado de fagos a 1 mL de la cultura receptora (incluyendo CaCl2 a 10 mM) calculado en el paso 3.1.2 y mezclar suavemente.
-
Selección de las transductants
- Una vez que las colonias han crecido en la placa de selección, les restreak LB + Kan solo colonias y crecen a 30 ° C hasta que las colonias solo aparecen.
4. suprimir el cassette de Kan
-
La transformación con el plásmido de recombinación
- Realizar el espectro de tensión de BL21ΔABCF Kan-resistente.
- Crece la tensión de una sola Colonia en fresco LB (5 mL) complementado con Kan (25 μg/mL) a 30 ° C hasta que el OD600 ~0.5 – 0,7.
- De aquí en adelante, llevar a cabo los siguientes pasos a 4 ° C o en hielo. Centrifugue las bacterias (5.000 x g por 10 min) y eliminar el sobrenadante.
- Lavar el precipitado con agua destilada helada de células dos veces repitiendo el paso anterior de centrifugación y, finalmente, Resuspender el precipitado de células en 100 μl de helada glicerol al 10%.
- Enfriar cubetas de electroporación de 1 mm en el hielo.
- Añadir 1 pg de ADN (pCP20) plásmido a la suspensión de células, mezclar suavemente la suspensión y transferir a una cubeta de electroporación refrescado.
- Establecer la electroporator a 1,8 kV y electroporate las células.
- Rescatar las células transformadas agregando 1 mL de medio SOC y creciendo por 1 h a 30 ° C.
- Placa de 100 μl de las células en LB + Amp placas y crecen las células en 30 ° C durante la noche.
- Realizar el espectro de tensión de BL21ΔABCF Kan-resistente.
-
Inducción de la recombinación
- Colonias solo Pick LB + amplificador de la placa e inoculan fresco LB omitiendo todos los antibióticos.
- Crecen las células a la temperatura no permisiva (43 ° C) durante la noche para inducir la expresión de recombinase FLP.
-
Selección de recombinantes
- Hacer diluciones seriadas y 50 μl de una dilución de6 5-10 10 en placas no selectivo de la placa y crecer durante la noche a 30 ° C.
5. verificación de la canceladura del Gene
-
Verificación de la pérdida del plásmido y recombinación exitosa
- Racha solo las colonias de las placas de preparado en el paso 4.3.1 en Kan, LB, LB + Amp y las placas LB sin antibióticos, en este orden. Para ayudar en las rayas, utilice una rejilla de Colonia (véase la archivo adicional 1). Crecen las placas a 30 ° C hasta que las colonias (~ 24 h).
- Recoger 10-20 clones que han crecido en la placa no selectivo pero no crecen en medios selectivos para mayor verificación.
-
Verificación adicional de colony PCR
- Realizar una colonia PCR con cebadores que flanquean el tamA secuencia de codificación (véase Tabla de materiales).
- Preparar una mezcla PCR master. La cantidad de mezcla necesitada depende del número de colonias a ser probado (ver ejemplo en tabla 1). Mezclar los reactivos en el hielo, añadir la polimerasa de la última.
- Homogeneizar la mezcla PCR master y dispensar 20 μl en tubos de una tira PCR. Recoger una pequeña cantidad de cada colonia para ser proyectada utilizando una pipeta estéril y añadir a un tubo. No olvide incluir la cepa receptora original para la comparación. Opcionalmente, también incluyen la cepa donante.
- Ejecutar una reacción de PCR (ver tabla 2 para el programa utilizado).
- Preparar un gel de agarosa al 1%.
- Para un gel de 50 mL, medir de 0.5 g de agarosa y añadir 50 mL de buffer TAE (40 mM Tris, ácido acético al 20 mM y 1 mM EDTA, pH 8.0). Calienta la mezcla en el microondas hasta que se disuelva la agarosa todos.
- Una vez que la agarosa se disuelva, enfriar la solución a aproximadamente 50 ° C y luego añadir 5 μL de ADN tinción colorante. Mezcle bien la solución y verterla en una bandeja de gel en una cámara de fundición. Introduzca una o más filas de peines bien hay pozos suficientes para todas las muestras, así como marcadores de tamaño molecular. Permita que el gel para 30 minutos.
- Una vez finalizado el plazo de la polimerización en cadena, añadir 4 μL de 6 x tinte carga de ADN a cada muestra. Colocar el gel en la cámara de electroforesis y añadir Tampón TAE hasta que los pozos están cubiertos.
- Aplicar 10-15 μl de cada reacción de PCR a un pozo en el gel. También añadir 5 μl del marcador de tamaño molecular. A continuación, ejecute el gel durante 30 minutos a 75 V.
- Una vez finalizado el plazo, el gel utilizando a un sensor de luz azul de la imagen.
6. otras técnicas
-
Expresión de la proteína
Nota: La expresión de proteínas de la prueba (YadA y EibD) ha sido descrito en detalle en otra parte23,24. Este es un breve resumen de los pasos principales.- Transformar BL21ΔABCF ΔtamA with la plásmidos necesaria (YadA pIBA2 y pEibD10 y la plásmidos de control correspondiente) y seleccione transformantes en LB + Amp.
- Para la expresión de la proteína, inocular 100 mL de medio LB + Amp con 1 mL de un cultivo durante la noche de bacterias transformadas y crecer a 30 ° C hasta la fase de registro medio, momento en el que, inducir la producción de proteína con cualquiera de los dos anhydrotetracycline (a 100 ng/mL) o thiogalactoside de isopropilo (a 0,5 mM).
- Después de 2 h de la inducción a 30 ° C, medir la turbidez de los cultivos y recolectar un número de células correspondientes a 50 mL en OD600 = 1.0 por centrifugación de los cultivos durante 15 min a 4.000 x g. Lavar el sedimento 1 x con 10 mM HEPES, pH 7,4 y luego o bien almacenarlo a-20 ° C o proceso adicional como en el paso 6.2.
-
Extracción de la membrana externa
Nota: La extracción de la membrana externa se realiza como se describe en detalle por León et al. 27. a continuación se resumen los pasos principales.- Resuspender el precipitado de células en 1 mL de 10 mM HEPES, pH 7,4, complementado con 10 mM MgCl2 MnCl2, lisozima (0,1 mg/mL) y una pizca de ADNasa I.
- Lyse las células (e.g., usando un batidor de grano).
- Centrifugar las células poco (2 min a 15.600 x g) para eliminar restos celulares y trasladar el sobrenadante a un tubo nuevo.
- Centrifugar las células durante 30 min a 16.000 x g, después de lo cual, Resuspender el pellet en 400 μL de 1% N-lauroil sarcosina en 10 mM HEPES, pH 7,4.
- Incubar las células con agitación durante 30 min a temperatura ambiente, después de lo cual, centrifugar por 30 min como el anterior.
- Lave el translúcido pellet 1 x con 200 μL de 10 mM HEPES, pH 7,4, resuspender en 30 μL de 10 mM HEPES y añade 10 μL de tampón de SDS-PAGE 4.
-
Ensayos de actividad
Nota: Realizar los análisis de SDS-PAGE y actividad para YadA y EibD descrito28. A continuación se resumen los pasos principales. -
SDS-PAGE
- Calentar las muestras a 50 ° C durante 5 minutos antes de cargar en un gel de poliacrilamida, para evitar la desnaturalización de las proteínas.
- Después de la separación en SDS-PAGE, transferir las proteínas a una membrana (PVDF) de difluoruro de polivinilideno.
- Después de la transferencia, bloquear la membrana con 2% de polvo de leche desnatada en PBS.
-
YadA-colágeno lejos-de western blot
- Después de los bloqueos, añadir colágeno bovino tipo que diluido en solución amortiguadora de bloqueo a una concentración de 10 μg/mL e incubar la membrana para 1 h.
- Lavar la membrana 2 x con PBS + 0.05% Tween20 (PBS-T).
- Añadir el anticuerpo primario (monoclonal anti colágeno COL-1) a la membrana, 1:2,000 diluido en solución amortiguadora de bloqueo.
- Después de incubar la membrana durante 1 hora, lavar 2 x como se menciona en el paso 6.3.2.2 y luego añadir el anticuerpo secundario [cabra anti-ratón IgG conjugado-rábano peroxidasa (HRP)], diluido 1: 10,000 en solución amortiguadora de bloqueo.
- Incubar la membrana durante 1 hora, luego lavar x 2 con PBS-T. Añadir un sustrato quimioluminiscente mejorado a la membrana según las instrucciones del fabricante y detectar la banda usando una cámara CCD.
-
IgG por EibD ensayo obligatorio
- Después de los bloqueos, añadir un anticuerpo secundario (cabra anti-conejo HRP), diluido 1:2,000 en solución amortiguadora de bloqueo.
- Incubar la membrana durante 1 hora, luego lavar x 2 con PBS. Realizar la detección quimioluminiscente como se menciona en el paso 6.3.2.5.
Resultados
Generación de un tamA Knock-out de BL21ΔABCF:
La estrategia mencionada anteriormente se ha utilizado para producir una cepa derivada de BL21(DE3), una cepa de laboratorio estándar utilizada para la producción de la proteína, que es optimizada para producción de proteínas de membrana externa y llamada BL21ΔABCF21. Esta cepa carece de cuatro genes que codifican para proteínas de membrana exte...
Discusión
Transducción de P1 es un método rápido, robusto y fiable para la generación de canceladuras del gene de e. coli. Esto se demuestra aquí por transducción de un mutante de eliminación de tamA de una cepa de Keio donante a un beneficiario derivado BL21. Las principales etapas en el proceso de transducción de señales son la producción de la transducción de lisado, la transducción de sí mismo, la supresión de los cassettes de resistencia de Kan y la verificación del knock-out por PCR. En total...
Divulgaciones
Los autores no tienen nada que revelar.
Agradecimientos
Se obtuvieron cepas de colección de Keio del proyecto nacional de BioResource (NIG, Japón): e. coli. Agradecemos a Dirk Linke (Departamento de ciencias biológicas, Universidad de Oslo) por su continuo apoyo. Este trabajo fue financiado por el Consejo de investigación de Noruega joven investigador subsidio 249793 (Jack C. Leo).
Materiales
| Name | Company | Catalog Number | Comments |
| Strains | |||
| E. coli BW25113 | NIG | ME6092 | Wild-type strain of Keio collection |
| E. coli BL21(DE3) | Merck | 69450-3 | Expression strain |
| E. coli BL21DABCF | Addgene | 102270 | Derived from BL21(DE3) |
| E. coli JW4179 | NIG | JW4179-KC | tamA deletion mutant |
| P1 vir | NIG | HR16 | Generally transducing bacteriophage |
| Plasmids | |||
| pCP20 | CGSC | 14177 | conditionally replicating plasmid with FLP |
| pASK-IBA2 | IBA GmbH | 2-1301-000 | expression vector |
| pEibD10 | N/A | N/A | for production of EibD; plasmid available on request |
| pET22b+ | Merck | 69744-3 | expression vector |
| pIBA2-YadA | N/A | N/A | for production of YadA; plasmid available on request |
| Chemicals | |||
| Acetic acid | ThermoFisher | 33209 | |
| Agar | BD Bacto | 214010 | |
| Agarose | Lonza | 50004 | |
| Ampicillin | Applichem | A0839 | |
| Anhydrotetracycline | Abcam | ab145350 | |
| anti-collagen type I antibody COL-1 | Sigma | C2456 | |
| Bovine collagen type I | Sigma | C9791 | |
| Calcium chloride | Merck | 102382 | |
| Chloroform | Merck | 102445 | |
| Di-sodium hydrogen phosphate | VWR | 28029 | |
| DNA dye | Thermo | S33102 | |
| DNA molecular size marker | New England BioLabs | N3232S | |
| DNase I | Sigma | DN25 | |
| dNTP mix | New England Biolabs | N0447 | |
| ECL HRP substrate | Advansta | K-12045 | |
| EDTA | Applichem | A2937 | |
| Glycerol | VWR | 24388 | |
| goat anti-mouse IgG-HRP | Santa Cruz | sc-2005 | |
| goat anti-rabbit IgG-HRP | Agrisera | AS10668 | |
| HEPES | VWR | 30487 | |
| Isopropyl thiogalactoside | VWR | 43714 | |
| Kanamycin | Applichem | A1493 | |
| Lysozyme | Applichem | A4972 | |
| Magnesium chloride | VWR | 25108 | |
| Manganese chloride | Sigma | 221279 | |
| N-lauroyl sarcosine | Sigma | L9150 | |
| Skim milk powder | Sigma | 70166 | |
| Sodium chloride | VWR | 27808 | |
| tamA forward primer | Invitrogen | N/A | Sequence 5'-GAAAAAAGG ATATTCAGGAGAAAATGTG-3' |
| tamA reverse primer | Invitrogen | N/A | Sequence 5'-TCATAATT CTGGCCCCAGACC-3' |
| Taq DNA polymerase | New England Biolabs | M0267 | |
| Tri-sodium citrate | Merck | 106448 | |
| Tryptone | VWR | 84610 | |
| Tween20 | Sigma | P1379 | |
| Yeast extract | Merck | 103753 | |
| Equipment | |||
| Agarose gel electrophoresis chamber | Hoefer | SUB13 | |
| Bead beater | Thermo | FP120A-115 | |
| CCD camera | Kodak | 4000R | |
| Electroporation cuvettes | Bio-Rad | 165-2089 | |
| Electroporation unit | Bio-Rad | 1652100 | |
| Gel imager | Nippon Genetics | GP-03LED | |
| Incubating shaker | Infors HT | Minitron | |
| Incubator | VWR | 390-0482 | |
| Microcentrifuge | Eppendorf | 5415D | |
| Microwave oven | Samsung | CM1099A | |
| PCR machine | Biometra | Tpersonal | |
| PCR strips | Axygen | PCR-0208-CP-C | |
| pH meter | Hanna Instruments | HI2211-01 | |
| PVDF membrane | ThermoFisher | 88518 | |
| SDS-PAG electrophoresis chamber | ThermoFisher | A25977 | |
| Tabletop centrifuge | Beckman Coulter | B06322 | |
| Vortex mixer | Scientific Industries | SI-0236 | |
| Water bath | GFL | D3006 | |
| Wet transfer unit | Hoefer | TE22 |
Referencias
- Blount, Z. D. The unexhausted potential of E. coli. eLife. 4, e05826 (2015).
- Hamilton, C. M., Aldea, M., Washburn, B. K., Babitzke, P., Kushner, S. R. New method for generating deletions and gene replacements in Escherichia coli. Journal of Bacteriology. 171 (9), 4617-4622 (1989).
- Link, A. J., Phillips, D., Church, G. M. Methods for generating precise deletions and insertions in the genome of wild-type Escherichia coli: application to open reading frame characterization. Journal of Bacteriology. 179 (20), 6228-6237 (1997).
- Sawitzke, J. A., Thomason, L. C., Costantino, N., Bubunenko, M., Datta, S., Court, D. L. Recombineering: in vivo genetic engineering in E. coli, S. enterica, and beyond. Methods in Enzymology. 421, 171-199 (2007).
- Datsenko, K. A., Wanner, B. L. One-step inactivation of chromosomal genes in Escherichia coli K-12 using PCR products. Proceedings of the National Academy of Sciences of the United States of America. 97 (12), 6640-6645 (2000).
- Muyrers, J. P., Zhang, Y., Stewart, A. F. Techniques: Recombinogenic engineering--new options for cloning and manipulating DNA. Trends in Biochemical Sciences. 26 (5), 325-331 (2001).
- Baba, T., et al. Construction of Escherichia coli K-12 in-frame, single-gene knockout mutants: the Keio collection. Molecular Systems Biology. 2, (2006).
- Yamamoto, N., et al. Update on the Keio collection of Escherichia coli single-gene deletion mutants. Molecular Systems Biology. 5, 335 (2009).
- Baba, T., Huan, H. -. C., Datsenko, K., Wanner, B. L., Mori, H. The applications of systematic in-frame, single-gene knockout mutant collection of Escherichia coli K-12. Methods in Molecular Biology. 416, 183-194 (2008).
- Chattopadhyay, M. K., Tabor, C. W., Tabor, H. Polyamines are not required for aerobic growth of Escherichia coli: preparation of a strain with deletions in all of the genes for polyamine biosynthesis. Journal of Bacteriology. 191 (17), 5549-5552 (2009).
- Xie, X., Wong, W. W., Tang, Y. Improving simvastatin bioconversion in Escherichia coli by deletion of bioH. Metabolic Engineering. 9 (4), 379-386 (2007).
- Maeda, T., Sanchez-Torres, V., Wood, T. K. Escherichia coli hydrogenase 3 is a reversible enzyme possessing hydrogen uptake and synthesis activities. Applied Microbiology and Biotechnology. 76 (5), 1035-1042 (2007).
- Meehan, B. M., Landeta, C., Boyd, D., Beckwith, J. The disulfide bond formation pathway is essential for anaerobic growth of Escherichia coli. Journal of Bacteriology. 199 (16), (2017).
- Niba, E. T. E., Naka, Y., Nagase, M., Mori, H., Kitakawa, M. A genome-wide approach to identify the genes involved in biofilm formation in E. coli. DNA Research. 14 (6), 237-246 (2008).
- Heinz, E., et al. Conserved features in the structure, mechanism, and biogenesis of the inverse autotransporter protein family. Genome Biology and Evolution. 8 (6), 1690-1705 (2016).
- Selkrig, J., et al. Discovery of an archetypal protein transport system in bacterial outer membranes. Nature Structural & Molecular Biology. 19 (5), 506-510 (2012).
- Stubenrauch, C., et al. Effective assembly of fimbriae in Escherichia coli depends on the translocation assembly module nanomachine. Nature Microbiology. 1 (7), 16064 (2016).
- Leo, J. C., Goldman, A. The immunoglobulin-binding Eib proteins from Escherichia coli are receptors for IgG Fc. Molecular Immunology. 46 (8-9), 1860-1866 (2009).
- Mühlenkamp, M., Oberhettinger, P., Leo, J. C., Linke, D., Schütz, M. S. Yersinia adhesin A (YadA) - Beauty & beast. International Journal of Medical Microbiology. 305 (2), 252-258 (2015).
- Studier, F. W., Moffatt, B. A. Use of bacteriophage T7 RNA polymerase to direct selective high-level expression of cloned genes. Journal of Molecular Biology. 189 (1), 113-130 (1986).
- Meuskens, I., Michalik, M., Chauhan, N., Linke, D., Leo, J. C. A new strain collection for improved expression of outer membrane proteins. Frontiers in Cellular and Infection Microbiology. 7, 464 (2017).
- Cherepanov, P. P., Wackernagel, W. Gene disruption in Escherichia coli: TcR and KmR cassettes with the option of Flp-catalyzed excision of the antibiotic-resistance determinant. Gene. 158 (1), 9-14 (1995).
- Grosskinsky, U., et al. A conserved glycine residue of trimeric autotransporter domains plays a key role in Yersinia adhesin A autotransport. Journal of Bacteriology. 189 (24), 9011-9019 (2007).
- Leo, J. C., et al. The structure of E. coli IgG-binding protein D suggests a general model for bending and binding in trimeric autotransporter adhesins. Structure. 19 (7), 1021-1030 (1993).
- Bertani, G. Studies on lysogenesis. I. The mode of phage liberation by lysogenic Escherichia coli. Journal of Bacteriology. 62 (3), 293-300 (1951).
- Hanahan, D. Studies on transformation of Escherichia coli with plasmids. Journal of Molecular Biology. 166 (4), 557-580 (1983).
- Leo, J. C., Oberhettinger, P., Linke, D. Assessing the outer membrane insertion and folding of multimeric transmembrane β-barrel proteins. Methods in Molecular Biology. , 157-167 (2015).
- Mikula, K. M., Leo, J. C., Łyskowski, A., Kedracka-Krok, S., Pirog, A., Goldman, A. The translocation domain in trimeric autotransporter adhesins is necessary and sufficient for trimerization and autotransportation. Journal of Bacteriology. 194 (4), 827-838 (2012).
- Thomason, L. C., Costantino, N., Court, D. L., Ausubel, F. M. E. coli genome manipulation by P1 transduction. Current Protocols in Molecular Biology. , (2007).
- Franklin, N. C. Mutation in gal U gene of E. coli blocks phage P1 infection. Virology. 38 (1), 189-191 (1969).
- Jeong, H., et al. Genome sequences of Escherichia coli B strains REL606 and BL21(DE3). Journal of Molecular Biology. 394 (4), 644-652 (2009).
- Greene, E. C. DNA Sequence Alignment during Homologous Recombination. Journal of Biological Chemistry. 291 (22), 11572-11580 (2016).
- Watt, V. M., Ingles, C. J., Urdea, M. S., Rutter, W. J. Homology requirements for recombination in Escherichia coli. Proceedings of the National Academy of Sciences of the United States of America. 82 (14), 4768-4772 (1985).
- Ho, T. D., Waldor, M. K. Enterohemorrhagic Escherichia coli O157:H7 gal mutants are sensitive to bacteriophage P1 and defective in intestinal colonization. Infection and Immunity. 75 (4), 1661-1666 (2006).
- Ikeda, H., Tomizawa, J. I. Transducing fragments in generalized transduction by phage P1. I. Molecular origin of the fragments. Journal of Molecular Biology. 14 (1), 85-109 (1965).
- Murooka, Y., Harada, T. Expansion of the host range of coliphage P1 and gene transfer from enteric bacteria to other Gam-negative bacteria. Applied and Environmental Microbiology. 38 (4), 754-757 (1979).
- Goldberg, R. B., Bender, R. A., Streicher, S. L. Direct selection for P1-sensitive mutants of enteric bacteria. Journal of Bacteriology. 118 (3), 810-814 (1974).
- O'Connor, K. A., Zusman, D. R. Coliphage P1-mediated transduction of cloned DNA from Escherichia coli to Myxococcus xanthus: use for complementation and recombinational analyses. Journal of Bacteriology. 155 (1), 317-329 (1983).
Reimpresiones y Permisos
Solicitar permiso para reutilizar el texto o las figuras de este JoVE artículos
Solicitar permisoThis article has been published
Video Coming Soon
ACERCA DE JoVE
Copyright © 2025 MyJoVE Corporation. Todos los derechos reservados