需要订阅 JoVE 才能查看此. 登录或开始免费试用。
Method Article
可抽税抗生素耐药盒 P1 转导制备大肠杆菌中的基因缺失
摘要
在这里, 我们提出了使用预先存在的抗生素耐药性-盒式删除结构作为一个基础, 使删除突变体在其他大肠杆菌菌株。这种删除突变可以调动和插入到相应的受体菌株的位置使用 P1 噬菌体转导。
摘要
第一种研究未知基因在细菌中的作用的方法是创建一个基因的敲除。在这里, 我们描述了一个健壮和快速的协议, 将基因缺失突变从一个大肠杆菌的菌株转移到另一个, 利用广义转导与噬菌体 P1。这种方法要求突变是可选择的 (例如,基于基因中断使用抗生素盒插入)。这种抗生素盒可以从捐献者的应变中调动出来, 并引入一个感兴趣的受体菌株, 迅速而容易地产生基因删除突变体。该抗生素盒可以设计为包括 flippase 识别网站, 允许通过特定地点的 recombinase 切除卡带, 以产生一个干净的敲出, 只有一个〜100基对长的疤痕序列在基因组。我们通过敲除 autotransporter 合成中涉及的一个装配因子的多摩基因来证明该协议, 并测试这一击出对两三联 autotransporter adhesins 合成和功能的影响。虽然 P1 转导基因的缺失有其局限性, 但其实施的简便性和速度使其成为其他基因缺失的一种有吸引力的替代方法。
引言
研究基因功能的一个共同的第一种方法是执行敲出诱变和观察结果表型。这也被称为反向遗传学。细菌大肠杆菌一直是分子生物学的主力, 在过去的70年左右, 由于它的培养和它的顺从的遗传操作1。在大肠杆菌中产生基因缺失的方法有好几种, 包括标记交换诱变2、3和最近的重组工程使用λ红色或 Rac ET 系统4,5,6。
在广泛使用的系统中, 单个基因的编码序列被一种抗生素耐药性盒取代, 以后可以从5、7号染色体中切除。编码序列被替换, 例如由卡那霉素 (菅直人) 抵抗卡带, 由 flippase (FLP) 识别目标 (首次登记) 站点在两边旁边。首次登记税的地点是由 recombinase FLP 认可的, 该处介导在首次登记税地点之间, 导致删除菅直人卡带的地点特定的重组。这样, 可以实现对给定基因编码序列的完全删除, 只留下大约100基对 (bp) 的最小疤痕序列 (图 1)。
就在十年前, 所谓的京王收藏被开发出来了。这是一个基于标准实验室大肠杆菌K12 菌株的细菌库, 几乎所有非必需基因都被λ红色重组7,8单独删除。此集合中的克隆每个都有一个编码序列, 替换为可抽税的坎电阻盒。在许多应用中, 京王系列已被证明是一个有用的工具9。其中一个应用是在其他大肠杆菌菌株中产生删除突变体。从一个给定的删除克隆的菅直人可以动员一般传感噬菌体, 如 P110,11,12,13,14。从这种菌株中制备的噬菌体可以用来感染受体大肠杆菌, 在低但可靠的频率下, 可以通过同源重组将菅直人盒包含的区域纳入受体基因组。(图 2)。Transductants 可以选择在含菅直人的培养基上生长。在此之后, 如果需要去除抗生素耐药性盒, FLP recombinase 可以提供给 transductant 菌株的反式。对含有氨苄西林 (amp) 抗性标记物的 FLP 质粒进行了筛选, 对其进行了筛查, 并对野型编码序列和菅直人盒的正确切除进行了菌落 PCR 验证。
在这里, 提出了一个详细的协议, 描述了根据上述战略生产出的大肠杆菌菌株的每一个步骤。作为一个例子, 一个删除的摩的基因是证明。多摩编码的外层膜β-桶蛋白是运输和组装模块 (TAM) 的一部分, 涉及某些 autotransporter 蛋白的合成和毛菌15,16,17。然后用这个敲出的菌株来考察多摩的删除对两个三联 autotransporter adhesins (TAAs)、合成耶尔森氏杆菌和大肠杆菌免疫球蛋白 (Ig) 结合黏附 TAA的影响。18,19。
研究方案
1. 菌株和质粒
-
菌株
- 使用大肠杆菌菌株 BW251135, JW4179 (BW25113摩:: 菅直人)7, BL21 (DE3)20, BL21ΔABCF21。有关详细信息, 请参阅材料表。
-
噬菌体
- 使用噬菌体 P1vir的一般转导。将噬菌体储存在少量氯仿滴下的液体储存中 (见步骤 2.2)。有关详细信息, 请参阅材料表。
-
质 粒
- 在本协议中使用以下质粒: pCP2022、pIBA2-YadA23和 pEibD1024。作为控制质粒, 使用 pASK-IBA2 和 pET22b (见材料表)。
-
生长条件
- 在含有 pCP20 的 BL21ΔABCF 和菌株的情况下, 以剧烈震动 (180–200 rpm) 在37摄氏度或30摄氏度的溶源性肉汤 (磅) 培养基25中传播细菌。
- 进行质粒固化43摄氏度。
- 对于固体培养基, 补充 LB 与1% 琼脂 (w/v)。
- 对于顶琼脂, 补充 LB 与0.7% 琼脂和10毫米 CaCl2和高压釜的培养基。使用 SOC 介质进行电穿孔26后的恢复。
- 使用以下抗生素浓度: 100 微克/毫升为安培和25微克/毫升为菅直人。
2. 制备噬菌体裂解液
-
供体菌株感染
- 生长捐赠者应变 JW4197 在5毫升 LB 介质补充与10毫米 CaCl2和可选与菅直人 (25 µg/毫升) 到光学密度在600毫微米 (OD600) ~ 1.0。使用分光光度计测量 OD600值。
- 在 LB 介质中做一个现有 P1 噬菌体库存的稀释系列: 推荐的稀释介于 10-3到 10-7之间。
- 混合 200 ul 的细菌悬浮和 100 ul 的特定噬菌体稀释在15毫升离心管或同等。准备尽可能多的管作为噬菌体稀释。在37摄氏度处孵育20分钟, 不摇晃。
- 添加〜3毫升的熔融顶琼脂 (~ 50 °c) 补充10毫米 CaCl2的管, 混合的内容, 通过涡流的管很快, 并倒入 prewarmed LB 板上的混合物, 使均匀层。
- 在37摄氏度一夜之间孵化盘子。
-
裂解制剂
- 第二天选择一个有半汇合生长的噬菌体斑块的盘子。在半汇合板上, 大约一半的板块表面是清晰的 (图 3)。
- 使用接种回路或类似的工具从这样的盘子刮上琼脂层, 并将顶琼脂放在离心管中。添加一毫升的 LB 和一滴氯仿和漩涡的管大力1分钟. 在通风罩中加入氯仿。
- 离心管15分钟, 在 4000 x克或更快的颗粒的琼脂和细菌细胞。
- 将上清移至新鲜的离心管, 避免从颗粒中携带任何碎片。加入2滴氯仿, 在4–10°c 储存裂解液。在通风罩中加入氯仿。不要冻结噬菌体裂解物, 因为这将导致显著减少感染微粒的数量。
-
测定裂解液滴度
- 生长 BW25113 在 LB 补充与10毫米 CaCl2在37°c, 直到文化达到 OD600的 ~ 1.0。
- 准备一个稀释系列的噬菌体裂解液在 LB (例如, 10-6–10-9)。
注意: 小心吸管从裂解液顶部取样, 避免将氯仿转移到稀释。 - 混合 200 ul 的细菌悬浮和 100 ul 的特定噬菌体稀释在15毫升离心管或同等。准备尽可能多的管作为噬菌体稀释。在37摄氏度处孵育20分钟, 不摇晃。
- 添加〜3毫升的熔融顶琼脂 (~ 50 °c) 补充10毫米 CaCl2的管, 混合的内容, 通过涡流的管很快, 并倒入 prewarmed LB 板上的混合物, 使均匀层。
- 在37摄氏度一夜之间孵化盘子。
- 第二天计算每个板块的斑块数量 (细菌垫中的清除区域), 并使用以下公式计算裂解液的滴度:

例子: 在板材与稀释 10-7, 10-8和 10-9, 分别有 231, 27 和2个匾。当 100 ul 每稀释被使用了为传染, 并且, 因为效价被表达作为斑块形成的单位 (pfu)/mL, 电镀因素是10。这些数字输入到公式中: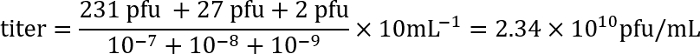
因此, 滴度是大约 2 x 1010传染性噬菌体粒子/毫升。
3. P1 转导
-
准备收件人单元格
- 增加接受者应变 BL21ΔABCF 在 LB 补充到光学密度在600毫微米 (OD600) 1.0。使用分光光度计测量 OD600值。
- 计算所需的噬菌体裂解液的体积, 以达到0.5 的感染多样性 (语言) 值。计算语言, 估计细菌数量的基础上600的文化。假设 OD600值1.0 对应于 ~ 109 cfu/毫升。根据已知的裂解液滴度计算所需的体积。
示例 (基于在步骤2.3.6 后的示例中计算的效度):
- 添加 CaCl2到接受菌株的文化, 以10毫米和混合。采取1毫升的文化进行转导。
-
执行转导
- 添加适当数量的噬菌体裂解到1毫升的接受者文化 (包括 CaCl2在10毫米), 计算在步骤3.1.2 和轻轻混合。
注意: 小心把样品从裂解液的顶部移除, 避免将氯仿转移到混合物中。 - 静态孵化混合20分钟, 在37摄氏度。
- 通过添加柠檬酸钠, pH 值 5.5, 到100毫米停止感染。
- 离心的细菌 (5000 x g 2 分钟) 和清除上清, 然后并用重悬他们在1毫升的新鲜磅补充100毫米柠檬酸钠, pH 5.5。
- 在步骤3.2.4 中清洗细胞两次, 以确保去除游离噬菌体和钙。
- 并用重悬1毫升新鲜磅的细菌补充100毫米柠檬酸钠, pH 5.5。用震动 (> 100 rpm) 在30摄氏度孵化细菌1小时。
- 收集细菌的离心 (5000 x g 2 分钟) 和并用重悬他们在 100 ul 的 LB 与100毫米柠檬酸钠, pH 5.5。
- 在 LB 板上传播细菌, 辅以25µg/毫升和10毫米柠檬酸钠, pH 5.5, 并将细菌生长在30摄氏度, 直到菌落出现 (~ 24 h)。
- 添加适当数量的噬菌体裂解到1毫升的接受者文化 (包括 CaCl2在10毫米), 计算在步骤3.1.2 和轻轻混合。
-
选择 transductants
- 一旦殖民地生长在选择板块上, restreak 他们在 LB + 菅直人为单一的殖民地和增长30摄氏度, 直到单一的殖民地出现。
4. 切除菅直人卡带
-
重组质粒的转化
- 使抗 BL21ΔABCF 应变 electrocompetent。
- 生长菌株从一个单一的蚁群在新鲜磅 (5 毫升) 补充与菅直人 (25 µg/毫升) 在30°c, 直到 OD600是 ~ 0.5–0.7。
- 从这里, 执行以下步骤在4°c 或在冰上。将细菌 (5000 x g 10 分钟) 离心, 取出上清液。
- 重复先前的离心步骤, 并在100µL 的冰冷10% 甘油中并用重悬细胞颗粒, 用冰冷的蒸馏水冲洗两次细胞颗粒。
- 冷却1毫米电穿孔小试管在冰上。
- 在细胞悬浮液中加入 1 pg 质粒 DNA (pCP20), 轻轻混合悬浮液, 将其转移到冷电穿孔试管。
- 将 electroporator 设置为1.8 伏, 并 electroporate 单元格。
- 通过添加1毫升的 SOC 培养基, 并在30摄氏度的1小时内生长, 来抢救转化后的细胞。
- 板100µL 的细胞在 LB + 安培板和增长的细胞在30°c 过夜。
- 使抗 BL21ΔABCF 应变 electrocompetent。
-
重组的诱导
- 从 lb + 安培板中挑选单一的菌落, 接种新鲜的 lb, 省略所有的抗生素。
- 在不允许的温度下 (43 摄氏度) 一夜之间生长细胞, 以诱导 FLP recombinase 的表达。
-
选择重组
- 使系列稀释和板材50µL 105-106稀释在非选择性板材和增长它隔夜在30°c。
5. 基因删除的验证
-
质粒丧失与成功重组的验证
- 在这一顺序中, 4.3.1 在 lb、lb、磅和无抗生素的 lb 板上进行的平板上的单菌落。要帮助裸奔, 请使用殖民地网格 (参见辅助文件 1)。将盘子生长在30摄氏度, 直到菌落出现 (~ 24 小时)。
- 选择在非选择性板上生长的10–20克隆, 但在选择性介质上无法生长以进行进一步的验证。
-
用菌落 PCR 法进行附加验证
- 执行一个群体 PCR 与引物侧翼的多重摩编码序列 (见材料表)。
- 准备一个主 PCR 组合。所需的混合量取决于要测试的菌落数 (参见表 1中的示例)。将试剂混合在冰上, 最后加入聚合酶。
- 将 pcr 大师组合彻底混合, 然后将20µL 放入 pcr 条的管中。选择一个小数量的每个殖民地使用不育的吸管提示, 并添加到管。请记住包含原始的收件人应变以进行比较。另外, 还包括捐助者的应变。
- 运行 PCR 反应 (见表 2所用的程序)。
- 准备1% 琼脂糖凝胶。
- 50毫升凝胶, 测量0.5 克琼脂糖和添加50毫升的泰缓冲器 (40 毫米三, 20 毫米醋酸, 1 毫米 EDTA, pH 值 8.0)。在微波炉中加热混合物, 直到所有琼脂糖都溶解。
- 一旦琼脂糖溶解, 冷却溶液约50摄氏度, 然后添加 5 ul 的 DNA 染色染料。把溶液搅拌好, 倒入浇注室的凝胶盘中。插入一个或多个行的好梳子, 以便有足够的水井为所有样品, 以及分子尺寸标记。允许凝胶设置为30分钟。
- 一旦 PCR 运行完成, 添加4µL 的 6x DNA 负载染料的每个样本。将凝胶放在电泳室中, 在井盖前加泰缓冲器。
- 应用10–15µL 从每个 PCR 反应到一个良好的凝胶。还添加5µL 的分子尺寸标记。然后, 在 75 v 上运行30分钟的凝胶。
- 一旦运行完成, 图像的凝胶使用蓝光成像仪。
6. 其他技术
-
蛋白表达
注: 测试蛋白 (亚 EibD) 的表达在其他地方23,24被详细描述。这是对主要步骤的简要总结。- 将所需的质粒 (pIBA2-YadA 和 pEibD10 及相应的控制质粒) 转换成 BL21ΔABCF Δ多摩 w, 并选择在 LB + Amp 上转化。
- 对于蛋白表达, 接种100毫升的 LB 中 + 安培与1毫升的一夜文化转化细菌和增长这些30°c, 直到中期记录阶段, 在这段时间, 诱导蛋白质生产与任何乳糖 (在 100 ng/毫升) 或异丙基-β-d (0.5 毫米)。
- 在 2 h 以后归纳在30°c, 测量文化的浊度和收集对应于50毫升的细胞在 OD600 = 1.0 通过离心文化为15分钟在 4000 x g。用10毫米 HEPES, pH 7.4, 然后将球团 1x, 然后将其存储在-20 摄氏度或进一步处理, 如步骤6.2。
-
外膜萃取
注: 外膜萃取是由 Leo等详细描述的。27. 下面概述了主要步骤。- 并用重悬细胞颗粒在1毫升10毫米 HEPES, pH 7.4, 补充10毫米氯化镁2和 MnCl2, 溶菌酶 (0.1 毫克/毫升), 和一捏 DNase i。
- 溶解细胞 (例如, 使用一个珠子搅拌器)。
- 将细胞尽快离心 (15600 x g2 分钟) 去除细胞碎片, 将上清移至新鲜管。
- 离心细胞为30分钟在 1.6万 x g, 在之后, 并用重悬颗粒在 400 ul 1% n-月桂肌氨酸在10毫米 HEPES, pH 7.4。
- 在室温下, 用搅拌30分钟孵育细胞, 然后将其离心30分钟以上。
- 清洗半透明颗粒1x 与 200 ul 10 毫米 HEPES, pH 7.4, 然后并用重悬它在 30 ul 的10毫米 HEPES 和添加 10 ul 4x SDS 页样本缓冲区。
-
活动化验
注: 执行 SDS 页和活动化验为亚多和 EibD 如28所述。主要步骤概述如下。 -
SDS 页
- 将样品加热50摄氏度5分钟, 然后再将其装入聚丙烯酰胺凝胶上, 以避免蛋白质变性。
- 在 SDS 页分离后, 将蛋白质转移到聚偏聚氟 (PVDF) 膜上。
- 转移后, 在 PBS 中用2% 脱脂奶粉堵住膜。
-
远西胶原蛋白印迹
- 阻断后, 加入牛胶原 i 型稀释在阻塞缓冲液中浓度为10微克/毫升, 并孵育膜1小时。
- 用 pbs + 0.05% Tween20 (pbs t) 冲洗膜2x。
- 将原抗体 (单克隆抗胶原 COL-1) 添加到膜中, 稀释 1:2, 000 在阻塞缓冲液中。
- 孵化后1小时的膜, 洗 2x, 如步骤6.3.2.2 中所述, 然后添加二次抗体 [山羊抗鼠 IgG-辣根过氧化物酶 (HRP) 共轭], 稀释1:10,000 在阻塞缓冲。
- 将膜孵化1小时, 然后用 PBS 清洗2x。根据制造商的指示, 在膜上添加增强的化学发光基板, 并使用 CCD 摄像机检测波段。
-
EibD IgG 结合试验
- 阻断后, 添加二次抗体 (山羊抗兔 HRP), 稀释 1:2, 000 在阻塞缓冲。
- 将膜孵化1小时, 然后用 PBS 冲洗2x。执行步骤6.3.2.5 中提到的化学发光检测。
结果
多摩BL21ΔABCF 的产生:
上面概述的战略以前曾被用来生产 BL21 (DE3) 的衍生物菌株, 这是用于蛋白质生产的标准实验室菌株, 它被优化用于外膜蛋白生产, 并被称为 BL21ΔABCF21。这种菌株缺乏四基因编码的丰富的外膜蛋白, 因此, 能够产生更多的 heterologously 表达外膜蛋白比野生型菌株。为了测试 TAM 是否参与了 TA...
讨论
P1 转导是一种快速、稳健、可靠的方法, 可在大肠杆菌中产生基因缺失。这在这里展示了传感一个摩中的删除突变从一个王庆会捐献者的应变 BL21-derived 接受。转导过程的主要阶段是传感裂解液的产生、转导本身、菅直人抗性盒的切除以及 PCR 的验证。总的来说, 这个过程需要大约1周的时间, 不需要使用分子生物学方法, 除了最终的 PCR 进行验证。因此, P1 转导可以在花费的努力和时间?...
披露声明
作者没有什么可透露的。
致谢
京王收集菌株来自国家 BioResource 项目 ( 日本 ) :大肠杆菌。我们感谢德克左翼 (奥斯陆大学生物科学系) 继续提供支持。这项工作由挪威青年研究员补助金 249793 (对杰克. 里奥) 的研究理事会资助。
材料
| Name | Company | Catalog Number | Comments |
| Strains | |||
| E. coli BW25113 | NIG | ME6092 | Wild-type strain of Keio collection |
| E. coli BL21(DE3) | Merck | 69450-3 | Expression strain |
| E. coli BL21DABCF | Addgene | 102270 | Derived from BL21(DE3) |
| E. coli JW4179 | NIG | JW4179-KC | tamA deletion mutant |
| P1 vir | NIG | HR16 | Generally transducing bacteriophage |
| Plasmids | |||
| pCP20 | CGSC | 14177 | conditionally replicating plasmid with FLP |
| pASK-IBA2 | IBA GmbH | 2-1301-000 | expression vector |
| pEibD10 | N/A | N/A | for production of EibD; plasmid available on request |
| pET22b+ | Merck | 69744-3 | expression vector |
| pIBA2-YadA | N/A | N/A | for production of YadA; plasmid available on request |
| Chemicals | |||
| Acetic acid | ThermoFisher | 33209 | |
| Agar | BD Bacto | 214010 | |
| Agarose | Lonza | 50004 | |
| Ampicillin | Applichem | A0839 | |
| Anhydrotetracycline | Abcam | ab145350 | |
| anti-collagen type I antibody COL-1 | Sigma | C2456 | |
| Bovine collagen type I | Sigma | C9791 | |
| Calcium chloride | Merck | 102382 | |
| Chloroform | Merck | 102445 | |
| Di-sodium hydrogen phosphate | VWR | 28029 | |
| DNA dye | Thermo | S33102 | |
| DNA molecular size marker | New England BioLabs | N3232S | |
| DNase I | Sigma | DN25 | |
| dNTP mix | New England Biolabs | N0447 | |
| ECL HRP substrate | Advansta | K-12045 | |
| EDTA | Applichem | A2937 | |
| Glycerol | VWR | 24388 | |
| goat anti-mouse IgG-HRP | Santa Cruz | sc-2005 | |
| goat anti-rabbit IgG-HRP | Agrisera | AS10668 | |
| HEPES | VWR | 30487 | |
| Isopropyl thiogalactoside | VWR | 43714 | |
| Kanamycin | Applichem | A1493 | |
| Lysozyme | Applichem | A4972 | |
| Magnesium chloride | VWR | 25108 | |
| Manganese chloride | Sigma | 221279 | |
| N-lauroyl sarcosine | Sigma | L9150 | |
| Skim milk powder | Sigma | 70166 | |
| Sodium chloride | VWR | 27808 | |
| tamA forward primer | Invitrogen | N/A | Sequence 5'-GAAAAAAGG ATATTCAGGAGAAAATGTG-3' |
| tamA reverse primer | Invitrogen | N/A | Sequence 5'-TCATAATT CTGGCCCCAGACC-3' |
| Taq DNA polymerase | New England Biolabs | M0267 | |
| Tri-sodium citrate | Merck | 106448 | |
| Tryptone | VWR | 84610 | |
| Tween20 | Sigma | P1379 | |
| Yeast extract | Merck | 103753 | |
| Equipment | |||
| Agarose gel electrophoresis chamber | Hoefer | SUB13 | |
| Bead beater | Thermo | FP120A-115 | |
| CCD camera | Kodak | 4000R | |
| Electroporation cuvettes | Bio-Rad | 165-2089 | |
| Electroporation unit | Bio-Rad | 1652100 | |
| Gel imager | Nippon Genetics | GP-03LED | |
| Incubating shaker | Infors HT | Minitron | |
| Incubator | VWR | 390-0482 | |
| Microcentrifuge | Eppendorf | 5415D | |
| Microwave oven | Samsung | CM1099A | |
| PCR machine | Biometra | Tpersonal | |
| PCR strips | Axygen | PCR-0208-CP-C | |
| pH meter | Hanna Instruments | HI2211-01 | |
| PVDF membrane | ThermoFisher | 88518 | |
| SDS-PAG electrophoresis chamber | ThermoFisher | A25977 | |
| Tabletop centrifuge | Beckman Coulter | B06322 | |
| Vortex mixer | Scientific Industries | SI-0236 | |
| Water bath | GFL | D3006 | |
| Wet transfer unit | Hoefer | TE22 |
参考文献
- Blount, Z. D. The unexhausted potential of E. coli. eLife. 4, e05826 (2015).
- Hamilton, C. M., Aldea, M., Washburn, B. K., Babitzke, P., Kushner, S. R. New method for generating deletions and gene replacements in Escherichia coli. Journal of Bacteriology. 171 (9), 4617-4622 (1989).
- Link, A. J., Phillips, D., Church, G. M. Methods for generating precise deletions and insertions in the genome of wild-type Escherichia coli: application to open reading frame characterization. Journal of Bacteriology. 179 (20), 6228-6237 (1997).
- Sawitzke, J. A., Thomason, L. C., Costantino, N., Bubunenko, M., Datta, S., Court, D. L. Recombineering: in vivo genetic engineering in E. coli, S. enterica, and beyond. Methods in Enzymology. 421, 171-199 (2007).
- Datsenko, K. A., Wanner, B. L. One-step inactivation of chromosomal genes in Escherichia coli K-12 using PCR products. Proceedings of the National Academy of Sciences of the United States of America. 97 (12), 6640-6645 (2000).
- Muyrers, J. P., Zhang, Y., Stewart, A. F. Techniques: Recombinogenic engineering--new options for cloning and manipulating DNA. Trends in Biochemical Sciences. 26 (5), 325-331 (2001).
- Baba, T., et al. Construction of Escherichia coli K-12 in-frame, single-gene knockout mutants: the Keio collection. Molecular Systems Biology. 2, (2006).
- Yamamoto, N., et al. Update on the Keio collection of Escherichia coli single-gene deletion mutants. Molecular Systems Biology. 5, 335 (2009).
- Baba, T., Huan, H. -. C., Datsenko, K., Wanner, B. L., Mori, H. The applications of systematic in-frame, single-gene knockout mutant collection of Escherichia coli K-12. Methods in Molecular Biology. 416, 183-194 (2008).
- Chattopadhyay, M. K., Tabor, C. W., Tabor, H. Polyamines are not required for aerobic growth of Escherichia coli: preparation of a strain with deletions in all of the genes for polyamine biosynthesis. Journal of Bacteriology. 191 (17), 5549-5552 (2009).
- Xie, X., Wong, W. W., Tang, Y. Improving simvastatin bioconversion in Escherichia coli by deletion of bioH. Metabolic Engineering. 9 (4), 379-386 (2007).
- Maeda, T., Sanchez-Torres, V., Wood, T. K. Escherichia coli hydrogenase 3 is a reversible enzyme possessing hydrogen uptake and synthesis activities. Applied Microbiology and Biotechnology. 76 (5), 1035-1042 (2007).
- Meehan, B. M., Landeta, C., Boyd, D., Beckwith, J. The disulfide bond formation pathway is essential for anaerobic growth of Escherichia coli. Journal of Bacteriology. 199 (16), (2017).
- Niba, E. T. E., Naka, Y., Nagase, M., Mori, H., Kitakawa, M. A genome-wide approach to identify the genes involved in biofilm formation in E. coli. DNA Research. 14 (6), 237-246 (2008).
- Heinz, E., et al. Conserved features in the structure, mechanism, and biogenesis of the inverse autotransporter protein family. Genome Biology and Evolution. 8 (6), 1690-1705 (2016).
- Selkrig, J., et al. Discovery of an archetypal protein transport system in bacterial outer membranes. Nature Structural & Molecular Biology. 19 (5), 506-510 (2012).
- Stubenrauch, C., et al. Effective assembly of fimbriae in Escherichia coli depends on the translocation assembly module nanomachine. Nature Microbiology. 1 (7), 16064 (2016).
- Leo, J. C., Goldman, A. The immunoglobulin-binding Eib proteins from Escherichia coli are receptors for IgG Fc. Molecular Immunology. 46 (8-9), 1860-1866 (2009).
- Mühlenkamp, M., Oberhettinger, P., Leo, J. C., Linke, D., Schütz, M. S. Yersinia adhesin A (YadA) - Beauty & beast. International Journal of Medical Microbiology. 305 (2), 252-258 (2015).
- Studier, F. W., Moffatt, B. A. Use of bacteriophage T7 RNA polymerase to direct selective high-level expression of cloned genes. Journal of Molecular Biology. 189 (1), 113-130 (1986).
- Meuskens, I., Michalik, M., Chauhan, N., Linke, D., Leo, J. C. A new strain collection for improved expression of outer membrane proteins. Frontiers in Cellular and Infection Microbiology. 7, 464 (2017).
- Cherepanov, P. P., Wackernagel, W. Gene disruption in Escherichia coli: TcR and KmR cassettes with the option of Flp-catalyzed excision of the antibiotic-resistance determinant. Gene. 158 (1), 9-14 (1995).
- Grosskinsky, U., et al. A conserved glycine residue of trimeric autotransporter domains plays a key role in Yersinia adhesin A autotransport. Journal of Bacteriology. 189 (24), 9011-9019 (2007).
- Leo, J. C., et al. The structure of E. coli IgG-binding protein D suggests a general model for bending and binding in trimeric autotransporter adhesins. Structure. 19 (7), 1021-1030 (1993).
- Bertani, G. Studies on lysogenesis. I. The mode of phage liberation by lysogenic Escherichia coli. Journal of Bacteriology. 62 (3), 293-300 (1951).
- Hanahan, D. Studies on transformation of Escherichia coli with plasmids. Journal of Molecular Biology. 166 (4), 557-580 (1983).
- Leo, J. C., Oberhettinger, P., Linke, D. Assessing the outer membrane insertion and folding of multimeric transmembrane β-barrel proteins. Methods in Molecular Biology. , 157-167 (2015).
- Mikula, K. M., Leo, J. C., Łyskowski, A., Kedracka-Krok, S., Pirog, A., Goldman, A. The translocation domain in trimeric autotransporter adhesins is necessary and sufficient for trimerization and autotransportation. Journal of Bacteriology. 194 (4), 827-838 (2012).
- Thomason, L. C., Costantino, N., Court, D. L., Ausubel, F. M. E. coli genome manipulation by P1 transduction. Current Protocols in Molecular Biology. , (2007).
- Franklin, N. C. Mutation in gal U gene of E. coli blocks phage P1 infection. Virology. 38 (1), 189-191 (1969).
- Jeong, H., et al. Genome sequences of Escherichia coli B strains REL606 and BL21(DE3). Journal of Molecular Biology. 394 (4), 644-652 (2009).
- Greene, E. C. DNA Sequence Alignment during Homologous Recombination. Journal of Biological Chemistry. 291 (22), 11572-11580 (2016).
- Watt, V. M., Ingles, C. J., Urdea, M. S., Rutter, W. J. Homology requirements for recombination in Escherichia coli. Proceedings of the National Academy of Sciences of the United States of America. 82 (14), 4768-4772 (1985).
- Ho, T. D., Waldor, M. K. Enterohemorrhagic Escherichia coli O157:H7 gal mutants are sensitive to bacteriophage P1 and defective in intestinal colonization. Infection and Immunity. 75 (4), 1661-1666 (2006).
- Ikeda, H., Tomizawa, J. I. Transducing fragments in generalized transduction by phage P1. I. Molecular origin of the fragments. Journal of Molecular Biology. 14 (1), 85-109 (1965).
- Murooka, Y., Harada, T. Expansion of the host range of coliphage P1 and gene transfer from enteric bacteria to other Gam-negative bacteria. Applied and Environmental Microbiology. 38 (4), 754-757 (1979).
- Goldberg, R. B., Bender, R. A., Streicher, S. L. Direct selection for P1-sensitive mutants of enteric bacteria. Journal of Bacteriology. 118 (3), 810-814 (1974).
- O'Connor, K. A., Zusman, D. R. Coliphage P1-mediated transduction of cloned DNA from Escherichia coli to Myxococcus xanthus: use for complementation and recombinational analyses. Journal of Bacteriology. 155 (1), 317-329 (1983).
转载和许可
请求许可使用此 JoVE 文章的文本或图形
请求许可This article has been published
Video Coming Soon
版权所属 © 2025 MyJoVE 公司版权所有,本公司不涉及任何医疗业务和医疗服务。