JoVE 비디오를 활용하시려면 도서관을 통한 기관 구독이 필요합니다. 전체 비디오를 보시려면 로그인하거나 무료 트라이얼을 시작하세요.
Method Article
Excisable 항 생 저항 카세트와 P1 변환 하 여 대장균 에서 생산 유전자 삭제
요약
여기 선물이 다른 대장균 변종에 삭제 돌연변이 만들기 위한 기반으로 기존의 항생제 저항-카세트 삭제 구문을 사용 하는 프로토콜. 이러한 삭제 돌연변이 동원 고 P1 살 균 소 변환을 사용 하 여 받는 사람 긴장의 해당 장소에 삽입 될 수 있습니다.
초록
박테리아에서 알 수 없는 유전자의 기능 연구에 대 한 첫 번째 접근은이 유전자의 녹아웃을 만드는 것입니다. 여기, 우리는 강력 하 고 빠른 살 균 소 P1 일반화 된 변환 사용 하 여 다른 한 대장균 긴장에서 유전자 삭제 돌연변이 전송 하기 위한 프로토콜을 설명 합니다. 이 방법은 돌연변이 선택 가능 (예: 항생제 카세트 삽입을 사용 하 여 유전자 중단에 따라) 해야 합니다. 이러한 항생제 카세트 기증자 긴장에서 동원 될 수 있습니다 관심 받는 사람 부담으로 신속 하 게 도입 하 고 쉽게 유전자 삭제 돌연변이 생성. 항생제 카세트 사이트별 recombinase 게놈에서 순서 ~ 100-기지-쌍-긴 흉터만 깨끗 한 녹아웃을 생산 하 여 카세트의 절단을 허용 하는 flippase 인식 사이트를 포함 하도록 디자인할 수 있습니다. 우리는 어셈블리 요소 autotransporter 속에 관련 된 인코딩 타 유전자를 노크 하 여 프로토콜을 설명 하 고는 속에이 노크 아웃의 효과 및 2 개의 trimeric autotransporter adhesins의 기능 테스트. P1 변환에 의해 유전자 삭제는 한계가, 비록 편리 하 고 속도 구현 하기가 유전자 삭제의 다른 방법에 매력적인 대안.
서문
유전자의 기능 연구에 대 한 일반적인 첫 번째 접근 노크 아웃 mutagenesis 수행 결과 표현 형을 관찰 하는 것입니다. 이것은 또한 반전 유전학을 불린다. 지난 70 년 동안 또는 이렇게, 그것의 경작 및의 순종 유전자 조작1의 용이성으로 인해 박테리아 대장균 분자 생물학의 주력 하고있다. 여러 가지 방법은 대장균, 등 표식 exchange mutagenesis2,3 , 더 최근에, λ 레드 또는 Rac 외 시스템4,5 를 사용 하 여 recombineering에에서 유전자 삭제를 생산 하기 위해 개발 되었습니다. , 6.
널리 사용 되는 시스템에서 개별 유전자의 코딩 시퀀스에서 염색체5,7excised 나중 수는 항생제 저항 카세트로 대체 됩니다. 코딩 시퀀스 대체 된다, 예를 들어 대 (칸) 저항 카세트, 양쪽에 flippase (FLP) 인식 대상 (FRT) 사이트에 의해 형벌 이다. FRT 사이트 recombinase 칸 카세트의 삭제로 이어지는 FRT 사이트 간의 사이트 재결합 중재 FLP에 의해 인식 됩니다. 이 방법에서는, 주어진된 유전자의 코딩 시퀀스의 전체 삭제 얻을 수 있습니다, 약 100의 기본적인 쌍 (bp) (그림 1)의 순서만 최소한의 흉터를 남겨두고.
전 10 년 넘게 소위 게이오 컬렉션 개발 되었다. 이것은 세균성 라이브러리 기반 표준 실험실 대장균 K12 변형, 거의 모든 비-필수 유전자 λ 빨간 재결합7,8에 의해 개별적으로 삭제 되었습니다. 이 컬렉션에서 클론 excisable 칸 저항 카세트 교체의 코딩 시퀀스 한 개 있다. 케이오 컬렉션9많은 응용 프로그램 대 한 유용한 도구가 될 입증 되었습니다. 하나는 같은 응용 프로그램 다른 대장균 변종에 삭제 돌연변이의 생산 이다. 일반적으로 살 균 소, P110,11,12,,1314등을 시험 하 여 주어진된 삭제 복제에서 칸 카세트를 동원 수 있습니다. 살 균 소 주식 같은 긴장에서 준비 사용할 수 있습니다 받는 사람의, E. 콜라이 긴장을 감염 하 어디 낮은 하지만 신뢰할 수 있는 주파수는 칸에서 카세트 포함 된 지역 포함 될 수 있습니다 받는 사람 게놈으로 동종 재결합에 의해 (그림 2)입니다. Transductants 칸 포함 된 매체에 성장에 대 한 선택할 수 있습니다. 이 따라 항생제 저항 카세트의 제거를 원하는 경우 트랜스 transductant 변형에 FLP recombinase 공급 수 있다. FLP 포함 된 플라스 미드, 암 피 실린 (Amp) 저항 마커를 운반, 경화 후 칸와 앰프-민감한 클론에 대 한 검사는 하 고 야생-타입 코딩 순서와 칸 카세트의 정확한 절단 식민지 PCR에 의해 확인 됩니다.
여기, 상세한 프로토콜 제공 됩니다, 각 녹아웃 대장균 스트레인 위에서 설명한 전략에 따라 생산 단계를 설명 하. 예를 들어, 타 마 유전자의 삭제는 보여 줍니다. 타 마 일부 전송 및 어셈블리 모듈 (TAM), 특정 autotransporter 단백질 및 pili15,,1617의 속에 포함 되는 외부 막 β 배럴 단백질을 인코딩합니다. 이 노크 아웃 긴장은 다음 2 개의 trimeric autotransporter adhesins (다시), 페스트 adhesin YadA와 대장균 면역 글로불린 (Ig)는 속에 다 마 삭제의 효과 검사 하는 데 사용 됩니다-TAA EibD 바인딩 18,19.
프로토콜
1. 긴장 및 플라스 미드
-
세균성 긴장
- E. 콜라이 긴장 BW251135, JW4179 (BW25113 tamA::kan)7,20, BL21(DE3) 및 BL21ΔABCF21을 사용 합니다. 자세한 내용은 테이블의 자료 를 참조 하십시오.
-
살 균 소
- P1비르 페이지를 사용 하 여 일반 변환에 대 한. 클로 프롬의 몇 방울과 액체 주식으로 페이지를 저장 (하십시오 단계 2.2 참조). 자세한 내용은 테이블의 자료를 참조 하십시오.
-
플라스 미드
- 다음 플라스 미드를 사용 하 여이 프로토콜에: pCP2022,23, pIBA2 YadA pEibD1024. 제어 플라스 미드로 pASK-IBA2 및 pET22b ( 재료의 표참조)를 사용 합니다.
-
성장 조건
- 전파는 lysogeny 국물 (파운드) 중간25 활기찬 동요 (180-200 rpm) 37 ° C 또는 BL21ΔABCF 및 pCP20를 포함 하는 긴장의 경우 30 ° C에 있는 박테리아.
- 43 ° c.에 치료 하는 플라스 미드를 수행
- 단단한 매체에 대 한 1 %agar (w/v)와 파운드를 보충.
- 최고 천 대 한 0.7 %agar 10mm CaCl2 와 압력솥 매체 LB 보충. Electroporation26후 회복 위한 SOC 매체를 사용 합니다.
- 항생제에 대 한 다음 농도 사용: 100 μ g/mL로 앰프와 칸에 대 한 25 μ g/mL
2. 준비 Lysate 살 균 소
-
기증자 스트레인의 감염
- 10 m m CaCl2 와 함께 선택적으로 칸을 보충 하는 파운드 매체의 5 mL에 기증자 스트레인 JW4197 성장 (25 µ g/mL) 600에서 광학 밀도를 ~ 1.0 nm (OD600). 분 광 광도 계를 사용 하 여 세600 값을 측정 합니다.
- LB 매체에는 기존 p 1 살 균 소 주식의 희석 시리즈를 만들기: 희석은 10-3 10-7을 권장.
- 세균성 펜션과 15 mL 원심 분리기 관 또는 이와 동등한 주어진된 살 균 소 희석의 100 μ의 믹스 200 μ. 살 균 소 희석으로 많은 튜브를 준비 합니다. 동요 하지 않고 37 ° C에서 20 분 동안 튜브를 품 어.
- 녹은 가기 천 (~ 50 ° C) 10mm CaCl2 관에 보충의 ~ 3 mL를 추가 하 고 내용을 철저 하 게 혼합 vortexing에 의해 관 곧, 심지어 레이어를 만들기 위해 prewarmed 파운드 접시에 혼합물을 부 어.
- 37 ° c.에 하룻밤 번호판을 품 어
-
Lysate 준비
- 다음 날 살 균 소 패의 세미 confluent 성장과 플레이트를 선택 합니다. 약 절반의 표면적 접시 반 confluent 접시에 분명 하다 (그림 3).
- 접종 루프 또는 비슷한 도구를 사용 하 여 같은 접시에서 최고 천 층을 긁어과 원심 분리기 튜브에 최고 agar를 놓습니다. 증기 두건에서 파운드의 1-2 mL 및 클로 프롬 및 소용돌이 적극적으로 ~ 1 분 추가 클로 프롬에 대 한 튜브의 한 방울을 추가 합니다.
- 4000 g x 또는 더 작은 공의 천 및 세균성 세포에서 15 분 동안 튜브 원심
- 신선한 microcentrifuge 튜브, 피하 펠 릿에서 어떤 파편 든 지 운반에 상쾌한을 이동 합니다. 클로 프롬의 2 방울을 추가 하 고 4-10 ° c.에 lysate 저장 증기 두건에서 클로 프롬을 추가 합니다. 마십시오 하지 고정 페이지 lysate로이 전염 성 입자 수의 뜻깊은 감소 귀 착될 것 이다.
-
Lysate titer를 결정
- BW25113 LB 문화 OD600 ~ 1.0에 도달할 때까지 37 ° C에서 10 mM CaCl2 보충에서 성장.
- 살 균 소 파운드 (예를 들어, 10-6– 10-9)에 lysate의 희석 시리즈 준비.
참고:은 희석을 클로 프롬을 전송 하지 않으려면 lysate의 위에서 피펫으로 샘플에 주의 해야 합니다. - 세균성 펜션과 15 mL 원심 분리기 관 또는 이와 동등한 주어진된 살 균 소 희석의 100 μ의 믹스 200 μ. 살 균 소 희석으로 많은 튜브를 준비 합니다. 동요 하지 않고 37 ° C에서 20 분 동안 튜브를 품 어.
- 녹은 가기 천 (~ 50 ° C) 10mm CaCl2 관에 보충의 ~ 3 mL를 추가 하 고 내용을 철저 하 게 혼합 vortexing에 의해 관 곧, 심지어 레이어를 만들기 위해 prewarmed 파운드 접시에 혼합물을 부 어.
- 37 ° c.에 하룻밤 번호판을 품 어
- 다음 날 플 라크 (세균성 매트에 명확한 지역) 각 접시의 수를 계산 하 고 다음 수식을 사용 하 lysate의 titer를 계산:

예: 접시 희석 10-7, 10-8및 10-9에 있다 231, 27, 2 패, 각각. 각 희석의 100 μ, 감염에 대 한 사용 되었습니다 그리고는 titer 플 라크 형성으로 표현 했기 때문에 단위 (pfu) / mL, 도금 요소는 10. 이 숫자를 수식에 입력 됩니다.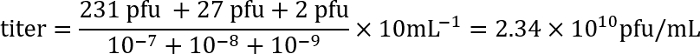
따라서,는 titer 약 2 × 1010 전염 성 살 균 소 입자/mL 이다.
3. P1 변환
-
받는 사람 세포 준비
- 받는 사람 변형 BL21ΔABCF 파운드 600에서 광학 밀도를 보충 성장 ~ 1.0 nm (OD600). OD600 값을 측정 하는 분 광 광도 계를 사용 합니다.
- 0.5의 감염 (MOI) 값의 다양성을 달성 하는 데 필요한 페이지 lysate의 볼륨을 계산 합니다. 나를 계산 하려면 문화권의 OD600 에 따라 박테리아의 수를 견적 한다. OD600 값이 1.0 ~ 109 cfu/mL에 해당 하는 가정 합니다. lysate의 알려진된 titer에 따라 필요한 볼륨을 계산 합니다.
예 (titer 단계 2.3.6 후 예제에서 계산에 따라):
- 10 m m 받는 사람 긴장 문화 CaCl2 를 추가 하 고 혼합. 변환에 대 한 문화의 1 mL를 가져가 라.
-
변환 수행
- 받는 사람 문화 (를 포함 하 여 10 m m CaCl2 ) 단계 3.1.2에서에서 계산한 1 mL를 파지 lysate의 적절 한 볼륨을 추가 하 고 부드럽게 혼합.
참고: 플라스틱 믹스에 클로 프롬을 전송 하지 않으려면 lysate의 상단에서 샘플을 주의 해야 합니다. - 정적으로 37 ° c.에 20 분을 위한 혼합을 품 어
- 100 m m 나트륨 구 연산 염, pH 5.5 추가 하 여 감염을 중지 합니다.
- 박테리아 (5000 x g 2 분) centrifuge 고 상쾌한, 다음 100 m m 나트륨 구 연산 염, pH 5.5로 보충 하는 신선한 파운드의 1 mL에 그들을 resuspend.
- 세포 단계 무료 페이지 및 칼슘의 제거를 보장 하기 위해 3.2.4 에서처럼 두 번 더 씻는 다.
- 100 m m 나트륨 구 연산 염, pH 5.5로 보충 하는 신선한 파운드의 1 mL에 박테리아 resuspend (> 100 rpm)를 흔들어 1 h 30 ° C에서 박테리아를 품 어.
- 원심 (5000 x g 2 분)에 의해 박테리아를 수집 하 고 100 m m 나트륨 구 연산 염, pH 5.5와 파운드의 ~ 100 μ에 그들을 resuspend.
- 25 µ g/mL와 10 m m 나트륨 구 연산 염, pH 5.5에서 칸으로 보충 하는 파운드 플레이트에 박테리아를 확산 하 고 식민지 (~ 24 h)이 나타날 때까지 30 ° C에서 박테리아를 성장.
- 받는 사람 문화 (를 포함 하 여 10 m m CaCl2 ) 단계 3.1.2에서에서 계산한 1 mL를 파지 lysate의 적절 한 볼륨을 추가 하 고 부드럽게 혼합.
-
transductants를 선택 하면
- 식민지는 선택 접시에 성장 했다, 일단 단일 식민지를 위한 파운드 + 칸에 그들을 restreak 단일 식민지 나타납니다까지 30 ° C에서 성장 하 고.
4. 절 개 칸 카세트
-
재조합 플라스 미드 전이
- 관 저항 BL21ΔABCF 긴장 electrocompetent를 확인 합니다.
- 신선한 파운드 (5 mL) 칸 보충에 단일 식민지에서 스트레인 성장 (25 µ g/mL) OD600 ~0.5-0.7까지 30 ° C에서.
- 여기에서에, 다음 단계 또는 얼음에 4 ° C에서 실시 합니다. Centrifuge 박테리아 (5000 x g 10 분) 하 고는 상쾌한을 제거 합니다.
- 이전 원심 분리 단계를 반복 하 여 두 번 차가운 증류수와 셀 펠 릿을 세척 하 고, 마지막으로, 얼음 10% 글리세롤의 100 µ L에서 셀 펠 릿을 resuspend.
- 1mm electroporation 큐 벳 얼음에 식혀.
- 세포 현 탁 액을 플라스 미드 DNA (pCP20)의 1 세를 추가 하 고 부드럽게, 현 탁 액을 혼합 냉각된 electroporation 베트에 그것을 전송.
- 1.8는 electroporator 설정 kV 및 electroporate 셀.
- SOC 매체의 1 mL을 추가 하 고 1 h 30 ° c.에 대 한 그들을 성장 하 여 변형 된 세포를 구출
- 파운드에 셀의 100 µ L 플레이트 + 앰프 플레이트와 하룻밤 30 ° C에서 세포를 성장.
- 관 저항 BL21ΔABCF 긴장 electrocompetent를 확인 합니다.
-
재결합의 유도
- 선택 단일 식민지 파운드 + 앰프에서 접시 하 고 접종 신선한 파운드 모든 항생제를 생략.
- FLP recombinase의 표현을 유도 하룻밤 비 허용 온도 (43 ° C)에서 세포 성장.
-
Recombinants 선택
- 직렬 희석 하 고 만들고, 비 선택적 접시에 105-106 희석의 50 µ L 플레이트 하룻밤 30 ° c.에서 그것을 성장합니다
5입니다. 유전자 삭제 확인
-
플라스 미드 손실 및 성공적인 재결합의 확인
- 격판덮개에서 행진 한 식민지 파운드 + 칸, 파운드 + 앰프, 그리고 파운드에 4.3.1 단계에서이 순서로 항생제 없이 접시 준비. 줄무늬에 투입, 식민지 표 사용 하 여 ( 보충 파일 1참조). 식민지 (~ 24 h)이 나타날 때까지 30 ° C에서 번호판을 성장.
- 비 선택적 접시에 성장 하지만 추가 확인에 대 한 선택적 미디어에 성장 하지 못했습니다 10-20 클론을 선택 합니다.
-
식민지 PCR에 의해 추가 확인
- 식민지 PCR 뇌관 순서 코딩 타 마 측면으로 수행 ( 재료의 표참조).
- 마스터 PCR 혼합을 준비 합니다. 필요 믹스 ( 표 1의 예를 참조) 테스트 하는 식민지의 수에 따라 다릅니다. 얼음에 시 약을 혼합, 마지막에 중 합 효소를 추가 합니다.
- 철저 하 게, PCR 마스터 믹스를 섞어 다음 20 µ L를 PCR 스트립의 튜브로 분배. 살 균 피 펫 팁을 사용 하 여 상영 될 각 식민지의 작은 금액을 선택 하 고 튜브를 추가. 비교를 위해 원래 받는 사람 긴장을 포함 하도록 하십시오. 선택적으로, 기증자 스트레인 포함.
- PCR 반응 실행 (사용 하는 프로그램에 대 한 표 2 참조).
- 1 %agarose 젤 준비.
- 50ml 젤 agarose의 0.5 g을 측정 하 고 태 버퍼 (40 m m Tris, 20 mM 아세트산 그리고 1 mM EDTA, pH 8.0) 50 mL를 추가 합니다. 모든 agarose 해산 했다 때까지 렌지에 혼합물을 열.
- agarose 해산 했다, 일단 약 50 ° C에 대 한 해결책을 냉각 하 고 염료를 착 색 하는 DNA의 5 μ를 추가 합니다. 솔루션을 잘 혼합 하 고 주조 챔버에 젤 트레이에 부 어. 분자 크기 마커 뿐만 아니라 모든 샘플에 대 한 충분 한 웰 스는 잘 빗의 하나 이상의 행을 삽입 합니다. 30 분 설정 하 젤을 허용 합니다.
- PCR 실행 완료 되 면 각 샘플을 DNA 로드 염료 x 6의 4 µ L를 추가 합니다. 젤 전기 이동 법 챔버에 배치 하 고 우물 덮여 때까지 태 버퍼를 추가 합니다.
- 젤에 잘 각 PCR 반응에서 10-15 µ L를 적용 합니다. 또한 분자 크기 마커의 5 µ L를 추가 합니다. 그런 다음 75 V에서 30 분 동안 젤을 실행 합니다.
- 실행 완료 되 면 젤 블루 빛 영상 장치를 사용 하 여 이미지.
6. 기타 기술
-
단백질 표정
참고: 테스트 단백질 (YadA와 EibD)의 표현 되었습니다23,24다른 곳에서 상세히 설명. 이것은 주요 단계에 대 한 간단한 요약입니다.- BL21ΔABCF Δ타 with 필요한 플라스 미드 (pIBA2-쫑 알, pEibD10 및 해당 제어 플라스 미드)를 변환 하 고 transformants 파운드 + 앰프에 대 한 선택.
- 단백질 표정에 대 한 변형 된 박테리아의 숙박 문화의 1 mL와 함께 파운드 매체 + 앰프의 100 mL를 접종 하 고 30 ° C에서이 시간에 중간 로그 단계까지 성장, (100 ng/mL)에서 어느 anhydrotetracycline와 단백질 생산을 유도 하거나 이소프로필 thiogalactoside (0.5 m m).
- 30 ° C에서 유도의 2 h 문화의 탁도 측정 하 고 OD600 에서 50 mL에 해당 하는 셀의 수를 수집 후 4000 x g에서 15 분 동안 문화 centrifuging 여 1.0 =. 펠 릿을 세척 1 x 10 mm HEPES, pH 7.4, 그리고 중-20 ° C 또는 프로세스 단계 6.2 에서처럼 추가 저장.
-
외부 막 추출
참고: 외부 막 추출 레오 외 에 의해 세부 사항에 설명 된 대로 수행 됩니다. 27. 주요 단계 아래 요약 되어 있습니다.- 10 mm HEPES, pH 7.4, resuspend 1 mL에 셀 펠 릿 10 mM MgCl2 , MnCl2, lysozyme (0.1 mg/mL), 및 DNase의 핀치 나 보충.
- (예를 들어, 구슬 때리는 사용 하 여) 셀 lyse
- 세포 파편을 제거 하 고 신선한 튜브에는 상쾌한 이동 곧 셀 (15,600 x g2 분)을 원심.
- 16000 x g에서 30 분 동안 셀 후 원심, 10 mM HEPES, pH 7.4에에서 1% N lauroyl sarcosine의 400 μ에 펠 릿을 resuspend.
- 실 온에서 30 분 동안 교 반 후 셀을 품 어, 위와 같이 30 분 동안 원심.
- 세척은 반투명 1 x 10 mm HEPES, pH 7.4, 200 μ와 작은 10 mm HEPES 30 μ에 resuspend 그리고 SDS 페이지 샘플 버퍼 x 4의 10 μ를 추가.
-
활동 분석 실험
참고: 설명28YadA와 EibD에 대 한 SDS 페이지 및 활동 분석 실험을 수행 합니다. 주요 단계는 아래에 요약 된다. -
SDS 페이지
- 단백질을 변성 시키기 방지 하려면 polyacrylamide 젤에 그들을 로드 하기 전에 5 분 동안 50 ° C에서 샘플을 열.
- SDS 페이지에서 분리 후 polyvinylidene difluoride (PVDF) 막 단백질을 전송 합니다.
- 송금 후 PBS에 2% 무 지방 우유 파우더 막을 차단 합니다.
-
뭐라 나 콜라겐 멀리 서쪽 오 점
- 차단, 후 10 μ g/mL의 농도에 블로킹 버퍼에 소 콜라겐 유형 나 희석을 추가 하 고 1 시간에 대 한 막 품 어.
- 막 2 워시 x PBS + 0.05 %Tween20 (PBS-T).
- 블로킹 버퍼에 막에 1 차적인 항 체 (단일 클로 널 항 콜라겐 COL-1), 희석된 1:2,000를 추가 합니다.
- 잠복기 1 h 막, 후 6.3.2.2 단계에서 언급 한 대로 2 배를 세척 하 고 이차 항 체 [염소 반대로 마우스 IgG-양 고추냉이 과산화 효소 (HRP) 켤레], 희석된 1:10,000 블로킹 버퍼에 추가 합니다.
- 1 시간에 대 한 막 품 어 다음 그것을 씻어 PBS. 2 x 향상 된 chemiluminescent 기판 제조업체의 지침에 따라 막에 추가 하 고 CCD 카메라를 사용 하 여 밴드를 감지.
-
EibD IgG 바인딩 분석 결과
- 차단, 이차 항 체 (염소 안티 토끼 HRP), 희석 1:2,000 블로킹 버퍼에 추가 합니다.
- 1 시간에 대 한 막 품 어 다음 그것을 씻어 PBS 가진 2 x. 6.3.2.5 단계에서 언급 한 대로 chemiluminescent 감지를 수행 합니다.
결과
세대는 타 마 노크 아웃 BL21ΔABCF의의:
위에서 설명한 전략은 이전 BL21(DE3), 외부 막 단백질 생산을 위한 최적화 이며 BL21ΔABCF21라는 단백질 생산에 사용 되는 표준 실험실 긴장의 파생 긴장을 생산 하기 위해 사용 되었습니다. 이 긴장 풍부한 외부 막 단백질을 코딩 하는 4 개의 유전자를 부족 하 고, 따라서,...
토론
P1 변환 대장균에 유전자 삭제를 생성 하기 위한 빠르고, 강력 하 고 신뢰할 수 있는 방법입니다. 이 여기 BL21 파생 된 받는 사람에 게 케이오 기증자 스트레인에서 타 마 삭제 돌연변이 시험에 의해 증명 됩니다. 변환 프로세스의 주요 단계는 lysate는 시험의 생산, 자체 변환, 칸 저항 카세트의 절단 및 PCR에 의해 녹아웃의 확인. 총에서 과정 약 1 주일 소요 하며 확인에 대 한 최종 PCR를 ?...
공개
저자는 공개 없다.
감사의 말
케이오 컬렉션 긴장 국가 유관 프로젝트 (검, 일본)에서 가져온: 대장균. 그의 지속적인 지원에 감사 더크 맺고 (생물 과학 부, 오슬로 대학교) 하 고. 이 작품은 노르웨이 젊은 연구원 부여 249793 (잭 C. 레오)의 연구 위원회에 의해 투자 되었다.
자료
| Name | Company | Catalog Number | Comments |
| Strains | |||
| E. coli BW25113 | NIG | ME6092 | Wild-type strain of Keio collection |
| E. coli BL21(DE3) | Merck | 69450-3 | Expression strain |
| E. coli BL21DABCF | Addgene | 102270 | Derived from BL21(DE3) |
| E. coli JW4179 | NIG | JW4179-KC | tamA deletion mutant |
| P1 vir | NIG | HR16 | Generally transducing bacteriophage |
| Plasmids | |||
| pCP20 | CGSC | 14177 | conditionally replicating plasmid with FLP |
| pASK-IBA2 | IBA GmbH | 2-1301-000 | expression vector |
| pEibD10 | N/A | N/A | for production of EibD; plasmid available on request |
| pET22b+ | Merck | 69744-3 | expression vector |
| pIBA2-YadA | N/A | N/A | for production of YadA; plasmid available on request |
| Chemicals | |||
| Acetic acid | ThermoFisher | 33209 | |
| Agar | BD Bacto | 214010 | |
| Agarose | Lonza | 50004 | |
| Ampicillin | Applichem | A0839 | |
| Anhydrotetracycline | Abcam | ab145350 | |
| anti-collagen type I antibody COL-1 | Sigma | C2456 | |
| Bovine collagen type I | Sigma | C9791 | |
| Calcium chloride | Merck | 102382 | |
| Chloroform | Merck | 102445 | |
| Di-sodium hydrogen phosphate | VWR | 28029 | |
| DNA dye | Thermo | S33102 | |
| DNA molecular size marker | New England BioLabs | N3232S | |
| DNase I | Sigma | DN25 | |
| dNTP mix | New England Biolabs | N0447 | |
| ECL HRP substrate | Advansta | K-12045 | |
| EDTA | Applichem | A2937 | |
| Glycerol | VWR | 24388 | |
| goat anti-mouse IgG-HRP | Santa Cruz | sc-2005 | |
| goat anti-rabbit IgG-HRP | Agrisera | AS10668 | |
| HEPES | VWR | 30487 | |
| Isopropyl thiogalactoside | VWR | 43714 | |
| Kanamycin | Applichem | A1493 | |
| Lysozyme | Applichem | A4972 | |
| Magnesium chloride | VWR | 25108 | |
| Manganese chloride | Sigma | 221279 | |
| N-lauroyl sarcosine | Sigma | L9150 | |
| Skim milk powder | Sigma | 70166 | |
| Sodium chloride | VWR | 27808 | |
| tamA forward primer | Invitrogen | N/A | Sequence 5'-GAAAAAAGG ATATTCAGGAGAAAATGTG-3' |
| tamA reverse primer | Invitrogen | N/A | Sequence 5'-TCATAATT CTGGCCCCAGACC-3' |
| Taq DNA polymerase | New England Biolabs | M0267 | |
| Tri-sodium citrate | Merck | 106448 | |
| Tryptone | VWR | 84610 | |
| Tween20 | Sigma | P1379 | |
| Yeast extract | Merck | 103753 | |
| Equipment | |||
| Agarose gel electrophoresis chamber | Hoefer | SUB13 | |
| Bead beater | Thermo | FP120A-115 | |
| CCD camera | Kodak | 4000R | |
| Electroporation cuvettes | Bio-Rad | 165-2089 | |
| Electroporation unit | Bio-Rad | 1652100 | |
| Gel imager | Nippon Genetics | GP-03LED | |
| Incubating shaker | Infors HT | Minitron | |
| Incubator | VWR | 390-0482 | |
| Microcentrifuge | Eppendorf | 5415D | |
| Microwave oven | Samsung | CM1099A | |
| PCR machine | Biometra | Tpersonal | |
| PCR strips | Axygen | PCR-0208-CP-C | |
| pH meter | Hanna Instruments | HI2211-01 | |
| PVDF membrane | ThermoFisher | 88518 | |
| SDS-PAG electrophoresis chamber | ThermoFisher | A25977 | |
| Tabletop centrifuge | Beckman Coulter | B06322 | |
| Vortex mixer | Scientific Industries | SI-0236 | |
| Water bath | GFL | D3006 | |
| Wet transfer unit | Hoefer | TE22 |
참고문헌
- Blount, Z. D. The unexhausted potential of E. coli. eLife. 4, e05826 (2015).
- Hamilton, C. M., Aldea, M., Washburn, B. K., Babitzke, P., Kushner, S. R. New method for generating deletions and gene replacements in Escherichia coli. Journal of Bacteriology. 171 (9), 4617-4622 (1989).
- Link, A. J., Phillips, D., Church, G. M. Methods for generating precise deletions and insertions in the genome of wild-type Escherichia coli: application to open reading frame characterization. Journal of Bacteriology. 179 (20), 6228-6237 (1997).
- Sawitzke, J. A., Thomason, L. C., Costantino, N., Bubunenko, M., Datta, S., Court, D. L. Recombineering: in vivo genetic engineering in E. coli, S. enterica, and beyond. Methods in Enzymology. 421, 171-199 (2007).
- Datsenko, K. A., Wanner, B. L. One-step inactivation of chromosomal genes in Escherichia coli K-12 using PCR products. Proceedings of the National Academy of Sciences of the United States of America. 97 (12), 6640-6645 (2000).
- Muyrers, J. P., Zhang, Y., Stewart, A. F. Techniques: Recombinogenic engineering--new options for cloning and manipulating DNA. Trends in Biochemical Sciences. 26 (5), 325-331 (2001).
- Baba, T., et al. Construction of Escherichia coli K-12 in-frame, single-gene knockout mutants: the Keio collection. Molecular Systems Biology. 2, (2006).
- Yamamoto, N., et al. Update on the Keio collection of Escherichia coli single-gene deletion mutants. Molecular Systems Biology. 5, 335 (2009).
- Baba, T., Huan, H. -. C., Datsenko, K., Wanner, B. L., Mori, H. The applications of systematic in-frame, single-gene knockout mutant collection of Escherichia coli K-12. Methods in Molecular Biology. 416, 183-194 (2008).
- Chattopadhyay, M. K., Tabor, C. W., Tabor, H. Polyamines are not required for aerobic growth of Escherichia coli: preparation of a strain with deletions in all of the genes for polyamine biosynthesis. Journal of Bacteriology. 191 (17), 5549-5552 (2009).
- Xie, X., Wong, W. W., Tang, Y. Improving simvastatin bioconversion in Escherichia coli by deletion of bioH. Metabolic Engineering. 9 (4), 379-386 (2007).
- Maeda, T., Sanchez-Torres, V., Wood, T. K. Escherichia coli hydrogenase 3 is a reversible enzyme possessing hydrogen uptake and synthesis activities. Applied Microbiology and Biotechnology. 76 (5), 1035-1042 (2007).
- Meehan, B. M., Landeta, C., Boyd, D., Beckwith, J. The disulfide bond formation pathway is essential for anaerobic growth of Escherichia coli. Journal of Bacteriology. 199 (16), (2017).
- Niba, E. T. E., Naka, Y., Nagase, M., Mori, H., Kitakawa, M. A genome-wide approach to identify the genes involved in biofilm formation in E. coli. DNA Research. 14 (6), 237-246 (2008).
- Heinz, E., et al. Conserved features in the structure, mechanism, and biogenesis of the inverse autotransporter protein family. Genome Biology and Evolution. 8 (6), 1690-1705 (2016).
- Selkrig, J., et al. Discovery of an archetypal protein transport system in bacterial outer membranes. Nature Structural & Molecular Biology. 19 (5), 506-510 (2012).
- Stubenrauch, C., et al. Effective assembly of fimbriae in Escherichia coli depends on the translocation assembly module nanomachine. Nature Microbiology. 1 (7), 16064 (2016).
- Leo, J. C., Goldman, A. The immunoglobulin-binding Eib proteins from Escherichia coli are receptors for IgG Fc. Molecular Immunology. 46 (8-9), 1860-1866 (2009).
- Mühlenkamp, M., Oberhettinger, P., Leo, J. C., Linke, D., Schütz, M. S. Yersinia adhesin A (YadA) - Beauty & beast. International Journal of Medical Microbiology. 305 (2), 252-258 (2015).
- Studier, F. W., Moffatt, B. A. Use of bacteriophage T7 RNA polymerase to direct selective high-level expression of cloned genes. Journal of Molecular Biology. 189 (1), 113-130 (1986).
- Meuskens, I., Michalik, M., Chauhan, N., Linke, D., Leo, J. C. A new strain collection for improved expression of outer membrane proteins. Frontiers in Cellular and Infection Microbiology. 7, 464 (2017).
- Cherepanov, P. P., Wackernagel, W. Gene disruption in Escherichia coli: TcR and KmR cassettes with the option of Flp-catalyzed excision of the antibiotic-resistance determinant. Gene. 158 (1), 9-14 (1995).
- Grosskinsky, U., et al. A conserved glycine residue of trimeric autotransporter domains plays a key role in Yersinia adhesin A autotransport. Journal of Bacteriology. 189 (24), 9011-9019 (2007).
- Leo, J. C., et al. The structure of E. coli IgG-binding protein D suggests a general model for bending and binding in trimeric autotransporter adhesins. Structure. 19 (7), 1021-1030 (1993).
- Bertani, G. Studies on lysogenesis. I. The mode of phage liberation by lysogenic Escherichia coli. Journal of Bacteriology. 62 (3), 293-300 (1951).
- Hanahan, D. Studies on transformation of Escherichia coli with plasmids. Journal of Molecular Biology. 166 (4), 557-580 (1983).
- Leo, J. C., Oberhettinger, P., Linke, D. Assessing the outer membrane insertion and folding of multimeric transmembrane β-barrel proteins. Methods in Molecular Biology. , 157-167 (2015).
- Mikula, K. M., Leo, J. C., Łyskowski, A., Kedracka-Krok, S., Pirog, A., Goldman, A. The translocation domain in trimeric autotransporter adhesins is necessary and sufficient for trimerization and autotransportation. Journal of Bacteriology. 194 (4), 827-838 (2012).
- Thomason, L. C., Costantino, N., Court, D. L., Ausubel, F. M. E. coli genome manipulation by P1 transduction. Current Protocols in Molecular Biology. , (2007).
- Franklin, N. C. Mutation in gal U gene of E. coli blocks phage P1 infection. Virology. 38 (1), 189-191 (1969).
- Jeong, H., et al. Genome sequences of Escherichia coli B strains REL606 and BL21(DE3). Journal of Molecular Biology. 394 (4), 644-652 (2009).
- Greene, E. C. DNA Sequence Alignment during Homologous Recombination. Journal of Biological Chemistry. 291 (22), 11572-11580 (2016).
- Watt, V. M., Ingles, C. J., Urdea, M. S., Rutter, W. J. Homology requirements for recombination in Escherichia coli. Proceedings of the National Academy of Sciences of the United States of America. 82 (14), 4768-4772 (1985).
- Ho, T. D., Waldor, M. K. Enterohemorrhagic Escherichia coli O157:H7 gal mutants are sensitive to bacteriophage P1 and defective in intestinal colonization. Infection and Immunity. 75 (4), 1661-1666 (2006).
- Ikeda, H., Tomizawa, J. I. Transducing fragments in generalized transduction by phage P1. I. Molecular origin of the fragments. Journal of Molecular Biology. 14 (1), 85-109 (1965).
- Murooka, Y., Harada, T. Expansion of the host range of coliphage P1 and gene transfer from enteric bacteria to other Gam-negative bacteria. Applied and Environmental Microbiology. 38 (4), 754-757 (1979).
- Goldberg, R. B., Bender, R. A., Streicher, S. L. Direct selection for P1-sensitive mutants of enteric bacteria. Journal of Bacteriology. 118 (3), 810-814 (1974).
- O'Connor, K. A., Zusman, D. R. Coliphage P1-mediated transduction of cloned DNA from Escherichia coli to Myxococcus xanthus: use for complementation and recombinational analyses. Journal of Bacteriology. 155 (1), 317-329 (1983).
재인쇄 및 허가
JoVE'article의 텍스트 или 그림을 다시 사용하시려면 허가 살펴보기
허가 살펴보기This article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. 판권 소유