È necessario avere un abbonamento a JoVE per visualizzare questo. Accedi o inizia la tua prova gratuita.
Method Article
Produzione di delezioni del Gene in Escherichia coli di trasduzione P1 con cassette di resistenza agli antibiotici soggetti ad accisa
In questo articolo
Riepilogo
Qui presentiamo un protocollo per l'uso di pre-esistente antibiotico resistenza-cassette eliminazione costrutti come base per la fabbricazione di mutanti di delezione in altri ceppi di Escherichia coli . Tali mutazioni di omissione possono essere mobilitati e inseriti il locus corrispondente di un ceppo destinatario mediante trasduzione del batteriofago P1.
Abstract
Un primo approccio per studiare la funzione di un gene sconosciuto nei batteri è quello di creare un knock-out di questo gene. Qui, descriviamo un protocollo robusto e veloce per trasferire le mutazioni di omissione del gene da un ceppo di Escherichia coli a altro mediante trasduzione generalizzata con il batteriofago P1. Questo metodo richiede che la mutazione sia selezionabile (ad es., basato su interruzioni di gene usando gli inserimenti cassetta antibiotico). Tali cassette antibiotici possono essere mobilitate da un ceppo di donatore e introdotto in un ceppo destinatario di interesse per rapidamente e facilmente generano un mutante di delezione del gene. La cassetta di antibiotica può essere progettata per includere siti di riconoscimento flippase che consentono l'asportazione della cassetta da una ricombinasi site-specific per produrre un knock-out pulito con solo una sequenza di ~ 100-base-coppia-lunga cicatrice nel genoma. Dimostriamo il protocollo battendo fuori il tamA gene codifica per un fattore di assemblaggio coinvolto nella biogenesi autotransporter e testare l'effetto di questo knock-out sulla biogenesi e funzione di due trimerico autotransporter adesine. Sebbene l'omissione del gene di trasduzione P1 ha i suoi limiti, la facilità e la velocità della sua attuazione rendono una valida alternativa ad altri metodi di omissione del gene.
Introduzione
Un primo approccio comune per studiare la funzione di un gene è quello di eseguire la mutagenesi di knock-out e osservare il fenotipo risultante. Questo è definito anche genetica inversa. Il batterio e. coli è stato il cavallo di battaglia della biologia molecolare negli ultimi 70 anni o così, a causa della facilità della sua coltura e la sua disponibilità a manipolazione genetica1. Sono stati sviluppati diversi metodi per produrre le eliminazioni genica in Escherichia coli, compreso indicatore cambio mutagenesi2,3 e, più recentemente, recombineering utilizzando il λ rosso o Rac ET sistemi4,5 , 6.
In un sistema ampiamente usato, sequenze di codificazione dei geni individuali vengono sostituiti da una cassetta di resistenza agli antibiotici che possa successivamente essere asportata dal cromosoma5,7. Le sequenze di codificazione sono sostituite, per esempio da una cassetta di resistenza alla kanamicina (Kan), fiancheggiata da siti di destinazione (FRT) riconoscimento flippase (FLP) su entrambi i lati. I siti FRT sono riconosciuti dalla ricombinasi FLP, che media site-specific di ricombinazione tra i siti FRT che conduce all'eliminazione della cassetta Kan. In questo modo, è possibile una cancellazione completa della sequenza codificante di un dato gene, lasciando dietro di sé solo una sequenza di cicatrice minima di circa 100 coppie di basi (bp) (Figura 1).
Poco più di un decennio fa, è stata sviluppata il cosiddetta collezione di Keio. Si tratta di una libreria batterica basata su un ceppo di e. coli K12, standard di laboratorio dove quasi tutti i geni non essenziali sono stati eliminati singolarmente da λ ricombinazione rosso7,8. I cloni all'interno della raccolta ogni hanno una sequenza di codificazione sostituito con una cassetta di resistenza Kan soggetti ad accisa. La collezione di Keio ha dimostrato di essere uno strumento utile per molte applicazioni9. Una tale applicazione è la produzione di mutanti di delezione in altri ceppi di Escherichia coli . La cassetta di Kan da un clone di eliminazione specificato possa essere mobilitata da generalmente transducing batteriofagi, ad esempio P110,11,12,13,14. Un brodo di fago preparato da una simile tensione quindi può essere utilizzato per infettare un destinatario Escherichia coli ceppo di interesse, dove ad una frequenza bassa ma affidabile il Kan regione contenente cassette possa essere incorporate nel genoma del destinatario tramite la ricombinazione omologa (Figura 2). Transductants possono essere selezionati per la crescita sul supporto di Kan-contenente. In seguito, se si desidera la rimozione della cassetta di resistenza agli antibiotici, la ricombinasi FLP possono essere forniti al ceppo transductant in trans. Dopo l'indurimento il plasmide contenente FLP, che trasporta un marcatore di resistenza all'ampicillina (Amp), Kan e Amp-sensibile cloni sono schermati per, e la corretta asportazione del selvaggio-tipo sequenza di codificazione e la cassetta di Kan sono verificate mediante PCR della Colonia.
Qui, un protocollo dettagliato è presentato, che descrive tutti i passaggi nella produzione di un knock-out ceppo di e. coli sulla base della strategia descritta sopra. Ad esempio, è dimostrata un'omissione del gene di tamA . tamA codifica per una proteina del β-barilotto di membrana esterna che è una parte del trasporto e montaggio modulo (TAM), che è coinvolto nella biogenesi di determinate proteine autotransporter e pili15,16,17. Questo ceppo di knock-out è stato poi utilizzato per esaminare l'effetto dell'eliminazione tamA sulla biogenesi di due trimerico autotransporter adesine (TAA), l'adesina di Yersinia YadA e l'immunoglobulina (Ig) di Escherichia coli -associazione TAA EibD 18,19.
Protocollo
1. ceppi e plasmidi
-
Ceppi batterici
- Utilizzare l'e. coli ceppi BW251135, JW4179 (BW25113 tamA::kan)7, BL21 (DE3)20e BL21ΔABCF21. Per ulteriori informazioni, vedere Tabella materiali .
-
Batteriofagi
- Utilizzare il fago P1vir per la trasduzione generale. Memorizzare il fago come un brodo liquido con poche gocce di cloroformio (Vedi punto 2.2). Per ulteriori informazioni, vedere Tabella materiali.
-
Plasmidi
- Utilizzare i plasmidi seguenti in questo protocollo: pCP2022, pIBA2-YadA23e pEibD1024. Come plasmidi di controllo, utilizzare pASK-IBA2 e pET22b (Vedi Tabella materiali).
-
Condizioni di crescita
- Propagare i batteri in Lisogenesi brodo (LB) medio25 con agitazione vigorosa (180 – 200 giri/min) a 37 ° C o 30 ° C nel caso di BL21ΔABCF e pCP20 dei ceppi.
- Eseguire il plasmide indurimento a 43 ° C.
- Per un mezzo solido, supplemento LB con agar 1% (p/v).
- Per top agar, supplemento LB con 0,7% agar e 10 mM CaCl2 ed autoclave il mezzo. Utilizzo medio SOC per il recupero dopo l'elettroporazione26.
- Utilizzare le seguenti concentrazioni di antibiotici: 100 μg/mL per Amp e 25 μg/mL per kan.
2. preparare un fago lisato
-
Infezione del ceppo donatore
- Crescere il ceppo donatore JW4197 in 5 mL di LB medium supplementato con 10 mM di CaCl2 e, facoltativamente, con Kan (25 µ g/mL) di densità ottica a 600 nm (OD600) di ~ 1.0. Misurare il valore di600 OD utilizzando uno spettrofotometro.
- Fare una serie di diluizioni di uno stock di fago P1 esistente nel medio LB: consigliato diluizioni sono tra 10-3 a 10-7.
- Mix 200 μL della sospensione batterica e 100 μL di una diluizione dei fagi dato in una provetta da centrifuga da 15 mL o equivalente. Preparare le provette come molti come diluizioni dei fagi. Incubare le provette per 20 min a 37 ° C senza agitazione.
- Aggiungere circa 3 mL di agar superiore fusa (~ 50 ° C) completati con 10 mM di CaCl2 ai tubi, mescolare il contenuto accuratamente nel Vortex le provette poco e versare le miscele sulle piastre LB preriscaldate a fare strati anche.
- Incubare le piastre durante la notte a 37 ° C.
-
Preparazione del lysate
- Il giorno seguente scegliere un piatto con un semi-confluente crescita delle placche dei fagi. Su una piastra semi-confluente, circa metà della superficie della piastra è chiara (Figura 3).
- Raschiare lo strato di agar superiore da tale piastra utilizzando un ciclo di inoculazione o uno strumento simile e posizionare l'agar superiore in una provetta da centrifuga. Aggiungere 1 – 2 mL di LB e una goccia di cloroformio e vortexare la provetta vigorosamente per ~ 1 min. Aggiungi il cloroformio in una cappa aspirante.
- Centrifugare la provetta per 15 min a 4.000 x g o più veloce per agglomerare le cellule batteriche e agar.
- Spostare il surnatante in una microcentrifuga fresca, evitando di trasportare più di eventuali detriti dal pellet. Aggiungere 2 gocce di cloroformio e memorizzare il lisato 4 – 10 ° c. Aggiungere cloroformio in una cappa aspirante. Non congelare il fago lisato come questo si tradurrà in una riduzione significativa del numero di particelle infettive.
-
Determinare il titolo lisato
- Crescere BW25113 in LB completati con 10 mM di CaCl2 a 37 ° C fino a quando la cultura raggiunge un OD600 di ~ 1.0.
- Preparare una serie di diluizioni del fago lisato in LB (ad es., 10-6-10-9).
Nota: Fare attenzione a dispensare il campione dalla parte superiore del lisato per evitare di trasferire il cloroformio per le diluizioni. - Mix 200 μL della sospensione batterica e 100 μL di una diluizione dei fagi dato in una provetta da centrifuga da 15 mL o equivalente. Preparare le provette come molti come diluizioni dei fagi. Incubare le provette per 20 min a 37 ° C senza agitazione.
- Aggiungere circa 3 mL di agar superiore fusa (~ 50 ° C) completati con 10 mM di CaCl2 ai tubi, mescolare il contenuto accuratamente nel Vortex le provette poco e versare il composto su piatti preriscaldati della libbra a fare strati anche.
- Incubare le piastre durante la notte a 37 ° C.
- Il giorno seguente contare il numero delle placche (regioni libere nella stuoia batterica) per ogni piatto e calcolare il titolo del lisato utilizzando la seguente formula:

Esempio: Su piastre con le diluizioni 10-7, 10-8e 10-9, ci sono 231, 27 e 2 placche, rispettivamente. Come 100 μL di ogni diluizione è stato usato per l'infezione, e perché il titolo è espresso come diformazione unità (pfu) / mL, il fattore di placcatura è 10. Questi numeri vengono inseriti nella formula: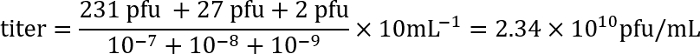
Così, il titolo è di circa 2 x 1010 infettiva dei fagi particelle/mL.
3. P1 trasduzione
-
Preparare le cellule recettive
- Crescere il destinatario ceppo BL21ΔABCF in LB completati a densità ottica a 600 nm (OD600) di ~ 1.0. Utilizzare uno spettrofotometro per misurare il valore di600 OD.
- Calcolare il volume del fago lisato necessario per raggiungere una molteplicità di valore di infezione (MOI) pari a 0,5. Per calcolare il MOI, stimare il numero di batteri basata sulla OD600 della cultura. Si supponga che un valore di600 OD di 1.0 corrisponde a ~ 109 cfu/mL. Calcolare il volume richiesto basato sul noto titolo del lisato.
Esempio (basato sul titolo calcolato nell'esempio dopo passo 2.3.6):
- Aggiungere CaCl2 alla cultura destinatario ceppo a 10 mM e mescolare. Prelevare 1 mL della cultura per la trasduzione.
-
Esecuzione di trasduzione
- Aggiungere il volume appropriato di lisato dei fagi a 1 mL di coltura destinatario (compreso CaCl2 a 10 mM) calcolata al punto 3.1.2 e mescolare delicatamente.
Nota: Fare attenzione a dispensare il campione dalla parte superiore del lisato per evitare di trasferire cloroformio al mix. - Incubare in modo statico il mix per 20 min a 37 ° C.
- Fermare l'infezione con l'aggiunta di citrato di sodio, pH 5.5, a 100 mM.
- Centrifugare i batteri (5.000 x g per 2 min) e rimuovere il supernatante, quindi li risospendere in 1 mL di LB fresco completati con citrato di sodio 100 mM, pH 5.5.
- Lavare le cellule due volte più come descritto al punto 3.2.4 per garantire la rimozione di fagi gratis e di calcio.
- Risospendere i batteri in 1 mL di LB fresco completati con citrato di sodio 100 mM, pH 5.5. Incubare i batteri a 30 ° C per 1 h con agitazione (> 100 giri/min).
- Raccogliere i batteri mediante centrifugazione (5.000 x g per 2 min) e li risospendere in ~ 100 μL di LB con citrato di sodio 100 mM, pH 5.5.
- Diffondere i batteri su una piastra di LB completati con Kan 25 µ g/mL e citrato di sodio di 10 mM, pH 5.5 e crescere i batteri a 30 ° C fino a quando le colonie apparire (~ 24 h).
- Aggiungere il volume appropriato di lisato dei fagi a 1 mL di coltura destinatario (compreso CaCl2 a 10 mM) calcolata al punto 3.1.2 e mescolare delicatamente.
-
Selezionando il transductants
- Una volta colonie cresciute sulla piastra di selezione, li restreak su LB + Kan per singole colonie e crescono a 30 ° C fino a singole colonie appaiono.
4. asportare la cassetta Kan
-
La trasformazione con il plasmide di ricombinazione
- Rendere il electrocompetent del ceppo di BL21ΔABCF Kan-resistente.
- Crescere il ceppo da una singola Colonia in fresco LB (5 mL) completati con Kan (25 µ g/mL) a 30 ° C fino a quando il OD600 è ~0.5 – 0,7.
- Da qui, procedere nel modo seguente a 4 ° C o su ghiaccio. Centrifugare i batteri (5.000 x g per 10 min) e rimuovere il surnatante.
- Lavare il pellet cellulare con gelida acqua distillata due volte ripetendo il passaggio precedente di centrifugazione e, infine, risospendere le cellule in 100 µ l di gelida glicerolo al 10%.
- Raffreddare le provette di elettroporazione di 1 mm sul ghiaccio.
- Aggiungere 1 pg del plasmide DNA (pCP20) per la sospensione di eritrociti, mescolare delicatamente la sospensione e trasferirlo in una provetta di elettroporazione raffreddato.
- Impostare il electroporator a 1,8 kV ed electroporate le cellule.
- Salvataggio di cellule trasformate aggiungendo 1 mL di terreno SOC e coltivarle per 1 h a 30 ° C.
- Piastra di 100 µ l di cellule su LB + Amp piastre e crescere le cellule a 30 ° C durante la notte.
- Rendere il electrocompetent del ceppo di BL21ΔABCF Kan-resistente.
-
Induzione della ricombinazione
- Pick singole colonie dal LB + Amp piastra e inoculare LB fresco omettendo tutti gli antibiotici.
- Crescere le cellule alla temperatura non permissivi (43 ° C) durante la notte per indurre l'espressione della ricombinasi FLP.
-
Selezionando ricombinanti
- Effettuare diluizioni seriali e piastra 50 µ l di una diluizione di6 105-10 su piastre non selettivi e crescere in una notte a 30 ° C.
5. Verifica della delezione genica
-
Verifica della perdita del plasmide e ricombinazione di successo
- Colonie di singola striscia da piastre preparate al punto 4.3.1 su Kan, LB, LB + Amp e piatti della libbra senza antibiotici, in questo ordine. Per facilitare le striature, utilizzare una griglia di Colonia (vedere complementare File 1). Crescere le piastre a 30 ° C fino a quando le colonie apparire (~ 24 h).
- Prendere 10 – 20 cloni che hanno cresciuto sulla piastra non selettiva, ma non è riuscito a crescere su terreni selettivi per ulteriori verifiche.
-
Verifica aggiuntiva di PCR della Colonia
- Eseguire una colonia PCR con primer che fiancheggiano il tamA sequenza di codificazione (Vedi Tabella materiali).
- Preparare un mix PCR master. La quantità di miscela necessaria dipende dal numero delle colonie da testare (vedere l'esempio in tabella 1). Mescolare i reagenti sul ghiaccio, aggiungere la polimerasi Ultima.
- Mescolare accuratamente il PCR master mix, quindi dispensare 20 µ l in tubi di una striscia PCR. Prendere una piccola quantità di ogni Colonia sarà proiettato utilizzando una pipetta sterile e aggiungerlo ad un tubo. Ricordati di includere il ceppo originario di destinatario per il confronto. Facoltativamente, includono anche il ceppo di donatore.
- Eseguire una reazione di PCR (Vedi tabella 2 per il programma utilizzato).
- Preparare un gel di agarosio all'1%.
- Per un gel 50 mL, 0,5 g di agarosio di misurare e aggiungere 50 mL di tampone TAE (40 mM Tris, acido acetico 20 mM e 1 mM EDTA, pH 8.0). Riscaldare la miscela in un forno a microonde fino a sciolta tutti i agarosio.
- Una volta che ha dissolto l'agarosio, raffreddare la soluzione a circa 50 ° C e quindi aggiungere 5 μL di DNA che macchia di tintura. Mescolare bene la soluzione e versarlo in un vassoio di gel in una camera di fusione. Inserire una o più righe di pettini bene in modo che ci sono pozzi sufficiente per tutti i campioni, così come marcatori di dimensioni molecolari. Lasciare che il gel da impostare per 30 min.
- Una volta che viene completata l'esecuzione PCR, aggiungere 4 µ l di 6 x tintura di caricamento del DNA di ciascun campione. Mettere il gel nella camera di elettroforesi e aggiungere tampone TAE finche ' i pozzi sono coperti.
- Applicare 10 – 15 µ l da ogni reazione di PCR ad un pozzo nel gel. Anche aggiungere 5 µ l di marcatore molecolare. Quindi, eseguire il gel per 30 min a 75 V.
- Una volta che viene completata l'esecuzione, l'immagine del gel usando un imager di luce blu.
6. altre tecniche
-
Espressione della proteina
Nota: L'espressione delle proteine di prova (YadA ed EibD) è stato descritto dettagliatamente altrove23,24. Si tratta di un breve riassunto delle principali operazioni.- Trasformare BL21ΔABCF ΔtamA with i plasmidi necessari (pIBA2-bla e pEibD10 e i corrispondente plasmidi di controllo) e selezionare per trasformanti su LB + Amp.
- Per l'espressione della proteina, inoculare 100 mL di LB medium + Amp con 1 mL di una coltura durante la notte di batteri trasformati e crescere questi a 30 ° C fino al medio-registro fase, momento in cui, inducono la produzione di proteine con entrambi anidrotetraciclina (a 100 ng/mL) o isopropilico thiogalactoside (a 0,5 mM).
- Dopo 2 h di induzione a 30 ° C, misurare la torbidità delle culture e raccogliere un numero di celle corrispondenti a 50 mL al OD600 = 1.0 mediante centrifugazione le culture per 15 min a 4.000 x g. Lavare la pallina 1x con 10 mM HEPES, pH 7.4 e quindi sia conservare a-20 ° C o processo esso più ulteriormente come descritto al punto 6.2.
-
Estrazione della membrana esterna
Nota: L'estrazione della membrana esterna è effettuata come descritto in dettaglio da Leo et al. 27. i passi principali sono riassunte di seguito.- Risospendere il pellet cellulare in 1 mL di 10 mM HEPES, pH 7.4, completati con 10mm MgCl2 e MnCl2, lisozima (0,1 mg/mL) e un pizzico di dnasi I.
- Lisare le cellule (per esempio, utilizzando un battitore di tallone).
- Centrifugare le cellule poco (2 min a 15.600 x g) per rimuovere i detriti cellulari e spostare il surnatante in una nuova provetta.
- Centrifugare le cellule per 30 min a 16.000 x g, dopo di che, risospendere il pellet in 400 μL di 1% N-lauroyl sarcosine in 10 mM HEPES, pH 7,4.
- Incubare le cellule con agitazione per 30 min a temperatura ambiente, dopo di che, li Centrifugare per 30 min come sopra.
- Lavare il traslucido a pellet 1x con 200 μL di 10 mM HEPES, pH 7.4 e quindi esso risospenda in 30 μL di 10 mM HEPES e aggiunga 10 μL di tampone di SDS-PAGE: 4x.
-
Analisi di attività
Nota: Eseguire analisi SDS-PAGE e di attività per YadA ed EibD come descritto28. I passaggi principali sono riassunte di seguito. -
SDS-PAGE
- Riscaldare i campioni a 50 ° C per 5 min prima di caricarle su un gel di poliacrilammide, per evitare di denaturare le proteine.
- Dopo la separazione in SDS-PAGE, è possibile trasferire le proteine ad una membrana di polivinilidene difluoruro (PVDF).
- Dopo il trasferimento, bloccare la membrana con latte scremato in polvere 2% in PBS.
-
Macchia occidentale lontano YadA-collagene
- Dopo il blocco, aggiungere il tipo di collagene bovino che diluito in tampone bloccante a una concentrazione di 10 μg/mL e incubare la membrana per 1 h.
- Lavare la membrana 2 x con PBS + 0.05% Tween20 (PBS-T).
- Aggiungere l'anticorpo primario (monoclonale anti-collagene COL-1) alla membrana, 1:2,000 diluito in tampone bloccante.
- Dopo incubazione la membrana per 1 h, lavare 2x come indicato al punto 6.3.2.2 e quindi aggiungere l'anticorpo secondario [coniugato con perossidasi (HRP) di capra anti-topo IgG-rafano], diluito 1: 10.000 in tampone bloccante.
- Incubare la membrana per 1 h, poi lavarlo 2 volte con PBS-T. Aggiungere un substrato chemiluminescente avanzato alla membrana secondo le istruzioni del produttore e rilevare la band utilizzando una telecamera CCD.
-
EibD-IgG analisi obbligatoria
- Dopo il blocco, aggiungere un anticorpo secondario (capra anti-coniglio HRP), diluito 1:2,000 in tampone bloccante.
- Incubare la membrana per 1 h, poi lavarlo 2 volte con PBS. Eseguire la rilevazione chemiluminescente come indicato al punto 6.3.2.5.
Risultati
Generazione di un tamA Knock-out di BL21ΔABCF:
La strategia delineata sopra è stata utilizzata in precedenza per produrre un ceppo derivato di BL21 (DE3), un ceppo di laboratorio standard utilizzato per la produzione di proteine, che è ottimizzata per la produzione di proteine di membrana esterna e denominata BL21ΔABCF21. Questo ceppo non dispone di quattro geni che codificano per proteine della...
Discussione
La trasduzione P1 è un metodo veloce, robusto e affidabile per la generazione di delezioni del gene dentro e. coli. Questo è dimostrato qui di trasdurre un mutante di delezione di tamA da un ceppo di donatore Keio a un destinatario BL21-derivato. Le principali fasi del processo di trasduzione sono la produzione della trasduzione lisata, trasduzione del sé, l'asportazione della cassetta Kan resistenza e per la verifica di knock-out mediante PCR. In totale, il processo richiede circa 1 settimana e non ...
Divulgazioni
Gli autori non hanno nulla a rivelare.
Riconoscimenti
Ceppi di Keio collezione sono stati ottenuti dal progetto nazionale BioResource (NIG, Giappone): e. coli. Ringraziamo Dirk Linke (dipartimento di scienze biologiche, Università di Oslo) per il suo continuo sostegno. Questo lavoro è stato finanziato dal Consiglio di ricerca della Norvegia Young Researcher grant 249793 (per Jack C. Leo).
Materiali
| Name | Company | Catalog Number | Comments |
| Strains | |||
| E. coli BW25113 | NIG | ME6092 | Wild-type strain of Keio collection |
| E. coli BL21(DE3) | Merck | 69450-3 | Expression strain |
| E. coli BL21DABCF | Addgene | 102270 | Derived from BL21(DE3) |
| E. coli JW4179 | NIG | JW4179-KC | tamA deletion mutant |
| P1 vir | NIG | HR16 | Generally transducing bacteriophage |
| Plasmids | |||
| pCP20 | CGSC | 14177 | conditionally replicating plasmid with FLP |
| pASK-IBA2 | IBA GmbH | 2-1301-000 | expression vector |
| pEibD10 | N/A | N/A | for production of EibD; plasmid available on request |
| pET22b+ | Merck | 69744-3 | expression vector |
| pIBA2-YadA | N/A | N/A | for production of YadA; plasmid available on request |
| Chemicals | |||
| Acetic acid | ThermoFisher | 33209 | |
| Agar | BD Bacto | 214010 | |
| Agarose | Lonza | 50004 | |
| Ampicillin | Applichem | A0839 | |
| Anhydrotetracycline | Abcam | ab145350 | |
| anti-collagen type I antibody COL-1 | Sigma | C2456 | |
| Bovine collagen type I | Sigma | C9791 | |
| Calcium chloride | Merck | 102382 | |
| Chloroform | Merck | 102445 | |
| Di-sodium hydrogen phosphate | VWR | 28029 | |
| DNA dye | Thermo | S33102 | |
| DNA molecular size marker | New England BioLabs | N3232S | |
| DNase I | Sigma | DN25 | |
| dNTP mix | New England Biolabs | N0447 | |
| ECL HRP substrate | Advansta | K-12045 | |
| EDTA | Applichem | A2937 | |
| Glycerol | VWR | 24388 | |
| goat anti-mouse IgG-HRP | Santa Cruz | sc-2005 | |
| goat anti-rabbit IgG-HRP | Agrisera | AS10668 | |
| HEPES | VWR | 30487 | |
| Isopropyl thiogalactoside | VWR | 43714 | |
| Kanamycin | Applichem | A1493 | |
| Lysozyme | Applichem | A4972 | |
| Magnesium chloride | VWR | 25108 | |
| Manganese chloride | Sigma | 221279 | |
| N-lauroyl sarcosine | Sigma | L9150 | |
| Skim milk powder | Sigma | 70166 | |
| Sodium chloride | VWR | 27808 | |
| tamA forward primer | Invitrogen | N/A | Sequence 5'-GAAAAAAGG ATATTCAGGAGAAAATGTG-3' |
| tamA reverse primer | Invitrogen | N/A | Sequence 5'-TCATAATT CTGGCCCCAGACC-3' |
| Taq DNA polymerase | New England Biolabs | M0267 | |
| Tri-sodium citrate | Merck | 106448 | |
| Tryptone | VWR | 84610 | |
| Tween20 | Sigma | P1379 | |
| Yeast extract | Merck | 103753 | |
| Equipment | |||
| Agarose gel electrophoresis chamber | Hoefer | SUB13 | |
| Bead beater | Thermo | FP120A-115 | |
| CCD camera | Kodak | 4000R | |
| Electroporation cuvettes | Bio-Rad | 165-2089 | |
| Electroporation unit | Bio-Rad | 1652100 | |
| Gel imager | Nippon Genetics | GP-03LED | |
| Incubating shaker | Infors HT | Minitron | |
| Incubator | VWR | 390-0482 | |
| Microcentrifuge | Eppendorf | 5415D | |
| Microwave oven | Samsung | CM1099A | |
| PCR machine | Biometra | Tpersonal | |
| PCR strips | Axygen | PCR-0208-CP-C | |
| pH meter | Hanna Instruments | HI2211-01 | |
| PVDF membrane | ThermoFisher | 88518 | |
| SDS-PAG electrophoresis chamber | ThermoFisher | A25977 | |
| Tabletop centrifuge | Beckman Coulter | B06322 | |
| Vortex mixer | Scientific Industries | SI-0236 | |
| Water bath | GFL | D3006 | |
| Wet transfer unit | Hoefer | TE22 |
Riferimenti
- Blount, Z. D. The unexhausted potential of E. coli. eLife. 4, e05826 (2015).
- Hamilton, C. M., Aldea, M., Washburn, B. K., Babitzke, P., Kushner, S. R. New method for generating deletions and gene replacements in Escherichia coli. Journal of Bacteriology. 171 (9), 4617-4622 (1989).
- Link, A. J., Phillips, D., Church, G. M. Methods for generating precise deletions and insertions in the genome of wild-type Escherichia coli: application to open reading frame characterization. Journal of Bacteriology. 179 (20), 6228-6237 (1997).
- Sawitzke, J. A., Thomason, L. C., Costantino, N., Bubunenko, M., Datta, S., Court, D. L. Recombineering: in vivo genetic engineering in E. coli, S. enterica, and beyond. Methods in Enzymology. 421, 171-199 (2007).
- Datsenko, K. A., Wanner, B. L. One-step inactivation of chromosomal genes in Escherichia coli K-12 using PCR products. Proceedings of the National Academy of Sciences of the United States of America. 97 (12), 6640-6645 (2000).
- Muyrers, J. P., Zhang, Y., Stewart, A. F. Techniques: Recombinogenic engineering--new options for cloning and manipulating DNA. Trends in Biochemical Sciences. 26 (5), 325-331 (2001).
- Baba, T., et al. Construction of Escherichia coli K-12 in-frame, single-gene knockout mutants: the Keio collection. Molecular Systems Biology. 2, (2006).
- Yamamoto, N., et al. Update on the Keio collection of Escherichia coli single-gene deletion mutants. Molecular Systems Biology. 5, 335 (2009).
- Baba, T., Huan, H. -. C., Datsenko, K., Wanner, B. L., Mori, H. The applications of systematic in-frame, single-gene knockout mutant collection of Escherichia coli K-12. Methods in Molecular Biology. 416, 183-194 (2008).
- Chattopadhyay, M. K., Tabor, C. W., Tabor, H. Polyamines are not required for aerobic growth of Escherichia coli: preparation of a strain with deletions in all of the genes for polyamine biosynthesis. Journal of Bacteriology. 191 (17), 5549-5552 (2009).
- Xie, X., Wong, W. W., Tang, Y. Improving simvastatin bioconversion in Escherichia coli by deletion of bioH. Metabolic Engineering. 9 (4), 379-386 (2007).
- Maeda, T., Sanchez-Torres, V., Wood, T. K. Escherichia coli hydrogenase 3 is a reversible enzyme possessing hydrogen uptake and synthesis activities. Applied Microbiology and Biotechnology. 76 (5), 1035-1042 (2007).
- Meehan, B. M., Landeta, C., Boyd, D., Beckwith, J. The disulfide bond formation pathway is essential for anaerobic growth of Escherichia coli. Journal of Bacteriology. 199 (16), (2017).
- Niba, E. T. E., Naka, Y., Nagase, M., Mori, H., Kitakawa, M. A genome-wide approach to identify the genes involved in biofilm formation in E. coli. DNA Research. 14 (6), 237-246 (2008).
- Heinz, E., et al. Conserved features in the structure, mechanism, and biogenesis of the inverse autotransporter protein family. Genome Biology and Evolution. 8 (6), 1690-1705 (2016).
- Selkrig, J., et al. Discovery of an archetypal protein transport system in bacterial outer membranes. Nature Structural & Molecular Biology. 19 (5), 506-510 (2012).
- Stubenrauch, C., et al. Effective assembly of fimbriae in Escherichia coli depends on the translocation assembly module nanomachine. Nature Microbiology. 1 (7), 16064 (2016).
- Leo, J. C., Goldman, A. The immunoglobulin-binding Eib proteins from Escherichia coli are receptors for IgG Fc. Molecular Immunology. 46 (8-9), 1860-1866 (2009).
- Mühlenkamp, M., Oberhettinger, P., Leo, J. C., Linke, D., Schütz, M. S. Yersinia adhesin A (YadA) - Beauty & beast. International Journal of Medical Microbiology. 305 (2), 252-258 (2015).
- Studier, F. W., Moffatt, B. A. Use of bacteriophage T7 RNA polymerase to direct selective high-level expression of cloned genes. Journal of Molecular Biology. 189 (1), 113-130 (1986).
- Meuskens, I., Michalik, M., Chauhan, N., Linke, D., Leo, J. C. A new strain collection for improved expression of outer membrane proteins. Frontiers in Cellular and Infection Microbiology. 7, 464 (2017).
- Cherepanov, P. P., Wackernagel, W. Gene disruption in Escherichia coli: TcR and KmR cassettes with the option of Flp-catalyzed excision of the antibiotic-resistance determinant. Gene. 158 (1), 9-14 (1995).
- Grosskinsky, U., et al. A conserved glycine residue of trimeric autotransporter domains plays a key role in Yersinia adhesin A autotransport. Journal of Bacteriology. 189 (24), 9011-9019 (2007).
- Leo, J. C., et al. The structure of E. coli IgG-binding protein D suggests a general model for bending and binding in trimeric autotransporter adhesins. Structure. 19 (7), 1021-1030 (1993).
- Bertani, G. Studies on lysogenesis. I. The mode of phage liberation by lysogenic Escherichia coli. Journal of Bacteriology. 62 (3), 293-300 (1951).
- Hanahan, D. Studies on transformation of Escherichia coli with plasmids. Journal of Molecular Biology. 166 (4), 557-580 (1983).
- Leo, J. C., Oberhettinger, P., Linke, D. Assessing the outer membrane insertion and folding of multimeric transmembrane β-barrel proteins. Methods in Molecular Biology. , 157-167 (2015).
- Mikula, K. M., Leo, J. C., Łyskowski, A., Kedracka-Krok, S., Pirog, A., Goldman, A. The translocation domain in trimeric autotransporter adhesins is necessary and sufficient for trimerization and autotransportation. Journal of Bacteriology. 194 (4), 827-838 (2012).
- Thomason, L. C., Costantino, N., Court, D. L., Ausubel, F. M. E. coli genome manipulation by P1 transduction. Current Protocols in Molecular Biology. , (2007).
- Franklin, N. C. Mutation in gal U gene of E. coli blocks phage P1 infection. Virology. 38 (1), 189-191 (1969).
- Jeong, H., et al. Genome sequences of Escherichia coli B strains REL606 and BL21(DE3). Journal of Molecular Biology. 394 (4), 644-652 (2009).
- Greene, E. C. DNA Sequence Alignment during Homologous Recombination. Journal of Biological Chemistry. 291 (22), 11572-11580 (2016).
- Watt, V. M., Ingles, C. J., Urdea, M. S., Rutter, W. J. Homology requirements for recombination in Escherichia coli. Proceedings of the National Academy of Sciences of the United States of America. 82 (14), 4768-4772 (1985).
- Ho, T. D., Waldor, M. K. Enterohemorrhagic Escherichia coli O157:H7 gal mutants are sensitive to bacteriophage P1 and defective in intestinal colonization. Infection and Immunity. 75 (4), 1661-1666 (2006).
- Ikeda, H., Tomizawa, J. I. Transducing fragments in generalized transduction by phage P1. I. Molecular origin of the fragments. Journal of Molecular Biology. 14 (1), 85-109 (1965).
- Murooka, Y., Harada, T. Expansion of the host range of coliphage P1 and gene transfer from enteric bacteria to other Gam-negative bacteria. Applied and Environmental Microbiology. 38 (4), 754-757 (1979).
- Goldberg, R. B., Bender, R. A., Streicher, S. L. Direct selection for P1-sensitive mutants of enteric bacteria. Journal of Bacteriology. 118 (3), 810-814 (1974).
- O'Connor, K. A., Zusman, D. R. Coliphage P1-mediated transduction of cloned DNA from Escherichia coli to Myxococcus xanthus: use for complementation and recombinational analyses. Journal of Bacteriology. 155 (1), 317-329 (1983).
Ristampe e Autorizzazioni
Richiedi autorizzazione per utilizzare il testo o le figure di questo articolo JoVE
Richiedi AutorizzazioneThis article has been published
Video Coming Soon