Bu içeriği görüntülemek için JoVE aboneliği gereklidir. Oturum açın veya ücretsiz deneme sürümünü başlatın.
Method Article
Escherichia coli Excisable antibiyotik direnci kasetleri ile P1 iletim tarafından üreten gen silme
Bu Makalede
Özet
Burada bir protokol antibiyotik direnç-kaset silme yapıları silme mutantlar diğer E. coli suşları yapmak için bir temel olarak önceden var olan kullanım için mevcut. Böyle silme mutasyonlar seferber ve büyük bir alıcı yük P1 bakteriyofaj iletim kullanarak odağı karşılık gelen eklenmiş.
Özet
Bakterilerde bilinmeyen bir gen işlevini incelemek için bir ilk yaklaşım bir knock-out bu gen oluşturmaktır. Burada, bakteriyofaj P1 ile jeneralize iletim kullanarak gen silme mutasyonlar bir Escherichia coli zorlanma diğerine aktarmak için sağlam ve hızlı bir iletişim kuralı açıklar. Bu yöntem mutasyon (antibiyotik kaset eklemeleri kullanarak gen kesintileri göre seçilebilirÖrneğin, ) olmasını gerektirir. Bu tür antibiyotik kaset bir donör yük seferber ve hızlı bir şekilde ilgi alıcı bir gerilim girmiştir ve kolayca bir gen silme mutant oluşturmak. Antibiyotik kaset kaset eksizyon izin flippase tanıma siteleri dahil etmek için temiz bir knock-out ile genom bir ~ 100 baz çifti uzun yara sırasına üretmek için siteye özgü bir recombinase tarafından dizayn edilebilir. Protokol autotransporter Biyogenez ilgili bir derleme faktör kodlama tamA gene dışarı knocking göstermek ve bu knock-out Biyogenez üzerinde etkisi ve iki trimeric autotransporter adhesins fonksiyonu test. Gen silme P1 iletim sınırlamaları olsa, kolaylık ve hız onun uygulama gene silme işlemini diğer yöntemleri için cazip bir alternatif olun.
Giriş
İşlev bir çalışmaya ortak bir ilk yaklaşım knock-out mutagenesis gerçekleştirmek ve elde edilen fenotip dikkat etmektir. Bu da geriye doğru genetik olarak adlandırılır. E. coli bakteri moleküler biyoloji beygir son 70 yıl ya da çok, kendi kültür ve onun amenability genetik manipülasyon1kolaylığı nedeniyle olmuştur. E. coliişaret exchange mutagenesis2,3 ve son zamanlarda, λ kırmızı veya Rac ET sistemleri4,5 kullanarak recombineering da dahil olmak üzere, gen silme üretmek için çeşitli yöntemler geliştirilmiştir , 6.
Yaygın olarak kullanılan bir sistemde, bireysel genlerin kodlama dizileri daha sonra kromozom5,7' den eksize bir antibiyotik direnci kaset değiştirilir. Kodlama dizileri, hangi flippase (FLP) tanıma hedef (FRT) siteleri iki tarafında çevrili örneğin bir sefaloridin (Kan) direnç kaset, değiştirilir. FRT siteleri recombinase siteye özgü rekombinasyon Kan kaset silinmesi için önde gelen FRT siteler arasında aracılık eder FLP tarafından tanınır. Bu şekilde, belirli bir genin kodlama dizisi tam bir silme, yalnızca yaklaşık 100 baz çifti (bp) (resim 1) en az yara dizisi bırakarak elde edilebilir.
Sadece bir on yıl kadar önce sözde Keio koleksiyonu geliştirilmiştir. Bu nerede hemen hemen tüm gerekli olmayan genler ayrı ayrı λ kırmızı rekombinasyon7,8tarafından silindi Standart laboratuvar E. coli K12 zorlanma, temel bakteriyel bir kütüphanedir. Klonlar bu koleksiyon içindeki bir excisable Kan direnç kaset ile yerine bir kodlama dizisi var. Keio koleksiyon birçok uygulamaları9için yararlı bir araç olduğu ispatlanmıştır. Böyle bir uygulama diğer E. coli suşları silme mutantlar yapımıdır. Kan kaset üzerinden verilen silme klon genellikle bacteriophages, P110,11,12,13,14gibi transducing tarafından seferber. Böyle bir yük hazırlanan bir fajının hisse senedi daha sonra alıcı faiz, e.coli suşu bulaştırmak için nerede bir düşük ama güvenilir frekansta Kan kaset içeren bölge alıcı Genom tarafından Homolog rekombinasyon dahil edilebilir kullanılabilir (Şekil 2). Transductants Kan içeren ortamda büyümesi için seçilebilir. Antibiyotik direnci kaset kaldırılmasını isterseniz, bunu, FLP recombinase trans transductant zorlanma sağlanabilir. Ampisilin (Amp) direnç işaret taşıyan FLP içeren plazmid kür sonra Kan ve Amp duyarlı klonlar için ekranlı ve vahşi-türü kodlama dizisi ve Kan kaset doğru eksizyon PCR colony tarafından doğrulandı.
Burada, her bir knock-out yukarıda özetlenen strateji dayalı E. coli zorlanma üreten adımları açıklayan ayrıntılı bir protokol sunulmuştur. Örnek olarak, tamA gen silinmesiyle gösterilmiştir. tamA bir parçası olan taşıma ve montaj modülü (TAM), bazı autotransporter proteinler ve pili15,16,17Biyogenez yer bir dış membran β-varil protein kodlar. Bu knock-out zorlanma sonra iki trimeric autotransporter adhesins (TAAs), Yersinia adhesin YadA ve E. coli immünglobulin (Ig) tamA silinmesi Biyogenez üzerinde etkisini incelemek için kullanılan-TAA EibD bağlama 18,19.
Protokol
1. suşları ve Plazmidlerin
-
Bakteri suşları
- E. coli suşları BW251135, JW4179 (BW25113 tamA::kan)7, BL21(DE3)20ve BL21ΔABCF21kullanın. Malzemeler tablo daha fazla bilgi için bkz:.
-
Bacteriophages
- Feyc P1vir genel iletim için kullanın. Kloroform birkaç damla ile sıvı bir hisse senedi olarak faj saklamak (bkz. Adım 2.2). Daha fazla bilgi için bkz: Tablo reçetesi.
-
Plazmid
- Bu protokol için aşağıdaki plazmid kullanın: pCP2022, pIBA2 YadA23ve pEibD1024. Denetim plazmid pASK-IBA2 ve pET22b ( Tablo malzemelerigörmek) kullanın.
-
Büyüme koşulları
- Dinç (180-200 devir/dakika) 37 ° C veya 30 ° C durumunda BL21ΔABCF ve pCP20 içeren suşları sallayarak bir lysogeny suyu (LB) orta25 bakteri yaymak.
- 43 ° C'de kür plazmid gerçekleştirmek
- Sağlam bir orta için LB %1 agar (w/v) ile ek.
- En iyi agar için LB %0,7 agar ve 10 mM CaCl2 ve otoklav orta ile ek. SOC orta Elektroporasyon26sonra kurtarma için kullanın.
- Aşağıdaki konsantrasyonları için antibiyotik kullanmak: 100 μg/mL Amp ve 25 μg/mL kan için
2. bir fajının Lysate hazırlanması
-
Enfeksiyon büyük donör yük
- 10 mM CaCl2 ve isteğe bağlı olarak Kan ile desteklenmiş LB orta 5 ml donör zorlanma JW4197 büyümek (25 µg/mL) için bir optik yoğunluk 600 nm ~ 1.0 (OD600). Bir spektrofotometre kullanarak OD600 değerini ölçmek.
- Varolan bir P1 fajının stok seyreltme dizi LB ortamda yapmak: dilutions 10-7için 10-3 arasında önerilir.
- Mix 200 μL bakteriyel süspansiyon ve verilen fajının seyreltme 15 mL santrifüj tüpü veya eşdeğeri 100 μL. Çok sayıda tüpler fajının dilutions hazırlayın. Tüpler 37 ° C'de 20 dk için sallayarak olmadan kuluçkaya.
- Tüpler için 10 mM CaCl2 ile takıma erimiş üst agar (~ 50 ° C) ~ 3 mL ekleyin, içeriği iyice kısa bir süre tüpler vortexing tarafından mix ve karışımları hatta katmanlar yapmak prewarmed LB plakalar üzerine dökün.
- Plakayı gecede 37 ° C'de kuluçkaya
-
Lysate hazırlık
- Ertesi gün bir tabak yarı konfluent büyüme fajının plaklar ile seçin. Yarı konfluent bir plaka üzerinde yaklaşık yarım yüzey alanı plaka açıktır (Şekil 3).
- Bir aşı döngü veya benzer bir aracı kullanarak böyle bir tabak üst agar katmandan scrape ve üst agar bir santrifüj tüpü yerleştirin. 1-2 mL lb ve kloroform ve girdap şiddetle ~ 1 dk. Ekle kloroform için tüp bir damla duman mahallede ekleyin.
- Tüp agar ve bakteri hücreleri cips için 4000 g x veya daha hızlı, 15 dk santrifüj kapasitesi.
- Süpernatant Pelet üzerinden herhangi bir enkaz üzerinde taşıyan kaçınarak bir taze microcentrifuge tüp taşıyın. Kloroform 2 damla ekleyin ve lysate 4-10 ° C'de depolayın Kloroform duman mahallede ekleyin. Faj-değil var dondurmak lysate gibi bu bulaşıcı parçacık sayısı önemli bir azalma sonuçlanır.
-
Lysate titresi belirleme
- BW25113 37 ° C'de 10 mM CaCl2 ile kültür bir OD600 ~ 1.0 ulaşır kadar takıma LB içinde büyümek.
- Feyc LB (Örneğin, 10-6–10-9) lysate bir seyreltme dizi hazırlamak.
Not: damlalıklı örnek üst lysate dilutions için kloroform aktarılmasını önlemek için dikkatli olun. - Mix 200 μL bakteriyel süspansiyon ve verilen fajının seyreltme 15 mL santrifüj tüpü veya eşdeğeri 100 μL. Çok sayıda tüpler fajının dilutions hazırlayın. Tüpler 37 ° C'de 20 dk için sallayarak olmadan kuluçkaya.
- Tüpler için 10 mM CaCl2 ile takıma erimiş üst agar (~ 50 ° C) ~ 3 mL ekleyin, içeriği iyice kısa bir süre tüpler vortexing tarafından mix ve hatta katmanlar yapmak prewarmed LB plakalar üzerine karışımı dökün.
- Plakayı gecede 37 ° C'de kuluçkaya
- Ertesi gün plaklar (bakteriyel mat açık bölgelerde) her plaka için sayma ve titresi lysate aşağıdaki formülü kullanarak hesaplar:

Örnek: dilutions 10-7, 10-8ve 10-9ile levha üzerinde orada 231, 27 ve 2 plaklar, sırasıyla. Her seyreltme 100 μL enfeksiyonu için kullanılan gibi ve titresi plak şekillendirme olarak ifade edilir çünkü birimleri (pfu) / mL, kaplama faktör 10. Bu sayılar formüle girilir: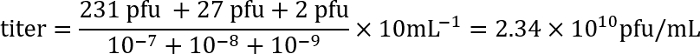
Böylece, titresi yaklaşık 2 x 1010 bulaşıcı fajının parçacıklar/ml'dir.
3. P1 iletim
-
Alıcı hücreler hazırlanıyor
- Alıcı zorlanma BL21ΔABCF 600 optik bir yoğunluk için takıma LB büyümek nm ~ 1.0 (OD600). Bir spektrofotometre OD600 değeri ölçmek için kullanın.
- Enfeksiyon (MOI) değeri 0,5 çokluğu sağlanması için gerekli lysate fajının hacmi hesaplamak. MOI hesaplamak için bakterileri OD600 kültür göre sayısını tahmin ediyoruz. Bir OD600 değerinin 1.0 ~ 109 cfu/mL karşılık geldiği varsayılmaktadır. Gerekli birim üzerinde lysate titresi bilinen dayalı hesaplamak.
Örnek: (-den sonra adım 2.3.6 örnekte hesaplanan titresi bağlı olarak)
- 10 mM için alıcı zorlanma kültürüne CaCl2 ekleyip karıştırın. Kültür iletim için 1 mL al.
-
İletim gerçekleştirme
- Feyc lysate uygun hacmi 1 mL (10 mm CaCl2 dahil) alıcı kültür 3.1.2. adımda hesaplanan ekleyin ve karışımı yavaşça.
Not: örnek lysate karıştırmak için kloroform aktarılmasını önlemek için üst pipette için dikkatli olun. - Statik olarak 37 ° C'de 20 dk için karışımı kuluçkaya
- Enfeksiyon için 100 mM sodyum sitrat, pH 5.5, ekleyerek durdurmak.
- Bakteri (5000 x g için 2 dk) santrifüj kapasitesi ve süpernatant kaldırın ve sonra onları taze LB ile 100 mM sodyum sitrat, pH 5.5 takıma 1 ml resuspend.
- İki kez daha fazla adım 3.2.4 ücretsiz phages ve kalsiyum kaldırılmasını sağlamak için olduğu gibi hücreleri yıkayın.
- 1 mL taze lb 100 mM sodyum sitrat, pH 5.5 ile desteklenmiş bakterilerde resuspend. Bakteri (> 100 rpm) sallayarak ile 1 h için 30 ° C'de kuluçkaya.
- Santrifüjü (5000 x g için 2 dk) tarafından bakteri toplamak ve onları LB ~ 100 μL 100 mM sodyum sitrat, pH 5.5 ile resuspend.
- 25 µg/mL ve 10 mM sodyum sitrat, pH 5.5, Kan ile desteklenmiş bir LB plaka üzerinde bakteri yaymak ve 30 ° C'de bakteri kolonileri (~ 24 h) görününceye büyür.
- Feyc lysate uygun hacmi 1 mL (10 mm CaCl2 dahil) alıcı kültür 3.1.2. adımda hesaplanan ekleyin ve karışımı yavaşça.
-
Transductants seçme
- Koloniler seçimi plaka üzerinde büyüdü sonra onları restreak LB + Kan için tek kolonileri ve tek kolonileri görünür kadar 30 ° C'de büyür.
4. Kan kaset bilincin
-
Rekombinasyon plazmid ile dönüştürme
- Kan dayanıklı BL21ΔABCF gerilme electrocompetent olun.
- Taze LB (5 mL) Kan ile desteklenmiş bir koloni gelen zorlanma büyümek (25 µg/mL) OD600 ~0.5 – 0,7 olana 30 ° C'de.
- Şu andan itibaren 4 ° C'de veya buz üzerinde aşağıdaki adımları uygulayın. Bakteri (5000 x g 10 dk) santrifüj kapasitesi ve süpernatant kaldırın.
- İki kez önceki Santrifüjü adımı tekrar ederek hücre Pelet buz gibi soğuk distile su ile yıkama ve son olarak, hücre Pelet buz gibi % 10 gliserol 100 µL içinde resuspend.
- 1 mm Elektroporasyon cuvettes buz üzerinde sakin.
- Plazmid DNA (pCP20) 1 pg hücre süspansiyon eklemek, süspansiyon karışımı yavaşça ve soğutmalı Elektroporasyon küvet aktarın.
- Electroporator 1.8 ayarla kV ve electroporate hücreleri.
- 1 mL SOC orta ekleyerek ve onları 30 ° C'de 1 h için büyüyen dönüştürülmüş hücreleri kurtarmak
- 100 µL LB hücrelerin plaka + Amp tabaklar ve 30 ° C'de hücreler gecede büyümek.
- Kan dayanıklı BL21ΔABCF gerilme electrocompetent olun.
-
Rekombinasyon indüksiyon
- Çekme tek kolonileri LB + Amp plaka ve taze LB tüm antibiyotiklere atlama aşılamak.
- Hücreleri, Sigara keyfi sıcaklığında (43 ° C) gece FLP recombinase ifade ikna etmek için büyür.
-
Rekombinasyonlar seçme
- Seri dilutions yapmak ve non-selektif plakaları 105-106 seyreltme 50 µL plaka ve gecede 30 ° C'de büyür
5. gen silme doğrulanması
-
Plazmid kaybı ve başarılı rekombinasyon doğrulama
- Çizgi tek kolonileri adım 4.3.1 LB + Kan, LB + Amp hazırlanan tabak ve LB plakaları olmadan antibiyotik, bu sırada. Çizgiler içinde yardım etmek için bir koloni kılavuzunu kullanın (bkz. ek dosya 1). Koloniler (~ 24 h) görününceye tabak 30 ° C'de büyümek.
- Non-selektif plaka üzerinde yetiştirilen, ancak daha fazla doğrulama için seçmeli medya büyümeye başarısız oldu 10-20 klonlar almak.
-
Koloni PCR tarafından ek doğrulama
- Bir koloni PCR sıra kodlama tamA kanat astar ile gerçekleştirmek ( Tablo malzemelerigörmek).
- Bir ana PCR karışımı hazırlayın. Gerekli mix ( Tablo 1' deki örneğe bakın) test edilecek kolonileri sayısını bağlıdır. Buzda reaktifleri mix, polimeraz son ekleyin.
- PCR ana karışımı iyice karıştırın, sonra 20 µL PCR şerit tüpler içine dağıtmak. Her koloni steril pipet ucu kullanarak taranması için küçük bir miktar almak ve bir tüp için ekleyin. Orijinal alıcı türe ürününü karşılaştırma listesine eklemeyi unutmayın. İsteğe bağlı, ayrıca donör zorlanma içerebilir.
- PCR reaksiyon çalıştırmak (bkz. Tablo 2 program için kullanılır).
- % 1'özel jel hazırlayın.
- 50 mL jel için özel 0.5 g ölçmek ve TAE arabellek (40 mM Tris, 20 mM asetik asit ve 1 mM EDTA, pH 8.0) 50 mL ekleyin. Tüm özel eriyene kadar karışımı fırın mikrodalga bir de ısı.
- Bir kez özel çözünmüş, çözüm için yaklaşık 50 ° C serin ve boya boyama DNA'ın 5 μL eklemek. Çözüm karıştırın ve bir döküm odası jel tepsisine içine dökün. Böylece tüm örnekleri yanı sıra moleküler boyutu işaretçileri için yeterli wells iyi tarak bir veya daha fazla satırı ekleyin. Jel 30 dk için ayarlamak izin verir.
- 6 x DNA yükleme boya 4 µL PCR çalışma tamamlandıktan sonra her örnek için ekleyin. Jel Elektroforez odasında yerleştirin ve kuyular kaplıdır kadar TAE arabellek ekleyin.
- 10-15 µL jel bir kuyuda her PCR reaksiyon uygulayın. Ayrıca moleküler boyutu işaretleyicinin 5 µL ekleyin. Daha sonra 30 dk için jel 75 V çalıştırın.
- Çalışma tamamlandıktan sonra bir mavi ışık Imager kullanarak jel görüntü.
6. diğer teknikleri
-
Protein ifade
Not: Test proteinler (YadA ve EibD) ifadesi oldu23,24başka bir yerde ayrıntılı olarak açıklanmıştır. Bu ana adımları kısa bir özetidir.- BL21ΔABCF ΔtamA wITH gerekli plazmid (pIBA2-YadA ve pEibD10 ve karşılık gelen denetim plazmid) dönüştürmek ve transformants LB + Amp için seçin.
- Protein ifade için bir gecede kültür dönüştürülmüş bakteri 1 mL ile 100 mL LB orta + Amp aşılamak ve bunlar 30 ° C'de orta log fazı, ne zaman kadar büyümek, her iki anhydrotetracycline (100 ng/mL) adlı protein üretim teşvik veya İzopropil thiogalactoside (0.5 mM) de.
- İndüksiyon 30 ° C'de 2 h kültürlerin bulanıklık ölçmek ve 50 mL OD600 karşılık gelen hücre sayısı toplamak sonra = 1.0 kültürler 4.000 x gde 15 dakika centrifuging. Pelet yıkayın 1 x 10 mM HEPES ile pH 7,4 ve sonra da-20 ° C veya işlem adım 6.2 olduğu gibi daha fazla bu depolamak.
-
Dış membran çıkarma
Not: Leo vd tarafından ayrıntılı olarak açıklandığı gibi dış membran ayıklama gerçekleştirilir 27. ana adımları aşağıda özetlenmiştir.- Resuspend hücre pelet 1 ml 10 mm HEPES, pH 7.4, takıma 10 mM MgCl2 ve MnCl2, lizozim (0.1 mg/mL) ve bir tutam DNaz ile ben.
- (Bir boncuk çırpıcı kullanarakÖrneğin,) hücreleri parçalayıcı.
- Kısa bir süre (15,600 x gde 2 dk) hücreleri hücre artıkları kaldırmak ve süpernatant temiz bir tüp taşımak için santrifüj kapasitesi.
- Hücreler için 16.000 x g, 30 dk sonra santrifüj kapasitesi, %1 N-lauroyl sarcosine 10 mm HEPES, pH 7.4 400 μL Pelet resuspend.
- Hangi sonra oda sıcaklığında 30 dakika ajitasyon hücrelerle kuluçkaya, onları yukarıdaki gibi 30 dk santrifüj kapasitesi.
- Yıkama saydam 1 x 10 mm HEPES, pH 7.4, 200 μL ile cips ve daha sonra 10 mM HEPES 30 μL içinde resuspend ve 4 SDS-sayfa örnek arabellek x 10 μL ekleyin.
-
Etkinlik deneyleri
Not: SDS-sayfa ve etkinlik deneyleri tarif28YadA ve EibD için gerçekleştirin. Ana adımları aşağıda özetlenmiştir. -
SDS-SAYFA
- Örnekleri 50 ° c 5 min için jel polyacrylamide proteinleri denaturing önlemek için, yüklemeden önce ısı.
- SDS-sayfa boşanmadan sonra polivinilidin difluoride (PVDF) membran proteinleri aktarın.
- Aktarımdan sonra membran PBS de %2 yağsız süt tozu ile engelleyin.
-
YadA-kollajen kadar western blot
- Belgili tanımlık kütük parçası sonra ben seyreltilmiş sığır kollajen türü engelleme arabellekte bir konsantrasyon 10 μg/mL için eklemek ve membran 1 h için kuluçkaya.
- Membran 2 yıkama x PBS + %0,05 Tween20 ile (PBS-T).
- Membran için birincil antikor (monoklonal Anti-kollajen COL-1), seyreltilmiş 1:2,000 engelleme arabellekte ekleyin.
- 1s için membran kuluçka sonra adım 6.3.2.2 belirtildiği gibi 2 x yıkayın ve ikincil antikor [keçi Anti-fare IgG-horseradish peroksidaz (HRP) eşlenik], seyreltilmiş 1:10,000 engelleme arabellekte ekleyin.
- Membran 1 h için kuluçkaya sonra yıka 2 x ile PBS-T. Membran üretici yönergelerine göre gelişmiş bir chemiluminescent substrat ekleyin ve grubun bir CCD kamera ile tespit.
-
EibD-IgG bağlama tahlil
- Engelleme sonra ikincil antikor (keçi Anti-tavşan HRP), seyreltilmiş engelleme arabelleği 1:2,000 ekleyin.
- Membran 1 h için kuluçkaya sonra yıka 2 x PBS ile. Chemiluminescent adım 6.3.2.5 belirtildiği gibi saptaması.
Sonuçlar
Bir tamA Knock-out BL21ΔABCF, nesil:
Yukarıda özetlenen strateji BL21(DE3), dış membran protein üretimi için en iyi duruma getirilmiş ve BL21ΔABCF21denilen protein üretimi için kullanılan bir Standart laboratuvar yük türetilmiş bir tür üretmek için daha önce kullanılmış. Bu zorlanma dört genler bol dış membran proteinlerinin için kodlama yoksun ve sonuç olarak, daha hetero...
Tartışmalar
P1 iletim gen silme E. coliüretmek için hızlı, sağlam ve güvenilir bir yöntemdir. Bu burada bir BL21 elde edilen alıcıya Keio donör zorlanma tamA silme mutant transducing tarafından gösterilmiştir. İletim işlemi büyük aşamasında bulunmaktadır lysate transducing üretim, iletim, Kan direnç kaset eksizyon ve knock-out PCR tarafından doğrulanması. Toplamda, süreci yaklaşık 1 hafta alır ve doğrulama için son PCR dışında kullanılmak üzere hiçbir moleküler biyoloji yöntem...
Açıklamalar
Yazarlar ifşa gerek yok.
Teşekkürler
Keio koleksiyonu suşları Ulusal BioResource (Zen, Japonya) projeden elde: E. coli. Dirk Linke (Biosciences bölümü, Oslo Üniversitesi) onun sürekli destek için teşekkür ederiz. Bu eser Norveç genç araştırmacı hibe 249793 (Jack C. Leo) araştırma kurumu tarafından finanse edildi.
Malzemeler
| Name | Company | Catalog Number | Comments |
| Strains | |||
| E. coli BW25113 | NIG | ME6092 | Wild-type strain of Keio collection |
| E. coli BL21(DE3) | Merck | 69450-3 | Expression strain |
| E. coli BL21DABCF | Addgene | 102270 | Derived from BL21(DE3) |
| E. coli JW4179 | NIG | JW4179-KC | tamA deletion mutant |
| P1 vir | NIG | HR16 | Generally transducing bacteriophage |
| Plasmids | |||
| pCP20 | CGSC | 14177 | conditionally replicating plasmid with FLP |
| pASK-IBA2 | IBA GmbH | 2-1301-000 | expression vector |
| pEibD10 | N/A | N/A | for production of EibD; plasmid available on request |
| pET22b+ | Merck | 69744-3 | expression vector |
| pIBA2-YadA | N/A | N/A | for production of YadA; plasmid available on request |
| Chemicals | |||
| Acetic acid | ThermoFisher | 33209 | |
| Agar | BD Bacto | 214010 | |
| Agarose | Lonza | 50004 | |
| Ampicillin | Applichem | A0839 | |
| Anhydrotetracycline | Abcam | ab145350 | |
| anti-collagen type I antibody COL-1 | Sigma | C2456 | |
| Bovine collagen type I | Sigma | C9791 | |
| Calcium chloride | Merck | 102382 | |
| Chloroform | Merck | 102445 | |
| Di-sodium hydrogen phosphate | VWR | 28029 | |
| DNA dye | Thermo | S33102 | |
| DNA molecular size marker | New England BioLabs | N3232S | |
| DNase I | Sigma | DN25 | |
| dNTP mix | New England Biolabs | N0447 | |
| ECL HRP substrate | Advansta | K-12045 | |
| EDTA | Applichem | A2937 | |
| Glycerol | VWR | 24388 | |
| goat anti-mouse IgG-HRP | Santa Cruz | sc-2005 | |
| goat anti-rabbit IgG-HRP | Agrisera | AS10668 | |
| HEPES | VWR | 30487 | |
| Isopropyl thiogalactoside | VWR | 43714 | |
| Kanamycin | Applichem | A1493 | |
| Lysozyme | Applichem | A4972 | |
| Magnesium chloride | VWR | 25108 | |
| Manganese chloride | Sigma | 221279 | |
| N-lauroyl sarcosine | Sigma | L9150 | |
| Skim milk powder | Sigma | 70166 | |
| Sodium chloride | VWR | 27808 | |
| tamA forward primer | Invitrogen | N/A | Sequence 5'-GAAAAAAGG ATATTCAGGAGAAAATGTG-3' |
| tamA reverse primer | Invitrogen | N/A | Sequence 5'-TCATAATT CTGGCCCCAGACC-3' |
| Taq DNA polymerase | New England Biolabs | M0267 | |
| Tri-sodium citrate | Merck | 106448 | |
| Tryptone | VWR | 84610 | |
| Tween20 | Sigma | P1379 | |
| Yeast extract | Merck | 103753 | |
| Equipment | |||
| Agarose gel electrophoresis chamber | Hoefer | SUB13 | |
| Bead beater | Thermo | FP120A-115 | |
| CCD camera | Kodak | 4000R | |
| Electroporation cuvettes | Bio-Rad | 165-2089 | |
| Electroporation unit | Bio-Rad | 1652100 | |
| Gel imager | Nippon Genetics | GP-03LED | |
| Incubating shaker | Infors HT | Minitron | |
| Incubator | VWR | 390-0482 | |
| Microcentrifuge | Eppendorf | 5415D | |
| Microwave oven | Samsung | CM1099A | |
| PCR machine | Biometra | Tpersonal | |
| PCR strips | Axygen | PCR-0208-CP-C | |
| pH meter | Hanna Instruments | HI2211-01 | |
| PVDF membrane | ThermoFisher | 88518 | |
| SDS-PAG electrophoresis chamber | ThermoFisher | A25977 | |
| Tabletop centrifuge | Beckman Coulter | B06322 | |
| Vortex mixer | Scientific Industries | SI-0236 | |
| Water bath | GFL | D3006 | |
| Wet transfer unit | Hoefer | TE22 |
Referanslar
- Blount, Z. D. The unexhausted potential of E. coli. eLife. 4, e05826 (2015).
- Hamilton, C. M., Aldea, M., Washburn, B. K., Babitzke, P., Kushner, S. R. New method for generating deletions and gene replacements in Escherichia coli. Journal of Bacteriology. 171 (9), 4617-4622 (1989).
- Link, A. J., Phillips, D., Church, G. M. Methods for generating precise deletions and insertions in the genome of wild-type Escherichia coli: application to open reading frame characterization. Journal of Bacteriology. 179 (20), 6228-6237 (1997).
- Sawitzke, J. A., Thomason, L. C., Costantino, N., Bubunenko, M., Datta, S., Court, D. L. Recombineering: in vivo genetic engineering in E. coli, S. enterica, and beyond. Methods in Enzymology. 421, 171-199 (2007).
- Datsenko, K. A., Wanner, B. L. One-step inactivation of chromosomal genes in Escherichia coli K-12 using PCR products. Proceedings of the National Academy of Sciences of the United States of America. 97 (12), 6640-6645 (2000).
- Muyrers, J. P., Zhang, Y., Stewart, A. F. Techniques: Recombinogenic engineering--new options for cloning and manipulating DNA. Trends in Biochemical Sciences. 26 (5), 325-331 (2001).
- Baba, T., et al. Construction of Escherichia coli K-12 in-frame, single-gene knockout mutants: the Keio collection. Molecular Systems Biology. 2, (2006).
- Yamamoto, N., et al. Update on the Keio collection of Escherichia coli single-gene deletion mutants. Molecular Systems Biology. 5, 335 (2009).
- Baba, T., Huan, H. -. C., Datsenko, K., Wanner, B. L., Mori, H. The applications of systematic in-frame, single-gene knockout mutant collection of Escherichia coli K-12. Methods in Molecular Biology. 416, 183-194 (2008).
- Chattopadhyay, M. K., Tabor, C. W., Tabor, H. Polyamines are not required for aerobic growth of Escherichia coli: preparation of a strain with deletions in all of the genes for polyamine biosynthesis. Journal of Bacteriology. 191 (17), 5549-5552 (2009).
- Xie, X., Wong, W. W., Tang, Y. Improving simvastatin bioconversion in Escherichia coli by deletion of bioH. Metabolic Engineering. 9 (4), 379-386 (2007).
- Maeda, T., Sanchez-Torres, V., Wood, T. K. Escherichia coli hydrogenase 3 is a reversible enzyme possessing hydrogen uptake and synthesis activities. Applied Microbiology and Biotechnology. 76 (5), 1035-1042 (2007).
- Meehan, B. M., Landeta, C., Boyd, D., Beckwith, J. The disulfide bond formation pathway is essential for anaerobic growth of Escherichia coli. Journal of Bacteriology. 199 (16), (2017).
- Niba, E. T. E., Naka, Y., Nagase, M., Mori, H., Kitakawa, M. A genome-wide approach to identify the genes involved in biofilm formation in E. coli. DNA Research. 14 (6), 237-246 (2008).
- Heinz, E., et al. Conserved features in the structure, mechanism, and biogenesis of the inverse autotransporter protein family. Genome Biology and Evolution. 8 (6), 1690-1705 (2016).
- Selkrig, J., et al. Discovery of an archetypal protein transport system in bacterial outer membranes. Nature Structural & Molecular Biology. 19 (5), 506-510 (2012).
- Stubenrauch, C., et al. Effective assembly of fimbriae in Escherichia coli depends on the translocation assembly module nanomachine. Nature Microbiology. 1 (7), 16064 (2016).
- Leo, J. C., Goldman, A. The immunoglobulin-binding Eib proteins from Escherichia coli are receptors for IgG Fc. Molecular Immunology. 46 (8-9), 1860-1866 (2009).
- Mühlenkamp, M., Oberhettinger, P., Leo, J. C., Linke, D., Schütz, M. S. Yersinia adhesin A (YadA) - Beauty & beast. International Journal of Medical Microbiology. 305 (2), 252-258 (2015).
- Studier, F. W., Moffatt, B. A. Use of bacteriophage T7 RNA polymerase to direct selective high-level expression of cloned genes. Journal of Molecular Biology. 189 (1), 113-130 (1986).
- Meuskens, I., Michalik, M., Chauhan, N., Linke, D., Leo, J. C. A new strain collection for improved expression of outer membrane proteins. Frontiers in Cellular and Infection Microbiology. 7, 464 (2017).
- Cherepanov, P. P., Wackernagel, W. Gene disruption in Escherichia coli: TcR and KmR cassettes with the option of Flp-catalyzed excision of the antibiotic-resistance determinant. Gene. 158 (1), 9-14 (1995).
- Grosskinsky, U., et al. A conserved glycine residue of trimeric autotransporter domains plays a key role in Yersinia adhesin A autotransport. Journal of Bacteriology. 189 (24), 9011-9019 (2007).
- Leo, J. C., et al. The structure of E. coli IgG-binding protein D suggests a general model for bending and binding in trimeric autotransporter adhesins. Structure. 19 (7), 1021-1030 (1993).
- Bertani, G. Studies on lysogenesis. I. The mode of phage liberation by lysogenic Escherichia coli. Journal of Bacteriology. 62 (3), 293-300 (1951).
- Hanahan, D. Studies on transformation of Escherichia coli with plasmids. Journal of Molecular Biology. 166 (4), 557-580 (1983).
- Leo, J. C., Oberhettinger, P., Linke, D. Assessing the outer membrane insertion and folding of multimeric transmembrane β-barrel proteins. Methods in Molecular Biology. , 157-167 (2015).
- Mikula, K. M., Leo, J. C., Łyskowski, A., Kedracka-Krok, S., Pirog, A., Goldman, A. The translocation domain in trimeric autotransporter adhesins is necessary and sufficient for trimerization and autotransportation. Journal of Bacteriology. 194 (4), 827-838 (2012).
- Thomason, L. C., Costantino, N., Court, D. L., Ausubel, F. M. E. coli genome manipulation by P1 transduction. Current Protocols in Molecular Biology. , (2007).
- Franklin, N. C. Mutation in gal U gene of E. coli blocks phage P1 infection. Virology. 38 (1), 189-191 (1969).
- Jeong, H., et al. Genome sequences of Escherichia coli B strains REL606 and BL21(DE3). Journal of Molecular Biology. 394 (4), 644-652 (2009).
- Greene, E. C. DNA Sequence Alignment during Homologous Recombination. Journal of Biological Chemistry. 291 (22), 11572-11580 (2016).
- Watt, V. M., Ingles, C. J., Urdea, M. S., Rutter, W. J. Homology requirements for recombination in Escherichia coli. Proceedings of the National Academy of Sciences of the United States of America. 82 (14), 4768-4772 (1985).
- Ho, T. D., Waldor, M. K. Enterohemorrhagic Escherichia coli O157:H7 gal mutants are sensitive to bacteriophage P1 and defective in intestinal colonization. Infection and Immunity. 75 (4), 1661-1666 (2006).
- Ikeda, H., Tomizawa, J. I. Transducing fragments in generalized transduction by phage P1. I. Molecular origin of the fragments. Journal of Molecular Biology. 14 (1), 85-109 (1965).
- Murooka, Y., Harada, T. Expansion of the host range of coliphage P1 and gene transfer from enteric bacteria to other Gam-negative bacteria. Applied and Environmental Microbiology. 38 (4), 754-757 (1979).
- Goldberg, R. B., Bender, R. A., Streicher, S. L. Direct selection for P1-sensitive mutants of enteric bacteria. Journal of Bacteriology. 118 (3), 810-814 (1974).
- O'Connor, K. A., Zusman, D. R. Coliphage P1-mediated transduction of cloned DNA from Escherichia coli to Myxococcus xanthus: use for complementation and recombinational analyses. Journal of Bacteriology. 155 (1), 317-329 (1983).
Yeniden Basımlar ve İzinler
Bu JoVE makalesinin metnini veya resimlerini yeniden kullanma izni talebi
Izin talebiThis article has been published
Video Coming Soon
JoVE Hakkında
Telif Hakkı © 2020 MyJove Corporation. Tüm hakları saklıdır